Method Article
Quantifizierung und Analyse der Bildung von HO-Endonuklease Stimulated chromosomalen Translokationen von Single-Strand Annealing in Saccharomyces cerevisiae
In diesem Artikel
Zusammenfassung
Die HO-stimulierte Translokation Assay überwacht Einzelstrang-Glühen nach der Gründung von DNA-Doppelstrangbrüche an mehreren Loci in diploiden Saccharomyces cerevisiae. Dieser Mechanismus kann Genom Umlagerungen in somatischen Zellen höherer Eukaryonten nach Exposition mit hohen Dosen ionisierender Strahlung Modell.
Zusammenfassung
Genetische Variation wird häufig durch genomische Rearrangements, die durch Interaktion entstehen zwischen verteilten repetitiven Elementen in jeder eukaryotischen Genoms vermittelt. Dieser Prozess ist ein wichtiger Mechanismus zur Erzeugung von Vielfalt zwischen und innerhalb von Organismen 1-3. Das menschliche Genom besteht aus ca. 40% sich wiederholenden Sequenz von Retrotransposon Herkunft, darunter eine Vielzahl von Linien und Sines 4. Exchange-Ereignisse zwischen diesen repetitiven Elementen können Genom-Umlagerungen, wie Translokationen, dass Gen-Dosierung und Ausdruck, der im Bereich der Autoimmun-und Herz-Kreislauf-Krankheiten 5, sowie bei Menschen Krebs 6-9 führen können stören führen kann.
Der Austausch zwischen repetitiven Elementen erfolgt in einer Vielzahl von Möglichkeiten. Der Austausch zwischen Sequenzen, die perfekte (oder fast perfekten) Homologie tritt durch einen Prozess namens homologe Rekombination (HR). Im Gegensatz dazu nicht-homologen Ende (NHEJ) verbraucht wenig-oder-Nein Sequenzhomologie zum Austausch 10,11. Der primäre Zweck des HR, in mitotischen Zellen, ist die Doppel-(DSBs) erzeugt endogen durch aberrante DNA-Replikation und oxidative Schädigungen oder durch Exposition gegenüber ionisierender Strahlung (IR), und andere exogene DNA schädigende Mittel zu reparieren.
In dem Test hier beschrieben, sind DSBs gleichzeitig erstellt angrenzenden Rekombination Substrate an zwei unterschiedlichen Loci in diploiden Zellen durch einen Galaktose-induzierbaren HO-Endonuklease (Abbildung 1). Die Reparatur der gebrochenen Chromosomen erzeugt chromosomalen Translokationen von Einzelstrang Annealing (SSA), sind ein Prozess, bei dem homologe Sequenzen angrenzend an die Chromosomen-Enden kovalent verbundenen Anschluss an Glühen. Eines der Substrate, his3-Δ3 ", enthält eine 3 'abgeschnitten HIS3-Allel und befindet sich auf einer Kopie des Chromosoms XV in der nativen HIS3-Locus befindet. Das zweite Substrat, his3-Δ5 ", ist an der LEU2-Locus auf eine Kopie des Chromosoms III befindet, und enthält eine 5 'abgeschnitten HIS3-Allel. Beide Substrate werden von einem HO-Endonuklease-Erkennungsstelle, die für Schnitt durch HO-Endonuklease gezielt eingesetzt werden kann flankiert. HO-Endonuklease-Erkennungsstellen ursprünglich aus dem MAT-Locus auf beiden Kopien des Chromosoms III, wurden in allen Stämmen wurde gelöscht. Dies verhindert, dass Wechselwirkungen zwischen der Rekombination Substraten und anderen gebrochenen Chromosomen-Enden von einer Einmischung in den Test. Die KAN-MX-markierte Galaktose-induzierbaren HO-Endonuklease Expressionskassette ist an der TRP1-Locus auf Chromosom IV eingefügt. Die Substrate Aktien 311 bp oder 60 bp des HIS3 codierende Sequenz, die von der HR Maschinen für die Reparatur von SSA eingesetzt werden kann. Zellen, die diese Substrate zu verwenden, um gebrochen Chromosomen von HR Reparatur Form einer intakten HIS3-Allel und ein TXV: III chromosomalen Translokation, die für die Fähigkeit, auf Medium ohne Histidin (Abbildung 2A) wachsen kann ausgewählt werden. Translokation Frequenz von HR wird durch Division der Anzahl der Histidin prototrophen Kolonien, die auf selektiven Medium entstehen durch die Gesamtzahl der lebenden Zellen, die nach dem Ausplattieren geeigneter Verdünnungen auf nicht-selektiven Medium (Abbildung 2B) auftreten berechnet. Eine Vielzahl von DNA-Reparatur-Mutanten wurden verwendet, um die genetische Kontrolle der Translokation Bildung Studie von SSA mit diesem System 12-14.
Protokoll
1. HO-stimulierte Translokation Frequenzen
- Impfen 10-20 unabhängige 1 ml Kulturen YPGly / Lac Medium (1% Hefeextrakt, 2% Pepton, 3% Glycerin und 3% Lactat) mit einzelnen Kolonien der gewünschten Genotyp. Inkubieren Sie die Kulturen über Nacht oder für eine ausreichende Zeit, um eine Zelldichte von ca. 5x10 7-1x10 8 Zellen / ml zu erreichen, bei 30 ° C auf einem Rotator, oder unter leichtem Schütteln.
- Add Galaktose, die Kulturen zu einer Endkonzentration von 2% auf HO-Endonuklease-directed DSBs am his3-Δ3 '(Chromosom III) und his3-Δ5' (Chromosom XV) Translokation Substrate zu induzieren.
- Inkubieren für 4 h bei 30 ° C auf einem Rotator, oder unter leichtem Schütteln.
- Nach 4 h, Platte geeigneter Verdünnungen der Kulturen auf YPD (1% Hefeextrakt, 2% Pepton, 2% Dextrose) zu ergeben rund 100 bis 200 Kolonien pro Platte, und eine ausreichende Anzahl von Zellen auf Medium ohne Histidin zu einem beobachtbaren Rendite Anzahl der Seine + rekombinanten Kolonien. Die Platten bei 30 ° C für zwei vor drei Tagen.
HINWEIS: Schritt 1.4 beschreibt die Bestimmung der Translokation Frequenzen unter selektiven Bedingungen durch Ausplattieren der Kulturen auf Medium ohne Histidin. Dieser Test kann auch unter nicht-selektiven Bedingungen durch Ausplattieren Kulturen auf YPD durchgeführt werden, und dann Replika-Plattierung der Kolonien, die auf-His-Platten entstehen zwei vor drei Tage später. Diese Methoden führen zu ähnlichen Frequenzen der Translokation (Abbildung 2C).
- Bestimmen Sie die Translokation Frequenz, indem die Anzahl von Histidin prototrophen Kolonien durch die Gesamtzahl der lebenden Zellen plattiert (bestimmt durch Ausplattieren Verdünnungen auf YPD). Bestimmen Sie den Median Translokation Frequenz und 95% Konfidenzintervall 15.
2. Plating Effizienz
- Nehmen Sie ein Aliquot von Zellen aus jeder Nacht-Kultur, und bestimmen Sie Zellzahl durch Hämazytometer count (Baxter Healthcare Corporation Katalogcode #:. B3178-1).
- Platte ca. 100 bis 200 Zellen mit Hilfe einer geeigneten Verdünnung auf YPD. Inkubieren zwei vor drei Tagen bei 30 ° C (zB für eine Kultur mit einer Zelldichte von 1x10 8 Zellen / ml, sollte eine Platte 100-200 ul einer 10-5 Verdünnung pro Platte).
- Fügen Sie 20% Galaktose, die Kulturen zu einer Endkonzentration von 2%.
- Nach 4 h, entfernen Sie ein Aliquot von Zellen aus jeder Kultur, zu bestimmen Zellzahl durch Hämazytometer zählen, und die Platte ca. 100 bis 200 Zellen mit Hilfe einer geeigneten Verdünnung auf YPD. Inkubieren zwei vor drei Tagen bei 30 ° C.
- Bestimmen Sie die Platierungseffizienz, indem die Anzahl der Kolonien, die auf YPD durch die Anzahl der Zellen ausplattiert und Multiplikation dieses Quotienten mit 100. Bestimmen Sie die prozentuale Zentralwert mit einem 95%-Konfidenzintervall.
3. Genomic Southern-Blot-Analyse
- Wählen Sie ein einzelnes Sein + rekombinanten Kolonie von jedem unabhängigen Studie und bereitet genomische DNA 16.
- Digest ca. 4 ug DNA mit BamHI Restriktionsendonuklease.
- Separate BamHI verdauten Fragmente auf einem 0,7% Agarosegel und Transfer zu einem positiv geladenen Nylonmembran (Hybond N +, GE Healthcare Product Code:. RPN303B) 17.
- Hybridisieren mit einer 32 P-markierten Sonde durch Random-Priming (Amersham Biosciences Product Code.: RPN1604) erhalten mit einem 1,8 kb BamHI / BamHI genomische Klon, der das HIS3-Gen.
- Visualisieren DNA-Fragmente durch Autoradiographie oder Phosphorimaging.
4. Chromosome-Blot-Analyse unter Verwendung Chromosomen getrennt in eine Kontur-geklemmt homogenes elektrisches Feld (CHEF):
- Bereiten Chromosomen aus ausgewählten His +-Rekombinanten in Agarose-Plugs 18:
- Wachsen eine flüssige Kultur des His + Kandidat TXV:: III-Chromosom-haltigen rekombinanten in 5 ml YPD auf ca. 1-2 x 10 8 Zellen / ml.
- Spin nach unten und waschen Sie die Zellen 2 mal mit 50 mM EDTA.
- Die Zellen in ca. 200 ul 50 mM EDTA, und warm bis 50 ° C.
- Fügen Sie dem gleichen Volumen von geschmolzenem 2% (w / v) Agarose mit niedrigem Schmelzpunkt auf 50 ° C, gründlich mischen.
- Dispense etwa 80 ul Aliquots in Plug Formen, und es ihnen ermöglichen, für 30 min bei 4 ° C abkühlen
- Extrude Stecker aus der Form zu einer 12-well Schale. Bis zu fünf Stecker können in jede Vertiefung gegeben werden.
- 3 ml frisch zubereitete Spheroplasting Lösung (14 mM 2-β-Mercaptoethanol, 20 mM EDTA, 0,5 mg / ml Zymolyase 20T, 10 mM Tris-HCl, pH 7,5, 1 M Sorbitol) in jede Vertiefung. Bei 37 ° C für 4 Stunden unter leichtem Schütteln.
- Entfernen Spheroplasting Lösung und ersetzen mit 3 ml LDS-Lösung (10 mM Tris-HCl, pH 8,0, 100 mM EDTA, 1% (w / v) Lithium-Dodecylsulfat, einstellbart pH bis 8,0). Bei 37 ° C für 15 min unter leichtem Schütteln.
- Entfernen LDS-Lösung und durch ein anderes ersetzen 3 ml Aliquot LDS. Bei 37 ° C über Nacht unter leichtem Schütteln.
- Entfernen Sie LDS und ersetzen mit 3 ml 0,2 X NDS (0,6 g Tris Base, 93 g Dinatrium-EDTA, 5 g N-Lauroylsarcosin, pH-Wert auf 8,0, brachte auf 500 ml mit dH 2 O). Inkubieren bei Raumtemperatur für 30 min unter leichtem Schütteln. Entfernen Sie NDS, und wiederholen Sie 2 mal.
- Remove NDS und ersetzen mit 3 ml TE. Wash unter leichtem Schütteln für 30 min bei Raumtemperatur. Wiederholen Sie die 4-fache.
- Shop-Stecker auf 4 ° C in 2 ml TE. Plugs können für immer bis zu einem Jahr.
- Separate Chromosomen auf einem 1% Agarosegel unter Verwendung eines Bio-Rad CHEF-DRII Apparatur bei 14 ° C (Catalog #: 170-3612).
Parameter: 1. Block: 70er Zeit umschalten, 15h am 6V/cm.
2. Block: 120s Zeit umschalten, 11h am 6V/cm
- Visualisieren Chromosomen durch Färbung mit 1μg/ml Ethidiumbromid für 30 min, Bestrahlung mit 60 mJ der UV in einem UV Stratalinker (Stratagene) und de-Färbung für 30 min in dH 2 O. Bestrahlung mit Ethidiumbromid gefärbten Chromosomen nicks der DNA, um eine effiziente Übertragung auf die Membran zu erlauben.
- Transfer-Chromosomen auf eine positiv geladene Membran (Hybond N +, GE Healthcare Product Code:. RPN303B) durch Kapillarwirkung in denaturierenden Bedingungen (0,4 N NaOH, 1,5 M NaCl).
- Hybridisieren mit einer 32 P-markierten Sonde durch Random-Priming (Amersham Biosciences Product Code.: RPN1604) erhalten mit einem 1,8 kb BamHI / BamHI genomische Klon, der das HIS3-Gen 17.
- Visualisieren Chromosomen durch Autoradiographie oder Phosphorimaging.
5. Repräsentative Ergebnisse:
Eine grafische Darstellung der Translokation Test ist bei der chromosomalen Ebene (Abbildung 1) dargestellt. Eine schematische Darstellung des experimentellen Verfahren wird auch angezeigt (Abbildung 2A). Sowohl Pre-und Post-Induktion plating Wirkungsgrade werden durch Teilung der gesamten Anzahl der lebensfähigen, Kolonie-bildenden Zellen durch die Gesamtzahl der Zellkörper in der Kultur durch Hämazytometer count (Abbildung 4B) bestimmt wird. Pre-und Post-Induktion plating Effizienz unterschieden sich nicht signifikant für Wildtyp-Zellen (p-Wert = 0,1400) (Tabelle 1).
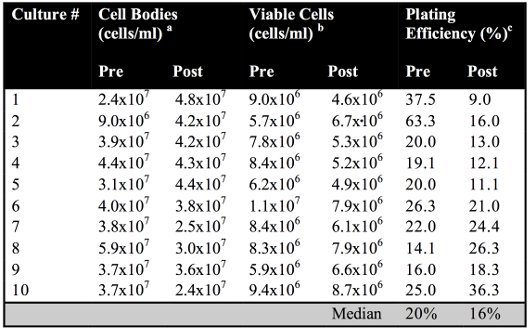
Tabelle 1. Pre-und Post-Induktion plating Effizienz in Wildtyp-Zellen.
a Die Zahl der Zellkörper pro Milliliter sind Hämazytometer count bestimmt.
b Die Anzahl der lebensfähigen Zellen pro Milliliter sind durch Ausplattieren geeigneter Verdünnungen auf nicht-selektiven Medium ~ 100-200 Kolonien produzieren bestimmt.
c Die Pre-und Post-Induktion plating Effizienz wird durch Division der Gesamtzahl der ermittelten lebensfähigen koloniebildenden, Zellen durch die Gesamtzahl der Zellkörper in die Kultur, die durch Hämazytometer count bestimmt.
Dies deutet darauf hin, dass das Vorhandensein oder Nichtvorhandensein von entweder Translokation Chromosom hat keinen Einfluss auf die Fähigkeit, DSB Bildung überleben. Die Häufigkeit von chromosomalen Translokationen können, indem die Anzahl von Histidin prototrophen Kolonien durch die Gesamtzahl der lebenden Zellen durch Ausplattieren auf YPD (Abbildung 2B) bestimmt berechnet werden. Dieser Test kann unter Verwendung Stämme von unterschiedlichen Genotyp zu ermitteln, wie Verlust der Funktion von Proteinen beeinflusst SSA (dh rad51Δ - / -). Rekombination Frequenzen in verschiedenen Stämmen bestimmt können dann grafisch dargestellt, um Unterschiede in der Fähigkeit dieser Stämme, die HO-Endonuklease induziert DSBs von SSA (Abbildung 2C) reparieren zu vergleichen. Translokation Frequenzen mit dem Wildtyp-diploide Stamm unter selektiven (2.2x10 -2) und nicht-selektive (2.17x10 -2) Bedingungen gewonnen wurden statistisch nicht voneinander unterscheiden (p-Wert = 0,9131), während der Translokation Frequenzen erhalten selektiv (6.0x10 -2) und nicht-selektiv (11.9x10 -2) mit dem rad51Δ - / - homozygot waren ähnlich, aber statistisch verschieden (p-Wert = 0,0089). Die Frequenzen erhalten sowohl selektiv (p-Wert = 0,0001) und nicht-selektiv (p-Wert = 0,0002) mit dem Rad51 ° - / - homozygot waren statistisch unterscheiden sich von denen unter Verwendung der entsprechenden Bedingungen mit dem Wildtyp-Stamm.
Vermeintliche Translokation tragenden Klone können durch genomische Southern-Blot-und chromosomale-Blot-Analysen (Abbildung 3) untersucht werden. Für Southern-Analyse ist die genomische DNA mit BamHI Endonuklease vor Agarosegelelektrophorese verdaut, Blotting und Hybridisierung zu einem 32P-markierten 1.8kb HIS3-Sonde der diagnostischen 0,8 kb his3Δ200, 1,7 kb his3-Δ3 ', 4 kb TIII:: XV, 5 kb TXV: III und 8 kb his3-Δ5' visualisieren Fragmente (Abbildung 3B.1) . Intakten Chromosomen können hergestellt werden, getrennt durch CHEF (Abbildung 3B.2), ausgelöscht zu Nylon und hybridisiert mit dem 32 P-markierten 1,8 kb HIS3 Sonde an die 1,1 Mb intakten Chromosom XV, 0,8 Mb TXV visualisieren: III Translokation Chromosom, 0,6 Mb TIII: XV Translokation Chromosom und 0,3 Mb intakten Chromosom III (Abbildung 3B.3). Eine grafische Darstellung Karte erwartet BamHI Endonuklease-verdaute genomische DNA-Fragmente, und Eltern und rekombinante Chromosomen, dargestellt (Abbildung 3A).

Abbildung 1. Formation der Translokation Chromosomen von Einzelstrang-Annealing (SSA). 1) DSBs sind am his3-Δ3 'und his3-Δ5 "Substrate (Chromosomen XV und III bezeichnet) durch HO-Endonuklease nach der Zugabe von Galaktose zu den Kulturen geschaffen. 2) DSBs werden verarbeitet, um 3 'single-Stränge an den Enden des gebrochenen Chromosomen zu erzeugen. 3) SSA Maschinen glüht komplementäre 311 oder 60 Nukleotid-Einzelstrang-HIS3-Sequenzen an jedem der Rekombination Substraten. Non-homologen Schwanz durch Tempern entstehen durch Endonukleaseverdau entfernt. Ergänzende vier bp Überhängen auf den verbleibenden chromosomale Fragmente von HO-Endonukleaseverdau gebildet kann auch glühen. 4) Ligation schließt die Schaffung eines intakten HIS3-Gens und TXV: III Translokation Chromosom von SSA. Zellen, die dieses Chromosoms kann durch ihre Fähigkeit, auf Medium ohne Histidin wachsen ausgewählt werden. Ligation kann auch erzeugen die gegenseitige TIII:: XV Translokation Chromosom von einem NHEJ-ähnlichen Mechanismus.
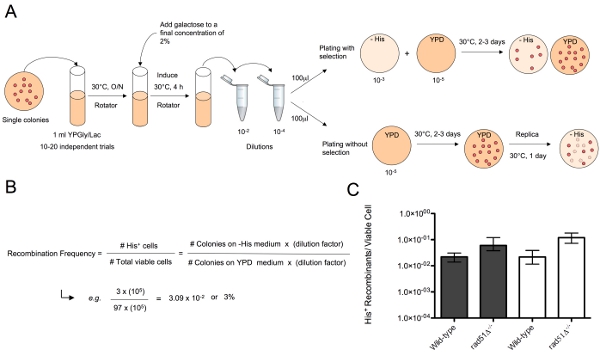
Abbildung 2. Assay zur Bestimmung der Häufigkeit der Translokation von SSA. A) Ein ml YPGly / Lac Kulturen werden mit einzelnen Kolonien von Zellen eines wählen Genotyp überimpft und eine angemessene Dichte von ca. 5x10 7-1x10 8 Zellen / ml. Galactose ist bis zu einer Endkonzentration von 2% bis DSBs bei der Rekombination Substrate auf den Chromosomen III und XV erstellen aufgenommen. Zur Durchführung des Tests unter selektiven Bedingungen sind entsprechende Verdünnungen hergestellt, so dass ungefähr 100 bis 200 Zellen auf YPD ausplattiert und eine ausreichende Anzahl von Zellen auf Medium ohne Histidin beschichtet, um eine beobachtbare Zahl Seiner + rekombinante Kolonien erhalten. Zur Durchführung des Tests ohne Selektion, sind etwa 100 bis 200 Zellen auf YPD vernickelt, angebaut für zwei vor drei Tagen bis zu einzelnen Kolonien und dann Replik auf Medium ohne Histidin ausplattiert. B) Translokation Frequenz kann durch Division der Anzahl der Kolonien, die auf-His-Platten wachsen, indem der Anteil, der auf YPD wachsen bestimmt werden. C) Die Translokation Frequenzen von Stämmen von unterschiedlichen Genotyp (dh Wild-Typ und rad51Δ - / -) können grafisch dargestellt, um Unterschiede in der Fähigkeit dieser Stämme, die DSBs von SSA Reparatur zu vergleichen.
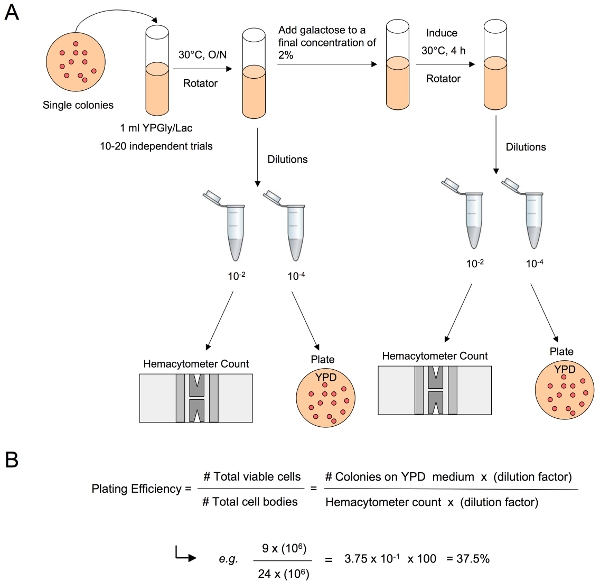
Abbildung 3. Bestimmen Platierungseffizienz. (A) Ein Aliquot von Zellen aus der Übernacht-Kultur vor und nach dem DSB-Induktion genommen werden geeignete Verdünnungen hergestellt, gefolgt von einem Hämacytometer verlassen, dass die Gesamtzahl der Zellkörper pro ml Kultur zu bestimmen. Geeignete Verdünnungen werden auf nicht-selektiven Medium ausplattiert, um die Gesamtzahl der lebenden Zellen pro ml zu bestimmen, durch Zählen der Kolonien, die auf YPD erscheinen. (B) Die Platierungseffizienz wird dann durch Division der Gesamtzahl der lebenden Zellen durch die Gesamtzahl der Zellkörper und Multiplikation dieses Quotienten mit 100 bestimmt.
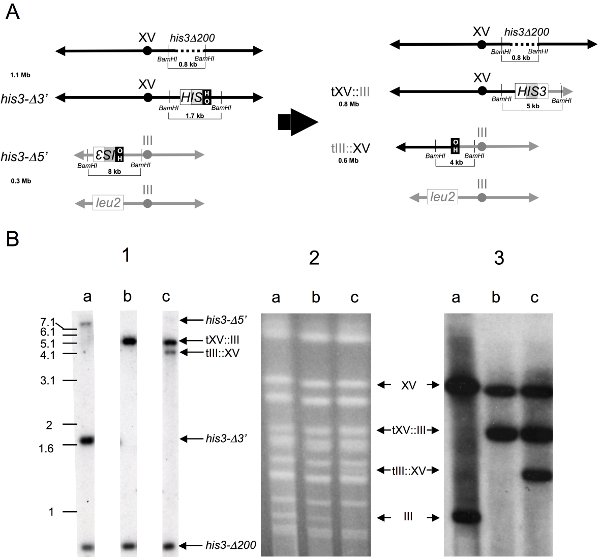
Abbildung 4. Nachweis von chromosomalen Translokation Ereignisse durch genomische Southern-Blot-und Chromosomen-Blot-Analysen.
A) Graphische Darstellung der relevanten Chromosomen vor (links) und nach (rechts) Translokation Bildung.
Die Größen der Eltern-und rekombinanten Chromosomen sind in Megabasen-Paaren (Mb) aufgeführt. Die Größen der Restriktionsfragmente mit entsprechenden Sequenzen von BamHI Verdau von genomischer DNA von den Eltern und rekombinanten Stämmen erzeugt, und zeigte auf Blots durch Hybridisierung mit einer 1,8 kb BamHI HIS3 genomischen Klon, sind in Kilobasen-Paaren (kb) aufgeführt. Chromosomen sind nicht maßstabsgetreu.
B) Physikalische Analyse von mutmaßlichen Translokation tragenden Klone.
(1) Genomic Southern-Blot-Analyse - Genomic DNA wurde gesammelt und mit BamHI Restriktionsendonuklease verdaut, gelelektrophoretisch, blotted zu Nylon und hybridisierte mit einer 32 P-markierten 1,8 kb HIS3-Sonde auf die folgenden Fragmente zu visualisieren: 0,8 kb his3Δ200, 1,7 kb his3-Δ3 ', 4 kb TIII:: XV, 5 kb TXV: III und 8 kb his3-Δ5 ". Lanes: a) Eltern diploid, b) Sein + nicht-reziproken Translokation rekombinanter, c) Sein + reziproken Translokation rekombinanter.
(2) CHEF Gele - Intact Chromosomen wurden in Agarose-Plugs bereit, getrennt durch CHEF, mit Ethidiumbromid angefärbt und unter UV-Licht. Lanes: Wie oben.
(3) Chromosome Blots - Separated Chromosomen wurden auf Nylon geblottet und hybridisiert mit dem 32 P-markierten 1,8 kb HIS3-Sonde auf die folgenden Chromosomen sichtbar: 1,1 Mb intakten Chromosom XV, 0,8 Mb TXV: III Translokation Chromosom, 0,6 Mb TIII: XV Translokation Chromosom und 0,3 Mb intakten Chromosoms III. Lanes: Wie oben.
Diskussion
Hohe Dosen ionisierender Strahlung stellen eine inhärente Risiko der Genom-Instabilität durch die Erzeugung einer großen Anzahl von DSBs 19. Eukaryotischen Genomen sind voll von repetitiven Sequenzen, die ausgezeichnete Substrate zur Erzeugung von Translokationen und andere genomische Rearrangements 20,21 sind. Chromosomalen Translokationen von HR sind häufig zu beobachten, wenn DSBs zwischen repetitiven Sequenzen 12,21,22 eingeführt werden. Überwältigende Beweise darauf hin, dass ein großer Teil der genomischen Instabilität mit Leukämien und Lymphomen können chromosomale Translokationen zurückzuführen verbunden, was die Bedeutung zu verstehen, wie dieser Mechanismus in Eukaryoten 22,23 auftritt. Wir haben ein System in Hefezellen entwickelt für die Untersuchung der Bildung von Translokation Chromosomen von DSB-induzierten HR zwischen kurz Regionen der Homologie auf verschiedenen Chromosomen, die eine ähnliche Größe, um sich wiederholende Elemente in der Hefe und dem menschlichen Genom verteilt sind.
In dem Test wird die his3-Δ3 "Translokation Substrat auf einer Kopie des Chromosoms XV gelegen. Die anderen his3 Allel (his3-Δ200) hat eine ~ 1kb Löschung des HIS3-Promotor und kodierender Sequenz, dass diese Sequenz verhindert als Vorlage für die Reparatur 24 verwendet. Die his3-Δ5 "Substrat an der LEU2-Locus auf eine Kopie des Chromosoms III befindet, mit der anderen Kopie des Chromosoms III enthält eine unveränderte LEU2-Allel (Abbildung 1). Ein Galaktose-induzierbaren HO-Endonuklease Expressionskassette mit KAN-MX gekennzeichnet war in den TRP1 Locus von Chromosom IV (:: GAL-HO-KAN-MX trp1) eingefügt. Jeder Translokation Substrat wird durch eine HO-Endonuklease Erkennungssequenz, dass für die Spaltung durch Induktion der Expression des HO-Gens durch die Zugabe von Galactose auf das Medium ausgerichtet werden kann flankiert. Nach HO-Endonuklease induzierte Spaltung an der his3-Δ3 'und his3-Δ5 "Substrate können Zellen effizient zu nutzen das gemeinsame kurz-Darm-Trakt des HIS3 Sequenzhomologie (311 bp oder 60 bp), um die gebrochenen Chromosomen von HR reparieren, wodurch eine Translokation Chromosom mit einem intakten HIS3 Allel 12-14,25.
Weil die Stammzellen eine intakte Kopie des HIS3-Gen fehlt, sind sie nicht in der Lage zu wachsen auf-His-Medium. Nur die Zellen, die Translokation Veranstaltung erfahren haben, können auf Medium ohne Histidin ausgewählt werden. Daher kann die Häufigkeit von chromosomalen Translokationen, indem die Anzahl von Histidin prototrophen Kolonien durch die Gesamtzahl der lebenden Zellen überzogen, durch Ausplattieren auf YPD bestimmt berechnet werden. Genomic DNA und intakte Chromosomen können dann von repräsentativen Sein + Kolonien, und das Vorhandensein einer Translokation Chromosom von genomischen Southern-Blot-Analysen und-Chromosom nachgewiesen isoliert werden.
Eine sorgfältige Analyse hat uns erlaubt, weitere wichtige Informationen über den Test 12 zu sammeln. Genomic Southern-Blot-Analyse hat Beweise dafür, dass es praktisch vollständige Trennen von Chromosomen XV und III nach 30 Minuten HO-Endonuklease Induktion vorgesehen, und somit gibt es keine signifikanten Hintergrund uncut chromosomale Substrate in der Bevölkerung (G. Manthey & A. Bailis, unveröffentlichte Ergebnisse). Genomic Süd-und Chromosomen-Blot-Analysen von Sein - Überlebende zeigt, dass die Zellen häufig verlieren, die anderen oder beide geschnitten Chromosomen und lebensfähig bleiben (L. Liddell & A. Bailis, unveröffentlichte Ergebnisse). Wichtig ist, dass die fast gleichwertigen Beschichtung Effizienz auf nicht-selektiven Medium vor und nach Induktion der Expression von HO-Endonuklease zeigt an, dass weder das Scheitern, um gebrochen Chromosomen reparieren, noch die fehlende Translokation Chromosomen behalten die Fähigkeit, DSB Bildung überleben beeinflusst. Im Einklang mit dieser, die TXV: III Translokation Chromosom hat sich gezeigt, instabil zu sein, in mitotischen Zellen in der Abwesenheit von Selektion. Dies wurde durch die wachsende TXV gezeigt: III enthalten sein + Rekombinanten Nacht nicht selektiv, Plattieren einzelne Kolonien auf nicht-selektiven Platten und Replika-Plattierung auf selektiven Medium ohne Histidin. : III Translokation Chromosom (N. Pannunzio & A. Bailis, unveröffentlichte Ergebnisse): Zehn bis 70% der Kolonien, die sich auf diesen Platten hatten die TXV verloren.
Translokation Chromosomen durch IR-Exposition beim Menschen erzeugt weisen eine ähnliche Instabilität 26. Dies deutet darauf hin, dass die Translokation Bildung kann zu frühen Ereignisse in der Tumorgenese durch die Förderung der Verlust der Heterozygotie beitragen. Zweitens schlagen umfangreiche genetische und molekulare Analysen, SSA, eine effiziente und zwingend nicht-konservativen Mechanismus der HR, der primäre Mechanismus der Translokation Bildung von HR nach der gleichzeitigen Schaffung von DSBs auf beiden Chromosomen 12,27,28 ist. Dies ist consistent mit der Feststellung, dass hohe Dosen von IR Ergebnis in einer Dichte von DSBs ausreichend Pausen neben mehreren repetitiven Sequenzen in der Hefe-Genoms zu erstellen, und eine hohe Frequenz von Translokation Bildung von HR. Zusammengenommen deuten diese Beobachtungen, dass die onkogene Wirkung der IR-Bestrahlung beim Menschen kann zum Teil ergeben sich aus der Reparatur von DSB durch einen effizienten Mechanismus des HR, dass Translokationen, die durch ihre inhärente Instabilität fördern genetischen Veränderungen, die Einführung Tumorgenese generiert. Da Strahlung wird oft verwendet, um Krebs, Genom Umlagerungen zu behandeln, die sich aus Reparatur von strahleninduzierten DSBs kann zur Bildung von sekundären Tumoren, die häufig auftreten bei Patienten beitragen. So kann dieses Modell uns dabei helfen, ein besseres Verständnis der genetischen und molekularen Grundlagen der eine wichtige klinische Ansprechen auf die IR-Behandlung.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch Mittel der National Institutes of Health und dem Beckman Research Institute der City of Hope unterstützt. Wir möchten den Gutachtern für ihre konstruktiven Kommentare, die Klarheit, das Manuskript aufgenommen danken.
Materialien

Referenzen
- Sen, S. K. Human genomic deletions mediated by recombination between Alu elements. The American Journal of Human Genetics. 79, 41-53 (2006).
- Han, K. Alu recombination-mediated structural deletions in the chimpanzee genome. Plos Genetics. 3, 1939-1949 (2007).
- Zhang, F. Copy number variation in human health, disease, and evolution. Annual Review of Genomics and Human Genetics. 10, 451-481 (2009).
- Goodier, J. L., Kazazian, H. H. Retrotransposons revisited: The restraint and rehabilitation of parasites. Cell. 135, 23-35 (2008).
- Lanktree, M., Hegele, R. Copy number variation in metabolic phenotypes. Cytogenet Genome Res. 123, 169-175 (2008).
- Mullighan, C. Genome-wide analysis of genetic alterations in acute lymphoblastic leukaemia. Nature. 446, 758-764 (2007).
- Mattarucchi, E. Microhomologies and interspersed repeat elements at genomic breakpoints in chronic myeloid leukemia. Genes Chromosomes Cancer. 47, 625-632 (2008).
- Stenger, J. Biased distribution of inverted and direct Alus in the human genome: implications for insertion, exclusion, and genome stability. Genome Res. 11, 12-27 (2001).
- Hedges, D., Deininger, P. Inviting instability: Transposable elements, double-strand breaks, and the maintenance of genome integrity. Mutat Res. 616, 46-59 (2007).
- Symington, L. Role of RAD52 epistasis group genes in homologous recombination and double-strand break repair. Microbiol Mol Biol Rev. 66, 630-670 (2002).
- Lewis, L., Resnick, M. Tying up loose ends: nonhomologous end-joining in Saccharomyces cerevisiae. Mutat Res. 451, 71-89 (2000).
- Pannunzio, N. R., Manthey, G. M., Bailis, A. M. RAD59 is required for efficient repair of simultaneous double-strand breaks resulting in translocations in Saccharomyces cerevisiae. DNA Repair (Amst). 7, 788-800 (2008).
- Nicholas, R., Pannunzio, G. M. M., Bailis, A. M. RAD59 and RAD1 cooperate in translocation formation by single-strand annealing in Saccharomyces cerevisiae. Curr Genet. 56, 87-100 (2010).
- Manthey, G. M., Bailis, A. M. Rad51 Inhibits Translocation Formation by Non-Conservative Homologous Recombination in Saccharyomyces cerevisiae. Plos One. 5, (2010).
- Knight, W. Confidence Intervals for the Median: Two sided Symmetric --95 or Better. , Available from: http://www.math.unb.ca/~knight/utility/MedInt95.htm (2006).
- Hoffman, C. S., Winston, F. A ten-minute DNA preparation from yeast efficiently releases autonomous plasmids for transformation of Escherichia coli. Gene. 57, 267-272 (1987).
- Sambrook, J., MacCallum, P., Russell, D. Molecular Cloning: A Laboratory Manual. , 3rd edition, Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY. (2001).
- Iadonato, S. P., Gnirke, A. RARE-cleavage analysis of YACs. Methods Mol. Biol. , 75-85 (1999).
- Argueso, J. L. Double-strand breaks associated with repetitive DNA can reshape the genome. Proc. Natl. Acad. Sci. U. S. A. , 11845-11850 (2008).
- Batzer, D. A. Alu repeats and human disease. Molecular Genetic Metabolism. 67, 183-193 (1999).
- Fasullo, M., Dave, P., Rothstein, R. DNA-damaging agents stimulate the formation of directed reciprocal translocations in Saccharomyces cerevisiae. Mutat. Res. 314, 121-133 (1994).
- Richardson, C., Jasin, M. Frequent chromosomal translocations induced by DNA double-strand breaks. Nature. 405, 697-700 (2000).
- Look, A. T. Genes altered by chromosomal translocations in leukemias and lymphomas. The Genetic Basis of Human Cancer. , McGraw Hill. (2002).
- Fasullo, M. T., Davis, R. W. Direction of chromosome rearrangements in Saccharomyces cerevisiae by use of his3 recombinational substrates. Mol. Cell. Biol. 8, 4370-4380 (1988).
- Manthey, G. M., Naik, N., Bailis, A. M. Msh2 Blocks an Alternative Mechanism for Non-Homologous Tail Removal during Single-Strand Annealing in Saccharomyces cerevisiae. PLoS One. 4, e7488-e7488 (2009).
- Muller, I. Time-course of radiation-induced chromosomal aberrations in tumor patients after radiotherapy. Int. J. Radiation Oncology Biol. Phys. 63, 1214-1220 (2005).
- Lin, F. L., Sperle, K., Sternberg, N. Model for homologous recombination during transfer of DNA into mouse L cells: role of DNA ends in the recombination process. Mol. Cell. Biol. 4, 1020-1034 (1984).
- Ivanov, E. L. Genetic Requirements for the single-strand annealing pathway of double-strand break repair in Saccharyomyces cerevisiae. Genetics. 142, 693-704 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten