Method Article
Ein-Tages-Workflow-Schema für bakterielles Pathogen-Erkennung und Resistenz gegen antimikrobielle Tests aus Blutkulturen
In diesem Artikel
Zusammenfassung
Die Konstruktion eines einfachen Ein-Tages-Workflow System für den bakteriellen Erreger-Diagnostik ermöglicht die schnelle Erkennung von Infektionen der Blutbahn. Die Aufnahme von acht klinisch relevanten bakteriellen Ziele und deren Antibiotikaresistenz Profile bietet dem Kliniker einen ersten Einblick am selben Tag, was zu mehr adäquate Therapie führen kann.
Zusammenfassung
Infektionen der Blutbahn sind mit hohen Sterberaten wegen der wahrscheinlichen Manifestation der Sepsis, schwere Sepsis und septischer Schock 1 zugeordnet. Daher ist die rasche Verabreichung geeignete Antibiotika-Therapie sind von besonderer Bedeutung bei der Behandlung von Infektionen des Blutkreislaufs. Das kritische Element in diesem Prozess ist das Timing, stark abhängig von den Ergebnissen der Identifizierung von Bakterien und Antibiotika-Empfindlichkeitsprüfung. Beide Parameter werden routinemäßig von Kultur-basierte Tests, die zeitaufwendig ist und dauert durchschnittlich 24-48 Stunden 2, 4 erhalten. Das Ziel der Studie war es, DNA-basierter Tests zur raschen Identifizierung von Infektionen des Blutkreislaufes, sowie eine rasche antimikrobielle Empfindlichkeitsprüfung zu entwickeln. Der erste Test ist ein eubakteriellen 16S rDNA-basierten Echtzeit-PCR-Assay mit Art-oder Gattungs-spezifischen Sonden 5 ergänzt. Mit Hilfe dieser Sonden, Gram-negative Bakterien einschliesslich Pseudomonas spp., Pseudomonas aeruginosaund Escherichia coli sowie gram-positive Bakterien einschließlich Staphylococcus spp., Staphylococcus aureus, Enterococcus spp., Streptococcus spp. und Streptococcus pneumoniae unterschieden werden könnten. Mit dieser Multiprobe Assay wurde eine erste Identifizierung des verursachenden Mikroorganismus nach 2 h angegeben.
Zweitens haben wir eine semi-molekularen Test zur Antibiotika-Empfindlichkeitsprüfung von S. aureus, Enterococcus spp. und (fakultativ) aerobe Gram-negative Stäbchen 6. Dieser Assay basiert auf einer Studie, in der PCR verwendet, um das Wachstum von Bakterien 7 zu messen beruht. Bakterien direkt aus dem Blut Kulturen geerntet werden für 6 h mit einer Auswahl von Antibiotika inkubiert und im Anschluss an eine Sybr Grün-basierte Echtzeit-PCR-Assay bestimmt Hemmung des Wachstums. Die Kombination dieser beiden Methoden konnte die direkte Wahl einer geeigneten Antibiotika-Therapie am selben Tag (Abbildung 1). Abschließend, Molekulare Analyse sowohl Identifikation und Empfindlichkeit gegenüber Antibiotika bietet eine schnellere Alternative für den Erregernachweis und könnte die Diagnose von Infektionen der Blutbahn zu verbessern.
Protokoll
TEIL I: Erregeridentifizierung
1. Probenvorbereitung
Hinweis: Der gesamte Workflow als molekulare wird im folgenden beschriebenen Protokoll sollte nach Empfehlungen für die Qualitätssicherung in der molekularen Diagnostik 3 durchgeführt werden.
- Fügen Sie ein 0,1 ml Aliquot der Blutkultur bis 0,9 ml 0,9% NaCl in ein 1,5 ml Reaktionsgefäß auf eine 1:10 verdünnte Probe zu werden. (Zu verdünnen, um qPCR-Hemmung zu verhindern).
- Zentrifuge Proben bei 13.400 × g für 5 min auf die bakterielle DNA Pellet.
- Resuspendieren Sie das Bakterienpellet in 100 ul sterilem demineralisiertem H 2 O.
- -Store der DNA-Probe bei 4 ° C bis zur weiteren Verwendung.
2. Identification Test: Real-time PCR-16S rDNA
- Planen der Reaktionsgemische wie folgt. Der Assay besteht aus vier getrennten Reaktionen pro Probe. Jede Mischung enthält 12,50 ul MasterMix, 0,9 um Forward-Primer (5-TCCTACGGGAGGCAGCAGT-3) 7, 0,6 um Reverse-Primer (5-GGACTACCAGGGTATCTAATCCTGTT-3) 8 und eine Serie von Sonden. Die Menge an Sonden für jede der vier getrennten Reaktionen in unten angegeben.
- Die erste Reaktion umfasst:
- 0,2 uM universelle Sonde (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1) 8
- 0,2 uM P. aeruginosa-Sonde (5-JOE-CCAAAACTACTGAGCTAGAGTACG-3-BHQ1)
- Die zweite Reaktion beinhaltet:
- 0,2 uM E. coli-Sonde (5-JOE-GGAGTAAAGTTAATACCTTTGCTCATT-3-BHQ1)
- 0,2 uM Pseudomonas spp. Sonde (5-NED-CCTTCCTCCCAACTTAAAGTGCTT-3-MGBNFQ)
- Die dritte Reaktion umfasst:
- 0,2 uM Staphylococcus spp. Sonde (5-NED-AATCTTCCGCAATGGGCGAAAGC-3-MGBNFQ)
- 0,2 uM S. aureus Sonde (5-FAM-AGATGTGCACAGTTACTTACACATAT-3-BHQ1)
- 0,2 uM Enterococcus spp. (5-JOE-TCCTTGTTCTTCTCTAACAACAGAG-3-BHQ1)
- Die vierte Reaktion umfasst:
- 0,2 uM universelle Sonde (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1)
- 0,3 uM Streptococcus spp. Sonde (5-NED-CCAGAAAGGGACSGCTAACT-3-MGBNFQ)
- 0,2 uM S. pneumoniae-Sonde (5-JOE-CCAAAGCCTACTATGGTTAAGCCA-3-BHQ1)
- In sterilem demineralisiertem H 2 O auf ein Gesamtvolumen von 20 ul zu erreichen. Je 20 ul jeder Reaktionsmischung auf die Vertiefungen einer 96-well PCR-Platte.
- 5 l der Probe in jede Vertiefung.
- Verwenden Sie eine Klebefolie, die 96-well PCR-Platte zu versiegeln.
- Führen Sie die Platte auf dem ABI PRISM 7900 HT Real Time PCR System mit den folgenden optimalen thermischen Bedingungen Radfahren:
- Vorheizung bei 50 ° C für 10 min
- Initiale Denaturierung bei 95 ° C für 15 min
- 42 Zyklen von
- Denaturierung bei 95 ° C für 15 s
- Annealing bei 60 ° C für 1 min
3. Analyse der Ergebnisse
Stellen Sie die Schwelle der CT-Analyse zu 0,1 auf der Registerkarte Analysis Settings. Grenzen Sie die Basiskonfigurationen zu Start (Zyklus): 6 und End (Zyklus): 15.
- Nehmen Sie die Schwellenwert-Zyklus (Ct)-Wert für alle Proben. Die Cut-Off-Wert, um ein PCR-Ergebnis positiv betrachten kann, um ein CT-Wert von 35 eingestellt werden. Die Menge der Bakterien in Blutkulturen reichten von 10 7 bis 10 11 KBE / ml, Erzeugen Ct-Werte unterhalb von 35.
TEIL II: Antibiotikaempfindlichkeit TESTS
4. Isolierung von Bakterien aus positiven Blutkulturen 9
- Saugen 5 ml Brühe aus einem positiven Blutkulturflasche und übertragen sie in eine Serumtrennröhrchen.
- Zentrifugieren Sie die Serumtrennröhrchen bei 2000 xg für 10 min.
- Verwerfen Sie den Überstand aus der Serumtrennröhrchen.
- Transfer-bacrien von der Gelschicht des Rohres mit einem sterilen Wattestäbchen in 0,9% Kochsalzlösung, bis eine 0,5 McFarland-Standard Suspension erhalten wird.
5. Inokulation von Mikrotiterplatten
- Verdünnen Sie die 0,5-McFarland-Suspension in doppelt konzentrierter Mueller Hinton II Bouillon um eine Suspension von 5 x 10 5 KBE / ml bilden.
- Dieses Suspension auf die Vertiefungen einer Mikrotiterplatte mit einer Auswahl von Antibiotika (Tabelle 1).
- Inkubieren Sie die Mikrotiterplatte bei 37 ° C für 6 Stunden.
- Bewahren Sie einen aliquoten Teil der Suspension bei 4 ° C (als negatives Wachstum Steuerung).
- Nach 6 h Inkubation, übertragen Sie den Inhalt jeder Vertiefung in ein steriles Röhrchen, sowie die negativen Wachstumskontrolle, die Probe wurde bei 4 ° C gelagert
- Zentrifugieren bei 16000 × g für 5 min.
- Entfernen Sie vorsichtig den Überstand, ohne das Bakterienpellet.
- Das Pellet in sterilem demineralisiertem H 2 O.
- Verdünnen Sie die Proben 10-fach in sterilem demineralisiertem H 2 O.
6. Real-time PCR-16S-rDNA-10
- Bereiten Sie den PCR-Ansatz wie folgt:
- 12,50 ul iQ SYBR Green Supermix
- 0,5 uM Vorwärtsprimer 16S-1-(5-TGGAGAGTTTGATCCTGGCTCAG-3) 11
- 0,25 uM Rückwärtsprimer 16S-2 (5-TACCGCGGCTGCTGGCAC-3) 11
- steril VE-H 2 O auf ein Gesamtvolumen von 20 ul
- In 20 ul PCR-Gemisch in die Vertiefungen einer 96-well PCR-Platte.
- 5 l der Probe in jede Vertiefung.
- Verwenden Sie eine Klebefolie, die 96-well PCR-Platte zu versiegeln.
- Führen Sie die Platte auf dem MyiQ einfarbige Real-Time PCR Detection System, mit Hilfe der folgenden optimalen thermischen Bedingungen Radfahren:
- Initiale Denaturierung bei 95 ° C für 4 min
- Anfängliche Anlassen bei 65 ° C für 30 s
- 35 Zyklen von
- Denaturierung bei 95 ° C für 15 s
- Annealing bei 60 ° C für 1 min
- Schmelzkurvenanalyse (60 bis 95 ° C in 20 Min. in Schritten von 0,57 ° C)
7. Analyse der Ergebnisse
- Berechnen Sie die Hell-Dunkel-Ct-Wert unter Verwendung einer der folgenden Formeln (je nach Art des Antibiotikums).
- Im Allgemeinen:
Cut-off-Wert = Ct Ct-Wert ein positives Wachstum Kontrolle + 0,5 x (Ct-Wert ein negatives Wachstum Kontrolle - Ct-Wert ein positives Wachstum Kontrolle) - Piperacillin, Piperacillin / Tazobactam und Ceftazidim in Gram-negative Stäbchen, Amoxicillin, Oxacillin und Trimethoprim / Sulfamethoxazol in S. aureus; Amoxicillin in Enterococcus spp. Cut-off-Wert = Ct Ct-Wert ein positives Wachstum Steuerung + 0,25 x (Ct-Wert ein negatives Wachstum Kontrolle - Ct-Wert ein positives Wachstum Kontrolle)
- Im Allgemeinen:
- Verwenden Sie die Probe mit sterilem demineralisiertem H 2 O als positive Wachstumskontrolle inkubiert.
- Abhängig von dem Mikroorganismus, verwenden Sie den entsprechenden negativen Wachstumskontrolle:
- Gram-negative Stäbchen Probe mit einer Mischung aus Antibiotika inkubiert
- Enterococcus spp. Probe bei 4 ° C gelagert
- S. aureus Probe bei 4 ° C gelagert
- Bestimmen der Empfindlichkeit (S) oder (R) des Stammes für die getestete Antibiotika wie folgt:
- Ein CT-Wert höher als der Cut-off-Ct-Wert gibt die Anfälligkeit
- Ein CT-Wert niedriger als der Cut-off-Ct-Wert gibt Widerstand
8. Repräsentative Ergebnisse
Zwei Modell-Organismen, also eine Gram-negativen E. coli und eines Gram-positiven S. aureus, ausgewählt sind, um das kombinierte Verfahren zum Nachweis und zur Identifizierung von pathogenen Bakterien und die Bestimmung ihrer antimikrobiellen Profil zu visualisieren. Der erste Teil des Protokolls umfasst die Erreger nachzuweisen. Specific Sonden zum Nachweis von acht klinisch relevanten Mikroorganismen gestalten. In Anwesenheit eines Ziels in der bakteriellen Platte enthalten sind, werden Amplifikationskurven erzeugt und Ct-Werte berechnet werden (Abb. 2). Die Cut-Off-Wert, um ein PCR-Ergebnis als positiv angesehen, um ein CT-Wert von 35 eingestellt. In 2A ist die Identifizierung Profil eines E. coli-infizierten Blutkultur gezeigt. Die 16S-Universal-Sonde wird in zwei getrennten Reaktionsmischungen enthalten und folglich generiert zwei Amplifikationskurven (Ct von 25,20 und 25,95). Das dritte Signal wird von der Sonde spezifisch für E. abgeleitet coli (Ct von 27,04). Die Identifizierung eines S. aureus-infizierten Blut Kultur wird in 2B gezeigt. Die 16S universellen Sonde hat Amplifikationssignale von 33,35 und 33,71. Die beiden verbleibenden Signale von den Sonden, die spezifisch für Staphylococcus spp. und S. aureus (Ct von 32,48 und 30,59).
Nach dem ersten Teil des Protokolls ist die verursachenden Mikroorganismus bekannt, und die antimikrobielle Profil bestimmt werden kann. Abbildung 3 ist ein Beispiel einer Antibiotika-Empfindlichkeitsprüfung Amplifikationsplot, als Vertreter der E. coli-Stamm, der auch gezeigt wurde in 2A. Jede Zeile steht für ein Antibiotikum, dass die Probe mit Bakterien inkubiert wurde. Eine Probe mit einer niedrigen Ct-Wert ist ein Beispiel, in dem Wachstum in Gegenwart eines Antibiotikums, der angibt, Resistenz gegen Antibiotika der getesteten aufgetreten ist. Im Gegenteil, stellt eine hohe Ct-Wert eine Probe, in der kein Wachstum, da der tatsächliche Funktionsweise des Antibiotikums, der angibt, Anfälligkeit für die getesteten Antibiotika aufgetreten ist. Tabelle 1 zeigt die Bestimmung der antimikrobiellen Profil des E. coli und S. aureus-Isolaten. Alle CT-Werte gemeldet werden und, nach den Formeln in der Protokoll-Text (7,1) erwähnt, zwei Cut-off-Ct-Werte sind errechnetlated zwischen Resistenz und Empfänglichkeit zu unterscheiden. Der Stamm ist resistent gegen das Antibiotikum wenn die gemeldete Ct-Wert niedriger ist als die berechnete Hell-Dunkel-Ct-Wert (und umgekehrt).
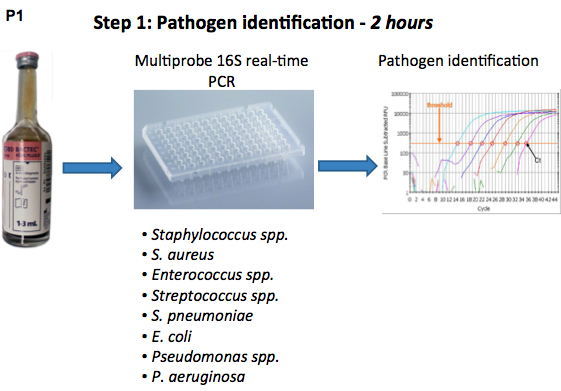
Abbildung 1. Ablaufschema Identifikation des Erregers und Empfindlichkeit gegenüber Antibiotika Testverfahren mittels real-time PCR-16S rDNA.
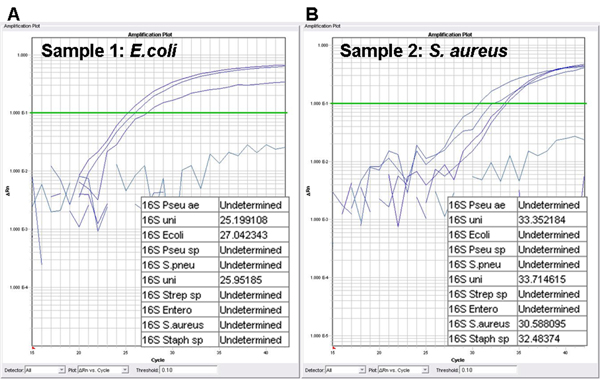
2 Identifizierung Assay. Amplifikationskurven und Zyklus Schwellenwerte (Ct-Werte) Eine positive Blutkultur, durch die allgemeine 16S rDNA-Sonde detektiert, während die spezifischen Sonden zur Identifizierung des ursächlichen Erreger verwendet werden.. A. Amplifikationsplot der Blutkultur mit E. coli, B. Amplifikationsplot der Blutkultur mit S. aureus, Pseudomonas ae, Pseudomonals aeruginosa; uni, 16S universellen Sonde; Ecoli, Escherichia coli-Sonde; Pseudomonas sp, Pseudomonas spp. Sonde; S. pneu, Streptococcus pneumoniae Sonde; Strep sp, Streptococcus spp. Sonde; Entero, Enterococcus spp. Sonde; S. aureus, Staphylococcus aureus-Sonde; Staph sp, Staphylococcus spp. Sonde.
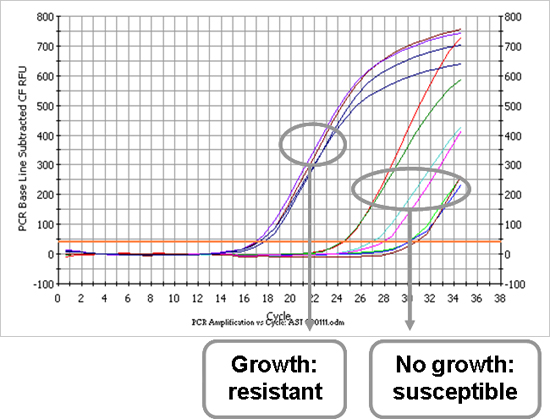
Abbildung 3. Amplifikationsplot von Antibiotika-Empfindlichkeitsprüfung eines E. coli-Isolat (Probe 1). Jede Kurve stellt ein Antibiotikum, dass der Stamm mit inkubiert. Ein frühes Signal wird durch einen hohen bakteriellen Belastung, so dass der Stamm in Gegenwart der getesteten Antibiotikum enthalten und ist somit gegen das Antibiotikum bedeutet, verursacht. Spät-Signale zeigen an, dass der Stamm nicht in Gegenwart des Antibiotikum enthalten, mit anderen Worten, ist sie anfällig.
| Beispiel 1: E. coli | Beispiel 2: S. aureus | ||||
| AST | Ct | R / S | AST | Ct | R / S |
| Amoxicillin 8 mg / L | 16,83 | R | Amoxicillin 0,25 mg / L | 21,03 | R |
| Amoxicillin-Clavulansäure 4.8 mg / L | 17,36 | R | Oxacillin 2 mg / l | 25,80 | S |
| Piperacillin 16 mg / L | 16,67 | R | Vancomycin 2 mg / l | 25,20 | S |
| Piperacillin-Tazobactam 16 /4 mg / L | 24,15 | S | Gentamicin 4 mg / L | 25,86 | S |
| Ciprofloxacin 1 mg / l | 29,72 | S | Trimethoprim-Sulfamethoxazol 2/38 mg / L | 24,62 | S |
| Ceftazidim 1 mg / l | 24,03 | S | |||
| Ceftazidim 8 mg / L | 26,58 | S | |||
| Gentamicin 4 mg / L | 29,83 | S | |||
| Trimethoprim-Sulfamethoxazol 2/38 mg / L | 27,60 | S | |||
| Negativen Wachstumskontrolle (Mischung von Antibiotika) | 30,41 | Negativen Wachstumskontrolle (Probe bei 4 ° C gelagert) | 27,42 | ||
| Positive Wachstums-Steuerung | 16,90 | Positive Wachstums-Steuerung | 20,22 | ||
| Cut-off-Ct-Wert 1 * | 21,76 | Cut-off-Wert Ct 1 *** | 23,82 | ||
| Cut-off-Ct-Wert 2 ** | 18,75 | Cut-off-Wert Ct 2 **** | 22,02 | ||
| * Für Amoxicillin, Amoxicillin-Clavulansäure, Ciprofloxacin, gentamicin, Trimethoprim-Sulfamethoxazol ** Für Piperacillin, Piperacillin-Tazobactam, Ceftazidim | *** Für Vancomycin und Gentamicin **** Für Amoxicillin, Oxacillin und Trimethoprim-Sulfamethoxazol |
Tabelle 1. Bestimmung der Antibiotika-Empfindlichkeitsprüfung der beiden Proben (E. coli und S. aureus). Ct-Werte der PCR-Assays wurden an diesem Excel-Datei kopiert, als denen kann berechnet automatisch die beiden cut-off Ct erte von der positiven und negativen Wachstumskontrolle, nach den Formeln in der Protokoll-Text angezeigt. Wenn ein Antibiotikum zeigt eine Ct-Wert niedriger als der Hell-Dunkel-Ct-Wert, der Stamm resistent gegen das Antibiotikum ist, wenn der Ct-Wert höher als der Hell-Dunkel, ist der Stamm anfällig.
Diskussion
Das hier beschriebene Protokoll ermöglicht die schnelle Identifizierung von Krankheitserregern und bietet eine funktionale antimikrobielle Profil, das auf die frühzeitige Verabreichung von Antibiotika ausreichend verbessert damit die Prognose von Patienten mit Infektionen der Blutbahn führen könnte. Je nach den gewünschten Bedingungen eines Tests, dh niedrige Kosten, hohe Durchsatz, minimal-umdrehen Zeit, Prüfbedingungen eingestellt werden kann. Das gesamte Verfahren kann innerhalb eines Arbeitstages durchgeführt werden. Darüber hinaus können die beiden Teile des Protokolls gleichzeitig durchgeführt werden, die reduziert die Durchlaufzeit deutlich. Wie hier dargestellt, ist die Identifikation Panel eine Auswahl der am meisten klinisch relevanten Bakterien in unserem Krankenhaus. Da der wichtigste Grundsatz zielt auf den 16S-Gen-Region, können spezifische Sonden für andere Mikroorganismen entwickelt und hinzugefügt werden, um den Test. Der komplette Test wurde ursprünglich für die schnelle Analyse von Blutkulturen vorgesehen, kann aber auch für die Verarbeitung von o verwendet werdenther Probenmaterialien. Dies ist auch der Fall bei den Antibiotika, die für Antibiotika-Empfindlichkeitsprüfung verwendet wurden: mehr oder andere Antibiotika können hinzugefügt, basierend auf lokalen Widerstand Mustern und Richtlinien werden.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde von der Profileringsfonds azM (PF245) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Natriumchlorid (NaCl) | Merck Chemicals | 106404 | 0,9% in Wasser |
| Vacutainer SST Serumtrennröhrchen 5 ml | BD Diagnostic Systems | 367986 | |
| Mueller-Hinton II Bouillon | BD Diagnostic Dystems | 212322 | 44 g / L in Wasser |
| Zentrifugieren ROTIXA 50 RS | Andreas Hettich GmbH & Co. KG | 4910 | |
| Centrifuge 5415 D | Eppendorf | Nicht mehr | |
| Grundierungen | Sigma-Aldrich | na | |
| Probes | Sigma-Aldrich/Applied Biosystems | na | |
| TaqManEnvironmental Master-Mix 2,0 | Applied Biosystems | 4396838 | |
| iQ SYBRGreen Supermix | Bio-Rad Laboratories BV | 170-8880 | |
| MicroAmp Optical 96-Well Drehmomentplatte | Applied Biosystems | N8010560 | |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | |
| iQ 96-Well PCR-Platten | Bio-Rad Laboratories BV | 223-9441 | |
| Microseal B Klebedichtungen | Bio-Rad Laboratories BV | MSB-1001 | |
| Real-time PCR Detection System | Applied Biosystems | ABI PRISM 7900 HT | |
| Real-Time PCR Detection System | Bio-Rad Laboratories BV | MyiQ einfarbige |
Referenzen
- Wallet, F. Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin. Microbiol. Infect. 16, 774 (2010).
- Beekmann, S. E., Diekema, D. J., Chapin, K. C., Doern, G. V. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J. Clin. Microbiol. 41, 3119 (2003).
- Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., Housni, H. E. l., Descheemaeker, P., De Schouwer, P., Franke, S., Hillen, F., Nollet, F., Soetens, O., Vankeerberghen, A. Molecular Diagnostics working group. Reflections and proposals to assure quality in molecular diagnostics. Acta. Clin. Belg. 66, 33 (2011).
- Peters, R. P. New developments in the diagnosis of bloodstream infections. Lancet Infect. Dis. 4, 751 (2004).
- Hansen, W. L., Beuving, J., Bruggeman, C. A., Wolffs, P. F. Molecular probes for the diagnosis of clinically relevant bacterial infections in blood cultures. J. Clin. Microbiol. 48, 4432-4432 (2010).
- Beuving, J. Antibiotic susceptibility testing of grown blood cultures by combining culture and real-time polymerase chain reaction is rapid and effective. PLoS ONE. 6, (2011).
- Rolain, J. M., Mallet, M. N., Fournier, P. E., Raoult, D. Real-time PCR for universal antibiotic susceptibility testing. J. Antimicrob. Chemother. 54, 538 (2004).
- Nadkarni, M. A., Martin, F. E., Jacques, N. A., Hunter, N. Determination of bacterial load by real-time PCR using a broad-range (universal) probe and primers set. Microbiol. 148, 257 (2002).
- Waites, K. B., Brookings, E. S., Moser, S. A., Zimmer, B. L. Direct susceptibility testing with positive BacT/Alert blood cultures by using MicroScan overnight and rapid panels. J. Clin. Microbiol. 36, 2052 ( ).
- Vliegen, I. Rapid identification of bacteria by real-time amplification and sequencing of the 16S rRNA gene. J. Microbiol. Meth. 66, 156 (2006).
- Hall, L., Doerr, K. A., Wohlfiel, S. L., Roberts, G. D. Evaluation of the MicroSeq system for identification of mycobacteria by 16S ribosomal DNA sequencing and its integration into a routine clinical mycobacteriology laboratory. J. Clin. Microbiol. 41, 1447 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten