Method Article
Genomweiten Screen für miRNA-Targets mit dem Missionsziel ID-Bibliothek
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Ziel-ID-Bibliothek ist ein Plasmid-basierte, Genom-weiten Sammlung von klonierten cDNA verwendet werden, um miRNA-Targets zu identifizieren. Hier zeigen wir, ihre Nutzung und Anwendung.
Zusammenfassung
Die Ziel-ID-Bibliothek wurde entwickelt, um zur Entdeckung und Identifizierung von microRNA (miRNA)-Ziele zu unterstützen. Die Ziel-ID-Bibliothek ist ein Plasmid-basierte, genomweiten cDNA-Bibliothek in der 3'UTR hinter der Dual-Auswahl-Fusionsprotein, Thymidinkinase-Zeocin (TKzeo) kloniert. Die erste Runde der Wahl ist für eine stabile Transformanten, gefolgt mit Einführung einer miRNA von Interesse, und schließlich die Auswahl für die cDNAs mit der miRNA-Ziel. Ausgewählte cDNAs werden durch Sequenzierung (siehe Abbildung 1-3 für die Ziel-ID-Bibliothek und Workflow-Details) identifiziert.
Um eine breite Abdeckung des menschlichen Transkriptom zu gewährleisten, wurden Ziel-ID-Bibliothek cDNAs über Oligo-dT-Priming mit einem Pool von Gesamt-RNA aus verschiedenen menschlichen Geweben und Zelllinien hergestellt generiert. Die resultierende cDNA Bereich von 0,5 bis 4 kb, mit einer durchschnittlichen Größe von 1,2 kb, und wurden in die p3TKzeo Dual-Auswahl Plasmid kloniert (siehe 4 für Plasmid-Karte). Die Gen-Targets in der Li vertretenthek kann auf der Sigma-Aldrich Webseite. Ergebnisse von Illumina Sequenzierung (Tabelle 3) zeigen, dass die Bibliothek 16.922 der 21.518 Gene, die in einzigartiger UCSC RefGene (79%) oder 14.000 Genen, die mit 10 oder mehr liest (66%) enthält.
Protokoll
1. Die Transfektion mit Ziel-ID-Bibliothek und Selektion auf stabile Zelllinien
1. Zeocin Tötungskurve
Zeocin wird verwendet, um stabil transfizierte Zellen auszuwählen. Jedoch verursacht überschüssiges Zeocin unerwünschten phänotypischen Reaktion in den meisten Zelltypen. Daher muss ein Kill-Kurven-Analyse durchgeführt, um die minimale letale Dosis zu ermitteln.
- Tafel 1,6 × 10 4 Zellen in Vertiefungen einer Platte mit 96 Vertiefungen in 120 ul Medien.
- Am nächsten Tag hinzuzufügen Zeocin in steigenden Konzentrationen im Bereich von 50 ug / ml bis 1 mg / ml in die entsprechenden Vertiefungen.
- Untersuchen Lebensfähigkeit alle 2 Tage.
- Tauschen Sie Medien mit Zeocin alle 3 Tage. Die minimale Konzentration des Selektionsmittels Reagens, das vollständige Zelltod nach der gewünschten Zeit sollte für diesen Zelltyp und Experiment verwendet werden. Unsere Ergebnisse zeigen, dass 500 ug / ml Zeocin Optimum liegt für A549, HeLa und MCF7-Zellen.
2.Bibliothek Transfektion und Selektion über Nucleofection
- Wählen Sie eine Zelllinie, die entweder nicht exprimiert oder äußert niedrigen Niveau Ihrer miRNA von Interesse. Die miRNA wird in Abschnitt B für die Zielauswahl werden, nachdem eine stabile Expression des Ziel-ID-Bibliothek eingeführt wird erreicht.
- Kultur / erweitern Zellen. Wir haben hervorragende Ergebnisse mit 2 x 10 7 Zellen pro Bibliothek Transfektion erhalten.
- Trypsinieren Zellen, die bei> 80% Konfluenz sind, und übertragen 2 x 10 7 Zellen in ein 15 ml sterilen Röhrchen Schraube Waggon.
- Pellet trypsiniert Zellen bei 200 × g für 5 min.
- Entfernen Sie das Medium und waschen Zellpellet mit HBSS oder 1X PBS.
- Zentrifuge bei 200 xg für 5 min und absaugen zu waschen.
- Wiederholen Sie Schritt waschen Zellpellet.
- Vorwärmen 6-Well-Platten mit 2 ml Vollmedium bei 37 ° C
- In 2 ug Ziel-ID-Bibliothek (nicht zu überschreiten 10 pl) pro 0,5 ml Röhrchen für jede Transfektion.
- Die Zellen in der 15 ml Tube von oben mit 100 ul Amaxa Nucleofection Solution (Zell-spezifische) je 2 x 10 6 Zellen. Zum Beispiel, für 10 Nucleofections 1 ml Reagenz.
- Eine Reaktion zu einer Zeit, fügen Sie 100 ul der Zellen an die 2 ug Plasmid. Mischen Sie mit Pipette.
- Übertragen Mischung auf eine Nucleofector Küvette.
- Küvette in Nucleofector Instrument und führen entsprechende optimiertes Programm für die Zell-Linie (High Efficiency über Lebensfähigkeit der Zellen bevorzugt).
- Füllen Sie Transferpipette mit vorgewärmten Medium. Nehmen Sie Zellen in derselben Pipette und Transfer zum 6-Well-Platte. Wiederholen Sie für jeden Nucleofection, einer pro Vertiefung.
- Zurück zur Klimakammer für Übernacht-Inkubation.
- Am nächsten Tag ersetzt Medium und Zellen ermöglichen für 3-5 Tage erholen.
- Ersetzen Medium mit Vollmedium mit den angemessenen Umfang der Zeocin, wie aus dem Kill-Kurve bestimmt.
- Monitor Zellen für Zeocin Auswahl (Zellen sterben).
- Ersetzen Medium mit Zeocin alle 2-3 Tage.
- Sobald konfluent in 6-Well-Platte, Passage, Pool und erweitern Zellen in größeren Flaschen.
- Länge der Zeit für die Expansion von Zellen ist benutzerfreundlich und Zelllinie abhängig, aber es wird dringend empfohlen, Zeocin resistente Zellen zu erweitern, um Kryo-Aktien für zukünftige Screening generieren (~ 2-3 Wochen; Zelllinie abhängig).
2. Transfizieren Bibliothek Cells mit miRNA-Expressions-Konstrukt, für stabilen Zelllinie auszuwählen, und wählen miRNA-Targets
Hinweis: Zeocin Auswahl ist nicht mehr erforderlich oder erwünscht ist. Exposition von Zellen gegenüber während der miRNA-Expression und Ganciclovir (Ziel-) Auswahl Zeocin kann den Verlust von miRNA-Zielen führen.
3. Puromycin, G418 und Ganciclovir Kill-Kurven
- Führen Sie ein Kill-Kurve für Ganciclovir-Zellen, die mit der Ziel-ID-Bibliothek und mit Puromycin oder G418 für WildtypZellen.
- Tafel 1,6 × 10 4 Zellen in Vertiefungen einer Platte mit 96 Vertiefungen mit 120 ul frisches Medium. Am nächsten Tag 0,1 bis 10 pg / ml puromycin/G418, oder 2 bis 32 um Ganciclovir an ausgewählte Brunnen hinzuzufügen.
- Untersuchen Lebensfähigkeit alle 2 Tage. Ersetzen Sie die Medien mit Selektionsreagenz alle 3 Tage. Die minimale Konzentration des Selektionsmittels Reagens, das vollständige Zelltod verursacht * nach der gewünschten Zeit, sollte für diesen Zelltyp und Experiment verwendet werden. Unsere Ergebnisse zeigen, dass 0,25 bis 1 mu g / ml Puromycin Optimum liegt für A549, HeLa und MCF7-Zellen, 0,3 pg / ml G418 für MCF7-Zellen und 8-16 uM Ganciclovir sind für eine optimale A549, HeLa und MCF7-Zellen.
* Hinweis: Bei Auswahl Ganciclovir, langsame oder gar kein Zellwachstum kann ohne vollständige Zelltod beobachtet werden. Beachten Zellen über mehrere Tage "Behandlung zu überprüfen, sie werden nicht aktiv teilenden. Wenn dies der Fall ist, Phenolrot in dem Medium bleibt rot. Es kann auch notwendig sein, eine ki durchführenll Kurve Analyse der Ziel-ID-Bibliothek transfizierten Zellen, wenn eine Veränderung der Empfindlichkeit beobachtet wird. Dies wird jedoch müssen auf einer zelltypspezifischen festgelegt werden.
4. miRNA-Transfektion über Nucleofection und Ziel-Auswahl
- Vergrößern / erweitern Target ID-Bibliothek Zellen zu 2 x 10 7 Zellen zu erreichen, und trypsinieren wenn Zellen bei> 80% Konfluenz sind.
- Transfer 2 x 10 7 Zellen in ein 15 ml sterile Schraube Spitze Rohr und Pellet bei 200 × g für 5 Minuten.
- Entfernen Sie das Medium und waschen Zellpellet mit HBSS oder 1X PBS.
- Zentrifuge bei 200 xg für 5 min und absaugen zu waschen.
- Wiederholen Sie Schritt waschen Zellpellet.
- Vorwärmen 6-Well-Platten mit 2 ml vollständigem Medium pro Vertiefung bei 37 ° C
- In 2 pg der miRNA-Expression Plasmid (nicht zu überschreiten 10 pl) pro 0,5 ml Röhrchen für jede Transfektion. Origene MicroRNA Expressionsplasmiden (Neomycin - G418 Auswahl) oder selbst geklont miRNA-Gene in pBABE-Puro (Plasmid 1764; Addgene) wurden erfolgreich eingesetzt. Für letztere wurde die miRNA Haarnadel mit ~ 200 bp auf jeder Seite PCR aus menschlicher DNA kloniert.
- Die Zellen in der 15 ml Tube von oben mit 100 ul Amaxa Nucleofection Solution (Zell-spezifische) je 2 x 10 6 Zellen. Zum Beispiel: Für 10 Nucleofections, 1 ml Reagenz.
- Eine Reaktion zu einer Zeit, fügen Sie 100 ul der Zellen an die 2 ug Plasmid. Mischen Sie mit Pipette.
- Übertragen Mischung auf eine Nucleofector Küvette.
- Küvette in Nucleofector Instrument und führen optimiertes Programm für die entsprechende Zelllinie (Hoher Wirkungsgrad über die Lebensfähigkeit der Zellen bevorzugt).
- Füllen Sie Transferpipette mit vorgewärmten Medium. Nehmen Sie Zellen in derselben Pipette und Transfer zum 6-Well-Platte. Wiederholen Sie für jeden Nucleofection, einer pro Vertiefung.
- Zurück zur Klimakammer für Übernacht-Inkubation.
- Ersetzen Medium und Zellen zu ermöglichen für 3-5 Tage zu erholen.
- Ersetzen Medium mit Vollmedium mit den angemessenen Umfang der Puromycin oder G418 als aus dem Kill-Kurve Test vor der Transfektion mit der miRNA-Konstrukt durchgeführt bestimmt.
- Überwachen Zellen für Puromycin / G418-Selektion (Zellen sterben).
- Ersetzen Medium mit Puromycin / G418 alle 2-3 Tage.
- Sobald konfluent in 6-Well-Platte, Passage, Pool und erweitern Zellen in größeren Flaschen.
- Länge der Zeit für die Expansion von Zellen ist abhängig von Benutzer, aber es wird dringend empfohlen Puromycin / G418-resistenten Zellen zu erweitern, um Kryo-Aktien für zukünftige Screening generieren (~ 2-3 Wochen; Zelllinie abhängig).
- Ersetzen Sie Medium mit den entsprechenden Ebenen von Ganciclovir (GCV) und Puromycin / G418, wie aus dem Kill-Kurve Test vor der Transfektion mit der miRNA-Konstrukt durchgeführt bestimmt.
- Überwachen Zellen zur Auswahl (Zellen dying, miRNA-Targeting und Knockdown von TK-ZEO).
- Erweitern Zellen in Gegenwart von GCV und Puromycin / G418.
- Planen Genomische DNA aus den ausgewählten Zellen GCV.
- PCR zu amplifizieren Einsätze mit Kit-Primer (siehe PCR Amplification).
- Clone in die TOPOTA Vektor (Invitrogen) für Standard-Sequenzierung oder unterbreiten PCR-Produkt für tief-Sequenzierung.
3. PCR-Amplify ausgewählten Bibliothek Inserts und Sequence
Hinweis: Dieses Verfahren wird durchgeführt, um PCR-Amplifikation die Bibliothek, die die Ziele Ganciclovir Auswahl überlebt haben.
5. Sammle Ganciclovir ausgewählten Zellen zu chromosomalen DNA vorzubereiten mit einem GenElute Mammalian Genomic DNA Miniprep Kit (Bestellnummer G1N10) oder gleichwertig. Wir empfehlen die Herstellung von DNA aus der übergeordneten Zell-Linie, die nicht enthalten Ziel-ID-Bibliothek, um als negative Kontrolle für den Vergleich mit DNA-Target verwenden von markierten Zellen in der PCR.
- PCR-Amvereinfachen die genomische DNA mit Amplifikationsprimer 1 und Primer 2 Amplifikation. Für die PCR (Bestellnummer P0982) - Eine erfolgreiche Amplifikation wurde mit JumpStart REDTaq ReadyMix Reaction Mix erhalten worden. Die Optimierung der Bedingungen kann es erforderlich sein, wenn eine andere Polymerase verwendet wird. Siehe Tabelle 1 für eine Probe PCR-Setup und Tabelle 2 für die PCR-Zyklus-Bedingungen.
- Auflösen 2-5 ul des PCR-Produkts auf einem 1% Agarosegel. Das erwartete Produkt sollte ein Abstrich mit einigen sichtbaren DNA-Banding sein. Vergleich zum PCR-Produkt von genomischer DNA aus Zellen ohne die Ziel-ID-Bibliothek zu steuern.
- Fahren Sie mit der Klonierung und / oder tiefen Sequenzierung von PCR-Produkt. Wenn kein Amplifikationsprodukt beobachtet wird, oder ist identisch mit DNA steuern, Optimierung der PCR-Amplifikation Bedingungen (dh, Primer-Konzentration, Annealing-Temperatur, und Zyklen).
6. Klonierung und Sequenzierung
Hinweis: Wir empfehlen die Klonierung der PCR-Produkts und dann die Durchführung Standard-Sequenzierung von mindestens 96 der Klone mit Hilfe der Amplifikationsprimer mit dem Kit mitgeliefert. Dieses Verfahren wurde erfolgreich durchgeführt mit 96-Loch-Nacht-Kulturen und Plasmid-Reinigungssystemen. Auch wenn tiefe Sequenzierung gewünscht wird, kann vorläufige Ergebnisse aus Klonen und die Standard-Sequenzierung als Qualitäts-Check, um festzustellen, ob die zusätzlichen Kosten der tiefen Sequenzierung gerechtfertigt ist verwendet werden.
- Verfolgen TA Cloning Kit-Protokoll Herstellung für die Klonierung PCR-Amplifikationsprodukte (oben).
- Transform-Klone in kompetente Bakterienzellen und wählen Sie über Nacht auf Antibiotika-haltigem Medium.
- Isolieren und wachsen einzelne Kolonien in Flüssigkultur mit einer angemessenen Antibiotika.
- Plasmid-DNA zu reinigen. 6.5. Führen Sie Sequenzierungsreaktionen mit Amplifikationsprimer.
- Identifizieren Gen-Targets durch BLAST-Alignment von Sequenzen mit menschlichen Transkriptom (für bekannte Transkripte) und des menschlichen Genoms (für Roman transcripts). *
* Cloning Fehlerbehebung: Wenn eine hohe Anzahl von Einsätzen aus Sequenzierung Plasmidsequenz sind, optimieren die Klonierung / Sequenzierung Bedingungen als solche:
- PCR-Amplifikation Produkt (Insert) Qualität und Quantität
- Ligationsreaktion
- Plasmidpräparation (Qualität und Quantität)
- Die Sequenzierung Reaktionsbedingungen
4. Repräsentative Ergebnisse
Bildschirm Bibliothek für miR-373 Ziele
Um die Leistung der Mission Ziel-ID-Bibliothek zu bewerten, wurden miR-373 Ziele aus MCF-7-Bibliothek exprimierenden Zellen ausgewählt. MCF-7 wurde gewählt, weil es wenig oder keine nachweisbare drückt miR-373 (Daten nicht gezeigt). miR-373 wurde für seine biologische Interesse gewählt. miR-373 Expression fördert Tumorinvasion und Metastasierung in MCF-7, in der Regel eine nicht-metastatischen Zelllinie [2]. Darüber hinaus miR-373 Orthologen aus der Maus miR-290-Cluster in embryonalen Stammzellen der Maus beteiligtZell-Wartung [3], und die miR-373 Familienmitglied, miR-372, fördert die Fibroblasten-Reprogrammierung zu induzierten pluripotenten Stammzellen [4]. Schließlich verwendeten wir Zinkfinger Nukleasen eine PGK-Promotor-miR-373 Expression in den AAVS1 Standort in MCF-7 Zellen konstruieren einzufügen, und generiert eine Liste potenzieller miR-373 Ziele durch RNA-Microarray-Analyse (Daten nicht gezeigt).
Die Ziel-ID-Bibliothek wurde in MCF-7-Zellen transfiziert, und eine stabile Population von Zellen wurde selektiert und amplifiziert in Zeocin-Medium. Die resultierenden Zellen (MCF-7-Bibliothek) wurden stabil mit einem miR-373 exprimiert Konstrukt transfiziert und in Ganciclovir-Medium für Zellen, die miR-373 Ziele zu bereichern. Wir beobachteten, dass die negative Kontrolle MCF-7-Zellen-Bibliothek (ohne miR-373) nicht zu trennen und abgerundet wie bei toten Zellen zu erwarten. Allerdings hat sie aufhören zu wachsen, das war leicht erkannt, weil der Phenolrot-haltigem Medium nicht drehen wollte orange-gelb als beobachtet for-Zellen, die miR-373 (Abb. 5). Die Zellen wurden in Ganciclovir-Medium expandiert und Ziel-Sequenzen wurden mittels PCR mit den Primern flankiert die Ziel-ID-Bibliothek cDNA-Inserts und DNA aus den überlebenden Zellen hergestellt isoliert. PCR-Produkte wurden sequenziert Illumina.
Wie in Tabelle 4 gezeigt, erhielten wir 17.740.719 cDNA liest, die 11.076 einzigartige kartiert. Von diesen einzigartigen Gene, wurden 2898 mit mehr als 40 mal gelesen erkannt, und deshalb wurden als zuverlässig Treffer. Die 13.106.469 Vektor liest wurde erwartet, weil die PCR-Primer verwendet, um cDNA-Inserts zu amplifizieren 40 sind und 300 Basen von der Insertionsstelle. Als solches wird erwartet, dass die meisten erhaltene Sequenzen aus dem Vektor würde im Gegensatz traditionellen tiefen Sequenzierung genomischer Material sein. Vierzehn Prozent nicht entweder Vektor oder cDNA zuzuordnen, aber mit NCBI nicht-redundanten Nukleotid-Datenbank (dh mit NR Hit) auszurichten, und nur 1% hatte keine cDNA-Insert.
Ein erster Vergleich ergab, dass 10 von den einzigartigen Gene mit der Ziel-ID-Bibliothek identifiziert sind auch in der Liste der zuvor identifizierten miR-373 Ziele in TarBase (Tabelle 5). Diese 10 miR-373 Ziele wurden zuvor von Microarray mit RNA aus HeLa-Zellen identifiziert transient mit einem synthetischen miR-373 nachzuahmen [5] transfiziert. Darüber hinaus entdeckten wir diese gleichen 10-Gene in MCF-7 Zellen, die miR-373 von der Seite AAVS1 (Daten nicht gezeigt) herunter reguliert. Deshalb sind diese 10 wahrscheinlich gültige Ziele von miR-373. Derzeit wird daran gearbeitet, die Liste der potenziellen Ziele und versuchen, ausgewählte Hits experimentell zu validieren durch RT-qPCR, Western Blot, und Luciferase-Reporter-Assay zu charakterisieren.
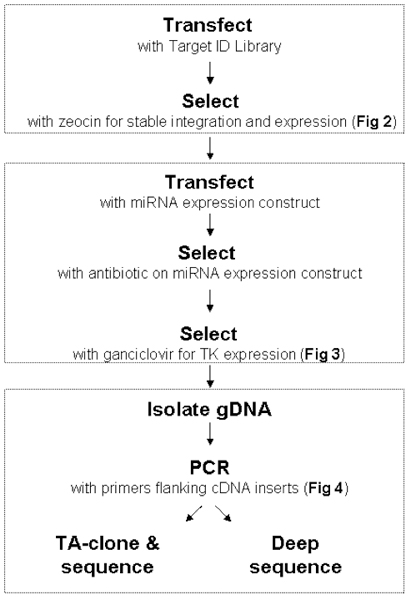
Abbildung 1. Workflow für Ziel-ID-Bibliothek. Abschnitte AC: auf Abschnitte in der Vorgehensweise finden. Jeder Schritt wird dargestellt und beschrieben im Detail in Bilder 2 und 3.

Abbildung 2. Transfektion und Zeocin Auswahl. Die Ziel-ID-Bibliothek ist ein Pool von Plasmiden (A), jeweils mit einer menschlichen cDNA in der 3'-UTR nach einem Thymidinkinase-Zeocin-Fusionsprotein (TKzeo; Abb. 4) eingesetzt ist. Die Zellen werden mit Ziel-ID-Bibliothek (B) transfiziert und man ließ sie für 3-5 Tage zu erholen. Konstrukte können in das Genom während dieser Erholungsphase (C) zu integrieren, und drücken die kodierten Transkript (D). Nach der Wiederherstellung werden die Zellen auf Zeocin (E) ausgesetzt. Zellen, die das Fusionsprotein aus TKzeo stabil integrierten Ziel-ID-Konstrukte überleben Zeocin Auswahl (F). Transfizierten Zellen sterben (G). Darüber hinaus beinhaltet alle Zellen ein Konstrukt, das ein Ziel für eine endogene miRNA ist oder eine andeER Faktor in der Zelle, die Expression von TKzeo hemmt aus der Ziel-ID-Konstrukt wird sterben (H) ausgedrückt.
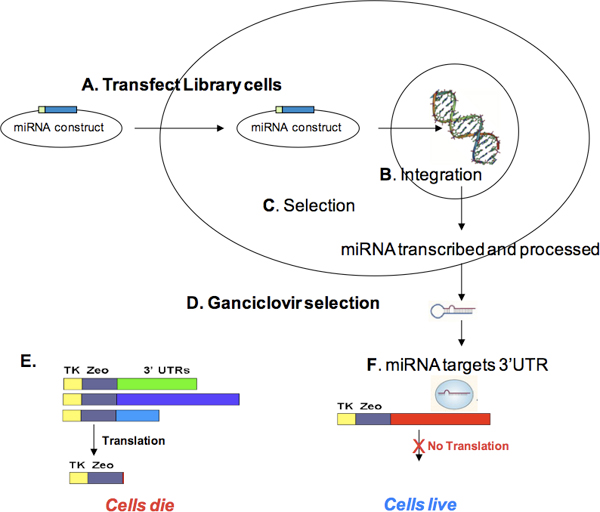
3. MiRNA Transfektion und Auswahl Ganciclovir. Zellen, welche die Ziel-ID-Bibliothek (dh Zeocin-markierten Zellen) mit einer wählbaren microRNA Expressionskonstrukt (A) transfiziert. Während der Wiederherstellung kann der miRNA-Expression Konstrukt integrieren (B) und drücken die Selektionsmarker auf der miRNA-Konstrukt kodiert. Nach Selektion auf stabile Integration (C) und Zellexpansion, werden Zellen mit Ganciclovir (D) behandelt. Zellen, die Thymidinkinase (TK) in Anwesenheit von Ganciclovir (dh Zellen, die nicht TKzeo Konstrukte durch die gezielte miRNA) wird (E) Auf der anderen Seite sterben Zellen, die Bibliothek mit miRNA Konstrukte Ziel s iten wird nicht zu TK, und daher wird die Auswahl Ganciclovir (F) zu überleben. Die überlebenden Zellen wachsen können, werden gDNA isoliert, und die cDNA enthaltenden miRNA Zielstellen PCR-amplifiziert unter Verwendung der Ziel-ID Amplifikationsprimer. PCR-Produkte können sequenziert und mit dem menschlichen Genom zu identifizieren miRNA Targets ausgerichtet werden.
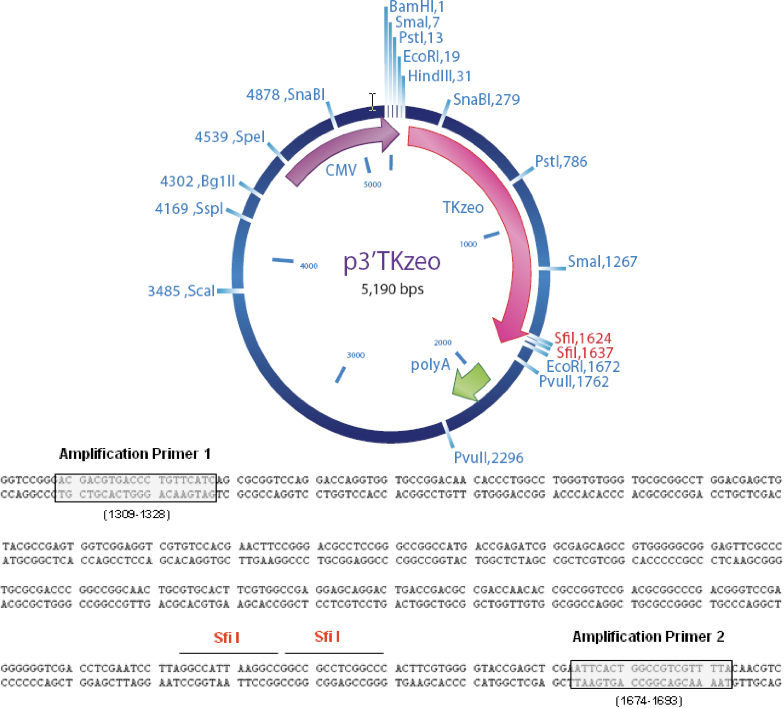
Abbildung 4. Plasmid-Karte und Lage von Amplifikationsprimer. Sfi I sind die Websites von cDNA Klonen.
Primersequenzen
MISSION Ziel-ID Amplification Primer 1
5 ACGACGTGACCCTGTTCATC 3
MISSION Ziel-ID Amplification Primer 2
5 TAAAACGACGGCCAGTGAAT 3
ig5.jpg "alt =" Bild 5 "/>
Bild 5. Ganciclovir Wirkung auf das Zellwachstum. Twenty-four-Well-Platten wurden mit 2 bis 100.000 MCF-7 Zellen oder MCF-7-Zellen, die das Ziel-ID-Bibliothek ausgesät. Vierundzwanzig Stunden später wurde das Medium durch Medium, enthaltend 0, 8 oder 16 uM Ganciclovir ersetzt. Nach 15 Tagen wurde die Platte fotografiert (6 oberen Brunnen), dann die Vertiefungen mit HBSS und gefärbt mit Brilliant Blue R-Färbelösung (B6529) (6 untere Brunnen) gewaschen. Beachten Sie, dass Ganciclovir keine Auswirkung auf MCF-7 Zellen hat, ohne Bibliothek, weil sie nicht exprimieren Thymidinkinase (TK). Auf der anderen Seite, zu tun MCF-7-Bibliothek exprimieren TK sind aber nicht vollständig von Ganciclovir mit 8 oder 16 uM getötet, wie durch die lebenden Zellen Aufnahme Brillinat blau angezeigt. Allerdings sind die Bibliothek Zellen aufhören zu wachsen Ganciclovir, als durch die Farbe des Phenolrot-Farbstoff in ihrem Medium belegt. Die verhafteten Zellen nicht säuern ihr Medium, und die Farbe bleibt anstatt sie zu verändern oder rötlichAnge-gelb.
| Reagens | Volumen / Reaktion |
| Jumpstart REDTaq ReadyMix Reaction Mix | 10 pl |
| Amplifikationsprimer-1 (25 uM) | 0,2 ul |
| Amplifikationsprimer-2 (25 uM) | 0,2 ul |
| Genomische DNA | 50-200 ng |
| Wasser-, Molekular-Grade | bis 20 ul |
Tabelle 1.
| Schritt | Temp. | Zeit | Cycles |
| Initiale Denaturierung | 95 ° C | 5 min. | 1 |
| Denaturierung | 95 ° C | 30 sek. | pan = "3"> 40 Zyklen |
| Glühen * | 62 ° C | 30 sek. | |
| Erweiterung | 68 ° C | 2 min. | |
| Letzte Extension | 68 ° C | 5 min. | 1 |
| Halten | 4 ° C | Halten |
Tabelle 2.
* Temperatur kann variieren, aber wir haben die besten Amplifikationsprodukte mit Glüh-Temperaturen zwischen 53 und 64 ° C beobachtet
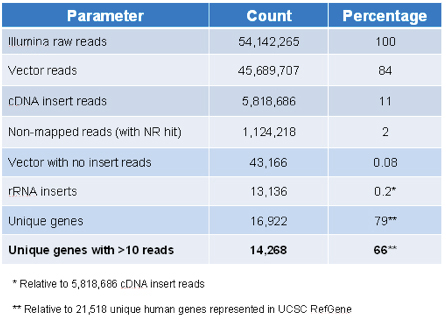
Tabelle 3. Ziel-ID-Bibliothek Inhalt von Illumina Sequenzierung.
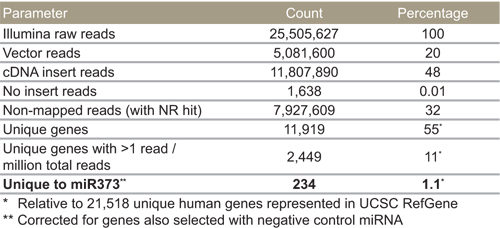
Tabelle 4. MiR-373 ausgewählte Ziele von Illumina Sequenzierung.
les/ftp_upload/3303/3303table5.jpg "alt =" Tabelle 5 "/>Tabelle 5. 10 Ziele zuvor von RNA Microarray identifiziert.
Diskussion
Mikro-RNAs sind 20-24 nt-RNAs die Genexpression regulieren posttranskriptional durch die Hemmung der mRNA-Translation und häufig, gezielte Destabilisierung der mRNA (Übersicht in [6]). Ein einzelner miRNA können regeln, mehrere hundert mRNAs einer Zelle Reaktion auf entwicklungs-und umweltpolitischen Signale steuern. Ermittlung und Validierung von Ziel-mRNAs ist von wesentlicher Bedeutung bei der Bestimmung einer miRNA die Rolle und Funktion in dieser Signalwege. Allerdings ist der Target-Identifizierung nicht einfach, weil, bei Tieren, miRNAs und deren Ziel-Seiten sind nicht vollständig komplementär. Der "Seed"-Region, Basen 2 bis 7 aus dem 5'-Ende der miRNA, ist in der Regel komplementär zu seinen Zielen. Allerdings gibt es viele Ausnahmen von der Regel Saatgut, und nachgelagerten Basenpaarung kann für eine unvollkommene Samen Match auszugleichen. Eine Reihe von Computer-Algorithmen wurden entwickelt, um miRNA-Targets auf Seed-Matching-und nachgelagerten Ausgleich, Zielstruktur basiert prognostizieren eind Position, Sequenzkonservierung, und verschiedene andere Parameter, die für experimentell validierten Targets (Übersicht in [7]) beobachtet wurden. Während in silico Vorhersagen bequem sind und nicht identifizieren viele gültige miRNA-Targets, die meisten Gene vorhergesagt ausfallen experimentelle Validierung Tests und vielen tatsächlichen Ziele sind nicht vorhersehbar. Da außerdem Computer-Algorithmen auf vorher bestimmten Ziel-Funktionen basieren, erlauben sie nicht Entdeckung von Zielen, die von dem, was bereits bekannt abweichen.
Eine Reihe von experimentellen Systemen wurden erfolgreich zu identifizieren oder zu entdecken funktionelle miRNA-Targets in lebenden Zellen eingesetzt. Diese globalen Screening-Methoden gehören Microarrays und RNA-Sequenzierung, RNA-Co-Immunopräzipitation (RIP) und Markierung mit stabilen Isotopen mit den Aminosäuren in der Zellkultur (SILAC), eine Proteomik-Verfahren (Übersicht in [7]). Jede Methode hat Vor-und Nachteile. Da miRNA-Targeting oft destabilisiert eine mRNA und führt zu dessen Abbau, Verlust or Gewinn an mRNA-Ebene nach der Vorstellung eines miRNA imitieren oder Inhibitor bzw. identifizieren können miRNA-Targets. Dieser Verlust oder Gewinn von mRNA wird leicht durch Microarray-oder tief-Sequenzierung nachgewiesen. Während mRNA Nachweismethoden einfacher und empfindlicher als Protein Nachweismethoden sind, wird mRNA-Nachweis verpassen keine miRNA-Targets, die nicht abgebaut werden. Jüngste Berichte aus dem Labor Bartell Vergleich miRNA Ziel ergibt sich aus RNA-Detektion mit denen aus SILAC [8] oder Ribosomen-Profiling [9] zeigen, dass miRNAs Säugetieren überwiegend durch Reduzierung Ziel-mRNA-Spiegel handeln. Allerdings haben viele andere Labore arbeiten mit einzelnen miRNA Targets Änderung Protein aber keine Änderung in der mRNA-Ebene nachgewiesen werden. Berichte von dem, was offenbar translationale Regulation ohne nachweisbare mRNA-Verlust sind: miR-10b auf HOXD10 [10], miR221/222 auf p27kip1 [11], miR-21 auf Pdcd4 [12], miR-126 auf p85β [13] , miR-34 auf SIRT1 [14], miR-21 auf PTEN [15], miR-302d auf Arid4b [16], miR-200c auf JAG1 [17], und miR-299, 297, 567, einemND 609 auf VEGFA [18]. Darüber hinaus Clancy et al [19] berichtete kürzlich, dass translationale Regulation von let-7 wurde nur erkannt, wenn einzelne mRNA-Isoformen, die die let-7-Ziel-Site enthalten, selektiv detektiert wurden. Letztere legt nahe, dass, zumindest in einigen Fällen, translationale Regulation in einem Verbundprofil übersehen werden - das heißt, wenn alle mRNA-Isoformen als eine mRNA nachgewiesen werden - wie bei vielen Mikroarray und Sequenzanalysen erhalten. Co-Immunpräzipitation von miRNA-mRNA-Komplexe über Argonaut (in der Regel Ago2) oder einem anderen assoziierten Protein (RNA Immunpräzipitation, oder RIP) wird unabhängig von miRNA-Targets Regulationsmechanismus zu isolieren. Darüber hinaus erkennt RIP endogenen und vermutlich biologisch relevante Wechselwirkungen. Es ist jedoch nicht bekannt, ob alle miRNA-mRNA Interaktionen funktionsfähig sind, und würde keine RIP-mRNA-Ziele, die nur vorübergehend zu verbinden oder auch schnell abgebaut zu verpassen. Darüber hinaus müssen spezifische miRNA-mRNA-Partnern geschlossen werden bioinformatitisch, weil alle miRNA-mRNA-Paare kogefällten zusammen. Schließlich SILAC direkt identifiziert das Endprodukt der miRNA-Verordnung, das Protein selbst, ist aber unempfindlich und daher verfehlt selten Proteinen und kleinen Falte Veränderungen im Protein-Spiegel.
Vor dem Hintergrund der aktuellen Einschränkungen mit miRNA Target-Identifizierung Methoden, fühlten wir uns eine zusätzliche globale Test auf funktionelle miRNA Targets durch einen alternativen Mechanismus identifizieren, benötigt wurde. Um diesem Bedarf gerecht zu werden, lizenziert wir eine Technologie, die von Joop Gaken und Azim Mohamedali von King 's College London erfunden. Die Erfindung ist ein Dual-Auswahl-Fusionsprotein, insbesondere ein Thymidinkinase-Zeocin-Fusion, reguliert von einem cDNA-Bibliothek potentieller miRNA-Zielsequenzen in der 3'UTR. Zellen, die stabil mit und exprimieren TKzeo-cDNA transfiziert mit Zeocin ausgewählt werden, wie in 2 dargestellt. Nach dem Ausdruck einer miRNA von Interesse, kann, dass miRNA-Ziele ausgewählt werden, with Ganciclovir, wie in Abb. 3 dargestellt. Ganciclovir tötet Zellen, die Thymidinkinase, das heißt, alle Zellen wird TKzeo-cDNA exprimieren, fehlt ein Ziel für die miRNA von Interesse. Gezielte cDNA durch PCR-Amplifikation von DNA aus Zellen, die Ganciclovir unter Verwendung von Primern, die Flanke der cDNA, PCR-Produkte sequenziert und zur Identifizierung von Zielen kann überleben isoliert werden.
Missionsziel ID-Bibliothek - Wir haben die King 's College-Technologie in ein neues Tool für die globale Identifikation und Entdeckung der funktionellen humanen miRNA-Targets entwickelt. Mit dem Ziel-ID-Bibliothek kann der Benutzer Zieltranskripte durch eine Reihe von Säugerzellen Transfektion und Arzneimittelselektion Schritte zu isolieren. Die Bibliothek ist umfangreich und enthält 66-79% der menschlichen Gene. Erste Ergebnisse zeigen, dass beide zuvor entdeckt sowie neue Ziele können von der Mission Ziel-ID-Bibliothek isoliert werden.
Obwohl das Protokoll ist von einiger Länge,Nutzung der Ziel-ID-Bibliothek erfordert Standard molekulargenetische Labor Techniken - Säugetier-Zellkultur, Transfektion, Medikamentenauswahl, PCR, Sequenzierung und - und sollte daher gut sein, im Rahmen der Möglichkeiten der meisten Biologen. Wir vermuten, dass genügend Aufmerksamkeit, um die ordnungsgemäße experimentelles Design bezahlt werden, die Optimierung der Kulturbedingungen, und am wichtigsten, Pre-Test jedes einzelnen neuen Zelltyp, um eine optimale Wirkstoffspiegel zur Auswahl Schritte (kill-Kurven), um Erfolg mit der Ziel-ID-Bibliothek gewährleisten zu bestimmen.
Wie bei allen Ziel-miRNA-Identifikations-Methoden, ist es sehr empfehlenswert, dass die Ziele durch Screening der Ziel-ID identifiziert Bibliothek mit einer zweiten Methode, wie Microarray, qRT-PCR, Reporter-Assay (dh Luciferase) oder Western-Analyse bestätigt werden.
Offenlegungen
Autoren erklären, finanziellen Beziehungen zu Wirtschaftsunternehmen, die ein Interesse an der eingereichten Arbeit zu haben.
Danksagungen
Wir danken der gesamten Sigma Life Science Mission Ziel-ID-Bibliothek Entwickler-Team, vor allem Kevin Gutshall für das Finden der Technologie und Verhandlung von Lizenz-und Vertragsbedingungen und Heather Holemon für ihre Unterstützung und Teilnahme an fruchtbaren Diskussionen zur Fehlerbehebung. Wir danken auch Dr. Joop Gäken von King 's College London für den Austausch frei unveröffentlichte Ergebnisse und Anregungen, und Qazi Hamid von RxBiosciences zur Herstellung einer großen Charge von cDNA und seine Beharrlichkeit in immer es geklont. Wir möchten Nan Lin und Scott Bahr aus SAFC anerkennen, zur Durchführung von cDNA-Arrays und Datenanalyse.
MISSION ist ein eingetragenes Warenzeichen von Sigma-Aldrich Biotechnology LP
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Zahl | Kommentare |
| JumpStart REDTaq ReadyMix | Sigma-Aldrich | P0982 | |
| Ganciclovir | Sigma-Aldrich | G2536 | |
| G418 | Sigma-Aldrich | G8168 | |
| Puromycin | Sigma-Aldrich | P9620 | |
| TOPO TA | Invitrogen | KNM4500-01 | |
| GenElute genomische Säuger-DNA-Miniprep-Kit | Sigma-Aldrich | G1N10 | |
| Zellspezifische Nucleofection kes | Lonza | ||
| Nucleofector | Lonza | AAD-1001 | |
| Zeocin | Invitrogen | R250-01 | |
| Ziel-ID-Bibliothek | Sigma-Aldrich | MREH01 |
Referenzen
- Huang, Q., Gumireddy, K., Schrier, M. The microRNAs miR-373 and miR-520c promote tumour invasion and metastasis. Nat. Cell Biol. 10 (2), 202-210 (2008).
- Lichner, Z., Pall, E., Kerekes, A. The miR-290-295 cluster promotes pluripotency maintenance by regulating cell cycle phase distribution in mouse embryonic stem cells. Differentiation. 81 (1), 11-24 (2011).
- Subramanyam, D., Lamouille, S., Judson, R. L. Multiple targets of miR-302 and miR-372 promote reprogramming of human fibroblasts to induced pluripotent stem cells. Nat. Biotechnol. 29 (5), 443-448 (2001).
- Lim, L. P., Lau, N. C., Garrett-Engele, P. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 433 (7027), 769-773 (2005).
- Huntzinger, E., Izaurralde, E. Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet. 12 (2), 99-110 (2011).
- Thomas, M. Desperately seeking microRNA targets. Nat. Struc. Mol. Biol. 17 (10), 1169-1174 (2010).
- Baek, D. The impact of microRNAs on protein output. Nature. 455 (7209), 71-71 (2008).
- Guo, H. Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature. 466 (3708), 835-840 (2010).
- Ma, L. Tumour invasion and metastasis initiated my microRNA-10b in breast cancer. Nature. 449 (7163), 682-688 (2007).
- Visone, R., et al. MicroRNAs miR-221 and miR-222, both overexpressed in human thyroid papillary carcinomas, regulate p27Kip1 protein levels and cell cycle. Endocrine-Related Cancer. 14 (3), 791-798 (2007).
- Lu, Z., et al. MicroRNA-21 promotes cell transformation by targeting the programmed cell death 4 gene. Oncogene. 27 (31), 4373-4379 (2008).
- Guo, C., et al. The noncoding RNA, miR-126, suppresses the growth of neoplastic cells by targeting phosphatidylinositol 3-kinase signaling and is frequently lost in colon cancers. Genes Chrom. Cancer. 47 (11), 939-946 (2008).
- Yamakuchi, M., et al. miR-34a repression of SIRT1 regulates apoptosis. Proc. Natl. Acad. Sci. U.S.A. 105 (36), 13421-13426 (2008).
- Wickramasinghe, N. S., et al. Estradiol downregulates miR-21 expression and increases miR-21 target gene expression in MCF-7 breast cancer cells. Nucleic Acid Res. 37 (8), 2584-2595 .
- Ciaudo, C., et al. Highly dynamic and sex-specific expression of microRNAs during early ES cell differentiation. PLoS Genetics. 5, e1000620 (2009).
- Vallejo, D. M., et al. Targeting Notch signaling by the conserved miR-8/200 microRNA family in development and cancer cells. EMBO J. 30 (4), 756-769 (2011).
- Jafarifar, F., et al. Repression of VEGFA by CA-rich element-binding microRNAs is modulated by hnRNAP. L. EMBO J. 30 (7), 1324-1334 (2011).
- Clancy, J. L., et al. mRNA isoform diversity can obscure detection of miRNA-mediated control of translation. RNA. 17 (6), 1025-1033 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten