Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Analyse der Trunk Neural Crest Cell Migration mit Hilfe eines modifizierten Zigmond Chamber Assay
In diesem Artikel
Zusammenfassung
Ein Ansatz, um die Migration von explantierten Zellen (Stamm Neuralleistenzellen) analysieren beschrieben. Dieses Verfahren ist kostengünstig, leicht, und in der Lage zu unterscheiden Chemotaxis sowohl Chemokinese und andere Einflüsse auf wandernde Polarität wie die von Zell-Zell-Wechselwirkungen innerhalb des primären Stamm Neuralleiste Zellkultur abgeleitet.
Zusammenfassung
Neuralleistenzellen (NCC) ist eine transiente Population von Zellen, die in Entwicklung von Wirbeltieren, dass aus dem dorsalen Neuralrohr (NT) auswandern nach Durchlaufen eines epithelial-mesenchymale Transition 1,2. Nach EMT, migrieren NCCs große Entfernungen entlang stereotypes Wege, bis sie ihre Ziele zu erreichen. NCCs differenzieren sich in einer Vielzahl von Zelltypen, einschließlich Neuronen, Glia, Melanozyten und Chromaffinzellen 1-3. Die Fähigkeit von NCCs zu erreichen und erkennen ihre richtigen Zielregionen für die entsprechende Ausbildung aller Strukturen mit trunk NCC-abgeleiteten Komponenten 3 grundlegend. Aufklärung der Mechanismen von Leitlinien für Trunk NCC Migration hat deshalb eine Angelegenheit von großer Bedeutung. Zahlreiche Moleküle nachgewiesen, dass NCC Migration 4 führen. Beispielsweise werden Trunk NCCs bekanntermaßen von negativen Führung Zeitgeber wie Semaphorin, Ephrin und Slit Liganden 5-8 abgestoßen werden. Allerdings sind nichtbis vor kurzem keine Lockstoffe des Rumpfes NCCs identifiziert worden 9.
Herkömmliche Ansätze zur in vitro Untersuchung der chemotaktischen Verhalten von adhärenten Zellen funktionieren am besten mit immortalisierten, homogen verteilt Zellen, sind jedoch schwieriger zu bestimmten primären Stammzellkulturen die zunächst fehlt eine homogene Verteilung und rasch zu unterscheiden (wie NCCs) anzuwenden. Ein Ansatz, um die Verteilung der Stamm NCCs für Chemotaxisstudien homogenisieren ist die Trunk NCCs aus primären NT Explantatkulturen isolieren, dann heben und replate ihnen zu fast 100% konfluent. Dies erfordert jedoch plating Ansatz erhebliche Mengen an Zeit und Mühe, um genügend Zellen Explantation, ist hart und vertreibt trunk NCCs in einem ungleichen Weise wie in in vivo-Bedingungen gefunden.
Hier berichten wir über eine in vitro Ansatz, der in der Lage, Chemotaxis und andere wandernde Reaktionen des Rumpfes NCCs ohne requirin bewerten istga homogene Zellverteilung. Diese Technik nutzt Zeitraffer-Aufnahmen von primären, unbeirrt trunk NCCs in einem modifizierten Zigmond Kammer (ein Standard Zigmond Kammer wird an anderer Stelle 10 beschrieben). Durch Belichten Trunk NCCs am Umfang der Kultur auf eine chemotactant Gradienten, die senkrecht zu ihrer vorhergesagten natürliche Richtung, können Veränderungen in wandernden Polarität durch die angelegte chemotactant Gradienten induzierte detektiert werden. Diese Technik ist kostengünstig, erfordert das Züchten von nur zwei NT je Wiederholung Explantate Behandlung vermeidet rauen Zelle Anheben (wie Trypsinisierung) verlässt Trunk NCCs in eine ähnliche Verteilung in vivo-Bedingungen, verkürzt sich die Zeitspanne zwischen Explantation und Experimente (was wahrscheinlich verringert das Risiko der Differenzierung), und ermöglicht Zeitraffer Auswertung zahlreicher wandernden Merkmale.
Protokoll
Ein. Tag 1: Isolierung von Stamm neuronalen Rohre für Übernacht-Kultur auf Deckgläsern
- Inkubieren chick Eier für 56 h bei 38 ° C. Entfernen Sie die Eier aus der Inkubation, leicht besprühen sie mit 70% Ethanol und dann trocknen lassen. Die Eier öffnen sich in einem UV-sterilisiert Glasschale.
- Extrahieren Sie jede Embryo aus Dotter und legen Sie sie in chick Ringer. Tun Sie dies, indem zuerst um seine Blutinseln mit gebogenen Schere, dann mit stumpfen Pinzette, wählen Sie die Embryos von seinem extraembryonalen Membran und legen Sie sie in einem sterilen Petrischale mit chick Ringer-Lösung.
- Isolieren den Kofferraum jedes Embryos durch Trimmen überschüssige extraembryonalen Membranen sowie Schädel-, vagalen und sakralen axialen Ebenen mit einem Wolfram-Nadel (Abb. 1). Wählen Sie zunächst ca. 9 Embryonen, die zwischen den Stufen HH15-17 11 sind. Für Bühnen HH15 oder höher, bilden die Vorderhirns und Rautenhirn Achsen einen spitzen Winkel und damit der Kopf scheint kaudal kippen. BeiBühne HH17, der Schwanz ist bud Gegenwart und kippt ventral aber noch nicht enthalten Somiten. Mit einer Wolfram-Nadel, schneiden Sie extraembryonalen Membranen bis etwa 2 mm aus dem Embryo und schneiden Sie embryonalen Geweben vorderen bis 10 Somiten. Entfernen Sie auch alle caudal embryonalen Geweben ab etwa dem fünften meisten neu gebildeten Somiten.
- Legen Sie die isolierten embryonalen Stämme in Dispase (0,24 U / ml DMEM) und inkubieren sie für 1 h 15 min bei 37 ° C und 5% CO 2. Sobald die Stämme der Inkubation beginnen, starten Herstellung von 6 Deckgläser (CS) für die Kultivierung NT Explantate (Schritte von 1,5 bis 1,8).
- Spülen 6 CS in 70% Ethanol (verdünnt in sterile, Reinstwasser) und dann trocknen lassen. Mit einem Labor-Marker, einen Kreis in der Mitte jeder CS, die etwa 1 cm im Durchmesser (dieser Kreis wird später helfen, herauszufinden, wo ein Fibronektin Mantel angewendet wurde) ist. Auf der gleichen Seite jedes CS, schreibt das Wort "be" (oder eine andere asymmetrische Wort oder Form) außerhalb des gezeichneten Kreis (dies y helfenou ermitteln, ob die markierte Seite des CS nach oben oder unten).
- Platzieren Sie jede CS in einem separaten 40 x 10 mm sterile Schale mit der markierten Fläche nach unten und lassen Sie das Gericht zu sitzen unter einem keimtötende UV-Lampe für 10 min geöffnet.
- Anwenden 60 ul Fibronektin (FN; 10 ug / ml DMEM) mit dem unmarkierten Oberfläche des CS gleichzeitig dafür der gesamte Bereich innerhalb des Kreises 1 cm beschichtet wird. Die Schalen werden bei 37 ° C für 30 min inkubieren und dann sorgfältig absaugen Fibronektin aus jedem CS.
- Eintragen von 250 ul "Kultur"-Medium [DMEM mit L-Glutamin (2 mM), Penicillin (100U/ml), Streptomycin (100 mcg / ml), 8% fetales Rinderserum (FBS)] zu dem FN-beschichtete Fläche die CS. Die Schalen werden mit jeder CS bei 37 ° C und 5% CO 2 wieder, bis die NTs isoliert wurden.
- Übertragen Sie alle bebrüteten Embryo Stämme zu einem 5 cm Glas-Petrischale mit L15 Medium und starten Sezieren aus jeder NT mit feinen Pinzette und eine Wolfram-Nadel (Abb. 1). Ct ein sorgfältig entlang der Grenze des NT und Somiten scheibe mit einem scharfen Wolframnadel während es nicht auf die vorsichtige NT beschädigen. Es ist oft einfacher zu beginnen Isolieren jeder NT aus dem kaudalen fast Ende des Rumpfes.
- Wählen Sie 6 der geradesten und längste NTs die Kultur über Nacht (NTs zwischen etwa 8 und 15 Somiten lange werden empfohlen). Verwendung einer Mikropipettenspitze mit Kulturmedium grundiert übertragen jeder der 6 NTs seinen eigenen zuvor hergestellten CS (ab Schritte 1.5 bis 1.8). Achten Sie darauf, dass das NT bleibt nicht an der Oberfläche schwimmen. Wenn das NT schwebt, tropft Medium auf ihn, bis er mit einer Mikropipette sinkt.
- Platzieren Sie jede Schale bei 37 ° C und 5% CO 2 über Nacht. Vorsichtig sein, um sicherzustellen, dass jedes NT ist innerhalb der FN-beschichteten Bereich seiner jeweiligen CS unmittelbar vor dem Platzieren der Schale in den Inkubator (mithilfe des Kreises im Schritt 1.5 als Referenz gezeichnet). Ein Mikropipette kann zur besseren Einstellung der Position jedes NT, wenn nötig.
- Place atmindestens 2 ml Kultur-Medium (ohne Serum) in ein steriles 15 ml Zentrifugenröhrchen eingewogen und bei 37 ° C und 5% CO 2 über Nacht. Lassen Sie die Kappe leicht abgeschraubt, um den pH-Wert des Mediums über Nacht anpassen können. Pre-Inkubieren des Mediums ist wichtig, um zu verhindern Blasenbildung in deine Kammer, die die Einrichtung eines molekularen Gradienten stören können. Solche "vorinkubierten" Medium sollte bei allen künftigen Maßnahmen verwendet werden. Wenn nicht verwendet wird, sollte dieses Medium bei 37 werden Inkubation ° C.
2. Tag 2: Laden des modifizierten Zigmond Kammer und Zeitraffer-Analyse der Zellmigration
- Von den 6 NTs kultiviert, wählen Sie die 3 Kulturen am besten für die Analyse geeignet. Im Allgemeinen sollte NCC Kulturen, die mindestens eine lange, gerade Kante haben, ausgewählt (2A) ist. Die 3 besten Kulturen zum Laden und Film 3 geändert Zigmond Kammern den ganzen Tag, jeder mit einer anderen Behandlung. Von den 3 Kulturen, wählen Sie ein für loading die erste Kammer und händigt die übrigen in den Inkubator für die spätere Verwendung.
- Mit einem Wattestäbchen, eine dünne, gleichmäßige Schicht Vaseline rund um die Stauseen und Brücke ein modifiziertes Zigmond Kammer.
- Mit einem Wolframnadel, entfernen die vom NT CS, während er die umgebenden NCCs an der Oberfläche des CS. Markieren Sie die Schale mit einem Stift, um die Ausrichtung der geradesten Rand des NCC Kultur erinnern.
- Legen Sie ein paar Tropfen vorinkubierten Medium auf der Brücke. Nehmen Sie den CS mit feinen Pinzetten, dab die Kante der CS gegen einen Kimwipe die meisten der alten Kulturmedium zu entfernen, dann sofort den CS auf der modifizierten Zigmond Kammer, so dass die gerade Kante der Kultur gefilmt werden über zentriert ist die Länge der Brücke und etwa senkrecht zu der Brücke-Reservoir Rand (2A, B).
- Mit einem inversen Mikroskop, bewegen Sie den geraden NCC Grenze auf der Seite der Brücke am nächsten th werdene Reservoir, das den mutmaßlichen chemotactant enthalten wird (Abb. 2B; für die Kontrollen wird dies auf den niedrigeren Reservoir geladen Sekunden entsprechen). Auch feiner auszurichten die gerade Kante der Kultur auf senkrecht zur Brücke-Reservoir Grenze sein.
- Sorgfältig, aber sicheren drücken CS in die Vaseline auf dem Zigmond Kammer ein, dass sie vollständig auf die Kammer versiegelt, dann stellen zusätzliche Vaseline entlang der Kante des CS weiter sicherzustellen, dass es luftdicht sein. Feineinstellung des Winkels des NCC Grenze wieder für jede Bewegung beim Siegeln zu korrigieren.
- Legen Sie den Behälter, die nicht enthalten, werden die vermuteten chemotactant ersten (Abb. 2B). Tun Sie dies, indem er ein 1-ml-Spritze (25 G x 1,5 in. Kanüle) mit rund 300 ul vorinkubierten mittel-und injizieren das Medium in den Behälter bis zur vollständigen (dabei darauf achten, dass keine Luftblasen in den Behälter zu erzeugen). Stecken des Reservoirs auf beiden Seiten mit einer ausr.iziente Menge Vaseline vor dem Laden des nächsten Behälter.
- Wiederholen Sie Schritt 2,7, außer dieser Zeit mit vorinkubierten Medium mit den Kandidaten chemotactant. Es ist wichtig, bei der Erzeugung eines molekularen Gradienten über die Kultur immer laden das Reservoir enthält das Molekül nach dem Laden das Reservoir fehlt den getesteten Molekül getestet werden.
- Setzen Sie den geladenen Zigmond Kammer bei 37 ° C für 1 h vor den Dreharbeiten zu inkubieren. Bild der geradeste Rand des NCC Kultur für 3 h bei 90 s-Intervallen während der Inkubation bei etwa 37 ° C (Abb. 2A, B). Vor dem Erstellen jeden Film ist, lesen Sie die Kamera so ausrichten, dass der Rand der Bilder zu erhalten und mit berühren den Rand der Brücke, die die letzte Reservoir geladenen grenzt (2B, obere Platte ausgerichtet sind; gestrichelten Kasten stellt ideale Position für imaging). Dies wird später Softwareanalyse durch Standardisieren die Direktionalität des molekularen angelegt, und th erleichterne Abstand von dem Reservoir in jedem Film gedreht hergestellt.
- Für Steuerelemente, die Schritte 2,2-2,9 für jede der zwei anderen Kulturen NCC ausgewählte (Schritt 2.1), aber erfüllen jedes Reservoir mit einem geeigneten Medium. Für eine Regelungsart füllen beide Reservoire mit vorinkubierten Medium ohne das Molekül getestet werden. Für eine zweite Kontrolle der Behandlung, prime die Brücke mit ein paar Tropfen vorinkubierten Medium mit dem Verdacht auf chemotactant vor der Montage der CS. Dann laden beide Reservoire mit dem gleichen Medium, enthaltend den mutmaßlichen chemotactant.
- Verwenden ImageJ (NIH) Manuelle Spur (rsb.info.nih.gov / ij / plugins / Track / track.html) und Chemotaxis und Migration Tool v1.01 (www.ibidi.de / applications / ap_chemo.html) plugins zu verfolgen die Migration von peripheren NCCs entlang der geraden Rand der Kultur just abgebildet und auf verschiedene Parameter der wandernden Trajektorien erhalten (2B-C) zu analysieren.
3. Repräsentative Ergebnisse:
Eine Probe von Zellen aus einem Film Trajektorien wo viele Stamm NCCs waren auf ein Kandidaten-chemoattraktive Verwendung des oben gezeigten Technik wird (2D). Die meisten Zellen in diesem Beispiel einer positiven Antwort erscheint eine Netto-Bewegung bis der Lockstoff-Gradienten (in Rot dargestellt). Trajektoriendaten verwendet, um andere Eigenschaften der Zellwanderung und analysieren.
Um visuell auswerten einer angelegten Gradienten in einer modifizierten Zigmond Kammer, eine Alexa Fluor 488 IgM-Konjugat (MW ~ 900 kDa) wurde in das zweite Reservoir eines modifizierten Zigmond Kammer (bei etwa 40 pg / ml H 2 O) geladen. Ein Gradient wurde durch 1 h festgelegt und noch etwas vorliegenden nach 26 h, aber stark von 50 h vermindert (Abb. 3). Wenn das Molekül zu testenden kleiner ist, dann wird die angewandte Gradienten will schneller abbauen als das, was angezeigt wird.

Abbildung 1. Explantation der Stamm-Ebene NTs über Nacht Kultivierung auf Fibronektin-beschichteten Deckgläschen. Weil Stamm NCCs delaminieren aus dem dorsalen NT befindet sich neben Somiten 8-28 wird dieses Segment des NT durch Mikrodissektion und über Nacht auf einem fibronektinbeschichtete CS für Auswanderung von NCCs aus dem NT Explantat ermöglichen isoliert. Isolated NTs, die zwischen 8-15 Somiten langen und relativ gerade sind am besten über Nacht Kultivierung geeignet, da sie an NCC Kulturen mit längeren geraden Grenzen ergeben neigen. Regionen des Neuralrohrs, die zu anderen Neuralleiste axialen Ebenen geben in einer kleineren Schrift dargestellt. s, Somiten.

Abbildung 2. Verfahren zur Auswertung der Migration von explantierten Trunk NCCsmit einem modifizierten Zigmond Kammer. (A) langgestreckten Rumpfes NCC Kulturen werden über Nacht kultiviert und von NTs resultierende NCC Kulturen mit wenigstens einem langen geraden Rand hergestellt werden für das Experiment ausgewählt. Die längste gerade Rand eines ausgewählten Kultur wird dann senkrecht zu der Brücke-Reservoir Rand positioniert ist, und somit parallel zum Vektor der Zukunft angewendet Gradienten. (B) Nach Feineinstellung der Position des NCC Kultur auf der Kammer und Abdichten Zigmond das Deckglas auf der Kammer, wird die Kammer geladen. Beim Testen Chemotaxis, das Reservoir, der nicht enthalten, werden die verdächtige chemotactant (-) geladen ersten und versiegelt. Dann wird das Reservoir mit dem anderen mutmaßlichen chemotactant (+) und versiegelten geladen. Peripheral NCCs entlang der zuvor ausgewählten Grenze kann dann abgebildet und verfolgt werden mit der manuellen Tracking-Plugin für ImageJ (unten). (C) Zahlreiche Zugvögel Eigenschaften als Reaktionum die aufgebrachte Gradient kann basierend auf Tracking-Daten beurteilt werden. Zum Beispiel kann ein Chemotaxis-Index durch Teilen einer Zelle Verschiebung entlang der x-Achse um die Gesamtdistanz es gewandert ableiten. (D) Ein Beispiel eines attraktiven Reaktion durch eine Zelle Trajektorie Plot ursprünglich von der Chemotaxis und Migration erzeugt wird gezeigt Tool Plugin für ImageJ. Der Ausgangspunkt jeder Trajektorie mit dem Ursprung (0,0) eingestellt. Man beachte, wie viel mehr Zellen in Richtung der Quelle anschließen chemoattraktive Massezentrum aller Zellen auf ihre endgültige Position zu migrieren (blaues Kreuz; alle Zellen gleich gewichtet) ebenfalls näher zu dem Chemoattractant Quelle. NCCs, Neuralleistenzellen; Red Spuren, Zellen, die zum Vorratsbehälter geladen mit migriert ein vermuteter chemoattraktive; Schwarz Spuren, Zellen, die auswärts migriert, (+), höhere chemotactant Konzentration; (-), niederes chemotactant Konzentration.

Abbildung 3. Intensitätsprofile über die Brücke eines modifizierten Zigmond Kammer zu verschiedenen Zeitpunkten nach der Zugabe eines Alexa Fluor 488 IgM Konjugat ist. Die Kammer wurde in ähnlicher Weise wie in dem Protokoll mit den wichtigsten Ausnahmen beschrieben geladen daß vorinkubierten Wasser (statt vorinkubierten Medium) benutzt, um den Antikörper zu 40 pg / ml zu verdünnen, und kleine Lufteinschlüsse waren an den Enden der Brücke (weg von der Scheibe, wo die oben Intensitätsprofile wo angenommen) vorhanden ist. Anfangs war kein Gefälle vorhanden in den meisten der Brücke. Mit 1 ha Gradient wurde gegründet und blieb bis 26 h vorhanden. Von 50 h die Anwesenheit des Gradienten inkonsistent zu unterschiedlichen Bereichen der Brücke war, und wenn vorhanden, wurde die Steilheit des Gradienten stark vermindert. Sämtliche Profile wurden von einer identischen scheibe über die Brücke (von einem brückenartigen Reservoir Rand zum anderen) mit AxioVision 4,6 Software generiert. Beachten Sie, dass auch während der LuftTaschen vorhanden waren, wurde die Steigung nicht gestört. Hohe, hohe Intensität, niedrig, niedriger Intensität, x-Achse, Abstand über die gesamte Breite der Brücke (2 mm), (+), mit dem Reservoir Alexa Fluor 488 IgM-Konjugat beladen; (-), Behälter nicht mit dem Konjugat beladen.
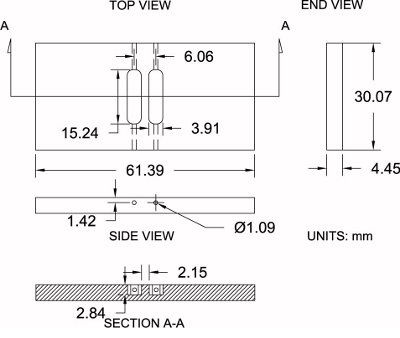
Abbildung 4. Modifizierte Zigmond Kammer Spezifikationen. Dargestellt ist ein Diagramm des modifizierten Zigmond Kammer welche hier zusammen mit seiner Maßangaben (± 0,2 mm). Die Messungen können moderat angepasst werden, um den individuellen Wünschen entsprechen.
Supplemental Protokoll: Herstellung eines Modified Zigmond Kammer
Bitte beachten Sie 4 als Referenz für das Protokoll in der folgenden Abbildung:
- Erwerben Sie ein Blatt 3/16 "dick poliert Acryl (4,45 mm tatsächliche Dicke).
- Mit einer Tischkreissäge, schneiden Kammer Rohlinge überdimensioniert, um diegroben Abmessungen von 33,25 mm x 64,57 mm. Dies ermöglicht 3,175 mm zusätzliches Material für die Bearbeitung.
- Die Kammer leer in einem Schraubstock. Mit einer Fräsmaschine und einer 6,35 mm (1/4 ") end mill bit, Fertigbearbeitung die Seiten der Kammer, um ihre genauen Abmessungen: 30,07 mm x 61,39 mm.
- Positionieren Sie die Kammer leer auf der Fräsmaschine und suchen Sie die Mitte des Rohlings entlang der x-und y-Achse mit einem Kantentaster, dann Null das Stadtzentrum.
- Erwerben die Kammer Höhe (z-Achse) durch Berühren des Schaftfräsers Bit auf der oberen Fläche und die Höhe Null.
- Verwendung einer 3,91 mm (0,154 ") Schaftfräser Bit, das Bit-Offset 3,03 mm entlang der x-Achse (positive Richtung) für das erste Reservoir. Beginnen Bearbeitung in die Kammer bis zu einer Tiefe von 2,84 mm während der Bewegung entlang der y-Achse (positive Richtung) bis 7,62 mm (0,300 ") und dann zu durchqueren, um 7,62 mm (0,300") in der entgegengesetzten (negativen) Richtung bis zu einem vollständigen Reservoir Länge von 15,24 mm (0,600 "). Offsetdruckdas Bit auf 3,03 mm (0,119 ") entlang der x-Achse (negative Richtung) und Wiederholen des gleichen Verfahrens für die zweite Reservoir.
- Positionieren der die Kammer an ihrem Rand und ein Loch mit einem 1,09 mm (0,043 Zoll) Bohrmeißel am Ende eines jeden Behälters (4 total), das das Ende des Reservoirs verbindet zu der Seite der Kammer für das Laden Medium während Experimentieren.
- Weichen Sie die Kammer auch in warmem Seifenwasser zu helfen, entfernen Sie keine chemischen Verunreinigungen.
- Spülen Sie die Kammer auch in doppelt destilliertem Wasser keine Seife entfernen. Die Kammern sind jetzt einsatzbereit wie oben beschrieben.
Diskussion
Dirigieren Chemotaxis Forschung am Stamm NCCs erwiesen Herausforderung für eine Reihe von Gründen. Trunk NCCs sind eine heterogene Stammzellen Bevölkerung unterscheiden, ob kultivierten langfristigen wird, daher trunk NCCs aus primären Explantation des Stamm-Ebene NT eingeholt werden muss. Herkömmliche Verfahren, die chemotaktische Reaktion von homogen verteilt Zellpopulationen in vitro zu untersuchen sind schwierig auf Trunk NCCs testen, da sie zuerst erforderlich, dass Zellen getrennt und homogen wiederp...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir geben besonderen Dank an Lino Kim, Steve Guzman und Ujit Satyarthi für die technische Unterstützung bei der Entwicklung dieser Methode. Myron Hawthorne, Richard Spengel und Roberto Rojas bearbeitet die Kammern verwendet hier und dringend benötigte technische Unterstützung. Bemerkenswert ist, produziert Roberto Rojas Abbildung 4. Wir sind auch dankbar für wertvolle Ratschläge Scott Fraser vor der Entwicklung der oben genannten Chemotaxis Assay. Diese Arbeit wurde teilweise durch eine NIH-MBRS SCORE-5S06GM048680-13 bis MEDB und durch eine Auszeichnung von der CSU, Northridge Diplomarbeit Support Program CW unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
| DMEM | Omega Scientific | DM-22 | |
| Penicillin Streptomycin-Lösung | Omega Scientific | PS-20 | 100X Lager Konzentration |
| L-Glutamin | Omega Scientific | GS-60 | 100X Lager Konzentration |
| Fötales Rinderserum | Omega Scientific | FB-11 | Lot # 105247 (oder eine andere, die vergleichbar ist) |
| Modified Zigmond Kammer | Hausgemachte | N / A | Behältervolumen: ~ 160 ul ea, für zusätzliche Spezifikationen, siehe Abb. 4 und die ergänzende Herstellung Protokoll |
| Zellkulturschale | Denville | T6040 | 40 x 10 mm |
| Fibronectin | BD | 354008 | 10X Lager vorbereitet durch Verdünnen von 1 mg FN in 1 ml H 2 O und 9 ml DMEM |
| Deckgläser | Fischer | 12-548-B | Vorgereinigt; 22 x 22 mm |
| L15 Medium | Thermo Scientific | SH30525.02 | |
| Petroleum Jelly | Comforts | 011110794642 | 100% |
| Zentrifugenröhrchen | Biologix | 10-9152 | 15 ml |
| Dispase | Cell Systems | 4Z0-850 | 10X Lager Konzentration |
| Spritze | BD | 309602 | 1 ml |
| Nadel | BD | 305127 | 25 G x 1,5 Zoll |
| Alexa Fluor 488-IgM | Ichnvitrogen | A21042 | Stock ist 2 mg / ml; 7 Mol Farbstoff / Mol IgM |
| Dissektionszangen | FST | Misc. | Dumont # 5 oder 55; geraden gekippt; Edelstahl oder Titan |
| Tungsten Needle | N / A | N / A | Hausgemachte; platziert in einem Stifthalter |
| Blunt Forceps | Tiemann | 160-18 | Wird für die Übertragung Embryonen Ringer aus Eigelb |
Supplemental Protokoll: Herstellung eines Modified Zigmond Kammer
Bitte beachten Sie 4 als Referenz für das Protokoll in der folgenden Abbildung:
- Erwerben Sie ein Blatt 3/16 "dick poliert Acryl (4,45 mm tatsächliche Dicke).
- Mit einer Tischkreissäge, schneiden Kammer Rohlinge überdimensioniert, um den groben Abmessungen von 33,25 mm x 64,57 mm. Dies ermöglicht 3,175 mm zusätzliches Material für die Bearbeitung.
- Die Kammer leer auf einer vise. Mit einer Fräsmaschine und einer 6,35 mm (1/4 ") end mill bit, Fertigbearbeitung die Seiten der Kammer, um ihre genauen Abmessungen: 30,07 mm x 61,39 mm.
- Positionieren Sie die Kammer leer auf der Fräsmaschine und suchen Sie die Mitte des Rohlings entlang der x-und y-Achse mit einem Kantentaster, dann Null das Stadtzentrum.
- Erwerben die Kammer Höhe (z-Achse) durch Berühren des Schaftfräsers Bit auf der oberen Fläche und die Höhe Null.
- Verwendung einer 3,91 mm (0,154 ") Schaftfräser Bit, das Bit-Offset 3,03 mm entlang der x-Achse (positive Richtung) für das erste Reservoir. Begin Bearbeitung in die Kammer bis zu einer Tiefe von 2,84 mm während der Bewegung entlang der y-Achse ( positive Richtung) bis 7,62 mm (0,300) ") und dann zu 7,62 mm (0,300 durchqueren" in der entgegengesetzten (negativen) Richtung bis zu einem vollständigen Reservoir Länge von 15,24 mm (0,600 "). Offset das Bit auf 3,03 mm (0,119 ") entlang der x-Achse (negative Richtung) und wiederholen Sie den Vorgang für das zweite Reservoir.
- Positionieren Sie die Kammer an ihrem Randund Bohren eines Loches mit einem 1,09 mm (0,043 Zoll) Bohrmeißel am Ende eines jeden Behälters (4 total), das das Ende des Reservoirs verbindet zu der Seite der Kammer für das Laden Medium während Experimentieren.
- Weichen Sie die Kammer auch in warmem Seifenwasser zu helfen, entfernen Sie keine chemischen Verunreinigungen.
- Spülen Sie die Kammer auch in doppelt destilliertem Wasser keine Seife entfernen. Die Kammern sind jetzt einsatzbereit wie oben beschrieben.
Referenzen
- Le Douarin, N. M. The avian embryo as a model to study the development of the neural crest: a long and still ongoing story. Mechanisms of Development. 121, 1089-1102 (2004).
- Baker, C. V. . Neural Crest and Cranial Ectodermal Placodes. , (2005).
- Gammill, L. S., Roffers-Agarwal, J. Division of labor during trunk neural crest development. Dev. Biol. 344, 555-565 (2010).
- Kulesa, P. M., Gammill, L. S. Neural crest migration: patterns, phases and signals. Dev. Biol. 344, 566-568 (2010).
- Wang, H. U., Anderson, D. J. Eph family transmembrane ligands can mediate repulsive guidance of trunk neural crest migration and motor axon outgrowth. Neuron. 18, 383-396 (1997).
- Krull, C. E. Interactions of Eph-related receptors and ligands confer rostrocaudal pattern to trunk neural crest migration. Curr. Biol. 7, 571-580 (1997).
- Gammill, L. S., Gonzalez, C., Gu, C., Bronner-Fraser, M. Guidance of trunk neural crest migration requires neuropilin 2/semaphorin 3F signaling. Development, Cambridge, England. , 133-199 (2006).
- De Bellard, M. E., Rao, Y., Bronner-Fraser, M. Dual function of Slit2 in repulsion and enhanced migration of trunk, but not vagal, neural crest cells. The Journal of cell biology. 162, 269-279 (2003).
- Kasemeier-Kulesa, J. C., McLennan, R., Romine, M. H., Kulesa, P. M., Lefcort, F. CXCR4 controls ventral migration of sympathetic precursor cells. J. Neurosci. 30, 13078-13088 (2010).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. The Journal of Cell Biology. 75, 606-616 (1977).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chicken embryo. J. Morph. 88, 49-52 (1951).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. The Journal of Experimental Medicine. 115, 453-466 (1962).
- Davis, E. M., Trinkaus, J. P. Significance of cell-to cell contacts for the directional movement of neural crest cells within a hydrated collagen lattice. Journal of Embryology and Experimental Morphology. 63, 29-51 (1981).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. Journal of Cell Science. 99, 769-775 (1991).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten