Method Article
Profiling Thiolredox Proteome Mit Isotopen-Massenspektrometrie Tagging
In diesem Artikel
Zusammenfassung
Reaktive Sauerstoffspezies Niveau angehoben wird, wenn Zellen begegnen Stressbedingungen. Hier zeigen wir das Beispiel von 3'-3 'Diaminobenzidin-Färbung sowie cysTMT Kennzeichnung und Massenspektrometrie, um die Redox-Proteom im Profil Pseudomonas syringae Behandelt Tomatenblättern.
Zusammenfassung
Pseudomonas syringae pv. Tomaten Stamm DC3000 verursacht nicht nur bakteriellen Krankheit in ipad Solanum lycopersicum sondern auch auf Brassica-Arten, ebenso wie von Arabidopsis thaliana, ein genetisch handhabbar Wirtspflanze 1,2. Die Akkumulation von reaktiven Sauerstoffspezies (ROS) in Kotyledonen mit DC3000 inokulierten deutet auf eine Rolle von ROS bei der Modulation der nekrotischen Zelltod während bakterielle Erkrankung der Speck Tomate 3. Wasserstoffperoxid, eine Komponente von ROS, wird nach der Inokulation von Tomatenpflanzen mit Pseudomonas 3 hergestellt. Wasserstoffperoxid kann unter Verwendung eines histochemische Färbung 3'-3 'Diaminobenzidin (DAB) 4 werden. DAB-Färbung mit Wasserstoffperoxid reagiert, um einen braunen Flecken auf der Blattgewebe 4 zu erzeugen. ROS hat eine regulatorische Rolle des zellulären Redox-Milieu, das den Redox-Status bestimmter Proteine 5 ändern können. Cystein ist eine wichtige Aminosäure, empfindlich gegenüberRedox-Änderungen. Unter milden Oxidation, dient reversible Oxidation von Cystein-Sulfhydrylgruppen als Redox-Sensoren und Signalgeber, die eine Vielzahl von physiologischen Prozessen 6,7 regulieren. Tandem-Massen-Tag (TMT) Reagenzien ermöglichen gleichzeitige Identifizierung und Quantifizierung von Proteinen Multiplex in verschiedenen Proben mittels Tandem-Massenspektrometrie 8,9. Die Cystein-reaktiven TMT (cysTMT) Reagenzien ermöglichen selektive Markierung und relative Quantifizierung von Cystein enthaltenden Peptiden von bis zu sechs biologischen Proben. Jede isobare cysTMT-Tag weist die gleiche nominale übergeordneten Masse und aus einem Sulfhydryl-reaktiven Gruppe, einem MS-neutralen Abstandhalterarm und einen MS / MS-Reporter 10 zusammengesetzt. Nach der Markierung wurden die Proben unter Proteaseverdau. Die Cystein-markierten Peptiden angereichert mit einem Harz, das Anti-TMT-Antikörper ist. Während MS / MS-Analyse, eine Reihe von Reporter-Ionen (dh, 126-131 Da) im Bereich geringer Masse ergeben, die Informationen über die relative Quantifizierung.Der Workflow ist wirksam zur Reduzierung der Komplexität Probe, die Verbesserung der Dynamik und dem Studium Cystein-Modifikationen. Hier haben wir Redox-Proteomanalyse der Pst DC3000 behandelt Tomaten präsentieren (Rio Grande) verlässt mit cysTMT Technologie. Diese High-Throughput-Verfahren hat das Potenzial, zu studieren andere Redox-regulierten physiologischen Prozesse angewendet werden.
Protokoll
1. Wachsende Sämlinge und Vorbereiten von Bakterien
- Sämlinge werden in Metromix 500 Boden-Gemisch (BWI Gesellschaften) in einer Klimakammer (160 pmol Photonen m -2 s -1 mit einer Photoperiode von 8 Stunden Licht bei 22 ° C gekeimt und 16 Stunden Dunkelheit bei 20 ° C für 1 Woche. Relative Luftfeuchtigkeit wurde auf 70% gesetzt. Sämlinge mit Leitungswasser bewässert wurden je nach Bedarf, um den Boden vor dem Austrocknen bewahren.
- Zwei Sämlinge von ähnlicher Größe werden dann 4 "Durchmesser Töpfe mit dem Metromix 500 Boden umgepflanzt und für weitere 3 Wochen in den gleichen Bedingungen wie 1.1.
- Pseudomonas syringae (PST) wird zunächst T-streifig auf Kings B Medium (KBM) Platten (pro Liter: 20 g Proteosepepton, 0,1975 g MgSO 4, 1% Glycerin, 1,5 g K 2 HPO 4, 18 g Agar) gewachsen und für zwei Tage bei 28 ° C Eine einzelne Kolonie wird gesammelt, in 300μl Flüssigkeit vermischt Kings B Medium (20 g Proteosepepton, 0,1975 g MgSO 4 , 10 ml Glycerin, 10 ml 100x K 2 HPO 4 Lager (1,5 g K 2 HPO 4 in 10 ml H 2 O), und die Ausbreitung auf ein Kings B Medium Platte. Dies wird über Nacht bei 28 ° C kultiviert
- Die Bakterien Inokulum wird durch Abkratzen der kultivierten Bakterien aus dem KBM Platte in 20 ml H 2 O. vorbereitet Eine OD 600 von 0,03 (~ 10 6 KBE / ml) wird in 1 l Impfung Puffer (10 mM MgCl 2 + 400 ul Silwett-70). Inokulationspuffer minus Bakterien wurde für die Kontroll-Lösung vorbereitet.
2. Inokulation von Tomaten mit Pst und H 2 O 2 Histochemische Stain
- Vier Wochen alten Pflanzen wurden durch Eintauchen in die oben Impflösung für 30 Sekunden inokuliert. Klare Plastiktüten wurden über die Pflanzen unmittelbar nach der Impfung gestellt.
- 3'-3 'Diaminobenzidin (DAB) zu färben 11 hergestellt wurde (1 mg / ml DAB in H 2 O, pH 3,8 (HCl)) drei days vor zu verwenden, um vollständige Solubilisierung zu erreichen. Die Lösung wurde gerockt, um bei Raumtemperatur zu mischen.
- Vierundzwanzig Stunden nach der Behandlung wurde der voll entfalteten Blätter entfernt, in DAB Fleck epidermalen Seite nach oben und Vakuum für 15 min infiltriert. Die Blätter wurden dann im Dunkeln bei Raumtemperatur über Nacht gelegt für die Färbung.
- Die Blätter wurden in 95% Ethanol für 10 min gekocht und dann gehalten in 75% Ethanol.
3. Protein Extraction
- Nach der Ernte wurden Tomatenblättern zu feinem Pulver mit flüssigem Stickstoff mit Mörser und Stößel zermahlen.
- Bei 0,5 g Gewebe, 1,25 ml hinzugefügt Tris pH 8,8 gepuffertem Phenol und 1,25 ml / 0,5 g Extraktionspuffer (0,1 M Tris-HCl pH 8,8, 10 mM EDTA, 0,9 M Saccharose), dann weiter Zerkleinerung für eine paar Minuten in Dunstabzug.
- Übertragen Sie das Extrakt zu Oakridge Zentrifugenröhrchen (Thermo Fisher Scientific Nalgene Company) und agitieren für 2 Stunden bei Raumtemperatur.
- Zentrifuge bei 5000 xgund 15 ° C für 10 min. Übertragen Sie die Top-Phenol-Phase in ein neues sauberes Röhrchen.
- Zurück zum Extrahieren der wässrigen Phase mit einem gleichen Volumen Phenol extrahiert Puffer; agitieren auf einem Schüttler für 30 min. Zentrifugieren und überweisen Sie den Extrakt auf ein neues Falcon-Röhrchen.
- Wiederholen Sie diesen Schritt noch einmal und überweisen Sie den Extrakt zu neuen Oakridge Rohr.
- Man fällt Phenol extrahiert Proteine durch Zugabe von 5 Volumina 0,1 M Ammoniumacetat in 100% Methanol (bei -20 ° C). Vortex und Inkubation bei -20 ° C zur Übernachtung.
- Sammeln der Protein-Pellet durch Zentrifugation bei 20.000 × g, 4 ° C für 20 min.
- Waschen des Pellets 2 Mal mit dem kalten 0,1 M Ammoniumacetat in Methanol und 2-mal mit 80% kaltem Aceton gewaschen. 1,5 ml kaltem 70% Ethanol zum Pellet-und aussetzen. Transfer zu einem 2 ml Mikrozentrifugenröhrchen (USA Scientific) und Zentrifuge bei 14.000 rpm, 4 ° C für 20 min. Ethanol entfernen.
- Trocknen Sie kurz das Pellet in einem SpeedVac Konzentrator und lösen sich in einem Protein extrMaßnahmen Puffer, zB ReadyPrep Protein Extraction Kit Reagenz 3 (8 M Harnstoff, 4% CHAPS, 40 mM Tris-Base, 2 M Thioharnstoff) (Bio-Rad). Zentrifuge bei 14.000 rpm und 20 ° C für 30 min, um ein Pellet zu bilden. Die überstehende Flüssigkeit.
- Messen Sie die Protein-Konzentration mit Hilfe eines CB-X-Protein-Assay nach Angaben des Herstellers manuell (Geno-Technologie).
4. Probenvorbereitung und Peptidmarkierung mit cysTMTs
- Planen Proteinprobe in einer Konzentration von 2-5 g / ul. Hier verwenden wir 100 yg Protein für jedes Masse-Tag, 20 ul-50 ul-Proben. Für dieses Experiment wurden alle 6-Tags verwendet wurden.
- In gleichen Volumen Alkylierung Puffer (100 mM Tris-HCl pH 7,5, 200 mM Iodacetamid) an das Protein Probe, um die freie Thiolgruppe zu blockieren. Buffer sollte frisch zubereitet werden. Alkylierung bei 37 ° C für 1 Stunde durchgeführt.
- Man fällt das Protein durch Zugabe von 1 ml kaltem 80% Aceton bei -20 ° C über Nacht. Pellet das Protein durch Zentrifugieren bei 14.000rpm, 4 ° C für 20 min. Waschen des Pellets 3 mal mit 80% Aceton Vortexen jeder Zeit. Entfernen Sie den Überstand und trocknen lassen, während auf Eis zu lüften.
- Das Pellet in 50 ul Lysepuffer (6 M Harnstoff, 50 mM Tris-Base, 1 mM EDTA). 0,5 ul 100 mM Tris (2-carboxyethyl) phosphin (TCEP) wird dann zugegeben, um die Disulfidbindungen durch Inkubieren für eine Stunde bei Raumtemperatur zu reduzieren.
- Bereiten Tags durch Zugabe von 20 ul von Acetonitril an die Reagenzgläser, um die Tags zu lösen. Vortex und Spin nach unten.
- Waschen Sie den zusätzlichen TCEP mit einem Microcon 3 kD liegen Zentrifugalfilter Gerät (Millipore). Dann werden 50 ul Probe auf Microcon 3 kD liegen Spalte und 50 ul Lysispuffer auf die Säule. Zentrifuge für 15 min bei 10.000 xg und 4 ° C. Wiederholen Sie diesen Schritt für insgesamt dreimal. Entfernen Sie die Spalte und schwenken in ein sauberes Röhrchen. Zentrifugieren Sie für 5 min bei 1.000 xg und 4 ° C.
- Prüfen Sie den pH-Wert. Falls erforderlich, den pH-Wert auf 7,0 bis 8,0 mit 1 M HCl.
- Fügen Sie 5 ul der cysTMT-Reagenz zu jeder Probe (Der cysTMT Reagenzien-Kit bietet 20 uL Tags für bis zu 500 ug Protein. Diese Methode verwendet 100 ug Protein, deshalb verwenden wir ein Fünftel des Tags.). Um die Reaktion für 2 Stunden bei Raumtemperatur ablaufen.
- Die Proben wurden mit Laemmli Probenpuffer (Bio-Rad) in einem Verhältnis von 1:1 kombiniert.
5. Entfernung von nicht reagierten Tag Probenfraktionierung
- Die Proben wurden 5 min gekocht und auf einem vorgefertigten 12% Polyacrylamidgel (Bio-Rad). Das Gel wurde dreimal in Intervallen von 5 Minuten mit gefilterter H 2 O gespült und mit Coomassie-Blue (Bio-Rad) für 1 Stunde. Das Gel wurde de-gefärbten über Nacht mit H 2 O.
- Zwölf Fraktionen wurden von jeder Gelspur gesammelt. Die Fraktionen wurden durch Protein-Bande-Konzentrationen bestimmt. Die Fraktionen wurden in 1 mm Stücke geschnitten und gesammelt in ein 1,5 ml Tube.
- Die Gelstücke wurden de-gefärbten unter Verwendung von 50% Acetonitril und 0,1 M Ammoniumbicarbonat in H 2 O. Dieser Schritt zu wiederholen, bis blaue Farbe Gelstücke (~ 3-4 mal, 15 min jeweils) entfernt wird.
- De-gefärbten Gel Stücke wurden getrocknet unter Verwendung von 100% Acetonitril (Cover Gelstücke) für 15 min, Überstand entfernt, und die Stücke getrocknet unter Verwendung einer SpeedVac.
- Man löst Trypsin (Promega) in 1:10 (Trypsin: Protein-Verhältnis) im gleichen Volumen (als gepoolte markierte Probe) von 25 mM Ammoniumhydrogencarbonat. Zum Beispiel muss 600 ug Protein-Probe mit einem Volumen von 500 ul 60 ug Trypsin in 500 ul 25 mM Ammoniumbicarbonat aufgelöst.
- Fügen Sie die Trypsin-Lösung für die Gel-Stücke und setzen auf Eis zu rehydrieren. Wenn die Stücke nicht abgedeckt werden mit 50 mM Ammoniumbicarbonat-Puffer. Nach Rehydratisierung bei 37 ° C für 12-16 h inkubiert.
- Entfernen Sie die Flüssigkeit Proteinextrakt aus den inkubierten Proben.
- Stoppen Sie die Reaktion, und entfernen Sie alle verbleibenden Proteinverdaus durch Abdecken Gelstückchen mit 5% Ameisensäure, 50% Acetonitril .. Schütteln Gelstücke für 20 min bei Raumtemperatur temperatur. Den Überstand in den extrahierten Protein Probenröhrchen von 5,7. Diese Extraktion zweimal. Trocknen Sie die extrahierten Peptide mit der SpeedVac.
6. Probenanreicherung von cysTMT markierter Peptide
- Fügen Sie ein Gefälle von Anti-TMT Harz bis 0,5 ml Röhrchen für eine 50% Gülle. (Gradient wurde von Band-Konzentration im Gel Fraktionierung bestimmt. Wenn ein Bruchteil Kollektion besteht aus einem dunklen Band, mehr Harz benötigt wird. Fraktionen mit schwächere Banden benötigen weniger Harz, da es weniger Protein.)
- Das Harz wird dann dreimal mit einem Säulenvolumen 1x Tris-gepufferter Salzlösung (TBS) (25 mM Tris, 0,15 M NaCl, pH 7,2) (Thermo Scientific Pierce Protein Research Products).
- Fügen Sie 200 ul 1xTBS zu jeder Probe. Die Proben werden dann auf Anti-TMT Harz (Thermo Scientific Pierce Protein Research Products) zugegeben und gerührt bei Raumtemperatur für 2 Stunden durch Schwenken über Nacht bei 4 ° C, gefolgt
- Probe in Spalte (dieRMO Scientific Pierce Protein Research Products).
- Waschen Sie jede Spalte dreimal mit 1x 200 ul TBS. Diese wird dann mit dreimaligem Waschen mit 0,05% CHAPS (gelöst in 1 × TBS) gefolgt.
- Die Säule wird dann dreimal mit 4 M Harnstoff in 1x TBS gewaschen. Zweihundert Mikroliter H 2 O wird dann verwendet, um die Säule dreimal waschen.
- Jede Probe wird dreimal mit 200 ul 50% Elutionspuffer (50% Acetonitril, 0,4% TFA) eluiert.
- Die Proben werden dann in einem Vakuum-Konzentrator getrocknet.
7. Massenspektrometrie-Analyse
- Resuspendieren Sie die Proben in 12 ul von 3% Acetonitril mit 0,1% Ameisensäure und injizieren 5 ul direkt auf ein Eksigent NanoLC-1D Hochdruck-Flüssigkeits-Chromatographie-Säule (AB SCIEX, USA).
- Peptide wird auf der Proteopep II C18-Säule 75 um ID getrennt werden x 20 cm (New Ziel, USA) unter Verwendung eines 4-60% Gradienten (A: 3% Acetonitril, 0,1% Ameisensäure, B: 97% Acetonitril, 0,1% Ameisensäure Säure)bei 3 ul / min über 60 min.
- Ein Thermo Scientific LTQ Orbitrap XL-Massenspektrometer wurde verwendet, um Peptide mit Hilfe eines Top-2 zu erfassen x 3 Experiment, bestehend aus einstufige MS gefolgt von Erwerb von 3 MS / MS-Spektren mit einer höheren Energie-C-Trap-Dissoziation (HCD) Fragmentierung von 3 MS gefolgt / MS mit Kollisions-induzierte Dissoziation (CID) für Protein-Identifizierung. Die Parameter in diesem Modus waren: Isolierung Breite: 3,0 m / z; Collision Energie: 50% (10% mit zwei Stufen). Nur doppelt-und dreifach geladene Peptide wurden zur Fragmentierung ausgewählt. Dynamische Ausschluss Parameter wurden eingestellt: Wiederholen count = 1; Wiederholen Dauer = 60; Ausschlussliste size = 500; Ausschluss Dauer = 28. Zielwerte sind wie folgt: MS = 5 e 5; MS / MS (HCD) = 1 e 5. Ion Transferzeiten wurden auf 500 und 300 für FTMS für MS / MS (HCD) eingestellt. Zwei microscans wurden für HCD-Spektren erforderlich.
8. Datenbank-Suche und Quantifizierung
- Die erworbenen CID und HCD Daten waren einnalyzed mit Thermo Scientific Proteome Discoverer-Software 1.2 (Thermo Scientific Pierce Protein Research Products) durch ein verzweigtes Workflow, der eine Reporter-Ionen-Quantizer (20 ppm Masse Toleranz von Fragmentionen) implementiert, um das Verhältnis zu quantifizieren. Ein eigenes Segment verarbeitet die MS 2-Spektren durch das Spektrum Selector, Spectrum Normalizer und Spectrum Grouper Knoten.
- Die Daten wurden dann gegen die SEQUEST Suchmaschine durchsucht. Ein 20 ppm Masse Toleranz verwendet wurde. Statische Änderungen wurden an der cysTMT Reagenziensatz (304,18 Da) und dynamische Änderungen enthalten Phosphorylierung und Methioninoxidation (15,99 Da). Eine benutzerdefinierte Datenbank für Solanum lycopersicum komponiert wurde unter Verwendung von RNA-Daten (mit über 350.000 Einträgen aus Harvard University) 13 wurde in beiden Fällen verwendet.
9. Repräsentative Ergebnisse
Ein repräsentatives Bild einer Kontrolle Tomatenpflanze Blatt und einer Pseudomonas-infizierten Blatt istin 1 gezeigt. Ein Unterschied zwischen Kontrolle behandelt und Pseudomonas behandelten Blätter zu beobachten ist. Nachdem die Blätter entfernt werden und gefärbt mit DAB, die de-Färbung erlaubt die histochemische Färbung auf Anzeichen von ROS im Blattgewebe (Abbildung 2) zeigen. 2A ist repräsentativ für eine Kontrolle Blatt ohne Färbung. 2B ist repräsentativ eines Blattes mit Pseudomonas und positive Färbung für H 2 O 2-Produktion behandelt. Ein Beispiel für Proteome Entdecken Datenausgang einer differentiell Redox regulierte Protein wird in 3 gezeigt. Dieses Protein ist ein Protein bekannten Redox-regulierten Ferredoxin-1-14 und ist gezeigt worden, um eine Rolle in der Verteidigung gegen Pseudomonas syringae pv tomato 15 zu spielen. Peak-Intensität zwischen Steuerung und inokuliert Proben wird zur relativen Quantifizierung, die wesentliche Änderungen in ferredox aufwies, zu erhaltenin-1-Redox-Regulation (p <0,05). Hochdruck-Peaks zeigen, dass dieses Protein in Reaktion auf das Pathogenbehandlung oxidiert wird. 4 ist ein Beispiel für Proteome Entdecken Datenausgang eines Proteins, das ähnliche Redoxregulation eines Proteins zwischen einem Steuerelement und inokulierte Probe aufweist. Peaks von ähnlicher Intensität auf das Vorliegen von Disulfidbindungen nicht durch eine Änderung in der Behandlung reguliert. Die Methode wird revolutionieren, wie Wissenschaftler Redox-Reaktion Cysteine und Disulfide 10 zu erfassen.

Abbildung 1. Ein repräsentatives Bild der inokulierten Tomatenblättern mit Control-Lösung (A) und Pseudomonas (B).

Abbildung 2. Ein repräsentatives Bild der DAB-Färbung der beimpften Tomatenblättern mit Control-Lösung (A) und Pseudomonas (B). Blätter wurden unter Verwendung 3'-3 'Diaminobenzidin. Chlorophyll wurde aus Blättern durch Kochen in 95% Ethanol entfernt. Dunkle Färbung zeigt die Anwesenheit von H 2 O 2. Nur verlässt geimpft mit Bakterien Kultur zeigte dunkle Färbung.
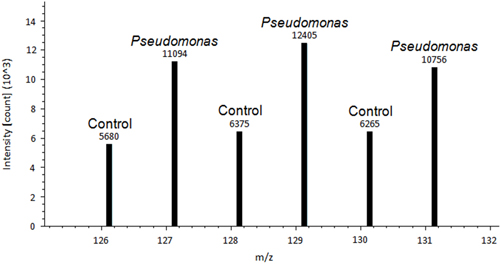
3. Ein Beispiel Proteome Entdecken Datenausgabe von Ferredoxin-1, differentiell Redox regulierte Protein 14. Peak-Intensität über jedem Peak wird zur absoluten Quantifizierung verwendet. Peak-Intensität zwischen Kontrolle und inokulierten Proben verwendet wird,um eine relative Quantifizierung durchführen.

4. Ein Beispiel Proteome Entdecken Datenausgang eines Proteins, das ähnliche Peak-Intensität zwischen einer Steuerung und inokulierte Probe aufweist. Peaks von ähnlicher Intensität auf das Vorliegen von Disulfidbindungen nicht durch eine Änderung in der Behandlung reguliert.
Diskussion
Dieses Protokoll gibt Auskunft über die Durchführung DAB-Färbung sowie cysTMT beschriftet Redox-Cystein-Quantifizierung. Diese Verfahren sind nützlich bei der Prüfung der Produktion von ROS sowie die Wirkung auf die Protein-Regulation bei Solanum lycopersicum mit Pseudomonas syringae wird geimpft. Die Methoden in diesem Protokoll vorgestellt bieten eine Möglichkeit, ROS in ganzen Blättern Proben untersuchen in einer Weise, die den geringsten Schaden an Blattgewebe verursacht. Das Markierungsverfahren bietet eine Möglichkeit, potentiell Redox regulierte Proteine unter Verwendung eines Cystein Markierungsverfahren untersuchen. Dies ist vorteilhaft, wenn die Prüfung eines frühen Stadium der Stressreaktion.
Verfahren wie Isotop-codierte Affinitäts-Tag (ICAT) und cysTMT in Prüfung potenzieller Redox regulierte Proteine in biologischen Proben verwendet werden. ICAT ermöglicht die Kennzeichnung und Vergleich von zwei Proben 12. Beide Methoden kennzeichnen freie Cysteine und kann für Protein quantific verwendet werdenation 10,12. Allerdings erlaubt die cysTMT Methode für einen Rückgang der experimentellen Variation sowie 10 Multiplexen. Die Anzahl der Tags zur Verfügung ermöglicht es den Forschern repliziert oder mehrere Proben in ihrem experimentellen Design gehören. Mit mehr Samples bietet das Potenzial für eine höhere Anzahl von Proteinen identifiziert. Ein großer Nachteil des cysTMT Technik ist, dass es die allgemeine Qualität von Protein-Identifizierung aufgrund der selektiven Anreicherung Schritte für cysTMT-markierten Peptiden (6,5-6,6) beeinträchtigt. Die Anzahl von Peptiden für die Identifizierung von Proteinen hängt weitgehend von der Anzahl der Cysteinreste in der Proteinsequenz. Dieses Problem kann erfolgen, indem ein Teil des tryptischen Probe vor der Anreicherung für die Massenspektrometrie Proteinidentifizierung überwunden werden.
Aufgrund der Natur des experimentellen Designs als auch die Markierung Mechanismus, dass der cysTMT Verfahren verwendet, bestimmte Schritte kritisch. Bei der Durchführung von Prä-Proteinpitation und Pellet Waschungen (3.9) ist es wichtig, auf Eis gekühlt, um Protein-Abbau zu reduzieren halten. Während cysTMT Kennzeichnung, ist die Entfernung des Reduktionsmittels (4,6) wichtig, weil Proben Reverse Kennzeichnung unterziehen können. Umgekehrt Kennzeichnung ist möglich, wenn Reduktionsmittel in der Probe zurückbleibt. Wenn die Proben nach der Markierung reduziert werden, kann die cysTMT-Tag entfernt werden. Wenn die Etiketten auf die Proben hinzugefügt, so muss der pH-Wert geprüft (4.7), um eine optimale Kennzeichnung Wirksamkeit besitzen. Darüber hinaus ist die Datenanalyse abhängig, was von dem Forscher und dem ultimativen Ziel im Umgang mit dem Protokoll erforderlich. Es ist auch abhängig von der verwendeten Software, da jede Software unterschiedliche Algorithmen.
Dieses Experiment verwendet Pathogen als Auslöser für eine verstärkte Produktion von reaktiven oxidativen Spezies in Tomaten, aber auch andere Redox geregelten Reaktionen kann entsprechend gemessen werden. Das experimentelle Design ist anpassbar an anderen pflanzlichen und tierischen Systemen.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren danken Dr. Greg Martin (Cornell University) und seine Gruppe für die Bereitstellung der DC3000 Stammes-, Tomaten-Samen, und Ratschläge danken. Sie möchte auch danken Dr. Zhonglin für Hilfe bei der DAB-Protokoll und der Proteomics Division bei UF Interdisziplinäres Zentrum für Biotechnologische Forschung für die Unterstützung bei der Methodenentwicklung Mou. Das Protokoll für Protein-Extraktion wurde von Tanaka Hurkman und 16 modifiziert. Das Protokoll über cysTMT Kennzeichnung, die Schritte 4 bis 6 wurde basierend auf dem ursprünglichen Thermo Fisher Scientific Pierce Produkthandbuch 17 angepasst. Diese Arbeit wurde vom National Science Foundation (MCB 0818051 bis S Chen) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| Metromix 500 | BWI Firmen | TX-500 | |
| 3,3 '-Diaminobenzidin | Sigma-Aldrich | D8001 | |
| ReadyPrep sequentielle Extraktion Kit Reagenz 3 | Bio-Rad | 163-2104 | |
| CB-X-Protein-Assay | Geno-Technologie | 786-12x | |
| cysTMT Reagenzien | Thermo Scientific Pierce Protein Research Products | 90071 | |
| Laemmli-Probenpuffer | Bio-Rad | 161-0737 | |
| Bio-Safe Coomassie (G-250-Färbung) | Bio-Rad | 161-0786 | |
| Microcon 3 kD liegen Spalte | Millipore | 42403 | |
| Immobilisierte anti-TMT-Harz | Thermo Scientific Pierce Protein Research Products | 90076 | |
| Zentrifugationssäule | Thermo Scientific Pierce Protein Research Products | 89896 | |
| Proteopep II C18-Säule | Neues Ziel | PFC7515-PP2-10 | |
| NanoLC-1D HPLC | AB Sciex | 90389 | |
| LTQ Orbitrap XL | Thermo Scientific | 0020137580 | |
| SpeedVac | Labconco | 7812013 | |
| Proteome Discoverer 1.2 Software | Thermo Scientific Pierce Protein Research Products | ||
| Trypsin | Promega | V5111 | |
| Oakridge Zentrifugenröhrchen | Thermo Scientific Nalgene Unternehmen | 3139-0050 | |
| Reaktionsgefäß (2ml) | USA Scientific | 1620-2700 | |
| 12% Mini-PROTEAN TGX Fertigteil-Gel | Bio-Rad | 456-1043 | |
| Top of Form > Bio-Safe Coomassie StainBottom der Form | Bio-Rad | 161-0786 | |
| TMT Bereicherung Kit | Thermo Scientific Pierce Protein Research Products | 90077 |
Referenzen
- Almeida, N. F. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Mol. Plant Microbe Interact. 22, 52-62 (2009).
- Preiter, K. Novel virulence gene of Pseudomonas syringae pv. tomato strain DC3000. J. Bacteriol. 187, 7805-7814 (2005).
- Apostol, I., Heinstein, P. F., Low, P. S. Rapid stimulation of an oxidative burst during elicitation of cultured plant cells: role in defense and signal transduction. Plant Physiol. 90, 109-116 (1989).
- Orozco-Cardenas, M., Ryan, C. A. Hydrogen peroxide is generated systemically in plant leaves by wounding and systemin via the octadecanoid pathway. Proc. Natl. Acad. Sci. U.S.A. 96, 6553-6557 (1999).
- Tonks, N. K. Redox redux: revisiting PTPs and the control of cell signaling. Cell. 121, 667-670 (2005).
- Alvarez, S., Wilson, G. H., Chen, S. Determination of in vivo disulfide-bonded proteins in Arabidopsis. J. Chromatogr. B. 877, 101-104 (2009).
- Apel, K., Hirt, H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction. Annu. Rev. Plant Biol. 55, 373-399 (2004).
- Dayon, L. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6- plex isobaric tags. Anal. Chem. 80, 2921-2931 (2008).
- Thompson, A. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Anal. Chem. 75, 1895-1904 (2003).
- Rosenblatt, M. A novel cysteine-reactive tandem mass tag reagent for subproteome labeling, enrichment and quantitation. , TP169 (2010).
- Clark, J. Phenotypic analysis of Arabidopsis mutants: diaminobenzidine stain for hydrogen peroxide. Cold Spring Harb. Protoc. , (2009).
- Sethuraman, M. Isotope-Coded affinity tag (ICAT) approach to redox proteomics: identification and quantification of oxidant-sensitive cysteine thiols in complex protein mixtures. J. Proteome Res. 3, 1228-1233 (2004).
- Asso, M. EPR and redox characterization of ferredoxins I and II from Desulfovibrio vulgaris Miyazaki. Biochem. Biophys. Res. Commun. 211, 198-204 (1995).
- Huang, H. e. Disease resistance to bacterial pathogens affected by the amount of ferredoxin-I proteins in plants. Mol. Plant Pathol. 8, 129-137 (2007).
- Hurkman, W., Tanaka, C. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis. Plant Physiol. 81, 802-806 (1986).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten