Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Einzel Oocyte Bisulfit-Mutagenese
In diesem Artikel
Zusammenfassung
Bisulfit-Mutagenese ist der Goldstandard für die Analyse von DNA-Methylierung. Unsere modifizierte Protokoll ermöglicht DNA-Methylierungs-Analyse auf der Ebene einzelner Zellen und wurde speziell für die einzelnen Eizellen entwickelt. Es kann auch für die Spaltung-Embryonen verwendet werden.
Zusammenfassung
Epigenetik umfasst alle vererbbar und reversible Modifikationen an Chromatin, die alter-Gen Zugänglichkeit und damit die primären Mechanismen für die Regulierung der Gentranskription 1 sind. DNA-Methylierung ist eine epigenetische Modifikation, die überwiegend fungiert als repressiv-Marke. Durch die kovalente Addition einer Methylgruppe an Cytosin in CpG-Dinukleotide, kann es zu rekrutieren weitere Proteine und repressiven Histon-Modifikationen, um Prozesse in kondensierenden Chromatin und Abschaltung von Genen 2 beteiligt zu initiieren. DNA-Methylierung ist wichtig für die normale Entwicklung, da sie eine entscheidende Rolle in der Entwicklungsbiologie Programmierung, Zelldifferenzierung, Unterdrückung von retroviralen Elementen, X-Chromosom-Inaktivierung und genomische Prägung spielt.
Eine der mächtigsten Methoden zur DNA-Methylierungs-Analyse ist Bisulfit-Mutagenese. Natriumbisulfit ist ein DNA-Mutagen, die Cytosine desaminiert in Uracile. Nach PCR-Amplifikation und seque ncing werden diese Umwandlung Ereignisse wie Thymine erkannt. Methylierte Cytosine werden aus Desaminierung geschützt und damit als Cytosine bleiben, welche die Identifizierung von DNA-Methylierung bei der einzelne Nukleotid-Ebene 3. Die Entwicklung des Bisulfit-Mutagenese-Assay wurde von den ursprünglich gemeldeten 4-6 gegen diejenigen, die mehr sensitive und reproduzierbare 7 sind fortgeschritten. Ein wichtiger Fortschritt wurde Einbetten kleiner DNA-Mengen in einem Agarose-Kügelchen, wodurch Schutz der DNA vor der rauen Bisulfit-Behandlung 8. Damit konnte die Methylierungsanalyse auf Pools von Eizellen und Embryonen im Blastozysten-9 durchgeführt werden. Die anspruchsvollste Bisulfit-Mutagenese-Protokoll auf dem neuesten Stand ist für einzelne Blastozyste-Embryonen 10. Da jedoch Blastozysten im Durchschnitt 64 Zellen (mit einem Gehalt von 120 bis 720 pg genomischer DNA) haben, ist diese Methode nicht wirksam für die Methylierung Studien zu einzelnen Eizellen oder Embryonen-Spaltung. Zelt "> Aufnehmen von Hinweisen aus Agarose Einbettung von Minuten DNA-Mengen einschließlich Oozyten 11, hier präsentieren wir eine Methode, bei der Eizellen direkt in einem Agarose-Lyse-Lösung und Wulst unmittelbar nach der Abruf und die Entfernung der Zona pellucida von der Eizelle eingebettet sind. Dies ermöglicht uns, Bypass die beiden wichtigsten Herausforderungen der einzigen Eizelle Bisulfit-Mutagenese:. Schutz einer winzigen Menge von DNA vor dem Abbau und anschließende Verlust während der zahlreichen Schritte Protokoll Wichtig ist, wie Daten aus einzelnen Eizellen gewonnen werden, die Frage der PCR-Bias-Pools innerhalb Außerdem entfällt. , unbeabsichtigte Kontamination Cumulus-Zellen nachweisbar ist, durch diese Methode, da jede Probe mit mehr als einem Methylierungsmuster von der Analyse ausgeschlossen werden kann 12. Dieses Protokoll bietet ein verbessertes Verfahren für eine erfolgreiche und reproduzierbare Analyse der DNA-Methylierung in der Ebene einzelner Zellen und ist ideal geeignet, für die einzelnen Eizellen sowie Embryonen-Spaltung.
Protokoll
TAG 1
Stellen Sie folgende Lösungen frisch auf den Tag der Eizellenentnahme mit sterilem, destilliertem Wasser wie GIBCO Wasser. Um die Möglichkeit von DNA-Kontamination zu verringern, die Handschuhe wechseln oft und Filterspitzen. Halten Röhrchen entfernt, wenn sie geöffnet abgewinkelt ist und Erinnerung alle Rohre, wenn nicht in Gebrauch ist. Wir empfehlen, dass Lösungen wie n +1 gemacht werden.
3% LMP-Agarose
30 mg niedrigem Schmelzpunkt (LMP) Agarose
bis zu 1 ml H 2 O GIBCO
lösen bei 70 ° C
Lysis Solution
8 ul Lysispuffer
1 ul Proteinase K
1 ul 10% IGEPAL
auf Eis bis zur Verwendung
2.01 Agarose: Lysis Solution (10 ul pro einzelne Eizelle, ist Betrag für 3 Oozyten)
20 ul 3% LMP-Agarose
10 pl Lysis Solution
mischen bei 70 ° C
SDS LySIS-Puffer (501 ul pro einzelnen Eizelle)
| 1x TE pH 7,5 | 450 ul |
| 10% SDS | 50 pl |
| Proteinase K | 1 ul |
| 501 ul |
1. Eizellenentnahme
- Legen Sie die Maus seziert Eileiter in M2-Medium, und reißen Sie die Ampullen, die Cumulus-Zell-Komplex zu extrahieren.
- Trennen Sie die Eizellen aus dem Cumulus-Zell-Komplex mit 0,3 mg / ml Hyaluronidase-Lösung in einem 30 ul Tropfen M2-Medium. Halten Sie die Oozyten in Lösung nur solange, wie es um die Cumulus-Zellen zu entfernen dauert, wie lange Exposition beschädigen könnten. Waschen Sie die Oozyten 3x in 30 ul Tropfen M2-Medium, das Entfernen Kumuluszellen in regelmäßigen Abständen.
- Entfernen Sie das Zona pellucida mittels saurer Tyrode-Lösung. Legen Sie die Eizellen in einem 30 ul Tropfen der Lösung zuerst, und dann auf einen anderen übertragen 30ul Tropfen, wie alle Medien mitgeführt wird die Säure zu verdünnen und seine Effizienz zu reduzieren. Halten der Oozyten in Lösung nur so lange wie es die Zona zu entfernen, wie lange Belichtungszeit beschädigen könnten erfolgt. Hinweis: eine erhöhte Konzentration von sauren Tyrode-Lösung oder Pronase kann für den menschlichen Proben verwendet werden, da das menschliche Zona pellucida ist gegenüber einer Behandlung mit sauren Tyrode-Lösung als die Maus.
- Waschen Sie die Eizellen noch einmal in einem 30 ul Tropfen M2-Medium.
2. Agarose Embedding und Lysis
- Um Agarose Einbettung durchführen, legen Sie die Lyse-Lösung auf einem 70 ° C Heizblock. Fügen Sie den vorgeheizten LMP-Agarose zur Lyse-Lösung, wodurch ein 2:1 Agarose: Lyse-Lösung.
- Legen Sie eine einzelne Eizelle auf einen sauberen Objektträger aus Glas in minimalen M2 Medien. Nehmen Sie bis 10 ul der Agarose: Lyse-Lösung in eine Pipettenspitze, und (unter dem Mikroskop) sanft pressen eine kleine Menge (ca. 1 mu l oder weniger) auf den Objektträger, so dass sie mit dem minim mischenal Medien. Vorsichtig holen die Eizelle in die Pipettenspitze und legen Sie alle 10 pl in ein Eppendorf-Röhrchen mit 300 ul Mineralöl so die Raupe einer Kugel bildet.
Hinweis: Dieser Prozess muss ziemlich schnell als der Agarose geschehen wird hart, wenn die Temperatur sinkt so wenig wie 5 ° C unter 70 ° C. - Das Röhrchen auf Eis für 10 Minuten. Zur Lyse durchführen, entfernen Sie die 300 ul Mineralöl und fügen Sie 500 ul des SDS Lysepuffer. Inkubieren über Nacht in einem 50 ° C Wasserbad.
Hinweis: Lyse-Lösung (Tabelle 1) kann auch für diesen Zweck verwendet werden.
TAG 2
Stellen Sie folgende Lösungen frisch am Tag der Bisulfit-Mutagenese. Um die Chance der DNA-Kontamination zu verringern, die Handschuhe wechseln oft und Filterspitzen. Halten Röhrchen entfernt, wenn sie geöffnet abgewinkelt ist und Erinnerung alle Rohre, wenn nicht in Gebrauch ist. Wir empfehlen, dass Lösungen wie n +1 gemacht werden.
| 3 M NaOH | 2,4 g NaOH in 20 ml autoklavierten ddH 2 O |
| 0,1 M NaOH | 0,5 ml 3M in 14,5 ml autoklavierten ddH 2 O |
| 0,3 M NaOH | 1,5 ml 3M in 13,5 ml autoklavierten ddH 2 O |
2,5 M Bisulfit-Lösung
- 3,8 g Natriumbisulfit
5,5 ml GIBCO destilliertem H 2 O
1 ml 3 M NaOH
auflösen @ Raumtemperatur - 110 mg Hydrochinon
1 ml GIBCO destilliertem H 2 O
aufzulösen bei 90 ° C (nur so lange wie es zu lösen hat, zu mischen regelmäßig)
Wenn vollständig aufgelöst ist, zu mischen Lösung (a) und (b)
* Fernhalten von Licht *
3. Bisulfit-Mutagenese
- Vollständig entfernen Sie die 500 ul SDS Lysepuffer und 300 ul Mineralöl (~ 20 Stunden). Jede Lysepuffer übrigen wird verdünnen the Agarose, wenn es erhitzt wird und der Wulst wird anfälliger für Auflösen in den nachfolgenden Schritten. Fahren Sie mit Bisulfit-Mutagenese sofort, oder bei -20 ° C für bis zu 5 Tage.
- Entfernen Sie gegebenenfalls Oozyten aus dem Gefrierschrank und tauen (nur bis Agarosekugel relativ lichtdurchlässig ist). Inkubieren für 2,5 Minuten auf 90 ° C Heizblock, nach dem Inkubieren auf Eis für 10 Minuten.
Hinweis: Verwenden Sie nicht gleichzeitig oder rühren, verlängern länger als 2,5 Minuten, oder schwanken Temperatur. - Zur Denaturierung durchführen, entfernen Sie die Mineralöl-und 1 ml 0,1 M NaOH in jedes Röhrchen, Flick und invertieren 5-6 mal.
- Inkubation: 15 Minuten bei 37 ° C im Wasserbad, Invertieren alle 3-4 Minuten. Der Wulst sollte in der NaOH schweben.
- Um Bisulfit-Behandlung durchzuführen, drehen Sie das Rohr vorsichtig, dann entfernen Sie die NaOH und 300 ul Mineralöl und 500 pl Bisulfit-Lösung. Das Röhrchen für 3,5 Stunden in einem 50 ° C im Wasserbad. * Fernhalten von Licht *
Hinweis: Die Länge der Inkubation Möglicherweise müssen empirisch für Gen von Interesse bestimmt werden. - Um Desulfonierung durchführen, Inkubation auf Eis für 3 Minuten, entfernen Sie dann die Mineralöl-und die Bisulfit-Lösung, spinnen sanft, und 1 ml 0,3 M NaOH. Flick und invertieren 5-6 mal.
- Inkubation: 15 Minuten bei 37 ° C im Wasserbad, Invertieren alle 3-4 Minuten. Der Wulst sollte in der NaOH schweben.
- Waschen Sie die Proben, indem Sie zunächst die Spinnerei sanft, dann entfernen Sie die NaOH und 1 ml 1x TE pH 7,5. Schütteln Sie für 5-10 Minuten bei Raumtemperatur (auf einem Schüttler). Spin sanft wieder, dann entfernen Sie die 1x TE. Wiederholen Sie diesen Vorgang zweimal Waschen.
- 1 ml autoklavierten ddH 2 O. Schütteln Sie für 5-10 Minuten bei Raumtemperatur (auf einem Schüttler). Spin sanft, dann entfernen Sie das H 2 O. Wiederholen Sie ddH 2 O Waschen zweimal.
- Den pH-Wert des Überstands; sollte pH 5,0 sein. Wenn immer noch zu einfach, nachwaschen mit H 2 O. Entfernen Sie das gesamte Überstand, so dass nur die Agarose seinad.
4. 1. und 2. Runde PCR-Amplifikation
- Bereiten 1. Runde PCR-Mix ** beim Waschen **
| 10 uM Primer vorderen äußeren | 0,5 ul |
| 10 uM Primer Reverse-Outer | 0,5 ul |
| 240 ng / ml tRNA | 1 ul |
| H 2 O | 13 ul |
In den Illustra Ready-to-Go Hot Start PCR Perlen
Schieben Sie die feste Agarose-Kügelchen in die PCR-Röhrchen (~ 10 ul)
Hitze bis 70 ° C und mischen
In 25 ul Mineralöl
Gesamt: 50 pl
- Verstärken
Hinweis: Ein Beispiel-Bedingungen für Maus Snrpn ist Denaturierung 2 min bei 94 ° C, gefolgt von 40 Zyklen von 30 Sekunden bei 94 ° C, 1 Minute bei 50 ° C und 1Minute bei 68 ° C und eine endgültige 10 Minuten Verlängerungsschritt bei 68 ° C Glühtemperatur für 1. Runde PCR für Maus und Peg3 H19 beträgt 50 ° C. - Bereiten 2. Runde PCR-Mix
| 10 uM Primer vordere innere | 0,5 ul |
| 10 uM Primer Reverse-Innere | 0,5 ul |
| H 2 O | 19 ul |
In den Illustra Ready-to-Go Hot Start PCR Perlen
Geben Sie 5 ul 1. Runde Produkt als Vorlage. Erhitzen Sie die 1. Runde Produkt bis 70 ° C für 1 Minute, um die Agarose zu erweichen. Stellen Sie sicher, unter der Schicht aus Mineralöl pipettieren.
In 25 ul Mineralöl
Gesamt: 50 pl
Hinweis: Verschachtelte Primersequenzen für Snrpn, H19 und Peg3 kann in Market-Urteil Velker et al 10 zu finden,12.
- Verstärken
Hinweis: Die Zyklusbedingungen für Maus Snrpn ist Denaturierung 2 min bei 94 ° C, gefolgt von 40 Zyklen von 30 Sekunden bei 94 ° C, 1 Minute bei 50 ° C und 1 Minute bei 68 ° C und eine endgültige Dehnung 10 Minuten Schritt bei 68 ° C 10. Maus H19 und Peg3 eine 50 ° C Annealing-Temperatur für 2 Runde PCR. - Als eines diagnostischen Tests können zweiten Runde Proben mit einem Restriktionsenzym, das Methylierung oder Stamm-spezifische geschnitten werden.
| 2. Runde Produkt | 4 ul |
| Restriktionsenzym | 1 ul |
| Puffer | 1 ul |
| H 2 O | 4 ul |
- Elektrophorese die Verdauungsprodukte auf einem 8% igen Acrylamidgel. Etwaige heterogenen Banden repräsentieren mehr als eine sequence.
5. TA Cloning und Kolonie-PCR
- Um 2. Runde Produkt zunächst erwärmt auf 70 klonen ° C für 1 Minute, um die Agarose, dann Ligat in den Vektor unter Verwendung des Promega pGEM-T Vektor-System (Fisher Scientific Cat # A1360) zu erweichen.
| 2. Runde PCR | 1 ul |
| pGEMT-easy-Vektor | 1 ul |
| Ligase | 1 ul |
| H 2 O | 2 ul |
| 2x Ligationspuffer | 5 ul |
Inkubieren über Nacht bei 4 ° C in PCR-Maschine.
- Auftauen kompetente E. coli Zellen auf Eis für 15 Minuten (Zymo Research Corp Cat # T3009). In 3 ul Ligationsreaktion zu 8 ul E. coli und inkubieren Ligation auf Eis für 15 Minuten.
- Hitzeschock für 40 Sekunden in einem 42 ° C im Wasserbad,und auf Eis für 2 Minuten inkubiert. In 60 ul SOC-Medium und Inkubation bei 37 ° C für 1 Stunde (im Shaker).
- Legen Sie alle der Reaktionsmischung auf eine LB / Agar / IPTG / X-Gal / Amp-Platte und inkubieren Platte bei 37 ° C über Nacht.
- Bereiten Kolonie-PCR-Mix
| 20 uM M13 Vorwärts-Primer | 0,7 ul |
| 20 uM M13 Reverse Primer | 0,7 ul |
| 5X Grüne Go Taq-Puffer | 7,0 ul |
| 10 mM dNTP | 0,7 ul |
| Taq DNA-Polymerase | 0,28 ul |
| H 2 O | 25,62 ul |
| Insgesamt 35 ul |
In 35 ul Colony PCR Master Mix in ein PCR-Tube. Wählen Sie einen weißen Bakterienkolonie von der Platte mit einer Pipettenspitze und wirbeln sie in die PCR-Reaktion.
- Verstärken mit Denaturierung für 10 Minuten bei 94 ° C, gefolgt von 30 Zyklen von 45 Sekunden bei 94 ° C, 30 Sekunden bei 57 ° C und 1 Minute bei 72 ° C und eine endgültige Dehnung 10 Minuten bei 72 ° C 4 ul Elektrophorese auf einem 1,5% Agarosegel unterworfen. Senden ~ 30 ul des PCR-Produkts für die Sequenzierung.
Hinweis: Oozyten, 5 Kolonie-PCR-Produkte sequenziert. - Sobald Sequenzierung erhaltenen Ergebnisse sind, können Methylierungsmuster zu lesen. Jede Original-CG, die als CG blieb wurde methyliert, und jedes Original CG, die heute ein TG wurde nicht methyliert.
6. Repräsentative Ergebnisse
In unserer Arbeit mit dem Aufdruck wir Assay-Methylierung in einzelnen Eizellen und Embryonen (Abbildung 1). Nach verschachtelten PCR-Amplifikation mit Primern konvertierter, ist es möglich, einen erfolgreichen Umwandlung durch Visualisierung einer korrekten Fragments die auf einem Agarosegel (2) zu bestätigen. Eine einzelne Eizellestellt einen elterlichen Allel, und in der Theorie, hat man aufgedruckten Methylierungsmuster. Als solches kann zweiten Runde PCR-Produkte für unbeabsichtigte Verunreinigungen geprüft werden. Ein Restriktionsenzym empfindlich auf DNA-Methylierung (wie HinfI oder DpnII) verwendet werden, um die zweite Runde PCR-Produkt verdauen zu beurteilen, ob ein methyliertes Allel oder unmethylierten (3) enthält. Ein methyliertes C innerhalb des Enzym-Erkennungssequenz gespalten wird, während ein unmethylierten C, die T umgewandelt wird nicht mehr durch das Enzym erkannt und ist nicht geschnitten. Jede Probe, die MII-Eizelle sowohl methylierter und nicht methylierter Allele sollte verworfen werden, da es indikativ für Cumulus Zellkontamination (Abbildung 3) ist. Nach Ligation und Transformation, kann eine erfolgreiche Kolonie PCR-Amplifikation werden auf einem Agarosegel visualisiert werden, um sicherzustellen, Proben mit dem richtigen Produkt zur Sequenzierung Größe gesendet werden (Abbildung 4). Schließlich wird die Sequenz von fünf einzelnen cldiejenigen aus einer MII Eizelle produzieren sollten fünf identische Methylierungsmuster und identisch nonCpG Conversion-Raten (Abbildung 5a). Alle Proben, die mehr als ein Muster enthalten, sollten verworfen werden (Abbildung 5b) werden. Seit ovulierten MII-Oozyten zwei Chromosom-Kopien oder eine angehängte Polkörper haben, gibt es eine Möglichkeit für den Erhalt von zwei ähnlichen Sequenz Muster (Abbildung 5c). Wir empfehlen Verwerfen von Daten aus Eizellen, die höchst unterschiedliche Methylierungsmuster haben seit Cumulus Zellkontamination kann nicht ausgeschlossen werden.

Abbildung 1. Schematische Darstellung der Einzel-Oocyte Bisulfit-Mutagenese-Assay.
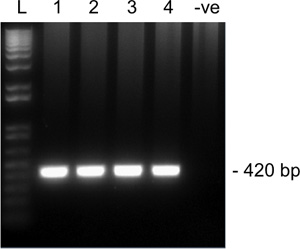
Abbildung 2. Repräsentative Ergebnisse aus 2. Runde Verstärkung für Snrpn aus einer singenle MII Oozyte auf einem 1,5% Agarosegel unterworfen. Spuren 1-4 sind vier Single-MII-Oozyten und Spur 5 ist eine Negativ-Kontrolle (keine Eizelle). Erwartete Fragmentlänge für Snrpn beträgt 420 bp. L, Leiter.
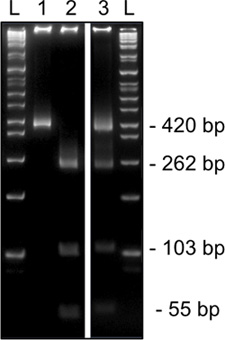
3. Repräsentative Ergebnisse von 2 Runde methylierungsspezifische Restriktionsverdau für Snrpn aus einer Eizelle MII auf einem 8% igen Acrylamidgel. HinfI diagnostischen Restriktionsverdau zeigt unmethyliert DNA, die ein T, das die Restriktionsstelle schafft trägt (420bp, Spur 1) oder methylierte DNA, die eine C innerhalb Erkennungsstelle enthält (Schnitt, 262, 103 und 54 bp, Spur 2). Die Verdauung in denen sowohl methylierter und nicht methylierter Restriktionsenzymstellen (Cut & Uncut Bands, Spur 3) sind bezeichnend für Cumulus-Zell-Kontamination. L, Leiter.
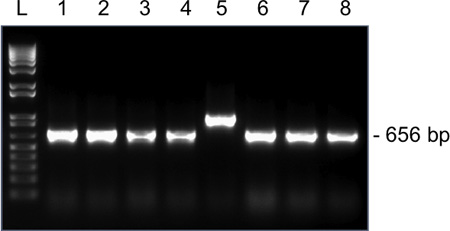
Abbildung 4. Repräsentative Ergebnisse für Kolonie-PCR-Amplifikation für Snrpn aus einer Eizelle MII auf einem 1,5% Agarosegel unterworfen. Voraussichtlich Fragmentlänge nach Ligation Snrpn in den pGEM-T Easy Vektor und unter Verwendung von M13 Vorwärts und Rückwärts-Primer ist 656 bp. Lane 1-8, 1-8 Amplikons aus Klonen. Clone 5 hat eine falsche Fragmentlänge und sollte nicht zum Sequenzieren geschickt werden.

Abbildung 5. Vertreter Sequenzierung Ergebnisse für Snrpn aus einer einzigen Eizelle MII. Snrpn wird in Oozyten methyliert. Schwarze Kreise zeigen methylierte CpG. Weiße Kreise zeigen unmethylierte CpGs. CpG Anzahl und die Platzierung ist repräsentativ für eine B6-Stamm weiblichen Maus. a) Erwartete Ergebnisse für die Sequenzierung Snrpn aus einer einzigen Eizelle MII. Nur ein einzelner DNA-Strang ist in allen fünf Klone zu amplifizieren. Oozyten mit einem einzigen Methylierungsmuster und identische nicht-CpG-Konvertierung Geplappern sollten Auswertung einbezogen werden (prozentuale Umwandlung von nicht-CpG angegeben auf der rechten Seite wurde die Anzahl der nicht-CpG Cytosine, die, wie in Prozent der gesamten nicht-CpG Cytosine Thymin berechnet). b) Die Sequenzierung Ergebnisse für Snrpn aus einer einzigen Eizelle MII mit Cumulus-Zell-Kontamination. Beachten Sie die Unähnlichkeit zwischen Methylierung Staaten und Muster, die Umwandlung Mehrfachsteckgliedern Verstärkung. c) Sequenzierung Ergebnisse für Snrpn aus einer einzigen Eizelle MII mit beiden Chromosomen-Kopien oder Polkörper Eingliederung.
Diskussion
Diese einzelne Eizelle Assay enthält viele Schritte mit einer Zahl, die kritisch sind und bedürfen einer besonderen Pflege. Die erste ist Oozyte Waschen. Es ist besonders wichtig zu waschen Jeder Oocyt mehrmals in frischem Medium fällt nach Hyaluronidase-Behandlung, so viele Kumuluszellen wie möglich zu entfernen. Darüber hinaus bei der Übertragung von Eizellen, um saure Tyrode-Lösung für Zona pellucida Entfernung sicherstellen umgebenden Medium ist klar, von Cumulus-Zellen. Die Eizelle ist sehr klebrig zona nac...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde von der University of Western Ontario, der Abteilung für Geburtshilfe und Gynäkologie unterstützt, und einen Zuschuss ER06-02-188 aus dem Ministryof Forschung und Innovation, Early Researcher Award. MMD wurde von einem CIHR-Trainings-Programm in der Reproduktion, frühe Entwicklung und die Auswirkungen auf die Gesundheit (REDIH) Graduate Scholarship unterstützt.
Materialien
Tabelle der spezifischen Reagenzien und Geräte.
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Eizellenentnahme | |||
| Hyaluronidase | Sigma | H4272 | |
| Saure Tyrode | Sigma | T1788 | |
| Proteinase K | Sigma | P5568 | |
| 10% IGEPAL | Bioshop | NON999.500 | |
| Lysis Solution | |||
| Tris, pH 7,5 | Bioshop | TRS001.5 | |
| LiCl | Sigma | L9650 | & Nbsp; |
| EDTA, pH 8,0 | Sigma | E5134 | |
| LiDS | Bioshop | LDS701.10 | |
| DTT | Invitrogen | P2325 | |
| SDS-Lysepuffer | |||
| TE pH 7,5 | Bioshop (Tris) Sigma (EDTA) | TRS001.5 E5134 | |
| 10% SDS | Bioshop | SDS001.500 | |
| Bisulfit-Konvertierung | |||
| Natriumhydroxid | Sigma | S8045 | |
| Natriumhydrogensulfit (Natriumbisulfit) | Sigma | 243973 | ; |
| Hydrochinon | Sigma | H9003 | |
| Niedrigen Schmelzpunkt (LMP) Agarose | Sigma | A9414 | |
| Mineralöl | Sigma | M8410 | |
| M2 Medium | Sigma | M7167 | |
| GIBCO destilliertes Wasser | Invitrogen | 15230-196 | |
| Autoklavierten doppelt destilliertes (dd) Wasser | |||
| PCR | |||
| Illustra Hot Start Mix RTG | GE Healthcare | 28-9006-54 | |
| 240 ng / ml Hefe-tRNA | Invitrogen | 15401-011 | |
| 5x Grün GoTaq Reaktionspuffer | ProfiMega- | M7911 | |
| Innere und äußere verschachtelten Primern | Sigma | ||
| Ligation | |||
| Promega pGEM-T Easy Vektor | Fisher Scientific | A1360 | |
| TA Cloning | |||
| Kompetente E. coli Zellen | Zymo Research Corp | T3009 | |
| Ausrüstung | |||
| Lupen-Mikroskop | |||
| 70 ° C und 90 ° C Hitze-Blöcke | |||
| 37 ° C und 50 ° C Wasserbäder (42 ° C für Transformationen) | |||
| Rocker | |||
| PCR-Maschine | |||
Referenzen
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten