Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung von Kardiomyozyten Kerne von Post-mortem Gewebe
In diesem Artikel
Zusammenfassung
Cardiac Kerne über Dichte Sedimentation isoliert und mit Antikörpern gegen immunmarkiert pericentriolar Material 1 (PCM-1) zu identifizieren und zu sortieren Kardiomyozyten Kerne durch Durchflusszytometrie.
Zusammenfassung
Identifizierung von Kardiomyozyten Kerne hat in Gewebeschnitten Herausforderung gewesen, da die meisten Strategien nur auf zytoplasmatische Markerproteine 1 verlassen. Seltene Ereignisse in Herzmuskelzellen wie Proliferation und Apoptose erfordern eine genaue Identifizierung von Kardiomyozyten-Kerne auf die Zellerneuerung in der Homöostase und bei pathologischen Zuständen 2 zu analysieren. Hier stellen wir eine Methode, um Kardiomyozyten Kerne aus post mortem Gewebe zu isolieren durch Sedimentation und Dichte Immunomarkierung mit Antikörpern gegen pericentriolar Material 1 (PCM-1) und anschließende Durchflusszytometrie-Sortierung. Diese Strategie ermöglicht einen hohen Durchsatz-Analyse und Isolation mit dem Vorteil bei der Arbeit ebenso gut auf frischen und gefrorenen Gewebe Archivalien. Dies macht es möglich, das Material bereits in Biobanken gesammelten studieren. Diese Technik ist und die Messung in einer Vielzahl von Arten und mehrfach Downstream-Anwendungen, wie Kohlenstoff-14-3, Zell-cycle-Analyse 4, Visualisierung von Thymidin-Analoga (zB BrdU und IdU) 4, Transkriptom-und epigenetische Analyse.
Protokoll
1. Die Isolierung des Herz-Kerne
- Mantel Ultrazentrifuge Rohre (Beckman Zentrifugenröhrchen # 363.664) mit 10 ml 1% BSA / PBS Beschichtungslösung. Verschließen Sie die Röhrchen und lassen Sie sie für 30 min in einem Rohr Rotator drehen. Entfernen Sie die Beschichtungslösung und lassen Sie die Zentrifugenröhrchen Luft trocknen lassen (eine Tube pro Maus Herz für die Analyse der einzelnen Mäuseherzen, abwechselnd bis zu 5 Mäuseherzen oder 1 g Herzgewebe von einer anderen Spezies erforderlich ist (z. B. Mensch) können verarbeitet werden In einem Röhrchen).
- Alle folgenden Schritte sollten auf Eis durchgeführt werden. Präparieren Sie die linke Herzkammer aus frischen oder gefrorenen Snap-Maus-Herz mit einem Skalpell. Man beachte, daß dieses Protokoll für Mäuseherz optimiert, sondern auch von Ratten oder menschlichen Herzens angepasst werden. Alternativ können bis zu 1 g des Herzgewebes von einer anderen Spezies.
- Schneiden Sie die Probe in kleinen Kabinen mit einem Skalpell.
- Übertragen der Gewebestücke in einen 50 ml-Falcon-Röhrchen mit 15 ml Lyse-Puffer gefüllt.
- Homogenisieren derHerzgewebe mit einem T-25 Ultra-Turrax-Homogenisator Sonde (IKA) bei 24.000 UpM für 10 sek.
- Verdünnen des Homogenats mit einem gleichen Volumen Lysepuffer bis 30 ml.
- Verwenden Sie ein Glas Douncer (40 ml) zur weiteren Homogenisierung des Gewebes und die Kerne frei. Führen Sie acht Schläge mit einem großen Abstand Pistill.
- Führen Sie das rohe Kerne durch ein 100 um und 70 um Nylon-Mesh Zellsieb (BD Biosciences), fortlaufend zu isolieren.
- Anzentrifugieren das rohe Kerne in einer gekühlten Zentrifuge (4 ° C) bei 700 × g für 10 min isoliert.
- Entfernen Sie den Überstand vorsichtig durch Umdrehen der Rohre und wischen Sie das Innere des Rohres mit einem Papiertuch. Achten Sie darauf, die Kerne Pellet stören.
- Man löst das rohe Kerne in 5 ml Saccharose-Puffer zu isolieren durch Pipettieren der Lösung mehrmals auf und ab. Fügen Sie eine weitere 25 ml Saccharose-Puffer auf die gelöste Pellet.
- 10 ml frisch zubereiteten Saccharose-Puffer auf die beschichtete Ultrazentrifuge Rohr (siehe Schritt 1.1).
- Sorgfältig überlagern den zusätzlichen 10 ml Saccharose-Puffer mit dem resuspendierten Pellet Kerne in Saccharose-Puffer von 1,9 Schritt aufgelöst.
- Gleichen Sie die Zentrifugenröhrchen, bevor Sie sie in eine JS13.1 frei schwingenden Rotor und platzieren Sie den Rotor in eine High-Speed-Zentrifuge (Beckman Avanti S-25).
- Drehen der Kerne Probe bei 13.000 × g bei 4 ° C für 60 min.
- Wenn die Spin abgeschlossen ist, entfernen die Röhrchen vorsichtig von dem Rotor und der Überstand verworfen durch Invertieren der Rohre und Abwischen der restlichen Ablagerungen aus dem Inneren der Rohre mit einem Papiertuch.
- Lösen Sie die Kerne Pellet in 1 ml Kerne Pufferspeicher (NSB Plus-Puffer). Hinweis: NSB plus enthält 1,5 mM Spermin als DNA-Stabilisator.
- Fahren Sie mit Schritt 2.1, Immunfärbung von Kardiomyozyten Kernen.
2. Immunfärbung für die Durchflusszytometrie
- Die negativen Kontrollen für Immunfärbung. Man entnimmt einen aliquoten von 20 ul aus den Kernen Probe und eineDD 980 ul von NSB und Puffer.
- In Anti-pericentriolar Material 1-Antikörper (Kaninchen-Anti-PCM-1, Atlas Antikörper) zu den Kernen Probe in einer Verdünnung von 1:500 bis Immunmarkierung Kardiomyozyten Kernen. In den Isotyp-Antikörper in der gleichen Verdünnung des Anti-PCM-1-Antikörper mit der Negativkontrolle, in Schritt 2.1 hergestellt.
- Inkubieren negativen Kontrolle und Probe-Röhrchen bei 4 ° C über Nacht.
- Waschen Sie Negativ-Kontrolle und Probe mindestens einmal mit NSB Plus-Puffer (Spin-Down Rohre in einer gekühlten Zentrifuge (4 ° C) bei 700 xg für 10 min. Überstand verwerfen und lösen sich die Kerne Pellet in 1 ml NSB Plus-Puffer).
- In Anti-Kaninchen-fluoreszierenden sekundären Antikörper (FITC oder APC) zu negativen Kontrolle und Probenröhrchen in einer Verdünnung von 1:1000.
- Inkubieren negativen Kontrolle und Probe-Röhrchen bei 4 ° C für 1 h.
- Waschen Sie Negativ-Kontrolle und Probe mindestens einmal mit NSB Plus-Puffer (Spin-Down Rohre in einer gekühlten Zentrifuge (4 ° C) bei 700 xg für 10 min. Überstand verwerfen und lösen sich die Kerne Pellet in 1 ml NSB Plus-Puffer).
- Fahren Sie mit der durchflusszytometrischen Analyse und Sortierung.
3. Durchflusszytometrie
- Coat Kerne Sammlung Röhrchen (Falcon 15 ml) mit 1% BSA / PBS-Lösung, bevor Sie die Durchflusszytometrie Sortierung wie in Schritt 1.1 beschrieben starten.
- Filtern Sie die Probe und die negative Kontrolle durch einen 30 um Zellsieb und laden Sie zuerst die negative Kontrolle der Durchflusszytometer (BD Zufluss). Definieren Sie die erste und zweite Tor zu Kernen und Singuletts (Einzel-Kerne) zu definieren, basierend auf Vorwärtsstreuung (FSC), Vorwärtsstreuung Impulsbreite (FS Pulsbreite) und seitliche Streulicht (SSC) (Abb. 2a und b). Hinzufügen eines DNA-Färbung (DRAQ5 (1:500)) zu der Probe kann helfen, die Kerne Bevölkerung zunächst zu identifizieren.
- Laden der Probe und immunmarkierten definieren das dritte Tor zu Kardiomyozyten Kerne (PCM-1-positve) aus nicht-Kardiomyozyten Kerne (PCM-1-negativen) zu isolieren. Starten Sie die Sortierung(Abb. 2c und d).
Optional: Um die Kern-DNA-Gehalt (Ploidie) zu analysieren und Zellzyklus-Analyse durchzuführen fügen Sie eine entsprechende DNA-Färbung der Kerne (zB Hoechst 33342 oder DRAQ5) (Abb. 2e).
- Nach der Durchflusszytometrie Sortierung, legen Sie die Kerne auf Eis und neu analysieren, um die Sortierung Reinheit (Abb. 3a und b) zu bestimmen.
- Drehen Sie den sortierten Kerne in den Sammelröhrchen bei 1500 xg in einer Kühlzentrifuge für 15 min.
- Man löst die Kerne Pellets in einer Pufferlösung mit dem Downstream-Anwendungen.
4. Repräsentative Ergebnisse
Kerne Morphologie und Integrität durch DNA Flecken beurteilt werden und visualisiert durch Mikroskopie (Abb. 1). Erfolgreiche PCM-1 Kennzeichnung kann durch Epifluoreszenzmikroskopie und mittels Durchflusszytometrie (Abb. 1 und Abb. 2c und d) beurteilt werden. PCM-1-Positive und negative Populationen sollten gut voneinander getrennt werden (Abb. 2c und d). In murinen linken Ventrikel ungefähr 30% aller Kerne Kerne sein sollte Kardiomyozyten (2d). Sortieren Reinheit kann durch neue Analyse der sortierten Kerne (Abb. 3a und b) beurteilt werden. Beide Kerne Bevölkerung sollte eine Sortierung Reinheitsgrad von mehr als 95%.
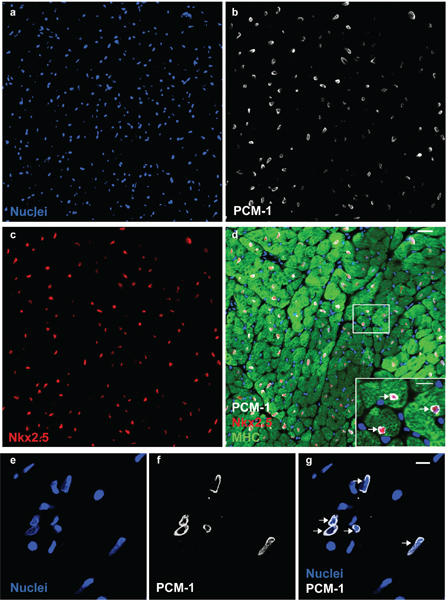
Abbildung 1. PCM-1 identifiziert Kardiomyozyten Kerne. Cardiac Kerne (a) mit Antikörpern gegen PCM-1 (b) und Nkx2.5 (c) in einer erwachsenen Maus Herzen gefärbt werden. (D) PCM-1-markierte Kerne werden von Kardiomyozyten Zytoplasma (Myosin heavy chain (MHC)) umgeben ist und zum Ausdruck des Transkriptionsfaktors Nkx2.5 und dokumentiert die genaue Identifizierung von Kardiomyozyten Kerne von PCM-1-Färbung (Maßstab 20 pm und 10 um (d, Einschub)). (E) Cardiac Kerne Isolate visualisiert mit dem DNA-Farbstoff DRAQ5. (F und g) CardiomyocYTE Kerne sind mit Antikörpern gegen PCM-1 (Maßstab 10 um) beschriftet. Beachte, dass die epinuclear Färbungsmuster von PCM-1 in Myozyten Kerne im Gewebe aufweist und in isolierten Kernen (Pfeile).
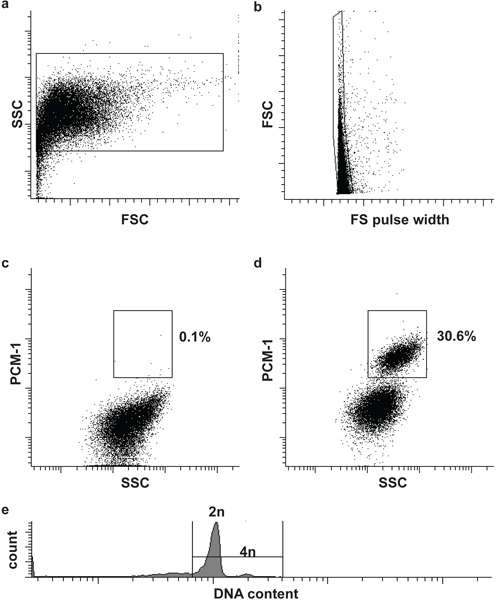
Abbildung 2. Durchflusszytometrische Sortierung von Kardiomyozyten Kernen. (A) Cardiac Kerne werden durch Vorwärtsstreuung (FSC) und Seitwärts-Streulicht (SSC) identifiziert. (B) ein zweites Tor identifiziert einzelne Kerne von FSC und FS Impulsbreite 5. (C, d) Fluoreszenz-Gating ermöglicht die Trennung von Kardiomyozyten Kerne (PCM-1-positiv) und nicht-Kardiomyozyten (PCM-1-negativen) Kerne von Herzgewebe. (E) Maus Kardiomyozyten meist (> 80%) diploid (2n), ist nur eine kleine Teilmenge tetraploiden (4n) 6. Hinweis enthalten menschlichen Kardiomyozyten eine höhere Frequenz von Polyploidie Kerne (> 2n) 7,8.

Abbildung 3. Reinheit Analyse der sortierten Kardiomyozyten und nicht-Kardiomyozyten Kerne. Re-Analyse der sortierten Nicht-Kardiomyozyten (a) und Kardiomyozyten Kerne (b). Beide Populationen zeigen eine Sortierung Reinheitsgrad von mehr als 99%.
Diskussion
Genaue Identifizierung der Kardiomyozyten Kerne ist entscheidend für die Analyse der regenerativen Vorgänge im Myokard 2,3. Herkömmliche Techniken zu Kardiomyozyten aus frischem Gewebe zu isolieren sind hauptsächlich auf enzymatischen Abbau von Proteinen der extrazellulären Matrix und die anschließende Reinigung in der interstitiellen Zellen durch Zentrifugation bei niedriger Geschwindigkeit basiert. Die weitere Reinigung des lebenden Kardiomyozyten aus embryonalen Stammzellen (ESC) Immunmarkierung mit ...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir möchten Marcelo Toro für die Unterstützung bei der Durchflusszytometrie anerkennen. Diese Studie wurde vom schwedischen Herz-und Lungen-Stiftung, EU-Kommission RP7 "CardioCell", Schwedischer Forschungsrat, AFA Versicherungen und ALF unterstützt. OB wurde von der Deutschen Forschungsgemeinschaft unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1. Lysispuffer | |||
| Name des Reagenzes | |||
| 0,32 M Saccharose | |||
| 10 mM Tris-HCl (pH = 8) | |||
| 5 mM CaCl 2 | |||
| 5 mM Magnesiumacetat | |||
| 2,0 mM EDTA | |||
| 0,5 mM EGTA | |||
| 1 mM DTT |
| 2. Saccharose-Puffer |
| Name des Reagenzes |
| 2,1 M Saccharose |
| 10 mM Tris-HCl (pH = 8) |
| 5 mM Magnesiumacetat |
| 1 mM DTT |
| 3. Kerne Speicherpuffer (NSB plus) |
| Name des Reagenzes |
| 0,44 M Saccharose |
| 10 mM Tris-HCl (pH = 7,2) |
| 70 mM KCl |
| 10 mM MgCl 2 |
| 1,5 mM Spermin |
| Chemikalien und Geräte | Firma |
| Kaninchen-IgG-Isotyp-ChIP Grade, # ab37415 | Abcam |
| Kaninchen anti-PCM-1-Antikörper, # HPA023374 | Atlas Antibodies |
| Donkey sek. Antikörper, Anti-Kaninchen Alexa 488 Fluor, # A-21.206 oder gleichwertige sek. Fluoreszenz-Antikörper | Life Technologies |
| DRAQ5 | Biostatus |
| Zelle Siebe 30 um, 70 um und 100 um | BD Biosciences |
| Glass Douncer (40 ml) undPistill "L" | VWR (Wheaton Industries Inc.) |
| T-25 Ultra-Turrax-Homogenisator | IKA Deutschland |
| Dispergierwerkzeug S25 N-18 G | IKA Deutschland |
| Beckman Avanti Zentrifuge | Beckman Coulter |
| Falcon-Tubes 15 ml und 50 ml | VWR |
| Beckman Zentrifugenröhrchen # 363664 | Beckman Coulter |
| JS13.1 frei schwingenden Rotor | Beckman Coulter |
| Influx Zytometer | Beckman Coulter |
| Tube Rotator | VWR |
Referenzen
- Ang, K. L. Limitations of conventional approaches to identify myocyte nuclei in histologic sections of the heart. American journal of physiology. Cell physiology. , 298-1603 (2010).
- Bergmann, O. Identification of cardiomyocyte nuclei and assessment of ploidy for the analysis of cell turnover. Experimental cell research. 327, 188-194 (2011).
- Bergmann, O. Evidence for Cardiomyocyte Renewal in Humans. Science. 324, 98-102 (1126).
- Walsh, S. Cardiomyocyte cell cycle control and growth estimation. Cardiovascular Research. , 1-31 (2010).
- Spalding, K., Bhardwaj, R. D., Buchholz, B., Druid, H., Frisén, J. Retrospective birth dating of cells in humans. Cell. 122, 133-143 (2005).
- Adler, C. P., Friedburg, H., Herget, G. W., Neuburger, M., Schwalb, H. Variability of cardiomyocyte DNA content, ploidy level and nuclear number in mammalian hearts. Virchows Arch. 429, 159-164 (1996).
- Herget, G. W., Neuburger, M., Plagwitz, R., Adler, C. P. DNA content, ploidy level and number of nuclei in the human heart after myocardial infarction. Cardiovascular Research. 36, 45-51 (1997).
- Adler, C. P., Friedburg, H. Myocardial DNA content. ploidy level and cell number in geriatric hearts: postmortem examinations of human myocardium in old age. Mol. Cell Cardiol. 18, 3953-39 (1986).
- Dubois, N. C. SIRPA is a specific cell-surface marker for isolating cardiomyocytes derived from human pluripotent stem cells. Nature. 29, 1011-1018 (2011).
- Hattori, F. Nongenetic method for purifying stem cell-derived cardiomyocytes. Nature Methods. 7, 61-66 (2010).
- Fransioli, J. Evolution of the c-kit-Positive Cell Response to Pathological Challenge in the Myocardium. Stem Cells. 26, 1315-1324 (2008).
- Elliott, D. A. NKX2-5(eGFP/w) hESCs for isolation of human cardiac progenitors and cardiomyocytes. Nature Methods. 8, 1037-1040 (2011).
- Laflamme, M. A. Evidence for Cardiomyocyte Repopulation by Extracardiac Progenitors in Transplanted Human Hearts. Circulation Research. 90, 634-640 (2002).
- Srsen, V., Fant, X., Heald, R., Rabouille, C., Merdes, A. Centrosome proteins form an insoluble perinuclear matrix during muscle cell differentiation. BMC cell biology. 10, 28 (2009).
- Spoelgen, R. A novel flow cytometry-based technique to measure adult neurogenesis in the brain. Journal of neurochemistry. 119, 165-175 (2011).
- Soonpaa, M. H., Kim, K. K., Pajak, L., Franklin, M., Field, L. J. Cardiomyocyte DNA synthesis and binucleation during murine development. The American journal of physiology. 271, H2183-H2189 (1996).
- Olivetti, G. Aging, cardiac hypertrophy and ischemic cardiomyopathy do not affect the proportion of mononucleated and multinucleated myocytes in the human heart. J Mol Cell Cardiol. 28, 1463-1477 (1996).
- Okada, S. Flow cytometric sorting of neuronal and glial nuclei from central nervous system tissue. Journal of cellular physiology. 226, 552-558 (2011).
- Matevossian, A., Akbarian, S. Neuronal Nuclei Isolation from Human Postmortem Brain Tissue. J. Vis. Exp. (20), e914 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten