Method Article
Multi-Target-Parallel Processing Ansatz zur Gene-to-Struktur Bestimmung der Influenza Polymerase PB2-Untereinheit
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Structure-based drug design spielt eine wichtige Rolle in der Medikamentenentwicklung. Verfolgen mehrere Ziele parallel erhöht die Chance auf Erfolg für Blei Entdeckung. Der folgende Artikel zeigt, wie die Seattle Structural Genomics Center for Infectious Disease eine Multi-Target-Ansatz für die Gen-to-Bestimmung der Struktur des Influenza-A-Untereinheit PB2 nutzt.
Zusammenfassung
Pandemic Ausbrüche der hoch virulenten Influenza-Stämme verursachen verbreitet Morbidität und Mortalität in menschlichen Populationen weltweit. In den Vereinigten Staaten allein, sind ein Durchschnitt von 41.400 Todesfälle und 1,86 Millionen Hospitalisierungen durch Influenza-Virus-Infektion verursacht jedes Jahr 1. Punktmutationen in der Polymerase-basic protein 2-Untereinheit (PB2) haben, um die Anpassung des viralen Infektion beim Menschen 2 verbunden. Erkenntnisse aus solchen Studien haben die biologische Bedeutung der PB2 als Virulenzfaktoren nachgewiesen, was hervorgehoben sein Potenzial als einem antiviralen Medikament Ziel.
Die strukturelle Genomik Programm, die weiter von der National Institute of Allergy and Infectious Disease (NIAID) stellt Mittel für Bio Smaragd und drei weitere Pacific Northwest Institutionen, die zusammen die Seattle Structural Genomics Center for Infectious Disease (SSGCID). Die SSGCID ist bestrebt, die wissenschaftliche Gemeinschaft mit dre gewidmetE-dimensionalen Proteinstrukturen von NIAID AC Krankheitserreger. Machen solche strukturellen Informationen der wissenschaftlichen Gemeinschaft dient zur Struktur-based drug design beschleunigen.
Structure-based drug design spielt eine wichtige Rolle in der Medikamentenentwicklung. Verfolgen mehrere Ziele parallel erhöht die Chance auf Erfolg für neue Blei Entdeckung durch Targeting einen Weg oder eine ganze Proteinfamilie. Smaragd Bio hat eine High-Throughput-, Multi-Target-parallele Verarbeitung Pipeline (MTPP) für die Gen-zu-Struktur Entschlossenheit, das Konsortium unterstützt entwickelt. Hier beschreiben wir die Protokolle verwendet werden, um die Struktur des PB2-Untereinheit von vier verschiedenen Influenza-A-Stämme zu bestimmen.
Protokoll
Ein Überblick über das Protokoll ist in Abbildung 1 dargestellt.
Molecular Biology
1. Construct Entwurf
Verwenden Gene Composer-Software, um Protein-Konstrukt und Codon entwickelt synthetische Gensequenzen entwerfen. Der Einsatz von Gen-Composer-Software im Detail an anderer Stelle 3 angeboten worden.
- Verwenden Sie die Alignment-Viewer-Modul und Konstruieren Design-Modul, um Protein-Alignments Vergleichen und definieren Protein Konstrukt. Richten Zielaminosäuresequenz sowohl für die Primär-und 3D-Strukturelemente von Homologen in der Protein Data Bank (PDB), sofern vorhanden (Abb. 2).
- Verwenden Ausrichtung Informationen zu Struktur-geführte Konstrukt Designs, indem Sie neue Termini auf die Erhaltung der primären Struktur und 3D-Strukturen von Homologen zu treffen.
- Design-Einsatz PCR (iPCR) und Vektor-PCR (VPCR) Amplimere (Klemme Primer).
- Mit Gene Composer die Protein-zu-DNA-Algorithmus zurück-übersetzen das Konstrukt Aminosäuresequenz in Codon engineered Nukleinsäuresequenz.
- Verwenden Sie die richtige Codonverwendungstabelle (CUT), um zur Expression in E. optimieren coli.
- Praktisch klonen insert into pET28 Vektor modifiziert, um eine N-terminalen 6x Histidin-Tag und Smt3/SUMO Fusionsprotein, das für eine einfache Reinigung ermöglicht integrieren.
- Zeigen synthetischen Gens um mit DNA 2.0 und Ordnung Primer von Integrated DNA Technologies.
2. Unvollständige Polymerase Primer Extension (PIPE) Cloning
- Bereiten Grundierungen und Gene
- Zentrifugieren Sie die vom Hersteller bereitgestellte Platten mit Primer bei 1.000 rpm für 1 min.
- Bringen Primer-Konzentration auf 100 pM und 50 ul TE-Puffer.
- Verdünnen Primer bis 10 um mit deionisiertem (DI) Wasser in einem 96-Well-v-Bodenplatte.
- Zentrifugieren Sie die vom Hersteller bereitgestellte Gen in einem 1,5-ml-Röhrchen bei 1300 rpm für 1 min.
- Verwenden TE-Puffer, bringen die DNA-Konzentration von jeder Röhre bis 50 ng / ul.
- In 1,5 ml Röhrchen, machen Verdünnungen von jedem Primer bis 10 ng / ul.
- Shop Primer und Gene bei -20 ° C, wenn sie nicht in Gebrauch ist.
- Bereiten Insert PCR (iPCR)
- Tauwetter ein Fläschchen mit Pfu Master Mix auf Eis, halten Gene und Primer bei Raumtemperatur.
- Erstellen Sie eine Platte Karte Zuweisen Brunnen zu einem Satz von Primer und konstruieren.
- In 13 ul DI Wasser in jede Vertiefung einer 96-Well-PCR-Platte.
- In 5 ul vorwärts und 5 ul Rückwärtsprimers zu jeder Reaktion in der 96-Well-Platte nach der Platte Karte, wodurch Tipps untereinander auch zu ändern.
- In 2 ul jeder Volllängengens seine entsprechenden auch nach der Platte Karte.
- In 25 ul Pfu Master-Mix in jede Vertiefung, die Gewährleistung um Tipps untereinander auch zu ändern.
- Zyklus die Reaktionen mit den folgenden PCR-Bedingungen:
- 95 ° C 2 min
- 95 ° C 30 sec
- 50 ° C 45 sec
- 68 ° C 3 min
- 4 ° C ∞
- Wiederholen Sie die Schritte bd für 25 Zyklen.
- Übertragen Sie 10 ul jeder iPCR Reaktion auf eine neue 96-well PCR-Platte.
- In 3 ul 6X Last Farbstoff zu jeder Probe.
- Trennen Sie die einzelnen Probe auf einem 1% TAE EtBr Agarosegel bei 110 V neben einem 100-500 bps DNA-Leiter zu fragmentieren Verstärkung bestätigen.
- Shop iPCR Produkt bei -20 ° C, wenn nicht in Gebrauch ist (Frost-Tau zu vermeiden, so viel wie möglich).
3. Bereiten Sie Vector PCR (VPCR)
- Starten Übernachtkultur von transformierten E. coli mit pET28 Vektorplasmid.
- Impfen zwei 5-ml-Röhrchen von 2-YT-Brühe mit 50 ug / ml Kanamycin.
- Wachsen Kulturen über Nacht bei 37 ° C in Schüttler bei 220 Umdrehungen pro Minute.
- Spin down Kulturen nach Wachstum über Nacht durch Zentrifugation bei 3.000 rpm für 15 min.
- Verwenden Sie eine Qiagen QIAprep Spin Miniprep Kit pET28 Vektor von Bakterienpellets Extrakt gemäß den Anweisungen des Herstellers.
- Setup-Restriktionsenzymverdaue der extrahierten pET28 Plasmid.
- In 2,2 ul 10X BamHI Puffer und 1 ul BamHI und HindIII bis 20 ul pET28 Vektor.
- Inkubieren Reaktion für 1 Stunde bei 37 ° C.
- Separate Spaltprodukt auf einem Gel.
- Siehe 2.2.10 fort.
- Geschnittenen Vektor Band aus Gel und reinigen Sie es mit dem QIAquick Gel Extraction Kit nach den Anweisungen des Herstellers.
- Mit einem NanoDrop, quantifizieren DNA-Konzentration.
- Verdünnen geschnitten Vektor bis 10 ng / ul. Lagerung bei -20 ° C, wenn sie nicht in Gebrauch ist.
- Bereiten VPCR Primer.
- Zentrifuge IDT geliefert oligonucliotides für 1 min bei 1.300 Umdrehungen pro Minute.
- Bringt Konzentration auf 100 pM mit DI-Wasser.
- Bereiten 10 uM Verdünnung von beiden Forward-und Reverse-Primer in einem 1,5-ml-Tube.
- Shop Primer und die Primer Verdünnungen bei -20 ° C.
- Auftauen Pfu Master Mix auf Eis und Tauwetter Template und Primer bei Raumtemperatur.
- Setup-VPCR Reaktionen in einer 96-Well-PCR-Platte:
- In der ersten Reihe einer 96-Well-Platte kombinieren 60 ul der beiden Forward-und Reverse-Primer VPCR und 24 ul verdaut pET28 Schablone (10 ng / ul).
- Mit einem 12-tip Multikanalpipette, übertragen 12 ul der Primer und Template-Master-Mix für jeden verbleibenden Vertiefung der Platte. Dies sollte in 12 ul Primer und Matrize Mastermix in jede Vertiefung der Platte führen.
- In 13 ul DI-Wasser in jede Vertiefung.
- In 25 ul Pfu Master Mix in jede Vertiefung.
- Zyklus die Reaktionen durch die PCR-Bedingungen in Schritt 2.2.7 verwendet.
- Pool alle VPCR Reaktionen in einem 15 ml Falcon-Röhrchen.
- Überprüfen Fragment Verstärkung durch die Trennung 10 ul gepoolten PCR-Produkt auf einem Gel (erwartete Länge der verdauten Vektor pET28beträgt ca. 6 kb).
- Siehe 2.2.10 fort.
- Bereiten Sie verschmelzen Platten.
- Aliquot 3 ul VPCR Produkt in jede Vertiefung einer 96-Well-v-Bodenplatte.
- Shop Platten bei -20 ° C bis Fusion mit iPCR Produkt.
4. Merge iPCR und VPCR Produkte
- Auftauen iPCR Produkte und vor-aliquotiert VPCR 96er verschmelzen Platte bei Raumtemperatur.
- In 3 ul jeder iPCR Produkt seiner jeweiligen und der Zusammenführung Platte.
- Verwandeln Sie verschmelzen Platte in Top Ten chemisch kompetenten Zellen.
- In 2 ul jeder Zusammenführung Reaktion in einem einzigen 50 ul Rohr Anbieter gelieferten chemisch kompetente Zellen und gehen mit einer vom Hersteller mitgelieferten Protokoll.
- Bereiten Übernacht-Kulturen für jedes Konstrukt aus der Transformation Platte.
- Aliquot 5 ml TB-Brühe (mit 50 ug / ml Kanamycin) von einem 25 ml sterile Reservoir in jede Vertiefung einer tiefen Brunnen Block.
- Mit Sterile Technik, nehmen eine isolierte Kolonie von jeder Transformation Platte und impfen die entsprechende Vertiefung der Tiefbrunnen Block.
- Bedecken Sie den Block mit einem Airpore Abdeckung.
- Schütteln bei 220 rpm Block bei 37 ° C über Nacht.
- Pellet-Zellen durch Zentrifugieren der Block für 30 min bei 4.000 Umdrehungen pro Minute.
- Abgießen des Überstandes und klopfen die Spitze der Block mit einem trockenen Papiertuch.
- Mini-Prep unter Verwendung eines Qiagen 96er Vakuumvorrichtung nach den Anweisungen des Herstellers.
5. Vorbereiten Glycerin Stocks erfolgreich geklonten Konstrukte
- Erfolgreich geklont Transform Sequenz validiert DNA in BL21 (DE3) chemisch kompetente Zellen nach der Anleitung des Herstellers.
- Für jedes Konstrukt, wählen Sie eine einzelne isolierte Kolonie aus dem BL21 (DE3) Transformation und impfen in 1 ml 2-YT-Bouillon (mit 50 ug / ml Kanamycin).
- Schüttelkulturen bei 220 rpm für 3-4 Stunden bei 37 ° C.
- Beschriften Sie eine 1,5 mlSchraubdose mit dem einzigartigen Konstrukt Identifikationsnummer Zellstamm und Datum. In 500 ul 50% Glycerin und 500 ul Zellkultur und invertieren mehrmals. Unmittelbar speichern Glycerin Lager auf Trockeneis oder in einem -80 ° C Gefrierschrank.
6. Expression Testing
| Lysis Buffe r | Waschpuffer | Elutionspuffer |
| 50 mM NaH 2 PO 4, pH 8,0 300 mM NaCl 10 mM Imidazol 1% Tween 20 2 mM MgCl 2 0,1 ul / ml Benzonase 1 mg / ml Lysozym | 50 mM NaH 2 PO 4, pH 8,0 300 mM NaCl 20 mM Imidazol 0,05% Tween 20 | 25 mM Tris, pH 8,0 300 mM NaCl 250 mM Imidazol 0,05% Tween 20 |
* Add Benzonase, Lysozym,und Protease-Inhibitor unmittelbar vor der Lyse.
- Streak eine Probe aus Glycerin Lager auf Kanamycin Selektivagar und Inkubation über Nacht bei 37 ° C.
- Starten Sie eine nicht-induzierenden Vorkultur in eine 96-Well rundem Boden block; impfen 1,2 ml Brühe TB (mit 50 mg / ml Kanamycin) mit 0,5% Glucose mit einer frisch gewachsenen E. ergänzt coli zu isolieren. Über Nacht wachsen Schütteln bei 220 rpm bei 37 ° C
- Nach Wachstum über Nacht, starten Induktion Kulturen durch Impfen 1,2 ml Brühe TB (mit 50 mg / ml Kanamycin) mit Novagen Overnight Express System 1 (nach dem Protokoll des Herstellers) mit 40 ul der Vorkultur ergänzt.
- Wachsen die kleinen Induktion Kulturen bei 20 ° C für 48 Stunden unter Schütteln bei 220 Umdrehungen pro Minute.
- Ernten Sie die Zellen durch Zentrifugation bei 4.000 rpm für 15 min, Überstand abgießen und bei -20 ° C für mindestens 1 Stunde vor der Verarbeitung.
- In der 96-Well-Block, resuspendieren Zellpellets in 300 ul Lysepuffer.
- Inkubieren Zellen in Lysepuffer bei Raumtemperatur für 30 min durch mechanische Lyse durch kräftiges Schütteln für 30 min bei Raumtemperatur.
- Klären Sie die Rohlysat durch Zentrifugation für 30 min bei 4.000 rpm bei 4 ° C.
- Verwenden Sie ein Multi-Kanal-Pipette bis 200 ul des geklärten Lysats (lösliche Fraktion) zu einer 96-well Flachboden Fach (Qiagen) zu übertragen. Für jede Vertiefung eine Probe enthält, fügen Sie 40 ul Ni-NTA magnetischen Kügelchen (Qiagen).
- Vorsichtig geschwenkt, die Platte auf einer Wippe für 1 h bei 16 ° C
- Legen Sie die Platte auf einem magnetischen Mastschild (Qiagen) und entfernen Sie die ungebundenen löslichen Fraktion. Achten Sie darauf, nicht Pipette aus einem der Ni-NTA-Perlen.
- Entfernen Sie die Platte von der Post Platte und vorsichtig resuspendiert die Perlen in 200 ul Waschpuffer. Pipette nach oben und unten für 30 Sekunden und dann wird die Platte wieder auf den Beitrag Teller.
- Entfernen Sie den Waschpuffer und wiederholen Sie Schritt 6.12.
- Entfernen Platte von Mastschild und eluieren Ni-NTA gebunden target Protein durch Waschen mit 50 ul Elutionspuffer für 5 min.
- Zurück flache Bodenplatte, um magnetische Mastschild und übertragen Sie die Elution in ein frisches 96-v-Bodenplatte.
- Übertragen Sie 20 ul der Elution in ein frisches 96-v-Bodenplatte und reagieren mit 1 ul Ulp1 Protease.
- Gemäß dem Protokoll des Herstellers, analysiert das eluierte und eluiert + Ulp1 Fraktion durch Kapillarelektrophorese unter Verwendung eines LabChip 90.
- Alternativ können alle Fraktionen, die von der Expression Tests über SDS-PAGE analysiert werden.
7. Large Scale Fermentation
- Verwenden Sie eine sterile Pipettenspitze ein Kratzen aus einem Glycerin Lager erhalten, impfen 100 ml Brühe TB (mit 50 mg / ml Kanamycin) und über Nacht wachsen. Schütteln bei 220 rpm und 37 ° C.
- Nach Wachstum über Nacht, erweitern Vorkultur durch Impfen 1 l Brühe TB mit EMD Autoinduktion Lösungen (siehe Protokoll des Herstellers) (mit 50 mg / ml Kanamycin) in einem 2 L Schikanekolben mit 10 ml derVorkultur (1:100 Verdünnung).
- Schütteln Sie die erweiterten 1-l-Kulturen bei 37 ° C; verändern die Temperatur der Schüttelinkubator bis 20 ° C, wenn eine optische Dichte von 0,6 (OD 600) erreicht wird.
- Nach Wachstum über Nacht, eine repräsentative 10 ml Aliquot von jedem Konstrukt für die Expression Tests.
- Ernte-Zelle durch Zentrifugation bei 5.000 rpm für 15 min fügen und Überstand verwerfen.
- Freeze-Zelle bei -80 ° C fügen
Proteinreinigung
Puffer:
| Lysis Buffer | Puffer A (Äquilibrierung) | Buffer B (Elution) | Sizing Column Buffer |
| 25 mM Tris, pH 8,0 200 mM NaCl 0,5% Glycerol 0,02% CHAPS 10 mM Imidazol 1 mM TCEP 50 mM Arginin 5 ul Benzonase 100 mg Lysozym 3 Protease Inhibitor Tabletten (EDTA-frei) | 25 mM Tris, pH 8,0 200 mM NaCl 10 mM Imidazol 1 mM TCEP 50 mM Arginin 0,25% Glycerol | 25 mM Tris, pH 8,0 200 mM NaCl 200 mM Imidazol 1 mM TCEP | 25 mM Tris, pH 8,0 200 mM NaCl 1% Glycerin 1 mM TCEP |
* In Benzonase, Lysozym und Protease-Inhibitor Tabletten zu jeder 150 ml Probe unmittelbar vor der Lyse.
8. Cell Lysis
- Als 2 l Lysepuffer; nicht hinzufügen Lysozym, Protease-Inhibitor Tabletten oder Benzonase (jedes Sample einzeln in 150 ml Lysepuffer werden).
- Tauwetter und resuspendieren Zellen in Lyse-Puffer bei einer 1:5-Masse fügen: Volumen-Verhältnis durch kräftiges Rühren für 30 min bei 4 ° C. Brechen Brocken lose von Seiten des Bechers mit einem sauberen Spatel. Während dieser Zeit herzustellen Ni und Dialysis Puffer
- Auf Eis, lysieren die Zellen unter Verwendung eines Misonix Ultraschallgerät (70% Leistung, 2 sec an / 1 s aus Impulse für 3 min) und schwenken Sie Behälter Überhitzung zu vermeiden. Speichern Sie eine kleine (200 ul) Aliquot Rohlysat für künftige Analysen.
- Klären Sie die Rohlysat durch Zentrifugation bei 18.000 g für 35 min bei 4 ° C, den Überstand und speichern Sie eine kleine (200 ul) Aliquot zur späteren Analyse. Laden Pellets bei 4 ° C bis bestätigt wird, das Protein in der löslichen Fraktion wurde lysiert.
9. Pre-run Setup-Protein Maker
- Mit dem Protein maker eingeschaltet und die Software zu öffnen, initialisieren Sie das Instrument.
- Einmal initialisiert, befestigen eine 5,0 ml GE Healthcare HisTrap FF Nickel-Chelat-Säule (Ni Spalte) in einer separaten Zeile der Gantry für jede der Proben.
- Führen Sie 3-4 Säulenvolumen (CV) von Equilibrierungspuffer durch jede Spalte.
- PRIME die Equilibrierung und Elutionspuffer Linien.
- Equilibraß die Spalten durch Absaugen Puffer A durch die Säule einmal.
10. Nickel 1 (Ni1) Spalte
- Waschen Sie jede Säule mit 20 ml Milli-Q Wasser Pufferspeicher entfernen. Führen Sie 5 ml Puffer B und 25 ml Puffer A für Äquilibrierung.
- Laden des geklärten Lysats mit solubilisierten Proteins in den Spalten mit einer Geschwindigkeit von 2 ml / min dann durch einen 15 ml Waschen mit Puffer A folgen
- Eluieren des gebundenen Proteins in einem Schritt Gradienten mit Puffer A und B durch die folgenden Verhältnisse respektvoll: 5 ml 95:5, 60:40 5 ml, 10 ml 0:100. Sammle jede Elutionsfraktion getrennt.
- Analysieren: eluierten Fraktionen, Rohlysat, geklärte Lysat und Durchfluss durch SDS-PAGE. Pool Fraktionen, die das Protein und verwenden Sie einen Nanodrop auf der A 280 zu messen, um grob die Menge des Proteins vorhanden.
11. Ulp1 Dekolleté
- Halten Sie ein kleines Aliquot (250 ul) des Ni1 Spalte Pool für die anschließende Gel-Analyse. Bringen Sie den Restder Ni1 Pool bis 10 ml und fügen Ubiquitin-ähnliche Protease 1 (Ulp1) bei 1 ul / 5 mg des gesamten Proteins, die His-Smt Affinitätstag entfernen.
- Dialysieren die Ni1 pool + Ulp1 gegen 2 l Puffer A für 4 Stunden bei 4 ° C in einer 10 kDa Molekulargewicht (MWCO) auf einer Rührplatte bei 4 ° C.
- Nach der Dialyse laufen SDS-PAGE von Ni1 Pool und Ni1 pool + Ulp1 um festzustellen, ob Ulp1 Spaltung war erfolgreich.
12. Nickel 2 (Ni2) Spalte
- Legen gespaltenen Protein gegenüber dem gleichen Ni Spalte und wiederholen Sie Schritt 9.3 zu einem reduzierten Fließgeschwindigkeit von 1 ml / min. Die gespaltene off-Tag wird an der Säule zu binden und die tagless Zielprotein wird nun Durchströmung. Sammeln Sie die Durchströmung in einem frischen Behälter.
- Waschen Ni Säule mit 3 ml Puffer A, gefolgt von 5 ml Puffer B für alle His-Tag und unspezifisch gebundenes Protein zu eluieren. Sammle jede Fraktion getrennt.
- Führen SDS-PAGE von Ni2 Flow-Through, waschen und Ni2 Elutionsfraktionen zu Ulp1 Spaltung Akkreditierung und dass protein ist in der Durchströmung. Verwenden Sie einen Nanodrop auf der A 280 zu messen, um etwa das Vorhandensein des Proteins.
13. Konzentration
- Konzentriere das Ni2 Flow-Through (und Ni2 Elution wenn Protein vorhanden ist) bis 5 ml mit einer Amicon Ultra 10 kDa MWCO Zentrifugenröhrchen. Spin in 10 Minuten-Intervallen bei 4.000 rpm bei 4 ° C. Mischen Sie mit einer Pipette zwischen jeder Drehung, um Protein-Konzentration von über entlang Membran zu verhindern.
14. Größenausschlusschromatographie (SEC)
- Richten Sie eine Sephacryl S-100 10/30 GL Säule (GE Healthcare) von ausgleichenden mit 200 ml SEC-Puffer bei einer Flussrate von 0,5 ml / min auf einem ÄKTApurifier System (GE Healthcare).
- Planen 10 ml Superloops zum Gebrauch auf der SEC-Säule gemäß den Anweisungen des Herstellers.
- Mit einem 5-ml-Spritze, laden Proben auf Superloops und beginnen die SEC run.
- Überwachen Sie die UV-Absorption bei 280 nm Spur beim Sammeln von kleinen Volumen frAktionen.
- Führen SEC Fraktionen über SDS-PAGE.
- Pool Der SEC-Fraktionen, welche die höchste Intensität Bands.
- Konzentrieren gepoolten Fraktionen SEC. Siehe 13.1 fort.
- Aliquot Protein in 100 ul-Proben, Flash freeze in flüssigem Stickstoff eingefroren und bei -80 ° C
KRISTALLISATION
15. Protein Kristallisation
- Pre-fill jedes Reservoir einer 96-Well-Compact Jr Kristallisationsplatte (Smaragd Bio) mit 80 ul der Kristallisation Bildschirm (Smaragd Bio) der Wahl.
- Verdünnen Protein mit Schlichte Puffer auf 2-20 mg / ml und auf Eis lagern.
- Dosieren 0,4 ul Protein und 0,4 ul der Kristallisation Bildschirm in jede der 96 Vertiefungen und decken mit kristallklarem Dichtband (Manco).
- Bewahren Sie die Platte bei 16 ° C während der Überprüfung auf Protein Kristallisation regelmäßig in den nächsten paar Wochen unter einem Binokular.
16. Kristall Harvesting
- Erstellen Sie ein Frostschutzmittel aus der Mutterlauge und Ethylenglykol. Schneiden Sie die durchsichtigem Klebeband für den gut mit dem Zielprotein Kristall. Um einem leeren gut, fügen 1,6 ul des entsprechenden kristallisierenden Zustand und verbinden sich mit 0,4 ul Ethylenglykol was eine Endkonzentration von 20% Ethylenglykol und 80% kristallisieren Zustand. Hinweis: Um Kristall Beugung zu optimieren versuchen, verschiedene Kryoprotektoren wie: Glycerin, Öle, niedrige MW Polyethylenglykole und / oder in unterschiedlichen Prozentsätze der Kälteschutzmittel.
- Vor der Ernte ab einer ALS-Stil Puck in einem Dewar mit flüssigem Stickstoff und Deckel mit Deckel gefüllt cool.
- Ernten Sie den Kristall, indem Sie einen CryoLoop mit dem Innendurchmesser passend zur Größe des Kristalls auf einem Magnetic Crystal Wand (Hampton Research) und schöpfen Sie es direkt aus dem Brunnen Lösung.
- Sofort tauchen die CryoLoop mit dem geernteten Kristall in der Kälteschutzmittel dann tauchen in der ALS-Stil Puck zu blinken einfrieren crystal. Wiederholen für eine gewünschte Anzahl von Kristallen.
17. Kristall Screening / Data Collection
- Nach der Ernte ist komplett mit einem Puck Zauberstab, um die magnetische Kryo Puck Deckel auf den Puck ALS platzieren. Mit gebogenen Zange, drehen Sie den Puck auf den Kopf.
- Übertragen Sie den Puck zu einem Rigaku SCHAUSPIELER Dewar, schrauben Sie den Puck Pusher auf den Puck, und Punsch den Deckel so dass es in dem Dewar mit Stiften nach oben zeigen.
- Mit JDirector Software, Bildschirm jeder Kristall unter den folgenden Parametern: Strahl Schlitz auf 0,5 Grad, Detektor-Abstand auf 50 mm, Bild Schritt bis 70 Grad, und die Belichtung Länge bis 30 Sekunden eingestellt.
- Führen Mosflm auf den Testbildern Sie mit JDirector geschossen, um zu bestimmen, was die beste Strategie für die Kristall-und Datenerfassung.
- Sammeln Sie eine komplette Datensatz auf Ihre Ergebnisse aus Mosflm basiert.
18. Data Processing / Strukturbestimmung
- Führen XDS / XSCALE 4, um die Datenmenge zu verarbeiten.
- Öffnen Sie die CCP4 Suite Software.
- Führen Phaser 5 einen molekularen Ersatz-Lösung mit einer hohen Homologie Suchmodell, wenn verfügbar berechnen. In diesem Fall haben wir das PDBID 3CW4 als Suchmodell 6.
- Führen Refmac 7 bis Ihr molekulares Modell gegen die beobachtete Reflexion in der Datenmenge gesammelt verfeinern. Endgültige Auflösung sollte aus der höchsten Schale beruhen und bestimmt durch die folgenden Parameter: R-Faktor> 50%, I / sigma> 2 und Vollständigkeit> 90%.
- Bauen Sie ein 3-Dimensional Elektronendichte Modell mit der molekularen Grafiksoftware COOT 8.
- Vor Aufbringen der Struktur in der PDB bestätigen Sie mit MolProbity 9-Software zu, dass die Qualität des Bauwerks zu überprüfen ist geeignet für die Abscheidung.
Ergebnisse
Die folgenden Ergebnisse zeigen die erwarteten Ergebnisse der beschriebenen Protokoll, und im Falle von PB2, die beobachteten Ergebnisse.
Mit Gene Komponist, fünf in voller Länge Ziel Aminosäuresequenzen des Influenza-Virus-Polymerase-Untereinheit PB2 entworfen wurden (Abbildung 2). Die Sequenzen wurden PB2 zurück übersetzt und einer viele technische Schritte 3, was in Codon harmonisierten Sequenzen für die Expression in E. optimiert coli. Vom iPCR Produkte (Abbildung 3b), insgesamt vierunddreißig Konstrukte wurden erfolgreich in einer modifizierten pET28 Vektor-System 10 mit einem N-terminalen 6x His-tag Smt Fusion mit PIPE Klonen 3, wie in 3a gezeigt geklont. Eine Zusammenfassung des Klonens Workflow ist in Abbildung 4 dargestellt.
Nach erfolgter Klonierung wurde Mikromaßstab Proteinexpression von jedem Konstrukt in BL21 (DE3) E. getestetcoli-Zellen. Die Zellen wurden in TB-Medium mit Novagen Overnight Express 1 Medium (nach dem Protokoll des Herstellers) für 48 Stunden ergänzt bei 20 ° C in einem Schüttelinkubator bei 220 rpm gewachsen. Nach dem Wachstum wurden die Zellen geerntet und für lösliche Protein-Expression mittels Kapillarelektrophorese Elektrophorese mit einem Caliper LabChip 90 getestet. Vierzehn der vierunddreißig PB2 Konstrukte führten zu löslichen Zielprotein und eingegeben großflächige Fermentation. Groß angelegte Kulturen von jedem Konstrukt wurden in TB-Medium mit Overnight Express Novagen die 1 Medium nach dem Protokoll des Herstellers ergänzt gewachsen. Nach Wachstum wurden die Zellen durch Zentrifugation geerntet und bei -80 ° C. Large-scale Proteinexpression von jeder Kultur wurde über SDS-PAGE-Analyse (Abbildung 5), bevor mit großen Reinigung bestätigt.
Das Protein Maker wurde verwendet, um parallel Reinigung der vierzehn PB2 Konstrukte durchzuführen. Die geklärten Lysaten von all vierzehn Konstrukte wurden durch eine Nickel-Chelat-Säule laufen. Nach der Bestimmung, welche Fraktionen enthaltenen Target-Protein mittels SDS-PAGE wurden die entsprechenden Fraktionen für jede Probe gesammelt und die Konzentration wurde jeweils durch einen A 280 Messwert bestimmt. Entfernen des 6x His-tag Smt wurde durch die Zugabe von Ulp1 durch Dialyse über Nacht und einer zweiten Nickel-Säule durchgeführt. Bestätigung der His-tag Smt Entfernung wurde durch SDS-PAGE (Abbildung 6) durchgeführt, und jede Probe wurde mit einer 10 kDa Amicon Ultra-Zentrifugenröhrchen konzentriert. Nach der Konzentration mit Hilfe der Amicon Ultra-Zentrifugen-Röhrchen wurde jede Probe über eine Dimensionierung Säule laufen, um kristallographische Reinheit zu erreichen. Eine zweite Konzentration wurde durchgeführt, um die Proteinkonzentration auf einer erforderlichen Höhe für die Kristallisation zu erhöhen. Alle vierzehn Konstrukte wurden erfolgreich gereinigt und trat Kristallisation Studien.
Die Kristallisation wurde durch Auftauen der zuvor fr eingeleitetozen Protein. Die Kristallisation wurde in einem klimatisierten Raum bei 16 ° C mit speziell entwickelten Platten (Smaragd Bio) für Sitting Drop Dampfdiffusion (Abbildung 7). Initial Screening wurde mit vier Sparse-Matrix-Bildschirme durchgeführt; JCSG +, Pakt, Wizard Full und CryoFull (Smaragd Bio), nach einer längeren Newman Strategie. 0,4 ul Protein-Lösung wurde dann mit 0,4 ul crystallant (oder Reservoir-Lösung) aus dem entsprechenden Behälter mit 96-Loch-Compact Jr Kristallisation Platten (Smaragd Bio). Von den vierzehn gereinigten Proben neun von ihnen lieferte geeignete Kristalle für die Beugung Studien (Abbildung 8). Eine Inhouse-Beugung Datensatz wurde auf fünf der neun Konstrukte bei Cu Ka Wellenlänge kristallisiert mit einem Rigaku SuperBright FR-E + rotierenden Anode X-ray-Generator mit Osmic VariMAX HF Optik und Saturn 944 + CCD-Detektor ausgestattet ist (Abbildung 9 gesammelt ). Jeder Datensatz wurde mit XDS / XSCALE 4 verarbeitet < / Sup> und skaliert, um eine endgültige Auflösung. Versuche, die Strukturen, die durch molekularen Ersatz zu lösen wurden mit Phaser 5 vom CCP4 Suite 7 durchgeführt. Die letzten Modelle wurden nach der Veredelung in REFMAC 7 und manueller Wiederaufbau mit Blässhuhn 11 erhalten. Die Strukturen wurden geprüft und korrigiert für Geometrie und Fitness mit MolProbity 9. Insgesamt vier Strukturen des PB2-Untereinheit bestimmt wurden (Abbildung 10) und hinterlegt in der PDB. Abbildung 11 zeigt das Gesamtergebnis auf jeder Stufe in der Pipeline MTPP.

Abbildung 1. Übersicht der SSGCID Gen-zu-Struktur Weg für Multi-Target-parallele Verarbeitung im Emerald Bio.
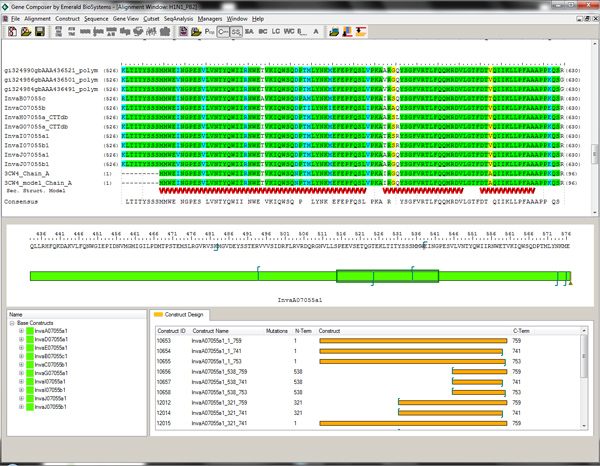
Abbildung 2. Alignment-Viewer und Protein Design-Modul in Construct Gene Composer-Software. Amino-Säure-Basis bauen Ziel ist in grün (Mitte Fenster) und die Struktur geführte Verkürzungen alternative Konstrukte sind in Gold (unteres Fenster) gezeigt. Ein Abgleich von mehreren viralen Grippe PB2 Sequenzen im Vergleich zu der Sequenz und sekundären Strukturelemente gezeigt der C-terminalen Domäne von PDBID 3CW4. Das Wissen um die Domänen-Struktur und sekundären Strukturelemente können N-terminale Verkürzungen innerhalb der Gene Komponist Design-Modul mit der rechten Maustaste auf den gewünschten Aminosäurerest gewählt werden. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
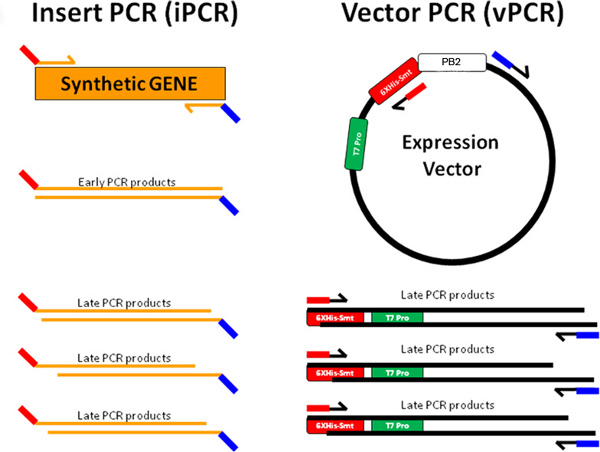
3a. PIPE Klonen dargestellt, wobei das synthetische Gen Einsatz (orange) von vorn (rot-orange Linien) entworfen und umgekehrt (orange-blaue Linien) Primer zur Erzeugung einfügen PCR Material verstärkt wird. Expressionsvektor mit Rückwärtsgang (rot-schwarze Linien wird verstärkt ) und vorne (blau-schwarze Linien) Primer PCR Vektor Material zu erzeugen. Die terminalen Sequenzen iPCR Produkte sind komplementär zu den terminalen Sequenzen VPCR Produkte (Rot iPCR ergänzt Rot VPCR und Blau ergänzt iPCR Blau VPCR). Dadurch können die iPCR und VPCR Produkte zu tempern Plasmide, nach Transformation in Wirtszellen BL21 (DE3) chemisch kompetente E. repliziert bilden coli-Zellen.

3b. Agarosegel Analyse iPCR Produktion ts vom PB2-Untereinheit. iPCR Ausfälle können als schwach oder schmierig Bands zu sehen, während erfolgreiche iPCR Produkte robust Bands vertreten sind. iPCR Produktqualität kann in der Regel mit dem Klonen Erfolg korreliert werden. Molekulargewichtsmarker sind in kiloDalton. Abbildung von Raymond et al., 2011, 12 wiedergegeben.

Abbildung 4. Gentechnik Schritte Ziel PB2 Proteine wurden unter Verwendung Gene Composer-Software. Nachdem das entwickelte Nukleinsäuresequenz für jedes Ziel gegründet wurde, wurden 6-7 alternative Protein-Konstrukte für jede gestalten. Multi-Target-parallele Verarbeitung in den ersten Schritten der Gen-Design und Klonierung in Folge 34 Konstrukte, von denen 14 tragfähige Ziele, die lösliche Proteine in E. produziert wurden coli.
re 5 "src =" / files/ftp_upload/4225/4225fig5.jpg "/>
Abbildung 5. Repräsentative SDS-PAGE-Analyse von großen Fermentation zeigt robust Proteinexpression (erwarteten Größe von 25,76 kDa), etwa 50% löslich (Bahn 4) und etwa 50% Spaltung von 6x His-tag von Smt eluierte Protein (Spur 7).
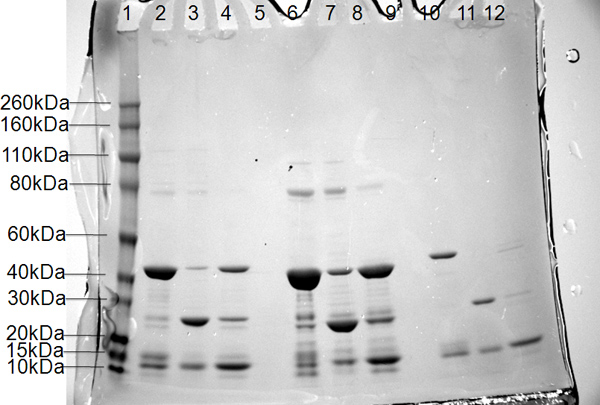
Abbildung 6. SDS-PAGE-Ergebnisse für drei Konstrukte der Polymerase-PB2-Untereinheit Spur 1, Molekulargewichts-Marker (beschriftet links in kDa). Spuren 2, 6 und 10, zusammengefaßt Protein aus Nickel 1 Spalte, Spuren 3, 7, und 11, Durchfluss von gespaltenen Proteins in Puffer A aus Nickel 2; Spuren 4, 8 und 12, die Entfernung von 6x His-tag Smt in Puffer B aus Nickel 2.
d/4225/4225fig7.jpg "/>
Abbildung 7. Eine schematische Darstellung der Dampfdiffusion durch die sitzen Drop-Methode. Die sitzen Drop-Methode für Protein Kristallisation fällt unter die Kategorie der Dampfdiffusion. Dieses Verfahren beinhaltet eine gereinigte Probe von Protein und Fällungsmittel mit einem größeren Reservoir mit ähnlichen Bedingungen in einer höheren Konzentration auszugleichen. Da das Wasser verdampft aus der Proteinprobe und überträgt zu dem Reservoir erhöht das Fällungsmittel Konzentration auf ein optimales Niveau für die Protein-Kristallisation.

Abbildung 8. Proteinkristall Polymerase PB2-Untereinheit von einem Stamm des Influenza-Virus.

Abbildung 9. Röntgen-Beugungsbild der Polymerase-PB2-Untereinheit aus einemStamm des Influenza-Virus.

Abbildung 10. Ribbon Diagramme von den Molekülen in der kristallographischen asymmetrischen Einheit von 4 PB2 Strukturen. Sekundäre Strukturen in farbigen Regenbogen-Muster mit entsprechenden PDB-Codes. (A) 3K2V (A/Yokohama/2017/2003/H3N2) (b) 3KHW (A / Mexiko / InDRE4487/2009/H1N1) (c) 3KC6 (A/Vietnam/1203/2004/H5N1) (d) 3L56 (A/Vietnam/1203/2004/H5N1).

Abbildung 11. Outcome-Analyse für Influenza PB2 Ziele durch die beschriebenen Methoden. Die strukturelle Bestimmung Pipeline wird in fünf Stufen dargestellt: Klonen, Löslichkeit, Reinigung, Kristallisation und Strukturaufklärung.
Diskussion
Multi-Target-Parallel Processing
Structure-based drug design spielt eine wichtige Rolle in der Wirkstoffforschung. Die SSGCID ist bestrebt, die wissenschaftliche Gemeinschaft mit dreidimensionalen Proteinstrukturen von NIAID AC Krankheitserreger gewidmet. Die Bereitstellung solcher strukturellen Informationen allgemein verfügbar wird letztlich dazu dienen, Struktur-basiertes Drug Design beschleunigen.
Der erste wichtige Schritt des MTPP Ansatz ist Konstrukt Design. Mehrere Konstrukte jedes Zielprotein erhöht die Wahrscheinlichkeit einer erfolgreichen Strukturbestimmung und erhöht die Turnaround. Es ist unvermeidlich, dass einige Protein-Konstrukte in Stufen der Pipeline scheitern. Die Umsetzung der PIPE Klonen Methode unterstützt die MTPP Verfahren, indem sie die Erzeugung von vielen Konstrukten in 96-Well-Format ohne arbeitsintensiv Reinigungsschritte. Pairing PIPE Klonen mit der Fähigkeit zur Proteinexpression in der gleichen 96-well-Format (Caliper Lab analysierenChip 90) weiter beschleunigt den Gesamtablauf. Die Paarung dieser Methoden ermöglicht eine schnelle Identifizierung von Konstrukten, lösliches Protein, die den Erfolg von großen Protein-Produktion und Reinigung gewährleistet produzieren.
Ein wesentlicher Aspekt für den Erfolg des MTPP hohem Durchsatz ist die Protein Maker (US-Patent Nr. 6.818.060, Smaragd Bio) Instrument. Das Protein Maker ist ein 24-Kanal-Parallel-Flüssig-Chromatographie-System speziell auf die Leistungsfähigkeit von High-Throughput-Protein-Produktion und die damit verbundenen strukturellen genomischen Forschung Pipeline Anwendungen steigern entwickelt. Mit dem zuvor beschriebenen Protokoll für die Protein-und Teekocher, sind die Vorteile im Vergleich zu einer einzelnen Zeile FPLC-System offensichtlich. Eine einzelne Person kann reinigen bis zu 48 Ziele parallel in einem Zeitraum von acht Stunden. Im Gegensatz dazu kann eine einzelne Person mit einer einzigen Linie FPLC System nur reinigen maximal vier Ziele innerhalb des gleichen Zeitraums. Das hohe Maß an Reinheit für jedes Zielerreicht mit dem Protein Maker sind ein entscheidender Faktor in den späteren Erfolg der wachsenden Protein-Kristalle für die Strukturanalyse.
Einschränkungen und Problembehebung
Lösen dreidimensionaler Strukturen durch Röntgenkristallographie ist ein mehrstufiges Anstrengungen mit vielen Herausforderungen, von denen die Unfähigkeit, um große Mengen von löslichen Zielprotein zu erhalten. Eine Strategie, die implementiert werden, um die Löslichkeit Problem überwunden werden kann, ist die Verwendung einer alternativen Expressionswirt wie E. coli-Zellen sind nicht in der Lage, einige wichtige eukaryotische post-translationale Modifikationen durchzuführen. Expression in verschiedenen Hefe-, Insekten und Säugerzellen, die in der Lage ist die Durchführung dieser post-translationale Modifikationen sind oft eine geeignete Alternative. Zielproteine sind manchmal ausgedrückt aber völlig unlöslich in den Standard Lysebedingungen. Das Protein Maker kann eine wertvolle Ressource für die schnelle Prüfung von alternativen Zelllyse Bedingungennach Smith et al. 2011 13. Diese Strategie ist oft notwendig, um Ziele bewegen durch die Rohrleitung. In jedem strukturelle Genomik Pipeline können standardisierte Protokolle nicht geeignet für jedes Ziel, das durch die Pipeline und Ziele können individuelle Optimierung benötigen kommt. Zum Beispiel haben wir uns entschieden, 20% Ethylenglykol für jeden Frostschutzmittel zu verwenden. In den Fällen, dass diese Bedingung nicht geeignet ist, können alternative Kryoprotektoren oder Konzentrationen getestet werden müssen.
Aufgrund der einzigartigen Natur der einzelnen Protein-Target ist der geschwindigkeitsbestimmende und unberechenbar Schritt bei der Bestimmung einer Struktur Kristallisation. Die MTPP Pipeline gleicht die allgemein niedrige Erfolgsquote der Kristallisation von Proteinen mit der Optimierung von den anfänglichen Sparse-Matrix-Bildschirme. Jede anfängliche Kristall aus kommerziell erhältlichen Sparse-Matrix-Bildschirme getroffen wird weiterhin mit einer E-Screen Builder (Smaragd Bio) optimiert. Die Optimierung Bildschirm ist so konzipiert arTo n den Zustand des ersten Kristalls Hit Änderung der Konzentration der Puffer, Salze und Zusatzstoffe enthalten. Erfolgreiche Optimierung Bildschirme liefern geeignete Kristalle für die Beugung Studien und Strukturbestimmung.
Die strukturelle Genomik Programm, die weiter von der National Institute of Allergy and Infectious Disease (NIAID) stellt Mittel für Bio Smaragd und drei weitere Pacific Northwest Institutionen, die zusammen die SSGCID (Smaragd Bio, SeattleBiomed, der University of Washington und Pacific Northwest National Laboratory) . Jedes Mitglied des Konsortiums wurde für ihr Know-how in der Anwendung state-of-the-art-Technologien für das Erreichen der Ziele der NIAID strukturelle Genomik-Programm benötigt gewählt. Bis heute hat SSGCID 461 Strukturen in der PDB-Ranking als der siebtgrößte Beitragszahler in der Welt abgeschieden und im Jahr 2011, die am produktivsten. Die Protokolle und Methoden der SSGCID werden mit der Absicht, in den Genuss der vorgesehenenWissenschaft und verewigen die Erforschung von Infektionskrankheiten.
Offenlegungen
Die Autoren sind Mitarbeiter von Emerald Bio, Inc.
Danksagungen
Die Autoren bedanken sich bei allen Mitgliedern des Konsortiums SSGCID danken. Erreichung der Ziele SSGCID Möglich wird dies durch die enormen Anstrengungen aller Team-Mitglieder im Emerald Bio. Diese Forschung wurde unter Federal Contract No HHSN272200700057C vom National Institute of Allergy and Infectious Diseases, der National Institutes of Health und dem Department of Health and Human Services finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
Referenzen
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten