Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
RNA Secondary Structure Prediction Mit High-Throughput-SHAPE
In diesem Artikel
Zusammenfassung
Hochdurchsatz-selektiven 2-Hydroxyl-Acylierung durch Primer-Extension (FORM) analysiert verwendet einen neuartigen chemischen Tastertechnologie, reverse Transkription, Kapillarelektrophorese und Sekundärstrukturvorhersage Software, um die Strukturen der RNAs, die von mehreren hundert bis zu mehreren tausend bestimmen Nukleotide an Einzel-Nukleotid-Auflösung.
Zusammenfassung
Das Verständnis der Funktion der RNA in biologischen Prozessen beteiligt erfordert eine gründliche Kenntnis der RNA-Struktur. Zu diesem Zweck wird die Methodik genannt "High-Throughput-selektive 2-Hydroxyl Acylierung durch Primer-Extension analysiert", oder SHAPE ermöglicht Vorhersage von RNA Sekundärstruktur mit Einzel-Nukleotid-Auflösung. Dieser Ansatz nutzt chemische Sondierung Mittel, die Acylierung bevorzugt einzelsträngig oder flexiblen Bereiche der RNA in wässriger Lösung. Seiten der chemischen Modifizierung durch reverse Transkription der RNA modifiziert detektiert und die Produkte dieser Reaktion sind fraktioniert automatisierten Kapillarelektrophorese (CE). Seit Reverse Transkriptase Pausen an diesen RNA Nukleotide durch die Form Reagenzien modifiziert, die resultierende cDNA-Bibliothek indirekt abbildet diese Ribonukleotiden die einzigen im Rahmen der gefalteten RNA gestrandet sind. Mit ShapeFinder Software werden die Elektropherogrammen durch automatisierte CE produziert verarbeitet und in nugonucleotid Reaktivität Tabellen, die sich in Pseudo-Energie-Einschränkungen in der RNAStructure (v5.3) Vorhersage-Algorithmus verwendet wird, umgewandelt. Die zweidimensionalen RNA-Strukturen durch die Kombination FORM Sondieren mit in silico RNA Sekundärstrukturvorhersage erhalten haben, wurde festgestellt, dass viel genauer als Strukturen erhalten mit beiden Methoden allein.
Einleitung
Um die Funktionen der katalytischen und nicht-kodierende RNAs bei der Regulation der Spleißen, Übersetzung, Virusreplikation und Krebs beteiligt zu verstehen, ist eine genaue Kenntnis der RNA-Struktur 1,2 erforderlich. Leider stellt genaue Vorhersage der RNA-Faltung eine gewaltige Herausforderung. Klassische Sondieren Mittel leiden viele Nachteile, wie Toxizität, unvollständige Nucleotidsequenz Abdeckung und / oder den Durchsatz beschränkt auf 100-150 Nukleotide pro Versuch. Unaided Sekundärstruktur Vorhersage-Algorithmen sind ähnlich nachteilig wegen Ungenauigkeiten aufgrund ihrer Unfähigkeit, effektiv unter energetisch ähnlichen Strukturen zu unterscheiden. Große RNAs insbesondere auch oft feuerfesten Verfahren zur 3D-Struktur Bestimmung wie Röntgenkristallographie und NMR-Spektroskopie (NMR), die aufgrund ihrer konformativen Flexibilität und große Mengen an hochreinem Proben für diese Techniken erforderlich.
HIGH-Durchsatz SHAPE löst viele dieser Probleme durch eine effektive, einfache Methode, um die Erforschung der Strukturen von großen RNAs bei single-nucleotide Auflösung. Darüber hinaus sind die Reagenzien für SHAPE verwendet sichere, einfach zu handhaben und im Gegensatz zu den meisten anderen chemischen Reagenzien Sondieren, mit allen vier Ribonukleotiden reagieren. Diese Reagenzien können auch zelluläre Membranen eindringen, was es ermöglicht, RNAs in ihre in vivo-Kontext (e) 3 sondieren. Ursprünglich in den Wochen 4 Labor entwickelt wurde, hat SHAPE verwendet worden, um eine Vielzahl von RNAs, die bemerkenswerteste Beispiel ist die Bestimmung der vollständigen sekundären Struktur des ~ 9 kb HIV-1-RNA-Genom 5 zu analysieren. Andere bemerkenswerte Erfolge mit SHAPE umfassen Aufklärung der Strukturen von infektiösen Viroiden 6, menschlichen langen nicht-kodierenden RNAs 7, Hefe Ribosomen 8 und 9 riboswitches sowie zur Identifizierung von Protein-Bindungsstellen in Virion-assoziierten HIV-1-RNA-3. While das Original und High-Throughput-Variationen des SHAPE-Protokoll wurde bereits an anderer Stelle veröffentlicht 10-12, stellt die vorliegende Arbeit eine detaillierte Beschreibung der RNA Sekundärstruktur Bestimmung durch High-Throughput-SHAPE mit fluoreszierenden Oligonukleotiden, die Beckman Coulter CEQ 8000 Genetic Analyzer, und ShapeFinder und RNAStructure (v5.3)-Software. Bisher unveröffentlichte technischen Details und Hinweise zur Fehlersuche sind ebenfalls enthalten.
Variationen von SHAPE
Das Wesen Form und seinen Varianten ist die Exposition von RNA in wässriger Lösung einer elektrophilen Anhydride, die selektiv acyliert 2'-Hydroxy-(2'-OH)-Gruppen Ribose, wodurch sperrige Addukte an den Stellen der Modifikation. Diese chemische Reaktion dient als Mittel zum Abfragen lokalen RNA strukturelle Dynamik, als einzelsträngige Nukleotide anfälliger für Konformationen förderlich elektrophilen Angriff durch diese Reagenzien erlassen sind, während basengepaarte oder architektonisch bebauteained Nukleotide sind weniger reaktiv oder 10. Seiten der Adduktbildung werden durch reverse Transkription initiiert von fluoreszent oder radioaktiv markierten Primer hybridisiert an eine spezifische Stelle auf der modifizierten RNA (die "(+)" Primer-Extension-Reaktion) detektiert. Als reverse Transkriptase (RT), um die acylierten Ribonukleotide durchqueren fehlschlägt, wird ein Pool von cDNA-Produkte hergestellt werden, deren Längen übereinstimmen mit Seiten der Modifikation. Ein Steuersystem, "(-)" Primerverlängerungsreaktion Verwendung RNA, die nicht Reagenz ausgesetzt wurde auch durchgeführt, so dass eine vorzeitige Beendigung der DNA-Synthese (dh "Haltestellen") aufgrund von Struktur, unspezifische RNA-Strang Bruch etc, kann RNA. Pausieren von durch chemische Modifikation hergestellt wurden, unterschieden werden. Schließlich werden zwei Dideoxy-Sequenzierungsreaktionen Einleiten von denselben Primern als Marker verwendet werden, um reaktive Nukleotide mit dem RNA-Primärsequenz nach Elektrophorese korrelieren.
In der ursprünglichen Anwendung SHAP(-), Und zwei Sequenzierungsreaktionen E, gleich 32 P-end-markiertem Primer für die (+), verwendet. Produkte dieser Reaktionen in benachbarten Vertiefungen in einer 5-8% igen Polyacrylamid-Slab-Gel geladen und fraktioniert durch denaturierende Polyacrylamid-Gelelektrophorese (PAGE; Abbildung 1). Die quantitative Analyse der Gel-Bilder durch herkömmliche Form hergestellt werden unter Verwendung SAFA, ein halbautomatisches Footprint-Analyse-Software 13 ist.
Im Gegensatz dazu verwendet Hochdurchsatz-FORM fluoreszenzmarkierten Primer und automatisierten Kapillarelektrophorese. Genauer gesagt, für jede Region von RNA untersucht, ein Satz von vier DNA Primer eine gemeinsame Sequenz, aber unterschiedliche 5'Fluoreszenzmarkierungen müssen synthetisiert oder gekauft werden. Diese unterschiedlich markierten Oligonukleotide dienen prime zwei SHAPE Reaktionen und zwei Sequenzierungsreaktionen, deren Produkte gebündelt und fraktionierte / erkannt durch automatisierte Kapillarelektrophorese (CE). Whereas die Reaktivität Profil von 100-150 nt von RNA aus einem Satz von vier Reaktionen mit dem ursprünglichen Ansatz erreicht werden kann, ermöglicht Hochdurchsatz-SHAPE Auflösung von 300-600 nt aus einer gepoolten Probe 3. Bis zu 8 Sätze von Reaktionen fraktioniert werden gleichzeitig, jedoch nicht weniger als 96 Proben für die Fraktionierung innerhalb von 12 aufeinander folgenden Läufen CE (2) hergestellt. Darüber hinaus ist die ShapeFinder entwickelte Software zur Verarbeitung und Analyse von Daten, die aus dem CEQ und andere genetische Analysatoren, stärker automatisiert und erfordern viel weniger Benutzereingriff als SAFA 13 oder Gel-Analyse-Pakete.
Weiter fortgeschrittene Hochdurchsatz-Methodik wurden kürzlich wie PARS (parallele Analyse von RNA-Struktur) 14 und Frag-Seq (Fragment-Sequenzierung) 15, die Struktur-spezifische Enzyme anstatt Alkylierungsmittel Verwendung in Verbindung mit der nächsten Generation Sequenzierung Techniken zu erhalten entstanden information über RNA-Struktur. Trotz der Attraktivität dieser Techniken, die viele Einschränkungen inhärent Nuklease Sondieren noch 16 bleiben. Diese Probleme können in Form Sequenzierung (Shape-Seq) 17-Protokoll, wobei Sequenzierung der nächsten Generation durch chemische Modifizierung und reverse Transkription der RNA in einer ähnlichen Weise wie in herkömmliche Form durchgeführt vorausgeht umgangen werden. Obwohl diese Methoden die Zukunft der RNA-Struktur Bestimmung darstellen kann, ist es wichtig, sich daran zu erinnern, dass der nächsten Generation Sequenzierung sehr teuer ist und bleibt für viele Labore.
SHAPE Data Analysis
Die Daten in der Genetic Analyzer erzeugt wird in Form einer Elektropherogramm dargestellt, wobei die Fluoreszenzintensität der Probe (n), der durch die Kapillare Detektor wird gegen einen anderen Zeitpunkt aufgetragen. Dieses Grundstück hat die Form von überlappenden Spuren entsprechend den vier Fluoreszenzkanals verwendet, um die verschiedenen Fluorophoren zu erfassen, und wobei jede Spur aus einzelnen Peaks, die cDNA oder Sequenzierungsprodukte besteht. Elektropherogramm Daten aus dem Genetic Analyzer als Tab-getrennte Textdatei exportiert und importiert in ShapeFinder Transformation und Analyse-Software 18.
ShapeFinder zunächst verwendet, um eine Reihe mathematischer Transformationen an den Daten durchführen, um sicherzustellen, dass die Migration Zeiten und Peakvolumina genau die Identität und Menge der Reaktionsprodukte sind. Peaks werden dann ausgerichtet und integriert, und die Ergebnisse zusammen mit dem primären RNA-Sequenz aufgelistet. A "Reaktivitätsprofil" für den betreffenden Abschnitt des RNA wird durch Subtrahieren Steuerwerte aus dem (+) RNA, die mit jedem Nukleotid verbunden ist, und das Normalisieren der Daten, wie unten beschrieben erhalten. Dieses Profil wird in RNAstructure importiert (v5.3)-Software 19,20, die den normierten Reaktivität val wandeltUES in Pseudo-Energie Einschränkungen, die in der RNA-Sekundärstruktur Faltungsalgorithmus eingebaut sind. Das Kombinieren der chemischen Sondieren und Falten Algorithmen auf diese Weise erheblich verbessert die Genauigkeit der Vorhersage Struktur im Vergleich zu beiden Verfahren allein 12,21. Der Ausgang des RNAstructure (v5.3) enthält Bilder von der niedrigsten Energie RNA Sekundärstrukturen mit der Form Reaktivitätsprofil (en), sowie den gleichen Strukturen in textueller dot-Klammern zu farbcodiert. Letzteres kann anschließend auf Software spezialisiert auf die grafische Darstellung der RNA Sekundärstruktur wie Varna 22 und 23 PseudoViewer exportiert werden.

Abbildung 1. Flussdiagramm der RNA-Struktur Bestimmung über SHAPE 4,10. (A) RNA may aus biologischen Proben oder durch in vitro-Transkription erhalten werden. (B) Je nach Quelle wird RNA gefaltet oder anderweitig verarbeitet und modifiziert mit SHAPE Reagenz. (C) reverse Transkription fluoreszierend oder radioaktiv markierte Primer verwenden. (D) cDNA-Produkte sind fraktionierten entweder über Kapillare oder Slab-Gel-basierten Elektrophorese. (E) Fragment-Analyse. (F) RNA-Struktur-Vorhersage. Klicken Sie hier für eine größere Abbildung zu sehen.
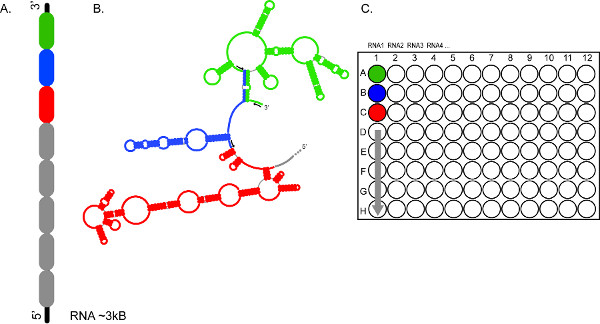
Abbildung 2. Die Hochdurchsatz-Charakter der CE-Form ermöglicht eine schnelle Analyse mehrerer RNAs, und / oder mehrere Segmente der gleichen RNA. (A) Gibt an, wie ein RNA in 300-600 nt Abschnitte (Farbe in grün, blau und rot kodiert) (B) Sektionen der RNA unabhängig sondiert mit verschiedenen Sätzen von fluoreszierenden Primer (schwarze Pfeile) (C) Sets unterteilt werden kann Reaktionen werden vereinigt und in die Vertiefungen A1, B1, C1, etc., die jeweils geladen, wodurch eine vollständige Abdeckung des ~ 3 kb RNA1. Reaktionsprodukte von RNAs 2, 3, 4, usw. können in ähnlicher Weise für die Fraktionierung in aufeinanderfolgenden Läufen Elektrophorese vorbereitet werden. Klicken Sie hier, um eine größere Abbildung anzuzeigen.
Protokoll
Primer-Design und Erweiterung des RNA 3'-Terminus
Um lange RNAs durch High-Throughput-SHAPE analysieren, sollte eine Reihe von Websites, Primer-Hybridisierung so gewählt, dass sie (i) von ~ 300 nt getrennt sind, (ii) sind 20-30 nt Länge, und (iii) dass RNA / werden DNA-Hybride durch Glühen DNA zu diesen Seiten erzeugt eine erwartete Schmelztemperatur> 50 ° C. Darüber hinaus sollte Segmente RNA, die voraussichtlich stark strukturiert werden, vermieden werden, obwohl eine solche Feststellung erfordert eine gewisse Vorkenntnis der RNA-Struktur, die oft nicht verfügbar ist. DNA-Primer, die zu diesen Websites hybridisieren sollte dann entwickelt werden, wobei darauf zu achten, dass sie nicht zu erwarten, dass stabile Dimere oder Intrastrang sekundären Strukturen bilden.
Einmal ausgelegt, so muss entweder Primer-Sets erworben werden (zB von Integrated DNA Technologies, Ames, Iowa) oder 24,25 synthetisiert. Primer mit Cy5, Cy5.5-markierten 5',WellRedD2 (Beckman Coulter) und IRDye800 (Lycor) / WellRedD1 (Beckman Coulter) sind am besten für die Beckman Coulter 8000 CEQ geeignet und bietet eine gute Signalintensität bei gleichzeitiger Minimierung Übersprechen. Markierten Oligonukleotiden kann auf unbestimmte Zeit in kleinen, 10 uM Aliquots bei -20 ° C gelagert werden, vermeiden Sie wiederholtes Einfrieren / Auftauen.
Durch die Verwendung von Primern, die in dieser Weise ausgebildet, ist es möglich, die Form für praktisch eine gesamte RNA von beliebiger Länge zu erhalten. Allerdings 'ist Terminus eines RNA immer unzugänglich Form, falls die RNA entwickelt, um eine 3 enthalten "die Sequenz am oder nahe dem 3'-Terminal-Erweiterung (zB ein" Struktur-Kassette "), an die ein Primer 4. Mai hybridisiert werden.
RNA-Präparation durch Kapillarelektrophorese
Obwohl RNAs aus biologischen Proben für das Hochdurchsatz-FORM verwendet werden kann, wird das Protokoll für die hier angegeben RNA durch in vitro Transkription hergestellt optimiert. Kommerzielle transcription Kits wie MEGAshortscript (Ambion) in Verbindung mit MEGAclear RNA Aufreinigung Spalten (Ambion) verwendet werden, gut geeignet, um große Mengen an reiner RNA erzeugen. RNAs sollte in TE-Puffer zwischen -20 ° C und -80 ° C gelagert werden Für beste Ergebnisse sollte RNAs erscheinen sowohl durch Denaturierung und nicht-denaturierenden Polyacrylamidgelelektrophorese homogen.
1. RNA-Faltung
- In einem 0,5 ml Mikrozentrifugenröhrchen, verdünnte 12 pmol RNA bis 18 ul mit Wasser und fügen Sie 2 ul 10X Renaturierungspuffer. Gut mischen.
- Erhitzen auf 85 ° C für 1 min, dann abkühlen auf 4 ° C mit einer Rate von 0,1 ° C / sec.
- 100 l Wasser und 30 ul 5X Faltungspuffer.
- Bei 37 ° C für 30-60 min, in Abhängigkeit von den RNA gefaltet. Im Allgemeinen erfordern Mg 2 +-abhängigen Faltung länger und strukturierte RNAs längere Inkubationszeiten.
- Übertragen einer 72 ul Aliquot auf jeweils zwei 0,5 ml Reaktionsgefäße: Modified (+) und Kontrolle (-).
2. Chemische Modifikation der RNA
Gut charakterisierte, gehören elektrophile Reagenzien SHAPE Isatosäureanhydrid (IA), N-methylisatoic Anhydrid (NMIA), 1-Methyl-7-Nitro-Isatosäureanhydrid (1M7) 26 und Benzoylcyanid (BzCN) 27. Von diesen sind die am häufigsten für das Hochdurchsatz-SHAPE verwendet 1M7 und NMIA, und nur die letztere ist im Handel erhältlich (Life Technologies). Die Endkonzentration Modifizierungsreagenz muss für jede RNA optimiert werden, um "Single-Hit" Modifikation Kinetik, dh der Zustand, in dem die meisten RNAs in Lösung einmal im Bereich der RNA analysiert 11 modifiziert. Diese optimale Konzentration kann durch die Durchführung mehrerer Reaktionen, bei denen die Konzentration des Reagenz in der Reihe (n) in der Tabelle in Abschnitt 2.1 unten verändert wird bestimmt. Verwenden Sie die Konzentration des Reagenz, das ein leicht nachweisbares Signal erzeugt, während minimizing den Unterschied in der Signalintensität zwischen langen und kurzen DNA-Synthese (z. B. Abbildung 3).
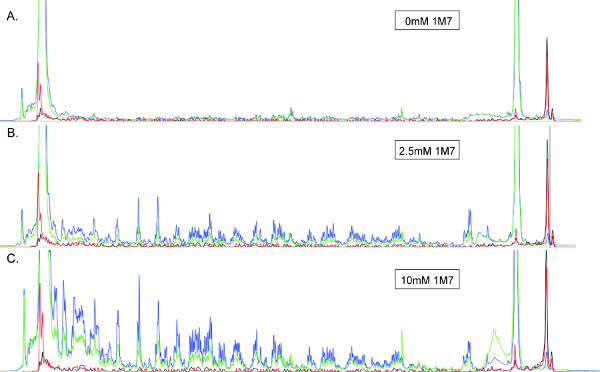
Abbildung 3. SHAPE Elektropherogrammen von einem ~ 360 nt RNA mit (A) 0 (B) 2,5 mm oder (C) 10 mM 1M7 behandelt produziert. Alle Elektropherogrammen sind auf der gleichen Skala angezeigt. Blau, grün, rot und schwarz Spuren auf (+) Reaktionsprodukte (Cy5), entsprechen (-) Reaktionsprodukte (Cy5.5), und die beiden Leitern Sequenzierung (WellRed D2 und IRDye800), beziehungsweise. Die RNA verwendet, um Bild (B) produzieren hat mit der optimalen Menge an 1M7 behandelt worden sind, zeigen gute Peak-Auflösung und Intensität, mit minimalem Signalabfall ganzen trace (links). Lesen Sie beträgt unter diesen Bedingungen maximal. Im Gegensatz dazu ist die Abwesenheit von Medium intensity schlägt gut aufgelöste Peaks in (A) eine sub-optimale Konzentration von 1M7. Umgekehrt zeigt die Signalabschwächung offensichtlich in (c) die einzigen Treffer Kinetik wird nicht beobachtet, und die RNA über modifiziert. In solchen Fällen, insbesondere bei RT nicht zu erwarten, dass das 5'-Ende der RNA-Matrize auftreten, gelesen Länge wird suboptimal.
- Bereiten 10X Lager von SHAPE Reagenz (NMIA oder 1M7). Dies geschieht am besten, indem Sie eine kleine Menge von Reagenz in ein 1,5 ml Mikrozentrifugenröhrchen, dann Zugabe von DMSO, um die gewünschte Konzentration zu erreichen erreicht. Achtung: SHAPE Reagenslösungen bleiben muss, bis wasserfreie mit RNA gemischt. Shop DMSO in einem Exsikkator bei Raumtemperatur und Stammlösungen unmittelbar vor der Verwendung, um die Exposition gegenüber Wasserdampf aus der Umgebung zu minimieren.
Reagenz Optimale Konzentration 10X (in DMSO) Time tovollständiger Abbau von Reagenz 27 NMIA 10-100 mM ~ 20 min 1M7 10-50 mm 70 sec
Tabelle 1. Elektrophile Reagenzien für RNA Modifikation verwendet. - In 8 ul 10x NMIA/1M7 oder wasserfreiem DMSO zu veränderten (+) und Kontrolle (-) mischt, bzw. Hinweis:. 2,5 mM hat sich als ein wirksames Startkonzentration sowohl für NMIA und 1M7 sein, unabhängig von der RNA analysiert.
- Bei 37 ° C für 50 min (NMIA) oder 5 min (1M7), als angemessen.
- Precipitate RNA durch Zugabe von 8 ul (0,1 Vol.) von 3 M NaOAc (pH 5,2), 8 ul 100 mM EDTA, 240 ul (3 Vol.) kaltem Ethanol und 1 ul 10 mg / ml Glykogen. Kühlschrank für 2 Stunden und dann zentrifugiert bei 14.000 g für 30 min bei 4 ° C. Waschen des Pellets zweimal mit kaltem 70% igem Ethanol. Achtung: Es ist wichtig,minimieren Kältetechnik Zeit, Zentrifugation Zeit und Geschwindigkeit, um Co-Fällung von Salz zu minimieren, da dies nachteilig beeinflussen können Peakauflösung während der Elektrophorese.
- Überstand entfernen mit einer Mikropipette und Luft trocknen Pellet für 5 min bei Raumtemperatur.
- Auflösen gefällte RNA in 10 ul TE-Puffer und Inkubation 5 min bei Raumtemperatur. Das ist genug für zwei RNA reverse Transkription Reaktionen gelöst. Bewahren Sie den unbenutzten Teil bei -20 ° C. Achtung: Mechanische Resuspension des Pellets ist in der Regel nicht notwendig und kann die RNA beschädigen.
3. Reverse Transkription
Dieser Schritt erzeugt die fluoreszenzmarkierten cDNA Produkte, die verwendet werden, um indirekt zu ermitteln, in welchem Maße RNA Nukleotide durch eine SHAPE Reagenz geändert wurden, sind. Für SHAPE, war die Leistung von Superscript III (Invitrogen) RT besser als alle anderen getesteten RTs, und ist das Enzym, das für den Einsatz mit ausgewähltenProtokoll. (-) Oligonukleotide mit Cy5 und Cy5.5 gekennzeichnet sind, um die prime (+) und verwendet Reaktionen sind. Für kürzere RNAs werden Primer hybridisiert an ein 3 'terminale Verlängerung des nativen RNA (z. B. ein "Struktur-Kassette"), um Informationen über die 3' zu erhalten Terminus 4 Preis. Von diesem Punkt durch CE, Proben von schützenden Licht.
- Planen (+) und (-)-Proben für die reverse Transkription in 0,5 ml Mikrozentrifugenröhrchen. Für die (+) RT-Reaktion, mischen 5 ul modifizierten RNA (+), 6 ul Wasser und 1 ul Cy5-markierten Primer (10 pM), für die (-) RT-Reaktion, mischen 5 ul Kontroll-RNA (-), . 6 ul Wasser und 1 ul Cy5-markierten Primer (10 pM) ACHTUNG: Sarstedt PCR-Röhrchen (REF 72.735.002) sind für diese Anwendung empfohlen.
- Die Röhrchen in einem Thermocycler und Glühen Primer an die RNA und die Vorbereitungen für die reverse Transkription, indem Sie das folgende Programm: 85 ° C, 1 min; 60 ° C, 5 min;35 ° C, 5 min; 50 ° C halten.
- Während der Glühschritt vorzubereiten genug 2.5x RT-Mix für die Anzahl von Reaktionen durchzuführen, plus 50% (z. B. zwei (+) und zwei (-) Reaktionen im Maßstab 4,5-fach). Eine Reaktion erfordert 8 ul, wie folgt: 4 ul 5x RT-Puffer, 1 ul 100 mM DTT, 1,5 ul Wasser, 1 ul 10 mM dNTPs, 0,5 ul SuperScript III RT. Keep on ice. Achtung: 5X RT-Puffer und 100 mM DTT mit dem SuperScript III RT zur Verfügung gestellt.
- Sobald die Temperatur der Glüh-Mixes erreicht 50 ° C, 8 ul 2.5X RT-Mix auf die (+) und (-)-Reaktionen. Empfehlung: Warm die RT-Mix auf 37 ° C für 5 min, bevor Sie auf die Reaktionen .
- Inkubation für 50 min bei 50 ° C, dann abkühlen auf 4 ° C und / oder auf Eis. Hinweis: Die Inkubation der RT-Reaktionen für länger als 50 min kann in abweichenden cDNA Produkten führen.
- Hydrolysieren RNA durch Zugabe von 1 ul 4 M NaOH und Erhitzen auf 95 ° C für 3 min. Kühle Reaktionen auf Eis und dann neutralisieren sie durch Zugabe von 2 ul 2 M HCl. Achtung: Das Weglassen dieser Schritt führt zu schlechter Qualität Trennung von cDNA-Produkte.
- Kombinieren (+) und (-)-Reaktionen und Ausfällen des cDNA durch Zugabe von 0,1 Vol. 3 M NaOAc, 0,1 Vol 100 mM EDTA, 1,5 Vol. kaltem Ethanol und 1 ul 10 mg / ml Glykogen. Kühlschrank für 2 Stunden, dann bei 14.000 xg zentrifugieren für 30 min bei 4 ° C. Waschen des Pellets zweimal mit kaltem 70% Ethanol Achtung:. Zentrifugation bei höheren Raten oder für einen längeren Zeitraum führt zu Schwierigkeiten Resuspendieren des Pellets (n).
- Sedimentierte cDNA in 40 ul deionisiertem Formamid durch Erhitzen auf 65 ° C für 10 min, durch kräftiges Vortexen für mehr als 30 min folgte. Achtung: Pellets kann unsichtbar sein. Fehlende oder schwaches Signal nach Elektrophorese kann das Ergebnis nicht angemessen das Pellet in dieser Phase.
Sequenzierungsleitern dienen als Marker zur Bestimmung der Nukleotid-Position während der Datenverarbeitung. Diese werden unter Verwendung eines USB-Cycle Sequencing Kit (# 78500), DNA mit der gleichen Sequenz wie die RNA untersucht, und der Primer mit WellRed D2 oder D1/Lycor 800 bezeichnet. Typischerweise wird DNA in dieser Reaktion eingesetzt werden, die als Vorlage für die Transkription der RNA in Frage verwendet. Obwohl die Reaktion Protokoll hier ähnelt, dass den Empfehlungen des Kit-Herstellers, die Reaktion wird bis Mehrfaches skaliert. Während ddA und DDT als Kettenabbrecher in den im Folgenden beschriebenen Reaktionen verwendet werden, kann jedes Paar von Terminatoren verwendet, um die Sequenzierung Leitern erzeugen.
- Mischen Sie 40 ul der ddA Kündigung Mix, 5 pmol DNA-Matrize, 4,6 ul 10X Sequenase Puffer, 10 ul WellRed D2 markierten Primer, 4,6 ul Sequenase und Wasser, um das Gesamtvolumen auf 82 ul zu bringen. In der letzten Sequenase. Prepawieder eine zweite Sequenzierungsreaktion in der gleichen Weise unter Verwendung DDT und Licor IR800 markierten Primer statt.
- Gehen Sie zur PCR-Amplifikation mit USB empfohlenen Bedingungen. Achtung: Die Zugabe von Mineralöl ist nicht erforderlich noch empfohlen für Protokolle / Thermocycler mit beheiztem Deckel verwenden.
- Kombinieren Sie die ddA und ddT Sequenzierungsreaktionen in einem 1,5 ml Mikrozentrifugenröhrchen (~ 164 ul insgesamt).
- Precipitate DNA wie folgt: In 16 ul 3 M NaOAc (pH 5,2), 16 ul 100 mM EDTA, 1 ul 10 mg / ml Glykogen, und 480 ul 95% Ethanol. Gut mischen, bei 4 ° C inkubieren 30 min und Zentrifuge bei 14.000 g für 30 min bei 4 ° C.
- Sedimentierte cDNA in 100 ul deionisiertem Formamid durch Erhitzen auf 65 ° C für 10 min, durch kräftiges Vortexen für mindestens 30 min folgte.
5. Fraktionierung von Reaktionsprodukte durch Kapillarelektrophorese
Kapillarelektrophorese ermöglicht die gleichzeitigeTrennung von cDNA-Synthese aus vier Reaktionen in einer einzelnen Probe gepoolt. Acht Proben fraktioniert werden kann gleichzeitig, während so viele wie 96 Proben können in einem einzigen Durchlauf (Abbildung 2) fraktioniert.
- Mix 40 ul von gepoolten Proben SHAPE mit 10 ul der gepoolten Sequenzierung Leitern, und Transfer zum 96-well Platten Probe. Achtung: Es ist zwingend notwendig, dass Beckman Coulter Reagenzien und Platten (einschließlich LPA-I-Gel-Laufpuffer, Mineralöl, Probe Laden Lösung und Probe und Puffer Platten) mit dem Beckman Coulter-CEQ 8000 Genetic Analyzer verwendet werden.
- Programm und bereiten Kapillarelektrophorese Instrument und initiieren laufen nach Herstellerangaben. Hinweis: Für eine optimale Auflösung von Proben, verwenden Sie die zuvor veröffentlichten CAFA Methodenparameter 28.
Idealerweise außerhalb des Primers und starke Hand Peaks, Signale für jeden Peak in allen vier Elektropherogramm tRennen soll im linearen Bereich liegen, eine allmähliche Abfall der Signal akzeptabel ist. Manchmal sind jedoch große Spitzen (Stationen) auch in der Kontrollreaktion ersichtlich, und diese können mit nachfolgenden Datenverarbeitung stören. Verkürzte cDNAs, die Anlass zu dieser Peaks kann das Ergebnis einer natürlichen Hindernis während der reversen Transkription (zB RNA Sekundärstruktur) oder RNA-Abbau ist. Im ersteren Fall könnte Zusatzstoffe wie Betain verbessern RT Prozessivität und reduzieren RT Pause / vorzeitige Kündigung.
Datenverarbeitung
ShapeFinder Software ermöglicht es dem Benutzer zu visualisieren und zu transformieren CE Spuren und wandeln sie in SHAPE Reaktivitätsprofile 18. Sobald Reaktivität Werte tabellarisch dargestellt werden, werden sie normalisiert und importiert in RNAStructure (v5.3) zu erzeugen und zu verfeinern sekundären strukturellen Modelle.
6. ShapeFinder Software
Eine Verlängerung der BaseFinder Spur Verarbeitung platForm 29, ist die veröffentlichte Version von ShapeFinder frei verfügbar für nicht-kommerzielle Zwecke 18. Detaillierte Anleitungen zur Datenverarbeitung in ShapeFinder werden mit der Software-Dokumentation.
- Elektropherogramme aus dem CEQ in ShapeFinder, wo sie eingestellt werden, um (i) fluoreszierenden Hintergrund zu korrigieren sind, importiert (ii) spektrale Überlappung zwischen fluoreszierenden Kanäle, (iii) Mobilität Verschiebungen durch unterschiedlich markierten Primer vermittelt, (iv) Unterschiede in Intensität der Fluoreszenz gängige Produkte mit verschiedenen Fluorophoren markiert ist, und (v) Signalabschwächung aufgrund vorzeitiger Beendigung der reversen Transkription.
- Das "Setup"-Funktion der "Ausrichten und Integrieren" Werkzeug in ShapeFinder vergibt automatisch Identitäten zu einzelnen Peaks und korreliert diese an die RNA-Sequenz von Benutzereingaben und den beiden Leitern Sequenzierung definiert. Obwohl erste Aufträge in der Regel sind unvollkommen, können Fehler manuell korrigiert werden mit Hilfe der Funktion "Steuern" vondas gleiche Werkzeug. Schließlich berechnet der "Fit"-Funktion die Bereiche unter den ausgerichteten (+) und (-)-Reaktion Peaks und tabellarisch diese Reaktivität Werte zusammen mit dem entsprechenden Nukleotid Nummer in einem Tab-getrennte Textdatei.
Hinweis: Die Analyse der Daten ist entscheidend für die Genauigkeit der SHAPE, und einige Überlegungen sind sehr wichtig in dieser Analyse, einschließlich:
- Signal-zu-Rausch: Die Signal-zu-Rausch-Verhältnis zu sein, dass die einzelnen Peaks sollte leicht erkennbar sein, auch für Positionen mit geringer Reaktivität. Obwohl ShapeFinder eine Datenglättung Möglichkeit, diese Alternative soll äußerst vorsichtig eingesetzt werden, wie es verfälschen können nachfolgende Analyse.
- Region Analyse: In der Regel zuverlässig Daten von cDNAs 300-600 nt lange erhalten werden, wobei an einem Bereich 40-80 nt entfernt von Primer 3 'Terminus und endet wie die Signalverläufe auf ein Niveau schwer von Hintergrundrauschen zu unterscheiden. Verwenden von Multischen Primer-Sets werden benötigt, um längere Strecken von RNA zu analysieren. In diesem Fall empfiehlt es sich, daß die Überlappung in zuverlässige Signalübertragung zwischen Primer-Sets in einem Bereich von 30-50 nt ist. Bei kürzeren RNAs, wobei reverse Transkriptase häufig das Ende der RNA-Matrize, ist darauf zu achten, diese Peaks, deren Signal-Rausch-Verhältnis wird durch die DNA-Synthese stark betroffen Anschlag auszuschließen.
- Signal Zerfall: Der Signalabschwächung in dem Ausmaß der RNA-Modifikation während des Versuchs sowie die unvollkommene Prozessivität RT stehen. Idealerweise sollte ein-hit Kinetik relativ zu dem Bereich der RNA analysiert erreicht werden, um zu maximieren Leselänge werden. ShapeFinder enthält ein Tool, das effektiv bei der Korrektur für Signal Zerfall ist, aber weil dies eher Fehler in der Analyse vorstellen - vor allem, wenn Single-Hit Kinetik nicht eingehalten werden, ist es am besten, wenn Signalabfall minimal ist (dh, wenn die Verteilung der Gipfel steht im Einklang mit Single-Hit kineTics). Kürzlich wurden verbesserte Algorithmen zur Umwandlung Signal Signalabfall wurde 30 veröffentlicht und sollte untersucht werden, ob Signalabfall ist von besonderer Bedeutung in einem bestimmten Experiment werden.
- Signalskalierung. Der wohl willkürliche Schritt in SHAPE Datenverarbeitung, sollte die Steuerung Profil so skaliert werden, dass die Peak-Intensitäten zwischen minimal reaktiven (+) und (-)-Spuren gleich sind. Skalierung der Kontrolle Spur zu einem zu großen Umfang werden in einer Fülle von negativen Reaktivität Werte im ersten Quartil führen (siehe Datennormalisierung unten). In diesem Fall sollte der Skalierungsfaktor entsprechend reduziert werden und die Daten reintegriert.
- Peaks Zuordnung. Im Allgemeinen funktioniert die automatisierte Version des Peakzuordnung gut. Wenn der Prozess ausfällt, ist es jedoch zwingend notwendig, dass der Benutzer, dass alle Peaks von der Software erkannt worden gewährleisten, insbesondere, wenn das Signal-zu-Rausch-Verhältnis niedrig ist. Schulterpeaks beispielsweise nicht immer erkannt wird, und G-reichen seFolgen sind häufig komprimiert.
7. Datennormalisierung
Um Nukleotid Reaktivitätsprofile in die sekundäre Struktur Algorithmus RNAStructure (v5.3)-Software verwendet zu integrieren und / oder zu Profilen von eng verwandten RNAs zu vergleichen, müssen SHAPE Daten in standardisierter Form 12 normiert werden. Dies beinhaltet (i) mit Ausnahme von Ausreißern aus folgenden Berechnungen, (ii) die Bestimmung der "effektive maximale" Reaktivität (dh der Durchschnitt der höchsten 8% der Reaktivität Wert, mit Ausnahme von Ausreißern) und (iii) Normalisierung indem alle Werte Reaktivität die "effektive maximale" wie folgt:
- Öffnen Sie die Registerkarte Textdatei nach der Ausrichtung und Integration erzeugt und kopieren Sie den Inhalt in eine Excel-Tabelle. Die rechte Spalte dieser Datei (RX.area-BG.area) enthält die berechnete absolute SHAPE Reaktivität Werte für jedes Nukleotid der RNA. Die linken Spalten beziehen diese Reaktivität der RNA sequENCE.
- Berechnen und Speichern der ersten und Drittel Quartil (dh der 25. und 75. Perzentile) Werte für (RX.area-BG.area) mit der Excel-Funktion "= QUARTILE (array, Quart)"
- Berechnen und speichern Sie die interquartile Unterschied "= QUARTILE (array, 3)-QUARTILE (array, 1)"
- Berechnen und lagern Sie den "Ausreißer Cutoff-Wert" unter Verwendung der Formel "= (QUARTILE, array, 3) +1,5 * ((QUARTILE (array, 3)-QUARTILE (array, 1))". Alle Reaktivität Werte größer als dieser Wert sind von weiteren Berechnungen ausgeschlossen werden.
- Kopieren Sie die Werte von Reaktivität (RX.area-BG.area) und fügen Sie sie in eine benachbarte, leere Spalte, dann sortieren Sie diese Werte, so dass der größte an der Spitze der Kolonne sind.
- In der neu geschaffenen "sortierten Werte der Spalte" Löschen Werte größer als die Ausreißer Cutoff-Wert.
- Berechne und speichere den Durchschnitt der größten 8% der Reaktivität Werte, die in dem "sortierten Werte column ". Dieser Wert ist die" effektive maximale "Reaktivität.
- Teilen Sie die unsortiert (RX.area-BG.area) jedes Nukleotid (einschließlich Ausreißer) durch den "effektiven maximalen" Reaktivität Wert auf die "normalisierte Reaktivität Werte" zu erhalten. Bewahren Sie diese in einer leeren Spalte, so dass eine leere Spalte auf der linken Seite. Dann kopieren Sie die Nukleotid-Nummern auf der linken Seite der Tabelle und fügen Sie sie in die leere Spalte direkt auf der linken Seite der "normierten Werte Reaktivität".
- Kopieren Sie die Nukleotid-Position-Reaktivität normalisierten Wert-Paare in einem Texteditor.
- Beseitigen Werte unterhalb -0.09 (dh, lassen Sie die Räume leer), da diese wahrscheinlich sind das Ergebnis der RT Pause während der cDNA Synthese aus anderen Gründen als chemische Modifikation der Vorlage. Darüber hinaus irgendwelche Reaktivität Werte für Nukleotide an denen starke Pause auf dem unveränderten Vorlage (wie durch visuelle Inspektion der "Ausrichten und Integrieren" ShapeFinder Profil bestimmt) beobachtet wird, ausgeschlossen werden sollte.
- Save die Datei mit einer. Shape-Erweiterung für den Einsatz in Strukturanalyse mit RNAstructure (v5.3)-Software.
8. Datenmodellierung
RNAstructure (v5.3)-Software wird verwendet, um experimentell unterstützt RNA Sekundärstruktur (s) sagen über die pseudo-freie Energie Zwänge von SHAPE Analyse 19 abgeleitet. Die Software bietet eine grafische Darstellungen der niedrigsten Energie 2D RNA-Strukturen sowie textuelle Darstellung dieser Strukturen in Dot-Notation Halterung. Letzteres kann in einer RNA-Struktur Betrachter von der Präferenz des Benutzers, z. B. 23 oder Pseudoviewer Varna 22, die Veröffentlichung hochwertige Bilder erzeugen importiert werden.
Hinweis: Es muss darauf geachtet, wenn man die Strukturen von der RNAstructure (v5.3)-Software hergestellt werden. Zum Beispiel kann die Software nicht beheben tertiären Wechselwirkungen wie Pseudoknoten und küssen Schleifen, noch kann es zu unterscheiden, ob Mangel anReaktivität in einer bestimmten Region ist durch Basenpaarung oder sterischen Schutz von Proteinen. Als Folge dieser Faktoren zusammen mit den Energien der einzelnen Strukturen berichtet, sind bei der Darstellung eines endgültigen Strukturmodell werden.
Ergebnisse
RNA, die die HIV-1-rev-Response-Element (RRE) und eine 3'-terminale Struktur Kassette 4 aus einem linearisierten Plasmid durch in vitro Transkription hergestellt, wonach es durch Erhitzen, Kühlen, und die Inkubation wurde bei 37 ° gefaltet C in Gegenwart MgCl 2. RNA wurde NMIA belichtet und dann von einem 5'-Ende markierten DNA-Primer hybridisiert das 3'-terminale Struktur Kassette revers transkribiert. Die resultierende Form cDNA-Bibliothek, zusammen mit Steuerungs-und Seque...
Diskussion
Wir stellen Ihnen hier eine detaillierte Protokoll für High-Throughput-SHAPE, eine Technik, die sekundäre Struktur Bestimmung erlaubt single-nucleotide Auflösung für RNAs jeder Größe. Außerdem Kopplung experimentellen Daten mit SHAPE Sekundärstruktur Vorhersage-Algorithmen ermöglicht Generation von RNA 2D-Modelle mit einem höheren Grad an Genauigkeit, als es mit beiden Methoden allein. Die Kombination von fluoreszenzmarkierten Primern und automatisierte CE bietet erhebliche Vorteile gegenüber dem herkömmlich...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch und SFJ Le Grice werden durch die Interne Research Program des National Cancer Institute, National Institutes of Health, USA unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
Referenzen
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten