Method Article
Eine erschwingliche HIV-1 Drug Resistance Überwachungsmethode für Ressourcen Begrenzte Einstellungen
In diesem Artikel
Zusammenfassung
Arzneimittelresistenz Tests für HIV-1-infizierten Personen in Ermangelung einer antiretroviralen Therapie (ART) können zukünftige Therapien führen und die Behandlungsergebnisse zu verbessern. Optimierung der individuellen und der Gesundheit der Bevölkerung Ergebnisse in hoher HIV-Prävalenz, aber mit beschränkten Ressourcen wird letztlich erfordern erschwinglich und zugänglich Resistenzen Genotypisierung und Interpretationsmethoden.
Zusammenfassung
HIV-1-Resistenzen hat das Potenzial, ernsthaft gefährden die Wirksamkeit und die Auswirkungen der antiretroviralen Therapie (ART). Wie ART-Programme in Afrika südlich der Sahara weiter ausbauen, sollten die Betroffenen auf ART eng für die Entstehung von Resistenzen überwacht werden. Überwachung der Arzneimittelresistenz übertragen, die Übertragung von Virusstämme bereits resistent gegen ART verfolgen ist ebenfalls kritisch. Leider ist Drogenresistenztests immer noch nicht in der Ressourcen begrenzte Einstellungen leicht zugänglich, weil die Genotypisierung ist teuer und erfordert anspruchsvolle Labor-und Datenmanagement-Infrastruktur. Ein offener Zugang genotypische Resistenzüberwachung Drogen Methode, um Personen zu verwalten und zu bewerten übertragen Resistenzen beschrieben. Das Verfahren nutzt kostenlose Open-Source-Software für die Auslegung des Arzneimittelresistenzmuster und die Erzeugung von individuellen Patientenberichte. Die Genotypisierung Protokoll weist eine Verstärkungsrate von mehr als 95% für die Plasmaproben mit avIRAL Last> 1.000 HIV-1 RNA Kopien / ml. Die Empfindlichkeit nimmt deutlich für Viruslast <1.000 HIV-1 RNA Kopien / ml. Das hier beschriebene Verfahren wurde gegen eine Methode der HIV-1-Resistenzen Prüfung durch den US-amerikanischen Food and Drug Administration (FDA), der ViroSeq Genotypisierungsmethode genehmigt validiert. Einschränkungen der hier beschriebenen Verfahren umfassen die Tatsache, daß es nicht automatisiert ist und daß es auch nicht zu den zirkulierenden rekombinanten Form CRF02_AG aus einer Gruppe von Validierungsproben verwendet, obwohl es verstärkt Subtypen A und B aus der gleichen Platte.
Einleitung
Die HIV-Epidemie im südlichen Afrika wurde rasch entwickelnden 1 mit einer gleichzeitigen exponentiellen Anstieg der Personen, die antiretrovirale Therapie (ART), vor allem in Südafrika, 2, 3. Als Beweise für die epidemiologische Auswirkungen von großen Behandlungsprogramme bei der Verringerung der Inzidenz 4 und der steigenden Lebenserwartung in begrenzten Ressource-Einstellungen (RLS) 5 weiterhin zu akkumulieren, werden Bemühungen um die Abdeckung zu erhöhen ART intensiviert werden. Die Entwicklung von Richtlinien zur Nutzung der Behandlung als Prävention Werkzeug 6, 7 unter der Test-und Behandlungsprogramme bedeutet, dass die absolute Zahl der Personen, auf die Behandlung wird weiter zunehmen. Eine große Anzahl von Personen auf ART für längere Zeit als die durchschnittliche Lebenserwartung der Menschen auf ART nähert, dass der HIV-infizierten Bevölkerung 8 sein. Die Entwicklung und Übertragung von HIV Resistenzen hat always worden, als eine Bedrohung für die Leistungen der ART 12.09. Somit besteht ein Bedarf für eine strengere Überwachung und Kontrolle der Arzneimittelresistenz als mehr Menschen auf ART eingeleitet.
Genotypische Resistenztestung Drogen (BRT) wurde erfolgreich in den entwickelten Ländern verwendet worden, sowohl für die Überwachung sowie die Überwachung der HIV-1-Resistenzen in Personen erhalten ART. In diesen Einstellungen hat GRT in die Kontinuität der Betreuung für HIV-1-infizierten Personen integriert. Die meisten internationalen Leitlinien empfehlen BRT für Erwachsene oder pädiatrische Patienten, bei denen ART (First-Line-und Second-Line) 13-15, anschließend infizierte pädiatrische Patienten, die Prävention der Mutter-zu-Kind-Übertragung (pMTCT) Therapien ausgesetzt, sondern 16, und in den Einstellungen mit Hochebenen übertragen Medikamentenresistenz bei akut infizierten Personen 13-15. Allerdings haben die Kosten, Technologie-und Infrastrukturanforderungen der Umsetzung beschränkttation von ähnlichen Ansätzen zur Arzneimittelresistenz Überwachung in RLS.
Die südafrikanische HIV-Behandlung und Überwachung von Richtlinien derzeit nicht empfehlen die Verwendung von BRT in Führungs Wahl der ART für Personen, andernfalls First-Line-Regimen 17. Personen überwiegend auf virologische (HIV-1-RNA-Viruslast) Parametern umgeschaltet. Doch im Jahr 2012, veröffentlichte der Southern African HIV Kliniker-Gesellschaft die ersten Southern African ARV Resistenzen Prüfrichtlinien 18. Diese Richtlinien empfehlen GRT Test für alle Erwachsenen in Ermangelung First-Line-und Second-Line-ART und für infizierte Säuglinge und Kinder bis 18 pMTCT ausgesetzt. Es wird jedoch nicht empfohlen GRT 18 für akut infizierten Personen, weil es keine aktuellen Belege für hohe übertragen Medikamentenresistenz im südlichen Afrika 19-29. Es wird erwartet, dass einige dieser Empfehlungen werden im Laufe der Zeit in die nationale tre integriert werdenatment Richtlinien und Überwachung der verschiedenen Länder in der Region. Bereits in den 2013 South African Behandlungsrichtlinien gibt es jetzt Empfehlung des GRT zum Zeitpunkt der Second-Line-Ausfall für Erwachsene und bei der ersten oder zweiten Linie PI-basierten Regime Ausfall für Kinder 30.
Es hat sich gezeigt, dass die Einbeziehung in die Behandlungsrichtlinien GRT in Südafrika würde möglicherweise kostenneutral sein. In Anbetracht der Kosten der zweiten Zeile Therapie Medikamente, die relativ teurer als die erste Zeile Drogen sind, mit GRT um Patienten, die wirklich brauchen, um zu Zweitlinientherapie identifizieren schaltet werden nicht führen keine zusätzlichen Kosten für das Programm. Darüber hinaus können GRT auch andere Gründe für das Scheitern zu identifizieren, zu sparen Behandlungsmöglichkeiten und generieren Informationen zu neuen Resistenzmuster 31. Daher ist es notwendig, die Kosten der Arzneimittelresistenz Überwachungsmethoden noch weiter, um den Zugang, die Qualität der Versorgung zu verbessern eine Reduzierungd Ergebnisse.
Hier präsentieren wir eine Methode entwickelt, um GRT generische (Open Source) Primer für Reverse Transkription Polymerase-Kettenreaktion (PCR) und Sequenzierung (Tabelle 1) sowie vor allem Open-Source-Software für die Medikamentenresistenz Interpretation zu verwenden. Für die klinische Management, wird das Protokoll durch eine umfassende Überprüfung und Berichterstattung Verfahren mit Fach Interpretation der Labor Resistenzen Bericht mit engen Einhaltung der nationalen Behandlungsrichtlinien ergänzt. Das Protokoll ist in vier Komponenten unterteilt: 1) HIV-Ribonukleinsäure (RNA)-Extraktion, 2) Reverse Transkription und Polymerase-Kettenreaktion (PCR) Amplifikation von viralen Targets 3) Sequenzierung und 4) Bioinformatics Verfahren zur Analyse von Chromatogrammen, Ausrichtung, Kuration und Interpretation der Sequenzdaten.
Protokoll
1. Ethylendiamintetraessigsäure (EDTA)-Vollblut-Verarbeitungs
HINWEIS: Unmittelbar nach Sammlung können bei 4 ° C für nicht länger als 24 h gelagert werden kann Blut verarbeitet werden.
- Die Arbeit in einem biologischen Sicherheitsschrank, damit der EDTA-Vollblut Probe auf Raumtemperatur zu erreichen.
- Für jede Probe beschriften genug Kryoröhrchen mit der Probe-Identifikation (ID), Speichermaterial (Plasma) und Datum.
- Zentrifugieren der Proben für 10 min bei 1.000 x g. Verwenden Sie keine Bremsen Zentrifuge stoppen. - Eine sehr dünne Schicht - und Erythrozyten, Thrombozyten einschließlich Plasma, Leukozyten (Buffy-Coat): Dies wird drei Schichten (von oben nach unten) zu erhalten.
- Den Überstand (Plasma) und 500 ml Aliquot vorsichtig absaugen in jedes Kryoröhrchen. Achten Sie darauf, um die Zellschicht (Buffy-Coat) zu stören oder übertragen alle Zellen.
- Lagern bei -80 ° C bis zur RNA-Extraktion benötigt oder sofort die RNA-Extraktion.
- Bereiten Sie eine Extraktion Arbeitsblatt mit den IDs der Proben extrahiert werden, die positive und negative Plasma-Kontrollen.
- Für jede Probe extrahiert werden, kennzeichnen eine 1,5 ml sterilen Mikrozentrifugenröhrchen mit der Probe-ID, Datum und Extraktion "RNA". Auch kennzeichnen einen zusammengesetzten Spalten-und Sammelrohr sowie eine 2 ml Mikrozentrifugenröhrchen mit Arbeits Lyse-Lösung mit entsprechenden Zahlen aus der Gewinnung Arbeitsblatt.
- Arbeiten in der Bio-Sicherheitsschrank, 200 ul Probe in die entsprechenden 2 ml Mikrozentrifugenröhrchen Arbeits Lyse-Lösung.
- Vortex gut und Inkubation für 10 min bei Raumtemperatur.
- Nach 10 min Zentrifugieren das Rohr kurz.
- Add 800 ml absolutem Ethanol zu jeder der Röhren.
- Mix von Puls Verwirbelung und kurz Zentrifuge.
- Transfer 600 ul dieser Lösung zu der entsprechenden Spalte / Sammelröhrchen-Baugruppe. Zentrifugieren bei 6.000 xg für 1 min.
- Übertragen Spalte an eine neue Sammelröhrchen und entsorgen Sie die alte Sammlung Rohr mit dem Filtrat. Wiederholen Sie den obigen Schritt 2.8 (oben) zweimal.
- In 500 ul Waschpuffer AW1 jeder Spalte und Zentrifuge bei 6000 g für 1 min.
- Entsorgen Sie das Filtrat und Sammelrohr und übertragen Sie die Spalte an eine neue Sammelröhrchen.
- In 500 ul Puffer AW2 war und Zentrifuge bei 20.000 g für 3 min. Wiederholen Sie Schritt 2.11.
- Zentrifuge in ein neues Auffanggefäß bei 20.000 × g für weitere 2 min.
- Entsorgen Filtrat und Ort Säule in 1,5 ml Mikrozentrifugenröhrchen.
- Man gibt 60 &mgr; l Puffer AVE (RNase-freies Wasser) zu der Mitte der Kolonne gewährleistet, dass man die Flüssigkeit nicht verzichten nicht auf der Seite der Säule.
- Inkubieren bei Raumtemperatur für 1 min.
- Zentrifugieren bei 6.000 g für 2 min.
- Entsorgen Sie die Spalte und die Kappe die 1,5-ml-Mikrozentrifugenröhrchen.
- Die Proben sind nun bereit für reverse Transkription.
- Wenn der Test bei 4 º C für 6 h durchgeführt werden, sofort, zu speichern. Wenn jedoch Tests verzögert werden dann bei -80 º C unmittelbar zu platzieren. NB: nicht einfrieren / auftauen die Proben mehr als 3x.
3. Vorbereitung der Reagenzien für Reverse Transkription
- Vor dem Start, berechnet das Volumen von jeder der für die Anzahl der Proben, die verarbeitet, einschließlich der positiven und negativen Kontrollplasmen benötigten Reagenzien. Fügen Sie auch ein Reagenz Kontrolle.
- Unter Verwendung der berechneten Mengen aus Schritt 3.1 (oben), bereiten die Desoxyribonukleotidtriphosphat (dNTP)-Primer-Mix in einer sauberen, sterilen 200 ul PCR-Röhrchen, gefolgt von kurzen Impuls Verwirbelung. Jede Probe sollte 0,5 ul der Reverse-Primer RT21 und 0,5 ul der dNTP haben, siehe Tabelle 2.
- Aliquot 1,0 ul der dNTP-Primer-Mix zu 200 ul PCR-Röhrchen.
- Vorbereitung der Reversen Transkriptase (RT) Enzymmischung durch Zugabe von 1 μ, L 10x Reverse Transkription Puffer, 1 &mgr; l 0,1 M DTT und 2 ul 25 mM MgCl 2 in ein steriles Röhrchen, gefolgt von Vortexen und kurz zentrifugiert, siehe Tabelle 3.
- In 0,5 ul jedes der Enzyme RNAseOUT und Superscript III Reverse-Transkriptase-Enzym-Mix auf die Röhre tippen Sie dann auf das Rohr vorsichtig mischen.
- Halten Sie die Rohre mit den dNTP-Primer-Mixe und Enzym-Mix auf einem kalten Block-und an der RNA-Station.
4. Reverse Transkription
- In 6 ul der RNA-Probe auf die dNTP-Grundierung, gefolgt von Mischrohr kurz Vortexen mischen.
- Nach der Zugabe der RNA, zu bewegen, um die PCR-Zimmer sowohl mit dNTP / Primer / RNA-Enzym-Mix und RT Rohre mischen an einem kalten Block oder Eis.
- Kurz zentrifugieren die dNTP / Primer / RNA-Mix Rohre (aus Schritt 4.2) und legen Sie sie in einem Thermocycler.
- Hitze bei 65 ° C für 5 min, um die RNA zu denaturieren.
- Schnelles Abkühlen auf 4 ° C, halten Sie für 2Minute
- Halten Sie den Thermocycler, während immer noch bei 4 ° C; nehmen Sie die Rohre.
- Schnelles Hinzufügen von 5 ul der Enzym-Mix, während die Rohre auf einem Kühlblock.
- Vorsichtig mischen, indem Sie den Schlauch dann kurz die Röhrchen zentrifugiert und zum Thermocycler.
- Halten der Röhrchen bei 50 ° C für 60 min, um Reverse Transkription der RNA-Enzym, gefolgt von einer Denaturierung bei 85 ° C für 5 min, um die reverse Transkription zu beenden.
- Kühlen auf 37 ° C Sobald die Temperatur wird auf 37 ° C, Pause und nehmen Sie den Schlauch aus dem Thermocycler.
- Schnelles Hinzufügen von 0,5 ul RNase H zu den Rohren und zum Thermocycler.
- Halten bei 37 ° C für 20 min und dann abkühlen auf 4 ° C
- Die komplementäre DNA (cDNA) kann sofort verwendet oder bei -20 ° C oder kälter gelagert, bis sie benötigt werden. Jedoch sollte die Langzeitlagerung von cDNA bei -80 ° C.
5. Vorbereitung der Reagenzien für die PCR
- BefoWiederstart Berechnen der Volumina von jeder der für die Anzahl der Proben, die verarbeitet und die Steuerelemente erforderlichen Reagenzien. Zusätzlich zu den drei Kontrollen (Positiv, Negativ, und Reagenz), können Sie auch eine PCR-Kontrolle (HIV DNA) hinzufügen. Die erste und die zweite Runde PCR Mischungen können gleichzeitig hergestellt werden, und das zweite Master-Mix bei -20 ° C gelagert, bis sie benötigt werden. Mixes kann für ca. 8 Stunden gespeichert werden.
- Hinzufügen 18,4 ul Wasser 2,5 ul 10x-Puffer, 1,0 &mgr; l MgCl 2, 0,5 &mgr; l dNTPs und 0,25 &mgr; l von jedem der Primer, wie in Tabelle 4 gezeigt, und Wirbel.
- In 0,1 ul Platinum Taq-Polymerase (5U/μl) und vorsichtig das Rohr durch Antippen.
- Aliquot 23 ul des Master-Mix zu 200 ul PCR-Röhrchen.
- Mit den Master-Mix Röhrchen auf einem Kühlblock oder Eis Umzug in die PCR-Zimmer.
6. Nested PCR
- In 2 ul der cDNA zu 23 ul der ersten Runde der PCR master mischen.
- Schließen der Rohre, legte die Proben im Thermocycler und die Verwendung der folgenden PCR-Zyklusbedingungen: 94 ° C für 2 min, 30 Zyklen von 95 ° C für 30 sec, 58 ° C für 20 sec, und 72 ° C für 2 min, gefolgt von einer abschließenden Verlängerung bei 72 ° C für 10 min wie in Fig. 1 gezeigt.
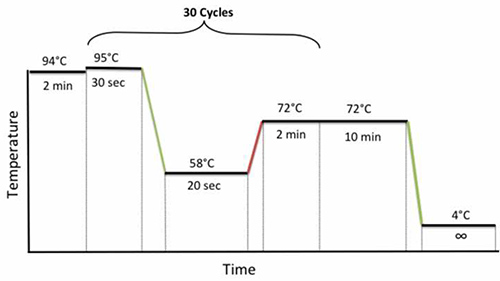
Fig. 1 ist. Nested PCR-Zyklusbedingungen. Klicken Sie hier für eine größere Ansicht.
- Fahren Sie mit der 2. Runde PCR Bühne oder die erste Runde der PCR-Produkte bei -20 ° C oder kälter, bis zu einem späteren Zeitpunkt erforderlich.
- Zum zweiten PCR-Runde, fügen Sie 2 ul der 1. Runde PCR-Produkt auf 23 ul der 2. Runde PCR Master Mix eind das gleiche PCR-Programm auf Abbildung 1.
7. Gel-Elektrophorese
- Gel-Vorbereitung
- Hinzufügen eines 0,5 g Agarose Tablette in einen 250 ml-Glaskolben eingewogen und mit 50 ml 1x TBE-Puffer in den Kolben gegeben.
- Hitze in der Mikrowelle zum Kochen; wirbeln häufig (ca. alle 30 Sekunden), bis vollständig solubilisiert. Verwenden Sie einen Silikongriff oder Silikonofenhandschuh, um die heiße Flasche greifen. Die Agarose-Lösung aus dem Kolben sehr leicht so eng kochen, diesen Prozess zu überwachen.
- Kühlen bei Raumtemperatur für etwa 10 min.
- Gießen Sie in ein Agarose Gelgießschale, die geeignete Größe Kamm; Gel ist bereit, in ca. 20-30 min zu verwenden.
- Zeigen Gel-Elektrophorese in Kammer und laufen, wie vom Hersteller empfohlen.
- Gel-Elektrophorese und Visualisierung.
- Vortex Novel Saft für 10 sec vor der Verwendung.
- Verdünnen Sie 1 ul der Novel-Saft mit 5 ul von DNA Probe und Mix.
- Verdünnen 3 ul Novel Saft mit 3 ul Molekulargewichtsmarker und mischen.
- Laden Sie die Mixe aus den Abschnitten 7.2.2 und 7.2.3 (oben) und führen Sie das Gel bei 100 V und 400 mA für 40 min, um die PCR-Amplifikation zu evaluieren.
- Positive Verstärkung kann unter UV-Licht als 1315 bp-Fragment, Abbildung 2 visualisiert werden.
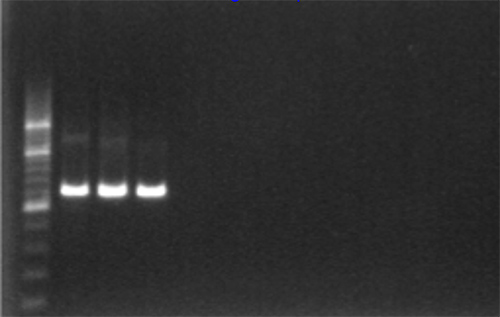
2. Gel Bestätigung der PCR-Amplifikation mit 1% Agarose-Gelelektrophorese und eine 200 bp Leiter. Klicken Sie hier für eine größere Ansicht.- Es sollte keine Verstärkung in den Negativ-und Reagenzienkontrollen sein, so dass keine Kontamination anzeigt.
8. PCR-Produktbereinigung
- In Vorbereitung der Sequenzierungsreaktion werden die positiven zweiten Runde PCR-Produkte unter Verwendung der Purelink PCR Purification Kit gereinigt.
- In 80 ul der Arbeits Binding Buffer High-Cutoff (B3) bis 20 ul PCR-Produkt und Pipette Mix.
- Hinzufügen der Probe mit dem Bindungspuffer auf eine Spin-Säule in ein Sammelröhrchen gemischt.
- Zentrifugieren der Säule bei 10.000 × g für 1 min. Übertragen Sie die Säule in ein neues Sammelröhrchen.
- Mit 650 ul Waschpuffer mit Ethanol Die Säule.
- Zentrifugieren der Säule bei 10.000 × g für 1 min. Übertragen Sie die Säule in ein neues Sammelröhrchen.
- Zentrifugieren Sie die Spalte bei maximaler Geschwindigkeit für 2-3 min, um restliche Waschpuffer zu entfernen.
- Setzen Sie die Spin-Säule in ein sauberes 1,7 ml Elution Rohr mit dem Kit geliefert.
- 40 &mgr; l Elutionspuffer zum Zentrum der Säule und Inkubieren der Säule bei Raumtemperature für 1 min.
- Zentrifugieren der Säule bei maximaler Geschwindigkeit für 2 min (> 10.000 x g).
- Die Elution Tube enthält Ihre gereinigte PCR-Produkt bereit für die Sequenzierung. Entsorgen Sie die Spalte.
- Bestimmen Sie die Konzentration und Qualität der DNA mit Hilfe eines Nanodrop.
- Wenn keine in-house-Sequenzierung Einrichtungen verfügbar sind, können die gereinigten PCR-Produkte zu einem kommerziellen Labor-Sequenzierung zu diesem Zeitpunkt gesendet werden.
9. Sequenzierungsreaktionen
- Die PCR-Produkte werden mit der großen Dye Terminator Kit-Version 3.1 und 4 Primer für jede Probe (zwei Vorwärts-und zwei Rückwärts) sequenziert. Die Primersequenzen sind in Tabelle 2 gezeigt. Deshalb wird nach der Sequenzierung Sicht wird jede Probe vier Sequenzen in ein Contig assembliert werden.
- Stellen Sie die Sequenzierungsreaktionen, wie in Tabelle 5 für jede der vier Primer angegeben.
- Mischen Sie die Sequenzierungspuffer und der Primer durch Vortexen vor dem Gebrauch.
- Mischen Sie dieWasser, Puffer und Grundierung vor der Zugabe des Big Dye-Sequenzierung. Mischen durch Vortexen.
- Vorsichtig mischen den Master-Mix nach der Zugabe des großen Farbstoff Sequenzierung Mix durch Umdrehen der Röhre oder leichte Schläge.
- 9 ul Aliquot des Master-Mix in eine 96-Well-Platte aus optischen.
- Um 24 Proben / Platte, bis die Platte wie unten 3 angedeutet laufen.
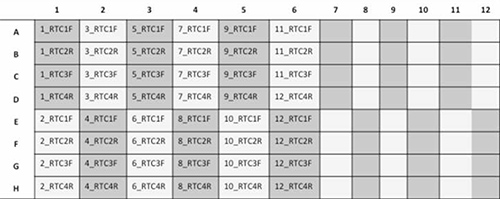
3. Schema-Darstellung einer 96-Well-Platte mit 12 Patienten, die Proben mit jeweils 4 Primer (RTC1F, RTC2R, RTC3F und RTC4R) sequenziert. Klicken Sie hier für eine größere Ansicht.
- 1.0 ul der DNA-Probe (~ 20-40 ng) wird die Platte mit einemn Kleber Aluminium-Deckel und dann vorsichtig mischen.
- Zentrifugieren bei 3000 g für 1 min. Entfernen Sie die Aluminiumabdeckung und fügen Sie ein Gummidichtungsmatte.
- Legen Sie die Platte auf dem Thermocycler und führen Sie das in Abbildung 4 dargestellt folgende PCR-Programm.
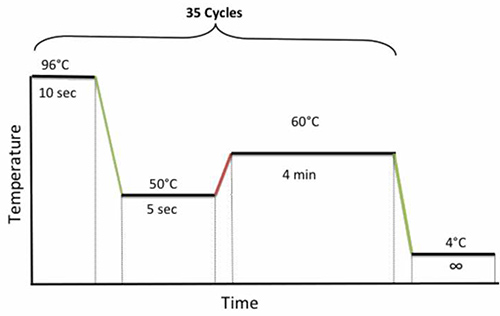
4. PCR-Zyklusbedingungen für die Sequenzierung. Klicken Sie hier für eine größere Ansicht.
- Wenn die PCR beendet ist, reinigen die Sequenzierung Produkt sofort.
10. Sequenzierung Cleanup
- Für jede Sequenzierungsreaktion, Mischung 50 ul absolutem Ethanol und 5 &mgr; l 3 M Natriumacetat.
- Mit einer Mehrkanal-Pipette 55 &# 956; l Natriumacetat / EtOH-Lösung zu jedem Well.
- Siegel Brunnen mit Alu Abdeckung, so dass jeder auch richtig abgedichtet.
- Zentrifugieren bei 3000 × g für 20 min.
- Nach 20 min Deckel abnehmen und die Platte umdrehen, in einer fließenden Bewegung, auf einem gefalteten Labor Gewebe (NICHT schlagen, um der Überstand loszuwerden, da dies die Pellets lösen!).
- Zentrifugieren der invertierten Platte auf demselben Gewebe bei 150 × g für 2 min.
- Sofort im 150 ul kaltem 70% EtOH. Zögern Sie nicht Zugabe von Ethanol bei diesem Schritt.
- Dichtung mit dem gleichen Klebstoff Aluminium-Deckel und Wirbel.
- Zentrifugieren bei 3000 × g für 5 min.
- Kehren Platte auf eine neue gefaltete Gewebe und Zentrifuge bei 150 g für 1 min invertiert.
- Nach der Zentrifugation in Thermocycler aufgedeckt und trocknen Sie es bei 50 ° C für 2 min.
- Sobald die Platten trocken ist, verschließen Sie diese mit Folie deckt, in Folie wickeln und bei -20 ˚ C bis bereit zu gehen with die Sequenzierung Elektrophorese.
- Wenn Sie bereit sind, um Sequenz auflösen gereinigt Sequenzierungsprodukte in 10 ml Hallo-Di Formamid, denaturieren und Belastung für die Elektrophorese.
11. Bioinformatik
- Sequence Assembly
- Starten Sie das Programm Geneious.
- Erstellen Sie einen Ordner, um die Arbeitsabläufe zu speichern.
- Importieren Sie die Dateien, die vom ABI-Sequenzierung Maschine in den Arbeitsordner mit dem Import-Tool generiert. Geneious Prozentsatz wird der Qualitätsfaktor für jede Sequenz importiert zuordnen.
- Offene Sequenzen mit Qualitätswerte> 70% durch einen Doppelklick auf sie.
- Jede Datei wird in einem neuen Fenster geöffnet. Die Software wird die Qualität bei jedem Nukleotid-Position des Chromatogramms der Sequenz Qualität mit hellblauen Balken zeigen. Je höher der Balken, desto besser die Qualität des Grund Anruf.
- Mit dem Cursor wählen Sie den mittleren Abschnitt der Sequenz lassen sich die Enden, die in der Regel von schlechter Qualität sind.
- Klicken Sie auf die Schaltfläche Extrakt, um die Region mit guter Qualität Folge zu extrahieren.
- Wählen Sie alle vier Sequenzen extrahiert für jede Probe und montieren sie gegen eine Referenzsequenz.
- Überprüfen Sie die Reihenfolge zusammengesetzt, um sicherzustellen, dass Sie in der richtigen Leserahmen sind. PQITLW: Wenn Sie in der richtigen Leserahmen sind, sollte der Beginn der Protease mit den folgenden Aminosäuren starten. Der Beginn der RT wird mit PISPIE starten.
- Entpacken Sie die Fläche zu Region, die den Beginn der PR auf den 300. RT-Codon. Während dieses Prozesses auch für Insertionen oder Deletionen überprüfen.
- Gehen Sie durch die Konsensus-Sequenz des extrahierten Fläche zu, die Identifizierung Unklarheiten und überprüfen Positionen mit gemischten Basen durch Einsicht Qualität (Symmetrie, Größe, Hintergrund und Schultern der flankierenden Regionen) der Grundgespräche.
- Wählen Sie die Konsensus-Sequenz und klicken Sie auf den Extrakt-Taste, um eine separate Datei von der Konsensus-Sequenz aus den vier Primer erstellen und label sie entsprechend.
- Exportieren Sie die Sequenz zu einem Backup-Speicher-Ordner auf dem Computer oder einem Netzwerkordner.
- Sequence Quality Assessment (HIVDB)
- Analysieren Sie die Sequenz mit der HIVDB Programm http://hivdb.stanford.edu .
- Prüfen Sie, ob Deletionen und Insertionen in den zusammengefassten Daten und sicherzustellen, dass die Sequenz umfasst alle 99 Protease (PR)-Codons und die ersten 300 RT-Codons.
- Überprüfen Sie alle markierten Qualitätssicherung (QA) Fragen sowohl in der PR-und RT-Regionen, wie Stop-Codons, Rahmenverschiebungen, mehrdeutig Positionen und ungewöhnliche Rückstände.
- Sequenzierung Qualitätskontrolle
- Sprengen Sie die neue Sequenz gegen eine lokale Sequenz-Datenbank von vorherigen Lauf.
- Wenn die neue Sequenz ist> 97% wie jede Sequenz in der Datenbank sollten alle Stufen des Protokolls überprüft werden, beginnend mit der Sequenzanalyse und geht zurück auf die RNA-Extraktion, dieausgee, dass es keine Verwechslungen (Probe-Switching, Falschetikettierung) oder Verunreinigung.
- Wenn keine Probleme erkannt werden, wiederholen Sie die Analyse sowohl der alte und neue Proben aus der RNA-Extraktion Bühne.
- Wenn die Sequenzen sind immer noch> 97% ähnlich, überprüfen Sie die Patientengeschichte, für jede epidemiologische Verbindung zwischen den Menschen zu beurteilen.
- Die phylogenetische Analyse
- Richten Sie alle Sequenzen aus der Datenbank mit ClustalW-Programm in Geneious.
- Manuelles Überprüfen Sie die Ausrichtung für falsch ausgerichtete Sequenzen, Deletionen und Insertionen und bearbeiten Sie entsprechend.
- Konstruieren Sie einen phylogenetischen Baum mit PHYML, Geneious Tree Builder oder anderen Baumbauer in Geneious.
- Untersuchen Sie den Baum für Proben mit kurzen Verzweigungslängen.
- Überprüfen Sie die Proben mit kurzen Verzweigungslängen für eine mögliche Kontamination.
12. REGA DB Informatik
- Sequence hochladen
- Melden Sie sich beim RegadB mit einem eindeutigen Benutzernamen und ein Passwort.
- Auf dem Dropdown-Menü, unter der Patienten-ID, wählen Sie "Beginnt mit".
- Fügen Sie die Patienten-ID, und wählen Sie die Person, deren Genotyp hochgeladen werden.
- Klicken Sie im Menü auf der linken Seite, wählen Sie "viralen Isolat".
- Von den Optionen unter viralen Isolat wählen "Hinzufügen".
- Geben Sie die Beispiel Datum, Proben-ID, ID-Sequenz und Sequenz Datum.
- Wählen Sie "Datei auswählen" und navigieren Sie zu der Datei fasta der Sequenz, die hochgeladen werden dann.
- Nach der Auswahl der Datei fasta, die hochgeladen werden, klicken Sie auf Upload.
- Sobald die hochgeladenen Sequenz erscheint in der Box unter den Nukleotid-Sequenz identifiziert und Daten, klicken Sie auf die OK-Taste an der rechten unteren Ecke des Fensters.
- Prüfen Sie, ob PR-und RT-Protein-Ausrichtung, indem Sie auf die Schaltfläche und wählen Protein entweder PR oder RT.
- Überprüfen Sie für das Medikament Resistenz-Mutation durch Klicken auf die Schaltfläche Widerstand. Das gibtSie die Resistenzprofile von drei Algorithmen: ANRS, Stanford und HIVDB RegaDB.
- Berichterstellung mit REGA
- Melden Sie sich beim RegaDB mit Ihrem persönlichen Benutzernamen und Passwort.
- Auf dem Dropdown-Menü, unter der Patienten-ID, wählen Sie "Beginnt mit".
- Fügen Sie die Patienten-ID, und wählen Sie die Person, deren Bericht generiert werden.
- Klicken Sie im Menü auf der rechten Seite, wählen viralen Isolat.
- Von den Optionen unter viralen Isolat klicken Sie auf "Ansicht".
- Klicken Sie doppelt auf das virale Isolat, für die Sie einen Bericht erstellen möchten.
- Auf der viralen Isolat Fenster klicken Sie auf der Registerkarte Virus Isolat Bericht.
- Wählen Sie die Algorithmen für die Interpretation des Genotyps aus dem Dropdown-Menü und wählen Sie Berichtsvorlage zu benutzen.
- Sobald der Algorithmus und der Vorlage ausgewählt haben, klicken Sie auf die Schaltfläche "erzeugen".
- Laden Sie die rtf-Dokument erzeugt.
- Öffnen Sie das tun, rtfcument als Word-Dokument.
- Ändern der Größe der Behandlungsgeschichte Diagramm.
- Nach der Tabelle, fügen Sie den Abschnitt "Klinische Chart und Widerstand Interpretation".
- Unter Verwendung der Daten auf der Widerstandstabelle und der klinischen Diagramm eine Beschreibung für Widerstandsprofil des Patienten, beginnend mit der Krankengeschichte des Patienten und die Arzneimittel, auf die die virale Isolat resistent. Fügen Sie auch eine Beschreibung der Viruslast des Patienten und die CD4 +-Zellzahl-Profile aus dem Diagramm.
- Senden Sie den Bericht zu den Infektionskrankheiten (ID) Spezialist für die Überprüfung und Empfehlungen für künftige Patientenmanagement. Dieser Prozess ist auch ein sehr wichtiger Qualitätssicherung Bühne. Alle Fehler im Erbgut oder Inkonsistenzen in der Behandlung der Geschichte, kann virologischen und immunologischen Profilen identifiziert und überprüft werden, bevor eine endgültige Bericht gesendet wird, mit all den Empfehlungen, die dem Kliniker die Verwaltung der Patienten.
Ergebnisse
Die validierte Verfahren war eine Modifikation eines früher berichteten Verfahren 20. Die ViroSeq Genotypisierung Methode, die von der FDA zugelassen wurde, wurde als Referenzmethode in der Validierung. Ein Panel von Eignungsprüfungsproben aus den Französisch Nationalen Agenturen für Forschung über Aids und Hepatitis (ANRS) erhalten in der Primär Vergleich zwischen den beiden Methoden. Die beiden Genotypisierung Methoden waren bei der Identifizierung aller klinisch relevanten Arzneimittelresistenz-assoziierten Mutationen wie von der HIVDB Programm für die Proben, die erfolgreich von beiden Methoden verstärkt wurden interpretiert 100% übereinstimm. Wie in Tabelle 6 gezeigt, waren die Nukleotidsequenzen der drei Paare 99,5% identisch. Die vorhergesagten Aminosäuresequenzen zu 100% identisch. Eine Probe von fünf nicht erfolgreich durch ViroSeq verstärkt werden. Neben der Probe nicht durch ViroSeq verstärkt, habe die Hausmethode, eine zweite Probe, die gezeigt wurde, verstärkenein umlauf rekombinanten Virus (CRF02_AG) durch ViroSeq sein. Die drei Proben, die mit beiden Methoden verstärkt waren Subtyp B (zwei Proben) und Subtyp A (eine Probe).
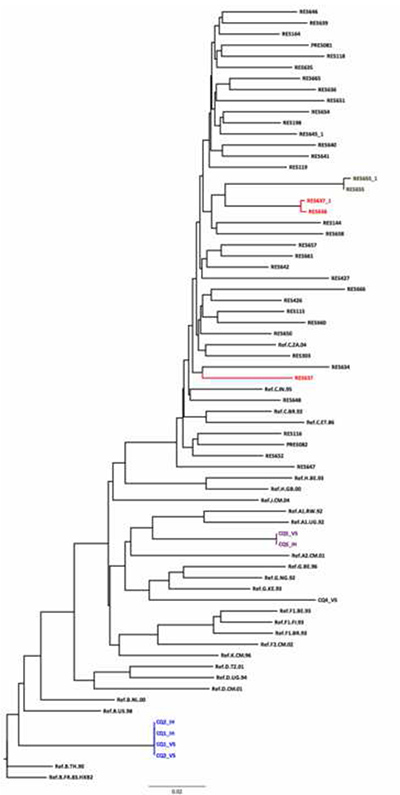
5. Verwendung eines HKY Nachbar Joining Baum als Teil der Qualitätssicherung Reihenfolge getan. Es gibt vier Paare / Cluster-Sequenz mit sehr kurzen genetischen Distanzen. Der genetische Abstand zwischen RES655 und RES655_1 (gleiche Proben an verschiedenen Tagen sequenziert) beträgt 0,003. Das ist eine potenzielle Fehler mit dem RES637_1/RES638 Paar als ihre genetische Distanz ist zu kurz (0,075) für Proben aus verschiedenen epidemiologisch nicht verknüpften Personen. Es ist ein weiteres RES637 auf dem Baum mit einem Abstand von 0,075 im Vergleich zu RES638_1. Die CQ01/CQ02 Cluster nahe, dass die zwei Probenvon der Platte sind Duplikate der gleichen Probe. Sie Cluster zusammen mit dem Subtyp B Referenzsequenz Bestätigung der Subtyp von der REGA Subtypisierung Werkzeug zugeordnet. CQ05 und CQ04 mit Subtypen A und G jeweils gruppierten, während die REGA Subtypisierung Werkzeug klassifiziert sie als A und CRF02_AG sind. Ein weiteres nützliches Tool für die HIV-Subtypen und Rekombination SCUEL, die an http://www.datamonkey.org ist. Klicken Sie hier für eine größere Ansicht.
Ein Panel von fünf Proben wurde verwendet, um die Genauigkeit der Hausmethode beurteilt. Zehn Wiederholungs Genotypen wurden für jede der fünf Proben erzeugt. Verwendung der Kapillare 16 3130xl Genetic Analyzer, 48 der 50 Genotypen von 24 Läufen erzeugt wird, am gleichen Tag hergestellt. Für alle fünf Proben wurden die vorhergesagten Aminosäuresequenzen zu 100% übereinstimm zwischen Wiederholungen. Für die Nukleinsäuresequenzen, there war> 99% paarweise Ähnlichkeit.
Während der ersten zwei Jahre der Anwendung dieses Verfahrens, sechzig Proben wurden zufällig aus RNA-Extraktion zur Sequenzierung wiederholt. Es gab keine statistisch signifikanten Unterschiede zwischen der Sequenz Qualitätsfaktor und der Anzahl der gemischten Basen zwischen den Wiederholungen. Sowohl die Nukleotid-und Aminosäurepaarweise Vergleiche der sechzig Paare waren mehr als 99% identisch ist. So waren die Arzneimittel-Resistenz-Mutationen für alle Paare 100% übereinstimm.
Kostenreduzierung
Die Reaktionsvolumina für RT-PCR und Sequenzierung wurden mindestens die Hälfte, bezogen auf das ursprüngliche Verfahren 20, 32 reduziert wird, ohne auf die Qualität der erzeugten Sequenzen. Dies ermöglichte eine Verringerung der Kosten von 50% für die RT-und PCR-Stufen.
Die neue Methode wurde ursprünglich entwickelt, um mit sechs Sequenzierprimer Sequenz arbeiten Alle 99 Codons des Protease-Gens und die ersten 300 Kodons des Reverse-Transkriptase-Gen 20, 32. Ähnliche Verfahren auch sechs bis acht Primer 33, 34. Einige kürzlich veröffentlichten Methoden sind weniger als sechs Primer verwendet, wenn auch manchmal die Sequenzierung der Protease-und RT-Gene seprately 35, 36. Wir versuchten, die Anzahl von Sequenzierungsprimer von sechs auf vier zu reduzieren (Fig. 6)
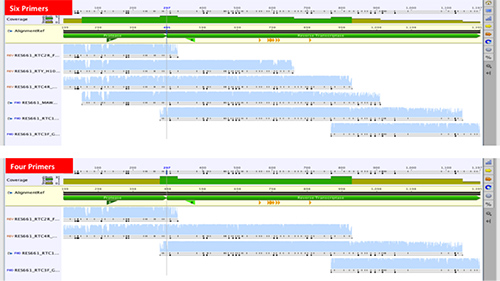
6. Vergleich von aufeinanderfolgenden Sequenzen von sechs vs vier Sequenzierungsprimer für die Erzeugung des 1197 bp pol-Sequenz, die alle 99 HIV-1-Protease-Codons und die ersten 300 Kodons des Reverse-Transkriptase-Gens.242/51242fig6highres.jpg "target =" _blank "> Klicken Sie hier für eine größere Ansicht.
Sequenzen aus einem Satz von 17 Proben von sechs Primern erzeugt wurden, Sequenzen nach dem Ausschluss von zwei Primern (MAW46 und RTY) erzeugt, verglichen. Die Subtypen waren 14 Subtyp C, zwei Subtyp B und einen Subtyp A. Es gab keine signifikanten Unterschiede in der Sequenz Qualitätswerte. Auch die durchschnittliche paarweise Identität zwischen den 17 Paaren von Nukleinsäure betrug 99% und 100% auf der Aminosäureebene. Daher ist die Verringerung der Sequenzierprimer von sechs auf vier zu einer Reduktion in der Sequenzierungskosten um fast ein Drittel.
Das einzige, proprietäre Software-Tool in diesem Protokoll verwendet wurde, war für Geneious Sequenz Montage. Die Medikamentenresistenz Interpretationswerkzeuge, sowie der Bericht Erzeugung Tools sind alle kostenlos, Open-Access-Tools. Dies verringert die Kosten durch den Wegfall der Kosten, die mit der Verwendung proprietärer Software verbunden. Ferner collectivE Verhandlungen erlaubt die Reagenzien für dieses Protokoll in einem Kit für den einfachen Zugriff von Life Technologies verpackt werden und ist als Saturn / Life Technologies Genotypisierung Verfahren 37 zur Verfügung. Darüber hinaus können Mitglieder Saturn die Reagenzien zu einem vergünstigten Preis zu gelangen.
Klinische Einstellung
Das beschriebene Protokoll wurde in der Kontrolle und Überwachung der Medikamentenresistenz in einer ländlichen Gemeinde in der Provinz KwaZulu-Natal umgesetzt. Insgesamt 604 Genotypen wurden aus klinischen Proben zwischen Dezember 2010 und Mai 2013 bei einer Verstärkung von 95% für Proben mit Viruslast> 1.000 RNA Kopien / ml erzeugt. Diese klinische HIV-Arzneimittelresistenz Studie wurde von der Ethikkommission Biomedical Research der Universität von KwaZulu-Natal (Ref. BF052/10) und des Gesundheitsforschungsausschuss der KwaZulu-Natal-Abteilung der Gesundheit (vgl. HRKM 176/10) zugelassen. Individuelle Patientenberichte wurden erzeugt und zurück zu den Kliniken geschicktzum Patientenmanagement.
Siebzig zwei (72) Genotypen wurden auch als Teil eines Überwachungs der übertragenen Resistenzen Studie erzeugt, innerhalb einer großen prospektiven populationsbasierten Studie HIV-Überwachung verschachtelt. Die primären Proben waren Nadelstich Vollblut in EDTA-Röhrchen gesammelt. Bei Genotypisierung gab es eine Verstärkungsrate von 79% 19. Ethik-Zulassung für die Genotypisierung von Proben aus der Surveillance-Studie wurde von der Universität von KwaZulu-Natal Biomedical Research Ethics Committee (siehe BE066107) erhalten.
| Primer Namen | Folge | Länge | Richtung | HXB2 Position | |
| MAW-26 | TTGGAAATGTGGAAA GGAAGGAC | 23 | Vorwärts | 2028-2050 | Ersten PCR-Runde |
| RT-21 | CTGTATTTCAGCTATC AAGTCCTTTGATGGG | 31 | Umge | 3539-3509 | Ersten PCR-Runde |
| Pro-1 | TAGAGCCAACAGCCC cacca | 20 | Vorwärts | 2147-2166 | 2. Runde PCR |
| RT-20 | CTGCCAATTCTAATTC TGCTTC | 22 | Umge | 3462-3441 | 2. Runde PCR |
| RTC1F | ACCTACACCTGTCAA CATAATTG | 23 | Vorwärts | 2486-2508 | Die Sequenzierung |
| RTC2R | TGTCAATGGCCATTG TTTAACCTTTGG | 27 | Umge | 2630-2604 | Die Sequenzierung |
| RTC3F | CACCAGGGATTAGAT ATCAATATAATGTGC | 30 | Vorwärts | 2956-2994 | Die Sequenzierung |
| RTC4R | CTAAATCAGATCCTAC ATACAAGTCATCC | 29 | Umge | 3129-3101 | Die Sequenzierung |
| RT-y | GTGTCTCATTGTTTAT ACTAGG | 22 | Umge | 2967-2946 | Die Sequenzierung |
| MAW-46 | TCCCTCAGATCACTC TTTGGCAACGAC | 27 | Vorwärts | 2251-2277 | Die Sequenzierung |
Tabelle 1. Reverse Transkription, PCR und Sequenzierung individuelle Primer bei der Erzeugung eines 1197 bp pol-Fragment, das alle 99 HIV-1-Protease-Codons und die ersten 300 Kodons des Rückwärts trascriptase Gens verwendet.
| RT21 (5pmol/ml) | 0,5 | 0,2 |
| dNTP (10 mM) | 0,5 | 0,4 |
| Gesamt | 1 |
Tabelle 2. dNTP / Primer mischen für die reverse Transkription Reaktion.
| Reagens | Volumen (ml) / Reaktion | Konzentration / Reaktion |
| First Strand Buffer (10x) | 1 | 1 |
| MgCl 2 (25 mM) | 2 | 4 |
| DTT (0,1 M) | 1 | 0,008 |
| RNaseOUT (40 U / ml) | 0,5 | 16 |
| Superscript Reverse Transcriptase III (200U/ml) | 0,5 | 8 |
| Gesamt | 5 |
Tabelle 3. Enzym-Mix für die reverse Transkription Reaktion.
| Reagens | Volumen (ml) / Reaktion | Endgültige Konzentration / Reaktion |
| DEPC behandeltem Wasser | 18,4 | - |
| PCR-Puffer (10x) | 2.5 | 1 |
| MgCl 2 (50 mM) | 1 | 2 |
| dNTP-Mix (10 mM) | 0,5 | 0,2 |
| Froward Primer (5 pmol / ml) | 0,25 | 0,05 |
| Rückwärts-Primer (5 pmol / ml) | 0,25 | 0,05 |
| Platinum Taq-Polymerase (5 U / ml) | 0,1 | 0,02 |
| Zwischensumme | 23 | - |
Tabelle 4. Master-Mix für die verschachtelte PCR.
| Reagens | Volumen (ml) / Reaktion | Konzentration / Reaktion |
| DEPC behandeltem Wasser | 6.1 | |
| Sequenzierungspuffer (5x) | 2 | 1 |
| Primer (3,2 pmol / ml) | 0,5 | 0,16 |
| Big Dye Terminator Sequencing-Mix | 0,4 | - |
| Gesamt | 9 |
Tabelle 5. Mastermix für den Sequenzierungsreaktionen.
| ViroSeq | Sonstige Innen | NA% Ähnlichkeit | |||||||
| Proben-ID | Unterart | Quality Score | PR-Mutationen | RT-Mutationen | Unterart | Quality Score | PR-Mutationen | RT Mutationen | |
| CQ01 | B | 99,9 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99,2 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ02 | B | 99,5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99,5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ03 | NA | NA | NA | NA | NA | NA | |||
| CQ04 | CRF02_AG | 98,4 | I54V, V82F, I84V | M41L, L74I, L210W, T215Y, V108I, Y181C | NA | NA | NA | NA | NA |
| CQ05 | A | 99,7 | K103N | A | 93 | K103N | 100 | ||
Tabelle 6. Vergleichende Results von einer parallelen Analyse zwischen dem ViroSeq Genotypisierung Methode und der Hausmethode unter Verwendung einer Reihe von Proben durch die ANRS Verfügung gestellt.
Diskussion
Mehrere kostengünstige Inhouse-Verfahren wurden in den Bemühungen, versuchen HIV-Arzneimittelresistenz Genotypisierung erschwinglicher 33, 34, 36, um beschrieben. Es besteht kein Zweifel an der Notwendigkeit, Drogenresistenztests in die Kontinuität der Betreuung für Personen, die antiretrovirale Therapie mit beschränkten Ressourcen zu integrieren. Allerdings konzentrieren sich die meisten der berichteten Verfahren über die Anwendung der Arzneimittel-Resistenz Genotypisierung bei der Überwachung der Arzneimittelresistenz bei einer Bevölkerungsebene. Der Saturn / Life Technologies Genotypisierungsmethode ist eine vollständig integrierte Protokoll für die Überwachung und Kontrolle der Medikamentenresistenz. Diese Methode wurde entwickelt, um eine kostengünstige Umsetzung Protokoll meist Open Source und Open Access Bioinformatik-Ressourcen für die Interpretation von Resistenzen und die Erzeugung von Berichten für die klinische Management.
Es wurde durch den Vergleich mit der FDA zugelassen ViroSeq Genotypisierungsmethode zu sein gezeigtpräzise bei der Identifizierung von Arzneimittelresistenzmutationen von einem Gremium von ANRS Eignungsprüfungen Proben in 100% der Laborproben Tafel, die erfolgreich verstärkt wurden. Die Genauigkeit wurde auch an klinischen Proben von Subtyp-C-Viren, die dominierende Subtyp im südlichen Afrika untersucht. Das Verfahren wurde so genau auf Subtyp C-Proben wie am Subtyp A und B. Wenn das Verfahren würde in anderen Teilen der Welt weit verbreitet ist, wo CRF02_AG verwendet werden, gibt es einen Bedarf zur Modifizierung der Primer da das Verfahren gegen eine der Plattenproben, die gezeigt wurde, haben CRF02_AG verstärken. Alternativ kann ein degenerierten Primer empfindlich auf alle Viren der Gruppe M 33, 36 könnten in Regionen eingesetzt werden, wo der Subtyp Verteilung heterogener ist 38.
Die Empfindlichkeit der reverse Transkription und PCR kann durch Extrahieren von RNA aus höheren Plasmavolumina wie 500 ml erhöht werden. Das Plasma kann CENTRIF werdenbei 21.000 g für 90 min uged, um die Viruspartikel, bevor mit dem Protokoll, wie durch die virale RNA-Extraktion QIAamp Mini Kit beschrieben fortfahren konzentrieren.
Wie gezeigt, hat das neue Verfahren den zusätzlichen Vorteil, dass sie umfassende Berichte zur individuellen Patientenbehandlung erzeugt. Diese Berichte sind eine Konsolidierung des Genotyps, der immunologischen und virologischen Überwachungsdaten sowie klinische und Behandlungsgeschichte von RegaDB. Dies wird durch eine detaillierte Interpretation der Laborresistenzprofil, gefolgt von einem ebenso detaillierte Überprüfung der Krankengeschichte des Patienten sowie Behandlungsempfehlungen begleitet. Die Verwendung von einem Facharzt, um die Berichte zu überprüfen und Behandlungsempfehlungen für die Patienten bietet die dringend benötigte Mentoring für Krankenschwester Praktiker sowie unerfahrene Kliniker, die zunehmend die Bereitstellung sind ART in Südafrika als Teil der WHO-Empfehlungen für die Aufgabenverschiebung. Diese klinischenBerichte haben gezeigt, dass effektive Lernhilfen für Ärzte mit wenig oder keiner Erfahrung in der Arzneimittelresistenzmanagement sein. Aus Patientensicht, reduziert unser Verfahren die Notwendigkeit, zur zentralen Standorten reisen, um HIV-Spezialist Dienste zuzugreifen.
Somit ist die beschriebene Protokoll als Ganzes bietet eine gute Plattform, durch die HIV-Arzneimittelresistenz-Management integriert werden können, zu einem erschwinglichen Preis, in der Kontinuität der Betreuung für HIV-infizierten Personen in Ermangelung ART. Die erzeugten Daten können für epidemiologische Zwecke verwendet werden, um die Entwicklung und die Übertragung von Resistenzen in der Gemeinde zu beurteilen. Die Größe des pol-Fragment erzeugt wird, ist gut genug für komplexere phylogenetischen Analyse, die ein besseres Verständnis der Epidemie auf die Bevölkerung produzieren wird.
Offenlegungen
Diese Arbeit wurde durch den Wellcome Trust (082384/Z/07/Z), Europäische Union (SANTE 2007 147-790), des US-Zentrums für Krankheiten unterstützt Kontrolle über CAPRISA (Projekttitel: Stärkung der Gesundheitssysteme und HIV Behandlungsfehler (HIV- TFC)) und die Schweizer South African Joint Research Programme (SSJRP) Forschungsförderung mit dem Titel "Swiss Prot / Südafrika: Protein Bioinformatics Resource Development für wichtige gesundheitsbezogenen Krankheitserreger." RL wird durch den Wellcome Trust unterstützt (Fördernummer 090999 / Z / 09 / Z). Die Geldgeber hatten keine Rolle in Studiendesign, Datenerfassung und Analyse, Entscheidung zur Veröffentlichung oder Vorbereitung des Manuskripts. Die Autoren erklären, keine finanziellen Interessen konkurrieren.
Danksagungen
Die Autoren danken allen Kolleginnen und Kollegen, die diese Arbeit möglich gemacht, vor allem Maya Balamane, Elizabeth Johnston Weiß, Sharon Sjoblom, Greg Ording Zakhona Gumede, Xolile Kineri, Phindile Mabaso, Lungisa Ndwandwe, James Garvey, Gavin Cobb, Senzo Maphanga, Terusha Chetty anerkennen , Kevi Naidoo, Andrew Skingsley, Katharine Stott und Lungani Ndwandwe. Die Autoren möchten auch alle Mitarbeiter der Abteilung für Gesundheit und Africa Centre Personal, das Hlabisa HIV-Behandlung und Pflege-Programm arbeiten zu danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Superscript III 1st strand Synthesis kit | Life Technologies | 18080051 | Reverse Transcription |
| SATURN/LiFE Technologies Custom Primers | Life Technologies | 4473517 | PCR |
| Platinum Taq | Life Technologies | 10966026 | PCR |
| PureLink QUICK PCR Purification Kit | Life Technologies | K310002 | PCR |
| Viroseq | ABBOTT | 4J94-20 | Reverse Transcription and PCR |
| Agarose Tablets (Dnase/Rnase free) | BIOLINE | BIO-41027 | PCR |
| TBE Buffer | MERCK | 1.06177.2500 | PCR |
| O'Range Ruler 200 bp DNA Ladder | Fermentas | FE SM0633 | PCR |
| Novel Juice | GeneDireX | LD001-1000 | PCR |
| MiniBis Bioimaging System | DNR Bioimaging Systems Ltd | Gel Documentation | |
| Power Pac 300 | BIORAD | Gel Electrophoresis | |
| Big Dye Terminator Kit version 3.1 | Life Technologies | 4337456 | Sequencing |
| Arrays | Life Technolgies | 4319899 | Sequencing |
| PoP | Life Technologies | 4363785 | Sequencing |
| 10x EDTA Buffer | Life Technologies | 402824 | Sequencing |
| Formamide | Life Technologies | 4311320 | Sequencing |
| 5x Sequencing Buffer | Life Tecgnologies | 4336697 | Sequencing |
| 3130 xl Genetic Analyzer | Life Technologies | Sequencing | |
| GeneAmp PCR System 9700 | Life Technologies | RT/PCR/Sequencing | |
| Centrifuge 5804 | EPPENDORF | Sample Processing | |
| Centrifuge 5415R | EPPENDORF | RNA Extraction | |
| Centrifuge 5415R | EPPENDORF | RT and PCR | |
| Centrifuge 5415D | EPPENDORF | PCR Product Clean up | |
| Centrifuge 5810 | EPPENDORF | Sequencing Clean up | |
| Picofuge | BIORAD | C1301-230V | RT and PCR |
| Vortex Genius 3 | IKA | RNA extraction and reagent preparation | |
| Vortex mixer | IKA | Sequencing Cleanup | |
| NanoDrop 2000 UV/VIS spectrophotometer | ThermoScientific | DNA quantification | |
| 3 M Sodium Acetate | MERCK | 567422 | Sequencing Clean up |
| Absolute Ethanol | MERCK | SAAR2233540LP | Sequencing Cleanup |
| 1.5 ml SARSTEDT Tubes | BIODEX | 72.692.005 | RNA Extraction |
| 2 ml SARSTEDT Tubes | BIODEX | 72.693.005 | RNA Extraction |
| 2 ml Collection tubes | SCIENTIFIC GROUP | MCT-200-NC/S | RNA Extraction |
| Optical MicroAmp 96-well reaction plates | Life Technologies | N8010560 | Sequencing |
| 200 µl 8 Strip StarPCR Tubes with attached flat caps | STAR Lab - supplied by CELTIC | A1402-3700 | RT and PCR |
| 200 µl PCR individual tubes | Scientific Group | CR/3745 | RT and PCR |
| Geneious | Biomatters | Sequence analysis | |
| Internet Access | Preferrably high speed | ||
| Web resources | |||
| hivdb.stanford.edu | Stanford University | Drug reistance analysis | |
| http://bioafrica.mrc.ac.za:8080/regadb-ui/RegaDB | SATuRN | database | |
| http://bioafrica.mrc.ac.za/tools/pppweb.html | SATuRN | Sequence quality tool | |
Referenzen
- Shao, Y., Williamson, C. The HIV-1 epidemic: low- to middle-income countries. Cold Spring Harbor Persp. Med. 2, (2012).
- Mutevedzi, P. C., et al. Scale-up of a decentralized HIV treatment programme in rural KwaZulu-Natal, South Africa: does rapid expansion affect patient outcomes. Bull. World Health Organ. 88, 593-600 (2010).
- Houlihan, C. F., et al. Cohort profile: Hlabisa HIV treatment and care programme. Int. J. Epidemiol. 40, 318-326 (2011).
- Tanser, F., Barnighausen, T., Grapsa, E., Zaidi, J., Newell, M. L. High coverage of ART associated with decline in risk of HIV acquisition in rural KwaZulu-Natal. South Africa. Science. 339, 966-971 (2013).
- Bor, J., Herbst, A. J., Newell, M. L., Barnighausen, T. Increases in adult life expectancy in rural South Africa: valuing the scale-up of HIV treatment. Science. 339, 961-965 (2013).
- Montaner, J. S., et al. The case for expanding access to highly active antiretroviral therapy to curb the growth of the HIV epidemic. Lancet. 368, 531-536 (2006).
- Granich, R. M., Gilks, C. F., Dye, C., De Cock, K. M., Williams, B. G. Universal voluntary HIV testing with immediate antiretroviral therapy as a strategy for elimination of HIV transmission: a mathematical model. Lancet. 373, 48-57 (2009).
- Johnson, L. F., et al. Life expectancies of South african adults starting antiretroviral treatment: collaborative analysis of cohort studies. PLoS Med. 10, (2013).
- Blower, S., Ma, L., Farmer, P., Koenig, S. Predicting the impact of antiretrovirals in resource-poor settings: preventing HIV infections whilst controlling drug resistance. Curr. Drug Targets. 3, 345-353 (2003).
- Geretti, A. M. Epidemiology of antiretroviral drug resistance in drug-naive persons. Curr. Opin. Infect. Dis. 20, 22-32 (2007).
- Larder, B. A., Darby, G., Richman, D. D. HIV with reduced sensitivity to zidovudine (AZT) isolated during prolonged therapy. Science. 243, 1731-1734 (1989).
- Erice, A., et al. Brief report: primary infection with zidovudine-resistant human immunodeficiency virus type 1. N. Engl. J. Med. 328, 1163-1165 (1993).
- Williams, I., et al. British HIV Association guidelines for the treatment of HIV-1-positive adults with antiretroviral therapy. HIV Med. 13 Suppl 2, 1-85 (2012).
- DHHS, . US Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. , (2012).
- Vandamme, A. M., et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS Rev. 13, 77-108 (2011).
- DHHS, . US Panel on Antiretroviral Therapy and Medical Management of HIV-Infected Children. Guidelines for the Use of Antiretroviral Agents in Pediatric HIV Infection. , (2012).
- . Department of Health. Clinical guidelines for the management of HIV & AIDS in adults and adolescents. , (2010).
- Conradie, F., et al. The 2012 southern African ARV drug resistance testing guidelines. S. Afr. J.HIV Med. 13, 162-167 (2012).
- Manasa, J., et al. Primary Drug Resistance in South Africa: Data from 10 Years of Surveys. AIDS Res. Hum. Retroviruses. 28, 558-565 (2012).
- Tshabalala, M., et al. Surveillance of transmitted antiretroviral drug resistance among HIV-1 infected women attending antenatal clinics in Chitungwiza, Zimbabwe. PLoS ONE. 6, (2011).
- Bartolo, I., et al. Antiretroviral drug resistance surveillance among treatment-naive human immunodeficiency virus type 1-infected individuals in Angola: evidence for low level of transmitted drug resistance. Antimicrob. Agents Chemother. 53, 3156-3158 (2009).
- Bartolo, I., et al. HIV-1 genetic diversity and transmitted drug resistance in health care settings in Maputo, Mozambique. J. Acquir. Immune Defic. Syndr. 51, 323-331 (2009).
- Hamers, R. L., et al. HIV-1 drug resistance in antiretroviral-naive individuals in sub-Saharan Africa after rollout of antiretroviral therapy: a multicentre observational study. Lancet Infect. Dis. 11, 750-759 (2011).
- Hamers, R. L., et al. HIV-1 Drug Resistance Mutations Are Present in Six Percent of Persons Initiating Antiretroviral Therapy in Lusaka, Zambia. J. Acquir. Immune Defic. Syndr. , (2010).
- Nwobegahay, J., Selabe, G., Ndjeka, N. O., Manhaeve, C., Bessong, P. O. Low prevalence of transmitted genetic drug resistance in a cohort of HIV infected naive patients entering antiretroviral treatment programs at two sites in northern South Africa. J. Med. Virol. 84, 1839-1843 (2012).
- Iweriebor, B. C., et al. Molecular epidemiology of HIV in two highly endemic areas of northeastern South Africa. Arch. Virol. 157, 455-465 (2012).
- Parboosing, R., Naidoo, A., Gordon, M., Taylor, M., Vella, V. Resistance to antiretroviral drugs in newly diagnosed, young treatment-naive HIV-positive pregnant women in the province of KwaZulu-Natal South Africa. J. Med. Virol. 83, 1508-1513 (2011).
- Nwobegahay, J. M., et al. Prevalence of antiretroviral drug resistance mutations and HIV-I subtypes among newly-diagnosed drug-naive persons visiting a voluntary testing and counselling centre in northeastern South Africa. J. Health Popul. Nutr. 29, 303-309 (2011).
- Nwobegahay, J., et al. Prevalence of drug-resistant mutations in newly diagnosed drug-naive HIV-1-infected individuals in a treatment site in the waterberg district, limpopo province). S. Afr. Med. J. 101 (2011), 335-337 (2011).
- Rosen, S., Long, L., Sanne, I., Stevens, W. S., Fox, M. P. The net cost of incorporating resistance testing into HIV/AIDS treatment in South Africa: a Markov model with primary data. J. Int. AIDS Soc. 14, 24 (2011).
- Dalai, S. C., et al. Evolution and molecular epidemiology of subtype C HIV-1 in Zimbabwe. AIDS. 23, 2523-2532 (2009).
- Chen, J. H., et al. In-house human immunodeficiency virus-1 genotype resistance testing to determine highly active antiretroviral therapy resistance mutations in Hong Kong. Hong Kong Med. 18, 20-24 (2012).
- Lee, C. K., et al. An in-house HIV genotyping assay for the detection of drug resistance mutations in Southeast Asian patients infected with HIV-1. J. Med. Virol. 84, 394-401 (2012).
- Aitken, S. C., et al. A Pragmatic Approach to HIV-1 Drug Resistance Determination in Resource-Limited Settings by Use of a Novel Genotyping Assay Targeting the Reverse Transcriptase-Encoding Region Only. J. Clin. Microbiol. 51, 1757-1761 (2013).
- Zhou, Z., et al. Optimization of a low cost and broadly sensitive genotyping assay for HIV-1 drug resistance surveillance and monitoring in resource-limited settings. PLoS One. 6, (2011).
- , Life Technologies. Life Technologies and SATuRN Collaborate to Increase Access to HIV Testing in Africa. , (2012).
- Lihana, R. W., Ssemwanga, D., Abimiku, A., Ndembi, N. Update on HIV-1 diversity in Africa: a decade in review. AIDS Rev. 14, 83-100 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten