Method Article
In-vivo-Abfrage des Zentralen Nervensystems durch Translatome Polyribosom Fraktionierung
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt wesentliche Änderungen Polyribosom Fraktionierung, um die translatome von in vivo ZNS-Proben zu untersuchen. Es ermöglicht umfassende Beurteilung der Übersetzung und der Transkriptionsregulation durch die Isolierung und der Vergleich der Gesamt-RNA gebunden Ribosomen-RNA-Fraktionen.
Zusammenfassung
Mehrere Verfahren sind in der Genexpression einschließlich Transkription, Translation und Stabilität von mRNAs und Proteine beteiligt. Jeder dieser Schritte sind fest geregelt und betrifft die letzten Dynamik von Proteinfluss. Verschiedene Regulationsmechanismen sind im Übersetzungsschritt, wodurch mRNA-Konzentrationen allein ein unzuverlässiger Indikator der Genexpression. Darüber hinaus werden lokale Regulation der mRNA-Übersetzung wurde insbesondere in neuronalen Funktionen beteiligt, Verlagerung 'translatomics', um den Fokus der Aufmerksamkeit in der Neurobiologie. Die vorgestellte Methode kann auf Brücke Transkriptomik und Proteomik eingesetzt werden.
Hier beschreiben wir wesentliche Änderungen an der Technik der Polyribosom Fraktionierung, die die translatome fragt auf der Grundlage der Vereinigung von aktiv übersetzt mRNAs mehrere Ribosomen und ihre differentielle Sedimentation in Saccharose-Gradienten. Traditionell, die Arbeit mit in-vivo-Proben, insbesondere des central Nervensystem (ZNS) ist schwierig, da es die begrenzten Mengen an Material und die Anwesenheit von Fettgewebe Komponenten bewährt. Um dieses Problem anzugehen, ist die beschriebene Protokoll für die Verwendung mit minimalem ZNS-Material optimiert ist, wie durch die Verwendung von einzelnen Mausrückenmark und Gehirn nachgewiesen. Kurz gesagt, werden ZNS-Gewebe extrahiert und übersetzen Ribosomen auf mRNAs mit Cycloheximid immobilisiert. Myelin Flotation wird dann durchgeführt, um lipidreichen Komponenten zu entfernen. Fraktionierung auf einem Saccharose-Gradienten in dem mRNAs werden nach ihrer ribosomalen Lade getrennt durchgeführt. Isolierten Fraktionen eignen sich für eine Reihe von Tests abwärts, einschließlich der neuen genomweiten Testtechnologien.
Einleitung
Genexpression wird durch die kombinierte Wirkung der Transkription, Translation und Stabilität von mRNA und Proteinen bestimmt, wobei die Translationslager vorherrschende Wirkung 1. Es ist nun klar, daß jeder dieser Schritte ist stark reguliert. Mikro-RNAs, die Bildung von RNA-Granulat, alternative und zytoplasmatische Polyadenylierung sind einige Beispiele der posttranskriptionelle Regulation der Genexpression 2,3. Jeder dieser Mechanismen entkoppelt Transkription von Übersetzung und beeinflusst das Proteom im biologischen System von Interesse. So überraschend, mRNA-Spiegel allein sind eine unvollkommene Auslesen der Protein-Ebene 4. Quantitative Proteomik bietet die direkte Beurteilung der Genexpression, aber trotz der jüngsten Fortschritte gibt es immer noch erhebliche Einschränkungen auf Empfindlichkeit und Auflösung Sequenz Protein. Daher Bewältigung der translatome, das Repertoire der Übersetzung mRNAs, bietet einen hervorragenden Kompromiss zwischen Studium derTranskriptom-und Proteom. Es ist genauer als bei der Beurteilung der letzten Transkriptomik Genexpression und bietet sowohl höhere Abdeckung und Reihenfolge Auflösung als Proteomik.
In Säugersystemen, die Mehrheit der Übersetzung Veranstaltungen beginnen über cap-abhängigen Initiation. Eine Gruppe von eukaryotischen Initiationsfaktoren zusammen mit der kleinen 40S ribosomalen Untereinheit zusammen am 5'-Kappe einer mRNA. Der Komplex dann scannt die mRNA und bei Erreichen der Start-Codon AUG, wirbt die 60S großen ribosomalen Untereinheit zu bilden, eine komplette 80S-Ribosomen. Die Dehnungsstufe fährt mit dem Ribosom sich entlang der mRNA, mit Elongationsfaktoren Unterstützung der Aufnahme von Aminosäuren aus geladenen tRNAs für die entstehende Peptidkette. Mehreren Ribosomen entlang einer einzigen mRNA gleichzeitig erfolgen und die Anzahl der zugehörigen Ribosomen wurde gezeigt, dass die Rate der Proteinsynthese 5,6 korrelieren. Dies macht ribosomalen Lade eine zuverlässige Indikation für translation und ermöglicht die Trennung von aktiv übersetzen mRNAs auf Basis Senkungsgeschwindigkeit. Neben der Quantifizierung der Übersetzung von mRNAs kann Sequenzinformationen erhalten werden, um Motive in der Translationsregulation beteiligt sind. Auch RNA-bindende Proteine und andere Faktoren können Übersetzung aus verschiedenen Fraktionen isoliert werden, erleichtern das Studium und die Entdeckung der damit verbundenen regulatorischen Proteinen.
Im Nervensystem hat Translationskontroll Prozessen wie mRNA, Lagerung, Transport und lokale Proteinsynthese verbunden. Wachstumskegeln haben gezeigt, dass eine bestimmte lokale Pool von mRNAs vom Rest des Axons 7 beherbergen. Außerdem Axone besitzen die Fähigkeit, lokal Proteine synthetisieren 8,9. Als Ergebnis lokale Steuerung der Übersetzung ist zu einem entscheidenden Thema der Studie in der Neurobiologie. Das Potential der Polyribosom Fraktionierung, um diese Adresse in mehreren Studien veranschaulicht worden, bei dem die Technik wurde verwendetuntersuchen Axon Führung in Rückenmark-Entwicklung, und zeigte die aktivitätsabhängige Übersetzung von BDNF im Gehirn 10,11.
Protokoll
Alle Tierversuche wurden gemäß den Richtlinien des Instituts des DKFZ durchgeführt.
Achtung: Um RNase Kontamination der Proben zu verhindern, nehmen grundlegenden Vorsichtsmaßnahmen zur Vermeidung von RNase-Kontamination und bereiten alle Puffer mit DEPC-behandeltem Wasser.
1. Vorbereitung der Saccharose-Gradienten
- Bereiten 17,5, 25,6, 33,8, 41,9 und 50% Saccharose-Lösungen (siehe Tabelle 1). Vorbereiten Gradienten durch langsame Zugabe von 2 ml 50% Saccharose-Lösung auf den Boden eines Polyallomer-Ultrazentrifugenröhrchen. Einfrieren der Schicht, indem Röhrchen für 20 min bei -80 ° C.
- Fügen Sie die nachfolgenden Schichten bei abnehm Saccharose Prozent mit Gefrierfach Schritte dazwischen, landen auf 17,5% Saccharose. Halten Gradienten auf Eis während der Zugabe von Saccharose Lösungen, um Auftauen der darunterliegenden Schichten zu verhindern.
- Bereiten Saccharosegradienten frisch einen Tag vor Gebrauch und halten bei 4 ° C Alternatively, Gradienten im Voraus vorbereiten, bei -80 ° C und Auftauen bei 4 ° C in der Nacht vor dem Experiment.
2. Gewebepräparation (Spinal Cord und / oder Gehirn)
- Betäuben Maus durch eine Überdosis von Xylazin und Ketamin in NaCl und transkardial mit 20 ml Hanks ausgewogener Salzlösung (HBSS), enthaltend 200 ug / ml CHX die Ribosomen zu assoziierten Transkripte immobilisiert Perfusion. Überprüfen Sie die Betäubung durch Nicht-Ansprechbarkeit bis Fuß drückt.
- Auszug Gewebe schnell. Öffnen Sie den Schädel von dorsal und extrahieren das Gehirn. Führen Laminektomie der gesamten Brust-und Lendenwirbelsäule und extrahieren das Rückenmark. Verwenden gesamte Gehirn (~ 400 mg) bzw. 4 cm großes Stück des Rückenmarks (~ 90 mg) auf.
- Übertragen Gewebe in eiskaltes Homogenisierung Puffer A (siehe Tabelle 1) (Rückenmark: 1 ml; Gehirn: 4 ml) und Würfel in kleine Stücke mit einem sauberen Skalpell, um erhöhte Aufnahme von Cycloheximid ermöglichen. Perform alle weiteren Schritte auf Eis.
- Inkubation: 15 min und homogenisieren Gewebe mechanisch mit einer Dounce Homogenisator. Sorgfältig kontrollierte Homogenisierung ist notwendig, um sicherzustellen, dass Kerne intakt bleiben. Behandeln Sie alle Proben genau die gleiche Art und Weise, um die Vergleichbarkeit zu gewährleisten. Hinweis: für Hirngewebe: Stören Gewebe um 5 Schläge mit einem dicht schließenden Stößel. Für Rückenmarksgewebe: brechen um 5 Schläge mit einem locker sitz Stößel, gefolgt von 5 weiteren Schläge mit einem dicht schließenden Stößel.
- Nehmen Aliquots (400 ul für Gehirn-und 200 ul für Rückenmark), Flash-Speicher, einfrieren und bei -80 ° C für die Gesamt-RNA Isolation.
- Kerne und ungestörten Zell-und Gewebefragmente zu entfernen durch Zentrifugation bei 500 × g für 10 min bei 4 ° C. Zentrifugation bei niedriger Geschwindigkeit verhindert den Verlust von Ribosomen.
- Lyse Membranfragmente in die Kerne freie Überstand für 30 min durch Zugabe von NP-40 und Natriumdeoxycholat Detergenzien (jeweils bis zu einer Endkonzentration von 1%).
3. Myelin Flotation
- Dieser Schritt entfernt Fettkomponenten aus der Probe, die sonst die Signalmaske wird in der Polyribosom Profil. Zuerst Pre-Chill-Ultrazentrifuge Eimern und Rotor bei 4 ° C und bereiten 2 M, 1,1 M und 0,9 M Saccharose-Lösungen (siehe Tabelle 1).
- Mix Lysat mit einem Volumen von 1,22 2 M Saccharose-Lösung und Transfer in einem Polyethylen-Ultrazentrifugenröhrchen. Füllen Sie alle Rohre zu gleichen Volumen von 10 ml mit 1,1 M Saccharose-Lösung, danach überlagern sie vorsichtig mit 0,9 M Saccharose-Lösung.
- Legen Sie die Ultrazentrifuge Rohre in vorgekühlte Ultrazentrifuge Eimern. Zentrifuge für 3 Stunden bei 100.000 × g bei 4 ° C. Während dieses Prozesses werden Ribosomen in der Pellet hinterlegt wohinFettKomponenten schweben und bleiben im Überstand.
4. Sucrose Gradient Fraktionierung
- Überstand entfernen und Pellet lösen sich in Homogenisierung Puffer B (siehe Tabelle 1 ). Die Extinktion bei 260 nm für jede Probe unter Verwendung eines Nanodrop oder eine äquivalente Vorrichtung und normalisieren beträgt nach dem Absorptionswert zu laden.
- Platzieren Saccharosegradienten (Herstellung wird in einem vorherigen Abschnitt beschrieben) in die vorgekühlte Ultrazentrifugeneimer. Schichtproben sorgfältig oben auf dem Gradienten. Fügen Sie eine leere Saccharose-Gradienten als die technische Kontrolle.
- Einstellen des Gewichts jeder Eimer mit Homogenisierungspuffer. Zentrifuge Proben für 1,5 Stunden bei 285.000 × g bei 4 ° C.
- Start der Vorbereitung der Isco Fraktionators 30 Minuten vor dem Ende der Zentrifugation. Wenn ein anderes Modell der Fraktionators verwendet wird, folgen Sie dem Protokoll des Herstellers.
- Stellen Sie die entsprechende Empfindlichkeit (0,2 AUFS für Gehirn-oder 0,05 AUFS für Rückenmark) für die UV-Lampe und schalten Sie es zum Aufwärmen. Montieren Sie die Rohrbohrer, verbinden Sie es über die Rollpumpe zu 60% Saccharose-Lösung durch das Rohr Piercer.
- Testen Sie das Setup durch Pumpen Saccharose (Fluss Rattee: 1 ml / min), insbesondere sicherzustellen, dass es keine Lecks in den Rohrleitungen, die Blasen in der Steigung vorstellen wird.
- Blank Grundabsorption durch manuelles Pumpen Gradientenpuffer in den UV-Detektor und die Korrektur der Grundlinie.
- Die Ultrazentrifuge Eimern, die die Saccharose-Gradienten Entfernen Sie vorsichtig mit sedimentierten Proben aus dem Rotor und legen sie auf Eis. Vermeiden Sie Anstoßen von Gradienten zum Verlust der Auflösung zu verhindern.
- Führen Gradienten auf Fraktionators. Führen Sie erste Grundabsorption beurteilen und eine ordnungsgemäße technische Setup leer Steigungen.
- Befestigen Gradienten der UV-Detektor und durchbohren die Unterseite des Rohres mit dem Rohr Dorn. Starten Sie den Pump von 60% Saccharose, die die Steigung mit sedimentierten Proben nach oben durch den UV-Detektor und in den Tropfenspender verdrängen wird.
- Die Absorption bei 260 nm dokumentiert die Absorptionsprofil für die Probe zu erzeugen, mit Spitzen, die die Sedimentationtion von mRNAs mit ribosomalen Untereinheiten, monosomes und anschließend zunehmende Anzahl von Ribosomen assoziiert.
- Die Proben werden in 20 Fraktionen über das Drop-Spender (jeweils 600 ul, in 2-ml-Röhrchen).
5. RNA-Isolierung aus einzelnen Fraktionen
- Werden 10% SDS bis zu einer Endkonzentration von 1% zu einzelnen Fraktionen und gut mischen, um Proteine zu entfalten und zu dissoziieren Ribosomen. An diesem Punkt können die Proben bei -80 ° C eingefroren werden, Je nach Fragestellung angegangen werden, können Fraktionen nach dem Absorptionsprofil gebündelt.
- Isolieren RNA mit saurem Phenol / Chloroform-Extraktion, die auch entfernt verunreinigenden Spuren von DNA.
- In einem Volumen von sauren Phenol / Chloroform (auf RT vorgewärmt) zu jeder Probe, Hitze für 10 Minuten bei 65 ° C und Lösedruck unter dem Abzug danach.
- Zentrifuge Proben für 20 min bei 17.000 × g bei RT. Wässrige Phase in ein neues 1,5 ml vorsichtig übertragenRöhre. Seien Sie sich bewusst von der Phaseninversion in dichter Saccharosefraktionen, wo die wässrige Phase kann an der Unterseite nach Zentrifugation sein.
- In einem Volumen Isopropanol, 1/9 Volumen Natriumacetat (pH 5,2) und 1 ul GlycoBlue zu jeder Probe, um die RNA zu fällen. Halten Proben wenigstens 1 Stunde bei -80 ° C.
- Zentrifugieren für 30 min bei 17.000 × g und 4 ° C. GlycoBlue bietet Visualisierung des Pellets. Überstand entfernen, waschen Pellet einmal mit eiskaltem 80% Ethanol und Pellet an der Luft trocknen. Das Pellet in RNase-freies Wasser.
- Quantifizieren RNA-Menge von Nanodrop und analysieren die Integrität der RNA von einem Bioanalyzer Chip. RNA, die durch das vorgestellte Protokoll erhalten wird, ist geeignet für alle hochmoderne Hochdurchsatz-Assays mit Microarray-und Sequenzierungs tief.
Ergebnisse
Abbildung 2 zeigt repräsentative Polyribosom Profile nach der Fraktionierung. Die Profile von Gehirn und Rückenmark charakteristische Absorptionskurven wie zuvor für Zelllinien beschrieben. mRNAs zu kleinen ribosomalen Untereinheiten (40S) Sediment am leichteren Fraktionen gebunden und erscheinen zunächst als ein Gipfel auf dem Profil, gefolgt von der großen ribosomalen Untereinheit (60S) und monosome (80S) gebunden mRNAs. mRNAs für mehrere Ribosomen gebunden sedimentieren schwereren Fraktionen, die später mit Peaks, die zunehmende Anzahl von gebundenen Ribosomen. Globale Übersetzung kann aus dem Profil beurteilt werden. Als Beispiel hat Hirngewebe eine höhere Polyribosom Verhältnis monosome als Rückenmarksgewebe, was aktiver Übersetzung.
RNA aus einzelnen Fraktionen extrahiert wurden von Bioanalyzer beurteilt, welche die Verteilung der 18S-28S rRNA und. 18S rRNA früher erscheint in dem Profil in Übereinstimmung mit der kleinen ribosomalen Untereinheiten Sedimentieren in leichter sucr ose Fraktionen. Typische Ausbeuten von Gesamt-RNA sind 10-20 ug für Gehirn-und 4.2 ug für Rückenmark. RNA-Ausbeuten aus den einzelnen Fraktionen bis zu 4 &mgr; g und 0,8 &mgr; g für Gehirn und Rückenmark sind, abhängig von der Fraktion. Eine leere Saccharose-Gradienten zeigt bereits einige Hintergrundabsorption bei 260 nm, durch die Anwesenheit von DTT. Dieser Hintergrund kann bei der Datenanalyse, um Probenwerte normalisieren abgezogen werden. EDTA-Behandlung brach die Polyribosom Gipfel, zeigt die Sedimentation Profil ist auf Übersetzung.
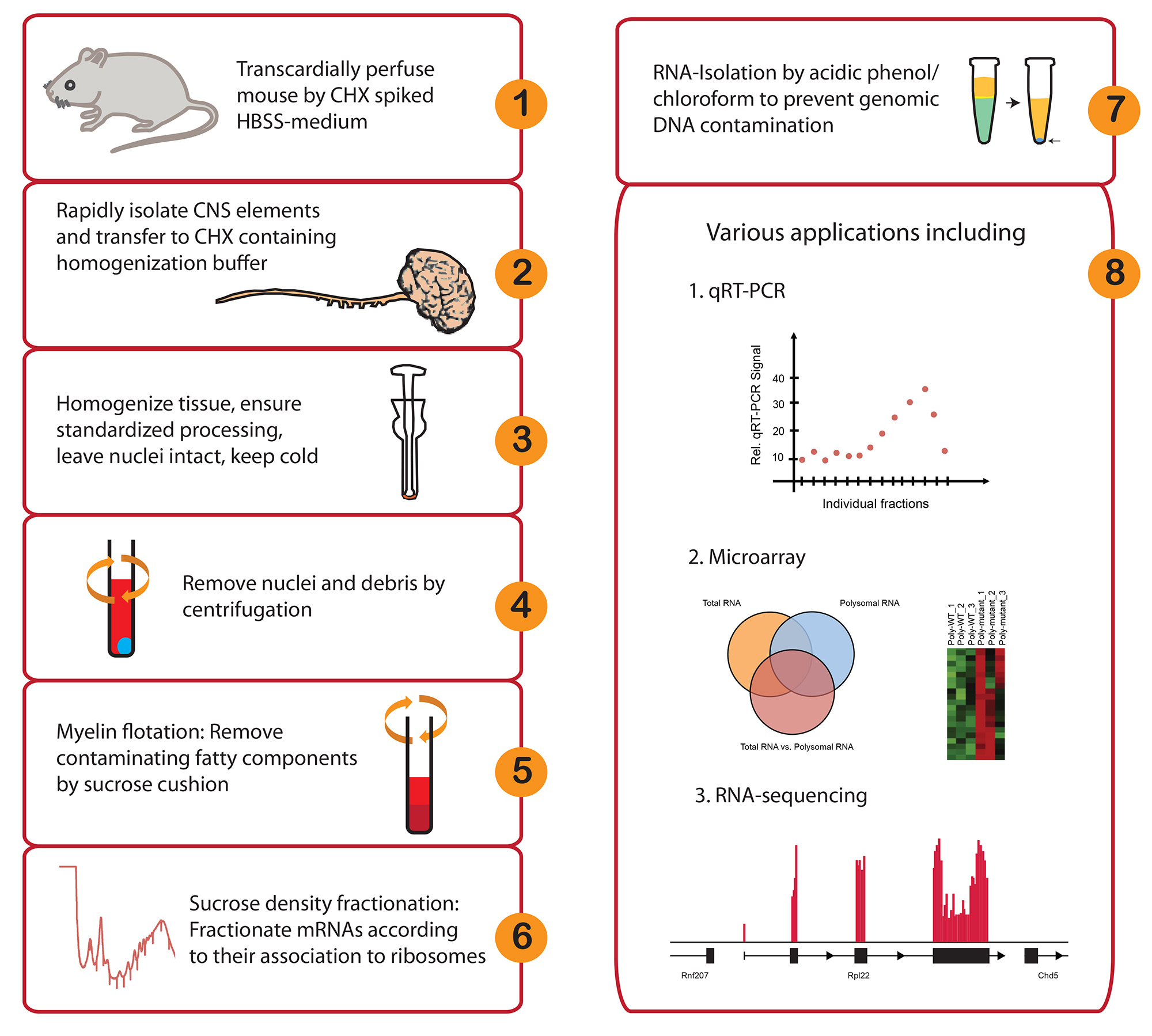
Abbildung 1. Workflow-und Anwendungsmöglichkeiten in vivo Polyribosom Fraktionierung.> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
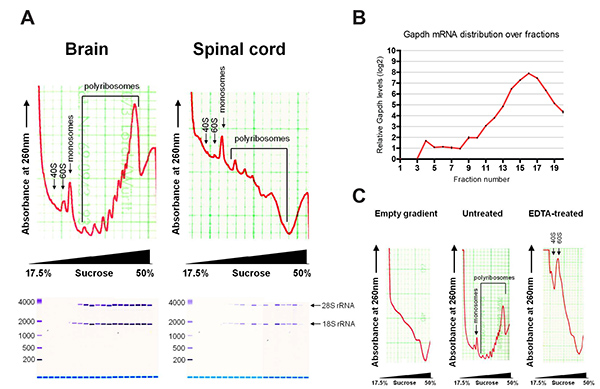
Abbildung 2. (A) Typische Polyribosom Profile von Gehirn und Rückenmark, mit der Beurteilung der extrahierten RNA durch Bioanalyzer. (B) Verteilung von mRNA über Gapdh Fraktionen durch qRT-PCR. (C) Polyribosom Profile von leeren Farbverlauf und das Gehirn mit und ohne EDTA-Behandlung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
| 1.1 | 2x Gradientenpuffer | 30 mM Tris-HCl pH 7,4, 30 mM MgCl 2, 600 mM NaCl, 200 &mgr; g / ml Cycloheximid (CHX), 2 mM Dithiothreitol (DTT) | |
| 1.1 | Saccharose-Lösung 17,5% | 8,67 g Saccharose, 25 ml 2x Gradienten Puffer auf 50 ml DEPC-H 2 0 | |
| 1.1 | Saccharose-Lösung 25,6% | 12,8 g Saccharose, 25 ml 2x Gradienten Puffer auf 50 ml DEPC-H 2 0 | |
| 1.1 | Saccharose-Lösung 33,8% | 16,9 g Saccharose, 25 ml 2x Gradienten Puffer auf 50 ml DEPC-H 2 0 | |
| 1.1 | Saccharose-Lösung 41,9% | 20,95 g Saccharose, 25 ml 2x Gradienten Puffer auf 50 ml DEPC-H 2 0 | |
| 1.1 | Saccharose-Lösung 50% | 25 g Saccharose, 25 ml 2x Gradienten Puffer auf 50 ml DEPC-H 2 0 | |
| 2.3 | HomogenisierungPuffer A | 0,25 M Saccharose, 50 mM Tris / HCl, pH 7,4), 5 mM MgCl 2, 25 mM KCl | 200 ug / ml CHX, 1x Roche komplette Proteaseinhibitor, 1 mM DTT, 1 mM phenylmethanesulfonylfluoride (PMSF), 100 U / ml RNAsin |
| 3.1 | 2M Saccharose-Lösung | 68,4% Saccharose, 50 mM Tris-HCl, pH 7,4, 5 mM MgCl 2, 25 mM KCl | 100 ug / ml CHX, 1x Roche komplette Proteaseinhibitor, 1 mM DTT, 1 mM PMSF |
| 3.1 | 1,1 M Saccharose-Lösung | 38,5% Saccharose, 50 mM Tris-HCl, pH 7,4, 5 mM MgCl 2, 25 mM KCl | 100 ug / ml CHX, 1x Roche komplette Proteaseinhibitor, 1 mM DTT, 1 mM PMSF |
| 3.1 | 0,9 M Saccharose-Lösung | 30,8% Saccharose, 50 mM Tris-HCl, pH 7,4, 5 mM MgCl 2, 25 mM KCl | 100 ug / ml CHX, 1x Roche komplette Proteaseinhibitor, 1 mM DTT, 1 mM PMSF |
| 4.1 | homogenization Puffer B | 0,25 M Saccharose, 50 mM Tris / HCl, pH 7,4, 5 mM MgCl 2, 25 mM KCl, 1% NP-40, 1% Natriumdesoxycholat | 200 ug / ml CHX, 1x Roche komplette Proteaseinhibitor, 1 mM DTT, 1 mM PMSF, 100 U / ml RNAsin |
Tabelle 1. Liste von Puffern und Lösungen.
Diskussion
Obwohl Polyribosom Fraktionierung ist keine neue Technik, bleibt es eine besondere Herausforderung ein. Auf der Grundlage der Input-Material, können erhebliche Optimierung notwendig sein. Dies ist insbesondere der Fall für die in vivo ZNS-Proben, wobei die Menge des Materials ist oft eine Einschränkung und Fettgewebe Komponenten behindern Isolierung übersetzen mRNAs. Die meisten veröffentlichten Fraktionierung Protokolle befassen sich mit Hefe-oder Säugerzelllinien, und es gibt etablierte Protokolle für das Gehirn 12,13,14. Im Gegensatz dazu gibt es kaum Publikationen, die Fraktionierung von Rückenmark und frühere Protokolle erfordern Rückenmark von einer großen Anzahl von Tieren zu 15 zusammengefasst werden. Aus diesen Gründen wurden einige wesentliche Änderungen vorgenommen, um die Fraktionierung Protokoll für ZNS-Geweben, einschließlich Einzelrückenmark der Maus anzupassen. Ribosom Immobilisierung mit Cycloheximid ist bei Tier Perfusion durchgeführt, um die Dissoziation der Ribosomen Durin vermeideng der lange Prozess der Gewebeentnahme. Anschließend werden polysomaler RNAs in einer bestimmten Art und Weise extrahiert, um Polyribosom Ausbeute zu maximieren. Zuerst kontrolliert die Homogenisierung des Gewebes durch Douncing, hält den Kern intakt und verhindert DNA-Kontamination. Der Kern wird dann zuverlässig durch Zentrifugation entfernt. Die Kombination der Wasch-NP-40 und Natriumdeoxycholat sorgt Lyse des endoplasmatischen Retikulums und Freisetzung der Membran assoziiert Ribosomen. Darüber hinaus UV-absorbierende Komponenten innerhalb der lipidreichen Myelin verschleiern die Polyribosom Profilen. Myelin Flotation ist daher notwendig, wo dichte Verbindungen wie Polyribosomen werden pelletiert und leichter Verbindungen wie Myelin Schwimmer und 16 werden entfernt. Polyribosome werden dann über eine 17,5% bis 50% Saccharose-Gradienten sedimentiert. Anstelle der manuellen Schichtung und Gefrier jedes Saccharose Schicht können auch Steigungen mit einem Gradienten-Mischer hergestellt werden. Extraktion von RNA aus den Fraktionen mit saurem Phenol / Chloroform ermöglicht DNA mit minimalem Verlust RNA entfernt werden. Allerdings kann die Phaseninversion bei dichten Saccharose-Fraktionen (oben Fraktion 16) auftreten.
Dieses Protokoll bietet Vorteile gegenüber anderen herkömmlichen Verfahren, die die Ebene der Translation zu adressieren. Beispielsweise sind sowohl die Messung der S6 phosphoryliert (ein prominenter Übersetzung Marker) sowie Kennzeichnung von naszierenden Ketten mit Radioisotopen geben Informationen über globale Übersetzung Ebenen, sondern offenbaren wenig auf, was speziell die übersetzt wird. Polyribosom Fraktionierung, andererseits, ermöglicht nicht nur Beurteilung globaler Übersetzung, sondern auch die Identität der Übersetzung mRNAs und die damit verbundenen regulatorischen Proteinen. Quantitative real-time PCR kann auf isolierte RNA für eine schnelle Auslesen der ausgewählten mRNAs durchgeführt werden und Microarrays und RNA-Sequenzierung der nächsten Generation für genomweite Studien durchgeführt werden. Die hier vorgestellten Optimierungen erlauben die Technik, um mit minimalen Mengen von ZNS-Gewebe verwendet werden, daher verringernten die Zahl der Tiere erforderlich ist und die Verbesserung der allgemeinen experimentellen Qualität durch Verringerung Gewebeverarbeitungszeit. Auf der anderen Seite kann diese Technik nicht unterscheiden gekippt Ribosomen übersetzen diejenigen. Transcripts Durchführung installiert Ribosomen werden in den schweren Fraktionen sedimentieren, und dies sollte bei der Interpretation der Daten aufbewahrt werden.
Es gibt neuere Techniken gemeldet, nämlich RiboTaq und TRAP, in denen Ribosomen sind mit Epitop-Tags oder Reportern, die jeweils in einer Zelltyp-spezifische Weise markiert und die zugehörigen mRNAs durch Immunpräzipitation 17,18 isoliert. Diese Technik kann auf die Fraktionierung Polyribosom hohe Auflösung für spezifische Zellpopulationen zu lesen bieten gekoppelt werden. Ribosom Fuß-Druck ist eine weitere neuartige Technik, Nucleaseverdau geht um kleine Fragmente zu erzeugen, oder "Footprints", von mRNAs, die von Ribosomen geschützt sind. Bibliotheken werden dann aus diesen Spuren erzeugt und sequenziert. Diese Methode provIdes ein Codon-spezifische Analyse des gesamten Genoms auf Übersetzung und in der Lage ist ins Stocken geraten Ribosomen zu identifizieren, nicht-AUG-Codons und kleinen vorgelagerten offenen Leserahmen, die nicht durch Fraktionierung Polyribosom 19 erreicht werden kann. Allerdings kann Polyribosom Fraktionierung Ribosom Profiling für die Isolierung von verdauten Fragmente mit einzelnen Ribosomen gekoppelt werden, und bereichert damit für die molekulare Spezies für Bibliotheks Vorbereitung, in denen hohe Reinheit der Probe ist oft erforderlich, benötigt wird. Zusammengenommen ist Polyribosom Fraktionierung eine flexible Methode mit bleibender Bedeutung und kann zu verschiedenen Downstream-Anwendungen einschließlich der genomweiten Untersuchungen wie Next-Generation-Sequenzierung gekoppelt werden.
Offenlegungen
Kein Interessenkonflikt erklärt.
Danksagungen
Diese Arbeit wurde durch das Bundesministerium für Bildung und Forschung (BMBF), der Systembiologie der Signaltransduktion in Cancer (Helmholtz-Allianz Systembiologie) und dem Deutschen Krebsforschungszentrum (DKFZ) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Hank’s balanced salts solution | Gibco | 14170-138 | |
| Tube, Thinwall, Polyallomer, 14 ml, 14 x 95 mm | Beckman Coulter | 331374 | |
| Diethylpyrocarbonate | Sigma | D5758 | caution |
| Cycloheximide | Sigma | C7698 | danger |
| Dithiothreitol | Sigma | 43815 | warning |
| Complete Protease Inhibitor Cocktail Tablets | Roche | 11697498001 | |
| Phenylmethanesulfonyl fluoride solution | Sigma | 93482 | danger |
| RNasin Plus RNase Inhibitor | Promega | N2611 | |
| Nonidet P-40 | Roche | 11332473001 | danger |
| Sodium deoxycholate | Sigma | D6750 | warning |
| Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1) | Ambion | AM9722 | danger |
| GlycoBlue | Invitrogen | AM9516 | |
| Equipment | |||
| Dounce Tissue Grinder, 1 ml/7 ml | Wheaton | 357538/357542 | |
| Nanodrop 2000 | Thermo Scientific | ||
| SW 40 Ti Rotor, Swinging Bucket, Titanium, 6 x 14 ml, 40,000 rpm, 285,000 x g | Beckman Coulter | 331302 | |
| Density Gradient Fractionator | Teledyne Isco | ||
| 2100 Bioanalyzer | Agilent Technologies |
Referenzen
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Lau, A. G., et al. Distinct 3'UTRs differentially regulate activity-dependent translation of brain-derived neurotrophic factor (BDNF). Proceedings of the National Academy of Sciences of the United States of America. 107 (36), 15945-15950 (2010).
- Piqué, M., López, J. M., Foissac, S., Guigó, R., Méndez, R. A combinatorial code for CPE-mediated translational control. Cell. 132 (3), 434-448 (2008).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Grolleau, A., et al. Global and specific translational control by rapamycin in T cells uncovered by microarrays and proteomics. The Journal of Biological Chemistry. 277 (25), 22175-22184 (1074).
- Rajasekhar, V. K., Viale, A., Socci, N. D., Wiedmann, M., Hu, X., Holland, E. C. Oncogenic Ras and Akt signaling contribute to glioblastoma formation by differential recruitment of existing mRNAs to polysomes. Molecular Cell. 12 (4), 889-901 (2003).
- Zivraj, K. H., et al. Subcellular profiling reveals distinct and developmentally regulated repertoire of growth cone mRNAs. The Journal of Neuroscience. 30 (46), 15464-15478 (2010).
- Brittis, P. a, Lu, Q., Flanagan, J. G. Axonal protein synthesis provides a mechanism for localized regulation at an intermediate target. Cell. 110 (2), 223-235 (2002).
- Tcherkezian, J., Brittis, P. a, Thomas, F., Roux, P. P., Flanagan, J. G. Transmembrane receptor DCC associates with protein synthesis machinery and regulates translation. Cell. 141 (4), 632-644 (2010).
- Colak, D., Ji, S. -J., Porse, B. T., Jaffrey, S. R. Regulation of axon guidance by compartmentalized nonsense-mediated mRNA decay. Cell. 153 (6), 1252-1265 (2013).
- Lau, A. G., et al. Distinct 3'UTRs differentially regulate activity-dependent translation of brain-derived neurotrophic factor (BDNF). Proceedings of the National Academy of Sciences of the United States of America. 107 (36), 15945-15950 (2010).
- Del Prete, M. J., Vernal, R., Dolznig, H., Müllner, E. W., Garcia-Sanz, J. A. Isolation of polysome-bound mRNA from solid tissues amenable for RT-PCR and profiling experiments. RNA. 13 (3), 414-421 (2007).
- Esposito, A. M., et al. Eukaryotic polyribosome profile analysis. J. Vis. Exp. (40), (2010).
- Sampath, P., Lee, Q. Y., Tanavde, V., et al. Identifying translationally regulated genes during stem cell differentiation. Current Protocols in Stem Cell Biology. Schlaeger, T. 1, (2011).
- Chiu, F. C., Smith, M. E. Studies on rat spinal cord polysomes: postnatal development and experimental demyelination. Journal of Neurochemistry. 31 (4), 835-844 (1978).
- Larocca, J. N., Norton, W. T., et al. Isolation of myelin. Current Protocols in Cell Biology. Bonifacino, J. S., et al. Chapter 3, (2007).
- Heiman, M., et al. A translational profiling approach for the molecular characterization of CNS cell types. Cell. 135 (4), 738-748 (2008).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Ingolia, N. T., Brar, G. a, Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature Protocols. 7 (8), 1534-1550 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten