Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Studieren von DNA-Looping von Einzelmolekül-FRET
In diesem Artikel
Zusammenfassung
Diese Studie stellt eine detaillierte experimentelle Verfahren zur Looping Dynamik der Doppelstrang-DNA mit Einzelmolekül-Fluoreszenz-Resonanz-Energie-Transfer (FRET) zu messen. Das Protokoll beschreibt auch, wie die Looping-Wahrscheinlichkeitsdichte genannt J-Faktor zu extrahieren.
Zusammenfassung
Biegen der Doppelstrang-DNA (dsDNA) mit vielen wichtigen biologischen Prozessen wie der DNA-Protein-DNA-Erkennung und Verpackung in Nukleosomen verbunden. Thermodynamik der dsDNA Biege wurde durch eine Methode namens Cyclisierung, die auf DNA-Ligase kovalent zu verbinden setzt kurz klebrigen Enden eines dsDNA sucht. Allerdings kann Ligationseffizienz durch viele Faktoren, die nicht verwandt sind dsDNA Looping, wie die DNA-Struktur rund um die klebrigen Enden verbunden, und Ligase betroffen sein können auch die scheinbare Schleifenrate beeinflussen durch Mechanismen wie unspezifische Bindung. Hier zeigen wir, wie man dsDNA Looping Kinetik ohne Ligase durch Erfassen transienter DNA-Schleifenbildung durch FRET (Fluorescence Resonance Energy Transfer) zu messen. dsDNA-Moleküle werden mit einem einfachen PCR-basiertes Protokoll mit einem FRET-Paar und einem Biotin-Linker konstruiert. Die Schleifenwahrscheinlichkeitsdichte als die J-Faktor bekannt ist, aus dem Looping Rate und der Kühlrate zwischen zwei abschaltung extrahiertted klebrigen Enden. Durch das Testen zwei dsDNAs mit verschiedenen intrinsischen Krümmungen zeigen wir, dass die J-Faktor ist sensibel für die Eigenform der dsDNA.
Einleitung
Das Verständnis der mechanischen Eigenschaften von dsDNA ist von grundlegender Bedeutung in der Grundlagenwissenschaften und technische Anwendungen. Die Struktur der dsDNA ist komplizierter als eine gerade, weil spiralförmigen Leiter Rolle, Neige-und Verdrehungswinkel zwischen aufeinanderfolgenden Basenpaare mit der Sequenz variieren. Temperaturschwankungen können dazu führen, dsDNA zu unterschiedlichen Modi Konformationsfluktuationen wie Biegen, Verdrehen und Strecken unterzogen werden. Übergänge wie Schmelzen und Knicken kann auch unter extremen Bedingungen auftreten.
Unter dieser Bewegungen hat dsDNA Biege die auffälligste biologische Wirkung ein. dsDNA Biegung mit Gen-Repression oder Aktivierung, indem zwei entfernten Standorten nahe einander zugeordnet. Es spielt auch eine wichtige Rolle bei der DNA-Verpackung im Zellkern oder ein virales Kapsid. Biegeverformung dsDNA kann experimentell durch die hochauflösende Mikroskopie (AFM-und TEM-2 3) und der ThermoDyn visualisiert werdenamics und Kinetik kann durch Looping-Assays, die chemisch verbinden nebeneinander liegenden Stellen des dsDNA sucht werden.
Ein solcher Test ist Ligase-abhängig Cyclisierung 4. In diesem Assay werden dsDNA-Molekülen mit "klebrig" (kohäsiven) Enden zirkularisiert oder durch DNA-Ligase dimerisiert. Durch Vergleichen der Raten der Kreis und die Dimer-Bildung, kann man eine effektive molare Konzentration von einem Ende der DNA in der Nähe des anderen Endes, die als J-Faktor bekannt ist, erhalten. Dieser Faktor ist dimensions J entspricht der Wahrscheinlichkeitsdichte des Findens ein Ende der DNA in einem kurzen Abstand von dem anderen Ende, und spiegelt damit die Flexibilität der DNA. Die Messung der J-Faktor als Funktion der DNA-Länge zeigt viele Merkmale über die DNA Mechanik einschließlich der Persistenz Länge 4,5.
Das wurmartige Kette (WLC)-Modell wurde weithin als der kanonischen Polymer-Modell für dsDNA Mechanik basierend auf dem Erfolg in Erklärungen angesehenining der Kraft-Dehnungs-Kurven in DNA ziehen Experimente 6 und richtig Vorhersage der J Faktoren dsDNAs länger als 200 bp 7 erhalten. Allerdings mit der Cyclisierung Assay an dsDNA-Moleküle so kurz wie 100 bp, Cloutier und Widom maßen die J Faktoren, um mehrere Größenordnungen höher als die WLC-Modell-Vorhersage 8 sein. Ein Jahr später, Du et al. J Faktoren erzeugt im Einvernehmen mit dem WLC-Modell mit dem Cyclisierung Assay mit niedrigeren Konzentrationen von Ligase und schrieb den anomalen Ergebnis aus der Gruppe Widom hohen Konzentrationen verwendet Ligase 9. Diese Kontroverse zeigt beispielhaft die unvermeidlichen Einfluss der DNA-Ligase auf Cyclisierung Kinetik bei Verwendung der herkömmlichen Assay 9. Darüber hinaus kann auch auf DNA-Ligase DNA-Struktur und Steifigkeit durch unspezifische Bindung 10,11.
Um die technischen Belange der Protein-abhängige Looping-Assays zu beseitigen, vor kurzem haben wir gezeigt, eine protein-kostenlos Looping Assay, der auf Fluoreszenz-Resonanz-Energie-Transfer (FRET) 12. In diesem Verfahren werden geschleift Konformationen von FRET zwischen dem Donor und Akzeptor in der Nähe der klebrigen Enden eines DNA-Moleküls gebunden nachgewiesen. Eine Totalreflexions-Fluoreszenzmikroskopobjektiv-Typ (TIRF) verwendet, um Bahnen der reversible Looping und unlooping Ereignisse von der Oberfläche immobilisierten DNA-Einzelmoleküle für einen längeren Zeitraum aufzuzeichnen. Diese Methode bietet PCR-basierten Aufbau von DNA-Molekülen an Mismatch-freie DNA-Moleküle, die eine entscheidende Verbesserung gegenüber einer ähnlichen Methode Vafabakhsh und Ha 13 zu generieren. Die Einzelmolekül-Aspekt dieses Protokoll ermöglicht die Messung von Verteilungen zusätzlich zu Durchschnittswerte Ensemble während der FRET-Aspekt ermöglicht es, DNA-Looping Dynamik wiederholt aus dem gleichen Molekül messen, auch unter Bedingungen, die Ligase-Aktivität beeinträchtigen können.
Die TIRFM Aufbau ist in Abbildung 1 dargestellt. Eine benutzerdefinierteGestalteten Probentisch wird über eine Olympus IX61 Mikroskop Körper platziert. 532 nm und 640 nm-Laser von der Seite eingeführt werden und durch kleine elliptische Spiegel 14 in den hohen NA Ziel reflektiert wird, um kritische Einfallswinkel, unter dem Deckglas-Wasser-Grenzfläche zu erreichen. Wir stellen fest, dass eine weit verbreitete Durch Ziel TIR mit dichroitischen Spiegeln oder Prismen TIR-basierte Setups können auch für diese FRET-Anwendung verwendet werden. Das Fluoreszenzbild durch das Mikroskop gebildet wird durch einen dichroitischen Spiegel in Donor-und Akzeptor-Bilder aufgeteilt. Sie werden dann auf beiden Hälften eines EMCCD erneut abgebildet. Weitere Langpassemissionsfilter verwendet werden, um Hintergrundsignal zu reduzieren.
Die Temperaturregelung ist unerlässlich für den Erwerb reproduzierbare kinetischen Daten. Zur Temperaturregelung wird das Ziel aus dem Mundstück des Mikroskopkörpers um die Wärmeübertragung und Wasser von einer Temperatur gesteuert Kühler / Heizung minimieren getrennt durch eine Messing-Kragen, die eng passt in Umlaufum die innere Metall unter der Ziel-Jacke. Diese Einrichtung ist in der Lage, stabile Temperaturregelung an der Deckfläche zwischen 15 und 50 ° C (Fig. 2) zu erreichen. In dieser Arbeit wurde die Probentemperatur auf 24 ° C gehalten wird
Das folgende Protokoll zeigt die Schritt-für-Schritt-Verfahren für die DNA-Konstruktion, die Einschätzung der DNA-Form, Einzelmolekül-Experiments, und J-Faktor Bestimmung.
Protokoll
1. DsDNA Probenvorbereitung
- Gestalten Sie global gekrümmt DNAs durch Wiederholung eines 10-mer-Sequenz. Zum Beispiel 5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X) 7 - TTTCATTCGAGCTCGTTGTTG-3 'ist ein 186-bp-DNA gekrümmten wobei X eine Zufalls zusätzlichen Basis und der flankierenden Sequenz die sich wiederholende 10-mer-Sequenz Adaptersequenzen.
HINWEIS:. In diesem Beispiel wurden zwei 10-mere mit gegenüberliegenden Präferenzen Nukleosom Bildung auf der Grundlage einer großen Nukleosom Belegung Studie von Kaplan et al 15 gewählt. Da die Schrauben Wiederholung der dsDNA ist nahe bei 10 bp, wird jede Netz Auslenkung der Schraubenachse des 10-mer zu akkumulieren, um eine Form wie ein Kreisbogen (Abbildung 3A) zu erzeugen. Da die Schrauben Zeit ist näher an 10,5 bp, wird eine zusätzliche Basis nach jeweils zwei Wiederholungen eingesetzt, um die gekrümmte Struktur so eben wie möglich zu halten. Diese Sequenzen kürzer als 200 bp aus Companie bestellt werdens Angebot, dass Gen-Synthese-Service. Es ist zweckmäßig, diese Sequenzen mit gemeinsamen Adaptersequenzen für nachfolgende Schritte flankieren. Das Verfahren ist in Fig. 3B schematisch dargestellt. - Führen Sie die PCR mit Primer 1 (GTGCCAGCAACAGATAGC) und Primer 2 (/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG). HINWEIS: Primer 2 mit der FRET-Donor Cy3 am 5'-Ende markiert. Ein typisches PCR-Protokoll Rezept und Radfahren sind in den Tabellen 1 und 2 dargestellt.
- Führen Sie die PCR mit Primer 3 (/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC) und Primer 4 (CAACAACGAGCTCGAATG). HINWEIS: Primer 3 mit dem FRET-Akzeptor Cy5 durch das Rückgrat und die Biotin-Linker am 5'-Ende markiert. PCR-Rezept und Radfahren Protokoll wie oben.
- Reinige die PCR-Produkte mit Hilfe eines PCR-Reinigung-Kit.
- Mischen die Cy3-markierten Produkt und die Cy5-markierte Produkt in einem Puffer für den Strangaustausch (100 mM NaCl, 10 mM Tris-HCl pH 7,0, 1 mM EDTA) in Endkonzentrationen von 0,4 undNr. 181, M und 0,1 uM. HINWEIS: Die überschüssige Cy3-DNA erhöht die Konzentration des Duplex Durchführung sowohl Cy3 und Cy5 als Ergebnis der Strangaustausch.
- Wechsel Stränge durch Inkubation bei 98,5 ° C für 2 Minuten, allmähliches Abkühlen bis 5 º C Anstiegsrate von 0,1 ° C / sec, und Inkubation bei 5 ° C für 2 Stunden.
2. Gel-Elektrophorese zu dsDNA Krümmung Detect
- Gießen Polyacrylamidgel 16,17 durch Mischen Acrylamid und Bisacrylamid Lösungen bei 29.2:0.8 Verhältnis von 5% (w / v) in 1 × Tris / Borat / EDTA (TBE)-Puffer bei pH 8,0. Hinweis: 10 ml Gel-Lösung enthält: 1,217 ml 40% Acrylamid, 0,667 ml 2% Bis-Acrylamid, 1 ml 10x TBE, 100 L Ammoniumpersulfat (APS), 10%, 10 l TEMED, und der Rest ist dH 2 O. Vollständige Verfestigung des Gels beträgt etwa 30 min.
- Laden der Polyacrylamid-Gel mit DNA-Proben und die DNA-Leiter in 1X-Beladungspuffer (5% Glycerin, 0,03% (w / v) Bromphenolblau blue) und führen Sie das Gel bei 5-8 V / cm bei 4 ° C für 45 Minuten oder bis die Farbstofffront nähert sich dem Ende des Gels.
- Färben Sie das Gel mit 1X TBE-Puffer, der 0,5 g / ml Ethidiumbromid für 30 min enthält. Identifizieren Sie die DNA-Banden unter UV-Beleuchtung. Vergleichen Bandpositionen mit der Größe Marker (100 bp DNA-Leiter), um die scheinbare Größe der DNA-Moleküle zu berechnen. HINWEIS: Gebogene DNAs bewegen in der Regel langsamer als gerade DNAs.
3. Durchflusszelle Vorbereitung
- Drill 6-7 Paare von Löchern, die entlang zwei gegenüberliegenden Rändern eines Glasobjektträgers (3'' × 1'') unter Verwendung einer Bohrmaschine und Diamantbohrer. Nach dem Bohren, reiben Sie die Folie in fließendem Wasser, um sichtbare Glaspulver zu entfernen. HINWEIS: Die Löcher dienen als Perfusions-und Auslässe. Beim Bohren ist Kühl die Folie mit Wasser wichtig Rissbildung zu vermeiden.
- Legen Sie die Folien aufrecht in ein Glas und füllen es mit Wasser. Beschallen für 15 min und übertragen sie in ein anderes Glas deAceton für Reinigungs dicated. Füllen Sie sie mit Aceton und beschallen für 15 min. Spülen Sie die Folien mit Ethanol mit einer Sprühflasche und dann mit Wasser. Legen Sie sie in einem Polypropylen-Glas, füllen Sie es mit 5 M Kaliumhydroxid und beschallen für 15 min. Schließlich beschallen die Folien in Wasser 15 min. Reinigen Sie die Deckgläser (Nr. 1, 24 x 40 mm) mit dem gleichen Protokoll. HINWEIS: Gereinigt Rutschen und Deckgläser können in dH 2 O für langfristige Gebrauch gespeichert werden.
- Mischen 1 mg Biotin-PEG-Silan (MW 3400) mit 80 mg mPEG-Silan (MW 2000) in 340 L 0,1 M Natriumhydrogencarbonat-Lösung. Gut mischen und zentrifugieren Sie die Mischung kurz auf von Blasen loszuwerden. HINWEIS: Die Funktionalisierung der Oberfläche mit Polyethylenglykol (PEG) trägt zur Verringerung unspezifischer Bindung von DNA an die Oberfläche.
- Setzen Sie 80 L der PEG-Lösung auf jeder Folie und senken Sie ein Deckglas darüber. Warten Sie 45 min. Trennen Sie das Deckglas aus der Folie mit einer Pinzette, spülen Sie sie mit reichlich dH 2 O und sei tSaum trocken im Freien.
- Legen Sie dünne Streifen von doppelseitigem Klebeband auf der Folie, um Kanäle zu bilden. Richten Sie ein Deckglas darüber und drücken Sie fest auf das Deckglas gegen den Schieber, um flüssigkeitsdichte Kanäle bilden. Wählen Sie mit 5 Minuten-Epoxy, um die Kanten der Kanäle zu versiegeln.
4. Vorbereitung der Trolox-Lösung
- Setzen ~ 30 mg Trolox und 10 ml dH 2 O in einem Kolben und verwenden Sie einen Magneten Rührstab, um die Lösung in der offenen Luft für 18 Stunden rühren.
- Filtern der Lösung unter Verwendung eines 0,2 &mgr; m-Filter und den pH auf 7 durch Zugabe von ~ 6 ul 1 M Tris-Base (pH 11). Hinweis: Trolox ist ein Anti-Reagenz blinkt die üblicherweise in Einzel-Molekül verwendet wird, untersucht 18. Die Wirkung von Trolox Antifading kommt aus seiner oxidierten Derivat, das in teilweise abgebaut Trolox-Lösung 19 vorhanden ist. Schnell Lösen Trolox in Methanol oder hohen pH-Tris-Lösung sollte durch ineffiziente Oxidation vermieden werden.
5. Einzel-molkül Imaging
- Injizieren 15 ul NeutrAvidin-Lösung (0,5 mg / ml) in den Kanal und Warten für 2 Minuten vor dem Spülen mit 100 ul T50-Puffer (10 mM Tris-HCl, 50 mM NaCl, pH 7,0).
- Injizieren 50 ul DNA-Probe (50-100 pM) in den Kanal. 5 min warten und spülen Sie die ungebundene DNA weg mit 100 L T50-Puffer. HINWEIS: Die DNA-Moleküle spezifisch an die Oberfläche binden durch die NeutrAvidin-Biotin-Wechselwirkung.
- Füllen Sie den Kanal mit der Bildpuffer, die eine sauerstoffbindende System 20 (100 mm Lochkreis, 5 mM PCA, 1 mM Trolox und 500 mM NaCl) enthält.
- Stellen Sie den Rahmen in EMCCD-Übertragungsmodus auf dem Computer mit 25 Bildern pro Sekunde streamen 2 x 2 klassierte Bilder (256 x 256).
- Setzen Sionsöl auf dem Mikroskopobjektiv, und befestigen Sie die Durchflusszelle auf dem Mikroskoptisch mit Probe-Clips. Grob den Fokus, indem man die Laser-Reflexionsmuster an der Wand. Feineinstellung des Fokus mit der Live-Ansicht der Fluoreszenz imAlter auf dem Monitor.
- Beginnt die Datenerfassung mit dem 532-nm-Laser auf. Überprüfen Sie die Live-Ansicht und stellen Sie den Fokus, wenn nötig. Stoppen Sie die Datenerfassung, wenn die meisten Moleküle haben gebleicht.
HINWEIS: In dieser Anlage wird ein Labor-C-Programm geschrieben, um die Mikroskopsteuerung und Anzeige von Live-Bildern auf dem Bildschirm, wie sie auf der Festplatte gespeichert sind, verwendet.
6. Bildverarbeitung und Datenanalyse
HINWEIS: Eine Zeitreihe von 256 x 256 Bilder werden mit einem MATLAB-Code verarbeitet, um Einzelmolekülzeitverläufe von Cy3-und Cy5-Intensitäten zu erzeugen. Um Pixel zwischen dem Spender und dem Akzeptor-Kanal Kanal des Split-View-Bild-Paare, 6-7 Paare von Cy3 und Cy5-Spots, wobei jedes Paar aus dem gleichen Molekül gleichmäßig über das Sichtfeld verteilt sind, werden von Hand gepflückt, und eine affine Transformation wird unter Verwendung der Koordinaten dieser Punkte als Ankerpunkte berechnet.
- Mit einem MATLAB-Skript, durch alle Einzelmolekül-Zeit zu suchen Spuren tHut zeigen mehrere Übergänge zwischen niedrigen und hohen FRET-Signale. Identifizieren Sie die geschleift und geloopten Staaten.
HINWEIS: Die FRET-Signal wird als das Cy5 Intensität (I a), dividiert durch die Summe der Cy3-und Cy5-Intensitäten (I a + I d). Geschleift Staat hat hohe FRET-Wert, während ungeloopte Zustand niedrigen FRET-Wert hat. Das Histogramm von FRET-Signale von einem einzelnen Molekül ist bimodal wegen reversible Looping und unlooping verteilt. - Finden Sie die Schwelle, die die beiden Verteilungen durch die Bestimmung der Schnittpunkt zwischen den beiden Einbau Gaußkurven trennt.
- Berechnen Sie die FRET-Effizienz als
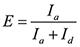 und weisen Sie die geschleift Staaten mit hohen FRET-Werten und den geloopten Staaten mit niedrigen FRET-Werte.
und weisen Sie die geschleift Staaten mit hohen FRET-Werten und den geloopten Staaten mit niedrigen FRET-Werte. - Mit einem MATLAB-Skript, analysieren die kumulative Anzahl der Moleküle (N (t)), die eingeschleift (odererreicht die hohe FRET-Zustand) zu verschiedenen Zeitabläufen seit Beginn der Datenerfassung. HINWEIS: Da eine dsDNA-Molekül kann in jeder Konformation des geloopten Zustand zu Beginn der Datenerfassung zu starten, die Steigerungsrate in der Bevölkerung spiegelt die geschlungenen Schleifendurchschnittsrate gemittelt über anfängliche Konformationen. Entpacken Sie die Looping Rate k-Schleife durch den Einbau N (t) mit einer Exponentialfunktion:
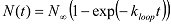 Wenn biphasisch steigt, kann es mit einem Doppel Exponentialfunktion montiert werden:
Wenn biphasisch steigt, kann es mit einem Doppel Exponentialfunktion montiert werden: 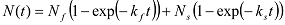 In diesem Fall ist k Schleife erhalten aus:
In diesem Fall ist k Schleife erhalten aus: 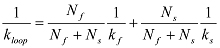 HINWEIS: Theoretisch ist die Überlebenswahrscheinlichkeit, dass ein Polymer nicht zum Zeitpunkt t geschlungen ist nicht eine einfache oder doppelte Exponentialfunktion 12. Exponential fitting wird als praktisches Mittel, um die durchschnittliche Kreiszeit zu extrahieren.
HINWEIS: Theoretisch ist die Überlebenswahrscheinlichkeit, dass ein Polymer nicht zum Zeitpunkt t geschlungen ist nicht eine einfache oder doppelte Exponentialfunktion 12. Exponential fitting wird als praktisches Mittel, um die durchschnittliche Kreiszeit zu extrahieren.
7. Bestimmung der J-Faktor
HINWEIS: Die J-Faktor darstellt, wie konzentriert das eine Ende eines dsDNA ist, um das andere Ende. Es kann durch Interpolieren der Konzentration von einem Ende des Segments der DNA, die die gleiche Reaktionsgeschwindigkeit mit dem anderen Ende des Segments gemessenen Schlingengeschwindigkeit erzeugen würde, bestimmt werden. Experimentell wird ein Endabschnitt auf der Oberfläche immobilisiert ist, und der andere Endabschnitt wird in einer bestimmten Konzentration c eingeführt. Wenn die gemessene Kühlrate zwischen den beiden Enden ist k Glühen, dann wird der Faktor 21 J gegeben durch 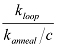 . Die Kühlrate-Konstante (k = k Glühen Glühen / C) ist unabhängig von der Sondenkonzentration.
. Die Kühlrate-Konstante (k = k Glühen Glühen / C) ist unabhängig von der Sondenkonzentration.
- Fluss 20 ul von 30-50 pM Biotin-Cy5 Oligo (Primer 3) into NeutrAvidin einer beschichteten Kanal. Spülen Sie den Kanal mit 100 ul T50 abzuwaschen ungebundenen Oligos.
- Vorbereitung des Bilderzeugungspuffer, wie in Teil 5 mit dem Zusatz von Cy3 Oligo (Primer 2) in einer Endkonzentration von 50 nM beschrieben. Fluss dieses Bildpuffer in den Kanal.
- Während die 532-nm-Laser auf, kurz drehen Sie den 640-nm-Laser auf Standorte der Oberfläche gebundenen Cy5 Oligos zu identifizieren. Schalten Sie die 640-nm-Laser und starten Sie die Überwachung des FRET-Signals.
HINWEIS: Bei der Hybridisierung eines Oligo Cy3 mit einem oberflächengebundenen Oligo Cy5, Cy5-Fluoreszenz wird von FRET auftreten. - Mit einem MATLAB-Skript, analysieren die Anzahl der Moleküle, die im ungebundenen Zustand (geringe Cy5-Intensität) zu starten, aber später in die geglühten Zustand (hohe Intensität Cy5) drehen als Funktion der Zeit aus den Cy5 Intensität Spuren.
- Zeichnen Sie diese Anzahl von geglüht Moleküle gegen die Zeit. Setzen Sie diese Kurve mit einer einzigen Exponentialfunktion (
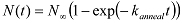 ), Um die Kühlrate (k Glühen) zu erhalten.
), Um die Kühlrate (k Glühen) zu erhalten. - Wiederholen dieses Experiment bei verschiedenen Cy3 Oligo-Konzentrationen (60, 100 und 180 nM), um die Linearität zwischen der Kühlrate und der Konzentration der Reaktanten zu bestätigen. Entpacken Sie die zweiter Ordnung Glühen Geschwindigkeitskonstante (k Glühen ) Von der Piste.
- Berechnen Sie die J-Faktor aus
 , Wobei k-Schleife ist der Schlingengeschwindigkeit in der gleichen Pufferbedingungen gemessen.
, Wobei k-Schleife ist der Schlingengeschwindigkeit in der gleichen Pufferbedingungen gemessen.
Ergebnisse
DNA-Moleküle für die Untersuchung verwendet Schleifen bestehen aus einem Duplexregion der variablen Sequenz und Länge und einzelsträngige Überhänge, welche komplementär zueinander sind. Die Überhänge, die 7-Basis lang sind, können miteinander zu glühen, um die Loop-Zustand zu erfassen. Jeder Überstand enthält entweder Cy3 oder Cy5, die in dem DNA-Rückgrat durch Amiditchemie verbunden ist. Die Cy5-Überhang ist auch mit Biotin-TEG (15-Atom-Tetra-Ethylen-Glykol-Abstandshalter) für die Oberflächen Immobilis...
Diskussion
Eine einfache Einzelmolekül-FRET-Assay, der auf Looping wurde verwendet, um die Kinetik der DNA von verschiedenen Eigenformen zu studieren. Gekrümmt DNAs können durch Wiederholen eines 10-mer-Sequenz in Phase mit dem Schrauben Zeitraum von 10,5 bp, hergestellt werden, und deren Krümmungen mit PAGE abgeschätzt werden. Diese dsDNAs werden mit klebrigen Enden entwickelt, um transienten Schleife Stabilisierung ermöglichen. Es extrahiert den Schlingengeschwindigkeit von der exponentiellen Anstieg in der Anzahl der Mole...
Offenlegungen
Die Autoren erklären, keine Interessenkonflikte.
Danksagungen
Wir danken James Waters, Gable Wadsworth und Bo Broadwater für das Manuskript kritisch zu lesen. Wir danken auch vier anonymen Gutachtern für die Bereitstellung von nützlichen Kommentare. Wir danken für die finanzielle Unterstützung von Georgia Institute of Technology, der Burroughs Wellcome Fund Career Award in der Scientific-Schnittstelle, und dem Studenten-Forschungsnetzwerk Zuschuss von NSF Physik lebender Systeme.
Materialien
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
Referenzen
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten