Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Sequenzspezifische Markierung von Nukleinsäuren und Proteinen mit Methyltransferasen und Cofaktor-Analoga
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
DNA und Proteine sequenzspezifisch mit Affinität oder fluoreszierenden Reportergruppen mit DNA- oder Protein-Methyltransferasen und synthetischen Cofaktor-Analoga bezeichnet. Abhängig von der Cofaktor-Spezifität der Enzyme, Aziridin- oder doppelt aktivierte Cofaktor-Analoga werden für ein- oder zweistufige Kennzeichnung verwendet.
Zusammenfassung
S -Adenosyl-L-Methionin (AdoMet oder SAM) -abhängige Methyltransferasen (MTase) katalysieren den Transfer des aktivierten Methylgruppe von AdoMet auf bestimmte Positionen in der DNA, RNA, Proteinen und kleinen Biomolekülen. Diese natürliche Methylierungsreaktion kann auf eine Vielzahl von Alkylierungsreaktionen unter Verwendung von synthetischen Cofaktor-Analoga ausgedehnt werden. Ersatz des reaktiven Sulfonium- Mitte AdoMet mit einem Aziridin-Ring führt zu Cofaktoren, die mit DNA von verschiedenen DNA MTasen koppelbar ist. Diese Aziridin Cofaktoren können mit Reportergruppen an verschiedenen Positionen des Adeninrestes ausgestattet und für S equence spezifischen M ethyltransferase- I L Abel ing der DNA (Smiling DNA) nduced verwendet werden. Als typisches Beispiel geben wir ein Protokoll für die Biotinylierung von pBR322 Plasmid-DNA an der 5'-ATCG A T-3'-Sequenz mit der DNA MTase M.BseCI und Aziridin Cofaktor 6BAz ineinem Schritt. Verlängerung der aktivierten Methylgruppe mit ungesättigten Alkylgruppen Ergebnisse in einer anderen Klasse von AdoMet-Analoga, die für m ethyltransferase gerichtete Nous Transfer von A ctivated G ruppen (MTAG) verwendet werden. Da die verlängerten Seitenketten durch Sulfonium Zentrum und der ungesättigten Bindung aktiviert werden diese Cofaktoren doppel aktiviert AdoMet Analoga genannt. Diese Analoga nicht nur als Cofaktoren für DNA MTasen wie Aziridin Cofaktoren, sondern auch für RNA, Proteinen und kleinen Molekülen MTasen. Sie werden typischerweise zur enzymatischen Modifizierung MTase Substrate einzigartige funktionelle Gruppen, die mit Reportergruppen in einem zweiten Syntheseschritt markiert verwendet. Dies wird in einem Protokoll für die Fluoreszenzmarkierung der Histon H3-Protein veranschaulicht. Eine kleine Propargylgruppe ist aus der Cofaktor analogen SeAdoYn an das Protein durch die Histon H3 Lysin 4 (H3K4) MTase Set7 / 9 gefolgt von Klick Kennzeichnung der übertragenealkinylierten Histon H3 mit TAMRA Azid. MTase vermittelte Markierung mit Cofaktor-Analoga ist eine Schlüsseltechnologie für viele spannende Anwendungen, einschließlich Identifizierung und funktionelle Untersuchung der MTase Substrate sowie DNA-Genotypisierung und Methylierungsnachweis.
Einleitung
Spezifische Markierung von Nukleinsäuren und Proteinen 1,2 3,4 ist von großem Interesse für die funktionelle Charakterisierung, der medizinischen Diagnostik und (Nano-) Biotechnologie. Hier stellen wir eine enzymatische Markierungsverfahren für diese Biopolymere, die auf S -adenosyl-L-Methionin (AdoMet oder SAM) -abhängige Methyltransferasen (MTasen) basiert. Diese Enzymklasse (EC 2.1.1.) Zielt einzelnen nucleophilen Stellen (Stickstoff, Sauerstoff, Schwefel und Kohlenstoffatomen) innerhalb spezifischer Reste von Nukleinsäuren und Proteinen, und überträgt die aktivierte Methylgruppe des Cofaktors AdoMet (1A) 5 natürlich. Darüber hinaus können MTasen synthetischen Cofaktor-Analoga zur spezifischen Markierung mit Affinitätsmarkern, Fluorophore oder andere Etiketten (Abbildung 1B) 6 zu nutzen. Zwei Klassen von AdoMet-Analoga entwickelt: Aziridin Cofaktoren für S equence spezifischen M ethyltransferase- I Nduced L abel ing (lächelnd) 7 und Doppel aktiviert AdoMet-Analoga für m ethyltransferase gerichteter Nous Transfer von A ctivated G ruppen (MTAG) 8.

Figur 1: Die Reaktionen von Methyltransferasen (MTasen) A. Methyl Gruppe von der natürlichen Cofaktor AdoMet (SAM) auf verschiedenen Substraten, einschließlich DNA, RNA, Proteinen und kleinen Biomolekülen B. Labeling / Funktionalisierung von Nukleinsäuren und Proteinen (NNNNN = katalysiert.. Basenpaare der DNA, Nukleotiden von RNA und Aminosäuren für Proteine; XXXXX = Erkennungssequenz der MTase mit Ziel Rückstand in grün) mit synthetischen Cofaktor-Analoga. Aziridin Cofaktoren, die eine Reportergruppe (blaue Kugel)an den Adenin gebunden sind sequenzspezifisch mit der Zielrest (links), und doppelklicken aktiviert AdoMet-Analoga gekoppelt führen zu erweiterter Alkylketten, der eine chemische Reporter Y (rechts), die von bioorthogonale Klick-Reaktion in einem zweiten Schritt bezeichnet werden kann übertragen. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Aziridin Cofaktoren funktionieren am besten mit DNA MTasen. Sie enthalten einen dreigliedrigen Ring mit einem Stickstoffatom, 9 (oder ein N -mustard 10,11) anstelle der Sulfonium- Zentrum als reaktive Gruppe. Protonierung des Stickstoffatoms aktiviert den Aziridinring für den nucleophilen Angriff durch die Zielnukleotidsequenz, die Kopplung des ganzen Cofaktor mit DNA kovalent führt. Durch das Anbringen Reportergruppen an den Adeninring die Aziridin Kofaktoren kann in Kombination mit DNA MTasen verwendet, um DNA in einem Schritt zu kennzeichnen (werden g> 1B, links) 7,12. Dies wird im Detail für die Biotinylierung von DNA mit 6BAz 13 gezeigt - 15 (Aziridin Cofaktor Biotin an der 6-Stellung der Adenin-Ring gebunden ist) und der Adenin-spezifische DNA MTase aus Bacillus stearothermophilus (M.BseCI) 16 (Abbildung 2, siehe Protokoll Abschnitt 2: Ein-Schritt-Markierung von DNA durch Aziridin Kofaktoren). Neben M.BseCI ("Erkennungssequenz, die DNA MTasen aus Thermus aquaticus (M.TaqI, 5'-TCG A -3 5'-ATCG A T-3)), aus Haemophilus heamolyticus (M.HhaI, 5 "-G C GC-3 ') und von Spiroplasma (M.SssI, 5'-C G-3') wurden erfolgreich eingesetzt, um DNA mit 6BAz 17 biotinylieren. Weiterhin kann Aziridin Cofaktoren für einstufige Fluoreszenz-DNA-Markierungs 18,19 eingesetzt werden.
NHALT "fo: keep-together.within-page =" always ">
Fig. 2: Sequenzspezifische einstufigen Biotinylierung von DNA mit M.BseCI und 6BAz Die DNA MTase M.BseCI erkennt die doppelsträngige DNA-Sequenz 5'-ATCG A T-3 'und natürlich methyliert die Aminogruppe des zweiten Adenin Rückstand (grün) mit AdoMet. Mit dem Aziridin Cofaktor 6BAz der Verlauf der Reaktion verändert wird und M.BseCI führt zu spezifischen DNA-Biotinylierung durch Kopplung des gesamten Cofaktor einschließlich Biotin (blau) mit der Ziel Adenin zu sequenzieren. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Doppel aktiviert AdoMet-Analoga enthalten verlängert ungesättigte Seitenketten statt einer Methylgruppe an der Sulfonium- Zentrum (1B , rechts) 20. Die ungesättigte Doppel- oder Dreifachbindung in β-Stellung zu dem Sulfoniumsalz Zentrum elektronisch kompensiert ungünstige sterische Effekte im Übergangszustand durch Konjugation Stabilisierung. Da sowohl die Sulfonium-Center und die ungesättigte Bindung aktivieren Sie die Seitenkette für die enzymatische Übertragung wurden diese Cofaktoren doppelt aktivierte AdoMet-Analoga genannt. Typischerweise werden sie verwendet, um Seitenketten mit einzigartigen chemischen Gruppen (chemischer Reporter), wie Amino, Alkin- und Azidgruppen in einem zweiten Schritt 8,21 zu übertragen, für die chemoselektive Etikettierung. In der Regel können mit einem Doppelklick aktiviert AdoMet-Analoga nicht nur als Co-Faktoren für die DNA-MTasen 8,20,21 sondern auch für RNA MTasen 22,23 und Protein MTasen 24-28 ermöglicht zusätzliche Kennzeichnung von RNA und Proteinen. Allerdings sind die verlängerten Seitenketten sterisch anspruchsvoller als eine Methylgruppe und eine Vergrößerung der MTase aktiven Zentren durch Protein-Engineering ist often erforderlich, um eine effiziente Übertragungsraten zu erzielen. Eine andere Lösung für dieses Problem ist es, eine AdoMet-Analogon mit einem kleinen Propargylgruppe (drei Kohlenstoffatome), wobei das terminale Alkin hat zwei Funktionen: 1. Stabilisierung des Übergangszustands bei der enzymatischen Übertragung und 2. Reaktivgriff für folgende chemische Modifikationen von Kupfer- katalysierte Azid-Alkin-Cycloaddition (CuAAC) Klickchemie. Es stellte sich heraus, dass die resultierende propargylische AdoMet analogen 29 ist unter neutralen oder leicht basischen Bedingungen und nur bedingt recht instabil. Dieser Nachteil kann durch den Ersatz des Schwefelatoms mit Selen fixiert werden. Der resultierende Cofaktor 5 '- [(Se) [(3S) -3-Amino-3-carboxypropyl] prop-2-ynylselenonio] -5'-Desoxyadenosin (SeAdoYn, Bild 3) durch Wildtyp-DNA erlaubt, RNA und Protein MTasen 30-32, die die Notwendigkeit für das Protein-Engineering in vielen Fällen aufheben. Dies wird durch Fluoreszenz pro exemplifiziert Protein Markierung mit dem Histon H3 Lysin 4 (H3K4) MTase Set7 / 9 33 (Abbildung 3, siehe Protokoll Abschnitt 3: Zweistufige Proteinmarkierung durch Doppelklick aktiviert Kofaktoren).
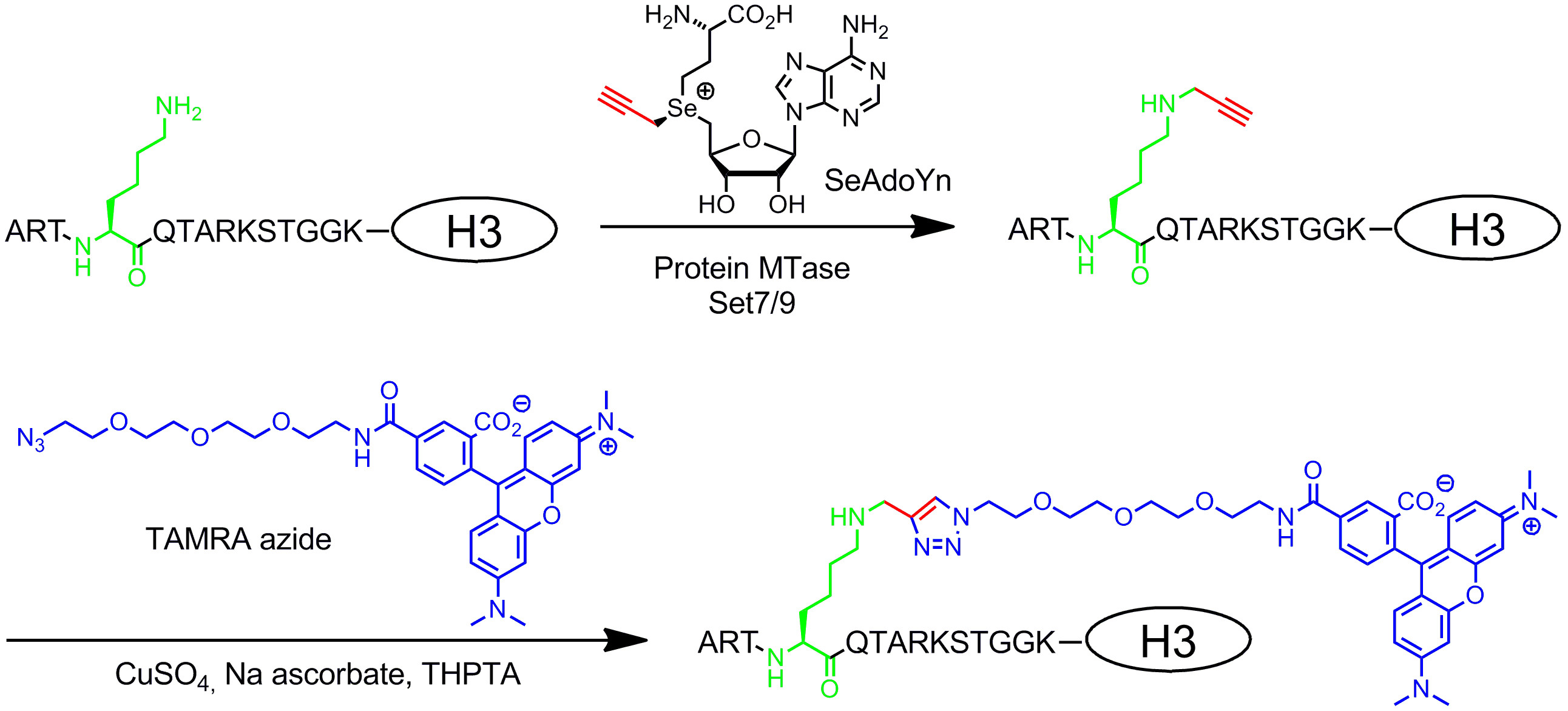
Fig. 3: Sequenzspezifische zweistufige Fluoreszenzmarkierung von Histon H3 mit Set7 / 9, SeAdoYn und TAMRA Azid Das Protein MTase Set7 / 9 natürlich methyliert die Aminogruppe des Lysins 4 in Histon H3 (H3K4, grün) mit AdoMet. Mit dem Doppel aktiviert Cofaktor SeAdoYn die MTase trägt eine kleine Propargylgruppe (rot) an den Lysinrest. Das angeschlossene Klemme Dreifachbindung wird dann selektiv in einer bioorthogonale Klick-Reaktion (Kupfer-katalysierte Azid-Alkin-Cycloaddition, CuAAC) mit Azid-derivatisierten TAMRA (Tetramethylrhodamin, blau) Fluorophor modifiziert.Last / 52.014 / 52014fig3highres.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Protokoll
1. Allgemeine Hinweise
- Speicher Aziridin Cofaktor 6BAz (in DMSO) und Protein MTase Set7 / 9 bei -80 ° C und alle anderen Reagenzien, wie doppel aktiviert Cofaktor SeAdoYn und DNA MTase M.BseCI (in 50% Glycerin) bei -20 ° C.
- Bestimmen Sie die Konzentration von 6BAz und SeAdoYn durch UV / Vis-Spektroskopie unter Verwendung des Extinktionskoeffizienten ε 269nm (6BAz) = 16.000 cm -1 M -1 und 260 nm ε (SeAdoYn) = 15.400 cm -1 M -1 in VE-Wasser. Man bestimmt die Konzentration der MTasen nach dem Bradford-Assay oder bei der Extinktionskoeffizient ist, durch direkte Absorption bei 280 nm.
- Versuchen Sie, um zu vermeiden, Blasen, die durch intensive Pipettieren oder Vortexen, um Verlust der Enzymaktivität zu verhindern. Stattdessen mischen durch vorsichtiges Auf- und Abpipettieren.
- Beim Hinzufügen Aziridin Cofaktoren aus Stammlösungen in DMSO sicherstellen, dass DMSO-Endkonzentration in dem Test wenigerals 5%. Immer auch 10 mM Magnesiumionen in dem Assay-Puffer, um nicht-spezifische Reaktionen mit DNA zu verhindern.
- Beim Hinzufügen Doppel aktiviert Cofaktoren aus sauren Stammlösungen verwenden kleine Mengen (hochkonzentrierten Stammlösungen) auf pH-Änderungen zu vermeiden und sicherzustellen, dass der pH-Wert der Testlösung nicht wesentlich ändert. Vermeiden Thiole, beispielsweise β-Mercaptoethanol oder Dithiothreitol (DTT), in dem Assaypuffer, da sie mit dem Klick-Reaktion durch Komplexbildung der benötigten Kupferionen stören können.
2. Ein-Schritt-Markierung von DNA durch Aziridin Cofaktoren
- Sequenzspezifischen Methyltransferase-induzierte Etiketten ing (lächelnd) von Plasmid-DNA mit DNA M.BseCI MTase und Aziridin Cofaktor 6BAz.
- Auftauen Cofaktor Lösung bei 20 ° C und bereiten die Reaktionsgemische auf Eis.
- Zusätzlich zur Durchführung des Assays eine "Cofaktor" -Steuerung, um alle nicht-spezifischen Modifikationen und eine & # visualisieren8220; Enzym "Kontrolle, um sicherzustellen, dass die MTase Zubereitung frei von natürlichen Cofaktor AdoMet.
- Für den Assay Mix 2 ul 10x Modifikation Puffer (enthaltend 100 mM Tris-HCl, 100 mM MgCl 2, 20 mM β-Mercaptoethanol, pH 7,4), 2 ul von pBR322 (0,5 ug / ul), 10 eq. M.BseCI pro Erkennungssequenz auf der DNA (1 Erkennungssequenz in pBR322) und dem Aziridin Cofaktor 6BAz bis zu einer Endkonzentration von 60 & mgr; M in einem Gesamtvolumen von 20 ul. Fügen Cofaktor und DNA MTase letzten.
HINWEIS: β-Mercaptoethanol ist giftig, ätzend und umweltschädliche. - Für die "Cofaktor" Kontrolle hinzufügen entsalztem Wasser anstelle von M.BseCI und für die "Enzym" Kontrolle hinzufügen entsalztem Wasser anstelle von 6BAz.
- Mischen Sie die Lösungen durch vorsichtiges Auf- und Abpipettieren.
- Die Röhrchen bei 55 ° C inkubieren 1 Std.
- Kurz zentrifugieren, um alle Flüssigkeit an dem Boden der Röhrchen zu sammeln.
- Restriktions-Modifikations-Assay zur DNA-Modifikation zu prüfen.
- Es wird eine Lösung durch Mischen von 10 ul 10x R.TaqI Puffer (enthaltend 100 mM Tris-HCl, 50 mM MgCl 2, 1 M NaCl, 1 mg / ml Rinderserumalbumin, pH 8,0), 80 & mgr; l deionisiertes Wasser und 3,3 ul der Restriktionsendonuklease (Rease) aus Thermus aquaticus (R.TaqI, 10 U / ul). Achten Sie auf die Rease im letzten Schritt hinzuzufügen.
- Zu jedem Röhrchen aus 2.1.7 hinzuzufügen 2 ul 10x R. TaqI-Puffer und 28 ul der Lösung von oben (2.2.1).
- Mischen Sie die Lösungen durch vorsichtiges Auf- und Abpipettieren.
- Die Röhrchen bei 65 ° C Inkubation 30 min.
- Kurz zentrifugieren, um alle Flüssigkeit an dem Boden der Röhrchen zu sammeln.
- Elektro Shift Assay (EMSA) mit Streptavidin funktionelle Änderung zu überprüfen.
- Entfernen von 25 ul aus jedem Röhrchen (2.2.5) und füge 2,4 ul einer Streptavidin-Lösung (1 mM in Bezug auf Streptavidin monomer in Streptavidin-Puffer, enthaltend 100 mM Na 2 HPO 4, 100 mM NaCl, pH 7,5; 4 Äquivalente Gesamt Biotin). Hinzufügen 2,4 ul Streptavidin-Puffer zu den verbleibenden Rohren.
- Alle Röhrchen bei 37 ° C inkubieren 1 Std.
- Analyse mittels Agarose-Gelelektrophorese.
- Mit 5 ul 6x Ladepuffer (0,25% Bromphenolblau, 30% Glycerin) in jedes Röhrchen.
- Mischen Sie die Lösungen vorsichtig.
- Last 10 ul jeder Probe in die Vertiefungen von einem Agarosegel (1% Agarose in 0,5x TBE-Puffer, enthaltend 1x GelRed von einem 10.000-Stammlösung).
- Führen Sie das Gel in 0,5 x TBE-Puffer mit 80 V für ca. 1 Std.
- Visualisieren DNA-Banden auf einem UV-Tisch (312 nm) mit einer CCD-Kamera mit einem Filter (540 ± 50 nm) ausgestattet ist.
HINWEIS: UV-Licht die Augen und die Haut zu beschädigen.
3. Zwei-Schritt-Protein Labeling über Doppel aktivierte Cofaktoren
- MethyltransfLöschen-Regie Übertragung von Aktivgruppen (MTAG) mit Set7 / 9 und doppelklicken aktiviert Cofaktor SeAdoYn für Histon H3 Lysin 4 Kennzeichnung (Modifikationsschritt).
- Tauen Sie die Komponenten und bereiten die Reaktionsgemische auf Eis. HINWEIS: Halten Sie immer SeAdoYn gekühlt, um den Abbau zu vermeiden.
- Zusätzlich zur Durchführung des Assays eine "Cofaktor" -Steuerung, um alle nicht-spezifischen Modifikationen zu visualisieren, und ein "Enzym" Kontrolle, um nicht-spezifische Reaktionen der fluoreszierenden Sonde auszuschließen.
- Vorbereiten einer Testlösung (20 ul), die Modifikationspuffer (50 mM Tris-HCl, 5% Glycerin, pH 8,5), 10 & mgr; M Histon H3, 10 uM Set7 / 9 und 600 & mgr; M SeAdoYn (Gemisch der beiden Epimeren in Selen). In den letzten Schritten hinzufügen Cofaktor und dann MTase.
- Für die "Cofaktor" Steuer bereiten eine Testlösung wie in 3.1.3 und fügen 60 mM AdoMet um mit dem synthetischen Cofaktor konkurrieren. Für die "Enzym" Kontrolle hinzufügen entsalztem Wasser anstelle von SeAdoYn.
- Mischen Sie die Lösungen durch langsames Auf- und Abpipettieren. Der pH-Wert durch Zugabe von 1 & mgr; l jeder Lösung in dem oberen Bereich eines pH-Streifen (pH 5 - 10).
- Bei 37 ° C für 2 Std.
- In der Zwischenzeit bereiten ein 12% SDS Polyacrylamidgel (Laufgel: 357 mM Bis-Tris, pH 6,5-6,8, 0,1% (w / v) APS, 0,04% (v / v) TEMED und 12% Acrylamid / Bisacrylamid 37,5: 1 ; Lade Gels: 357 mM Bis-Tris, pH 6,5-6,8, 0,1% (w / v) APS, 0,04% (v / v) TEMED und 5% Acrylamid / Bisacrylamid 37,5: 1).
HINWEIS: Acrylamid / Bisacrylamid ist giftig und gesundheitsgefährdenden. Während dieses Vorgangs Handschuhe tragen.
- Chemische Kennzeichnung alkinylated Lysin 4 in Histon H3 durch Kupfer-katalysierte Azid-Alkin-Cycloaddition (CuAAC) (Markierungsschritt).
- Kurz vor dem Ende der Modifizierungsreaktion bereiten 5x Klick Mischung, enthaltend 3 mM CuSO 4, 3 mM Tris (3-hydroxypropyl-triazolylmethyl) amin (THPTA), 250 mM Natriumascorbat und 6 mM TAMRA Azids mit einemGesamtvolumen von 20 ul.
- Mit 5 ul des frisch hergestellten 5x Klick Mischung zu jedem Röhrchen, um die CuAAC beginnen und löschen den Modifikationsreaktion.
- Vorsichtig mischen nach oben und unten.
- Schützen Sie alle Rohre mit Aluminiumfolie vor Licht auf Photobleaching des Fluorophors zu vermeiden.
- Inkubiere bei 20 ° C für 1 Stunde.
- Proteinfällung, um überschüssige freie TAMRA Fluorophor zu entfernen.
- Um Überstrahlen des fluoreszierend markierten Histon H3 durch intensive in-Gel-Fluoreszenz der freien TAMRA Fluorophor zu vermeiden, entfernen Sie überschüssige Fluorophor durch Fällung von Proteinen (3.3.2 - 3.3.4) 34.
- In 75 ul Methanol, 18,8 ul Chloroform und 50 ul entionisiertem Wasser zu jedem Röhrchen und vortexen kurz nach jeder Zugabe. Zentrifugieren bei 16.000 × g für 5 min. Die obere Phase ohne Störung der Grenzschicht, die das Protein enthält.
- In 56,3 ul Methanol zu der verbleibenden Phase in jedes Rohr, Wirbel und Zentrifuge bei 16.000 × g für 5 min, um das Protein zu pelletieren. Entfernen Sie den Überstand. Wiederholen Sie diesen Schritt, um das Pellet zu waschen.
- Decken Sie die offene Rohre mit einem fusselfreien Tuch und lassen Sie sie für 15 trocken - 30 min.
- Analyse über SDS-PAGE.
- Man löst die ausgefällten Proteine aus 3.3.4 in 20 ul SDS-Ladepuffer (50 mM Tris-HCl, 2,5% (w / v) SDS, 10% (v / v) Glycerin, 320 mM β-Mercaptoethanol und 0,05% (w / v) Bromphenolblau, pH 6,8). Achten Sie darauf, um das Pellet vollständig auflösen durch Spülen der Wände der Rohre mit einer Pipette.
- Inkubieren der Proben bei 95 ° C für 10 min und ließ sie abkühlen auf 20 ° C.
- Kurz zentrifugieren, um alle Flüssigkeit an dem Boden der Röhrchen zu sammeln.
- Legen der gesamten Menge von jeder Probe in die Vertiefungen eines SDS-Polyacrylamidgels (3.1.7). Benutzen 50 mM MOPS, 50 mM Tris-X (Tris-Base), 5 mM EDTA, 0,1% (w / v) SDS als Laufpuffer für die Elektrophorese.
- Führen Sie das Gel mit 120 V für ca. 90 min.
- Visualisierung der im Gel auf eine Fluoreszenz-UV-Tabelle (312 nm) mit einer CCD-Kamera mit einem Filter (540 nm ± 50 nm) ausgestattet ist.
HINWEIS: UV-Licht die Augen und die Haut zu beschädigen.
Ergebnisse
Ein-Schritt-Markierung von DNA durch Aziridin Cofaktoren
Dieses Beispiel Reaktion mit der DNA MTase M.BseCI, die den zweiten Adenin innerhalb des doppelsträngigen 5'-ATCG A-T-3'-Sequenz modifiziert ist und eine Erkennungsstelle auf dem Plasmid pBR322 (4A) durchgeführt. Um das Plasmid Kennzeichnung zu testen, wird pBR322 mit der Restriktionsendonuklease (ReaSE) R.TaqI (5'-TCGA-3 '), herausgefordert. R.TaqI sieben Stellen am pBR322 u...
Diskussion
Ein-Schritt-Markierung von DNA mit DNA MTasen und Aziridin Cofaktoren (lächelnd DNA) ist eine robuste Methode, aber einige Aspekte sollten bei der Planung des Experiments werden.
Aziridin Cofaktor: Der 6BAz Konzentration zur DNA-Markierung mit M.BseCI betrug 60 uM. Bei Verwendung anderer DNA MTasen sollte die Cofaktor-Konzentration optimiert werden, zum Beispiel so niedrigen Konzentrationen wie 20 & mgr; M wurden mit der DNA MTase M.TaqI 19 eingesetzt. Low 6...
Offenlegungen
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Danksagungen
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
Materialien
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Referenzen
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten