Method Article
Automatisierung von ChIP-seq Experimente Epigenetische Profile auf 10.000 HeLa-Zellen zu generieren
In diesem Artikel
Zusammenfassung
Methods for mapping in vivo protein-DNA interactions are becoming crucial for every aspect of genomic research but they are laborious, costly, and time consuming. Here a commercially available robotic liquid handling system that automates chromatin immunoprecipitation for mapping in vivo protein-DNA interactions with limited amounts of cells is presented.
Zusammenfassung
Chromatin immunoprecipitation followed by next generation sequencing (ChIP-seq) is a technique of choice for studying protein-DNA interactions. ChIP-seq has been used for mapping protein-DNA interactions and allocating histones modifications. The procedure is tedious and time consuming, and one of the major limitations is the requirement for high amounts of starting material, usually millions of cells. Automation of chromatin immunoprecipitation assays is possible when the procedure is based on the use of magnetic beads. Successful automated protocols of chromatin immunoprecipitation and library preparation have been specifically designed on a commercially available robotic liquid handling system dedicated mainly to automate epigenetic assays. First, validation of automated ChIP-seq assays using antibodies directed against various histone modifications was shown, followed by optimization of the automated protocols to perform chromatin immunoprecipitation and library preparation starting with low cell numbers. The goal of these experiments is to provide a valuable tool for future epigenetic analysis of specific cell types, sub-populations, and biopsy samples.
Einleitung
Als Next-Generation Sequencing (NGS) Technologien haben sich weit verbreitet und leichter zugänglich, die primäre Methode für genomweite Kartierung von Protein-DNA-Wechselwirkungen ist nun Chromatin Immunopräzipitation gefolgt von NGS-Erkennung (ChIP-seq), die die Entdeckung des Transkriptionsfaktors erlaubt Bindungsstellen oder Muster von Histonmodifikationen. ChIP-seq ist vorteilhaft bei der Bereitstellung von Hochdurchsatzdaten des gesamten Genoms, das für die quantitative und qualitative Analyse der Protein-DNA-Wechselwirkungen, die durch die Messung der angereicherten DNA-Fragmenten verwendet werden können. Es gibt jedoch einige Nachteile in Standard ChIP-seq Experimente wie die Schwierigkeit, genügend Material, um eine Sequenzierung Bibliothek zu erstellen.
ChIP-Experimente sind in sechs Grundschritte, einschließlich 1) Vernetzung von Protein-DNA-Bindungsbereiche 2) Probenvorbereitung, die Zelllyse und Scheren der Chromatin durch Beschallung, 3) Bildung der Immunkomplexe umfaßt unterteilt,4) Präzipitation der Immunkomplexe, 5) Waschen der Immunkomplexe, und 6) die Elution des angereicherten Materials und Analyse durch qPCR und NGS.
Der Erfolg eines ChIP-Test ist abhängig von drei Faktoren: ein gutes Chromatin Zubereitung, die Menge an Antigen in der ursprünglichen Probe und der Spezifität und Affinität des Antikörpers für sein zugehöriges Antigen. Eine Hauptbeschränkung ist die Voraussetzung für hohe Mengen an Ausgangszellzahlen um genügend angereicherte DNA um eine Bibliothek zu erstellen Sequenzierung zu erhalten. Für Wissenschaftler, die mit begrenzten Probenmengen, wie zum Beispiel Biopsieproben oder Zellunterpopulationen arbeiten, sind ChIP-seq Experimente sehr anspruchsvoll. Jüngste Studien haben gezeigt, dass ChIP-seq-Assays durchgeführt werden kann, wenn mit einer geringen Anzahl von Zellen 1, 2. Diagenode eine Roboterflüssigkeitshandhabungssystem, das vollständig automatisieren ChIP-seq Experimente beim Starten mit einer begrenzten Anzahl von Zellen entwickelt.
Automation bietetviele Vorteile gegenüber manuellen Erstellung von ChIP-Seq-Proben, wie es menschliche Fehler verringert, reduziert die Variabilität und reduziert experimentellen Kosten. Halbautomatische Protokolle für Chromatin-Immunopräzipitation und Bibliothek Vorbereitung berichtet worden, aber keiner dieser Studien wurde bei der Verwendung von niedrigen Zellzahlen 3, 4, 5, 6-Daten angezeigt.
In diesem Beitrag wird sowohl für Chromatin-Immunopräzipitation und Bibliothek Vorbereitung Tests in einem Roboter-Liquid-Handling-System, das Magnetic-Bead-basierten Technologie verwendet und können mehrere Parameter in der Protokolloptimierung Adresse ein kompletter automatisierter Workflow beschrieben. Hier automatisierten ChIP-seq Experimente wurden erfolgreich auf eine begrenzte Anzahl von Zellen mit dem Ziel der Vereinfachung, Standardisierung und eine zuverlässige Lösung zur epigenetischen Profile in kleinen Zellpopulationen durchgeführten Studie. Die in diesem Dokument beschriebenen automatisierten ChIP Protokoll auf HeLa-Zellen unter Verwendung von spezifischen Histon-Antikörper und Reagenzien b optimiertut des Workflows können auf andere Zelllinien und Antikörper mit entsprechenden experimentellen Optimierung angepasst werden.
Protokoll
1. Standard Chip-Experimente
- Zellsammlung und DNA-Protein-Quervernetzung.
- Wachsen HeLa-S3 Zellen auf eine Konfluenz von 80% -90%. Entfernen Kulturmedium, waschen zweimal die Schale mit 10 ml 1x phosphatgepufferter Kochsalzlösung (PBS), und fügen Sie Trypsin-EDTA (1x) auf den kultivierten Platte. Inkubieren für höchstens 2 min, um die Zellen von der Schale abzulösen. Sammeln Zellen und waschen zweimal mit 10 ml PBS.
HINWEIS: Längere Inkubationszeiten wird zu Zellschäden führen. - Zentrifugiere die Zellen 5 min bei 500 × g und Resuspendieren der Zellen in 20 ml PBS. Gehen Sie, um die Zellen zu zählen.
- Zentrifugieren Sie die Zellen für 5 Minuten bei 500 · g, den Überstand verwerfen und fügen Sie 500 ul PBS. Die optimale Anzahl von Zellen für den Fixierungsschritt wird auf 10 Millionen Zellen pro 500 & mgr; l PBS.
- Hinzufügen 13,5 ml frisches 37% iges Formaldehyd in jedem Aliquot von 500 ul der Zellsuspension. Fixieren der Zellen für 8 Minuten bei RT.
- In 57 ul von 1,25 M Glycin solutIonen, die Aufzeichnung zu stoppen. Inkubation für 5 min bei RT unter ständigem Mischen durch leichtes Wirbel. Die Arbeiten auf dem Eis ab diesem Zeitpunkt.
- Zentrifugiere die Zellen bei 500 g für 5 min bei 4 ° C und der Überstand verworfen, ohne das Zellpellet zu stören.
- Mit 1 ml PBS Waschen Sie die Zellen zweimal. Entsorgen Sie vorsichtig den Überstand und halten das Zellpellet auf Eis.
- Wachsen HeLa-S3 Zellen auf eine Konfluenz von 80% -90%. Entfernen Kulturmedium, waschen zweimal die Schale mit 10 ml 1x phosphatgepufferter Kochsalzlösung (PBS), und fügen Sie Trypsin-EDTA (1x) auf den kultivierten Platte. Inkubieren für höchstens 2 min, um die Zellen von der Schale abzulösen. Sammeln Zellen und waschen zweimal mit 10 ml PBS.
- Zell-Lyse und Chromatin Scher
- 10 ml eiskaltem Lysepuffer iL1 zu dem Zellpellet (1 ml Lysepuffer pro 1 Million Zellen ist das optimale Verhältnis). Pipette auf und ab mehrmals und Inkubation für 10 min bei 4 ° C unter leichtem Rühren.
- Zentrifugieren Sie das Lysat für 5 Minuten bei 500 xg und 4 ° C. Überstand verwerfen.
- 10 ml eiskaltem Lysepuffer iL2 den Lysaten und vorsichtig mischen durch Auf-und Abpipettieren. Die Lysate 10 min Inkubation bei 4 ° C.
- Zentrifugieren Sie für 5 Minuten bei 500 xg und 4 ° C und den Überstand verwerfen.
- Prepare den gesamten Scherpuffer Hinzufügen der 200x-Protease-Inhibitor-Cocktail (PIC) auf die iS1 Scher Puffer. Halten Sie den Puffer auf Eis für 5 min auf Eis und arbeiten danach. 1 ml des kompletten iS1 Scherpuffer in jede 10 Millionen Zellen zu pelletieren und vorsichtig mischen durch Auf-und Abpipettieren. Vor der Beschallung inkubiert die Proben auf Eis für 10 min, um die Viskosität der Probe zu verringern.
- Scheren 300 ul Aliquots von Chromatin durch Beschallung unter Verwendung eines Wasser Ultraschallbad für 2 bis 3 Sätze von 10 Zyklen. Ein Zyklus besteht aus 30 s "EIN" und 30 s "AUS" auf einem hohen Leistungseinstellung. Als Alternative kann ein pico-Beschallung Vorrichtung mit einer kürzeren Beschallungszeit von 5 bis 10 Zyklen von 30 sec "EIN" 30 sec "OFF". Vortexen und drehen die Rohre zwischen den Läufen. Bei der Verwendung von anderen Arten von Beschaller, folgen entsprechende Herstellerangaben für Chromatin Scher.
- Zentrifugieren Sie die geschert Chromatin bei 16.000 · g für 10 min und collect den Überstand sofort in das IP-Schritt verwendet werden. Alternativ speichern Sie die Chromatinstruktur bei -80 ° C für bis zu 2 Monate für die zukünftige Verwendung.
- Analysieren Sie den Chromatin Scher Effizienz vor Immunpräzipitation Schritt unter Verwendung von 1 bis 1,5% igen TAE-Agarose-Gelen oder bionalyzer. Optimale Chromatin Fragmentgrößen liegen zwischen 100 bis 600 bp.
2. Niedrige Zell ChIP Experimente
- Zellsammlung und DNA-Protein-Vernetzung
- Wachsen HeLa-S3 Zellen auf eine Konfluenz von 80% -90%. Entfernen Kulturmedium, waschen zweimal die Schale mit 10 ml 1x phosphatgepufferter Kochsalzlösung (PBS), und fügen Sie 1x Trypsin-EDTA zu der kultivierten Platte. Inkubieren für höchstens 2 min, um die Zellen von der Schale abzulösen.
HINWEIS: Längere Inkubationszeiten wird zu Zellschäden führen. - Sammle die Zellen durch Zugabe von 1 ml Kulturmedium, das Serum in einer 1 ml-Zentrifugenröhrchen. Zählen Sie die Zellen.
- Zentrifugiere die Zellen 5 min bei 500 x g. Bringen Sie die Zellzahl10.000 Zellen pro ml Kulturmedium für die Fixierung.
- In 27 ul von 36,5% frisch zubereiteten Formaldehyd in jedes Röhrchen für die Fixierung. Das Röhrchen zwei oder drei Mal, Inkubation 10 min bei RT.
- Fügen Sie 115 ul von 1,25 M Glycin-Lösung auf die Probe, das Röhrchen zwei oder drei Mal und inkubieren 5 min bei RT. Die Arbeiten auf dem Eis ab diesem Zeitpunkt.
- Zentrifugiere die Zellen bei 300 g für 10 min bei 4 ° C. Überstand verwerfen langsam.
- Waschen Sie die Zellen mit 1 ml eiskaltem HBSS mit PIC (200x, Endkonzentration 1x). Das Röhrchen zwei oder drei Mal, um die Zellen und Zentrifuge für 10 min bei 4 ° C resuspendieren bei 300 g. Entsorgen Sie vorsichtig den Überstand und halten das Zellpellet auf Eis.
- Wachsen HeLa-S3 Zellen auf eine Konfluenz von 80% -90%. Entfernen Kulturmedium, waschen zweimal die Schale mit 10 ml 1x phosphatgepufferter Kochsalzlösung (PBS), und fügen Sie 1x Trypsin-EDTA zu der kultivierten Platte. Inkubieren für höchstens 2 min, um die Zellen von der Schale abzulösen.
- Zell-Lyse und Chromatin Scher
- Man gebe 25 & mgr; l der vollständigen Lysis Buffer tL1 (Lysepuffer tL1 + PIC) pro 10.000 Zellen und rühren manuell den Boden der Röhre, um die Zellen zu resuspendieren. Inkubieren auf Eis für 5 min.
- In 75 ul von kompletten HBSS (HBSS + PIC) Puffer in jedes Aliquot, enthaltend 10.000 Zellen.
- Schere 100 ul Aliquots von 10.000 Zellen durch Beschallung für 5 Sätze von jeweils 5 Zyklus. Ein Zyklus besteht aus 30 s "EIN" und 30 s "AUS" auf dem Hochleistungseinstellung. Alternativ können Sie einen Pico-Ultraschall-Gerät mit einer kürzeren Beschallungszeit von 5 Zyklen von 30 s "EIN" 30 s "AUS" verwendet. Optimale Chromatin Fragmentgrößen liegen zwischen 100 bis 600 bp. Beachten Sie, dass Chromatin Präparate, Zelltypen und unterschiedlichen Beschaller erfordern separate Scheroptimierungsversuche.
- Zentrifugieren Sie die geschert Chromatin bei 14.000 · g für 10 Minuten, um das unlösliche Material zu verwerfen und den Überstand zu sammeln, um sofort in der IP-Schritt verwendet werden. Alternativ speichern Sie die Chromatinstruktur bei -80 ° C für bis zu 2 Monate für die zukünftige Verwendung.
- Analysieren Sie den Chromatin Scher Effizienz vor Immunpräzipitation Schritt unter Verwendung von 1 bis 1,5% igen TAE-Agarose-Gelen oderdie bionalyzer. Treat Proben mit RNase vor der Gel-Analyse, um die visuelle Beurteilung der Scher verbessern Agarose. Optimale Chromatin Fragmentgrößen liegen zwischen 100 bis 600 bp.
3. Chromatin Immunopräzipitation und Bibliothek Prep
- Für Standard-automatisierte Chip-Experimente
- Fügen Sie 120 ul von ChIP Puffer H (ChIP Puffer H + PIC) zu 100 ul geschert Chromatin. Verwenden Sie 200 ul für die IP und halten Sie 2 ul bis 20 ul als Eingangsabtastung.
- Wählen Sie das automatisierte ChIP 200 ul-Protokoll in der Automatisierungsinstrument. Der Durchsatz des Protokolls ist 1 bis 16 Proben pro Durchlauf.
- In einem System mit ChIP Experiment mit, Chromatin entspricht auf 1-2 Millionen Zellen, 1-2 ug anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 und H3K9me3 ChIP-seq Note polyklonale Kaninchen-Antikörper. Optimale Antikörper Mengen variieren je nach Histonmodifikationen und der Affinität und Spezifikation abhängigificity des entsprechenden Antikörpers.
- Verwenden gleiche Mengen von nicht-immunen Kaninchen-IgG als Isotypkontrollantikörper. Alternativ können Sie unbeschichteten Perlen oder spezifische Antikörper blockiert, wie ChIP Kontrollen. In 20 ul Protein-A-beschichteten magnetischen Kügelchen für jede Reaktion.
- Verwenden Sie die automatische Histon ChIP-seq Kitreagenzien automatisierte ChIP Experimente mit Anti H3K79me3 und -H3K4me2 polyklonalen Antikörpern durchzuführen. Nutzen Sie die ideale ChIP-seq Kit-Reagenzien zu ChIP Experimente mit Anti H3K27me3, -H3K4me3, -H3K9ac durchzuführen, -H3K9 / 14ac, -H3K36me3 und -H3K9me3.
- Wählen Sie das automatisierte ChIP-Protokoll wird die im Automatisierungsgerät implementiert Software-Anweisungen. Stellen Sie den Chip Versuchsparameter zu 4 h für die Antikörperbeschichtungsschritt und 15 Stunden für die Immunpräzipitation Schritt. Der Rückwärtsvernetzungsschritt in der automatisierten Instrument erfolgt bei 65 ° C für 4 h.
- Reinige den Rückwärts vernetzten DNA auf dem automatisierten System. Wählen Sie automated Protokolle für DNA-Reinigung mit ein Protokoll oder Kit mit magnetischen Kügelchen auf Basis der DNA-Aufreinigung. Eluieren der DNA in 25 ul Wasser.
- Quantifizierung der immunpräzipitierten DNA durch Extraktion von 10% der immunpräzipitierten DNA. Die immunpräzipitierten DNA Ausbeute hängt von der Qualität des Chromatins und der Antikörper, Zelltyp und dem Ziel Histonmodifikation. Quantifizierung der DNA unter Verwendung eines Assay-Kits nach den Anweisungen des Herstellers.
- Analysieren der Qualität der immunpräzipitierten DNA durch quantitative PCR unter Verwendung von Primern für wenigstens 1 positive und negative Kontrolle 1 genomischen Regionen. Verwenden Sie nicht mehr als 10% des gesamten immun DNA für die Bewertung ChIP Anreicherungen.
- Bereiten Sie die qPCR Reaktionen. Zugabe von 10 ul einer 2x SyberGreen qPCR-Master-Mix, 1 ul Primer-Gemisch, 1-5 ul immunpräzipitierten oder Eingabe DNA und steriles Wasser bis zu 20 ul Endreaktionsvolumen. Die qPCR Programm einen anfänglichen Denaturierungsschritt bei 95 ° C5-10 min je nach Anbieter des Taq-Polymerase und die Glühtemperaturen sollte entsprechend der gewählten Primer eingestellt werden.
- Verwenden automatisierten Bibliotheks prep Protokolle mit den im Handel erhältlichen Illumina ChIP-seq Bibliothek Vorbereitung Reagenzien, um die Bibliotheken sowohl mit ChIP DNA als auch die gespeicherten Eingabe DNA aus der gleichen Chromatin Vorbereitung bauen kompatibel. Verwenden Sie 10 bis 20 ng immunpräzipitierten DNA aus jeder Antikörper für Bibliothek Vorbereitung. Bereiten Sie bis zu 16 automatisierten Bibliotheken pro Lauf.
- Sequenz, die Bibliotheken und erzeugen Cluster nach Illumina Herstellers. Führen primären bioinformatische Analyse (Cluster-Filterung, Basis Berufung, etc.) im Anschluss an die Standard-Illumina-Pipeline, Filter und richten Sie das liest den neuesten menschliche Genom-Baugruppe (aktuelle Version ist GRCh38) mit dem ELAND Ausrichter. Verwenden SICER 7 oder 8 MACS für Spitzen Berufung und führen Sie die Downstream-Analysen der Spitzen with Homer 9, 10 oder bedtools bevorzugte Software.
- Für niedrige Zellzahl automatisierte Chip-Experimente
- Fügen Sie 120 ul von kompletten ChIP Puffer tC1 (ChIP Buffer tC1 + PIC) zu 100 ul geschert Chromatin. Verwenden Sie 200 ul für die IP und halten 20 & mgr; als Eingabe.
- Wählen Sie automatische ChIP 200 ul-Protokoll in das Automatisierungssystem. Der Durchsatz des Protokolls ist 1 bis 16 Proben pro Durchlauf.
- In einem System mit ChIP-seq Experiment mit automatisierten ChIP Reagenzien und ChIP Grade Antikörper optimiert, um auf niedrige Mengen Chromatin zu arbeiten. Verwenden Chromatin entsprechend 10.000 Zellen und 100.000 Zellen, 0,5 ug anti-H3K27me3, 0,25 ug -H3K4me3, 0,1 ug -H3K27ac, 0,25 ug -H3K9me3 Kaninchen Premium-ChIP-seq Grade polyklonalen Kaninchenantikörpern. Optimale Antikörpermengen variieren je nach Histonmodifikation und die Affinität und Spezifität des entsprechenden Antikörpers abhängig.
- Verwenden der gleichen Menge von nicht-immune rabBit-IgG als Isotypkontrollantikörper. Alternativ können Sie unbeschichteten Perlen oder spezifische Antikörper blockiert, wie ChIP Kontrollen. Zugabe von 10 & mgr; l Protein-A-beschichteten magnetischen Kügelchen für jede Reaktion.
- Wählen Sie das automatisierte ChIP-Protokoll wird die im Automatisierungsgerät implementiert Software-Anweisungen. Stellen Sie den Chip Versuchsparameter zu 4 h für die Antikörperbeschichtungsschritt und 15 Stunden für die Immunpräzipitation Schritt. Der Rückwärtsvernetzungsschritt in der automatisierten Instrument erfolgt bei 65 ° C für 4 h.
- Reinige Rückwärts vernetzten DNA mittels Spinsäulen den Anleitungen des Herstellers und eluieren in Mengen von 6 & mgr; l bis 25 & mgr; l Wasser.
- Quantifizierung der DNA mit einem handelsüblichen Test-Kit. Analysieren Sie die Ergebnisse von qPCR Verwendung von Primern für positive und negative Kontrollregionen, die Spanqualität zu bewerten.
- Verwenden Sie eine Bibliothek Vorbereitung Kit mit optimierten Bibliothek Vorbereitung Reagenzien, um Bibliotheken mit niedriger DNA quan vorbereitenmengen. Verwenden Sie 30 pg bis 300 pg von ChIP DNA (entsprechend 10.000 und 100.000 Zellen Experimente beziehungsweise) für Bibliotheks Vorbereitung. Bereiten Sie Bibliotheken mit Hilfe der automatisierten Protokoll mit Bibliothek Vorbereitung Reagenzien kompatibel. Die automatisierte Bibliothek prep Durchsatz 1-48 Bibliotheken pro Lauf.
- Nach dem Ende Reparatur der Doppelstrang-DNA-Matrizen, ligieren spaltbaren Stamm-Schleifenadaptern, die die Sequenzierungsprimer-Sites. Nach der DNA-Verlängerungsschritt, verstärken die Probe mit der in der Bibliothek Vorbereitung Kit Protokoll beschrieben high fidelity Amplifikationsverfahren.
- Nach Bibliothek Verstärkung, zu quantifizieren und zu reinigen, die Bibliotheken nach der Vorbereitung Kit Leitlinien Bibliothek. Beachten Sie, dass Größenauswahl nach der Reinigung ist nicht erforderlich.
- Sequenz, die Bibliotheken und erzeugen Cluster gemäß den Anweisungen des Herstellers. Führen primären bioinformatische Analyse (Cluster-Filterung, Basis Berufung, etc.) nach dem Standard Hersteller Pipeline, Filter, und richten Sie das liest den neuesten menschliche Genom-Baugruppe (aktuelle Version ist GRCh38) mit dem ELAND 7 Ausrichter. Verwenden SICER 8 oder 9 MACS für Spitzen Berufung und führen Sie die Downstream-Analysen von Gipfeln mit Homer, bedtools 10, oder eine bevorzugte Software.

Abbildung 1: Screenshots der Software zeigt, wie automatische Chip-Experimente in der IP-Stern-Vertrag einzurichten. Die Software bietet die Flexibilität, die Menge an Proben pro Durchlauf sowie zu den wichtigsten Versuchsparameter (Antibody Coating, IP und wäscht ändern wählen ) gemäß den Forscher braucht. Das automatisierte Verfahren erlaubt das Testen verschiedener Bedingungen parallel (dh verschiedene Typen und Mengen von Antikörpern verschiedener Arten und Mengen von Zellen und sogar unterschiedliche typen und Mengen von magnetischen Kügelchen in einem Testansatz. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.

Abbildung 2: Screenshots der Software zeigt, wie Sie automatisierte Bibliothek Vorbereitung der nächsten Generation Sequenzierung unter Verwendung des Bibliothek-Kit in das Automatisierungssystem. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Ergebnisse
Optimierung automatisierter ChIP-seq Experimente für acht verschiedene Histon-Marker
Für die erfolgreiche Entwicklung und Validierung von automatisierten ChIP Protokolle ChIP-seq Grade Antikörper, die zuvor in Hand ChIP-Seq Experimente validiert wurden (Daten nicht gezeigt) ausgewählt wurden. Anti H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, -H3K9 / 14ac, -H3K36me3 und -H3K9me3: Die folgenden ChIP-seq Grade Antikörper wurden für diese Studie ausgewählt. Die Spezifität aller ChIP-seq relevanten Antikörpern wurde zuvor durch Dot-Blot, Peptid-Arrays und Western Blot Experimente bestätigt (Daten nicht gezeigt). Steuer-Chip-qPCR-Experimente mit steigender Antikörpermengen wurden durchgeführt, um die Empfindlichkeit der Antikörper (Figur 3) zu bestimmen. qPCR mit mindestens zwei positive und zwei negative Kontrollziele analysiert und Profile mit Anreicherungen von positiv in negativ Ziel höher als verfünffacht werden für die Sequenzierung exper qualifiziert Formen. Es ist wichtig, Chip und ChIP-seq Experimente mit einer hohen Qualität der geschert Chromatin durchzuführen. Alle in dieser Publikation enthaltenen ChIP Experimente wurden unter Verwendung frischer Chromatin. Es ist auch möglich, die fixierten Zellen bei -80 ° C einfrieren und fahren mit dem Chromatin Zubereitung und Scherung an einem anderen Tag. Allerdings Chromatin aus gefrorenen fixierten Zellen hergestellt wird, kann vom Chromatin frisch zubereitet und deshalb Beschallung Bedingungen müssen möglicherweise für jede Chromatin Vorbereitung optimiert werden unterschiedlich verhalten. Bei der Arbeit mit verschiedenen Zelltypen können Scher Puffer mit verschiedenen Reinigungsmittelzusammensetzungen (SDS) verwendet werden. Zelltypen, wie primäre Zelllinien oder Zellen in Suspension gezüchtet sind schwierig Zellen zu scheren und hoher SDS-Konzentrationen (1%), während die Zelllinien, die leicht zu scheren, wie HeLa niedrig SDS-Konzentrationen (0,1%) in die erforderlich sind, erfordern Scher Puffer.
res.jpg "/>
Abbildung 3. Validierung von ChIP Grade Antikörper mit dem Automatisierungssystem. Chip wurde mit Anti-H3K79me3, -H3K27me3, -H3K4me3, -H3K4me2, -H3K9ac, H3K9 / 14ac, durchgeführt -H3K36me3 und -H3K9me3 polyklonalen Kaninchenantikörpern auf geschert Chromatin aus 1 Million HeLa-S3 Zellen in Abhängigkeit von der Histon-Modifikationen. Automatisierte ChIP Protokolle mit 200 ul Arbeitsvolumen wurden bei der Automatisierung Instrument für eine Antikörper-Titration Experimente verwendet. Antikörper Mengen von 1, 2, 5 und 10 ug wurden pro Chip-Experiment getestet und 2 ug IgG wurden als negative Kontrolle in jedem Experiment verwendet. Anreicherungen wurden von qPCR bewertet. Die Ergebnisse sind als% der Eingang (der relativen Menge an immun DNA im Vergleich zum Ausgangs-DNA nach der qPCR-Analyse) angezeigt. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Nach der Prüfung und Ermittlung optima l Mengen ChIP-grade Antikörper in dem Automatisierungssystem eingesetzt werden, wurden automatisierte ChIP-seq Versuche zur Sequenzierung Profile für jeden Histonmodifikation (Figur 4) zu erzeugen, durchgeführt wird.

Abbildung 4. Die Histon-ChIP-seq Profile durch automatisierte ChIP-seq Experimente erzeugt werden. Die Abbildung zeigt die ChIP-seq Profile in verschiedenen genomischen Regionen für H3K4me3, H3K9ac und H3K9 / 14acH3K4me2 H3K79me3 und H3K36me3. 4A zeigt die Spitzenverteilung entlang der kompletten X -chromosome und 4B die Verteilung in einer 75 kb Umgebung des GAPDH-Gens. 4C zeigt die Profile der H3K27me3, H3K36me3 und H3K4me3 in einem 500 kb Bereich um den Myt1 Gen und 4D zeigt die Verteilung der H3K9me3 in einem 200 kb Bereich um ZNF12.large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Histone epigenetische Profile für sechs verschiedene Histon-Modifikationen mit Genexpression assoziiert wurden erzeugt (H3K4me3, H3K9ac, H3K9 / 14ac, H3K4me2, H3K79me3 und H3K36me3). 4A zeigt ChIP-seq Profile entlang der X-Chromosom für die verschiedenen Histon-Marker. Die zwischen den 6 verschiedenen Histon Profile beobachtet hochSpitzenKorrelations zeigt die Fähigkeiten von automatisierten System, genaue und zuverlässige Daten zu generieren. 4B, 4C und 4D zeigen die Verteilung von Spitzen für verschiedene Histon-Modifikationen an spezifischen genomischen Regionen.
Automatisierte Chromatin-Immunopräzipitation Experimente bis zu 200 Zellen
Die Mindestmenge an Zellen, die in der Chip-Experimenten verwendet werden können, hängt von der Qualität des Chromatins, die specificity und der Empfindlichkeit des Antikörpers und die Fülle des Histon-Modifikation oder Protein untersucht. Auswahl guter ChIP-seq relevanten Antikörpern ist wichtig, wenn mit geringen Mengen der Probe und der Auswahl von optimalen Reagenzien und verschiedene Träger verbessert die Effizienz der DNA-Rückgewinnung und zum Erfolg des Chip-Experiment. Zur Bestimmung der Mindestmenge von Zellen, die das automatisierte ChIP-Protokoll verarbeiten können unterschiedliche Mengen von Chromatin, Antikörper und magnetischen Kügelchen wurden in der IP-Stern automatisiertes System mit ChIP Reagenzien speziell optimiert, um mit geringen Mengen von Chromatin arbeiten getestet.
Zuerst wurde das Chromatin von 10.000 Zellen wie in dem Protokoll beschrieben beschallt. Der Chip-Ergebnisse wurden durch qPCR (5A), die signifikante Anreicherungen mit H3K4me3 Antikörper in positive Kontrollregionen und vernachlässigbar Signal in negativer Kontrollregionen zeigt bestätigt. Zum Vergleich und der Nachweis der Konsistenz, additional Daten mit H3K27ac, H3K9me3 erhalten und H3K27me3 Antikörper mit 10.000 Zellen ist.
Automatisierte ChIP-Experimente wurden durchgeführt, um die Fähigkeiten des automatisierten Systems zu demonstrieren, mit geringen Mengen von Zellen unter Verwendung der gleichen H3K4me3 Antikörper funktionieren. Automatisiertes ChIP gut entwickelt, die durch eine Reihe von zehn IP Reaktionen, die reproduzierbar und sehr vergleichbar mit den manuellen ChIP Ergebnisse (5B) wurden nachgewiesen. Manuelle und automatisierte Experimente wurden durchgeführt und die Vorteile der automatisierten Protokolle wurden bei der Verringerung der Versuch, um die Variabilität (5C) experimentieren gesehen.
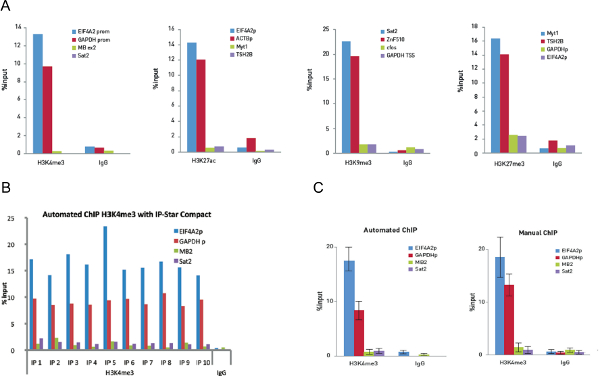
Abbildung 5. Optimierung der Chip und Auto ChIP Experimente an 10.000 Zellen Manuelle ChIP Experimente wurden an 10.000 Zellen durchgeführt und unter Verwendung von 0,25 ug H3K4me3, 0,1 ug H3K27ac, 0,5 ug H3K9me3 und 0,25 ug H3K27me3 Antikörper. Identische Mengen an Kaninchen-IgG wurden als Kontrolle verwendet. Die qPCR wurde mit Primern für zwei positive und zwei negative Loci Loci für jeden Chip-Assay durchgeführt. 5A zeigt die Erholung, ausgedrückt als Prozentsatz der Eingang (der relativen Menge des immunpräzipitierten DNA im Vergleich zur Ausgangs-DNA nach qPCR-Analyse). 5B zeigt 10 ChIP Reaktionen auf die IP-Stern-Vertrag mit 0,25 ug H3K4me3 polyklonalen Antikörper und 0,25 ug Kaninchen-IgG-Antikörper als negative Kontrolle ausgeführt werden. Dann wurde qPCR-Analyse mit Primern für die positive loci EIF4A2 Promotor und GAPDH TSS und der negativen loci Myoglobin exon2 und Sat2 geführt. Die Figur zeigt die Gewinnung, ausgedrückt als Prozent der Eingang (relative Menge der immunpräzipitierten DNA im Vergleich zur Ausgangs-DNA nach qPCR-Analyse). 5C zeigt H3K4me3 Chip-Daten von 10 Hand ChIP-Experimente im Vergleich zu 10 automatisiert ChIP-Experimente. Fehlerbalken stellen s tandard Abweichungen von jedem der zehn Wiederholungen. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
Um die Empfindlichkeit der automatischen ChIP Protokolle zu verstehen, wurden Experimente durchgeführt unter Verwendung einer Menge von Zellen, die von 100.000 bis 200 Zellen pro IP reichten. Das Anti H3K27me3 Antikörper wurde verwendet, da es eine sehr häufige Histonmodifikation. Die Verwendung anderer Histon oder Nicht-Histon-Antikörper können mehr oder weniger Zellen in Abhängigkeit von der Häufigkeit des Epitops und die Qualität des Antikörpers. Die Experimente wurden durch quantitative PCR bestätigt und es wurde beobachtet, dass durch die Reduzierung Mengen von Perlen und Antikörper-Hintergrund in den Experimenten reduziert und die erfolgreiche ChIP-qPCR Ergebnisse mit so wenig wie 200 Zellen Antikörper (Figur 6) wurde.
iles / ftp_upload / 52.150 / 52150fig6highres.jpg "/>
Abbildung 6. Automatisierte ChIP Assays auf 200 Zellen. Hela-S3-Zellen und Antikörper gegen H3K27me3 gerichtet. Chromatin wurde von 1 Million Zellen und Reihenverdünnungen dieser Chromatin (von 100.000 auf 200 Zellen Äquivalente) pro Chip Reaktion verwendet geschert. 1 ug H3K27me3 und 10 ul Protein-A-beschichteten Magnetperlen wurden auf 100.000 Zellen Experiment verwendet wurde, 0,5 ug H3K27me3 und 10 ul Perlen auf 10.000 und 1.000 Zellen und 0,25 ug H3K27me3 und 5 & mgr; l Kügelchen, die mit 500 und 200 Zellen. 1 & mgr; g und 0,5 & mgr; g Kaninchen-IgG wurden als negative Kontrollantikörper bei der Durchführung von Experimenten mit 100.000 Zellen und 1.000 Zellen verwendet wurden. 6A zeigt die Belegung von TSH2B und GAPDH-Gene in% gegenüber dem Eingang. 6B zeigt relative Belegung TSH2B gegenüber negativen GAPDH Kontrolle genomischen Region.
Downstream Analyse ChIP-seq Ergebnisse auf10.000 Zellen
Um die globale Qualität der automatisierten ChIP-seq Experimente mit niedrigen Ausgangszellzahlen, automatisierte ChIP-seq Assays wurden mit 0,25 ug des H3K4me3 Antikörper auf 10.000 HeLa-Zellen und ChIP Experimente beurteilen auf 100.000 HeLa-S3-Zellen wurden verwendet, als positive Kontrolle für das Experiment. Die automatisierten Bibliotheken wurden unter Verwendung der MicroPlex Bibliothek Vorbereitung Kit Reagenzien bereit Bibliotheken mit niedriger DNA-Mengen angepasst vorbereitet. Beachten Sie, dass, auch wenn es möglich ist, erfolgreich automatisiert ChIP-Experimente mit weniger als 10.000 Zellen durchführen, werden die Beträge der nach unten gezogen DNA nicht aus, um Bibliotheken mit den Kit-Reagenzien vorzubereiten. Cluster Generation und Sequenzierung wurden nach den Anweisungen des Herstellers durchgeführt. Die Bioinformatik-Analysen nach der Sequenzierung zeigen hervorragende Ergebnisse von den niedrigen Zellzahl Splitterproben. Die 30 pg Datensatz (entsprechend 10000 Zellen des Ausgangsmaterials ) Enthalten geringe Hintergrundgeräusche und sehr zuverlässig Bereicherung Spitzen, die sowohl von der 300 pg Datensatz (entspricht 100.000 Zellen des Ausgangsmaterials) und der H3K4me3 Datensatz vom Broad Institute des ENCODE Projekt, das als externe Referenz verwendet wurde erzeugt bestätigt werden. Es ist wichtig zu beachten, die Top 40 Überdeckung Daten, die zu einer Standardmethode in der ENCODE-Projekt 11, in die das ChIP-seq wird als reproduzierbar, wenn Vergleich von zwei Datensätzen gibt es zumindest eine Überlappung der besten 40% 80% bezieht sich der Gipfel von Bedeutung Score rangiert. Die 30 pg Datensatz erfüllt diese Kriterien im Vergleich zu sowohl der 300 pg Datensatz (unter Berücksichtigung aller seiner Gipfel, nicht nur die besten 40%) und die Grundzüge der Institute Daten (Tabelle 1). Die 300 pg Datensatz zeigt fast identische Gipfel, um Broad Institute Daten mit einem 98% Top 40 Überdeckung (Abbildung 7).
ighres.jpg "/>
Abbildung 7. ChIP Assays und Bibliothek Generation auf 10.000 Zellen ChIP-seq Experimente wurden an 10.000 und 100.000 HeLa-S3-Zellen mit H3K4me3 Antikörper (0,25 ug / ul) erzeugt. Wurden die 35 bp-Tags, um das menschliche Genom mit dem ELAND Ausrichter abgebildet. Bei der anschließenden Spitzen Aufruf SICER verlässlich identifizieren die Anreicherungen von niedrigen Zellzahlen als auch von Millionen von Zellen. Die Datensätze wurden analysiert und miteinander verglichen und auf die vom Broad Institute erzeugten Referenzdaten. Die niedrigen Zellproben stimmen und haben eine sehr hohe Ähnlichkeit. Die 30 pg Probe erfüllt das ENCODE Kriterien 11 (min. 80% der oberen 40% der Spitzen überlappen). Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
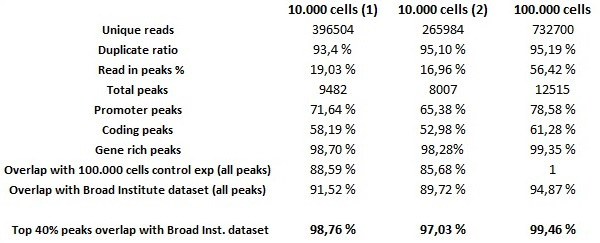
Tabelle 1.
Diskussion
Chromatin-Immunopräzipitation gefolgt von Sequenzierung ist heute ein Standardverfahren. Hier ein automatisiertes ChIP-seq Protokoll, Chromatin epigenetische Profile mit nur 10.000 Zellen des Ausgangsmaterials erzeugen kann, wird dargestellt.
Automatisierung von Chip und Bibliothek Vorbereitung Assays ermöglicht die Standardisierung der ChIP Optimierungsverfahren und die Verringerung der experimentellen Variabilität. Die Liquid-Handling-System hier vorgestellten beseitigt viele der manuellen Verfahren mit Chip reduziert die Hände auf Zeit, nur 30 Minuten verbunden ist, minimiert Probenverluste und ermöglicht eine genaue ChIP-seq mit nur wenigen Picogramm Bibliothek Eingang. Um eine erfolgreiche automatisierte ChIP-seq Experimenten zu erreichen, ist es auch von entscheidender Bedeutung, um qualitativ hochwertige geschert Chromatin Präparate und ChIP-seq Grade Antikörper bei jedem Versuch das System verwenden verwendet Magnetic Bead-basierte Technologie und bietet Flexibilität, Hauptversuchsparameter wie Inkubation ändern Zeit für die Antikörperbeschichtunging und Immunpräzipitation Schritte oder Änderung der Wasch Bedingungen, die die Forscher auf, alle notwendigen Versuche für ChIP-seq-Optimierung durchzuführen. Das automatische System ist ein "offenes" -Plattform, die auch ermöglicht Vergleich mehrerer Reagenzien parallel zur Optimierung der experimentellen Bedingungen für die einzelnen Zelllinie und Antikörper und ermöglicht den direkten Vergleich der verschiedenen Typen und Konzentrationen von Chromatin, verschiedenen Antikörpern und sogar verschiedene Typen magnetischer Perlen.
Eine der Einschränkungen des automatisierten Systems ist die Notwendigkeit der Automatisierung aller Protokolle in Mengen, die von 5 & mgr; l bis 200 & mgr; l liegen. Allerdings ist die Miniaturisierung der Experimente in diesem automatisierten Plattform ermöglicht auch die Kosten in Reagenzien sparen.
Zusätzlich zu den in dieser Studie beschriebenen Protokollen ist das System flexibel und automatisiert eine Vielzahl von anderen magnetischen Kügelchen basierte Anwendungen wie Immunpräzipitation a auchnd Erfassung von methylierter DNA (MeDIP und MethylCap Technologien), Immunpräzipitation hydroxylmethylated DNA (hMEDIP), sequentielle Chromatin-Immunopräzipitation (ReChIP), RNA Immunpräzipitation (RNA-IP), Bisulfit-Konvertierung und DNA-Reinigung-Assays.
Offenlegungen
The authors of this article, at the time of its writing, are employed by Diagenode S.A. and Diagenode, Inc., the manufacturer of the automated system described.
Danksagungen
This work was supported by the BLUERPINT EU grant (BLUEPRINT – A BLUEPRINT of Haematopoietic Epigenomes). We also thank the Walloon Region (DG06) for its financial support.
Materialien
| Name | Company | Catalog Number | Comments |
| PBS | Life technologies | 14190-094 | |
| Trypsin-EDTA | Sigma | T3924-100ML | |
| Formaldehyde 37% | Sigma | F8775-25 | |
| 1.25 M Glycine Solution | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL2 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Shearing Buffer iS1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Protease Inhibitors Mix (200x) | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| HBSS (no calcium, no magnesium, no phenol red) | Life technologies | 14175-053 | |
| Lysis Buffer tL1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ChIP Buffer tC1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ideal ChIP-seq kit | Diagneode | C01010051 | |
| ChIP-Buffer H | Diagenode | C01010020 | Component of the Auto Histone ChIP-seq kit |
| Auto Histone ChIP-seq kit | Diagenode | C01010020 | |
| Auto True Micro ChIP kit | Diagenode | C01010130 | |
| H3K79me3 polyclonal antibody-Classic | Diagenode | C15310068 | |
| H3K27me3 polyclonal antibody-Classic | Diagenode | C15410069 | |
| H3K4me3 polyclonal antibody-Classic | Diagenode | C15410030 | |
| H3K4me2 polyclonal antibody-Classic | Diagenode | C15410035 | |
| H3K9ac polyclonal antibody-Premium | Diagenode | C15410004 | |
| H3K9/14ac polyclonal antibody-Premium | Diagenode | C15410200 | |
| H3K36me3 polyclonal antibody-Premium | Diagenode | C15410058 | |
| H3K9me3 polyclonal antibody-Premium | Diagenode | C15410193 | |
| Rabbit IgG | Diagenode | C15410206 | |
| Protein-A coated paramagnetic beads | Diagenode | C01010020 | |
| Auto IPure | Diagenode | C03010010 | |
| MicroChIP DiaPure columns | Diagenode | C03040001 | |
| Universal SyberGreenMaster Mix 1.25 ml | Diagenode | DMMLD2D100 | |
| Quant-IT dsDNA | Invitrogen | Q32854 | |
| Illumina Sample Preparation kit fro Genomic DNA | Illumina | FC-121-3001 | |
| Illumina True-seq kit ChIP library Prep kit | Illumina | IP-202-1012 | |
| MicroPlex Library Preparation Kit | Diagenode | C05010010 | |
| Agencourt AMPure XP beads | Beckman Coulter | A63881 | |
| Illumina Library prep Quantification kit | Kapa Biosystems | KK4844 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | |
| Bioruptor Plus | Diagenode | B01020001 | |
| Bioruptor Pico | Diagenode | B01060001 | |
| Qubit system | Invitrogen | Q32857 | |
| Illumina Hiseq systems | Illumina |
Referenzen
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Adli, M., Bernstein, B. E. Whole-genome chromatin profiling from limited numbers of cells using nano-ChIP-seq. Nature Protocols. 6, 1656-1668 (2011).
- Blecher-Gonen, R., et al. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nature Protocols. 8, 539-554 (2013).
- Aldridge, S., et al. AHT-ChIP-seq: a completely automated robotic protocol for high-throughput chromatin immunoprecipitation. Genome Biology. 14, 124 (2013).
- Farias-Hesson, E., et al. Semi-automated library preparation for high-throughput DNA sequencing platforms. Journal of Biomedicin., & Biotechnology. 2010, (2010).
- Callejas, S., Alvarez, R., Benguria, A., Dopazo, A. AG-NGS: a powerful and user-friendly computing application for the semi-automated preparation of next-generation sequencing libraries using open liquid handling platforms. BioTechniques. 56, 28-35 (2014).
- Zang, C., et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics. 25, 1952-1958 (2009).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9, 137 (2008).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Molecular Cell. 38, 576-589 (2010).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics (Oxford, England). 26, 841-842 (2010).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten