Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Der Nachweis von exosomalen Biomarker von Electric Field-induzierte Freisetzung und Messung (Efirm)
In diesem Artikel
Erratum Notice
Zusammenfassung
Exosomes are microvesicular structures found within biofluids that potentially carry important disease discriminatory biomarkers. Here, a novel method is used to specifically extract exosomes and rapidly test the exosomal cargo for both RNA/protein targets following the disruption of exosomes using non-uniform electric cyclic square waves.
Zusammenfassung
Exosomen sind mikrovesikulären Strukturen, die eine Vermittlerrolle in der interzellulären Kommunikation spielen. Es ist von Interesse, um die innere Ladung von Exosomen studieren, um festzustellen, ob sie Krankheit diskriminierenden Biomarker tragen. Zur Durchführung exosomalen Analyse ist es notwendig, ein Verfahren zur Extraktion und Analyse von Exosomen Ziel biofluids ohne Beschädigung der internen Inhalte zu entwickeln.
Elektrisches Feld-induzierte Freisetzung und Messung (Efirm) ist ein Verfahren zur spezifischen extra Exosomen aus biologischen Flüssigkeiten, Entladen ihre Ladung, und testen ihre internen RNA / Proteingehalt. Verwendung eines anti-human CD63 spezifische Antikörper magnetische Mikroteilchen werden Exosomen zuerst aus Biofluiden fällt. Nach der Extraktion, Niederspannungs-Elektro zyklischen Rechtecksignale (CSW) angewendet, um die Vesikelmembran stören und Fracht entladen. Der Inhalt der Exosom, mit DNA-Primer oder Antikörper auf einer Elektrodenoberfläche für qua immobilisierten hybridisiertenntification molekularer Inhalte.
Die Efirm Methode ist für die Extraktion von Exosomen und Entladen von Fracht zur Analyse ohne Lysepuffer vorteilhaft. Dieses Verfahren ist in der Lage, die spezifische Detektion von beiden RNA und Protein Biomarker Ziele in der Exosomen. Efirm extrahiert Exosomen speziell auf ihre Oberflächenmarker basiert im Gegensatz zur Größe basierte Techniken.
Transmissionselektronenmikroskopie (TEM) und Assay zeigen die Funktionalität des Verfahrens für die Exosom Erfassung und Analyse. Die Efirm Methode wurde die Analyse von 9 Mäusen mit menschlichen Lungenkrebs H640-Zellen injiziert (eine Zelllinie transfiziert, um die Exosom Marker menschlichen CD63-GFP exprimieren) um ihre exosome Profil gegen 11 Mäusen, die Kochsalzlösungskontrollen getestet exosomalen. Erhöhte exosomalen Biomarker (Referenz-Gen GAPDH und Proteinoberflächenmarker CD63 Menschen-GFP) gefunden für das H640 injizierten Mäusen in beiden Serum und Speichelproben. Außerdem saliva und Serumproben wurde gezeigt, dass die Linearität (R = 0,79) haben. Diese Ergebnisse lassen auf die Lebensfähigkeit der Speichel exosome Biomarker zum Nachweis von distalen Krankheiten.
Einleitung
Exosom Forschung ist ein aufstrebendes Gebiet der Untersuchung, die Lipid-Mikrobläschen, die RNA-1, 2-DNA und Protein 3 Fracht transportieren sucht. Frühere Untersuchungen von Exosom Biologie haben, um die Identifizierung von Exosomen in Körperflüssigkeiten wie Blut 4, 5 Urin, Muttermilch 6, und Speichel 7 geführt. Studien haben gezeigt, dass Exosomen spielen eine Rolle bei verschiedenen zellulären Wege, fernmedi Kommunikation zwischen verschiedenen Systemen des Körpers 8. Aufgrund der Rolle in Exosomen der interzellulären Kommunikation spielt, wird die Hypothese aufgestellt, dass sie Biomolekül Targets (Protein, RNA und DNA) mit Krankheitszuständen korreliert verpacken. In vitro 3 und Tiermodellstudien 9 angezeigt, um diese Hypothese zu bestätigen. Bei der Untersuchung exosomalen Inhalte für die Entdeckung neuer Biomarker, ist es notwendig, eine Methode für die selektive Exosom Isolation von Körperflüssigkeiten zu entwickeln, induzierte expulsiauf der Ladung von Exosomen und Quantifizierung von Exosom Biomolekülen. In dem Umfang der vorliegenden Arbeit wird Exosomen als eine Struktur mit einem Durchmesser von ca. 70-100 nm und besitzen Oberflächenmarker CD63 definiert werden.
Forscher in der Regel zunächst zu reinigen Exosomen durch Ultrazentrifugation 10 und dann exosomalen Inhalte durch den Einsatz von Lysepuffer Sätze zu verarbeiten. Usage Lysepuffer Verfahren erfordert Inkubationszeiten reichen von Minuten bis Stunden. Dieser Vorgang kann möglicherweise Schaden zufügen Exosom Ladung und führen zu einer Verschlechterung zu probieren. B. Speichel exosome RNA über Lysepuffer in die umgebende extrazelluläre Umgebung freigesetzt besitzt eine Halbwertszeit von weniger als 1 min, wodurch Messung exosomalen RNA post Lysepuffer eine besonders schwierige Aufgabe, ohne den Zusatz von Stabilisierungsreagenzien 11. Das compoundierte Wirkung der Zugabe verschiedener Reagenzien zur Lyse und Stabilisierung können Mittel, die erschweren einzuführen und stören die analysis von exosomalen Inhalt. Eine alternative Vorgehensweise kann hilfreich für die schnelle Entladung exosomalen Inhalt und sicher die Erhaltung der Ladung zur Charakterisierung sein.
In dieser Arbeit schlagen wir die Verwendung eines nicht-gleichmäßiges elektrisches Feld für die Freigabe exosomalen Inhalt. Elektrofelder bekannt, um die Fähigkeit zu polarisieren und stören die Lipid-Doppelschicht, die Zellmembranen bildet durchzuführen. Unsere experimentelle Arbeit untersucht Nutzung ungleichmäßige zyklischen Rechtecksignale (CSW) zur Unterbrechung der microvesicle Struktur der Exosomen und Freigabe geführt Fracht. Bei dieser Methode werden Spannungen in der mehrere hundert Millivolt-Bereich, was bedeutet, dass die meisten Biomoleküle nicht gestört werden. Wir zeigen, dass die Verwendung einer zyklischen Rechteckwellen in der Lage ist, um die Freisetzung von Speichel exosome mRNA-Gehalt in das umgebende Fluidumgebung zu betätigen. Diese Version von exosomalen Inhalt ist nahtlos mit einem Elektrodensystem, die verwendet werden können, um die Biomarker-Expressionsniveaus zu quantifizieren integriert 12,13. Dieses vorgeschlagene Verfahren ermöglicht eine schnelle, empfindliche und Lysepuffer kostenlose Analyse Exosom Inhalt.

Abbildung 1. Übersicht über Efirm-Workflow.. Die Efirm Verfahren wird im Großen und Ganzen in die drei Hauptphasen, die zur Reinigung und Analyse von Exosomen notwendig sind geteilt.
Das CSW auf Basis exosomalen Content-Release und Analyseverfahren wird in Verbindung mit CD63-spezifischen magnetischen Mikrokügelchen für Exosom Isolierung verwendet. Diese CD63-Affinität Perlen ermöglichen die selektive Isolierung von Exosomen aus Speichelproben (und andere Körperflüssigkeiten). Nach der Inkubation und Gewinnung von Exosomen mit den magnetisierten Kügelchen werden die Perlen mit dem elektrochemischen Sensor-System für die CSW basierte Content-Release und Analyse Teil des Experiments migriert. Abbildung 1 gibt einen Überblick über die Arbeitfließt der Efirm Verfahren.
Protokoll
1. Magnetic Bead-basierten Exosom Extraction
- Pipettieren gut gemischte Lösung von 5 & mgr; l Streptavidin-beschichteten magnetischen Mikropartikeln in 495 ul phosphatgepufferter Salzlösung (PBS) -Puffer in einem Mikrozentrifugenröhrchen, um die Perlen zu resuspendieren. Verwendung eines Magnetgestell Waschen und Resuspendieren der Kügelchen mit 500 ul PBS dreimal. Das Gestell ist eine Anordnung von Magneten auf der Seite einer Gehäuseeinheit, die die Probe-Mikrozentrifugenröhrchen halten kann.
- Für jede Waschanlage, lassen Sie uns zuerst die Rohre sitzen auf dem Rack für 1 min, und dann mit einem Pipettenspitze vorsichtig den Überstand Puffer ohne die Perlen zu stören.
- Die Röhrchen auf einer regulären Zahnstange ohne Magnete an der Seite. Fügen Sie 500 ul PBS in die Rohre, und verwenden Sie die Pipette, um die Lösung und Perlen zusammen mischen. Dann legen Sie die Rohre wieder auf dem Magnetgestell wieder trennen Sie die Perlen aus der Lösung.
- Führen Sie diese Entfernung der Puffer über Magnetisierung und Resuspension in PBS eininsgesamt dreimal. Dies führt eine erste Wasch der magnetischen Teilchen.
- Resuspendieren der Kügelchen in 490 & mgr; l PBS-Puffer, mit dem Rohr auf dem nicht-magnetisierten Teil der Magnetgestell gestellt. Pipette 5 ul biotinylierten Maus-anti-Mensch-CD63-Antikörper mit 1,0 mg / ml Ausgangskonzentration in dem Gemisch von Perlen. Verwenden Sie die Pipette, um die Perlen und Antikörper in Lösung zu mischen.
- Legen Sie die Reaktionsgefäße mit Perle und biotinyliertem Antikörper-Gemisch auf eine Probe-Rotator. Festlegen der Rotator Parametern für die Probe für reziproke Rotator Drehung bei 90 ° Neigung für 5 sec und Vibrieren bei 5ºC für 1 sec. Drehen der Probe-Perlen-Mischung Röhrchen bei diesen Parametern für 30 min bei RT.
- Entfernen ungebundener Antikörper nach der Konjugation.
- Nach 30 Minuten Dreh bei RT, legen Sie die Rohre wieder in den Magnetträger für 5 min.
- Führen Sie drei Wäschen von Perlen durch Entfernen der flüssigen Phase mit einer Mikropipette und wäscht mit 500 μ; L PBS. Nach dem dreifachen Waschen, resuspendieren die Perlen in 490 ul von Casein-PBS und auf den unmagnetisierten Teil des Racks.
- Exosom Extraktion mit Antikörper-beschichteten Perlen.
- Beschriften Sie jedes Rohr mit gezielten Proben-ID. Pipettieren Sie 10 ul-Probe von Serum oder Speichel in das Reaktionsgefäß. Verwenden Sie die Pipette, um die Probe und magnetischen Beads durch mehrmaliges Pipettieren mischen.
- Die Röhrchen mit der Probe und anti-Mensch-CD63-Antikörper-Perlen auf Rotator und drehen für 2 h bei RT. Verwenden Sie die gleichen Rotator Parameter wie in Schritt 1.2 beschrieben.
- Nach 2 h der Probe drehen, führen eine dreifache Wasch durch Magnetisieren getrennte Kügelchen aus der Lösung, Entfernung flüssiger Phase mit Mikropipette und Resuspendieren Perlen in 500 ul Tris-HCl-Puffer. Die erhaltenen Perlen sind jetzt auf die Exosomen gebunden und sind bereit für das elektrische Feld Release und Messung.
2. Elektrisches Feld induzierte Freigegeben einnd Messung exosomalen Inhalt
- Erste Beschichtung von Elektroden mit GADPH Primer
- Anwenden eines Kunststoff auch eine Elektrodenanordnung, um eine Kreuzkontamination der einzelnen Elektroden zu verhindern. Für dieses Experiment wurde mit einem 16-Sensor-Elektrodenanordnung mit jeder Einheit Elektrode in der Anordnung, die aus einem Arbeits-, Gegen- und Bezugselektrode des nackten Gold.
- Bereiten Sie eine Mischung aus Lager 100 nM DNA-Sonde, 0,3 M KCl und 10 mM Pyrrol durch Pipettieren Lager Reagenzien in ein Rohr mit hochreinem destilliertem Wasser. Gründlich mischen durch Vortexen.
HINWEIS: Für diese Studie entspricht die DNA-Sonde so ausgewählt, daß der GAPDH Referenzgens, die bekanntlich innerhalb von Exosomen vorliegen. Die Sondensequenz verwendet wird: 5'-Biotin-AGGTCCACCACTGACACGTTG-3 '. Verwenden Sie diese Mischung auf alle Elektroden. - Pipette 60 ul der Monomer-DNA Sondenmischung auf der Oberfläche jeder Goldelektrode. Untersuchen der Elektroden zu gewährleisten, dass eine ausreichende Abdeckung der Arbeits, counter und Referenzelektroden durch die flüssige Mischung.
- Electropolymerize Monomer-Sondenmischung, um eine leitende Polymerschicht auf der Elektrodenoberfläche durch Anlegen einer zyklischen Rechteck (CSW) elektrische Feldprofil an die Elektrodenoberfläche zu schaffen. Dieses elektrische Feld besteht in der Anwendung 350 mV für 9 sec und sofort die Umstellung auf 950 mV für 1 s. Wenden Sie dieses zyklischen Rechteckprofil an die Elektrode 10 Zyklen, für insgesamt 100 sec der angelegten elektrischen Feldes.
- Spülen Sensoroberfläche 3 mal mit destilliertem Wasser und mit Stickstoffgas trocken um Flüssigkeit von der Oberfläche der Elektrode zu entfernen. Sicherzustellen, daß die Flüssigkeit ordnungsgemäß von der Elektrode entfernt.
- Exosom Güter Entladen
- Last 5 ul 1 uM eines Detektors Sonde in 495 & mgr; l der Kügelchen Exosom Gemisches und mit einer Pipette mischen.
HINWEIS: Der Fühler ist ein DNA-Primer-Sequenz an eine Fluorescein-Molekül am 3'-Ende konjugiert. Der Detektor probe Sequenz für diese Untersuchung verwendet wird, entspricht dem GAPDH mRNA in Exosomen gefunden. Die Sequenz des Fluorescein konjugierten Detektorsonde: 5'-GCAGTGGGGACACGGAAGGCC-Fluorescein-3 '. - Pipette 60 ul der Sonde und sicken Exosom Mischung auf eine Goldelektrode mit einer Magnetanordnung darunter. Diese Magnetanordnung besteht aus sechzehn 2,54 mm Durchmesser Neodymmagneten ausgerichtet, um den Arbeitselektroden des Sensors entsprechen. 2A veranschaulicht die Anordnung der Magnete und sicken exosome Lösung.
- Sobald Probe auf der Elektrodenoberfläche geladen wird, gelten 20 Zyklen des CSW elektrisches Feld mit 9 sec bei -300 mV und 1 s bei +200 mV (200 s insgesamt). Die exosomalen Ladung, die freigesetzt wird, wird an die Primer auf der Oberfläche der Elektrode zu hybridisieren. Wenn Oberflächenmarker der Exosom sind Gegenstand der Untersuchung, überspringen Sie diesen Teil des Experiments. 2B veranschaulicht diesen Prozess.
- Wash-Off die ungebundenen Analyten auf der Elektrodenoberfläche durch dreifache Spülung der Elektrodenoberfläche mit destilliertem Wasser. Trocknen Sie die Elektrode mit Stickstoffgas.
- Last 5 ul 1 uM eines Detektors Sonde in 495 & mgr; l der Kügelchen Exosom Gemisches und mit einer Pipette mischen.
- Reporter Antikörper und Readout
- Man gibt 60 & mgr; l von 150 Einheiten / ml Anti-Fluorescein-Antikörper, konjugiert an Meerrettichperoxidase (HRP in 1: 1000 Verdünnung) in Casein / PBS verdünnt.
- Verwenden eines ein elektrisches Feld angetrieben Konjugation an Komplex Anti-Fluorescein-HRP zu der Sonde Sandwich. Anwenden -200 mV für 1 Sekunde und +500 mV für 1 Sekunde für 5 Zyklen auf die Elektrodenoberfläche. 2A zeigt die Fänger- und Detektorsonde-Komplexe sowohl für ein Protein und Nukleinsäure-System.
- Dreibettzimmer Waschsensoroberfläche mit destilliertem Wasser und mit Stickstoffgas trocken.
- Nach dem Abwaschen von ungebundenem überschüssigem anti-Fluorescein-Antikörper, mit 60 & mgr; l 3,3 ', 5,5'-Tetramethylbenzidin (TMB) -Substrat. Laden Sie dieses Substrat auf jede Sensorfläche mit einer Mehrkanalpipette.
- Führen amperometrischen Auslesen des Stroms durch Messen des Elektrodenstroms bei -200 mV für 60 sec unter Verwendung einer elektro Potentiostat fähig gleichzeitige Messung von 16 Kanälen. 2C ist ein Beispiel der Stromverlauf während des Auslesens.
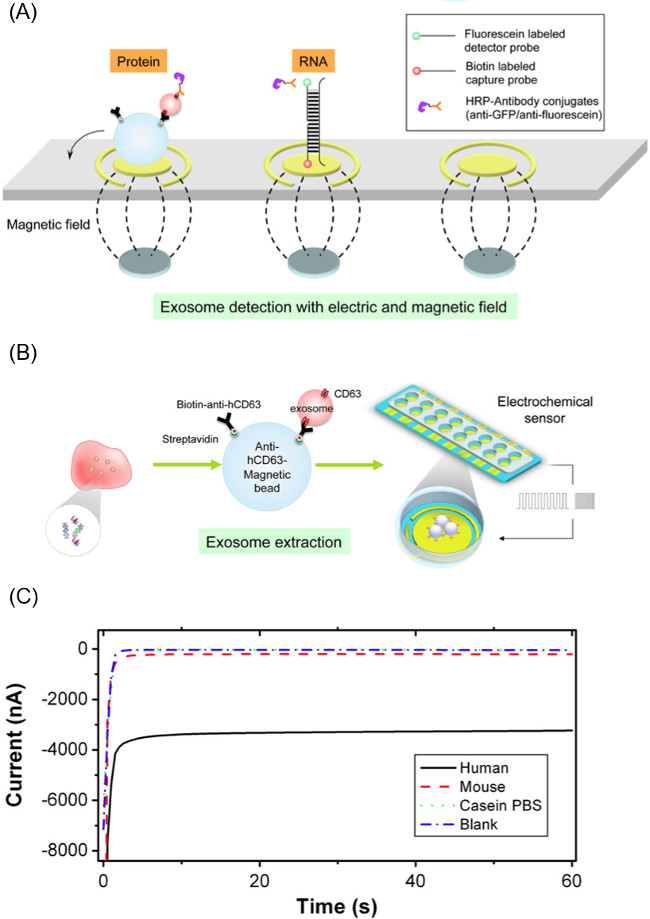
Abbildung 2. Komponenten Efirm Methode. (A) Verfahren zur Extraktion von Exosomen aus Biofluid mit anti-human CD63 beschichteten magnetischen Mikropartikeln und dann Entladen Exosom Fracht mit zyklischen Rechtecksignale an das Partikel-Exosom angewendet. (B) Schema der Elektrode Biosensor zum Nachweis von RNA / DNA / Protein-Targets von der frei Exosom verwendet. (C) repräsentatives Beispiel für amperometrische Auslesen aus dem Efirm Methodik, wo größere Stromstärke entspricht to höherer Gehalte eines Biomoleküls. Diese Zahl ist von Wei et al. 14 Klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Ergebnisse
Validierung Exosom Erfassung von Perlen Mit TEM
Isolierung der Exosomen aus Speichel unter Verwendung von Anti-Mensch-CD63 magnetischen Kügelchen wurde folgender Extraktionsvorschrift unter Verwendung der Transmissionselektronenmikroskopie (TEM) bestätigt. TEM zeigt magnetische Beads mit 70-100 nm Granulat unmittelbar benachbarten (siehe 3A und 3B), was mit der bekannten Profil der Exosomen. Keine 70-100 nm Granulat wurde für die magnetis...
Diskussion
Wie die Ergebnisse zeigen, sind anti-human CD63-beschichteten magnetischen Nanopartikel in der Lage, spezifisch einzufangen kleine Partikel, die eine Größe im Bereich von 70 bis 100 nm haben. Diese erfassten Teilchens entspricht der zuvor beobachteten Profil Exosomen. Weiterhin ist die Verwendung von Niederspannungs CSW nach der Abscheidung der Teilchen dargestellt, um sie von der Perlenoberfläche zu entfernen und verursachen DNA Abbauprofile ähnlich zu derjenigen eines herkömmlichen Lysepuffer basiertes Verfahren ...
Offenlegungen
David Wong ist Mitbegründer RNAmeTRIX Inc., ein Molekulardiagnostik-Unternehmen. PeriRx LLC lizenziert geistigen Eigenschaften, die zur molekularen Diagnostik von RNAmeTRIX. David Wong ist Berater PeriRx.
Danksagungen
Diese Arbeit wurde von der National Center for Research Resources und der National Center for Advancing Translational Sciences, National Institutes of Health, durch Zuschuss UL1TR000124 (FW) unterstützt; die Felix & Mildred Yip Stiftungsprofessur und die Barnes Familie Fonds (zu DTWW), dem National Institute of Dental und kraniofaziale Forschung der National Institutes of Health unter Preis Anzahl T90DE022734 (MT). Der Inhalt ist allein in der Verantwortung der Autoren und nicht unbedingt die offizielle Meinung der National Institutes of Health.
Materialien
| Name | Company | Catalog Number | Comments |
| Helios 16-Channel Reader System with Chip Interface | Genefluidics, USA | RS-1000-16 | |
| 16x Sensor Chip, Bare Gold, pack of 5 chips | Genefluidics, USA | SC1000-16X-B | |
| Biotinylated anti-human CD63 Antibody | Ancell, USA | 215-030 | |
| Dynabeads MyOne Streptavidin T1 | Invitrogen, USA | 65601 | |
| Neodynium Magnetics (1/10" dia. x 1/32" thick) | K&J Magnetics, USA | DH101 | |
| Ultrapure Distilled Water | Life Technologies, USA | 10977-023 | |
| Mettler Toldeo 3 M KCl Solution | Fisher Scientific, USA | 1911512 | |
| Pyrrole | Sigma-Aldrich, USA | W338605-100g | |
| Anti-Fluorescein-POD, Fab fragments | Roche, Germany | 11426346910 | |
| 3,3′,5,5′-tetramethylbenzidine substrate (TMB/H2O2, low activity) | Neogen, Usa | 330175 | |
| Phosphate Buffered Saline Solution | Life Technologies, USA | 10010023 | |
| Casein/PBS | Fisher Scientific, USA | 37532 |
Referenzen
- Rabinowits, G., Gerçel-Taylor, C., Day, J. M., Taylor, D. D., Kloecker, G. H. Exosomal MicroRNA: A Diagnostic Marker for Lung Cancer. Clinical Lung Cancer. 10 (1), 42-46 (2009).
- Thakur, B. K., et al. Double-stranded DNA in exosomes: a novel biomarker in cancer detection. Cell Research. 24 (6), 766-769 (2014).
- Lau, C. S., Wong, D. T. W. Breast Cancer Exosome-like Microvesicles and Salivary Gland Cells Interplay Alters Salivary Gland Cell-Derived Exosome-like Microvesicles In. Vitro. PLoS ONE. 7 (3), e33037 (2012).
- Bala, S., et al. Circulating microRNAs in exosomes indicate hepatocyte injury and inflammation in alcoholic, drug-induced, and inflammatory liver diseases. Hepatolog. 56 (5), 1946-1957 (2012).
- Dear, J. W., Street, J. M., Bailey, M. A. Urinary exosomes: A reservoir for biomarker discovery and potential mediators of intrarenal signalling. Proteomics. 13 (10-11), 1572-1580 (2013).
- Lässer, C., et al. Human saliva, plasma and breast milk exosomes contain RNA: uptake by macrophages. Journal of Translational Medicine. 9 (1), 9 (2011).
- Palanisamy, V., et al. Nanostructural and Transcriptomic Analyses of Human Saliva Derived Exosomes. PLoS ONE. 5 (1), e8577 (2010).
- Camussi, G., et al. Exosomes/microvesicles as a mechanism of cell-to-cell communication. Kidney International. 78 (9), 838-848 (2010).
- Lau, C., et al. Role of pancreatic cancer-derived exosomes in salivary biomarker development. The Journal of Biological Chemistry. 288 (37), 26888-26897 (2013).
- Théry, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and Characterization of Exosomes from Cell Culture Supernatants and Biological Fluids. Current Protocols in Cell Biology. 3 (22), (2001).
- Park, N. J., Li, Y., Yu, T., Brinkman, B. M. N., Wong, D. T. Characterization of RNA in Saliva. Clinical Chemistry. 52 (6), 988-994 (2006).
- Wei, F., et al. Bio/Abiotic Interface Constructed from Nanoscale DNA Dendrimer and Conducting Polymer for Ultrasensitive Biomolecular Diagnosis. Small. 5 (15), 1784-1790 (2009).
- Wei, F., et al. Electrochemical Sensor for Multiplex Biomarkers Detection. Clinical Cancer Research. 15 (13), 4446-4452 (2009).
- Wei, F., Yang, J., Wong, D. T. W. Detection of exosomal biomarker by electric field-induced release and measurement (EFIRM). Biosensors and Bioelectronics. 44, 115-121 (2013).
Erratum
Formal Correction: Errata: Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM)
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM). The disclosures were updated.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten