Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Planar Gradient Diffusionssystem, um Chemotaxis in einem 3D-Kollagen-Matrix Untersuchen
In diesem Artikel
Zusammenfassung
Cell migration is an important part of human development and life. In order to understand the mechanisms that can alter cell migration, we present a planar gradient diffusion system to investigate chemotaxis in a 3D collagen matrix, which allows one to overcome modern diffusion chamber limitations of existing assays.
Zusammenfassung
The importance of cell migration can be seen through the development of human life. When cells migrate, they generate forces and transfer these forces to their surrounding area, leading to cell movement and migration. In order to understand the mechanisms that can alter and/or affect cell migration, one can study these forces. In theory, understanding the fundamental mechanisms and forces underlying cell migration holds the promise of effective approaches for treating diseases and promoting cellular transplantation. Unfortunately, modern chemotaxis chambers that have been developed are usually restricted to two dimensions (2D) and have complex diffusion gradients that make the experiment difficult to interpret. To this end, we have developed, and describe in this paper, a direct-viewing chamber for chemotaxis studies, which allows one to overcome modern chemotaxis chamber obstacles able to measure cell forces and specific concentration within the chamber in a 3D environment to study cell 3D migration. More compelling, this approach allows one to successfully model diffusion through 3D collagen matrices and calculate the coefficient of diffusion of a chemoattractant through multiple different concentrations of collagen, while keeping the system simple and user friendly for traction force microscopy (TFM) and digital volume correlation (DVC) analysis.
Einleitung
Die bevorzugte Bewegung von Zellen in Richtung eines Konzentrationsgefälles Chemotaxis bekannt, spielt eine wichtige Rolle bei pathologischen und physiologischen Prozessen im Körper. Solche Beispiele sind Haut- und Schleimhaut Wundheilung 1, 2 Morphogenese, Entzündungen 3, und das Tumorwachstum 4,5. Es wurde auch gezeigt, daß Krebszellen durch beide individuelle und kollektive Zellmigrationsstrategien 6 migrieren. Darüber hinaus können Diffusionsinstabilität Mechanismen die Trennung von einzelnen oder gruppierten Zellen aus einer Tumor Körper / Objekt zu induzieren und dann kann auf eine Quelle von Nährstoffen einzuwandern und so dringen größere Gebiete und Gewebe 7.
Ferner hat es sich gezeigt, dass die vielfältigen Migrationsmechanismen aktiv in 2D und 3D werden, wegen der unterschiedlichen Rollen von Adhäsionsmolekülen 8. Daher ist ein Umzug in physiologisch relevanten in-vitro-Tests an Zellbeweglichkeit in einem measureabl untersuchene und einfache Weise von Bedeutung für das Verständnis Zellbewegung Phänomene 9. Leider ist die Schwierigkeit bei der Analyse von Zellmigration, eine umfassende quantifizierbare Chemotaxis-Assay erfordert in der Regel eine lange mühsame Methode, auf der Messung der unparteiische Zellbeweglichkeit und Transportphänomene Modelle gegründet.
Vergangenen experimentellen Ansätzen, um die Zellchemotaxis zu untersuchen sind der Boyden-Kammer 10 und der unter Agarose-Assay 11. Doch innerhalb dieser frühen Tests, die Zellmigration Experimente nicht die Bewegung in Bezug auf die Zeit zu überwachen. Weitere, besonders wichtig, die Konzentrationsgradienten für die Experimente verwendet wurden, nicht gut definiert oder vollständig verstanden, während nur die Erhaltung der Signalisierung für nicht mehr als ein paar Stunden. Weiterhin frühen Chemokammer Versuche beschränkt Zellmigration auf zwei Dimensionen und nicht erlauben, die Kinetik der Migration 12 überwachen. Mit Blick auf die Boyden-Kammer, ein Endpunkt-Assaywürde nicht zulassen, dass der Forscher, um die Migration visuell zu beobachten und nicht direkt zu unterscheiden Chemotaxis (Bewegungsrichtung) von Chemokinese (Zufallsbewegung). Darüber hinaus mehrere Variablen-Unterschiede in der Porengröße und Dicke der Membranen verursachten die Kammer sehr schwierig, einfach zu reproduzieren und verdeckte die Wander Reaktion von Zellen auf Chemokine 13,14.
Mit dem neuen Verständnis der Mikrofluidik, haben neue Kammern und Mikrogeräten als Instrument untersucht worden, um Zellbewegung unter Zwischenströmungsbedingungen zu untersuchen oder Chemotaxis 15,16. Unter diesen neuen Geräten wurden neue Zell Metriken eingeführt und untersucht, wie die Wirkung von Scherspannung auf eine Zelle 17,18. Leider vergangenen und aktuellen mikrofluidischen Chemotaxiskammern begrenzt Untersuchungen von Zellmigration, um 2D-Substrate eine wichtige Rückschlag seit vielen biologischen Prozessen wie Tumorzellinvasion und Metastasierung, und imImmunzellmigration, beinhalten 3D-Migration.
Direkte Beobachtung Kammern, wo-a chemoattractant Lösung in Kontakt mit einer 3D-Gel enthaltenden Zellen wurden ebenfalls berichtet 19,20. Diese Kammern haben zwei Kammern, von denen eine Lockstoff und eine mit Zellen, die nebeneinander horizontal 21 oder als konzentrische Ringe 22 verbunden sind. Diese Systeme werden in die richtige Richtung, aber nicht über ein Chemotaxis-System zu halten für eine ausgedehnte Zeitperiode.
Darüber hinaus haben Forscher auch durch Kollagenmembranen in Dialysezellen sowie die Diffusion von Tracermolekülen durch Kollagenproben gegen hydrostatischen Druck unterworfen 23-25 geprüft Leitfähigkeit. Einige Diffusionsexperimente in Kollagengelen beruhen auf physikalischen und chemischen Modifikationen des Gels mit Magnetfeldern und chemischer Einbau 26. Eine beliebte Methode zur Modellierung von Diffusionsvermögen in collaGenous Gewebe beruht auf der Fluoreszenzabbildung der kontinuierlichen Punkt Photobleichen. Dieses Verfahren wurde Anisotropie der Diffusionskoeffizienten von Makromolekülen in orientierten Kollagengewebe offenbart. Dennoch hat Photobleichens im Gelenkknorpel benutzt und nicht-Kollagen-Matrices. Zwar ähnlich, müssen die erforderlichen Modellversuche durch speziell Verständnis der Diffusionskoeffizient von Kollagengelen erfolgen. Noch wichtiger ist, die Systeme nicht ein Verfahren zur Messung Zellkrafterzeugung nutzen.
Leider sind die meisten Systeme scheinen auf ein oder zwei Schlüsselelemente für ein ideales System fehlen: das ermöglicht der Zelle Tracking, einem Diffusionsgefälle Verständnis mit einem chemotaktischen Faktor durch die Matrix, einem relativ einfachen Aufbau mit einer Leichtigkeit der Reproduzierbarkeit, der Minimierung von Zell-Zell-Wechselwirkungen, sowie die Fähigkeit, Dimensionseinheiten zur Quantifizierung (dh, die Geschwindigkeit, Kraft, spezifische Konzentrations) zu messen. Moghe et al. 27 ein System vorgeschlagen, das weitgehend die Anforderungen in der Zellen wurden zunächst im ganzen Gel dispergiert anstatt auf der Filteroberfläche konzentriert erfüllt, ist aber schwierig zu Kräften, die die Zelle erzeugt messen.
Zu diesem Zweck stellen wir eine planare Gradienten-Diffusionssystem zur Chemotaxis in einem 3D-Kollagen-Matrix, die eine moderne Diffusionskammer Beschränkungen vorhandener Assays, die auf Zeitraffermikroskopie beruht, in Verbindung mit Bildanalysetechniken zu Zelle messen zu überwinden ermöglicht untersuchen Kräfte in einer 3D-Umgebung. Dieses Protokoll sieht eine einfache, aber innovative Art der Erstellung einer einfachen 3D-Diffusionskammer, die verwendet werden können, um 3D-Chemotaxis in verschiedenen Zellen zu untersuchen.
Protokoll
1. 3D-Formenbau und Parts
- Schimmel
- Vor der Arbeit, eine Silikonelastomer-Kit, ein Live Cell Imaging Kammer, eine 22 mm Deckglas und einer bearbeiteten Aluminium-Metall-Würfel mit den Abmessungen 10,07 mm x 3,95 mm x 5,99 mm. Bereiten Sie das Live Cell Imaging Kammer zur Formung, indem das Deckglas in der unteren Halterung und Montage der Rest der Kammer, wie den Angaben des Herstellers.
- Als nächstes wird mit einer Pinzette, legen Sie die bearbeiteten Aluminium-Metall-Würfel in der Mitte des Live Cell Imaging Kammergehäuse und auf der Oberseite des Deckglases und dann beiseite stellen.
- Mischungssiliconelastomerlösungen gemäß dem Protokoll des Herstellers zu 5 ml Elastomer bilden.
- Mit einem Einweg-Labor Spatel, gießen Siliconelastomer-Lösung in Live Cell Imaging Kammer-Setup und stellen Sie sicher nicht platziert bearbeitetem Aluminium Metallwürfel bewegen. Stellen Sie die Anlage auf dem Labortisch, an einem sicheren Ort O / N für die Heilung.
- Am nächsten Morgen, dekonstruierendas Live Cell Imaging Kammer, wie vom Hersteller empfohlen wird, und ziehen Sie Form mit einer Pinzette. Mit einer Pinzette vorsichtig heraus das bearbeitete Aluminium-Metall-Würfel von Schimmel. Gehen Sie auf die Wanne und spülen Sie die Form mit VE-Wasser. Legen Sie die Form auf einem Papiertuch, um zu trocknen.
- Nach dem Trocknen mit einem Hobby-Messer, schneiden Schlitze durch die Form, Abstand 2,34 mm von jedem Längsende, in der Silikonform. Achten Sie darauf, die Form bleibt in einem sicheren und trockenen Ort, bis Sie bereit sind, um das System für ein Experiment zu konstruieren sind.
- Hydrophilen und hydrophoben Deckgläser Vorbereitung
- Damit der Kollagenmatrix zu einer Oberfläche haften, zu schaffen hydrophilen Deck:
- Unter Verwendung einer Einwegpipette, abmessen 150 ul 3-Aminopropyltrimethoxysilan und gießen Lösung in ein 50 ml Röhrchen. 30 ml 100% Ethanol auf die 50-ml-Röhrchen mit einem zweiten Einwegpipette und den Deckel schließen. Vortex die Lösung für 2 min, wodurch eine vollständige Durchmischung. Punsere Lösung in einer Glaspetrischale und beiseite stellen.
- Gießen Sie 15 ml 100% Ethanol in eine zweite Petrischale und beiseite stellen (stellen Sie sicher, um Gerichte zu markieren).
- Mit Hilfe einer neuen Einweg-Pipetten, abmessen 30 ml VE-Wasser und gießen Lösung in einen 50-ml-Tube. Als nächstes wird mit einer neuen Einweg-Pipetten Maßnahme aus 1.875 ul Glutaraldehyd und gießen Lösung in den gleichen 50-ml-Tube und den Deckel schließen. Vortex die Lösung für 2 min, wodurch eine vollständige Durchmischung. Die Mischung in Drittpetrischale, und beiseite stellen.
- Mit einer Pinzette, nehmen Sie eine 22 mm runden Deckglas und spülen Sie beide Seiten mit 100% Ethanol-Gemisch mit einer Einwegpipette.
- Platzieren gespült Deckglas in 3-aminopropyl-trimethoxysilan mit 30 ml 100% ige Ethanollösung Schüssel und zu ermöglichen, in Lösung 5 min stehen lassen.
- Mit einer Pinzette herausnehmen Deckglas und spülen wieder mit 100% Ethanol-Lösung mit Einwegpipette.
- Drop gespült Deckglas in 1875ul Glutaraldehyd und 30 ml entionisiertem Wasser-Gemisch und beiseite für 30 min eingestellt.
- Nach 30 Minuten, entfernen Sie Deckglas mit einer Pinzette und spülen mit VE-Wasser und legen Sie auf das trockene Gewebe O / N bei RT trocknen.
- Wiederholen Sie die Deckeintauchen und Bewegen für so viele Deckgläser als mit den gleichen erzeugten Lösungen benötigt.
- Machen hydrophoben Deckgläser durch das gegebene Protokoll.
- Gib 500 ul Tridecafluor-1,1,2,2-tetrahydrooctyl wurden 100 ul Essigsäure und 19,4 ml Hexan in einem 50 ml-Röhrchen und Vortex für 2 min. Gießen Lösung in Glaspetrischale und beiseite stellen.
- Mit sauberen Pinzette, nehmen Sie ein Deckglas-and-Drop in die vorbereitete gemischte Lösung für 2 Minuten. Nach 2 Minuten vergangen sind, nehmen Sie Deckglas mit einer Pinzette und spülen mit VE-Wasser unter Verwendung einer Einwegpipette und Ort Deckglas auf das trockene Gewebe O / N bei RT trocknen. Shop Deckglas in Kunststoff-Petrischalen bis zur Verwendung.
- Wiederholen Sie die Deck dipping und Bewegen für so viele Deckgläser als mit den gleichen erzeugten Lösungen benötigt.
- Damit der Kollagenmatrix zu einer Oberfläche haften, zu schaffen hydrophilen Deck:
2. Mold Assembly
- Vor der Verwendung von Formen, mit einem Einweg-Pipetten spülen Sie die Form mit 90% Ethylalkohol in einem Behälter für Chemieabfälle. Als nächstes legen Sie die Form in einer Petrischale mit entionisiertem Wasser gefüllt und lassen Sie O / N sitzen.
- Nehmen Sie Form aus der Lösung mit einer Pinzette und auf einem Handtuch an der Luft trocknen, bevor Sie den Formaufbau.
- Während die Form Lufttrocknung, schneiden Sie die quadratische hydrophile Deckgläser in zwei Rechtecke etwas größer als 3,95 mm x 5,99 mm mit einem hochpräzisen Diamantritzwerkzeug. Schieben Sie die Schnitt Rechteck Deckglas in die in die Silikonform geschnitten Slots.
- Drehen Sie die Form auf den Kopf und gelten Vakuumfett auf dem Boden der Silikonform mit einer Einwegpipette. Als nächstes drehen Sie die Form wieder rechten Seite nach oben und drücken Sie den Boden der Form auf die kreisförmige hydrophile GlasabdeckungenLippe, um eine Dichtung zu schaffen.
- Mit Einweghandschuhe, nehmen Sie den zusammengesetzten Form und in einen Einweg-Petrischale und dann in eine Bio-Haube. Schalten Sie UV Bio-Haube-Lampe für 1 Stunde, um die Form zu sterilisieren, bevor Experimente auftritt.
3. Kollagen Mixture und 3D-Matrix
- Vor der Herstellung der Kollagenmischung, Färbung und Vorbereitung der Zellen zum Abbilden gemß Standardprotokollen 28.
- Verwendung von Standard-Labortechniken und Protokolle 29,30, in einem bio-Haube, mischen Zellmedien und chemische Lockstoffe in einer 15 ml Tube, um die gewünschte Konzentration chemoattractant. Pipette 5 ml spezifischer Zellmedien chemoattractant Konzentration Lösung in einen 15-ml-Röhrchen und bewegen Sie die Lösung in ein beheiztes Wasserbad.
- Mit einer Pipette extrahieren 5 ml nur Zellmedium in einen zweiten 15-ml-Tube und in ein erhitztes Wasserbad zum Aufheizen der Lösung für die Mikroskopie Experimente.
- In einem bio-Haube, Pipette 30 ul 10x Phosphatpuffered Lösung (PBS) in Mikrozentrifugenröhrchen. In 6 ul 1 N Natriumhydroxid (NaOH) in derselben Mikrozentrifugenröhrchen und Vortex für 15 Sekunden.
- Zu erhalten Kollagen I Rattenschwanz-Lösung aus dem Kühlschrank und zu bewegen, um die bio-Haube mit Standard-Labortechniken und Protokolle 29. Stellen Sie sicher, das Kollagen noch kalt ist. Pipettieren 168,3 ul Kollagen in derselben Mikrozentrifugenröhrchen.
- Als Nächstes fügen Sie 18 ul der gelb-grün fluoreszierende Carboxylat-modifizierte Mikrosphären mit einer Pipette auf die gleiche Mikrozentrifugenröhrchen. Wirbel das Mikrozentrifugenröhrchen mit allen Lösungen für 30 sec.
- In 77,7 ul der gewünschten Zellmedien-Zellgemisch (ca. 2 x 10 6 Zellen / ml-Konzentration) in Mikrozentrifugenröhrchen und mischen mit Pipettenspitze 20 Sek. Wie nötig, ändern Sie die Matrixdichte, indem Sie die benutzerdefinierte Collagen Mischung für den Zusatz von fluoreszierenden Kügelchen und die Lebensfähigkeit der Zellen Tabelle (Tabelle 1) angepasst.
- Pipette 300 & #181; l von Kollagen-Zellen-Mischung in der Mitte gut (gut # 2) des hergestellten Formanordnung. Legen Sie die Form auf einem Einweg-Petrischale und verschieben Sie sie in ein Standard-Inkubator für 20 min bei 37 ° C mit 5% CO 2.
- Entfernen System von Inkubator und legen wieder in bio-Haube. Mit einer Pinzette, entfernen Sie die beiden hydrophoben Deckgläser durch Klemmen auf der Oberseite jedes Deckglas und Ziehen nach oben und weg von der Form.
- Bewegen des gesamten Systems auf die gewünschte Mikroskopie und Bild sicherzustellen Kollagen Kokillenseiten noch direkt auf eine planare Diffusionsgradienten ermöglichen.
- Mit dem System immer noch unter dem Mikroskop, mit einem 100 & mgr; Einkanalpipette, fügen Sie 100 ul der Zellmedien in gut # 1. Dann wurde unter Verwendung eines 100 & mgr; Einkanalpipette fügen 100 ul Zellmedium mit den gewünschten chemoattractant Konzentration in gut # 3.
- Unmittelbar nach der Zugabe von Lösungen in beiden Brunnen, beginnen Bildgebung.
4. Imaging und DiffusionModellieren
- Wenn man die Diffusionskoeffizienten für die spezifische Kollagendichte, die spezifische Konzentration berechnen finden will, folgen Sie der unten Protokoll:
- Folgen Sie der oben angegebenen Protokoll mit dem Titel "Collagen Mixture und 3D-Matrix", anstatt Erzeugung Zellmedium mit den gewünschten chemoattractant Konzentration, machen 5 uM Rhodamin in einer 15-ml-Röhrchen mit Standard-Berechnungen und Protokolle 30.
- Bild der 3D-Kollagen-Matrices mit einem konfokalen System auf einem inversen Mikroskop unter Verwendung eines Argon-Laser (488 nm), um die Fluoreszenz zu erfassen montiert. Bild alle 2 oder 3 sec für bis zu 7 Std.
- Für Diffusion Modeling: Mit Hilfe der unten mathematisches Modell, einen Rechensoftwarecode für die Verarbeitung und Analyse mit diesen allgemeinen Schritten.
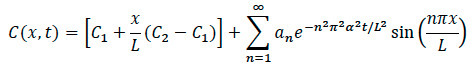
- Import von Bildern in Computersoftware für Skalierung und Normalisierung durch die maximale intensity. Konvertieren von Bildern in einer grauen Farbe-Karte des Bildes, um die Kante zu wählen. Pick eine Achse entlang der Mitte des Bildes, auf der rechten Seite der Mitte und links von der Mitte.
- Verwendung der Rechensoftware Code plotten die normalisierte Intensität zusammen mit der wahre Intensität und der Unterschied zwischen den beiden.
- Danach geben die Parameter für die Montage (Konzentration, Zeit und Länge) notwendig.
- Plotten der normierten Konzentration gegen die Zeit und das Konzentrationsprofil in der x-Achse mit einem Polynom-Anpassung an die Daten.
- Berechnen der Diffusionskoeffizient mit Hilfe der Rechensoftwarecode.
5. Experimentelle Messungen
- Rückbezug auf die Diffusionsgleichung, schreiben die Gleichung, um die spezifische Konzentration an jedem Ort innerhalb des Systems zu finden, wie in unten gezeigt.
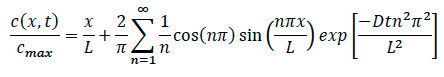
Wo:
L = Länge der the Kollagenmatrix
x = Lage in Untersuchung
D = Diffusionskoeffizient
t = verstrichene Zeit, - Während Chemo Experimenten sicherzustellen, um die spezifische Zeit, die das Moment Chemokin wurde System hinzugefügt aufzuzeichnen. Sobald die Zellmigration Bewegung gefunden wird, um sicherzustellen, konfokale Zeitrafferaufnahme aufzeichnen, und beachten Sie den genauen Zeitpunkt und Ort vom Rand, wo Bildgebung aufgetreten.
- Bei der Datenanalyse, schließen die erwähnte abgelaufene Zeit, Standort und der Diffusionskoeffizient für die spezifische Zelle, in der Gleichung auf die normierte spezifische Konzentration an jedem Punkt innerhalb der Kollagen sowohl Diffusion und Zellexperimenten gefunden.
6. Tracking-Zellmigration Verwendung TFM
- Zu verfolgen, die Zellmigration mit TFM / DVC-Techniken folgen dem Protokoll im Handumdrehen:
- Nach Zugabe der gewünschten Konzentration chemoattractant in Vertiefung 3, Start Abbilden der 3D-Kollagen-Matrices mit einem konfokalen System auf einem invertierten MICR montiertenOscope zu finden, um eine Zelle zu untersuchen.
- Sobald eine Bewegung Zelle gefunden wird, setzen Sie die Zelle, in der Mitte des Sichtfeld und nutzen eine piezoelektrische Motor (für Bild in der z-Achse), Bild alle 1 oder 2 min.
- Für die Berechnung Zellkrafterzeugung und Verformung, erzeugen eine Rechencode, um die Verschiebungen der Fluoreszenz Perlen nach den TFM / DVC-Protokolle 31 zu finden.
Ergebnisse
Die Fähigkeit des Assays, genau zu beurteilen die Migration der Zellen beruht auf einem guten Aufbau des Systems. Daher ist es wichtig für Sie sicher, dass die Diffusion System Form genau entwerfen und große Sorgfalt bei der Platzierung sowohl hydrophobe und hydrophile Deckgläsern, wie in Abbildung 1 veranschaulicht. Wenn das System ordnungsgemäß entworfen und während des Diffusionsmodellierungsstufe gewährleistet eine sehr finden gute lineare Startlinie, ist man in der Lage schön zu erreichen ...
Diskussion
Die wichtigsten Schritte für eine erfolgreiche Diffusionsexperimente mit oder ohne Zellen sind: richtig Einrichten der Formanordnung; Entwicklung der notwendigen Fingerfertigkeit, um bei der Extraktion von hydrophoben Deckschäden zu vermeiden; gewährleisten eine sehr gute lineare Startlinie, um den Diffusionskoeffizienten korrekt berechnen zu finden; korrigieren experimentelle Berechnungen von sowohl Kollagen und chemische Lockstoffe; von Live Cell Imaging System, um sicherzustellen, Matrix trocknet nicht aus richtig...
Offenlegungen
The authors declare that they have no competing financial interests.
Danksagungen
The authors would like to acknowledge Drs. Jonathan Reichner and Angle Byrd for cell experiment insight. The National Science Foundation Graduate Research Fellowship Program (GRFP) supported this work.
Materialien
| Name | Company | Catalog Number | Comments |
| Silicone elastomer kit | Dow Corning Corp | 182 SIL ELAST KIT .5KG | a two-part misture with a 10:1 mix |
| Live cell imaging chamber | Live Cell Instrument | CM-B18-1 | CMB for 18 mm round coverslips |
| 22 mm Glass Coverslip | Fisher Scientific | NC0180281 | Neuvitro Corp. cover slip 22 mm 1.5 |
| Machined aluminum metal cube | |||
| Hobby utility knife | X-Acto | X3201 | |
| 3-(aminopropyl) trimethoxysilane | Sigma-Aldrich | 281778-5ML | |
| Glutaraldehyde | Polysciences, Inc | 00216A-10 | Glutaraldehyde, EM Grade, 8% |
| 50 ml tube | Fisher Scientific | 14-432-22 | Standard floor model and tabletop centrifuges |
| Glass Petri dish | Fisher Scientific | 08-747A | Reusable Petri Dishes: Complete (60 x 15 mm) |
| Forceps | Fisher Scientific | 22-327-379 | Fine Point Forceps |
| Cover glasses | Fisher Scientific | 12-518-105A | Rectangle; 30 x 22 mm; Thickness No. 1 |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl | Gelest | SIT8174.0 | |
| Acetic acid | Sigma-Aldrich | 320099 | Acetic acid ACS reagent, ≥99.7% |
| Hexane | Sigma-Aldrich | 296090 | anhydrous, 95% |
| Ethyl alcohol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| High-precision diamond scribing tool | Lunzer | PV-081-3 | Straight extended tip scribe, .020" (.50 mm) diameter by .200" (5.0 mm) tip length |
| Vacuum grease | Dow Corning | 14-635-5C | High-Vacuum Grease |
| 15 ml tube | Fisher Scientific | 14-959-49D | 15 ml conical centrifuge tubes with hydrophobic, biologically inert surface |
| 10x phosphate buffered solution | Fisher Scientific | BP399-500 | 1.37 M Sodium Chloride, 0.027 M Potassium Chloride, and 0.119 M Phosphate Buffer |
| 1 N sodium hydroxide | Sigma-Aldrich | 38215 | Sodium hydroxide concentrate |
| Collagen I, rat tail | BD Biosciences | 354236 | Rat tail |
| Micro centrifuge tube | Fisher Scientific | 02-681-332 | Volume: 2.0 ml; O.D. x L: 13 x 40 mm; sterile; single-wrapped |
| [header] | |||
| Carboxylate-modified microspheres | Invitrogen | F-8813 | Carboxylate-modified microspheres, 0.5 µm, yellow-green fluorescent (505/515), 2% solids |
| Rhodamine | Sigma-Aldrich | 83689 | Rhodamine B for fluorescence |
Referenzen
- Adzick, N. S. The Molecular and Cellular Biology of Wound Repair. Ann Surg. 225 (2), 236 (1997).
- Reddi, A. H. Bone morphogenetic proteins: an unconventional approach to isolation of first mammalian morphogens. Cytokine Growth F. R. 8 (1), 11-20 (1997).
- Coussens, , Werb, Z, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Vital-Lopez, F. G., Armaou, A., Hutnik, M., Maranas, C. D. Modeling the effect of chemotaxis on glioblastoma tumor progression. AIChE J. 57 (3), 778-792 (2011).
- Hughes-Alford, S. K., Lauffenburger, D. A. Quantitative analysis of gradient sensing: towards building predictive models of chemotaxis in cancer. Curr. Opin. Cell Biol. 24 (2), 284-291 (2012).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol Cell Bio. 10 (7), 445-457 (2009).
- Cristini, V. Morphologic Instability and Cancer Invasion. Clin. Cancer Res. 11 (19), 6772-6779 (2005).
- Lammermann, T., et al. Rapid leukocyte migration by integrin-independent flowing and squeezing. Nature. 453 (7191), 51-55 (2008).
- Parent, C. A., Devreotes, P. N. A cell's sense of direction. Science. 284 (5415), 765-770 (1999).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. J. Exp. Med. 115, 453-466 (1962).
- Nelson, R. D., Quie, P. G., Simmons, R. L. Chemotaxis under agarose: a new and simple method for measuring chemotaxis and spontaneous migration of human polymorphonuclear leukocytes and monocytes. J. Immunol. 115 (6), 1650-1656 (1975).
- Rosoff, W. J., McAllister, R., Esrick, M. A., Goodhill, G. J., Urbach, J. S. Generating controlled molecular gradients in 3D gels. Biotechnol. Bioeng. 91 (6), 754-759 (2005).
- Wells, A., Kassis, J., Solava, J., Turner, T., Lauffenburger, D. A. Growth factor-induced cell motility in tumor invasion. Acta Oncol. 41 (2), 124-130 (2002).
- Wilkinson, P. C. Assays of leukocyte locomotion and chemotaxis. J. Immunol. Methods. 216 (1-2), 139-153 (1998).
- Bonvin, C., Overney, J., Shieh, A. C., Dixon, J. B., Swartz, M. A. A multichamber fluidic device for 3D cultures under interstitial flow with live imaging: development, characterization, and applications. Biotechnol. Bioeng. 105 (5), 982-991 (2010).
- Noo, L. J., et al. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device. Nat. Biotechnol. 20 (8), 826-830 (2002).
- Shao, J., et al. Integrated microfluidic chip for endothelial cells culture and analysis exposed to a pulsatile and oscillatory shear stress. Lab Chip. 9 (21), 3118-3125 (2009).
- Haessler, U., Kalinin, Y., Swartz, M. A., Wu, M. An agarose-based microfluidic platform with a gradient buffer for 3D chemotaxis studies. Biomed. Microdevices. 11 (4), 827-835 (2009).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat. Rev. Cancer. 3 (5), 362-374 (2003).
- Sixt, M., Lammermann, T. In vitro analysis of chemotactic leukocyte migration in 3D environments. Methods Mol. Biol. 769, 149-165 (2011).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. J. Cell Biol. 75 (2 Pt 1), 606-616 (1977).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. J. Cell. Sci. 99 (Pt 4), 769-775 (1991).
- Gilbert, D. L. Macromolecular diffusion through collagen membranes. Int. J. Pharm. 47 (1-3), 79-88 (1988).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys. J. 83 (3), 1650-1660 (2002).
- Weadock, K., Silver, F. H., Wolff, D. Diffusivity of 125I-calmodulin through collagen membranes: effect of source concentration and membrane swelling ratio. Biomaterials. 7 (4), 263-267 (1986).
- Erikson, A., Andersen, H. N., Naess, S. N., Sikorski, P., Davies, C. d. L. Physical and chemical modifications of collagen gels: impact on diffusion. Biopolymers. 89 (2), 135-143 (2008).
- Moghe, P. V., Nelson, R. D., Tranquillo, R. T. Cytokine-stimulated chemotaxis of human neutrophils in a 3-D conjoined fibrin gel assay. J. Immunol. Methods. 180 (2), 193-211 (1995).
- Sundd, P., et al. Live cell imaging of paxillin in rolling neutrophils by dual-color quantitative dynamic footprinting. Microcirculation. 18 (5), 361-372 (2011).
- Sanders, E. R. Aseptic Laboratory Techniques: Volume Transfers with Serological Pipettes and Micropipettors. J. Vis. Exp. (63), 2754 (2012).
- Stephenson, F. H. . Calculations for Molecular Biology and Biotechnology: A Guide to Mathematics in the Laboratory 2e. , (2010).
- Bar-Kochba, E., Toyjanova, J., Andrews, E., Kim, K., Franck, C. A Fast Iterative Digital Volume Correlation Algorithm for Large Deformations. Exp. Mech. In press, (2014).
- Shenoy, V., Rosenblatt, J. Diffusion of Macromolecules in Collagen and Hyaluronic Acid, Rigid-Rod-Flexible Polymer Composite Matrixes. Macromolecules. 28 (26), 8751-8758 (1995).
- Leddy, H., Guilak, F. Site-Specific Molecular Diffusion in Articular Cartilage Measured using Fluorescence Recovery after Photobleaching. Ann. Biomed. Eng. 31 (7), 753-760 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten