Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein ELISA-basierte Bindung und Wettbewerbsverfahren zu schnell zu bestimmen, Ligand-Rezeptor-Wechselwirkungen
In diesem Artikel
Zusammenfassung
The presented protocols describe two enzyme-linked immunosorbent assay (ELISA) based techniques for the rapid investigation of ligand-receptor interactions: The first assay allows the determination of dissociation constant between ligand and receptor. The second assay enables a rapid screening of blocking peptides for ligand-receptor interactions.
Zusammenfassung
A comprehensive understanding of signaling pathways requires detailed knowledge regarding ligand-receptor interaction. This article describes two fast and reliable point-by-point protocols of enzyme-linked immunosorbent assays (ELISAs) for the investigation of ligand-receptor interactions: the direct ligand-receptor interaction assay (LRA) and the competition LRA. As a case study, the ELISA based analysis of the interaction between different lambda interferons (IFNLs) and the alpha subunit of their receptor (IL28RA) is presented: the direct LRA is used for the determination of dissociation constants (KD values) between receptor and IFN ligands, and the competition LRA for the determination of the inhibitory capacity of an oligopeptide, which was designed to compete with the IFNLs at their receptor binding site. Analytical steps to estimate KD and half maximal inhibitory concentration (IC50) values are described. Finally, the discussion highlights advantages and disadvantages of the presented method and how the results enable a better molecular understanding of ligand-receptor interactions.
Einleitung
Ein umfassendes Verständnis der Signalwege erfordert detailliertes Wissen über die Ligand-Rezeptor-Wechselwirkung. Die meisten Methoden zur Beurteilung der Wechselwirkung eines bestimmten Liganden mit seinem spezifischen Rezeptor sind teuer, zeitaufwendig, arbeitsintensiv und erfordern eine spezielle Ausrüstung und Know - how 1.
Dieser Artikel beschreibt zwei schnelle und zuverlässige Punkt-für-Punkt-Protokolle den Liganden-Rezeptor-Interaktion an ein Enzym linked immunosorbent assay (ELISA) basieren, zu untersuchen: den direkten Liganden-Rezeptor-Wechselwirkung-Assay (LRA) und den Wettbewerb LRA. ELISA ist ein sehr empfindlich, spezifisch und leicht verfügbare Technik routinemäßig in fast jedem Labor eingesetzt. ELISA kann in verschiedener Weise durchgeführt und angepasst werden. Die vorgestellten Protokolle werden für die Untersuchung der Wechselwirkung zwischen verschiedenen lambda Interferone (INFLs) und deren Rezeptor optimiert.
Die direkte LRA ermöglicht eine quantificatiauf der Ligand-Rezeptor in Bezug auf Ligandenkonzentration Bindung und somit eine Bindungskurve ergibt. Verwendung eines geeigneten Modells für den Liganden-Rezeptor - Interaktion können die Daten analysiert werden , weiter die Dissoziationskonstante (K D) zu schätzen.
In dem vorgestellten Protokoll wird die allgemein verwendete Hill-Gleichung, die Ligand-Rezeptor-Bindung an Modell angewendet. Obwohl andere Verfahren wie beispielsweise die Oberflächenplasmonresonanz - Technologie 2,3 die Bestimmung der Bindungsaffinitäten zwischen zwei Proteinen zu ermöglichen, ist diese Technik oft arbeitsintensiv, teuer und erfordert eine spezielle Laborausrüstung.
Der Wettbewerb LRA ermöglicht das Screening von inhibitorischen Peptide: Die Ligand-Rezeptor-Bindung wird in Bezug auf Peptidkonzentration quantifiziert. Dies ergibt eine Dosis-Wirkungs-Kurve, die die hemmende Wirkung des Peptids beschreibt. Die Daten können weitere 50 die Hälfte der maximalen Hemmkonzentration (IC abzuschätzen analysiert werden ) des Blockierungspeptids.
Beide ELISA-Protokolle sind einfach zu bedienen und kann auf ein breites Spektrum an Forschungsfragen angepasst werden. Rekombinante Proteine jeglicher Art verwendet werden, um zuverlässig und schnell die Wechselwirkungsteile bestimmen. Darüber hinaus kann der Wettbewerb LRA verwendet werden, um kritische Interaktionsstellen von Liganden und Rezeptoren zu bestimmen durch Verwendung von blockierenden Peptiden, die ausgelegt sind, entweder den Liganden oder den Rezeptor zu imitieren. Wenn das Blockierungspeptid effiziente und spezifische Hemmung zeigt, nimmt das Peptid eine kritische Interaktionsstelle des Liganden (wenn das Peptid-Mimetika der Rezeptor) oder des Liganden (wenn das Peptid-Mimetika der Ligand).
Das erste Protokoll der K D -Wert Bestimmung verschiedener INFLs beschreibt und die alpha - Untereinheit von ihrer Rezeptor, dh der Interleukin-28 - Rezeptor (IL28RA) die direkte LRA verwenden. Als nächstes zeigt das zweite Protokoll, wie die Fähigkeit eines langen Peptid 20 Aminosäuren zu bestimmen,die INFL-IL28RA Wechselwirkungen hemmen. Das Peptid wurde entwickelt mit IFNLs an ihren Rezeptor-Bindungsstelle konkurrieren, und ermöglicht somit ein Molekular Verständnis der Wechselwirkung. Darüber hinaus kann dieses Peptid verwendet werden IL28RA in in vitro - Experimenten zu blockieren , die Auswirkungen auf nachgeschalteten Signaleffekte 4 zu bestimmen.
Protokoll
1. Vorbereitung der Reagenzien
- Zur Herstellung von Karbonat Beschichtungspuffer, lösen sich 0,36 g Na 2 CO 3 und 0,84 g NaHCO 3 in 100 ml destilliertem Wasser; Sterilfilter der Puffer durch ein Vakuum von 0,22 um Polyethersulfon (PES) Membranfilter und lagern bei RT bis zur Verwendung angetrieben werden.
- Bereiten Waschlösung durch Zugabe von 0,05% v / v Tween 20 in phosphatgepufferter Salzlösung (PBS).
- Bereiten Sie eine 5% Rinderserumalbumin (BSA) (Blocking-Lösung) in PBS-Lösung durch Auflösen von 5 g BSA (≥98%) in 100 ml PBS und lagern bei 4 ° C.
- Rekombinante Rezeptor, Liganden und Blocking Peptide
- Reconstitutethe rekombinanten humanen Interleukin-Rezeptor-alpha-Untereinheit (IL28RA) und rekombinante His-markierten Liganden des menschlichen IFN (IFNL1-3) gemäß den Anweisungen des Herstellers und bei -80 ° C. Blockierende Peptide synthetisieren und verwendet wie zuvor 4 beschrieben. Verwenden PBS auf verschiedene Konzentrationen von Ligand vorbereitens und Peptide zur Verwendung in den Assays.
- An den primären Antikörper herzustellen, verdünnte 6x His monoklonalen Maus-Antikörper in PBS mit 0,1% BSA auf 1: 1.000-Verdünnung. Meerrettich-Peroxidase (HRP) konjugiertes Ziege-anti-Maus-IgG (H + L) in PBS mit 0,1% BSA in 1 an den sekundären Antikörper herzustellen, verdünnter: 10.000-Verdünnung.
- Bereiten Sie TMB-Lösung durch die Reagenzien A und B Mischen gemäß den Anweisungen des Herstellers.
- Bereiten Sie Stop - Lösung durch Zugabe von 5 N Schwefelsäure (H 2 SO 4) in destilliertem Wasser und lagern bei RT.
2. Enzyme-linked immunosorbent assays (ELISAs)
HINWEIS: Die direkte Ligand-Rezeptor - Wechselwirkung ELISA (direct LRA, 1) können die Rezeptor-Ligand - Dissoziationskonstante (K D), als Maß für die Rezeptor-Liganden - Bindungsaffinität zu messen , verwendet werden. Der Wettbewerb Ligand-Rezeptor - Wechselwirkung ELISA (Wettbewerb LRA, Abbildung 2) alleOWS Screening von Peptiden (und andere blockierende Verbindungen), die mit der Wechselwirkung zwischen Ligand und Rezeptor zu interferieren handeln. Das Basisprotokoll , die zuvor 5 veröffentlicht wurde , wurde weiter optimiert.
Hinweis: In beiden ELISA Methoden Mehrkanalpipette nach Lösungen zu den Vertiefungen von 96-Well-Platte in jedem Schritt hinzugefügt wird. In Lösung dekantieren oder Waschschritte, werfen die Lösungen direkt in die Spüle.
- Direkter Ligand-Rezeptor-Interaktion Assay (direkte LRA)
HINWEIS: Eine Abbildung des Workflows (siehe Abbildung 1).- Lackplatte mit rekombinanten Rezeptor
- Verdünne den rekombinanten Rezeptor in Carbonat-Puffer auf eine Endkonzentration von 100 ng / & mgr; l. Coat Wells von 96-Well-Mikrotiterplatte mit festen Rezeptor-Konzentration (100 ng / ul) durch Pipettieren von 100 ul in jede Vertiefung einer Mehrkanalpipette mit. Ausschließen Außenwände der Platte gut Kante Artefakt zu vermeiden. Decken Sie die Platte mit einem Deckel und brüten diebei 4 ° CO / N Platte.
- Sperren und Zusatz von Liganden
- Am nächsten Tag, entfernen Sie die Beschichtungslösung durch die Platte gegen das Waschbecken kippen und die Platte 3-mal mit Waschlösung (PBS + 0,05% v / v Tween 20) waschen.
- Blockieren Sie die freien Rezeptor-Bindungsstellen in der beschichteten Platte unter Verwendung von 200 ul 5% BSA-Lösung in jede Vertiefung einer Mehrkanalpipette mit und inkubieren Sie die Platte für 2 Stunden bei RT.
- Entsorgen Sie die Blockierungslösung (siehe Schritt 2.1.2.1.) Und waschen Sie die Platte 3-mal mit Waschlösung.
- Bereiten Sie die rekombinante His-markierte Liganden bei verschiedenen Konzentrationen (beispielsweise 8 & mgr; g / ml, 4 ug / ml, 2 ug / ml, 1 & mgr; g / ml, 0,5 ug / ml, 0,25 & mgr; g / ml, 0,125 & mgr; g / ml, 0,063 & mgr; g / ml, 0,031 & mgr; g / ml, 0,0 ug / ml) in PBS. In nur PBS in den leeren Brunnen.
- Füge 100 ul jeder Ligandenkonzentration in die Vertiefungen in zweifacher Ausfertigung und inkubiere die Platte für 2 h bei RT ermöglicht Rezeptor-LigandenInteraktion.
- Inkubation mit Antikörper
- Nach der Inkubation mit den Liganden, waschen Sie die Platte 3-mal mit Waschlösung.
- Je 100 & mgr; l der primären anti-His monoklonalen Maus-Antikörperlösung (1: 1000) in jede Vertiefung.
- Die Platte bei RT für 2 h; nach der Inkubation, entsorgen Sie die Antikörperlösung (siehe Schritt 2.1.2.1.) und waschen Sie die Platte 3-mal mit Waschlösung.
- Füge 100 & mgr; l HRP gekoppelten Ziege-anti-Maus-IgG-sekundärem Antikörper-Lösung (1: 10.000) in jede Vertiefung. Die Platte 45 min bei RT.
- Entsorgen Sie die Antikörperlösung (siehe Schritt 2.1.2.1.) Und waschen Sie die Platte 3-mal mit Waschlösung.
- Zugabe von Substrat und Entwicklung
- Bringen Sie die TMB Substratlösungen auf RT und bereiten TMB Substratlösung A und B in 1: 1-Verhältnis. 100 l frisch hergestellte Substrat in jede Vertiefung und halten die Platte bei RT für 15-30 min. Nach ausreichender col oder Entwicklung werden 50 & mgr; l Stopplösung.
- Das Lesen der Platte und Datenanalyse
HINWEIS: Die beschriebene Protokoll wird auf der Annahme basiert, dass das gemessene Signal von spezifischen Bindungs ansteigt. Es könnte erforderlich sein, den Beitrag der unspezifischen Bindung an das Signal zu schätzen, aber dies ist aus dem Geltungsbereich dieses Protokolls.- Die Absorption (optische Dichte, OD) direkt bei 450 nm.
- Ziehen Sie das Hintergrundsignal aus den gemessenen OD-Werte und normalisieren. Wandeln Sie alle Werte der Ligandenkonzentration auf logarithmische Skala (Basis 10, log 10).
- Plot der normierten und Hintergrund korrigierten OD - Werte (Y-Achse entspricht dem Anteil der besetzten Rezeptor - Bindungsstellen) gegen den Logarithmus der Ligandenkonzentration (X-Achse, 10 Skala log).
- Um den K D - Wert zu schätzen, passen die Daten an die folgende Form der Hill - Gleichung:
tp_upload / 53575 / 53575eq1.jpg "/>
HINWEIS: Hier Y bezeichnet den Anteil der besetzten Rezeptor - Bindungsstellen und Y max die maximale Bindung; [L] die Konzentration an freiem Liganden und der Hill-Koeffizient. Wenn es nur eine Bindungsstelle für den Liganden ist, ist der Hill-Koeffizient n = 1. Bei Systemen mit mehr als einem Ligandenbindungsstelle, die Bindungs weist positive Kooperativität, wenn n> 1 ist, negativen Kooperativität wenn n <1 und keine Kooperativität wenn n = 1. die mikroskopische Dissoziationskonstante wird bezeichnet und entspricht der Hälfte der maximalen wirksamen Konzentration EC 50 6. Die scheinbare Dissoziationskonstante K d = (K D) n. Im einfachsten Fall , in dem n = 1, die Dissoziationskonstante entspricht dem Ligandenkonzentration , bei der die Hälfte der Rezeptorbindungsstellen besetzt sind , und K d = K D. Dieses Modell geht davon aus Massenaktion unter Gleichgewichtsbedingungen verbindlich, sowie, dass nur ein kleiner Bruchteildie zugegebene Liganden an den Rezeptor gebunden, das heißt, [L] >> [RL].
- Lackplatte mit rekombinanten Rezeptor
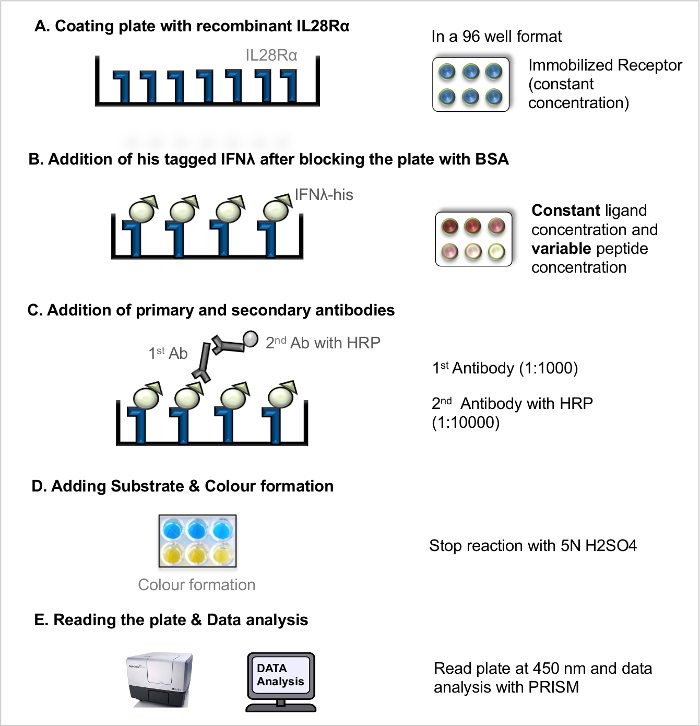
Abbildung 1. Direkte Ligand-Rezeptor-Interaktion Assay (direkte LRA). Schritt- für -Schritt - Protokoll für die direkte LRA. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
- Wettbewerb Ligand-Rezeptor-Interaktion Assay (Wettbewerbs LRA)
HINWEIS:. Für eine Darstellung des Workflows siehe Abbildung 2 Der Wettbewerb LRA Verfahren folgt die gleichen Schritte wie die direkte LRA (Beschichtung der Platte, Antikörperinkubation, Entwicklung Platte) , mit Ausnahme wichtige Veränderungen in den Liganden und Peptiden Additionsschritt. Die richtige Negativkontrollen sind für diesen Test von wesentlicher Bedeutung. In einer früheren Screening-Studie 4, das verschlüsselte Blockierung Peptid zeigte keine antagonistische Wirkungen.- Blocking - Zusatz von Liganden und Blocking Peptide
- Am nächsten Tag, entfernen Sie die Beschichtungslösung und waschen Sie die Platte (siehe 2.1.2.1).
- Blockieren Sie die beschichtete Platte durch Zugabe von 200 ul 5% BSA-Lösung in jede Vertiefung und inkubieren Sie die Platte für 2 Stunden bei RT.
- Bereiten Sie die rekombinante His-markierte Liganden (IFNL1-3) bei einer festen Konzentration (2x-20 ng / ml) in PBS.
- Bereiten Sie die blockierende Peptid (siehe Tabelle 3) mit unterschiedlichen Konzentrationen von 10 nM bis 100 uM in PBS Bereich eine Dosis-Wirkungs-Kurve zu gewährleisten.
ANMERKUNG: Dies ermöglicht die anschließende Bestimmung des IC 50 -Wertes für das Blockierungspeptid. In Kontrollvertiefungen, fügen Sie nur feste Ligand-Konzentration ohne Peptid das Maximum (100%) Bindung abzuleiten. In dem leeren, fügen Sie nur PBS ohne Ligand oder Peptid. - Je 50 ul der Liganden (IFNL1-3) und 50 ul jeder peptide Konzentration auf die Vertiefungen in Duplikaten.
- Inkubieren der Platte für 2 h bei RT.
- Das Lesen der Platte und Datenanalyse
HINWEIS: Die beschriebene Protokoll wird auf der Annahme basiert, dass das gemessene Signal von spezifischen Bindungs ansteigt. Es könnte erforderlich sein, den Beitrag der unspezifischen Bindung an das Signal zu schätzen, aber dies ist aus dem Geltungsbereich dieses Protokolls.- Die Absorption (optische Dichte, OD) direkt bei 450 nm.
- Ziehen Sie das Hintergrundsignal aus den gemessenen OD-Werte und normalisieren. Wandeln Sie alle Werte der Peptidkonzentration auf logarithmische Skala (Basis 10, log 10).
- Plot der normierten und Hintergrund korrigierten OD - Werte (Y-Achse entspricht dem Anteil der besetzten Rezeptor - Bindungsstellen) gegen den Logarithmus der Ligandenkonzentration (X-Achse, 10 Skala log).
- Um die IC - 50 - Wert zu schätzen, passen die Daten an die folgende Gleichung:
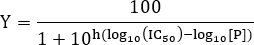
HINWEIS: Hier [P] ist die Peptidkonzentration und der Hill-Anstieg. Der Hügel Steigung beschreibt die Steilheit der Dosis-Wirkungs-Kurve. Der IC 50 entspricht der Inhibitorkonzentration , bei der 50% Inhibierung der Bindung zwischen Ligand und Rezeptor beobachtet wird.
- Blocking - Zusatz von Liganden und Blocking Peptide

Abbildung 2. Wettbewerb Ligand-Rezeptor-Interaktion Test (Wettbewerb LRA). Schritt- für -Schritt - Protokoll für den Wettbewerb LRA. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Ergebnisse
Die Dissoziationskonstanten zwischen INFL1-3 und deren Rezeptor alpha-Untereinheit IL28RA wurden unter Verwendung des direkten LRA bestimmt. Die Ergebnisse sind in Abbildung 3: Der Anteil an besetzten Bindungsstellen aufgetragen gegen den Logarithmus der jeweiligen IFN - Konzentration. Die Scatchard-Plot der Daten wird in der rechten unteren Ecke des Bildschirms angezeigt. Die Ergebnisse zeigen , dass die direkte LRA eine Bindungskurve ergibt, was die K D -Wer...
Diskussion
ELISA ist ein Standard und gut etablierte Methode für viele Laboratorien. Wir haben weiter modifiziert und verbessert eine zuvor Methode 5,7 veröffentlicht. Die aufgezeigte Schritt- für -Schritt - Protokoll zeigt , wie es in einfacher Weise verwendet werden können , um die K d -Werte von Ligand-Rezeptor - Wechselwirkungen zu bestimmen. Darüber hinaus ist die IC50 eines Blockierungspeptid, das bestimmt werden kann, mit dem Liganden-Rezeptor-Wechselwirkung stört.
We...
Offenlegungen
A.E. was supported by a research grants from the "SNSF Ambizione Score" program (PZ00P3_154709), "Forschungsfond, Förderung strategischer Projekte" University of Basel, Stiftungsinfektionskrankheiten Basel, and Bangeter Rhyner Stiftung. J.L. acknowledges support by an iPhD fellowship of the SystemsX.ch initiative in systems biology program (9th call).
Danksagungen
We thank Prof. J. Stelling (Department of Biosystems Science and Engineering, ETH Zurich and Swiss Institute for Bioinformatics, Basel, Switzerland) for his critical review of the manuscript.
Materialien
| Name | Company | Catalog Number | Comments |
| Nunc-Immunoplate (F96 Maxi sorp) | Thermo Scientific | 442404 | ELISA plate |
| Sodium carbonate (Na2CO3) | Merck | 497-19-8 | For ELISA plate coating buffer |
| Sodium hydrogen carbomnate(NaHCO3) | Merck | 144-55-8 | For ELISA plate coating buffer |
| Bovine Serum Albumin (BSA) | Sigma | A7030-100G | 5% BSA in PBS for Blocking |
| rhIL-28Rα/IFNλR1 | R&D systems | 5260-MR | Recombinant human interlukin-28 Receptor alpha |
| rhIL-29/IFNλ1 | R&D systems | 1598-IL/CF | Recombinant human interlukin-29/Carrier free/C-terminal 10-His tag |
| rhIL-28A/IFNλ2 | R&D systems | 1587-IL/CF | Recombinant human interlukin-28A/Carrier free/C-terminal 6-His tag |
| rhIL-28B/IFNλ3 | R&D systems | 5259-IL/CF | Recombinant human interlukin-28B/Carrier free/C-terminal 6-His tag |
| 6x His Monoclonal antibody (Mouse) | Clontech | 631212 | Primary antiboy to capture His tagged Ligands |
| Goat anti-Mouse igG (H+L) | Jackson Immuno Research | 115-035-166 | Horseradish Peroxidase conjucated secondary antibody |
| BDoptEIA TMB reagent set | BD Biosciences | 555214 | ELISA - TMB substrate solution |
| Sulfuric acid (H2SO4) | Fulka | 84720 | 5 N H2SO4 (Enzyme reaction stop solution) |
| Synergy/H1 - Microplate reader | BioTeK | ELISA plate reader |
Referenzen
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods in enzymology. 545, 103-125 (2014).
- Rossi, G., et al. Biosensor analysis of anti-citrullinated protein/peptide antibody affinity. Analytical biochemistry. 465, 96-101 (2014).
- van der Merwe, P. A., Barclay, A. N. Analysis of cell-adhesion molecule interactions using surface plasmon resonance. Curr Opin Immunol. 8, 257-261 (1996).
- Egli, A., et al. IL-28B is a key regulator of B- and T-cell vaccine responses against influenza. PLoS Pathog. 10, e1004556 (2014).
- Rosenbluh, J., et al. Positively charged peptides can interact with each other, as revealed by solid phase binding assays. Analytical biochemistry. 352, 157-168 (2006).
- Goutelle, S., et al. The Hill equation: a review of its capabilities in pharmacological modelling. Fundamental & clinical pharmacology. 22, 633-648 (2008).
- Levin, A., et al. Peptides derived from HIV-1 integrase that bind Rev stimulate viral genome integration. PLoS One. 4, e4155 (2009).
- Egli, A., Santer, M. D., O'Shea, D., Tyrrell, D. L., Houghton, M. The impact of the interferon-lambda family on the innate and adaptive immune response to viral infections. Emerging infectious diseases. , e51 (2014).
- Gad, H. H., Hamming, O. J., Hartmann, R. The structure of human interferon lambda and what it has taught us. J Interferon Cytokine Res. 30, 565-571 (2010).
- Folch, B., Rooman, M., Dehouck, Y. Thermostability of salt bridges versus hydrophobic interactions in proteins probed by statistical potentials. Journal of chemical information and modeling. 48, 119-127 (2008).
- Yuzlenko, O., Lazaridis, T. Interactions between ionizable amino acid side chains at a lipid bilayer-water interface. The journal of physical chemistry. B. 115, 13674-13684 (2011).
- Tissot, A. C., Vuilleumier, S., Fersht, A. R. Importance of two buried salt bridges in the stability and folding pathway of barnase. Biochemistry. 35, 6786-6794 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten