Method Article
Einrichtung von Genom bearbeitet humanen pluripotenten Stammzelllinien: Von Targeting zur Isolation
In diesem Artikel
Zusammenfassung
Genome editing of human pluripotent stem cells (hPSCs) can be done quickly and efficiently. Presented here is a robust experimental procedure to genetically engineer hPSCs as exemplified by editing the AAVS1 safe harbor locus to express EGFP and introduce antibiotic resistance.
Zusammenfassung
Genom-Bearbeitung von menschlichen pluripotenten Stammzellen (hPSCs) bietet eine genetisch gesteuert und klinisch relevante Plattform, um die menschliche Entwicklung zu verstehen und zu untersuchen, die Pathophysiologie der Erkrankung. Durch die Verwendung von ortsspezifischen Nukleasen (Sozialversicherungsnummern) für die Genom Bearbeitung, wird die schnelle Ableitung neuer HPSC Linien beherbergen spezifische genetische Veränderungen in einem ansonsten isogenen Einstellung möglich. Zinkfinger-Nukleasen (ZFNs), Transkriptionsaktivator artigen Effektormolekülen Nukleasen (Talens) und Clustered regelmäßig beabstandeten kurzen palindromischen Repeats (CRISPR) / Cas9 sind die am häufigsten verwendeten Sozialversicherungsnummern. Alle diese Nukleasen funktionieren durch Einführen eines doppelsträngigen DNA-Bruch bei einem bestimmten Ort und damit eine präzise Bearbeitung Gens fördern bei einer genomischen Locus. SSN-meditierte Genombearbeitung nutzt zwei der endogenen DNA-Reparaturmechanismen der Zelle, nicht-homologen Ende (NHEJ) und Homologie gerichtet Reparatur (HDR), um entweder einzuführen Einfügen / Löschen Mutationen oder alter das Genom mit einer homologen Reparatur-Vorlage an der Stelle des Doppelstrangbruch. Elektroporation von hPSCs ist ein effizientes Mittel zur Transfektion Sozialversicherungsnummern und Reparatur-Vorlagen, die Transgene beinhalten, wie beispielsweise fluoreszierende Reporter und Antibiotikaresistenz-Kassetten. Nach der Elektroporation, ist es möglich, nur die hPSCs dass die Reparaturkonstrukt durch Selektion auf Antibiotika-Resistenz einge isolieren. Mechanisches Trennen HPSC Kolonien und Bestätigung angemessene Integration am Zielort durch Genotypisierung ermöglicht die Isolierung des korrekt ausgerichtet und genetisch homogenen Zelllinien. Die Gültigkeit dieses Protokolls ist hier durch Verwendung aller drei SSN-Plattformen zu integrieren EGFP und einem Puromycin-Resistenz-Konstrukts in der AAVS1 Safe-Harbor-Locus in der menschlichen pluripotenten Stammzellen nachgewiesen.
Einleitung
Genome Editing-Technologien entwickeln sich rasant in die Standardwerkzeuge für Molekular- und Zellbiologie 1. Gentechnik menschlichen pluripotenten Stammzellen (hPSCs) ist von besonderem Interesse, da hPSCs stellen eine selbst erneuernden Quelle genetisch intaktes primären menschlichen Zellen. hPSCs kann in verschiedene Zelltypen für Krankheitsmodelle oder als Quelle für den Transplantationstherapien 2,3 unterscheiden. Hier dargestellt ist ein Protokoll, das drei verschiedene Arten von ortsspezifischen Nukleasen (SSNs) in Verbindung mit endogenen DNS-Reparaturmechanismen zur gezielten Integration eines Reporterkonstrukt im AAVS1 Locus verwendet. Nach Transfektion von Sozialversicherungsnummern in hPSCs zeigen wir, wie man isogene Zellpopulationen zu isolieren beherbergen den Reporter.
Die Möglichkeit, Genome zu manipulieren, spezifisch pluripotente Stammzellgenome ist mit SSNs kein neues Phänomen, wie die Nützlichkeit der Zinkfinger-Nukleasen (ZFNs) und Transkriptions betätigendesoder artigen Effektormolekülen Nukleasen (Talens) für die Gen-Editing wurde vor einigen Jahren 4-10 demonstriert. Doch mit dem Aufkommen von S. pyogenes CRISPR / Cas9 Technologie 11-13, Gen-Editing hat allgemein zugänglich 14 geworden. Alle SSNs Einführung einer doppelsträngigen DNA Bruch (DSB) am angegebenen Zielort 1,4,5,11, die durch endogene zelluläre Mechanismen entweder unter Verwendung nicht-homologen Ende (NHEJ) oder Homologie gerichteten Reparatur (HDR) 15 repariert wird. NHEJ ist fehleranfällig und kann Frameshift-Mutationen, was zum Verlust der Genfunktion einzuführen, während HDR ermöglicht neuartige Elemente, die durch die Co-Transfektion eines Reparatur Schablone mit der SSN eingeführt werden. Während die zugrunde liegenden Prinzipien der DNA-Reparatur, die Gen-Bearbeitung zu erleichtern sind gedacht, um weitgehend die gleichen für jede SSN gewährleisten, sind gewisse Unterschiede zwischen den Plattformen zu beachten. De-novo-Design ZFNs ermöglicht Flexibilität und Nuklease Optimierungs 16 jedoch die Verwendung von öffentlichvorhandenen Montage Bibliotheken und Screening-Tools, um einzelne ZFNs entwerfen kann zeitaufwendig sein. Sobald die gewünschte Stelle für ZFN-vermittelte Targeting bestimmt ist, kann ZFN Paare mit der Online-Werkzeug ZiFit 17 ausgebildet sein. Nach dem Design, können ZFNs modular über mehrere Runden von Plasmid-Klonen 18 zusammengebaut werden. Alternativ gibt es viele im Handel erhältliche, vorvalidierter ZFNs 19. TALE Nukleasen können mit Online-Tools und öffentlich zugängliche Bauteile 17,20 gestaltet werden. Beispielsweise kann TALENS schnell von Blöcken von fünf GESCHICHTE wiederholt zusammengebaut werden, durch FLASH-Baugruppe 21 oder unter Verwendung von PCR basierte hierarchische Golden Gate-Anordnung 22. Einfache SSN Design und Geschwindigkeit des Aufbaus mit CRISPR / Cas9 haben Genom hergestellt Bearbeitung einer breiten Masse zugänglich Werkzeug. Der Kurzführer RNA-vermittelte Targeting CRISPR / erlaubt Cas9 auch zum Multiplexen von Führungs RNAs zu mehreren Loci mit einem einzigen Konstrukt 14 zielen. Das Designder Cas9 zur Gen Bearbeitung erfordert nur die Identifikation eines protospacer benachbarten Motiv (PAM, ein NGG Trinukleotid von S. pyrogenes Cas9) proximal zu dem Zielort. Durch Einfügen eines Oligonukleotids, der dem 20 Basenpaare 5 'von der PAM in den px330 Plasmid 14 kann das Konstrukt in einen Klonierungsschritt gebaut werden. Zusätzlich zu S. pyogenes Cas9, Cas9 von N. meningitidis (NmCas9), die eine 5'-NNNNGATT-3 '(PAM) erkennt wurde gezeigt, dass für eine effiziente Gen-Bearbeitung in hPSCs 23 ermöglichen.
Zusätzlich zu den Unterschieden in einfache SSN Ausführung weist jede Plattform spezifischen Eigenschaften. Zum Beispiel ZFNs und Talens nutzen die FokI Nucleasedomäne, die eine vier Nukleotid-5'-Überhang 24 erzeugt, während Cas9 wird gedacht, um vor allem zu erzeugen blunt ended DSBs. ZFNs, Talens, und Cas9 unterscheiden sich in ihrer Proteinstabilitäten, on-off-Rate auf Ziel-DNA, und die Art der DNA-Scannen, von denen c alleOuld theoretisch in kleine Unterschiede in der Bearbeitung Ergebnis 1 führen. Während weitere Studien erforderlich, um die Folgen dieser Unterschiede verstehen werden, beschreiben wir hier ein Protokoll, das sehr robust in allen drei Plattformen ist und kann verwendet werden, um leicht zu generieren gentechnisch veränderten hPSCs.
Unabhängig von SSN Wahl, ist die Elektroporation ein robustes Verfahren, um Sozialversicherungsnummern und Homologie Reparatur Vorlagen in hPSCs 25 transfizieren. Die Anzahl der überlebenden Kolonien nach der Selektion auf Antibiotika-Resistenz hängt von ortsspezifischen Parameter und der Bearbeitungsstrategie (zB Größe transgener Einsatz und die Art der Auswahl). Das hier beschriebene Protokoll führt in der Regel etwa 150 bis 400 Einzelzell-abgeleiteten Kolonien.
Gen-Schnitt an der AAVS1 Locus unter Verwendung dieses Protokolls wurde früher verwendet, um die Wirksamkeit der SSNs 4,5 demonstrieren. Die AAV-CAGGS-EGFP-Reparatur-Vorlage verwendet eine Gen-Trap-strategie zu Puromycin Resistenz gegen in einem Ort gezielt. Kurz gesagt, enthält das Reparaturschablone eine Spleißakzeptorstelle stromaufwärts des promotor Puromycin-Resistenz-Kassette. Auf korrekte Integration in den ersten Intron des Gens PPP1R12C am AAVS1 Locus wird die Resistenzkassette aus der bearbeiteten Gen Promoter ausgedrückt. Die Robustheit dieses spezifischen AAVS1 Assay ermöglicht es uns, die Effizienz jedes SSN Plattform zu vergleichen.
Gene Editing mit Sozialversicherungsnummern ist mächtig angesichts der Möglichkeit, zu stören und / oder zu verändern theoretisch jedes Gen. Anwendung dieser Strategie hPSCs bietet Vielseitigkeit hPSCs können anschließend in einer Vielzahl von menschlichen Zelltypen, wie Neuronen 26 differenziert, Hepatozyten 27 und Kardiomyozyten 28. Außerdem ist die Verwendung von Patienten stamm pluripotenten Stammzellen ermöglicht die Reparatur oder der Einführung von bekannten Krankheit verursachenden Mutationen in einem Patienten-spezifischen genetischen Hintergrund 29 Und bietet eine Plattform, um Krankheitsmechanismen und Test Therapeutika unter Verwendung von patienteneigenen Zellen 30 zu untersuchen. Zusammenfassend ist Gen-Bearbeitung in hPSCs ein effizienter und vielseitiger Ansatz zur Untersuchung der biologischen Grundlagen der menschlichen Entwicklung und Krankheit 31.
Protokoll
Die in dieser Handschrift beschriebenen Verfahren wurden überprüft und von der UC Berkeley Stammzellforschung Oversight Committee genehmigt.
1. Bereiten Sie Stammzellen für die Bearbeitung
- Wachsen und Kultur menschlichen pluripotenten Stammzellen (hPSCs) auf einer Platte mit 6 Vertiefungen, die 2,4 × 10 6 Zellen / Platte von Mitomycin C-inaktivierten embryonalen Maus-Fibroblasten (MEF) Zubringer auf Gelatine 32 gewachsen. Pflegen hPSCs in 3 ml humanen embryonalen Stammzellmedien pro Vertiefung (hESC Medien) und wachsen in eine 37 ° C-Inkubator mit 3% O 2/5% CO 2.
Anmerkung: Für den Erfolg dieses Protokoll ist es nicht notwendig, hPSCs in einer sauerstoffarmen Inkubator aufrecht zu erhalten; aber es ist wichtig zu beachten, dass hPSCs vermehren sich schneller in einer hohen O 2 Umwelt, so Zeiten und Zellzahlen entsprechend angepasst werden. Es sollte auch darauf hingewiesen, dass hPSCs in niedrigen Sauerstoff gehalten haben niedrigere Rate der spontanen Differenzierung 33 werden.- To machen 500 ml hESC Medien kombinieren, 380 ml DMEM / F12, 75 ml fötalem Rinderserum (FBS), 25 ml KnockOut Serum Ersatz (KSR). Hinzufügen Glutamin (1 mM Endkonzentration), 5 ml 100x Nicht-essentielle Aminosäuren, 100 Einheiten / ml Penicillin-Streptomycin (P / S), basischen Fibroblastenwachstumsfaktor (bFGF) (4 ng / ml Endkonzentration) und 2- Mercaptoethanol (5,5 & mgr; M Endkonzentration) bestimmt.
- Verändern Medien durch Entfernen gesamte Volumen der Medien (3 ml) unter Verwendung einer Glaspipette und Vakuum. Ersetzen Sie mit 3 ml warmem hESC Medien mit einer serologischen Pipette. Wiederholen Sie die Medien verändern jeden Tag bis hPSCs sind ca. 50% konfluent (Tag -1).
- Einen Tag vor Targeting (Tag -1), ändern hESC Medien, Entfernen der alten Medien und Zugabe von warmen hESC Medien mit 10 & mgr; Y-27632 ergänzt.
- Auch am Tag -1, bereiten 1.59 6-Well oder 10-cm-Platten von resistenten MEF Nährzellen von DR4-Mäusen (2,4 · 10 6 Zellen / Platte) 34.
Hinweis: In der Regel sind 6-Well-Platten adhaften über 10 cm-Platten, da sie Platz für mehr Medien. 6-Well-Platten auch sicherstellen, dass die verschiedenen Klone aus verschiedenen Vertiefungen sind unabhängig. Wird jedoch eine 10-cm-Platte einfacher Kommissionierung erlauben, abhängig von der für diesen Prozess zur Verfügung Mikroskop.
2. Bearbeiten von pluripotenten Stammzellen
- Vorbereitung Transfektionslösungen durch Pipettieren von je 5 ug ZFN 1 und 2 TALEN 1 und 2 bzw. 15 ug des CRISPR / Cas9 px330 kodierenden Plasmid (Figur 1) in ein 1,5 ml Röhrchen. Pipette 30 ug des Reparatur Plasmid in diesem Rohr als auch. Schließlich Pipette 1x Phosphatgepufferte Kochsalzlösung (PBS) in die Röhre, um die Lautstärke bis zu 300 & mgr; l zu bringen.
Anmerkung: Bereiten Plasmiden als Midiprep (jeder Bausatz ist) mit einer Konzentration von mindestens 300 ng / & mgr; l, um das Gesamtvolumen der Transfektionslösung unter 300 & mgr; l zu halten. Es ist nicht notwendig, Phenol / Chloroform-Extraktion durchzuführen, um das Plasmid zu linearisieren, oder eine endoto verwendenxin freien Plasmid Prep Kit. - Entfernen Sie das Medium aus hPSCs Verwendung einer Glaspipette und Vakuum. Pipette 2 ml warmem 1x PBS in jede Vertiefung, um die Zellen zu waschen.
- Entfernen Sie die PBS sofort mit einer Glaspipette und Vakuum. 0,5 ml 0,25% Trypsin-EDTA-Lösung direkt auf Zellen in jeder Vertiefung. Legen in den Inkubator (37 ° C / 5% CO 2/3% O 2) für ca. 10 min. oder bis Feeder-Schicht beginnt, heben Sie die Platte.
- 2 ml warmem esWash Medium (470 ml DMEM / F12, 25 ml FBS, 100 Einheiten / ml P / S) in jede Vertiefung, um das Trypsin Reaktion zu stoppen.
- Sicherzustellen, dass die Feeder-Zellen aus wie ein Blatt zu kommen. Pipettieren Sie die Inhalte jeder Vertiefung in einem einzigen 50 ml konischen Röhrchen, die Kombination aller Brunnen. Man reibt Zellen unter Verwendung einer 10 ml serologische Pipette. Es ist nicht notwendig, brechen die Feeder-Schicht; hPSCs wird weg von der Feeder-Schicht durch leichtes Verreiben kommen. Es sollte etwa 15 ml der Zellsuspension werden.
- 25 ml esWash Medien, um das Rohr, um die volu bringenme bis zu 40 ml insgesamt. Erlauben große Brocken Zuführung am Boden des Röhrchens für 1-2 Minuten absetzen. Entfernen Sie den Überstand (~ 38 ml) aus dem Rohr mit einer serologischen Pipette und Einzahlung auf ein neues 50 ml konischen Röhrchen.
- Spin down für 5 Minuten bei 190 x g. Den Überstand aus dem Röhrchen unter Verwendung einer Glaspipette und Vakuum. Achten Sie darauf, das Zellpellet nicht stören. Resuspendieren der Zellen in 500 ul 1x PBS. Kombinieren Sie mit früheren vorbereitet Plasmidtransfektion Lösung. Zähle die Zellen in diesem Schritt. Verwenden Sie 5-10 Million Zellen pro Elektroporation.
- Pipettieren Sie die gesamte 800 ul Suspension in eine 4 mm Elektroporationsküvette, setzen auf Eis für 3-5 min. Elektroporieren Zellen unter Verwendung des Exponential-Programm auf der Elektroporation System mit den folgenden Parametern: 250 V, 500 uF, ∞ Beständigkeit und 4 mm Küvette Größe. Nach der Elektroporation platzieren Küvette wieder auf Eis für 3 min.
Hinweis: Beachten Sie die Zeitkonstante der Elektroporation auf der Elektroporation System. Die Zeitkonstantevariiert mit der Anzahl von Zellen und DNA-Reinheit. Erfolgreiche Transfektionen haben in der Regel eine Zeit zwischen 10 bis 14 ms konstant, wenn Sie die genannten Bedingungen und dem Gene Pulser II. Unter Elektroporation Effizienz kann bei Zeitkonstanten variiert von diesen Werten. - Resuspendieren elektroporierten Zellen in 18 ml warmem hESC Medien, ergänzt mit 10 & mgr; M Y-27632. Platte 3 ml dieser Einzelzellsuspension in jede Vertiefung einer Platte mit 6 Vertiefungen mit DR4-Feeder-Zellen. Zurück zum Inkubator (37 ° C / 5% CO 2/3% O 2).
3. Auswahl der Positive Kolonien
- Tag 2, entfernen Sie alle Medien unter Verwendung einer Glaspipette und Vakuum. Ersetzen Sie mit 3 ml warmem hESC Medien mit 10 & mgr; Y-27632 ergänzt. Tag 3, entfernen Sie alle Medien unter Verwendung einer Glaspipette und Vakuum. Ersetzen Sie mit 3 ml warmem hESC Medien ohne einschließlich Y-27632. Tag 4, entfernen Sie alle Medien unter Verwendung einer Glaspipette und Vakuum. Ersetzen Sie mit 3 ml warmem hESC Medien mit Antibiotika für selecti ergänztam. Zurück zum Inkubator (37 ° C / 5% CO 2/3% O 2).
Hinweis: Der Typ des Antibiotikums verwendet wird, hängt die Resistenzkassette hängt im Reparaturschablone enthalten. Beim Arbeiten mit WIBR # 3 hPSCs (NIH Register 0079), 0,5 & mgr; g / ml Puromycin, 70 & mgr; g / ml G418 (Geneticin) und 35 ug / ml Hygromycin wurden erfolgreich verwendet. Die Konzentrationen für die Auswahl sollte empirisch durch die Gründung der minimalen Konzentration des Antibiotikums notwendig, um Wildtyp-Zellen innerhalb von etwa einer Woche zu töten bestimmt werden. - Tage 5-12, ändern Medien täglich, den Austausch alter Medien jedes Mal mit warmem hESC Medien mit Antibiotika ergänzt.
Hinweis: Erwarten Sie eine große Menge an Zelltod. Einzelne Kolonien werden offensichtlich um Tag 8-10. Regelmäßige hESC Medien ohne Antibiotika kann nach 12-14 Tagen kontinuierlicher Selektion verwendet werden. Wenn die Zelldichte hoch ist oder Zelltod langsam ist, sollte Ansäuerung des Mediums vermieden werden und es kann notwendig sein, um die Medien zu erhöhen Volume (bis zu 4-5 ml) während der ersten paar Tage der Selektion.
4. Heben ausgewählten Kolonien (Tag 12-14)
- Beachten Kolonien am Tag 12-14. Beachten Sie die Kolonien, die bereit sind, auf einem Dissektionsmikroskop holen und sicherzustellen, dass sie Zellen, die beginnen, zu differenzieren nicht enthalten sind. Die ungefähre Größe sollte 800-1.200 um betragen. Wenn Kolonien erreichen diese Größe vor der 12. Tag, ist es empfehlenswert, dass sie dann abgeholt werden.
Hinweis: Für jede Ziel Experiment holen so notwendig wie viele Kolonien, um sicherzustellen, dass der gewünschte Genotyp isoliert. Als ein Beispiel hat AAVS1 Bearbeiten mit dem AAV-CAGGS-EGFP Reparaturschablone konsistent und stabilen Integration gezeigt und erfordert nur 12-24 Kolonien ausgewählt, um etwa 5-10 heterozygot gezielte Klone erhalten. Andere Targeting Experimente können mehr Kolonien verlangen abgeholt werden (Tabelle 1). Die Häufigkeit der korrekten Ausrichtung Ereignisse hängt von Faktoren wie der Effizienz des SSN einen DSB, die Größe des Einsatzes und die Auswahlstrategie einzuführen. Im Fall der Gen-Trap-Ansatz zur AAVS1 Locus hier präsentiert wird, der Selektionsmarker nur exprimiert, wenn sie richtig an der Zielstelle integriert, wodurch die Anzahl der Kolonien erforderlich ist, um eine korrekt gezielte Klon erhalten. - Einen Tag vor Kommissionierung, bereiten 12-Well-Platten von MEFs (2,4 x 10 6 Zellen / Platte, wird man auch verwendet werden, für jede Kolonie abgeholt).
- Am Tag der Kommissionierung, entfernen Sie alle Medien aus den 12 und MEF-Platten unter Verwendung einer Glaspipette und Vakuum und Ersetzen mit 1 ml hESC Medien. Auch am Tag der Kommissionierung, ändern hESC Medien über die 6-Well-Platten, die gehen, abgeholt werden.
- Ziehen Glaspipetten für die mechanische Dissoziation von einzelnen Kolonien von der Feeder-Schicht. Senken Sie die Pipetten über eine in-Kapuze Bunsenbrenner und an der Expansionspunkt erweicht, bis Glas ist formbar. Schnell zu entfernen, von der Flamme, und ziehen Sie die Pipette auseinander Schaffung einer Winkelpunkt. Breche diePunkt etwa 2 cm von dem gebogenen Achse, so dass ein schmaler Kanal. Polieren des Endes des Kanals durch Aussetzen Flamme für 1-2 sec.
Hinweis: Optional abholen Kolonien eine p20 Pipettenspitze, jedoch kann dies die Klonalität der Kultur zu reduzieren, wie die stumpfe Spitze kann größere Mengen von Zellen aus jeder Kolonie in die Medien zu vertreiben, potenziell verunreinigende nachfolgenden Beute. - Montieren Aufnahmevorrichtung, indem sie einen p1000 Filterpipettenspitze und Befestigen eines Saugball um das schmale Ende. Legen Sie die herausgezogen Glaspipette in das breite Ende.
- Legen Sie eine 6-Well-Platte von hPSCs auf der Bühne eines Dissektionsmikroskop in einer Gewebekultur Haube montiert abgeholt werden. Komprimieren Sie die Glühbirne der Abzugseinrichtung und vorsichtig herauszuschneiden und schneiden Sie eine Einzelkolonie in 10-20 gleich große Stücke, dabei nicht in Stücke in der Medienmitteilung. Nehmen Sie herausgeschnitten HPSC Stücke der Kolonie in die Pipette durch Lösen der Lampe. Versuchen Sie, so wenig Medien wie möglich während der Übertragung.
- Transfer die Einzelkolonie auf eine einzelne Vertiefung einer 12-Well-MEF Platte, komprimiert die Glühbirne wieder direkt in den Brunnen, die Freigabe der nun gebrochen Kolonie. Beschriften Sie jedes gut auf eindeutige Identifikation von Einzelzell-abgeleiteten Klonen zu erlauben. Wiederholen Sie dies für so viele Kolonien wie erforderlich, die Änderung der Glaspipette jeder Zeit.
Hinweis: Die Auswahl Kolonien kann einige Zeit dauern, um zu lernen. Es wird empfohlen, den Experimentator Praxis an einigen Kontrollzellen, bevor Sie versuchen, um gezielte Kolonien zu isolieren. - Rückkehr 12 Well-Platten in den Inkubator (37 ° C / 3% O 2/5% CO 2), sanftes Schütteln der Platte zunächst um eine Ansammlung von Zellen, die in der Mitte jeder Vertiefung zu vermeiden.
- Am nächsten Tag und jeden weiteren Tag, nehmen Sie volle Volumen von Medien unter Verwendung einer Glaspipette und Vakuum und Ersetzen mit 1,5 ml warmem hESC Medien, bis Zellen sind etwa 50% konfluent (dies dauert in der Regel 10-12 Tage).
- Nach 10-12 Tage, nehmen 1-2 Kolonien aus jeder Vertiefung und Transfer zum neuen 12-Well-Platten neu MEFholenden Schritte 4,1 bis 4,8, um ein Replikat Platte zu erzeugen.
- Extrahieren DNA (siehe unten) von den verbleibenden Kolonien in jeder Vertiefung der ursprünglichen MEF Platten.
- Entfernen Sie das Medium aus allen Vertiefungen mit einer Glaspipette und Vakuum. Pipette 1 ml 1x PBS auf jede Vertiefung, um Zellen zu waschen. Entfernen PBS mit Glaspipette und Vakuum. Pipette 250 ul Zelllysepuffer (Endkonzentrationen in H 2 O: 10 mM Tris HCl, 5 mM EDTA, 0,2% SDS, 200 mM NaCl, 0,08 mg / ml Proteinase-K) auf jede Vertiefung. Platz im Inkubator (37 ° C / 3% O 2/5% CO 2) O / N.
- Am nächsten Tag, Pipetten Inhalt der Vertiefungen in einzelne 1,5 ml Gefäße. Pipette 250 ul Isopropylalkohol in jedes Rohr, um die DNA auszufällen. Schütteln Sie Rohr kräftig. Ein weißer Niederschlag sichtbar sein soll.
- Spin jedes Rohr nach unten für 3 min bei 13.000 rpm in einer Tischzentrifuge. Nach dem Spin-Down, Überstand verwerfen durch Dekantieren in flüssige Abfälle. Ein kleiner DNA-Pellet sollte zum Boden stecken bleibtdas Rohr. Pipette 250 ul 70% Ethanol in jedes Rohr, um das DNA-Pellet zu waschen. Kräftig schütteln. Spin jedes Rohr nach unten für 3 min bei 13.000 rpm in einer Tischzentrifuge.
- Nach Stillstand kommen, zu verwerfen Überstand durch Dekantieren in flüssige Abfälle. Ein kleiner DNA-Pellet sollte klebte an der Unterseite des Rohres verbleiben. Pipette, um den Rest des Überstandes, so gibt es keine Flüssigkeit in dem Rohr belassen. Lassen Rohr offen auf Benchtop 5-10 min trocknen. Nach dem Trocknen resuspendieren DNA in 250 ul TE Puffer. Bei 37 ° C für 6 Stunden, damit DNA zu lösen.
- Genotyp jede Probe unter Verwendung eines vorab optimierten PCR oder Southern-Blot-Strategie.
Hinweis:.. Für AAVS1 Targeting mit dem AAV-CAGGS-EGFP-Reparatur-Vorlage kann der Southern-Blot-Strategie in Hockemeyer finden et al 2009 können 4 Eine umfassende Southern-Blot-Protokoll im Süden gefunden werden 2006 35 Für Multiplex-PCR Genotypisierung. Das gleiche Experiment, Primer und Bedingungen könnenin der Tabelle 2 36 gefunden. - Entsorgen Brunnen, die nicht richtig ausgerichtet sind und weiter verändernden hESC Medien zielgerichtet Zellen jeden Tag. Frieren Sie sich die Zellen, wenn sie etwa 50% konfluent sind, siehe unten für das Einfrieren Protokoll.
- Einen Tag vor dem Einfrieren, ändern hESC Medien, Entfernen der alten Medien und Zugabe von warmen hESC Medien mit 10 & mgr; Y-27632 ergänzt.
- Am Tag der Gefrierpunkt vorzubereiten jede 0,5 ml Lösung A und Lösung B pro Vertiefung einer 12-Well-Platte, die sich in zwei 15 ml konischen Röhrchen eingefroren wird. Ort-Lösungen auf Eis. Lösung A: 50% hESC Medien und 50% FBS; Lösung B: 80% FBS und 20% Dimethylsulfoxid (DMSO).
- Distanzieren HPSC Kolonien in einzelne Zellen vor dem Einfrieren. Führen Sie die Schritte 2,2 bis 2,8, dies zu tun, Skalierung Volumen entsprechend. Komplett resuspendieren Zellpellet in 0,5 ml Lösung A.
- 0,5 ml Lösung B zu Zellsuspension, Pipette nach oben und unten Zellsuspension homogen zu machen. Take das Gesamtvolumen der Zellsuspension (~ 1 ml) und Einlage in eine 2 ml Kryoröhrchen. Schraubverschluss fest zu.
- Platz Kryoröhrchen in einem -80 ° C Gefrierschrank O / N. Der Tag nach dem Einfrieren, entfernen gefrorenen Zellen von -80 ° C Gefrierschrank und sofort in flüssigem Stickstoff Tank für langfristige Lagerung zu geben.
Ergebnisse
Hier zeigen wir ein Protokoll mit drei verschiedenen SSN-Plattformen, um gentechnisch HPSC Linien schaffen kompatibel. Wir gezielte WIBR # 3 humanen embryonalen Stammzellen am AAVS1 Locus mit früher veröffentlichten ZFNs 4, 5 und Talens CRISPR / Cas9s 37 mit Hilfe eines Reparatur-Vorlage, die eine EGFP Reporter und ein Puromycin-Resistenz-Kassette 4 führt.
Wir kultiviert unseren hPSCs auf MEFs für eine Zellkultur-Workflow ermöglicht den Erhalt und Ausbau der undifferenzierten hPSCs (Abbildung 2), das ist auch kostengünstig und skalierbar. Wenn Zellen länger als notwendig geworden besteht ein Risiko erhöhter Differenzierung, die Verringerung der Anzahl von pluripotenten Zellen, die transfiziert sind und folglich die Anzahl der korrekt ausgerichtet HPSC Kolonien erhalten.
Wir electroporated 5,0 x 10 6 Zellen pro SSN Plattform und plattiert die Zellen aus jeder Ausrichtung auf einer einzelnen Platte mit 6 Vertiefungen von DR4 MEFs. Nach Auswahl der gewünschten Folge jede Plattform in EGFP-positiven Kolonien (Figur 3) und eine Kombination von ungezielte, homozygot gezielte und heterozygot gezielte Klone (4A, B; Tabelle 3). Unter den hier vorgestellten Bedingungen, so finden wir, daß die AAVS1 TALENS ergaben den EGFP-positiven Klone. Die Reparaturschablone für dieses Experiment verwendet wird, besteht aus einer Spleißakzeptorstelle stromaufwärts von der EGFP Reporter und Puromycin-Resistenz-Kassette. Mit diesen "gene-trap" -Strategie (Abbildung 1), sollte das Konstrukt in das erste Intron des AAVS1 Locus einzufügen, mit dem endogenen Promotor die Expression des Puromycin-Resistenz-Kassette zu fahren. Der Mangel an einem Promotor, der die Expression des Puromycin-Resistenzgen im Reparatur Vorlage sollte die Expression in die verhindern Falle der zufälligen Integration.
Daher erwarten wir, dass alle Puromycin-resistenten Klone würde am AAVS1 Website ausgerichtet werden. Es sei darauf hingewiesen, dass eine Untergruppe von Klonen führen aberrant Integrationen im AAVS1 Locus, durch Southern-Blot unter Verwendung einer internen Sonde nachweisbar sind, aber nicht durch die meisten PCR-Strategien (1; 4C) 4. Diese Integrationsereignisse sind wahrscheinlich das Ergebnis von heterologen Targeting-Ereignisse, die zu mehreren Integrationen des Donorplasmids 4. Wir wählten 24 Kolonien von jeder SSN Experiment und festgestellt, dass alle Plattformen hatte sehr hohe Wirkungsgrade Targeting und zeigte nur geringe Unterschiede. Wie durch PCR verhört, ergab CRISPR / Cas9 die meisten korrekt ausgerichtet, während die Klone TALEN Plattform hatte die meisten homozygot gezielte Klone (Tabelle 3).
/files/ftp_upload/53583/53583fig1highres.jpg "width =" 700 "/>
Abbildung 1. Schematische des Gens bearbeitet AAVS1 Locus mit dem AAV-CAGGS-EGFP-Reparatur-Vorlage. Geändert von Hockemeyer et al., 2009 Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
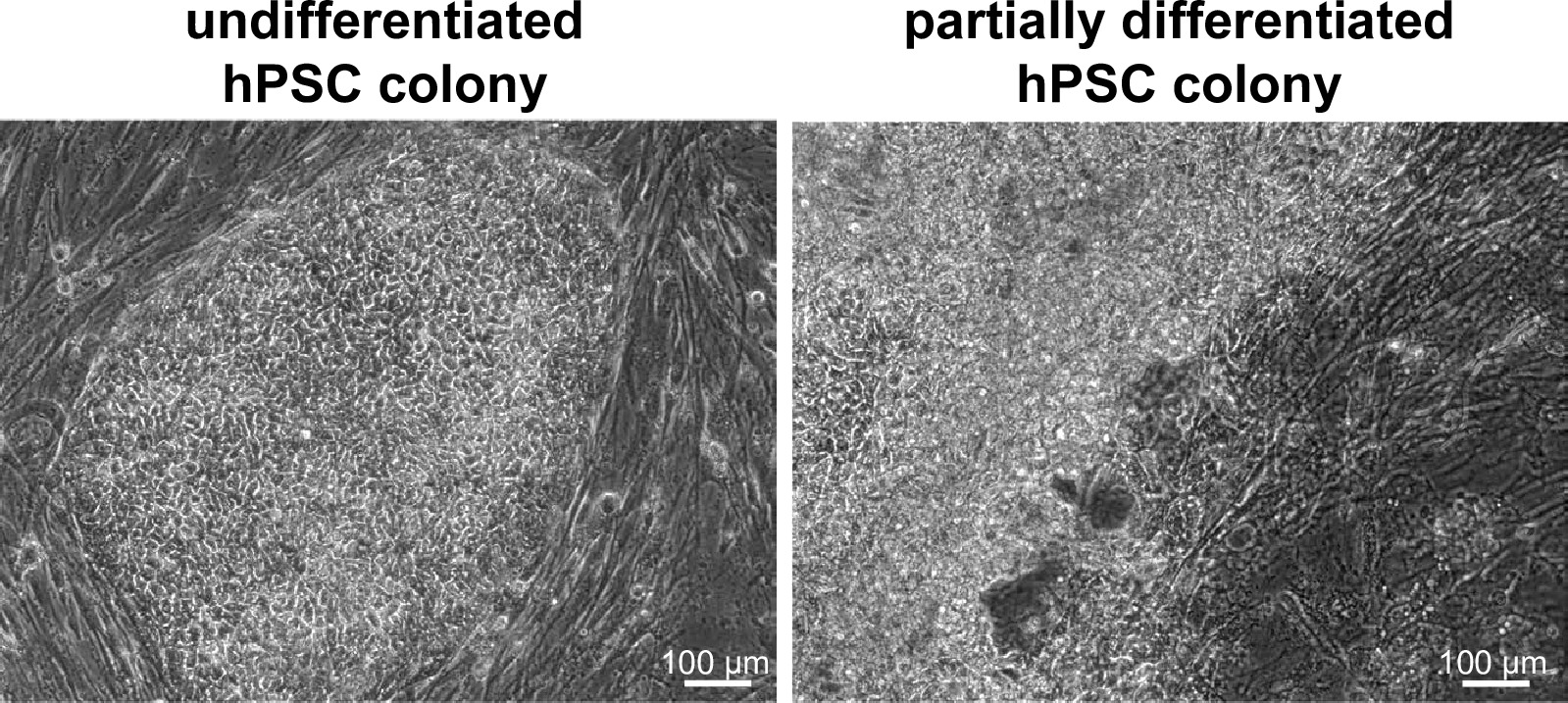
Abbildung 2. Kolonien von WIBR # 3-Zellen. Repräsentative Hellfeldbilder von Kolonien von WIBR # 3 humanen embryonalen Stammzellen vor Targeting. Beachten Sie die fehlende Differenzierung und die klare Trennung von der Feeder-Schicht in einer idealen Kolonie (links) im Vergleich zu einer nicht-idealen (rechts). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
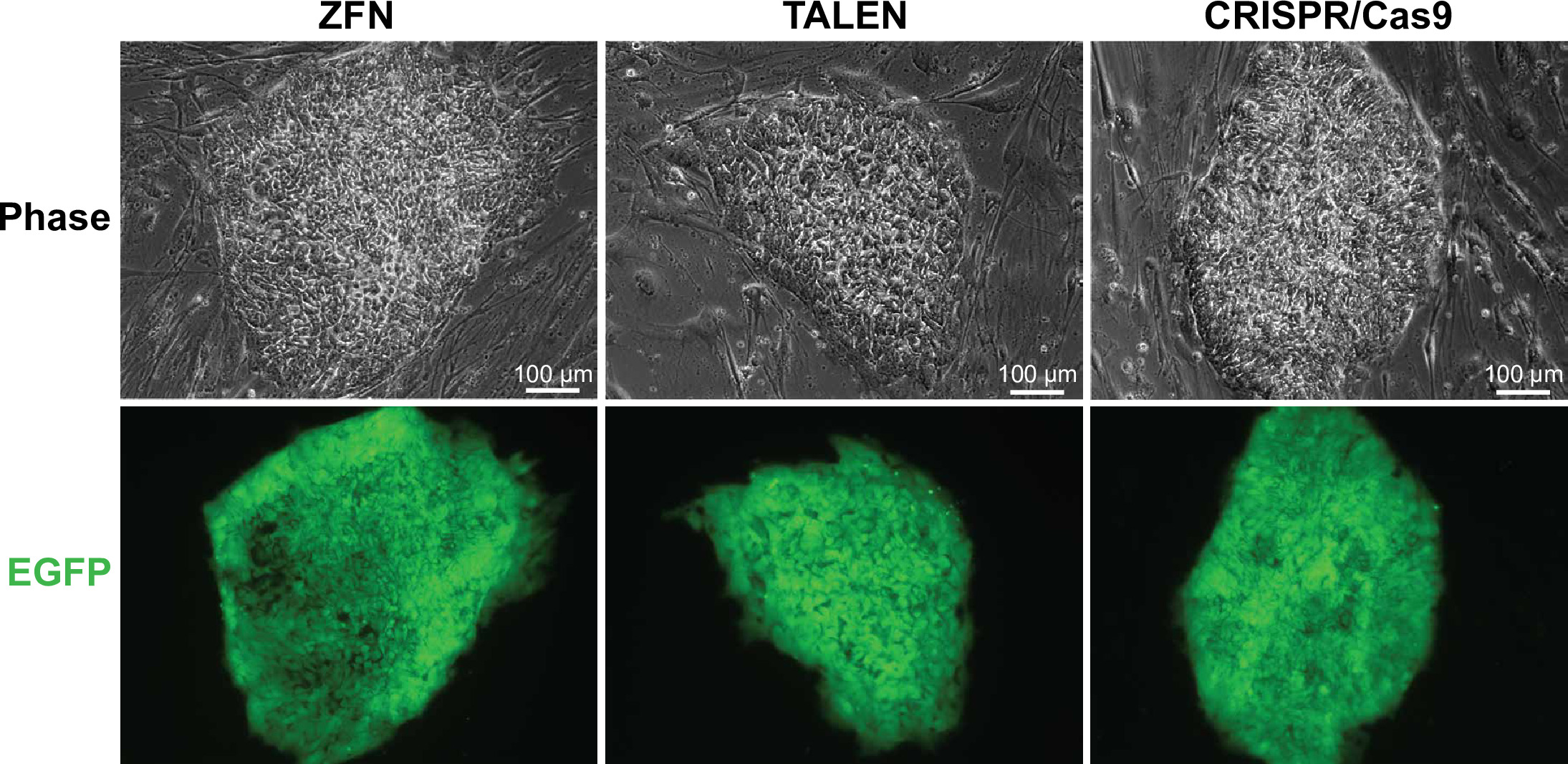
Abbildung 3. EGFP-positive WIBR # 3-Zellen. Repräsentative Bilder der WIBR # 3-Zellen mit einem EGFP-exprimierenden Reparatur Vorlage am AAVS1 Locus ausgerichtet. Bilder repräsentativer Kolonien mit ZFNs editiert, Talens und CRISPR / Cas9 angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
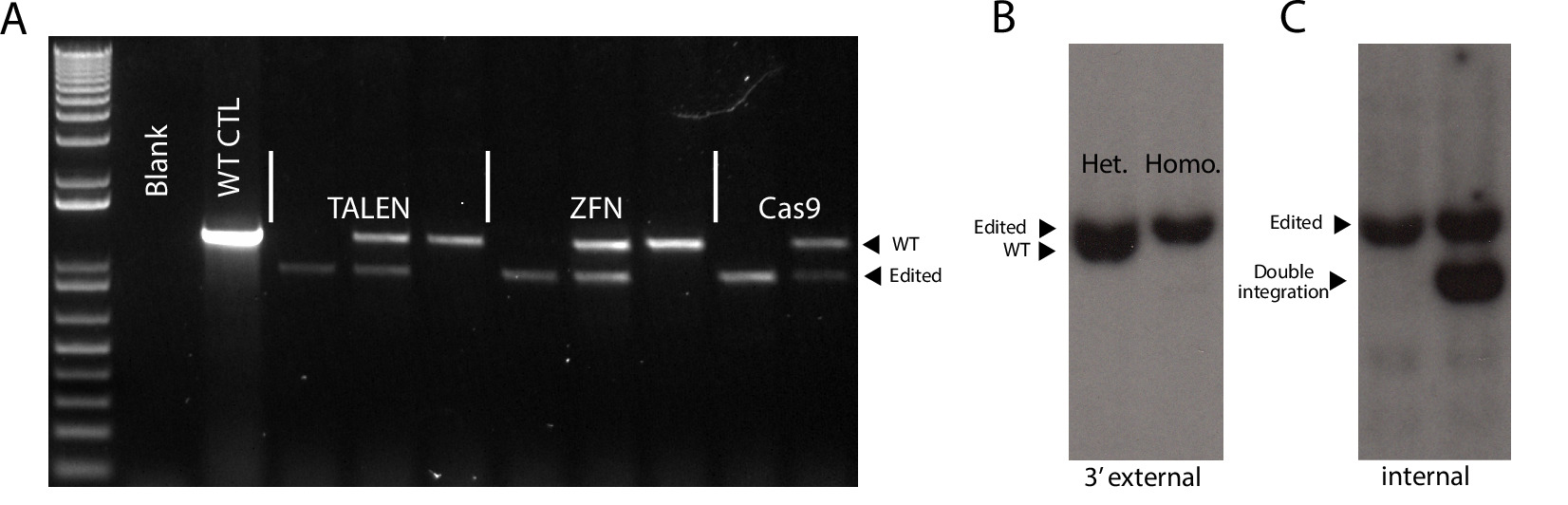
Abbildung 4. Genotypisierung Strategien korrekte Ausrichtung zu bestätigen. (A) Repräsentative PCR Genotypisierung Ergebnisse zeigen ungezielte, heterozygote und homozygote gezielte Klone über die drei SSN-Plattformen. WT CTL = Wildtyp-Kontrolle. (B) Repretreter Southern-Blot-Ergebnisse, die eine heterozygote gezielte Klon und eine homozygote mit einem 3 'externe Sonde nachgewiesen gezielte Klon. Fragmentgrößen: WT-6,5 kb, Bearbeitet-6,9 kb. (C) Repräsentative Southern-Blot-Ergebnisse, die einen richtig bearbeitet Klon und eine heterozygote Klon mit einer nicht-zufälligen Doppel Integration. Fragmentgrößen:. Richtig bearbeitet-6,9 kb, anomale zusätzliche Integration-5 kb Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
| Studie | Gene Gezielte | Plattform | Reparatur Vorlagentyp | Anzahl Klone gepickt | Targeting-Effizienz |
| Sexton et al., 2014 | TPP1 | ZFN | GFP-Puro | nicht gemeldet | nicht gemeldet |
| Sexton et al., 2014 | TERT | ZFN | Hygromycin | nicht gemeldet | nicht gemeldet |
| Hockemeyer et al., 2009 | POU5F1 | ZFN | GFP-Puro | 31 | 39,0% |
| Hockemeyer et al., 2009 | PITX3 | ZFN | GFP-Puro | 74 | 14,9% |
| Hockemeyer et al., 2011, | POU5F1 | TALEN | GFP-Puro | 68 | 91,0% |
| Hockemeyer et al., 2011, | PITX3 | TALEN | GFP-Puro | 96 | 13,0% |
| Merkle et al., 2015 | VASA | Cas9 | Reporter-Geneticin | 139 | 94,0% |
| Merkle et al. </ em> 2015 | CRH | Cas9 | Reporter-Geneticin | 30 | 9.30% |
| Merkle et al., 2015 | HCRT | Cas9 | Reporter-Geneticin | 154 | 92,0% |
| Merkle et al., 2015 | HMX2 | Cas9 | Reporter-Geneticin | 11 | 45,0% |
| Forster et al., 2014 | LRG5-Nterm | ZFN | GFP-Puro | nicht gemeldet | 30,0% |
| Forster et al., 2014 | LRG5-Cterm | ZFN | GFP-Puro | nicht gemeldet | 14,0% |
| Soldner et al., 2011, | SNCA | ZFN | Puro | 96 | 1,0% |
Tabelle 1. Beschreibung von anderen Genen gezielte Verwendung dieser Methode mit correchenden Zielwirkungsgrade von bisher veröffentlichten Studien entnommen. 4,5,38-41
| Ort | Sequenz | Hinweise |
| AAVS1-F-Primer | CTCTAACGCTGCCGTCTCTC | PCR-Bedingungen: T m = 57 ° C, 35 Zyklen |
| AAVS1-WT-R Primer | GCTTCTCCTCTTGGGAAGTG | WT-Band: 1273 bp |
| AAVS1-Targeted-R Primer | CGTCACCGCATGTTAGAAGA | Gezielte Band: 992 bp |
| T2-Cas9-Führer | GGGCCACTAGGGACAGGAT | Mali et al., 2013 |
| AAVS1-ZFN-Rechts | TAGGGACAGGAT | Von Hockemeyer et al., 2009 |
| AAVS1-ZFN-Linker | TGGGGTGTCACC | Von Hockemeyer et al., 2009 |
| AAVS1-TALEN-Rechts | TCCTAACCACTGTCTTT | Aus Hockemeyer et al., 2011, |
| AAVS1-TALEN-Linker | CCCCTCCACCCCACAGT | Aus Hockemeyer et al., 2011, |
Tabelle 2. Liste der Primer und SSN Targeting-Sequenzen.
| Targeting-Konstrukt | Anzahl von EGFP + Kolonien | Gezielte abgeholt Klone (PCR überprüft) | Zurück Berichtet Correct Targeting Effizienz (durch Southern-Blot) |
| ZFN | 150 | 86,9% (73,9% het / 13,0% homo) | 56% (50% het / 6% homo) |
| TALEN | 412 | 91,3% (47,8% het / 39,1% homo) | 47% (37,5% het / 9,3% homo) |
| CRISPR-Cas9 | 235 | 95,7% (69,5% het / 26,3% homo) | nicht gemeldet |
Tabelle 3 Vergleichszahlen EGFP-positive und ziel TALEN, ZFN und CRISPR / Cas9 menschlichen Stammzellenkolonien. PCR verifiziert Integrationen am AAVS1 Locus für dieses Experiment sind Southern-Blot verifiziert korrekte Einzel Integrationen in früheren Experimenten 4,5 verglichen.
Diskussion
Die zur Isolierung homogene Populationen von Gen-bearbeitet menschlichen pluripotenten Stammzellen hier vorgestellte Methode ist ein leistungsfähiger Ansatz zur Erzeugung von isogenen HPSC Linien, die nur an der geplanten Ortskurve abweichen. Diese Zellen sind ein ideales System für die Erforschung der Mechanismen des menschlichen zellulären Differenzierung und Entwicklung als auch für das Verständnis der Pathophysiologie von monogenen Erkrankungen, in einer kontrollierten genetischen Umgebung. Wie hier gezeigt, ist es möglich, drei unabhängige SSN Designstrategien (ZFNs, Talens, und CRISPR / Cas9) verwenden, um gezielte Integration an der AAVS1 Locus zu erreichen. Jede dieser Methoden hat seine eigenen Vorteile und Nachteile. Ein potentieller Vorteil der ZFNs und zu einem gewissen Grad ist TALENS ihre Designflexibilität, die iterative Technik, um die DNA-Bindungsdomänen von Einzel Nukleasen 42 verbessern kann. Diese Nuklease-Optimierung konnte die Spezifität der ZFNs und Talens hinaus, was ist erreichbar mit dem CRI erhöhenSPR / Cas9 Systems. Solche Selektivität kann wichtig für klinische Anwendungen, die einen hohen Grad an Zielspezifität. Der Hauptvorteil des CRISPR / Cas9 Systems ist seine Benutzerfreundlichkeit. Obwohl TALEN und ZFN Baukästen wurden der Öffentlichkeit zur Verfügung (dh durch Addgene 21) gemacht, sind CRISPR / Cas9 basierten Sozialversicherungsnummern deutlich einfacher zu konstruieren, als die einzige notwendige Anpassung ist eine 20-Basenpaar-Oligonukleotid (bei Verwendung des px330 Plasmid Design-14). Diese Einfachheit erweist für Forschungslabors suchen, um Genom Editieren in ihren Studien sind vorteilhaft.
Es gibt alternative Transfektionstechniken, einschließlich Nukleofektion 43, um Gen-bearbeitet HPSC Linien zu schaffen; jedoch Elektroporation hat sich gezeigt, konsistente und kostengünstige 4,5,25 sein. Nukleofektion kann verwendet werden, um direkt zu transfizieren Cas9-Führungs RNA Ribonukleoproteinkomplexe in den Zellkern, die Erhöhung der Effizienz SSN eind Treue 44. Wachsenden hPSCs auf MEFs ist ein robustes und kostengünstiges Verfahren zur hPSCs in einem pluripotenten Zustand ohne übermäßige Differenzierung aufrecht zu erhalten. Darüber hinaus erlaubt es die einfache Trennung von genetisch identischen Kolonien. Alternativ ist es möglich, ohne Kultur hPSCs MEFs jedoch diesen Kulturbedingungen können teurer als feeder basierend Kulturen. Darüber hinaus ist das ganze Verfahren skalierbar, so dass für die Isolierung von sehr seltenen Bearbeitungs Ereignisse oder die Erzeugung von vielen verschiedenen Zelllinien in parallele Bearbeitung Experimenten.
Das hier beschriebene Protokoll ist robust; jedoch gibt es mehrere wichtige Schritte, die den Wirkungsgrad, mit dem korrekt bearbeitet Klone erhalten werden beeinflussen. Die kritischste Komponente für diese Methode ist mit hoher Qualität MEFs und arzneimittelresistente DR4 MEFs. Das Überleben der einzelnen hPSCs ist dürftig, und von geringer Qualität MEFs wird die Isolierung von undifferenzierten HPSC Linien zu behindern. Zweitens ist auch die Verwendung von Y-27632Schlüssel zur Einzelzellüberleben, ohne einen Selektionsdruck für Zellen mit einer abnormen Karyotyp 45 zu ermöglichen. Drittens, Kommissionierung gut verteilte Kolonien sichergestellt, genetische Homogenität der Zelllinien. Schließlich ist es wichtig, Glaspipetten vorzubereiten, so dass die Öffnung klein genug, um die Kolonie in viele kleinere Stücke brechen ist, sicherzustellen, dass mehrere Kolonien in der neuen gut zu wachsen. Dies ermöglicht die Kommissionierung von gut verteilte Subklone für eine Replik Platte in Kultur haben, so dass die Originalplatte isolierte Kolonien für die Genotypisierung. Eine anspruchsvolle Rolle in diesem Protokoll ist das manuelle Ziehen der Glaspipetten; Dies sollte im Vorfeld geübt werden. Es sei darauf hingewiesen, dass es mehrere Techniken des Klons Kommissionierung, die nicht einer Glaspipette erfordern werden. Experimentatoren sind aufgefordert, die eine, die für sie am besten funktioniert.
Gibt es Einschränkungen bei diesem Protokoll, das von einfachen Modifikationen überwunden werden können. Ein Experiment, um o bestimmtnly stören den Locus von Interesse, ohne Reparatur oder eine, deren Reparatur Vorlage keinen Selektionskassette enthält, muss ein anderes Verfahren zum Anreichern von Zellen, die bearbeitet wurden, zu verwenden. Man beachte, dass die Zahl der Kolonien, die aufgenommen werden müssen, um einen positiven Klon zu finden stark erhöht in Abwesenheit der Selektion. Eine Strategie, um die Effizienz zu verbessern, ist Cotransfektion eines nicht-integrierenden Plasmid, das für ein fluoreszierendes Protein exprimiert. Nachdem man die Zellen für zwei Tage erholen können Zielzellen auf positive Fluoreszenz durch Fluoreszenz-aktivierte Zellsortierung sortiert und wieder ausplattiert 29. Dieses Verfahren bereichert für Zellen, die mit den Plasmiden durch Elektroporation transfiziert wurden, und erhöht die Wahrscheinlichkeit einer gleichzeitigen Bearbeitung Ereignis in der Zelle, wodurch.
Die hier beschriebenen Methoden kann erweitert werden, um mehrere Führungs RNAs verwenden, um gleichzeitig mehrere Ziel loci 14,46,47. Viele Protokolle wurden eingerichtet, um hPSCs unterscheidenin bestimmte Zelltypen, wodurch verschiedene genetische Manipulationen in Zelltypen von Interesse 30. Insgesamt haben wir das Potenzial für die effiziente Bearbeitung im Genom hPSCs unabhängig von SSN Wahl demonstriert. Wir schlagen vor, dass diese Technik angepasst, um isogene HPSC Linien jeder genomischen Locus, die Gen-bearbeitet haben zu schaffen.
Offenlegungen
Die Autoren erklären, keine konkurrierenden finanziellen Interessen.
Danksagungen
Diese Arbeit wurde von einem Brain Research Foundation Seed Grant (BRFSG-2014-02), um Helen Bateup unterstützt. Dirk Hockemeyer ist ein New Scholar in Altern der Ellison Medical Foundation und wird von der Glenn-Stiftung sowie der The Shurl und Kay Curci Stiftung. DH wird auch von NIH Zuschusses 1R01CA196884-01 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Life Technologies | 11320082 | |
| Fetal Bovine Serum (HI) | Life Technologies | 10082-147 | |
| Knockout Serum | Life Technologies | 10828-028 | |
| Fibroblast Growth Factor - basic | Life Technologies | PHG0261 | |
| Pen/Strep | Life Technologies | 15140-122 | |
| Glutamine | Life Technologies | 25030-081 | |
| MEM NEAA | Life Technologies | 11140-050 | |
| 2-mercaptoethanol | Life Technologies | 21985-023 | |
| Y-27632 | Calbiochem | 688000 | |
| 6-well plates | Corning | 3506 | |
| 12-well plates | Corning | 3512 | |
| 4 mm Electroporation cuvettes | Bio-rad | 165-2081 | |
| X-cel gene pulser II | Bio-rad | 165-2661 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| 10× Phosphate buffered saline (PBS) pH7.4 | Life Technologies | 70011-044 | |
| Puromycin | Life Technologies | A11138-02 | |
| Pasteur pipettes, plugged | VWR | 14672-412 | |
| Tris-HCl | Sigma-Aldrich | S5941 | |
| NaCl | Sigma-Aldrich | S9888 | |
| SDS | Sigma-Aldrich | L3771 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Proteinase-K | Life Technologies | AM2544 | |
| Ethanol | VWR | TX89125-172SFU | |
| Isopropyl Alcohol | VWR | MK303216 | |
| TE Buffer | Life Technologies | 12090-015 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| 1.8 ml Cryotubes | ThermoScientific | 377267 |
Referenzen
- Carroll, D. Genome engineering with targetable nucleases. Annu Rev Biochem. 83, 409-439 (2014).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Matsa, E., Burridge, P. W., Wu, J. C. Human stem cells for modeling heart disease and for drug discovery. Sci Transl Med. 6 (239), 239ps6 (2014).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat Biotechnol. 27 (9), 851-857 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol. 29 (8), 731-734 (2011).
- Bozas, A., Beumer, K. J., Trautman, J. K., Carroll, D. Genetic analysis of zinc-finger nuclease-induced gene targeting in drosophila. Genetics. 182 (3), 641-651 (2009).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Urnov, F. D., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 435 (7042), 646-651 (2005).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jinek, M., East, A., Cheng, A., Lin, S., Ma, E., Doudna, J. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. the new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/cas systems. Science. 339 (6121), 819-823 (2013).
- Jasin, M. Genetic manipulation of genomes with rare-cutting endonucleases. Trends Genet. 12 (6), 224-228 (1996).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 11 (9), 636-646 (2010).
- Sander, J. D., Maeder, M. L., Reyon, D., Voytas, D. F., Joung, J. K., Dobbs, D. ZiFiT (zinc finger targeter): An updated zinc finger engineering tool. Nucleic Acids Res. 38, W462-W468 (2010).
- Carroll, D., Morton, J. J., Beumer, K. J., Segal, D. J. Design, construction and in vitro testing of zinc finger nucleases. Nat Protoc. 1 (3), 1329-1341 (2006).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39 (12), e82 (2011).
- Reyon, D., Tsai, S. Q., Khayter, C., Foden, J. A., Sander, J. D., Joung, J. K. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol. 30 (5), 460-465 (2012).
- Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F. A transcription activator-like effector toolbox for genome engineering. Nat Protoc. 7 (1), 171-192 (2012).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from neisseria meningitidis. Proc Natl Acad Sci U S A. 110 (39), 15644-15649 (2013).
- Podhajska, A. J., Szybalski, W. Conversion of the FokI endonuclease to a universal restriction enzyme: Cleavage of phage M13mp7 DNA at predetermined sites. Gene. 40 (2-3), 175-182 (1985).
- Costa, M., et al. A method for genetic modification of human embryonic stem cells using electroporation. Nat Protoc. 2 (4), 792-796 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Cai, J., et al. Directed differentiation of human embryonic stem cells into functional hepatic cells. Hepatology. 45 (5), 1229-1239 (2007).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Soldner, F., et al. Parkinson's disease patient-derived induced pluripotent stem cells free of viral reprogramming factors. Cell. 136 (5), 964-977 (2009).
- Wen, Z., et al. Synaptic dysregulation in a human iPS cell model of mental disorders. Nature. 515 (7527), 414-418 (2014).
- Chiba, K., Hockemeyer, D. Genome editing in human pluripotent stem cells using site-specific nucleases. Methods Mol Biol. 1239, 267-280 (2015).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. J Vis Exp. , (2012).
- Ezashi, T., Das, P., Roberts, R. M. Low O2 tensions and the prevention of differentiation of hES cells. Proc Natl Acad Sci U S A. 102 (13), 4783-4788 (2005).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25 (18), 3745-3746 (1997).
- Southern, E. Southern blotting. Nat Protoc. 1 (2), 518-525 (2006).
- Henegariu, O., Heerema, N. A., Dlouhy, S. R., Vance, G. H., Vogt, P. H. Multiplex PCR: Critical parameters and step-by-step protocol. BioTechniques. 23 (3), 504-511 (1997).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Sexton, A. N., et al. Genetic and molecular identification of three human TPP1 functions in telomerase action: Recruitment, activation, and homeostasis set point regulation. Genes Dev. 28 (17), 1885-1899 (2014).
- Merkle, F. T., et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus. Cell Rep. 11 (6), 875-883 (2015).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Forster, R., et al. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2 (6), 838-852 (2014).
- Miller, J. C., et al. Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nat Methods. , (2015).
- Hohenstein, K. A., Pyle, A. D., Chern, J. Y., Lock, L. F., Donovan, P. J. Nucleofection mediates high-efficiency stable gene knockdown and transgene expression in human embryonic stem cells. Stem Cells. 26 (6), 1436-1443 (2008).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat Biotechnol. 25 (6), 681-686 (2007).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4, 5400 (2014).
- Ousterout, D. G., Kabadi, A. M., Thakore, P. I., Majoros, W. H., Reddy, T. E., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy. Nat Commun. 6, 6244 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten