Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verfahren für Adaptive Labor Entwicklung von Mikroorganismen Mit einem Chemostat
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
mit Chemostatkultur Hier präsentieren wir ein Protokoll adaptive gerichtete Evolution von Mikroorganismen unter Bedingungen zu erhalten. Auch genomische Analyse des entwickelten Stamm wird diskutiert.
Zusammenfassung
Natural evolution involves genetic diversity such as environmental change and a selection between small populations. Adaptive laboratory evolution (ALE) refers to the experimental situation in which evolution is observed using living organisms under controlled conditions and stressors; organisms are thereby artificially forced to make evolutionary changes. Microorganisms are subject to a variety of stressors in the environment and are capable of regulating certain stress-inducible proteins to increase their chances of survival. Naturally occurring spontaneous mutations bring about changes in a microorganism's genome that affect its chances of survival. Long-term exposure to chemostat culture provokes an accumulation of spontaneous mutations and renders the most adaptable strain dominant. Compared to the colony transfer and serial transfer methods, chemostat culture entails the highest number of cell divisions and, therefore, the highest number of diverse populations. Although chemostat culture for ALE requires more complicated culture devices, it is less labor intensive once the operation begins. Comparative genomic and transcriptome analyses of the adapted strain provide evolutionary clues as to how the stressors contribute to mutations that overcome the stress. The goal of the current paper is to bring about accelerated evolution of microorganisms under controlled laboratory conditions.
Einleitung
Mikroorganismen können auf unterschiedliche Umgebungen überleben und sich anzupassen. Unter starkem Stress, Anpassung kann 1-3 über Erwerb von positiven Phänotypen durch statistische genomische Mutationen und anschließende positive Selektion auftreten. Daher kann mikrobiellen Zellen anzupassen durch metabolische oder regulatorische Netzwerke für ein optimales Wachstum zu ändern, die "adaptive Evolution" bezeichnet wird. Vor kurzem sind wichtige mikrobielle Tendenzen, wie Ausbrüche von Superbugs und dem Auftreten von robusten mikrobielle Stämme, sind sehr eng miteinander verbunden Evolution unter Stressbedingungen zu adaptiv. Unter definierten Laborbedingungen, sind wir die Mechanismen der molekularen Evolution zu studieren können und sogar die Richtung der mikrobiellen Evolution für verschiedene Anwendungen zu steuern. Im Gegensatz zu mehrzelligen Organismen sind einzellige Organismen gut geeignet, um adaptive Labor Entwicklung (ALE) aus den folgenden Gründen: sie regenerieren schnell, sie pflegen große Populationen, und es ist leicht hom zu erstellen und zu pflegenogeneous Umgebungen. In Kombination mit den jüngsten Fortschritten in der DNA-Sequenzierungstechniken und Hochdurchsatz-Technologien, ermöglicht ALE für die direkte Beobachtung von genomischen Veränderungen, die zu einer systemischen regulatorischen Veränderungen führen. Mutations Dynamik und Vielfalt der Bevölkerung sind auch zu beobachten. Die Gentechnik - Strategien können aus der Analyse der ALE - Stämme 4,5 bestimmt werden.
Chemostatkultur ist eine Methode verwendet Steady-State - Zellen zu erhalten und die Produktivität bei Fermentationsprozessen 6 erhöhen. Frisches Medium wird zugesetzt und Kulturbrühe wird während des Verfahrens geerntet (letzteres enthält Medium und Biomasse). Langzeit Chemostat - Kultur, ändert jedoch den steady-state Produktivität der Kultur und bewirkt die Anhäufung von spontanen Mutationen und Selektion während der Kultivierung (Abbildung 1a). Unter verschiedenen Selektionsdruck (Stressoren), wird die Akkumulation von Mutationen verbessert. Eine allmähliche Zunahme der Spannung in einem langfristigen Chemostat sorgt für eine kontinuierliche Auswahl von Mutationen , die gegen den gegebenen Stressoren arbeiten, wie beispielsweise Temperatur, pH, osmotischer Druck, Nährstoffmangel, Oxidation, toxische Endprodukte usw. Colony Übertragung von einem festen Medium und serielle Übertragung aus einem flüssigen Medium (wiederholte Batch - Kultur) erlauben auch Forscher entwickelten Mikroorganismen (Abbildung 1b und 1c) zu erhalten. Obwohl Chemostatkultur komplizierte Methoden erfordert, ist der Pool der Vielfalt (Anzahl der Replikationen und Populationsgröße) höher als die durch Kolonie-Transfer und serielle Übertragungstechniken erhalten. Die stabile Stressbelastung auf einzelne Zellen und verringerte Veränderung der zellulären Zustand während Chemostatkultur (Steady State) sind weitere Vorteile von ALE im Vergleich zu Batch-Kultur-basierte Techniken. Stress-induzierten ALE von Escherichia coli zu hoher Succinat Bedingungen unterworfen wird in diesem Artikel eingeführt.
iles / ftp_upload / 54446 / 54446fig1.jpg "/>
Abbildung 1: Methoden der adaptiven Labor Evolution (A) Chemostat;. (B) eine serielle Übertragung; (C) Kolonie übertragen. Die Top - Zahlen das Konzept der Methoden für das ALE illustrieren, und die unteren Zahlen die Anzahl der Zellen darstellen , die während ALE wuchs. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Protokoll
1. Vorbereitung der Ausrüstung
- Besorgen Sie sich ein Chemostat Glas (150-250 ml) oder einem Erlenmeyerkolben (250 ml) mit einer Einlassöffnung und eine Austrittsöffnung enthält. Verbinden Sie die Anschlüsse mit Silikonschlauch so dass für Flussraten von 10 bis 100 ml / h. Optional kann ein Entlüftungsventil, eine Luftaustrittsöffnung, und temperiertes Wasser Einlass- und Auslasskanäle verwenden.
- Besorgen Sie sich eine Vorrichtung, die geeignet für die chemostat Glas, die für das Rühren und Temperaturkontrolle bietet (oder einen Dreh Schüttelinkubator verwenden).
- Erhalten zwei peristaltische Pumpen, um frisches Medium zu liefern und die Kultur zu sammeln.
- Besorgen Sie sich ein Reservoir Glas (10-20 L) eine mittlere Austrittsöffnung und eine Lufteinlassöffnung enthält.
- Erhalten Silikonschlauch geeignet für die Verdünnungsrate (dh ID 0,8 mm, Durchflussbereich von 0,06 bis 36 ml / min; L / S 13 Schläuche).
2. Medium Vorbereitung und Sterilisation
- Initial Medium
- Man löst 0,3 g Glucose, 0,08 g NH 4Cl, 0,05 g NaCl, 0,75 g Na 2 HPO 4 · 2H 2 O und 0,3 g KH 2 PO 4 in 90 ml destilliertem Wasser (DW) in einem Chemostat jar.
- Seal die chemostat Glas zusammen mit dem Schlauch mit Schellen. Sie nicht die Lüftungsöffnung abzudichten.
- Sterilisieren Sie die chemostat Glas in einem Autoklaven bei 121 ° C für 15 min. Nach der Sterilisation, speichern Sie die chemostat Glas bei Raumtemperatur.
- Man löst 0,02 g MgSO 4 · 7H 2 O, 0,01 g CaCl 2 und 0,1 mg Thiamin in 10 ml DW (Lösung A).
- Filterlösung A eine Spritze und eine vorge sterilisierten Spritzenfilter unter Verwendung von (a 0,45 um Porenfilter).
- Fügen Sie die Lösung A Filtrate zum Chemostat Glas.
- Stress-Medium
- Man löst 30 g Glucose, 8 g NH 4 Cl, 5 g NaCl, 75 g Na 2 HPO 4 · 2H 2 O, 30 g KH 2 PO 4 und 300 g Dinatriumsuccinat Hexahydrat (Na 2 · Succinat · 6H 2 O; der Stressor in diesem Experiment verwendet) in 9,9 L DW in einem Reservoir jar.
- Verschließen Sie den Behälter Glas zusammen mit dem Schlauch mit Schellen. Sie nicht die Lüftungsöffnung abzudichten.
- Sterilisieren des Behälters jar in einem Autoklaven bei 121 ° C für 15 min. Nach der Sterilisation, speichern Sie das Gefäß bei Raumtemperatur.
- Man löst 2 g MgSO 4 · 7H 2 O, 1 g CaCl 2 und 10 mg Thiamin in 100 ml DW (Lösung A).
- Filter Lösung A mit einer Spritze und einem vorher sterilisierten Spritzenfilter (0,45 & mgr; m ein Porenfilter).
- Fügen Sie die Lösung A Filtrate zum Reservoir Glas.
- Aseptisch schließen Sie das sterilisierte Silikonschlauch mit dem Reservoir Glas und die peristaltische Pumpen befestigen.
- High-Stress-Mittel
- Bereiten Sie das Medium , wie in Abschnitt 2.2, aber mit einer größeren Konzentration von Stressor (dh 3-5 g / l höher in der Succinat - Anpassung).
Hinweis: Dieses Protokoll zur Anpassung an eine Stress than kann über das Medium geliefert werden. Im Falle von physikalischen Stressoren, wie Temperatur, Rühren oder Beleuchtung, die Kultivierung sollte entsprechend ausgelegt werden.
- Bereiten Sie das Medium , wie in Abschnitt 2.2, aber mit einer größeren Konzentration von Stressor (dh 3-5 g / l höher in der Succinat - Anpassung).
3. Erste Anbau
- Impfen eine einzige Kolonie von Wildtyp E. coli in einem 15 ml Teströhrchen mit 4 ml des anfänglichen Mediums.
- Inkubieren des Teströhrchens in einem Schüttelinkubator 12 Stunden bei 37 ° C und 220 Umdrehungen pro Minute.
- Aseptisch 1 ml der Vorkultur in Chemostat Glas.
- Inkubieren Sie die chemostat Glas und bietet für die Belüftung (Luft 50 ml / min) und Rühren (200 Umdrehungen pro Minute), bei 37 ° C für 6 Stunden.
4. Stress-Anpassung
- Aseptisch schließen Sie das Ende des Silikonschläuche von den Pumpen zum Chemostat Glas.
- Starten Sie den Ausgang Pumpe (10 ml / h oder höher) und sammeln Kultur.
Hinweis: Die Kultur in der exponentiellen Phase sein sollte, in der Regel 4-8 Stunden nach der anfänglichen Kultivierung. - CHeck der optischen Dichte (600 nm) der Kultur aus dem Auslassschlauch.
- Starten Sie den Zulaufpumpe (10 ml / h, auf eine Rate von Verdünnung von 0,1 h -1 entspricht).
- Überprüfen Sie die optische Dichte der Kultur bei 600 nm von dem Auslass alle 24 h Schläuche.
- Betreiben Sie den chemostat für 96 Stunden (9,6-fache Umsatz) oder mehr. Wenn die optische Dichte stabil ist, tauschen Sie das Reservoir, das den hochbelasteten Medium enthält. Wenn die optische Dichte von weniger als 0,2 ist, stoppen Sie die Fütterung Zulaufpumpe für 6 Stunden. Starten Sie den Zulaufpumpe und prüfen, ob die optische Dichte über 0,2 ist.
- Nach und nach die Konzentration des Stressor erhöhen, indem sie zu einem Reservoir Ändern einer höheren Stressor Konzentration enthält.
- Nehmen Sie Proben der angepassten Kultur , wenn es einen Meilenstein erreicht (zB ein Stamm angepasst bis 100 g / L Succinat Stress) und Speicher für weitere Genomanalyse.
- Für die Probenlagerung, mischen Sie die Kulturprobe (0,5 ml) mit einer sterilisierten 80% Glycerin solutiauf (0,5 ml) und lagern Sie es bei -80 ° C.
Hinweis: Wenn der Mikroorganismus die Fähigkeit erwirbt die Stressoren während der ALE-Verfahren abzubauen, die Stressor Konzentration in der Fermentations Glas wie in der frischen Reservoir ist nicht das Gleiche.
5. Einzelkolonie Isolierung des Stress angepasst Dehnungs
- Bereiten Agarplattenmedium (1,6% Agar), enthaltend die gleichen Stressor und bei der gleichen Konzentration des Mediums.
- Platte die Austritts Kultur (0,1 ml) aus dem Chemostat und inkubieren bei 37 ° C für 16 Stunden.
- Pick einzelne Kolonien von der Platte einen sterilen Zahnstocher und inokulieren sie in 15 ml Teströhrchen das gleiche Stressor enthält, und bei der gleichen mittleren Konzentration wie im Chemostat, und Inkubation für 6 Stunden.
- 1 ml der Kulturbrühe in einen 250 ml Erlenmeyerkolben mit 50 ml Medium enthält. Ernte 0,5 ml der Kulturbrühe alle 1 h und Messung der OD bei 600 nm. Vergleichen Sie die Wachstumsrate des angepassten strain derjenigen des Wildtyp-Stamm der Stressor gegeben.
Ergebnisse
Für High-Succinat Stress Anpassung, die Wildtyp - E coli - W3110 - Stamm war in einem Chemostat bei D kultiviert = 0,1 h -1 für 270 Tage (Abbildung 2).
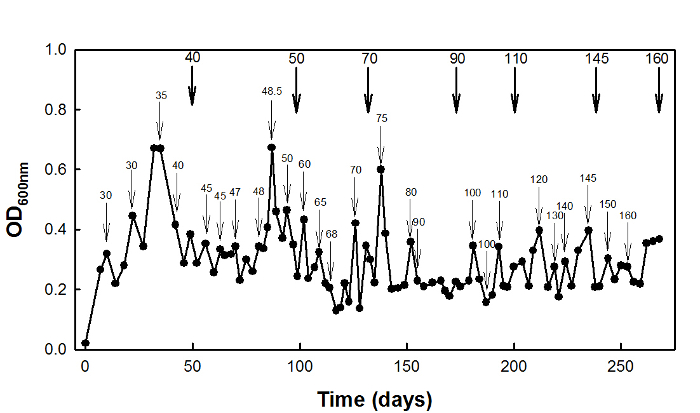
Abbildung 2: High-Succinat Stress Anpassung von E. coli W3110 unter Verwendung Chemostat - Kultur. Dünne Pfeile zeigen die Zeiten , bei denen die Konzentrati...
Diskussion
Mikroorganismen sind in der Lage aufgrund ihrer schnellen Wachstumsrate und der genetischen Vielfalt zu fast allen Umgebungen anzupassen. Adaptive Laborentwicklung ermöglicht Mikroorganismen unter Bedingungen entwickelt, zu entwickeln, die eine Möglichkeit der Auswahl der einzelnen Organismen beherbergt spontane Mutationen bereit, die unter den gegebenen Bedingungen von Vorteil sind.
Die chemostat Technik ist robuster für den folgenden Gründen künstlich angetrieben Evolution als Übertr...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This study was financially supported by the Korean Ministry of Science, ICT and Future Planning (Intelligent Synthetic Biology Center program 2012M3A6A8054887). P. Kim was supported by a fellowship from the Catholic University of Korea (2015).
Materialien
| Name | Company | Catalog Number | Comments |
| Mini-chemostat fermentor | Biotron Inc. | - | manufactured by special order |
| silicon tubing | Cole-Parmer | Masterflex L/S 13 | tubing size can be varied depending on the dilution rate and the size of fermentor jar. |
| reservoir jar | Bellco | Media storage bottle | 20 L |
| chemicals | Sigma-Aldrich | - | reagent grade |
| glucose | Sigma-Aldrich | G5767 | ACS reagent |
| NH4Cl | Sigma-Aldrich | A9434 | for molecular biology, suitable for cell culture, ≥99.5% |
| NaCl | Sigma-Aldrich | 746398 | ACS reagent, ≥99% |
| Na2HPO4·2H2O | Sigma-Aldrich | 4272 | 98.5-101% |
| KH2PO4 | Sigma-Aldrich | 795488 | ACS reagent, ≥99% |
| MgSO4·7H2O | Sigma-Aldrich | 230391 | ACS reagent, ≥98% |
| CaCl2 | Sigma-Aldrich | 793639 | ACS reagent, ≥96% |
| thiamine·HCl | Sigma-Aldrich | T4625 | reagent grade, ≥99% |
| Na2·succinate·6H2O | Sigma-Aldrich | S2378 | ReagentPlus, ≥99% |
Referenzen
- Rando, O. J., Verstrepen, K. J. Timescales of genetic and epigenetic inheritance. Cell. 128, 655-668 (2007).
- Kim, H. J., et al. Short-term differential adaptation to anaerobic stress via genomic mutations by Escherichia coli strains K-12 and B lacking alcohol dehydrogenase. Front Microbiol. 5, 476 (2014).
- Mendizabal, I., Keller, T. E., Zeng, J., Yi, S. V. Epigenetics and evolution. Integr Comp Biol. 54, 31-42 (2014).
- Lee, J. Y., Seo, J., Kim, E. S., Lee, H. S., Kim, P. Adaptive evolution of Corynebacterium glutamicum resistant to oxidative stress and its global gene expression profiling. Biotechnol Lett. 35, 709-717 (2013).
- Lee, J. Y., et al. Artificial oxidative stress-tolerant Corynebacterium glutamicum. AMB Express. 4, 15 (2014).
- Narang, A. The steady states of microbial growth on mixtures of substitutable substrates in a chemostat. J Theor Biol. 190, 241-261 (1998).
- Kwon, Y. D., Kim, S., Lee, S. Y., Kim, P. Long-term continuous adaptation of Escherichia coli to high succinate stress and transcriptome analysis of the tolerant strain. J Biosci Bioeng. 111, 26-30 (2011).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nat Rev Genet. 14, 827-839 (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25, 2078-2079 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten