Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Definieren von Substratspezifität für Lipase und Phospholipase-Kandidaten
In diesem Artikel
Zusammenfassung
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Zusammenfassung
Mikroorganismen produzieren ein breites Spektrum von (phospho) Lipasen, die um sezerniert werden für den Organismus externe Substrate zur Verfügung zu stellen. Alternativ können andere (phospho) Lipasen physikalisch mit dem produzierenden Organismus verursacht einen Umsatz von intrinsischen Lipiden assoziiert sein können und häufig zu einem Remodeling der Zellmembranen führt. Obwohl Potential (phospho) Lipasen mit einer Anzahl von Algorithmen vorhergesagt werden, wenn das Gen / Protein-Sequenz verfügbar ist, experimentelle Nachweis der Enzymaktivitäten, Substratspezifitäten, und potentielle physiologische Funktionen ist häufig nicht erhalten werden. Dieses Manuskript beschreibt die Optimierung der Testbedingungen für Interessenten (phospho) Lipasen mit unbekannten Substratspezifität und wie diese optimierten Bedingungen bei der Suche nach dem natürlichen Substrat eines jeweiligen (phospho) Lipase zu verwenden. Mit künstlichen chromogene Substrate, wie p - Nitrophenyl - Derivate, kann dazu beitragen , eine kleinere zu erkennenenzymatische Aktivität für eine vorhergesagte (phospho) Lipase unter Standardbedingungen. Eine solche geringfügige enzymatische Aktivität angetroffen wird, können die unterschiedlichen Parameter einer Enzymassay, um einen effizienteren Hydrolyse des künstlichen Substrat zu erhalten, variiert werden. Nachdem die Bedingungen arbeitet ein Enzym gut unter denen sie festgestellt haben, sollte eine Vielzahl von potentiellen natürlichen Substrate für deren Abbau getestet werden, ein Prozess, der verschiedene chromatographische Methoden gefolgt Einsatz werden können. Die Definition von Substratspezifitäten für neue Enzyme, liefert oft Hypothesen für eine mögliche physiologische Rolle dieser Enzyme, die dann experimentell überprüft werden kann. Nach diesen Richtlinien, konnten wir eine Phospholipase C (SMc00171) , die Phosphatidylcholin, für den Umbau von Membranen in dem Bakterium Sinorhizobium meliloti auf phosphor limitierenden Bedingungen des Wachstums in einem entscheidenden Schritt phosphocholin und Diacylglycerin verschlechtert zu identifizieren. Für zwei vorhergesagten patatin-wie Phospholipasen (SMc00930 und SMc01003) des gleichen Organismus, könnten wir ihre Substratspezifität neu zu definieren und zu klären, dass SMc01003 ein Diacylglycerol Lipase ist.
Einleitung
Glycerol-basierten Lipiden wie Triglyceriden und (glycero) Phospholipide sind wichtige und wohl die bekanntesten Lipidklassen 1. Triacylglycerolen (TAGs) sind Fette oder Öle, die üblicherweise als Speicherlipiden funktionieren und daher als potentielle Energie und Kohlenstoffquellen. TAGs können durch Lipasen abgebaut werden, die häufig durch den produzierenden Organismus ausgeschieden werden externe TAGs zu verdauen und sie als Kohlenstoffquellen zur Verfügung zu stellen. Auch wurden Lipasen weit über die Jahre studiert aufgrund ihrer wichtigen biotechnologischen Anwendungen 2.
Auf Grund ihrer amphiphilen Natur und ihrer nahezu zylindrischen Form, (glycero) Phospholipide weisen membranbildenden Eigenschaften und in der Regel die Hauptlipidkomponenten einer doppelschichtigen Membran 3 bilden. In einfachen Mikroorganismen wie dem Bakterium Escherichia coli, nur drei Hauptkopfgruppe Varianten, Phosphatidylglycerin (PG), Cardiolipin (CL) und phosphatidylethanolamine (PE) angetroffen werden , obwohl man sich bewusst sein sollte , dass jeder von ihnen kann 4 mit einer beträchtlichen Anzahl unterschiedlicher Fettacylketten an der sn - 1 oder sn - 2 - Position, die zu einer großen Anzahl von verschiedenen molekularen Spezies ersetzt werden . Andere Bakterien könnten haben andere Phospholipide zusätzlich oder statt. Zum Beispiel Sinorhizobium meliloti, ein Bodenbakterium, das mit der Leguminosen Luzerne (Medicago sativa) , um eine Stickstoff-fixierenden Knöllchen Symbiose zu bilden, enthält zusätzlich zu PE ein zweites zwitterionisches Phospholipid, Phosphatidylcholin (PC) 5 in der Lage ist. Auch Lipide nicht, die Phosphor oder Glycerin können amphiphile und bilden einen Teil der Zellmembran sein. Beispielsweise von Phosphor beschränkende Wachstumsbedingungen in S. meliloti, (glycero) Phospholipide sind weitgehend durch Membranlipide ersetzt , die nicht Phosphor enthalten, dh Sulfolipide, Ornithin Lipide und diacylglyceryl trimethylhomoserine (DGTS) 6. In Bakterien ist DGTS von Diacylglycerin (DAG) in einem zweistufigen Weg gebildet 7 , aber die Quelle für DAG Generation war nicht klar. Pulse-Chase - Experimente vorgeschlagen , dass PC ein Vorläufer für 8 DGTS sein könnte und mit Hilfe der Methodik in diesem Manuskript beschrieben wir eine Phospholipase C identifizieren konnte (PLCP, SMc00171) , die unter Phosphor einschränkenden Bedingungen gebildet wird , und die PC in DAG umwandeln und Phosphocholin 8.
In einer separaten Studie entdeckten wir , dass eine Acyl-CoA - Synthetase (FADD) defizienten Mutante von S. meliloti oder von Escherichia coli freien Fettsäuren akkumuliert , wenn 9 stationären Wachstumsphase eintritt. Obwohl diese Fettsäuren schienen von Membranlipiden abgeleitet werden, die genaue Quelle für die freien Fettsäuren oder die Enzym (e) zu befreien, sie waren nicht bekannt. Auch in diesem Manuskript die Strategie skizziert den Einsatz, zwei Patatin-like 10 (phospho) Lipasen (SMc00930 und SMc01003), die zur Bildung von freien Fettsäuren in S. beigetragen meliloti 11 vorhergesagt wurden. Überraschenderweise verwendet SMc01003 DAG als Substrat Umwandlung in Monoacylglycerin und schließlich 11 Glycerin und freien Fettsäuren. Daher ist SMc01003 eine DAG-Lipase (DGLA).
Obwohl eine Reihe von Algorithmen zur Vorhersage Potential (phospho) sind Lipasen 12,13, deren genaue Funktion und physiologische Rolle ist in der Regel nicht bekannt. Hier beschreiben wir ein Protokoll, zu klonen und überexprimiert vorhergesagt oder Potential (phospho) Lipasen. Diese Handschrift wird erläutert, wie Enzymassays können unter Verwendung von künstlichen chromogene Substrate für die überexprimiert (phospho) Lipase entwickelt und optimiert werden. Wir liefern Beispiele, wie mit einem optimierten Enzymtest die reale (phospho) Lipasesubstrats begegnet werden kann und wie diese Erkenntnisse könnten unser Verständnis der mikrobiellen Physiologie bereichern.
Protokoll
1. Klon und überexprimiert Strukturgens für Prognostizierte Lipase
- Unter Verwendung der Polymerasekettenreaktion (PCR) 14 und spezifischen Oligonukleotiden (Tabelle 1) 15, zu amplifizieren , die das Gen von Interesse (smc01003, smc00930 oder smc00171), vorhergesagt für eine Lipase kodieren oder Phospholipase, aus der genomischen DNA des Wirtsorganismus (dh , S. meliloti).
- Einführung spezifischer Restriktionsstellen (mit der entworfenen Sequenz der Oligonukleotide). Digest des amplifizierten DNA - Fragments mit den entsprechenden Restriktionsenzymen und klonieren es in einen Expressionsvektor , wie beispielsweise Plasmide der pET - Serie 16.
- Die korrekte DNA - Sequenz für das geklonte Gen, Transformieren des Vektors in eine Expressionsstamm, wie Escherichia coli BL21 (DE3) pLysS 16 , nachdem geprüft.
- Bereiten Sie eine Nacht vor der Kultur des Expressionswirt E. coli BL21 (DE3) pLyss, Beherbergung des jeweiligen pET - Vektor mit dem geklonten Gen oder dem leeren Vektor, in 100 ml Kulturflaschen , die 20 ml Luria Bertani - Brühe (LB) 17 sowie den erforderlichen Antibiotika. Kultur die Zellen bei 30 ° C (oder im üblichen Wachstumstemperatur des Bakteriums , von dem die Lipase stammt).
- Mit den über Nacht Vorkulturen, impfen 500 ml vorgewärmtem LB - Medium (plus die erforderlichen Antibiotika) in 2 l Kulturflaschen eine anfängliche optische Dichte bei 620 nm (OD 620) = 0,05 zu erhalten. Folgen Wachstum der Kulturen und bei einer OD 620 = 0,3, fügen Isopropyl-β-D-thiogalactosid (IPTG) bis zu einer Endkonzentration von 100 uM, und Inkubieren unter Rühren bei 30 ° C für einen Zeitraum von 4 Stunden.
- Am Ende der Inkubationsperiode übertragen jeder Kultur in einen 500 - ml - Zentrifugenröhrchen und zentrifugiere bei 5.000 × g bei 4 ° C für 30 min. Resuspendieren Bakterienzellpellets in 5 ml Suspensionspuffer (zB SMc00930- und SMc01003-exprimierenden Zellen in 50 mM Tris-HCl pH 8,0 und SMc00171-exprimierenden Zellen in 50 mM Diethanolamin-HCl pH 9,8). Speichern der Zellsuspensionen bei -80 ° C bis zur Verwendung.
2. Bereiten Sie die zellfreie Proteinextrakte und Protein-Konzentration bestimmen
- Tauen Bakterienzellsuspensionen und lagern auf dem Eis. Passieren Zellsuspensionen dreimal durch eine Kaltdruckzelle bei 20.000 lb pro in 2. Entfernen intakten Zellen und Zelltrümmer durch Zentrifugation bei 5.000 xg für 30 min bei 4 ° C.
- Nach Zentrifugation herzustellen Aliquots von 100 und 500 & mgr; l aus dem Überstand für eine nachfolgende Analyse und speichert sie bei -80 ° C bis zur Verwendung.
- Verwenden Sie eines der 100 ul aliquoten Proteinkonzentration von verschiedenen zellfreien Extrakten durch ein Verfahren der Wahl , um zu bestimmen oder als 18 beschrieben.
3. Verwenden Sie künstliche Substrate für Optimizing EnzymAktivitäten von (Phospho) Lipasen
- Für eine erste Abdeckung der unterschiedlichen Enzymaktivitäten verwenden künstliche Substrate , die ein gefärbtes Produkt bei der Hydrolyse ergeben, wie beispielsweise p - Nitrophenol (p -NP).
- Für Enzymassays optimiert bereits mit Kunst p - Nitrophenylester Substrate (umrissenen für Phospholipase C PLCP (SMc00171) sowie für den vorhergesagten Patatin-like Phospholipasen SMc00930 und SMc01003) beschriebenen Verwendung Pipettieren Schemata in Tabelle 2.
- Wenn ein neues Potential (phospho) Lipase erforschen, mit einem ersten Standard - Enzym - Assay , das 50 mM Tris-HCl, 8,5, 100 mM NaCl, 0,05% Triton X-100, 0,5 mM p - Nitrophenyl-enthaltende Verbindung (p - Nitrophenylphosphat pH vorzubereiten , Bis p - Nitrophenylphosphat, p - Nitrophenyl- decanoate oder p - Nitrophenyl Palmitat) und zellfreie Proteinextrakt (Check 1, 3, 10, 30, 100, 300 und 1000 ug) in einem Gesamtvolumen von 1 ml in 1 ml Kunststoff cuvettes.
HINWEIS: Die Verwendung alkalischen pH (Abbildung 1) , wenn folgende p - Nitrophenylester Hydrolyse in einem kontinuierlichen Assay. Alternativ können Sie auch einzelne Zeitpunkt - Assays für einen Bereich von pH - Werten, Zugabe von NaOH am Ende der Inkubationszeit die Enzymreaktion zu beenden und sicherzustellen , dass alle p -NP in der Phenolat Form vorliegt. - Folgen den Zeitverlauf für eine Zunahme der Absorption bei 405 nm aufgrund der Bildung von p -NP, in einem Spektrophotometer bei 30 ° C über einen Zeitraum von 5 min. Quantifizierung der anfänglich linearen Bildung von p -NP durch die anfängliche Steigung der Zunahme der Extinktion pro Zeit bestimmen.
- Berechnen Sie die Änderung der Konzentration (& Delta; c) für p -NP mit dem Gesetz von Lambert-Beer (& Delta; A = ε & Delta; c d) 1.
HINWEIS: & Dgr; A ist die lineare Änderung der Extinktion bestimmt, ε der molare Extinktionskoeffizient bei der jeweiligen Wellenlänge (in Einheiten von M -1 cm -1) ist , d die Länge des Lichtweges (1 cm), und & Dgr; c ist die Änderung der Konzentration (in Einheiten von M) bestimmt werden.- Bedenkt man, dass die Assay - Volumen von 1 ml ist, berechnen die Menge an p -NP gebildet.
HINWEIS: Betrag = Konzentration x Volumen. - Berechnung der Enzymaktivität durch die Menge an p -NP durch die Zeit gebildet Aufteilen in dem es gebildet wird. Bestimmung der spezifischen Enzymaktivität von Enzymaktivität durch die Menge an Protein Dividieren (in mg), die zur Erzeugung von diese Aktivität verantwortlich war.
- Bedenkt man, dass die Assay - Volumen von 1 ml ist, berechnen die Menge an p -NP gebildet.
- Vergleichen Absorptionsänderungen hervorgerufen durch Proteinextrakte , in denen ein Kandidat - Gen (smc00171, smc00930 oder smc01003) hatte mit Extrakten zum Ausdruck gebracht , die nur einen leeren Vektor enthalten .
ANMERKUNG: Um mit den folgenden Schritten fortzufahren, die spezifischen Aktivitäten, die durch Proteinextrakte in dem ein Kandidatengen exprimiert worden waren, mindestens zweimal oder m seinErz als die Werte für die spezifischen Aktivitäten, die durch Proteinextrakte gewonnen, die nur einen leeren Vektor enthalten. - Für weitere Experimente, wählen jene Bedingungen , unter denen die Hydrolyse des p - Nitrophenyl-enthaltende Verbindung ist minimal mit zellfreien Extrakten (dh Leervektor) und für die der stärksten Bildung von p -NP und der p -nitrophenolate Anion (Fig 1) beobachtet werden kann , wenn die Proteinextrakte verwendet werden, in denen ein Kandidat - Gen exprimiert worden war.
- die anfängliche Enzymaktivität in 3.1, Optimierung der Testbedingungen für das jeweilige Enzym nach der Bestimmung von pH, Puffertyp variierenden Pufferstärke, Konzentrationen von NaCl, Detergenzien, wie Triton X-100, und die Abwesenheit oder Anwesenheit von verschiedenen zweiwertigen Kationen.
- Für unterschiedliche Konzentrationen der einzelnen Variablen, die spezifische Enzymaktivität bestimmen (siehe 3.1.4.2) (die höchste Zahl erhalten definiert die Bedingungder maximale Enzymaktivität). Verwenden Sie die Kombination der optimalen Bedingungen für jede Variable angetroffen ein optimiertes Enzymtest zu definieren, in der jede Variable in seiner optimalen Konzentration vorhanden ist.
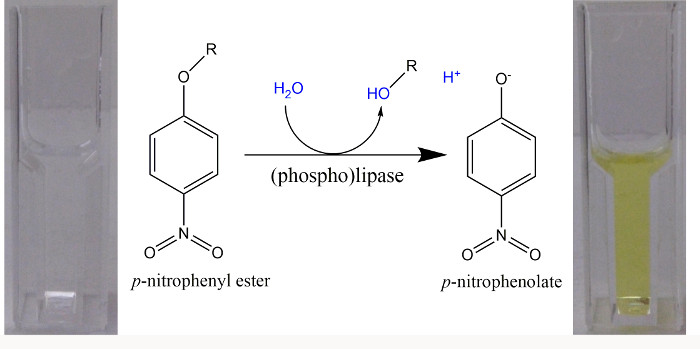
Abbildung 1 p - Nitrophenyl Ester als künstliche Substrate für (phospho) Lipasen in einem spektrophotometrischen Assay. Bei der Hydrolyse von p - Nitrophenyl Ester, eine Säure (R-OH) und p - Nitrophenol (p -NP) gebildet werden . Aufgrund der pK a = 7,2 für die Dissoziation des + phenolischen H aus p -NP, bei einem pH - Wert> 9,2 mehr als 99% in der hellen Form gelb p -nitrophenolate sind und einem molaren Extinktionskoeffizienten von 18.000 M -1 cm - 1 kann bei einer Wellenlänge verwendet werden von 405 nm für die Quantifizierung von freiem p -nitrophenolate 22. Wenn Puffer mit einem pH - Wert von 8,5 verwendet wurden, wurde die Absorption bei 400 nm und einem molaren Extinktionskoeffizienten von 14.500 M -1 cm -1 bestimmt wurde 23 verwendet. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
HINWEIS: Nachdem die optimalen Bedingungen für die Aktivität des Enzyms von Interesse definiert zu haben, begeben sich auf die Suche nach dem real / physiologischen Substrat dieser Lipase. Grundsätzlich nehmen zwei, oft komplementäre Ansätze , dieses Ziel zu erreichen, eine in vivo - Ansatz oder ein in vitro - Ansatz.
4. In vivo Identifizierung des physiologisches Substrat einer Lipase
ove_content "> . ANMERKUNG: In einem In - vivo - Ansatz, die Lipase von Interesse in einem Wirtsorganismus 8,11 um auszudrücken über die Zeit zu registrieren , ob die Expression der Lipase das Profil host's Lipid verändert In einem anderen in vivo Ansatz, erzeugen ein mutanten - defizienten des Gens von Interesse 8,11 und untersuchen , ob seine Lipidprofil von der Wildtyp - Version 6,8,11 unterscheidet. um eine quantitative Beurteilung eines Organismus Lipidprofil, eine einfache Methode besteht darin , die radioaktive Markierung von zellulären Verbindungen zu erhalten , Extrahieren der Lipide, sie durch Chromatographie und die Quantifizierung der radioaktiv markierten Lipiden getrennt trennt.- Die radioaktive Markierung von Lipiden.
- Bereiten Sie eine Nacht Vorkultur eines Organismus von Interesse (E. coli oder S. meliloti) in 5 ml des gewünschten Kulturmedium (komplexes Medium oder definierte Minimalmedium) und wachsen bei 30 ° C.
- Von der Vorkultur impfen in 20 ml des same frisches Medium in einer 100 ml Kulturflasche eine anfängliche OD 620 = 0,3 für die Kultur zu erhalten.
- Einen aliquoten (1 ml) der Kultur unter sterilen Bedingungen und in ein 14 ml sterilen Polystyrol-Rundbodenrohr.
- 1 & mgr; Ci von [1- 14 C] Acetat (60 mCi pro mmol) zu der 1 ml Kultur.
- Inkubieren der flüssigen Kultur unter Rühren bei 30 ° C für einen Zeitraum von 24 Stunden.
- Am Ende der Inkubationsperiode Übertragung der Kultur in ein 1,5 ml Mikrozentrifugenröhrchen und zentrifugiere bei 12.000 xg bei Raumtemperatur für 5 min.
- Resuspendieren des Pellets in 100 ul Wasser. An diesem Punkt, speichern Sie die Zellsuspension bei -20 ° C oder unmittelbar mit der Gewinnung von polaren Lipiden (Abschnitt 4.2) fortzusetzen.
- Die Extraktion von polaren Lipiden.
HINWEIS: Die Methode , die hier im Wesentlichen beschrieben folgt dem Verfahren berichtet von Bligh und Dyer 19.- Zu den 100 ul wässriger Zelle suspension, fügen 375 ul Methanol: Chloroform-Lösung (2: 1; vol / vol).
- Vortex für 30 Sekunden und 5 Minuten bei Raumtemperatur inkubiert.
- Zentrifuge 5 min bei 12.000 xg bei Raumtemperatur.
- Den Überstand in ein neues 1,5 ml Mikrozentrifugenröhrchen.
- In 125 ul Chloroform und 125 ul Wasser, Wirbel 30 sec.
- Zentrifuge 1 min bei 12000 xg bei Raumtemperatur.
- Übertragen Sie die untere Chloroform-Phase in ein frisches Röhrchen und trocken mit einem Strom von Stickstoffgas.
- Auflösen getrockneten Lipide in 100 ul Chloroform: Methanol-Lösung (1: 1; vol / vol).
HINWEIS: Zu diesem Zeitpunkt wurde ein Aliquot von 5 & mgr; l der Lipidlösung kann durch Flüssigscintillationszählung quantifiziert werden. - Für dünnschichtchromatographischen (TLC) Analyse, trocknen unten die restlichen 95 & mgr; l mit einem Strom von Stickstoffgas abgeblasen, getrockneten Lipide in 20 ul Chloroform: Methanol-Lösung (1: 1; v / v). Verwenden Sie ein 3-ul-Aliquotfür DC-Analyse.
- Trennung von polaren Lipiden durch Dünnschichtchromatographie (TLC).
HINWEIS: Klassen auf den Lipid Je analysiert werden, verschiedene Kombinationen von festen und mobilen Phasen kann für die Trennung eingesetzt werden. Hier ist eine typische Trennung für geladene polare Lipide und andere, geeignet für neutrale polare Lipide, unter Verwendung von Hochleistungs-Dünnschicht-Chromatographie (HPTLC) Kieselgel Aluminiumbleche als feste Phase, skizziert.- Trennung von geladenen polaren Lipiden durch zweidimensionale Dünnschichtchromatographie (2D-TLC).
- Eine 3 & mgr; l Aliquot von Lipidprobe in einer Ecke eines HPTLC Kieselgel Aluminiumblech (10 x 10 cm), 2 cm vom Rand der Platte.
- Bereiten Sie und mischen Sie die mobile Phase (140 ml Chloroform, 60 ml Methanol und 10 ml Wasser) für die Trennung in der ersten Dimension.
- Mantel eine TLC Entwicklungskammer intern mit Chromatographie-Papier.
Hinweis: Dies ist, um sicherzustellen, dass die Gasphase der Kammer(Innerhalb von 30 min) wird schnell gesättigt werden, nachdem die mobile Phase für die erste Dimension wurde in die Kammer gegeben, und die Kammer wurde mit einer Glasplatte verschlossen. - Bereiten Sie und mischen Sie die mobile Phase (130 ml Chloroform, 50 ml Methanol und 20 ml Eisessig) für die Trennung in der zweiten Dimension und die Übertragung auf eine zweite TLC Entwicklungskammer intern mit Chromatographie Papier aufgetragen und die Kammer sättigen lassen.
- Übertragen sorgfältig die HPTLC Kieselgel Aluminiumblech mit dem getrockneten Lipidprobe in die erste Kammer und zu entwickeln , um die Platte für 60 min in der geschlossenen Kammer in der ersten Dimension 5 (dh Chromatographie durchzuführen).
- Entfernen Sie die Platte aus der Kammer und lassen Lösungsmittel für 30 Minuten in einem Flow-Haube trocknen.
- Nach dem Drehen auf dem die Platte um 90 Grad in Bezug auf die vorherigen Chromatographie, übertragen die HPTLC Kieselgel Aluminiumblech wurden die Lipide in einer Dimension getrennt wurden, zum SECONd Kammer und entwickeln , um die Platte für 60 Minuten in der zweiten Dimension 5.
- Entfernen Sie das Blatt aus der Kammer und lassen die Lösungsmittel für mindestens 2 Stunden in einem Flow-Haube trocknen.
- Die Trennung von neutralen polaren Lipiden.
- Anwenden 3 ul Aliquots von Lipidproben auf einer HPTLC Kieselgel Aluminiumblech beginnend 2 cm von den Rändern der Platte. Wenn mehrere Proben in einem eindimensionalen Chromatographie analysiert werden, halten Sie einen Abstand von mindestens 1,5 cm zwischen den verschiedenen Beispielanwendung Flecken.
- Bereiten Sie und mischen Sie die mobile Phase (140 ml Hexan, 60 ml Diethylether, und 8 ml Essigsäure) aufnehmen und in einem TLC Entwicklungskammer intern beschichtet mit Chromatographiepapiers und bedeckt mit einer Glasplatte in die Kammer sättigen zu lassen (30 min).
- Übertragung der HPTLC Kieselgel Aluminiumblech mit den getrockneten Lipidproben in die Kammer und zu entwickeln, um die Platte für 30 min in der geschlossenen Kammer.
- Entfernen Sie die Platte aus der Kammerund lassen Sie die Lösungsmittel für 2 Stunden in einem Flow-Haube trocknen.
- Trennung von geladenen polaren Lipiden durch zweidimensionale Dünnschichtchromatographie (2D-TLC).
- Die Quantifizierung und Visualisierung von getrennten polaren Lipiden.
- Sobald die entwickelte DC-Blatt trocken ist, brüten sie mit einem photostimulierbaren Lumineszenz (PSL) auf dem Bildschirm in einer geschlossenen Kassette für 3 Tage.
- Setzen Sie die inkubiert Bildschirm zu einem PSL-Scanner und erwerben ein virtuelles Bild der getrennten radiomarkierten Lipiden.
- Führen Sie die Quantifizierung mit PSL - Software 20.
- Visualisierung und Isolierung einzelner polaren Lipide Klassen.
- Inkubieren entwickelten TLC Blatt für 10 min in einer Chromatographiesäule Kammer in Gegenwart von 1 g Jodkristalle.
HINWEIS: Getrennt lipidischen Verbindungen wird das Jod und erscheinen als bräunliche Flecken aufzulösen. - Kreisen Sie die Flecken mit einem Bleistift, vergleichen sie mit der relativen Mobilität (R f) von Standard - Verbindungen (dh 1,2-Dipalmitoyl- sn - glycerin, Dipalmitoyl-L-α-phosphatidylcholine, DL-α-Monopalmitin oder Palmitinsäure) und identifizieren, zu der Lipidklasse sie angehören könnten.
- In einem Abzug ließ das Jod aus dem DC-Blatt verdunsten.
- Mit Hilfe eines Spachtels, kratzen die Kieselgel die Verbindung von Interesse aus der Folie enthält, und extrahieren Sie die Verbindung aus dem Kieselgel mit einem Gemisch aus 100 ul Wasser und 375 ul Methanol: Chloroform-Lösung (2: 1; vol / vol).
- Fahren Sie mit der Extraktion nach Bligh und Dyer wie skizziert (4.2.2 ff).
- Store gereinigt Lipidklasse in 100 ul Chloroform: Methanol - Lösung (1: 1; vol / vol) bei -20 ° C bis zur Verwendung.
- Inkubieren entwickelten TLC Blatt für 10 min in einer Chromatographiesäule Kammer in Gegenwart von 1 g Jodkristalle.
5. In - vitro - Identifizierung des physiologisches Substrat einer Lipase
HINWEIS: In einem in vitro - Ansatz untersuchen , ob die Lipase von Interesse eine Mischung aus isolierten Lipide oder einzelne reine Lipide in die entsprechende hydrol umwandeln kannlyse Produkte unter den als optimal definierten Bedingungen in 3.2.
- Verwenden Sie Pipettieren Schemata für Enzymtests gemäß Tabelle 3 für PC-spezifischer Phospholipase C SMc00171 (siehe 5.2), Phospholipase A (siehe 5.3) und DAG - Lipase SMc01003 (siehe 5.4) Aktivität.
- Bestimmung von PC-spezifischen Phospholipase - C - Aktivität (Tabelle 3).
- In ein 1,5 ml Mikrozentrifugenröhrchen, fügen 5.000 Zählimpulsen pro Minute (cpm) von insgesamt 14 C-markiertem PC und eine Lösung von Triton X-100.
- Mischen Sie und trocken unter einem Stickstoffstrom.
- Hinzufügen Diethanolamin-HCl, pH 9,8 Puffer, sowie NaCl und MnCl 2 Lösungen und bidestilliertem Wasser auf ein Endvolumen von 99,5 & mgr; l zu erhalten. Vortex für 5 Sekunden.
- Mit 0,5 & mgr; l Enzym (5 ug Protein) (dh einen zellfreien Extrakt , bei dem überexprimiert SMc00171 vorhanden ist) , um die Reaktion zu starten. Mischen Sie kurz.
- Inkubieren bei 30 ° C für 4 Stunden.
- Die Reaktion durch dieZugabe von 250 ul Methanol und 125 ul Chloroform.
- Extrahieren Lipide wie zuvor beschrieben (siehe 4.2).
- Separate Lipide durch eindimensionale (1D) -TLC (siehe 4.3.2 und 4.4), und analysieren sie durch PSL-Bildgebung.
- Bestimmung der Phospholipase A - Aktivität (Tabelle 3).
- Zu einem 1,5 ml Mikrozentrifugenröhrchen, fügen Sie 5000 cpm von insgesamt 14 C-markierte Phospholipide und eine Lösung von Triton X-100.
- Mischen Sie und trocken unter einem Stickstoffstrom.
- Für eine endgültige 100 & mgr; l Assay, fügen Tris-HCl, pH 8,5 Puffer, NaCl-Lösung und Wasser. Vortex für 5 Sekunden.
- Zugabe von 5 & mgr; l Enzym (50 ug Protein) (dh einen zellfreien Extrakt , bei dem überexprimiert SMc00930 oder SMc01003 vorhanden ist).
- Inkubieren bei 30 ° C für 5 Stunden.
- Beenden Sie die Reaktion durch Zugabe von 250 ul Methanol und 125 ul Chloroform.
- Extrahieren Sie Lipide, wie zuvor beschrieben (siehe 4.2), separatessie von 1D-TLC unter Verwendung von 130 ml Chloroform, 50 ml Methanol und 20 ml Eisessig als mobile Phase, und analysieren sie durch PSL-Bildgebung.
- Bestimmung von Diacylglycerin (DAG) Lipase-Aktivität.
- Herstellung von 14 C-markiertem DAG.
- Radioaktive Markierung S. meliloti Kulturen (siehe 4.1) und extrahieren polare Lipide (siehe 4.2) , wie beschrieben. Separate S. meliloti Gesamtlipidextrakte von 1D-TLC in Chloroform: Methanol: Essigsäure (130: 50: 20; vol / vol) Bedingungen für die Trennung in 4.3.1 in der zweiten Dimension beschrieben ist .
- Visualisieren PC durch Jod-Färbung und mit einem Bleistift die Lokalisierung von Phosphatidylcholin (PC) zu markieren.
- Isolieren radiomarkierten PC wie in 4.5 beschrieben.
- Quantifizieren extrahierter PC durch Szintillationszählung.
HINWEIS: Über 320.000 cpm PC wird erwartet. - Behandlung von PC (250.000 cpm) mit 0,1 U von Phospholipase C aus Clostridium perfringens in 50 mM Tris-HCl, pH 7,2, 0,5% Triton X-100 und 10 mM CaCl 2 für 2 Stunden in einem Gesamtvolumen von 100 ul und die Reaktion durch Zugabe von 250 ul Methanol und 125 ul Chloroform stoppen.
- Extrahieren Lipide wie zuvor und getrennt von ihnen durch 1D-TLC beschrieben (siehe 4.3.2).
- Isolieren Diacylglycerin von der Kieselgelplatte und quantifizieren durch Szintillationszählung (wie in 4.2 beschrieben)
- Diacylglycerin - Lipase - Assay (Tabelle 3).
- In ein 1,5 ml Mikrozentrifugenröhrchen, fügen 5000 cpm von 14 C-markiertem DAG und eine Lösung von Triton X-100.
- Mischen Sie und trocken unter einem Stickstoffstrom.
- Für eine endgültige 100 ul Assay, fügen Tris-HCl (pH 9,0) Puffer, eine NaCl-Lösung und bidestilliertes Wasser. Vortex für 5 Sekunden.
- Initiieren der Reaktion durch Zugabe von 5 & mgr; l Enzym (50 & mgr; g Protein von zellfreier Extrakt).
- Inkubieren bei 30 ° C für 4 Stunden.
- Stoppen Sie die Reaktion durch die Zugabe von 250 ul Methanol eind 125 ul Chloroform und Extrakt Lipide wie zuvor beschrieben (siehe 4.2).
- Analysieren neutrale polare Lipide von 1D-TLC (siehe 4.1.3.2) und anschließender PSL-Bildgebung.
- Herstellung von 14 C-markiertem DAG.
Ergebnisse
Aktivität von PC-spezifische Phospholipase C SMc00171 mit Bis- p - Nitrophenylphosphat
Zellfreie Extrakte von E. erhalten coli BL21 (DE3) pLysS x, die smc00171 hatte ausgedrückt, wurden auf ihre Fähigkeit untersucht bis- p - Nitrophenylphosphat Ester zu hydrolysieren, eine spektrophotometrische enzymatischen Assay unter Verwendun...
Diskussion
In den letzten 20 Jahren haben die Genome vieler Organismen sequenziert worden, und obwohl eine Fülle von Daten Genomsequenz generiert wurde, ist funktionale Auslegung Rückstand und damit behindert unser Verständnis der Genomfunktion. Genfunktionen in Genomen basieren häufig auf Ähnlichkeit mit Genen bekannter Funktion oder Auftreten von konservierten Motive zugeordnet. Jedoch ist die genaue Funktion eines bestimmten Gens häufig nicht bekannt. Insbesondere vorhergesagte strukturelle Gene für Enzyme können nicht ...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde durch Zuschüsse aus Consejo Nacional de Ciencias y Tecnología-México (CONACyT-Mexico) (82.614, 153.998, 253.549 und 178.359 in Investigación Científica Básica sowie 118 in Investigación en Fronteras de la Ciencia) und von Dirección General de unterstützt Asuntos de Personal Académico-Universidad Nacional Autónoma de México (UNAM DGAPA-; PAPIIT IN202616, IN203612).
Materialien
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Referenzen
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten