Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
G-Protein-selektive GPCR Konformationen gemessen mit FRET-Sensoren in einer Live Cell Suspension Fluorometer Assay
In diesem Artikel
Zusammenfassung
Simple methods to detect the selective activation of G proteins by G protein-coupled receptors remain an outstanding challenge in cell signaling. Here, Fӧrster resonance energy transfer (FRET) biosensors have been developed by pairwise tethering a GPCR to G protein peptides to probe conformational changes at controlled concentrations in live cells.
Zusammenfassung
Fӧrster Resonanzenergietransfer (FRET) -basierte Studien immer häufiger bei der Untersuchung von GPCR-Signal geworden sind. Unsere Arbeitsgruppe entwickelte ein intramolekularen FRET-Sensor die Wechselwirkung zwischen Ga-Untereinheiten und GPCRs in lebenden Zellen nach Agonist Stimulation zu erkennen. Hier stellen wir ausführlich das Protokoll für die Veränderungen in der FRET zwischen dem β 2 -adrenergen Rezeptor und dem Gαs C-Terminus - Peptid bei der Behandlung mit 100 & mgr; M Isoproterenolhydrochlorid Erfassen gekennzeichnet zuvor 1. Unsere FRET-Sensor ist ein einzelnes Polypeptid, das aus seriell von einer Volllängen-GPCR, einem FRET-Akzeptor-Fluorophor (mCitrine), eine ER / K SPASM (systematische Protein-Affinitätsstärke modulation) Linker, einem FRET-Donor-Fluorophor (mCerulean) und eine G & agr; C -terminalen Peptid. Dieses Protokoll wird ausführlich Zellpräparation, Transfektionsbedingungen, Geräte-Setup, Testausführung und Datenanalyse. Dieses experimentelle Design erkennt kleine changes in FRET indikativ für Protein-Protein-Wechselwirkungen und kann auch verwendet werden, die Stärke der Interaktion zwischen den Liganden und GPCR-G-Protein-Paarungen zu vergleichen. Um das Signal-Rausch-in unseren Messungen zu verbessern, Dieses Protokoll erfordert erhöhte Präzision in allen Schritten, und wird hier präsentiert, um reproduzierbare Durchführung ermöglichen.
Einleitung
G-Protein-gekoppelte Rezeptoren (GPCRs) sind sieben-Transmembran-Rezeptoren. Das menschliche Genom enthält etwa 800 allein Gene kodierend für GPCRs, die durch eine Vielzahl von Liganden, einschließlich Licht, Geruchsstoffe, Hormone, Peptide, Arzneimittel und andere kleine Moleküle aktiviert werden. Fast 30% aller Arzneimittel auf dem Markt Ziel GPCRs , weil sie eine große Rolle spielen bei vielen Krankheitszuständen 2. Trotz jahrzehntelanger intensiver Arbeit an dieser Rezeptorfamilie getan, gibt es weiterhin erhebliche offene Fragen auf dem Gebiet, vor allem im Hinblick auf die molekularen Mechanismen, die GPCR-Effektor-Wechselwirkung fahren. Bis heute hat nur eine hochauflösende Kristallstruktur veröffentlicht worden sind , einen Einblick in die Wechselwirkung zwischen dem β 2 -adrenergen Rezeptor Bereitstellung (β 2 -AR) und das Gs - Protein 3. Zusammen mit den umfangreichen Forschung in den letzten drei Jahrzehnten, wiederholt sie eine spezifische strukturelle Komponente, die in dieser kritischen istInteraktion: der C-Terminus G & agr; -Untereinheit. Diese Struktur ist wichtig für die G - Protein - Aktivierung durch den GPCR 4 und G - Protein - Auswahl 5-6. Daher stellt die G & agr; C-Terminus eine entscheidende Verbindung zwischen Ligand Stimulation des GPCR und selektive Protein-Aktivierung G.
Forschung im letzten Jahrzehnt legt nahe, dass GPCRs eine breite Konformationsänderungen Landschaft bevölkern, mit Ligand-bindenden Untergruppen von GPCR Konformationen zu stabilisieren. Während verschiedene Techniken, einschließlich Kristallographie, NMR und Fluoreszenzspektroskopie und Massenspektrometrie sind verfügbar , um die GPCR Konformationsänderung Landschaft zu untersuchen, gibt es einen Mangel von Ansätzen ihre funktionelle Bedeutung in Effektor Auswahl 7 zu erläutern. Hier beschreiben wir eine Fӧrster Resonanzenergietransfer (FRET) -basierte Ansatz G-Protein-selektive GPCR Konformationen zu erkennen. FRET stützt sich auf die Nähe und die parallele Ausrichtung von zwei Fluorophore mit überlappenden Emissions (Donor) einnd Anregung (Akzeptor) Spektren 8. Als Donor und Akzeptor - Fluorophore kommen einander näher als Ergebnis entweder Konformationsänderung im Protein oder ein Protein-Protein - Wechselwirkung erhöht die FRET zwischen ihnen, und 8 mit einer Reihe von Verfahren gemessen werden. FRET-basierten Biosensoren wurden 9 ausführlich in der GPCR - Bereich eingesetzt. Sie wurden durch Einfügen Donor und Akzeptor in der dritten intrazellulären Schleife und GPCR C-Terminus in der GPCR Sonde Konformationsänderungen verwendet; Sensoren wurden durch getrennte Markierung des GPCR und Effektorzellen (G - Protein - Untereinheiten / Arrestine) mit einem FRET - Paar 10 zur Sonde GPCR und Effektor - Wechselwirkung entwickelt; Einige Sensoren auch Konformationsänderungen in dem G - Protein 11 erfassen. Diese Biosensoren haben das Feld aktiviert eine Vielzahl von offenen Fragen zu stellen , einschließlich Konformationsänderungen in der GPCR und Effektorzellen, GPCR-Effektor - Wechselwirkung Kinetik und allosterische Liganden 12. Unsere Gruppewar besonders daran interessiert, einen Biosensor zu schaffen, die G-Protein-spezifischen GPCR Konformationen unter Agonisten getriebenen Bedingungen erkennen konnte. Dieser Biosensor beruht auf einer neu entwickelten Technologie SPASM (systematische Protein - Affinitätsstärke Modulation) 13 genannt. SPASM beinhaltet Tethering die Interaktion Proteindomänen ein ER / K-Linker mit, die ihre wirksamen Konzentrationen kontrolliert. Flankierend den Linker mit einem FRET - Paar von Fluorophoren schafft ein Werkzeug , das den Zustand der Wechselwirkung zwischen Proteinen 12 mitteilen. Zuvor 1 wurde das SPASM Modul verwendet , um die G & agr; C-Terminus mit einem GPCR anbinden und überwachen ihre Wechselwirkungen mit FRET Fluorophore, mCitrine (bezeichnet in diesem Protokoll , das von seiner allgemein bekannten Variante Yellow Fluorescent Protein (YFP), Anregungs- / Emissions - Peak bei 490/525 nm) und mCerulean (bezeichnet in diesem Protokoll, das von seiner allgemein bekannten Variante Cyan Fluorescent Protein (GFP), Anregungs- / Emissions-Peak 430/475 nm). Vom N- zum C-Terminus, tseine genetisch kodierte einzelnes Polypeptid enthält: ein in voller Länge GPCR, FRET-Akzeptor (mCitrine / YFP), 10 nm ER / K-Linker, FRET-Donor (mCerulean / CFP) und der G & agr; C-Terminus-Peptid. In dieser Studie Sensoren sind als GPCR-Linker-Länge-G & agr; Peptid abgekürzt. Alle Komponenten werden von einer unstrukturierten (Gly-Ser-Gly) 4 Linker getrennt , die eine freie Drehung jeder Domäne ermöglicht. Die detaillierte Charakterisierung solcher Sensoren wurde durchgeführt , die zuvor zwei prototypische GPCRs mit: β 2 -AR und Opsin 1.
Dieser Sensor ist transfiziert transient in HEK-293T-Zellen und Fluorometer basierten lebenden Zellen Experimente messen Fluoreszenzspektren des FRET-Paares in willkürlichen Einheiten der Zählungen pro Sekunde (CPS) in Gegenwart oder Abwesenheit eines Liganden. Diese Messungen werden verwendet , um ein FRET - Verhältnis zwischen den Fluorophoren (YFP max / CFP max) zu berechnen. Eine Änderung in der FRET (ΔFRET) wird dann durch Subtraktion des durchschnittlichen FRET-Verhältnis berechnetder unbehandelten Proben aus dem FRET Verhältnis von Ligand behandelten Proben. ΔFRET kann über Konstrukte (β 2 -AR-10 nm-Gαs Peptid im Vergleich zu ß 2 -AR-10 nm-kein Peptid) verglichen werden. Hier stellen wir ausführlich das Protokoll, um diese Sensoren in Live-HEK-293T-Zellen exprimieren, überwachen deren Ausdruck und die Einrichtung, Durchführung und Analyse des Fluorometers-basierten Live-Cell-FRET-Messung für unbehandelte gegen Arzneimittel-behandelten Bedingungen. Während dieses Protokoll für den β 2 -AR-10 nm-Gαs Peptidsensor mit 100 & mgr; M Isoproterenol Bitartrat spezifisch ist, kann es für verschiedene GPCR-G & agr; Paare und Liganden optimiert werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. DNA-Präparation
- Design-Sensor konstruiert ein modulares Klonens Schema verwendet. Beziehen Sie sich auf den β 2 -AR Sensordesign zuvor 1 beschrieben.
- Bereiten Sie DNA nach Handels Minipräparationskit Protokoll und eluieren in 2 mM Tris-HCl - Lösung, pH 8, bei einer Konzentration von ≥ 750 ng / & mgr; l, A 260 / A 280 von 1,7-1,9, A 260 / A 230 von 2,0 bis 2,29.
2. Zellkultur Vorbereitung
- Kultur HEK-293T-Flp-n-Zellen in DMEM, das 4,5 g / L D-Glucose, supplementiert mit 10% FBS (hitzeinaktiviert) (v / v), 1% L-Glutamin zu ergänzen, 20 mM HEPES, pH 7,5 bei 37 ° C in angefeuchteter Atmosphäre mit 5% CO 2. Griff Zellen in biologischen Schutzhaube für den nachfolgenden Schritten.
- Lassen Zellen zu einer konfluenten Monoschicht zu wachsen , bevor Passagierung in Sechs-Well - Gerichte. Zeit Konfluenz zu erreichen, hängt von anfänglichen Plattierungsdichte. Verwenden Platten, diekommen zu Konfluenz in 1 - 2 Tage Plattierung für Sechs-Well-Beschichtung. Eine konfluente 10 cm Gewebekultur-behandelte Schale hat Zelldichte von etwa 4 x 10 6 Zellen / ml. Siehe Abbildung 1 für Bild von Zellkulturwachstum.
- Waschen Sie die Zellen mit 10 ml PBS und trypsinize mit 0,25% Trypsin (siehe Diskussion, Absatz 2). Platte 8 x 10 5 Zellen / Vertiefung in 2 ml Medium in Gewebekultur-behandelten sechs - Well - Schalen und ermöglichen 16 anhaften - 20 Stunden.
3. Transfektionsbedingungen
- Stagger Transfektionen für Konstrukte, die unterschiedliche Mengen an Zeit erfordern eine optimale Expression zu erreichen (zwischen 20 - 36 h). Synchronisieren Sie Bedingungen für eine einheitliche Versuchszeit. Auch haben eine nicht-transfizierte Kontrollvertiefung bei äquivalenten Zelldichte für die Hintergrundgeräusche und Streu Subtraktion während der Analyse verwendet werden.
- Bringen Sie Transfektionsreagenzien auf Raumtemperatur: reduzierte Serum Medien, DNA, Transfektionsreagenz.
- In einembiologische Schutzhaube Reagenzien in einem sterilen Mikrozentrifugenröhrchen in der folgenden Reihenfolge kombinieren: mit 100 & mgr; l reduziert Serum Medien 2 ug DNA mischen. Spike 6 ul Transfektionsreagenz in Medium / DNA-Gemisch ohne Oberfläche Mischung zu berühren oder die Seite des Röhrchens. Richten Sie eine Transfektionsreaktion pro Vertiefung. Transfektionsbedingungen können (1 - 4 & mgr; g DNA, 3 - 6 ul Transfektionsreagenz) optimiert werden konsistente Expressionsniveaus zu erreichen. Siehe Tabelle 1 für weitere optimierte Verhältnisse.
- Inkubieren Mischung bei RT in biologischen Schutzhaube für 15 - 30 min. Nicht Reaktion verwenden, wenn länger als 30 min inkubiert.
- In Reaktion auf die Zellen in einer tropfenweise Art und Weise über gut und sanft schütteln sechs Vertiefungen Durchmischung zu gewährleisten. In einer Reaktion pro Vertiefung.
- unter Verwendung von Gewebekulturfluoreszenzmikroskop Nach 20 Stunden des Ausdrucks, Monitor Fluoreszenz. Beurteilen Bevölkerung Ausdruck mit 10X-Objektiv und Proteinlokalisierung in einer Zelle bei 40X. Observe für die Proteinexpression in der Plasmamembran (PM). Wenn wesentliche Internalisierung festgestellt wird, überwachen die Transfektion, bis eine signifikante Expression bei PM erfasst wird.
4. Reagenz und Vorbereitung der Ausrüstung
- Bereiten 100 mM Pharma - Aktien und bei -80 ° C: Isoproterenol Bitartrat (100 mM in dH 2 O , enthaltend 300 mM Ascorbinsäure). Machen Sie auf Eis / in kalten Raum, und Flash-Freeze sofort. Aliquots können bis zu einem Jahr hergestellt und verwendet werden.
- Bereiten Sie Zellenpuffer (~ 2 ml / Zustand) und Speicher in einem 37 ° C Wasserbad. Machen Sie jeden Tag frisch. Referenztabelle 2 für Zellenpuffer Bestandteile.
- Bereiten Drug-Puffer (10 ml) und bei Raumtemperatur lagern. Referenztabelle 2 für Drogen Buffer Bestandteile.
- Waschen mit Säure Küvetten konzentrierter HCl. Neutralisieren mit einer schwachen Base (1 M KOH) und gründlich waschen Küvetten mit dH 2 O.
- Bereiten Sie Arbeitsstation um Fluorometer mit mehrerenKisten von 10, 200 und 1000 ul Pipettenspitzen, einem Timer mit 10 Minuten Countdown, eine zugängliche Vakuumleitung mit Tipps für Küvetten - Reinigung, heikle Aufgabe Tüchern und Spritzflasche mit Reinstwasser H 2 O eingestellt
- Wärmebad externe Wasser für Fluorometer und Wärmeblock auf 37 ° C.
- Schalten Sie Fluorometers; Fluoreszenzerfassungsprogramm für GFP Sammlung Set 430 nm bis Anregung, Bandpass 8 nm; Emissionsbereich von 450 nm - 600 nm, Bandpass 4 nm. Für YFP Sammlung nur als Sensorsteuerung (siehe Diskussion) Satz Anregung auf 490 nm, Bandpass 8 nm, Emissionsbereich von 500 bis 600 nm, Bandpass 4 nm. GFP Sammlung Einstellungen werden verwendet, um ein FRET-Spektrum in diesem Experiment zu erwerben.
- Legen Sie zwölf 1,5 ml Mikrozentrifugenröhrchen in Wärmeblock , wie in Abbildung 2 gezeigt. Diese Rohre sind Halterungen für die Zell aliquoten Rohre (500 & mgr; l-Mikrozentrifugenröhrchen.) Legen Sie ein kleines Stück Gewebe in Haltern 1 und 7, um die Küvetten hier platziert abzufedern.
Hinweis: Verwenden Sie getrennte Küvetten für unbehandelten Zustand und Drogen Zustand eine Kreuzkontamination zu vermeiden.

Abbildung 2. Microcentrifuge Gefäß Einrichten und Position Referenz in Wärmeblock Cuvette für unbehandelten Proben ist in Position . 1; Zelle aliquoten Rohre sind in den Positionen 2 - 6. Cuvette für Proben Medikament behandelt in der Position 7; Zelle aliquoten Rohre sind in den Positionen 8 - 12. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
- Füllen Halter 2 bis 6 und 8 bis 12 mit ~ 750 & mgr; l Wasser mit einer mini 37 ° C Wasserbad zu schaffen.
- Legen Sie zehn 500 ul-Mikrozentrifugenröhrchen für Zellaliquote in Mini-Wasserbäder (Halter 2 - 6, 8 bis 12). Jedes Rohr wird eine einzelne Wiederholung der Bedingung (5 untreated, 5 Arzneimittel behandelt).
- Überwachen Sie Zellen für die Expression (siehe Schritt 3.6).
5. Experiment & Data Collection
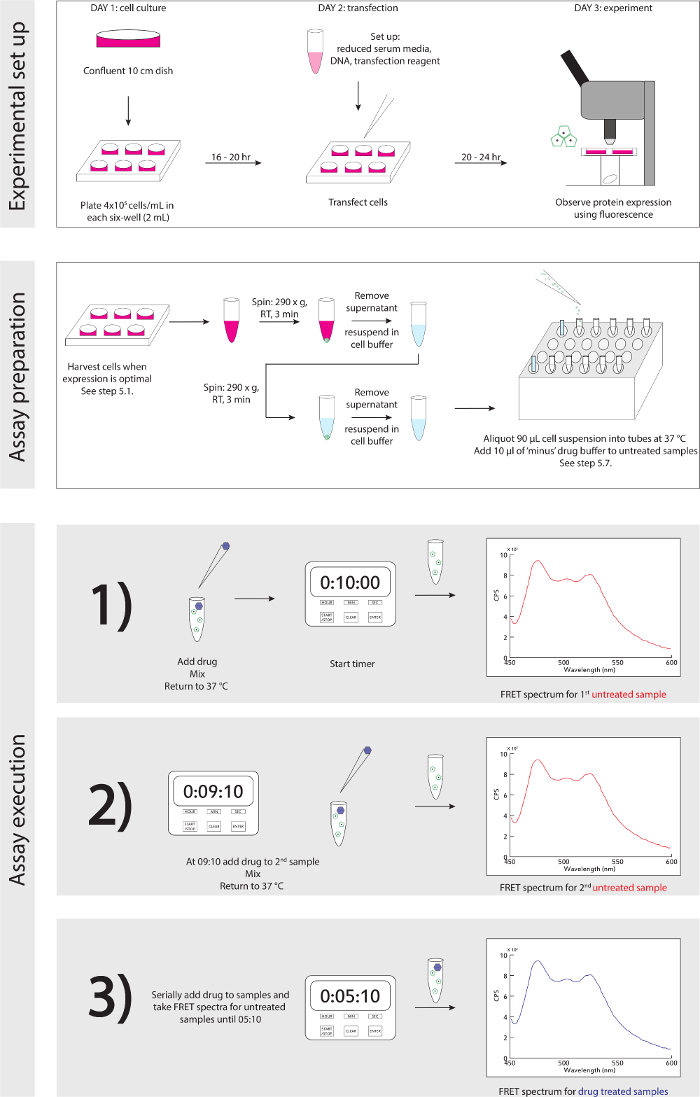
Abbildung 3. Experimentelle Schematic. Eine ausführliche schrittweise Anleitung für experimentelle Aufbau und Ausführung. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
- Referenz 3 für experimentelle Schema. Wenn Zellen bereit sind, geerntet werden, bezogen auf die Proteinexpression mit Fluoreszenzmikroskop nachgewiesen werden (siehe Diskussion, Absatz 4): in der Haube biologische Sicherheit, sanft entfernen ~ 1 ml Medium, resuspendieren Zellen in ihrer Kultur mit einem P1,000 und übertragen Aufwirbelung in ein 1,5 ml Mikrozentrifugenröhrchen.
Hinweis: Vermeiden unter Verwendung von Trypsin, wie es die N-Terminus verdauen und /oder Bindungstasche des GPCR-Sensor. - Zählen von Zellen geeignete Zelldichte in Resuspension zu gewährleisten. Optimieren Resuspensionsvolumen für 4 x 10 6 Zellen / ml.
- Spin-Zellen in Schwingbecherzentrifuge bei Raumtemperatur 290 g für 3 min. Überstand entfernen nach der Zentrifugation.
- GENTLY Zellen in 1 ml Zellpuffer (gelagert bei 37 ° C) und wiederholen Sie Schritt 5.3 resuspendieren. Während zweite Spin, sammeln 100 mM Medikament Lager aliquoten von -80 ° C. Machen Sie 1: 100 Verdünnung in Drug-Puffer für 1 mM Arbeits Lager und halten bei RT.
- Nach der zweiten Zentrifugation entfernen Überstand und vorsichtig in 1 ml der Zellenpuffer resuspendieren Zellen (4 x 10 6 Zellen / ml). Maßnahme OD 600 der Probe in dem Spektrophotometer unter Verwendung von 1 ml der Zellen und 1 ml der Zellenpuffer als Rohling. Dispense-Zellen in einer Einweg-Kunststoff-Küvetten und Transfer zurück zu einem Mikrozentrifugenröhrchen unmittelbar nach Spektrophotometrie.
- Für eine Kontrolle untransfizierten cell Zustand Spektrum, sanft untransfizierten Zellen in 1 ml Zellenpuffer mit P1,000 Pipette resuspendieren, fügen Sie 90 ul der Zellen FRET-Spektrum bei Anregung 430 nm, Bandpass 8 nm, Emission 450 bis Küvette und erwerben - 600 nm, Bandpass 4 nm. Sammeln Sie 3 bis 5 wiederholen Spektren mit frischen 90 & mgr; l der Zellen. Halten Sie Lager von Zellen bei 37 ° C zwischen specs, resuspendieren sanft mit P1,000 zwischen jedem Probenaliquothandhabungsvorrichtung und spülen Küvette mit Reinstwasser H 2 O zwischen den Proben.
- Für Versuchsbedingungen aliquote 90 ul von transfizierten Zellen zu jeder der 500 & mgr; l Röhrchen in Halter 2 bis 6, 8 bis 12 in dem Heizblock. resuspendieren vorsichtig Lager von Zellen mit P1,000 pipettieren zwischen jedem aliquoten Teil.
- Nachdem die Zellen aliquotiert werden, fügen Sie 10 ul Drug Buffer 2 Röhren - 6 für unbehandelten Zustand Proben.
- Beginnen Experiment durch Zugabe von 10 ul 1 mM Wirkstofflösung in das Rohr 8, starten Sie den Timer von 10 Minuten mit dem Countdown, und vorsichtig mischen Rohr mit P200 pipettieren. Schließen Rohr und Rückkehr zu 37° C Wärmeblock.
- Pick sofort bis Rohr 2, vorsichtig mischen mit P200 (eine neue Spitze verwenden), fügen Sie 90 ul der Zellsuspension in unbehandeltem Zustand Küvetten und in Fluorometer.
- Erwerben FRET-Spektrum bei Anregung 430 nm, Bandpass 8 nm, Emission 450 bis 600 nm, Bandpass 4 nm.
- Bei 9 min - 10 sec, Spike Rohr 9 mit 10 ul 1 mM Wirkstofflösung, vorsichtig mit einem P200 (verwenden Sie neue Spitze) mischen, und das Rückrohrblock zu erhitzen.
- Wiederholen Sie die Schritte 5,10-5,11 mit Rohr 3 und 5.12 mit Rohr 10.
- Wiederholen Sie die Schritte 5,10-5,13 in 1 - Minuten - Intervallen (8.10, 7.10, etc.) bis Spektren für alle unbehandelten Zustand Proben gesammelt werden, und Droge wurde auf alle Arzneimittel Zustand Proben hinzugefügt. Verwenden Sie eine frische Spitze für jeden Pipetten Schritt eine Kreuzkontamination zu verhindern.
- Bei 5 min - 10 sec, beginnen Mischrohr 8 (drug Zustand) sanft mit P200 pipettieren, fügen Sie 90 ul der Zellsuspension in getrennten Küvetten für Arzneimittel behandelten Probe und in Fluorometer.
- erwerben FRET-Spektrum (siehe Schritt 5.11 für Einstellungen).
- Wiederholen Sie die Schritte 5,15-5,16 in 1 - Minuten - Intervallen (04.10, 03.10, etc.) für die verbleibenden Arzneimittel Zustand Proben (Rohre 9 bis 12).
- Nach dem Ende des Tests Projektdateien zu speichern, gründlich Küvetten mit Reinstwasser H 2 O waschen und für den nächsten Zustand Röhren-Lager wieder. Achten Sie darauf, eine Kreuzkontamination in dem Waschschritt zu verhindern. Ändern Sie die Spitze auf dem H 2 O - Flasche sowie auf der Vakuumleitung zwischen Wäschen.
6. Datenanalyse
- Speichern und Exportieren von Daten-Dateien in SPC formatiert werden für die Analyse verwendet werden. Analyseprogramme sind zum Download auf der Sivaramakrishnan Lab Veröffentlichung Website zur Verfügung.
- Erstellen Sie Pfad-Dateien für Analyse-Software, die die Analyseprogramme (v9, v15) umfassen, untransfizierten Proben Dateien (siehe Schritt 5.6 für untransfizierten Zelle Spektrum-Sammlung), OUTPUT Datendatei, und durch Komma getrennte Werte (CSV) Dateien für die Dateneingabe.
- Geben Sie folgende Informationenin CSV - Datei und benennen jeweiligen Bedingungen für jede Probe (Probe in Tabelle 3 zu sehen), einschließlich:
Dateiname - individuelle SPC Graph-Dateien
Rezeptor - designierter Konstrukt , das GPCR wurde getestet (zB Β2)
Binder - bezeichnen , die Peptidvariante des Konstrukts wurde untersucht (beispielsweise S)
Agonist - bezeichnen unbehandelten (N) oder Arzneimittel behandelt (D) Bedingungen
Directory - der Pfad Ordner, in dem SPC-Dateien gespeichert werden, in der Regel nach Datum sortiert
OD - aufgezeichnet optische Dichte der Probe aus Spektrophotometer - Geben Sie Dateinamen für untransfizierten Proben (Schritt 5.6) Puffer zu subtrahieren und Streuungsrauschen von Proben.
- Geben Sie Bedingungen in Analyseprogramm.
- Ausführen von Programmen zu analysieren Proben innerhalb der einzelnen Bedingungen (v9) und über Bedingungen (v15).
- Ausschließen Beispieldateien, die offensichtlich Ausreißer im Datensatz sind, oder für die Subtraktion einstellen durch Erhöhen oder Verringern OD-Wert von indiviDual-Dateien.
- Exportieren von Daten in der Ausgabedatei für den Zugriff auf berechneten FRET-Verhältnisse (525 nm / 475 nm).
- Berechnen ΔFRET, indem die durchschnittliche FRET-Verhältnis für unbehandelten Zustand aus den einzelnen FRET-Verhältnisse für behandelt (Arzneimittel) Bedingungen abzieht.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Eine verallgemeinerte schematische Darstellung des Versuchsaufbau und die Ausführung ist in Abbildung 3 beschrieben.
Um eine FRET Änderung in dem schmalen Dynamikbereich des Sensors zu erfassen, ist es entscheidend für die Nuancen des Systems anhaften einzuhalten. Zellqualität ist zwingend notwendig , um die Proteinexpression sowie die Konsistenz bei der Probenahme. Abbildung 1 zeigt Bilder von kultivierten Zellen in einer konsistenten einschichtigen (10...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die enge Dynamikbereich von FRET-Messungen in diesem System verstärkt die Notwendigkeit, sensible Qualitätskontrolle bei jedem Schritt dieses Protokolls. Die wichtigsten Schritte ein erfolgreiches FRET Experiment sind 1) die Zellkultur, 2) Transfektion 3) Proteinexpression und 4) zeitnah, präzise Koordination während des Testausführung zu gewährleisten.
Zell Gesundheit und Pflege / Beschichtung Qualität kann einen erheblichen Einfluss auf das Signal-zu-Rausch des experimentellen Syste...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
RUM wurde von der American Heart Association Pre-Doc-Stipendium finanziert (14PRE18560010). Forschung wurde von der American Heart Association Scientist Entwicklung Grant (13SDG14270009) & NIH (1DP2 CA186752-01 & 1-R01-GM-105646-01-A1) finanziert SS
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| B2-AR-10 nm-Gas peptide sensor | Addgene | 47438 | https://www.addgene.org/Sivaraj_Sivaramakrishnan/ |
| GeneJET Plasmid Miniprep Kit | Fermentas/Fisher Sci | FERK0503 | Elute in 2 mM Tris elution buffer |
| HEK-293T-Flp-n cells | Life Technologies | R78007 | |
| Trypsin (0.25%) | Life Technologies | 25200056 | |
| DMEM- high glucose | Life Technologies | 11960-044 | Warm in 37 °C water bath before use |
| FBS, certified, Heat inactivated, US origin | Life Technologies | 10082147 | |
| Glutamax I 100x | Life Technologies | 35050061 | |
| HEPES | Corning | MT25060CL | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media; Bring to RT before use |
| XtremeGene HP transfection reagenet | Roche | 6366236001 | Highly recommended for its consistency. Bring to RT before use |
| FluoroMax 4 | Horiba | Use with FluorEssence V3.8 software | |
| 3-mm path length quartz cuvette | Starna | NC9729944(16.45F-Q-3/z8.5) | May require cuvette holder/adaptor for use in Fluorometer, available from Starna |
| Sc100-S3 Heated Circulating water bath pump | Fisher Scientific | 13-874-826 | Warm to 37 °C before use |
| Thermomixer Heat Block | Eppendorf | 22670000 | Warm to 37 °C before use |
| Ultrapure DNA/RNAse free water | Life Technologies | 10977015 | Use at RT |
| D(+)-glucose, anhydrous | Sigma | G5767 | |
| aprotinin from bovine lung | Sigma | A1153 | |
| leupeptin hemisulfate | EMD | 10-897 | |
| L-ascorbic acid, reagent grade | Sigma | A0278 | |
| (-)-isoproterenol (+)-bitartrate | Sigma | I2760 | Use fresh aliquot each experiment |
Referenzen
- Malik, R. U., et al. Detection of G Protein-selective G Protein-coupled Receptor (GPCR) Conformations in Live Cells. J. Biol. Chem. 288, 17167-17178 (2013).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat. Rev. Mol. Cell Bio. 9, 60-71 (2008).
- Rasmussen, S. G., et al. Crystal structure of the β2 adrenergic receptor-Gs protein complex. Nature. 477, 549-555 (2011).
- Alexander, N. S., et al. Energetic analysis of the rhodopsin-G-protein complex links the α5 helix to GDP release. Nat. Struct. Mol. Biol. 21 (1), 56-63 (2014).
- Conklin, B. R., Farfel, Z., Lustig, K. D., Julius, D., Bourne, H. R. Substitution of three amino acids switches receptor specificity of Gqα to that of Giα. Nature. 363, 274-276 (1993).
- Conklin, B. R., et al. Carboxyl-Terminal Mutations of Gqα and Gsα That Alter the Fidelity of Receptor Activation. Mol. Pharmacol. 50, 855-890 (1996).
- Onaran, H. O., Costa, T. Where have all the active receptor states gone. Nat. Chem. Bio. 8, 674-677 (2012).
- Jares-Erijman, E. A., Jovin, T. M. FRET imaging. Nat Biotechnol. 21 (11), 1387-1395 (2003).
- Lohse, M. J., Nuber, S., Hoffman, C. Fluorescence/bioluminescence resonance energy transfer techniques to study G-protein-coupled receptor activation and signaling. Pharmacol Rev. 64 (2), 299-336 (2012).
- Vilardaga, J. P., Bünemann, M., Krasel, C., Castro, M., Lohse, M. J. Measurement of the millisecond activation switch of G protein-coupled receptors in living cells. Nat. Biotechnol. 21, 807-812 (2003).
- Bünemann, M., Frank, M., Lohse, M. J. Gi protein activation in intact cells involves subunit rearrangement rather than dissociation. Proc. Natl. Acad. Sci. USA. 100 (26), 16077-16082 (2003).
- Stumpf, A. D., Hoffman, C. Optical probes based on G protein-coupled receptors - added work or added value. Brit. J. Pharmacol. 173, 255-266 (2016).
- Sivaramakrishnan, S., Spudich, J. A. Systemic control of protein interaction using a modular ER/K α-helix linker. Proc. Natl. Acad. Sci. USA. 108, 20467-20472 (2011).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2 (12), 905-909 (2005).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten