Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Durchflusszytometrische Analyse von natürlichen Killerzellen lytische Aktivität in menschlichem Vollblut
In diesem Artikel
Zusammenfassung
This work describes an advanced workflow for the accurate and fast determination of NK (Natural Killer) cell count and NK cell cytotoxicity in human blood samples.
Zusammenfassung
NK-Zell-Cytotoxizität ist eine weit verbreitete Maßnahme, um die Wirkung der Intervention von außen auf NK-Zell-Funktion zu bestimmen. Jedoch kann die Genauigkeit und Reproduzierbarkeit des Assays instabil betrachtet werden, entweder weil der Benutzer-Fehler oder wegen der Empfindlichkeit von NK-Zellen auf experimentelle Manipulation. Um diese Probleme zu beseitigen, wird ein Workflow, der sie auf ein Minimum reduziert wurde etabliert und wird hier vorgestellt. Zu veranschaulichen, erhalten wir Blutproben zu verschiedenen Zeitpunkten, von Läufern (n = 4), die zu einem intensiven Trainingseinheit vorgelegt wurden. Zunächst werden NK-Zellen gleichzeitig identifiziert und isoliert durch CD56-Tagging und magnetische-basierte Sortierung, direkt aus Vollblut und von so wenig wie ein Milliliter. Die sortierten NK-Zellen sind von irgendwelchen Reagenz oder Capping-Antikörper entfernt. Sie können, um gezählt werden, eine genaue NK-Zellzahl pro Milliliter Blut zu schaffen. Zweitens können die sortierten NK-Zellen (Effektoren Zellen oder E) mit 3,3'-Diotade gemischt werdencyloxacarbocyanine Perchlorat (DIO) markierte K562-Zellen (Zielzellen oder T) bei einem Test-optimal 1: 5 T: E-Verhältnis, und analysiert, um eine Bildgebungs Durchflusszytometer verwenden, die für die Visualisierung jedes Ereignisses und die Beseitigung aller falschen positiven erlaubt oder falsch-negative Ergebnisse (wie Dubletten oder Effektor-Zellen). Dieser Arbeitsablauf kann in etwa 4 h vollendet werden, und ermöglicht eine sehr stabile Ergebnisse, selbst wenn sie mit menschlichen Proben arbeiten. Wenn verfügbar, Forschungsteams können mehrere experimentelle Eingriffe am Menschen zu testen und zu vergleichen, Messungen über mehrere Zeitpunkte, ohne die Datenintegrität zu beeinträchtigen.
Einleitung
Natürliche Killerzellen sind ein wesentlicher Bestandteil des angeborenen Immunsystems. Während sie sehr geregelt werden, haben sie die Fähigkeit , zu erkennen und abnormalen Zellen durch Zell-zu-Zell - Kontakt und ohne vorherige Aktivierung 1 beseitigen. Als solche bilden sie eine schnelle Verteidigungslinie gegen Infektionen. Exercise, besonders intensiv, wurde gezeigt , dass die transient das Immunsystem 2, 3, 4, 5 drücken. NK - Zellen sind besonders anfällig für diese Wirkung 4, 6, 7, effektiv ein Fenster mit erhöhter Empfindlichkeit gegenüber Krankheiten zu schaffen. Daher, bevor die Untersuchung von Interventionen, während oder nach intensivem Training mit dem Ziel, ihre Auswirkungen zu verringern auf NK-Zell-Funktion ist von besonderem Interesse für das Wohlbefinden der Athleten post-Wettbewerben.
Allerdings ist die Untersuchung solcher Eingriffe durch zahlreiche Faktoren erschwert: 1) NK - Zellen sparse sind 8, bei etwa 1% der weißen Blutkörperchen Fach; 2) NK-Zellen sind sehr empfindlich gegenüber Stress und verlassen sich auf ständige Exposition gegenüber physiologischen Bedingungen während des Experimentierens lebensfähig und stabil zu bleiben; und 3) Standardassays NK - Zell - Cytotoxizität zu bestimmen, wie beispielsweise Ficoll - Gradienten 9 und Freisetzungsassays 10, sind unzuverlässig und inkonsistent. Die inhärente Variabilität der menschlichen Proben nur diese Fragen Verbindung. Frischen menschlichen Proben bei Eingriffen gesammelt sind ziemlich geregelten und schwer zu beschaffen, zumindest im Vergleich zu den Tierproben oder immortalisierten Zelllinien. Dies reduziert die Möglichkeiten Experimente zu wiederholen, oder fügen Sie die Teilnehmer der Studie Kohorte signifikanten statistischen Schwellenwerte zu erreichen. Zusammengefasst unterstützen diese Fragen die Notwendigkeit für eine optimierte Protokoll, das fo erlaubtr beide Hochdurchsatz und eine Hochzuverlässigkeitsanalyse von NK-Zell-lytische Aktivität in menschlichen Proben.
Wir haben ein Workflow, der die notwendige Zeit zu identifizieren, zu isolieren und zu testen NK-Zellen aus menschlichem Vollblut verkürzt, während der Exposition gegenüber äußeren Faktoren zu minimieren. Das Verfahren optimiert die Verwendung von zwei Instrumenten, eine Magnetbasis Zellsortierer und eine Abbildungs Durchflusszytometer und eine Assay-spezifische, optimierte T: E-Verhältnis die Detektion abnimmt oder zunimmt von NK-Zell-Cytotoxizität zu ermöglichen.
Protokoll
HINWEIS: Alle Blutsammelverfahren wurden in Übereinstimmung mit den Richtlinien durchgeführt von der Appalachian State University (ASU) Institutional Review Board (IRB) dargelegt.
1. Vollblut-Sammlung
- Haben Sie einen zertifizierten phlebotomist ziehen Blut nach Angaben der Weltgesundheitsorganisation Richtlinien.
- Zeichnen Sie Blut in ein 4 ml Blutentnahmeröhrchen , das Di-Kalium Ethylendiamintetraessigsäure (K 2 EDTA). Invert-Blutsammelröhrchen nach den Anweisungen des Herstellers. Halten Sie Blutentnahmeröhrchen bei Raumtemperatur auf einem Benchtop-Rocker bis zur Trennung.
2. Vorbereitung der OVI-markierten Zielzellen
- Wachsen K562-Zellen in Komplett Iscove modifiziertem Dulbecco-Medium (IMDM), ergänzt mit 10% fötalem Rinderserum (FBS) und 1% Penicillin-Streptomycin mehrere Wochen lang vor dem Assay auf die Gesundheit der Zellen zu gewährleisten. Stellen Sie die KonzentratIonen der Zellen bis 3 x 10 5 Zellen / mL der Fertigstellung einer Zelle täglich durch Zählen und anschließende Passage von Zellen. Führen Sie eine vollständige Medienwechsel alle 2 bis 3 Tage.
- Am Tag des Tests, führen Sie eine 1 ein Zellenzahl: 1 - Hämozytometer.
- Entfernen 10 & mgr; l aus dem K562 Kolben aufnehmen und in 1,5 ml-Röhrchen.
- Zugabe von 10 ul Trypanblau-Farbstoff in der gleichen 1,5-ml-Röhrchen für einen Verdünnungsfaktor von 1: 1.
- Sanft flick Rohr K562-Zellen und Trypanblaufarbstoff zu mischen.
- Erlauben K562 Zellen Trypanblau für 1 min bei Raumtemperatur inkubieren.
- Entfernen 15 & mgr; l von gefärbten K562-Zellen aus dem Rohr.
- Pipette auf Hemocytometer für Zellzahl.
- Zählen von Zellen in den vier Eckquadrate sowie den mittleren Platz für insgesamt fünf Plätzen.
- Berechnen Zellzahl unter Verwendung der Gleichung:
Insgesamt Zellen / ml = Gesamt Zellen gezählt X (Verdünnungsfaktor / Anzahl der Quadrate) X 10000 / mL
- Resuspendieren K562 - Zielzellen zu einer endgültigen Dichte von 1 x 10 6 Zellen / ml in serumfreiem IMDM - Kulturmedium.
- Für ungefärbte K562 - Zielzellen, 10 ml von K562 - Zielzellen zu einem 15 ml - Röhrchen in einer Dichte von 1 x 10 6 Zellen / ml für insgesamt 10 x 10 6 K562 - Zellen. In eine 37 ° C - Inkubator mit 5% CO 2 bis zur Verwendung.
- Dio gefärbten K562 - Zielzellen, mit 10 ml K562 - Zellen zu einem 15 ml - Röhrchen mit einer Dichte von 1 x 10 6 Zellen / ml für insgesamt 10 x 10 6 K562 - Zellen abzielt.
- 1 & mgr; l der OVI Zellmarkierungslösung pro ml Zellsuspension in 15-ml-Röhrchen Dio-Färbung bezeichnet und sanft Wirbel. Beispielsweise 10 & mgr; l Lösung DiO Zellmarkierungs bis 10 x 10 6 K562 Zellen / ml in einem Volumen von 1 x 10 6 Zellen / ml hinzu.
- Inkubieren K562-DIO - Lösung für 20 min bei 37 ° C mit 5% CO 2 in einem 15 ml Röhrchen.
- Folgening Inkubation 3 ml Raumtemperatur phosphatgepufferter Salzlösung (PBS) auf K562-DIO-Lösung 15 ml Röhrchen mit.
- Zentrifuge für 10 min bei 135 xg bei Raumtemperatur.
- Entfernen Sie vorsichtig Überstand, ohne mit einem 1000 ul Volumen verstellbare Pipette das Zellpellet zu stören.
- In 10 ml frischem IMDM zu Zellpellet enthaltenden 15 ml-Röhrchen.
- Sanft Wirbelrohr um die Zellen zu resuspendieren.
- Wiederholen Sie die Schritte 2,7-2,10 zwei weitere Male.
- Store - Zellen in einem 37 ° C - Inkubator mit 5% CO 2 bis zur Verwendung.
HINWEIS: Die Zellen können bis zu 24 h im Brutschrank gelagert werden, aber es ist bevorzugt, sie am selben Tag zu verwenden.
3. Herstellung von Kontrollen
- Übertragen Sie die folgenden in getrennte und in geeigneter Weise 1,5 - ml - Röhrchen beschriftet:
- Add 500 ul frischem IMDM, enthaltend erneut suspendiert DiO-markierte K562-Zellen in den "Double Positive" bezeichnet 1,5 ml-Röhrchen.
- In 500 ml frisches IMDM, enthaltend erneut suspendiert DiO-markierte K562-Zellen in den "DiO nur" etikettiert 1,5 ml-Röhrchen.
- In 500 ul frischem IMDM enthält, erneut suspendiert unmarkierten K562-Zellen in den "Propidiumiodid (PI) nur" etikettiert 1,5 ml-Röhrchen.
- Der doppelte Positive und PI nur Röhrchen in einem 55 ° C Wasserbad für 5 min.
- Nach dem 5 Minuten verstrichen ist, entfernen Sie Rohre und wischen Sie mit 70% Ethanol nach unten.
- In 10 l PI an das Doppel Positive und PI nur 1,5 ml-Röhrchen.
- Platzieren alle drei K562 Zielzelle steuert in dem Inkubator bei 37 ° C für 30 min.
- Nach der 30 min Inkubation Ablauf Zentrifuge alle Steuerelemente Zielzelle K562 drei für 2 min bei 163 x g.
- Entfernen Sie vorsichtig Überstand ohne das Zellpellet zu stören.
- Resuspendieren jede Steuerung mit 20 & mgr; l frischem IMDM Zellkulturmedien, und lassen Sie in der 37 ° C - Inkubator mit 5% CO 2 für mindestens30 min für eine optimale DiO Signal.
HINWEIS: Die Kontrollen sind nun bereit, cytometer durch die Abbildungsfluss ausgeführt werden.
4. NK-Zellen Automatisierte Trennung
- Schalten Sie Zellseparator und lassen Sie den Start-up-Zyklus zu beenden.
- Stellen Sie sicher, dass alle Flüssigkeit Flaschen Illuminationen sind grün und dass die Abfallflasche ist leer.
- Besorgen Sie sich eine Raumtemperatur 15 ml-Röhrchen-Rack.
HINWEIS: Die Auswahl basiert auf Probenvolumina. Zum Beispiel für ein Volumen von weniger als 3 ml verwenden, um ein 15 ml Röhrchen Zahnstangen- und für ein Volumen von mehr als 3 mL verwenden, um eine 50 ml-Röhrchen Rack. - Label-Probenröhrchen entsprechend (Wiederholung pro Probe / Teilnehmer): Teilnehmer 1 Vollblut-Probe; Teilnehmer 1 Negativ-Fraktion; Teilnehmer 1 Positive Fraktion.
- Pipette vorsichtig 1,5 ml Vollblut aus Schritt 1.2 in "Vollblut-Probe" 15 ml-Röhrchen.
- Legen Sie entsprechend markierten 15 ml "Vollblut-Probe" Röhrchen aus Schritt 4.5 und markierte 15 ml "Negative Fraktion" und"Positive Fraktion" Rohre aus Schritt 4.4 in der Rohrgestell. Verwenden Sie thefollowing Probenrack Set-up: Reihe (R1) A: Vollblut-Probe, Reihe (R2) B: Negativ, unmarkierten Fraktion, Reihe (R4) C: Positiv, magnetisch markierten Fraktion.
- Legen Sie die Separator-Rack auf den Minisampler für autolabeling.
- Wählen Sie "Reagenz" auf dem Menü und markieren Sie die Position, wo das Fläschchen wird in dem Separator Rack platziert werden.
- Wählen Sie "Read-Reagenz", um den 2D-Code-Leser zu aktivieren.
- Legen Sie das entsprechende Reagenzabfüllung vor dem 2D-Code-Leser zwischen 0,5-2,5 cm von dem Codeleser Abdeckung.
HINWEIS: Zum Beispiel ist die erforderliche Reagenz für NK Zelltrennung CD56. - Halten Reagenzabfüllung in einem Winkel vor 2D-Code-Leser für die optimale Lese.
- Legen Sie die Reagenzabfüllung in die richtige Trennzeichen Rackposition.
- Markieren Sie die gewünschten Positionen unter der Trennung Registerkarte auf dem Bildschirm.
- Aus dem Labeling Untermenü zuweisen ein Autolabeling Programm.
- die Zellseparation Reagenz CD56 MicroBeads Weisen Positionen Rack 1, 2, 3 und 4.
- Wählen Sie den "posselwb" Trennung Programm.
- Wählen Sie den "spülen" Waschprogramm.
- Legen Sie ein Probenvolumen von 1.500 & mgr; l in der "Lautstärke" Untermenü mit der Zifferntastatur.
- Wählen Sie die "Enter" auf der Tastatur.
- Wählen Sie "Ausführen", um die Zelltrennung starten.
- Wählen Sie "OK", um zu bestätigen, dass genügend Puffer für die Bearbeitung aller Proben zur Verfügung steht.
5. NK Cell Count Nach Zellseparation
- Unmittelbar im Anschluss an die Zelltrennung mit dem Zellseparator, rufen Sie die positive Fraktion. Lassen Sie bei Raumtemperatur. Diese Fraktion enthält die gewünschten reinen NK-Zell-Population.
- Für jede einzelne Probe, führen Sie eine Zellzahl einer Zählkammer gemäß Schritt 2.2 verwendet wird .
- Nach Berechnungen, die Zellenzahl aufzuzeichnen.
6. Zytotoxizitätstest Probenvorbereitung
- Bereiten Sie und beschriften entsprechend 1,5 ml Röhrchen für jede Probe / Teilnehmer.
- Pipette gewünschte Verhältnis von NK-Zellen und DiO-labled K562-Zellen in jedes Röhrchen.
HINWEIS: Zum Beispiel ist das gewünschte Verhältnis von K562-Zielzellen und Effektorzellen NK 1: 5. - Zentrifuge für 5 Minuten bei 135 x g.
- Entfernen Sie vorsichtig Überstand ohne das Zellpellet zu stören.
- Resuspendieren NK-DiO markierten K562 Zellgemisch in 500 & mgr; l von NK-Zell-Medium ohne Interleukin-2 (IL-2) und 2-Mercaptoethanol (2-ME) (incomplete NK Zellkulturmedien).
HINWEIS: Die unvollständige NK Zellkulturmedien ist, Minimum Essential Medium Eagle mit Natriumbicarbonat, ohne L-Glutamin, Ribonukleosiden und Desoxyribonukleosiden. - Werden 5 ul PI zu jedem Röhrchen.
- Zentrifuge für 2 min bei 163 x g.
- Inkubiere Zellen bei 37 ° C für 2 h.
- Nach 2 h Inkubation centrifuge für 2 min bei 163 x g.
- Entfernen Sie vorsichtig Überstand ohne das Zellpellet zu stören.
- Resuspendiere Zellen in 25 ul unvollständiges NK Zellkulturmedien.
7. Herstellung von Spontaneous ( "S") Probe
- Pipette 500 ul DiO-markierte K562 - Zellen (Konzentration von 1 x 10 6 Zellen / ml) in 1,5 ml - Röhrchen.
- Zugabe von 10 & mgr; l von PI zu jedem Röhrchen.
- Zentrifugenröhrchen für 2 min bei 163 x g.
- Inkubiere Zellen bei 37 ° C für 2 h.
- Folgenden 2 h Inkubation Zentrifuge für 2 min bei 163 x g.
- Entfernen Sie vorsichtig Überstand ohne das Zellpellet zu stören.
- Die Zellen in 25 ul unvollständig alpha-Minimum Essential Medium (α-MEM) Zellkulturmedien.
8. Datenerfassung mit Imaging Durchflusszytometer
- Drücken Sie die grüne Taste in der vorderen Tür des Abbildungs Durchflusszytometer auf das Gerät einzuschalten.
- Schalten Sie all Computer mit der Imaging-assoziierten Durchflusszytometer.
- Starten Sie die Imaging-Durchflusszytometer Software.
- Klicken Sie auf die "Inbetriebnahme", um das System zu spülen und die Probenleitung vorzubereiten.
- Sobald die "Inbetriebnahme" abgeschlossen ist, schließen Sie das "Calibra" Fenster.
- Weisen Sie Kanäle: auf der oberen linken Seite, klicken Sie auf jedem Kanal, um sie zu vergeben.
- Auf der rechten Seite klicken Sie auf den Streudiagramm-Taste 4 Scatterplots zu erstellen: Rohmaterial Max Pixel _MC_6 vs Area_M06, Rohmaterial Max Pixel _MC_2 vs Area_M02, Rohmaterial Max Pixel _MC_5 vs Area_M05 und FieldArea_M01 vs AspectRatio_M01.
- Beginnen Analyse von Proben , indem zuerst die "Double Positive" Steuerung.
- Klicken Sie auf "Laden".
- Legen Sie die 1,5-ml-Röhrchen mit dem "Double Positive" Probe aus den Schritten von 3,4 bis 3,9 in den Probenlader.
- Wählen Sie das 40X-Objektiv unter der "Vergrößerung" aus.
- Schalten Sie die 405 mW und642 mW Laser.
- Schalten Sie das "Hellfeld" Kanal.
- Klicken Sie auf "Wählen Sie Intensität."
- Basierend auf dem "Double Positive" Kontrollprobe, bestimmen die gewünschte Intensität für die 405 mW Laser so dass der Detektor nicht überlastet wird.
Hinweis: Zum Beispiel wurde bei 11 mW die optimale Intensität für dieses Experiment eingestellt.
- Nachdem die gewünschte Set-up erreicht, Daten erfassen.
- Im Rahmen der "Acquisition Datei", geben Sie in einem benutzerdefinierten Dateinamen Text. Wählen Sie einen Ordner zum Speichern der Datendatei (en).
- Geben Sie die Anzahl der Zellen neben "Collect" zu erwerben. Typischerweise variiert diese Zahl zwischen 1.000 bis 10.000.
- Klicken Sie auf "Acquire".
HINWEIS: Wenn die gewünschte Anzahl von Zellen gewonnen wird, wird die Datendatei in dem zuvor ausgewählten Ordner automatisch gespeichert.
- Nach dem Erwerb abgeschlossen ist , legen Sie die nächste Kontrollprobe - DiO nur Kontrolle.
- Klicken Sie auf "Laden".
- Legen Sie die 1,5-ml-Röhrchen mit dem "DiO nur" Probe in die Probenzuführung.
- Lassen Sie das 40X-Objektiv unter der "Vergrößerung" Registerkarte ausgewählt.
- Lassen Sie das 405-mW-Laser eingeschaltet.
- Schalten Sie den 642-mW-Laser und das "Hellfeld" Kanal.
HINWEIS: Nun, da die gewünschte Set-up erreicht wurde, können die Daten erfasst werden. - Unter dem "File Acquisition", geben Sie in einem benutzerdefinierten Dateinamen Text und wählen Sie die Datendatei einen Ordner für das Speichern (s). Geben Sie die Anzahl der Zellen neben erwerben "sammeln.". Typischerweise ist diese Zahl 1000.
- Klicken Sie auf "Acquire".
HINWEIS: Wenn die gewünschte Anzahl von Zellen, die Datendatei automatisch in dem zuvor ausgewählten Ordner gespeichert wird, erfasst wird.
- Wiederholen Sie Schritt 8,10 für die "PI nur" Kontrollprobe. Die experimentellen Proben sind nun bereit, gesammelt werden.
- Behandeln Sie die remaining experimentellen Proben, einschließlich der "Spontane" S "Samples" wie folgt:
- Lassen Sie das 40X-Objektiv unter der "Vergrößerung" Registerkarte ausgewählt.
- Schalten Sie die 405 mW und 642 mW Laser.
- Schalten Sie die "Hellfeld" Kanal.
- Klicken Sie auf "Set Intensität."
- Unter dem "File Acquisition", geben Sie in einem benutzerdefinierten Dateinamen Text und wählen Sie die Datendatei einen Ordner für das Speichern (s). Geben Sie in einem benutzerdefinierten Dateinamen Text.
- Geben Sie die Anzahl der Zellen zu erwerben neben "sammeln."
- Klicken Sie auf die Schaltfläche "Erfassen".
HINWEIS: Wenn die gewünschte Anzahl von Zellen, die Datendatei automatisch in dem zuvor ausgewählten Ordner gespeichert wird, erfasst wird.
- Wiederholen Sie Schritt 8.12 für alle experimentellen Proben.
- Nachdem alle experimentellen Daten und Dateien gesammelt wurden, klicken Sie auf die Schaltfläche "Shutdown" um das System zu sterilisieren.
9. Imaging Durchflusszytometer Probenanalyse
- Öffnen Sie die Imaging-Durchflusszytometer-Analyse-Software-Anwendung.
- Unter "Datei", öffnen Sie eine experimentelle .RIF Datei.
- Erstellen Sie eine neue Matrix, die die einzelnen Farb .RIF Dateien mit (DIO-only steuern und PI-only-Steuerung, erstellt während der Schritte 8.10 und 8.11), indem Sie "Erstellen einer neuen Matrix" unter der Kompensation Registerkarte in der Abbildungs Durchflusszytometer Software.
HINWEIS: Die Software fordert für die Auswahl der einzelnen Farbdateien und kombiniere sie mit einer Matrix-Datei (.ctm Dateierweiterung) zu erstellen, dh ausgewählt werden Kanalkompensation anzuwenden. - Erstellen Sie Dot - Plots unter Verwendung der "Bausteine" Funktion der Software.
- Erstellen Sie ein Punkt-Plot (BrightFieldGradient_RMS vs Frequenz) die fokussierten Zellen Tor. Rufen Sie das Tor "Focus" (Abbildung 1A).
- den "Focus" Tor benutzen, ein Punkt-Diagramm von Bright Field Bereich vs Hell Feld Seitenverhältnis g schaffen aßen die Singulett-Zellen. Rufen Sie das Tor "Single" (1B).
- Mit dem "Single" Tor, erstellen Sie ein Punktdiagramm von Intensity_MC_Ch02 vs Intensity_MC_Ch5. Verwenden Sie diese Handlung zu identifizieren und Gate DiO-positive Zellen nur (Ziele, lebendig) und PI-DIO doppelt positiven Zellen (Targets, tot) (1C).
HINWEIS: Alle Grundstücke in den Schritten 9.4.1, 9.4.2 und 9.4.3 kann unter Verwendung der "Bausteine" Funktion der Software erstellt werden.
- Klicken Sie auf die Statistik-Funktion des Dot-Plot zu Zellzahlen von jedem Gate zugreifen.
- Berechnen Sie den Prozentsatz der toten Ziele in der spontanen Probe und experimentellen Proben unter Verwendung folgender Formel:
% Tote Ziele in Probe = (#dead Ziele x 100) / (# leben Ziele + #dead Ziele) - Berechnen Zytotoxizität unter Verwendung der folgenden Formel:
% Zytotoxizität = [(Experimental dead-Spontane tot) / (100-Spontane tot)] x100
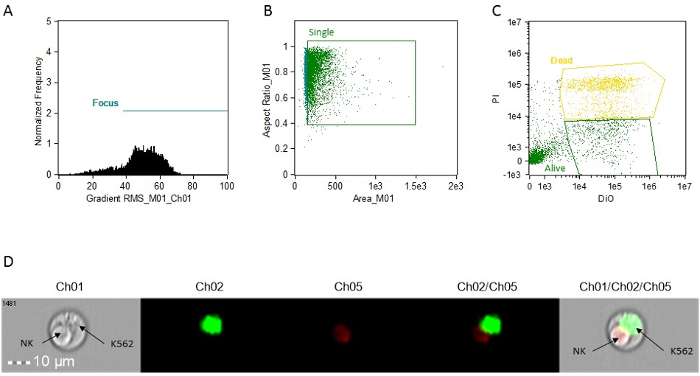
Abbildung 1: Repräsentative Histogramme, Streudiagramme und Bilder für zytotoxische Aktivität Analyse. (A) Fokus Zellanalyse. (B) Einzelzellanalyse. (C) Zielzellfärbung Analyse. Alle Bestimmungen werden unter Verwendung des Bildes zu jedem Ereignis angebracht gemacht. Dies kann durch einfaches Klicken auf die Veranstaltung in den Diagrammen in der Analyse-Software zugegriffen werden. (D) repräsentatives Bild eines Dubletts Ereignis, eine apoptotische NK - Zellen und eine Live - K562 Ziel zeigt. Ch01, Hellfeld. Ch02, OVI. CH05, PI. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Ergebnisse
Bestimmung der NK-Zellzahl
Die Wirkung von schweren Lauf auf NK - Zellzahl im Vollblut wurde gemessen, die Übung Protokoll in Abbildung 2 beschrieben ist . Blutproben wurden vor dem Training gezogen, unmittelbar nach dem Training, 1,5 Stunden nach dem Training, und schließlich 24 und 48 Stunden nach der ersten Blutentnahme. Die Konzentration von NK - Zellen pro Milliliter Vollblut wurde für jede Kufe (3A) gemessen und im Schnitt (Figur ...
Diskussion
Das Verfahren in dieser Studie beschriebenen Maßnahmen direkt auf die spezifische Aktivität eines NK-Zellen des Individuums als Reaktion auf Reize (in diesem speziellen Fall längerer Übung). Typischerweise werden NK-Zellen aus einem das Blut unter Verwendung von Dichtegradienten oder eine Zelle durch die Verwendung einer Kombination von Markern Sortierung isoliert. Obwohl diese Verfahren weit verbreitet sind, haben sie viele Nachteile: sie zeitraubend sind, beinhalten mehrere Manipulationen und sind stark Benutzer a...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This project was supported by Agriculture and Food Research Initiative Competitive Grant no. 2100-68003-30395 from the USDA National Institute of Food and Agriculture.
Materialien
| Name | Company | Catalog Number | Comments |
| K-562 lymphoblasts | ATCC | CCL-243 | |
| Iscove's Modified Dulbecco's Media | ATCC | 30-2005 | High glucose, with L-Glutamine, with HEPES, Sterile-filtered |
| Alpha Minimum Essential medium | ATCC | CRL-2407 | Without ribonucleosides and deoxyribonucleosides but with 2 mM L-glutamine and 1.5 g/L sodium bicarbonate |
| Trypan Blue Solution 0.4% | Amresco | K940-100ML | Tissue culture grade |
| Propridium Iodide Staining Solution | BD Pharmingen | 51-66211E | |
| Vybranto DiO cell-labeling solution | Vybranto | V-22886 | |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | |
| autoMACS Running Buffer | Miltenyi Biotec | 130-091-221 | |
| autoMACS Washing Buffer | Miltenyi Biotec | 130-092-987 | |
| autoMACS Columns | Miltenyi Biotec | 130-021-101 | |
| Whole Blood CD56 MicroBeads, human | Miltenyi Biotec | 130-090-875 | |
| ImageStream X Mark II Imaging Flow Cytometer | EMD Millipore | ||
| Speedbeads | Amnis Corporation | 400030 | |
| 0.4-0.7% Hypochlorite (Sterilizer) | VWR | JT9416-1 | |
| Coulter Clenz | Beckman Coulter | 8546929 | |
| 70% Isopropanol (Debubbler) | EMD Millipore | 1.3704 | |
| D-PBS (Sheath fluid) | EMD Millipore | BSS-1006-B (1X) | No calcium or magnesium |
| INSPIRE Software | EMD Millipore | Version Mark II, September 2013 | |
| Ideas Application Software | EMD Millipore | Version 6.1, July 2014 |
Referenzen
- Cerwenka, A., Lanier, L. L. Natural killer cells, viruses and cancer. Nat Rev Immunol. 1 (1), 41-49 (2001).
- Nieman, D. C. Exercise, infection, and immunity. Int J Sports Med. 15, 131-141 (1994).
- Romeo, J., Warnberg, J., Pozo, T., Marcos, A. Physical activity, immunity and infection. Proc Nutr Soc. 69 (3), 390-399 (2010).
- Nieman, D. C. Marathon training and immune function. Sports Med. 37 (4-5), 412-415 (2007).
- Simpson, R. J., Kunz, H., Agha, N., Graff, R. Exercise and the Regulation of Immune Functions. Prog Mol Biol Transl Sci. 135, 355-380 (2015).
- Walsh, N. P., et al. Position statement. Part one: Immune function and exercise. Exerc Immunol Rev. 17, 6-63 (2011).
- Timmons, B. W., Cieslak, T. Human natural killer cell subsets and acute exercise: a brief review. Exerc Immunol Rev. 14, 8-23 (2008).
- Westermann, J., Pabst, R. Distribution of lymphocyte subsets and natural killer cells in the human body. Clin Investig. 70 (7), 539-544 (1992).
- Boyum, A. Isolation of leucocytes from human blood. Further observations. Methylcellulose, dextran, and ficoll as erythrocyteaggregating agents. Scand J Clin Lab Invest Suppl. 97, 31-50 (1968).
- McMillan, R., Scott, J. L. Leukocyte labeling with 51-Chromium. I. Technic and results in normal subjects. Blood. 32 (5), 738-754 (1968).
- Berk, L. S., et al. The effect of long endurance running on natural killer cells in marathoners. Med Sci Sports Exerc. 22 (2), 207-212 (1990).
- McAnulty, L. S., et al. Effect of blueberry ingestion on natural killer cell counts, oxidative stress, and inflammation prior to and after 2.5 h of running. Appl Physiol Nutr Metab. 36 (6), 976-984 (2011).
- Millard, A. L., et al. Brief Exercise Increases Peripheral Blood NK Cell Counts without Immediate Functional Changes, but Impairs their Responses to ex vivo Stimulation. Front Immunol. 4, 125 (2013).
- Janeway, C. A., Travers, P., Walport, M., Shlomchik, M. J. . Immunobiology: The Immune System in Health and Disease. 5th edn. , (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten