Method Article
3D-Drucktechnologie Verwendung MRI mit Histologie fusionieren: Ein Protokoll für Hirn Sectioning
In diesem Artikel
Zusammenfassung
Das übergeordnete Ziel dieses Protokolls ist es, genau zu der Magnetresonanztomographie (MRT) Bildvolumen mit Histologieschnitten über die Erstellung von kundenspezifischen 3D-gedruckten Gehirn Halter und Slicer Boxen ausrichten.
Zusammenfassung
Magnetic resonance imaging (MRI) allows for the delineation between normal and abnormal tissue on a macroscopic scale, sampling an entire tissue volume three-dimensionally. While MRI is an extremely sensitive tool for detecting tissue abnormalities, association of signal changes with an underlying pathological process is usually not straightforward. In the central nervous system, for example, inflammation, demyelination, axonal damage, gliosis, and neuronal death may all induce similar findings on MRI. As such, interpretation of MRI scans depends on the context, and radiological-histopathological correlation is therefore of the utmost importance. Unfortunately, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, thus complicating the comparison between histology sections and MRI. This article presents novel methodology for accurately sectioning primate brain tissues and thus allowing precise matching between histology and MRI. The detailed protocol described in this article will assist investigators in applying this method, which relies on the creation of 3D printed brain slicers. Slightly modified, it can be easily implemented for brains of other species, including humans.
Einleitung
In vivo MRI provides a noninvasive and sensitive measure of tissue integrity at the macroscopic level. Changes in MRI signal intensity seen in vivo are outcome measures in many ongoing clinical trials.1 While the intensity changes seen via MRI can identify areas of abnormality in the context of the whole brain, they are often not sufficiently specific to differentiate pathological processes. This is especially true of dynamic processes involving multiple pathologies. For example, in multiple sclerosis (MS) or its animal model, experimental autoimmune encephalomyelitis (EAE), inflammation, edema, myelin degradation, axonal destruction, gliosis, and neuronal death overlap. 2, 3 To obtain the necessary specificity regarding the underlying pathology, context must be taken into account, together with knowledge of the histology of the MRI-identified abnormal tissues.
However, even in well-controlled animal experiments, matching histology with in vivo MRI is fundamentally challenging for various reasons. First, the difference in dimensional scales between histology sections and MRI is of several orders of magnitude.4 Second, for proper comparison, the orientation of MRI slice plane must match the sectioning plane of the brain tissue when cut. Due to the shape of the brain, it is very difficult to make consistently straight and accurate cuts when the brain is sitting on a flat surface. Third, the large size of the brain relative to a potentially small area of interest (lesion, tumor, etc.) creates a "needle-in-a-haystack" scenario for the pathologist processing the tissue. Fourth, even when the target tissue is found, it is commonly processed in such a way as to render virtually impossible an association with the original MRI data. Finally, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, further complicating the comparison between histology sections and MRI images.
Previous attempts to overcome these challenges relied on the use of deformational algorithms to coregister the data and/or placement of fiducial markers within or around the tissue as a reference.5, 6, 7, 8 The former approach requires complex computational models that are particularly susceptible to complications due to data formatting, imaging artifacts, and changes caused by tissue processing.4 On the other hand, the latter approach introduces the possibility of contaminating or otherwise harming the tissue itself.9
The approach described here improves the transition between modalities through the use of postmortem MRI to bridge the gap between in vivo MRI and histology. Postmortem MRI provides three-dimensional (3D) images of the brain at higher resolution than can be achieved in vivo and furthermore provides the data needed for producing a morphologically accurate model of the brain surface. This digital model can then be used to create a 3D-printed custom holder for the brain. With careful positioning, the brain holder allows for precise, MRI-oriented brain sectioning, reducing the need for complex mathematical algorithms, and enables a focus on specific regions for targeted sampling.
Our laboratory recently introduced new methods for creating custom brain holders and slicers using postmortem MRI and 3D-printing technology for human10 and marmoset brains.4 The two methods allow for a more accurate correlation between MRI and histology in a research setting, and ultimately allow a deeper understanding of the specific pathology underlying MRI abnormalities. Carefully designed experiments, in which the brain is sampled repeatedly over time in vivo, can provide context for interpretation of the pathology, which in turn can add specificity to interpretation of the MRI. Here, we present a modified protocol in a unified framework that can be applied to any brain tissue, whether it derive from nonhuman primates, rodents, or humans. We provide detailed instructions, and a corresponding video, for the marmoset sectioning. Although the overall protocol applies to any type of brain, due to differences in MRI acquisition and tissue size, as well as the challenges encountered when dealing with specific brain types, there are some differences in the approach depending on the type of brain being processed. In this presentation, sections with "human" will denote differences in protocol specific to the human brain.
Protokoll
Alle Tier Handhabung und hier beschriebenen Verfahren wurden in Übereinstimmung mit einem Protokoll, das vom Nationalen Institut für Neurologische Erkrankungen und Schlaganfall Animal Care und Use Committee genehmigt durchgeführt. Die Gehirne wurden von Weißbüschelaffen (Callithrix jacchus) gesammelt induzierte EAE zu entwickeln. 11 Die Gehirne wurden in 10% Formalin für zwischen 3 Wochen und 1 Jahr nach der Euthanasie durch transcardial Perfusion mit 4% Paraformaldehyd gespeichert.
1. Postmortem MRI Vorbereitung und Akquisition
- Marmoset Gehirn
- Bereiten einer Workstation mit Baumwollgaze, ein 50 ml-Zentrifugenröhrchen, kleine Spatel, ~ 30 ml eines fluorierten Öl, Paraffin und Marmoset Gehirn.
- Füllen Sie das Rohr mit dem fluorierten Öl und Gaze auf die 20-ml-Markierung. Komprimieren Sie die Gaze Luftblasen auf dem Weg zu entfernen.
- Vorsichtig trocken Formalin von der Oberfläche des Gehirns mit einem Papiertuch. Legen Sie das Gehirn mit dem frontalen Pol in Richtung der Unterseite desdas Rohr. Sorgfältig sichern mehr Gaze um die Seiten mit seiner Position zu fixieren, das Gehirn in die Röhre. Siehe Zusatzabschnitt 11 für ein Verfahren zur Herstellung eines MRT des Gehirns Wiege für die Einrichtung von zusätzlichen MRI-Scans zu schaffen.
- Füllen Sie den Rest des Rohres mit Gaze und fluorierte Öl. Vorsichtig Luftblasen auf dem Weg zu entfernen. Befestigen Sie die Kappe und die Dichtung um das Rohr mit Paraffin.
- Markieren Sie die Kappe im Einklang mit der Interhemisphärenspalt. Wickeln Sie das Rohr in einem Papiertuch und legen Sie sie in die Spule mit der Markierung oben in der Mitte.
- Erwerben Sie 2D-Echo T2 Spin. Die Parameter sind in Tabelle 1 angegeben.
- Öffnen Sie die 10 anatomischen 150 Mikron T2-weigted Akquisitionen in Mipav und registrieren zum 6. Akquisition.
HINWEIS: Die Registrierung ist eine optimierte automatische Registrierung 3.5D-Algorithmus mit 9 Freiheitsgrade, Fenster sinc-Interpolation, normalisierte Kreuzkorrelation Kostenfunktion, mit einem Aufruf des Powell Brent Suchalgorithmus. Rotationen wurden aus 10 Proben6; bis -10 ° mit groben Umdrehungen Inkrementieren 5 Grad und Fein Umdrehungen Erhöhen 1 °. Dann Mittelwert der registrierten Bilder: Utilities, Bildrechner-Bulk-Bilder, Durchschnitt.
- Menschliches Gehirn
- Trennen Sie die Vorderhirns vom brainstem einen Schnitt auf der Ebene des Mittelhirns verwenden. 10 Die Hemisphären kann auch mit einem Schnitt entlang der Mittellinie getrennt werden.
- Positionieren Sie den Vorderhirns in einem zylindrischen Rohr mit einer hemisphärischen Kuppel an einem Ende und einem Auslauf am anderen Ende.
- Füllen Sie das Rohr mit einem fluorierten Öl durch den Auslauf. Entfernen Sie Luftblasen mit sanften Sog für ~ 30 min durch den Auslauf.
- Erwerben Sie 3D-T1-MPRAGE. Die Parameter sind in Tabelle 1 angegeben.
2. Extrahieren von Gehirn-Oberfläche: Mipav 7.2
- Öffnen Sie die MRT in der koronalen Orientierung.
- Wählen Sie Algorithmen, Transformation Tools, Transformieren, Resampling. Wählen Sie Benutzerdefinierte Größe und Resampling zu isotRopic Voxel: 0,1 x 0,1 x 0,1 mm. Speichern Sie das neu abgetastete MRI als Brain_MRI_Resampled. Mensch: Resampling zu isotropen Voxel 0,3 x 0,3 x 0,3 mm.
- Wählen Sie Algorithmen, Filter (räumliche), Nonlinear Rauschunterdrückung. Verwenden Sie die Standardeinstellungen, klicken Sie auf OK.
- Wählen Zeigt Lookup-Tabelle und klicken Sie auf den Dual-Schwellwert-Taste. Ziehen Sie den Regler auf der grafischen Darstellung das gesamte Gehirn zu decken.
- Wählen Sie Algorithmen, Segmentierung, Schwelle, Schwelle mit min / max. Geben Sie die in der linken unteren Ecke Wert des Intensitätsgraphen (knapp unterhalb der Skala) in die "Untere Grenze" -Box. Wählen Sie "binäre" in für die Ausgabebildtyp und deaktivieren Sie "Invert-Schwelle."
- Wählen Sie Algorithmen, Morphologische, Löcher füllen. Überprüfen Sie "Prozess in 2.5D."
- Denn dies ist ein MRI des vollen Gehirn ist, gibt es einige leere Raum zwischen dem Hinterhirn und Kortex, die gefüllt werden muss. Das menschliche Gehirn: diesen Schritt überspringen.
- Mit Linie VOI, ziehen in eine Verbindungzwischen dem Rautenhirns und Rinde auf beiden Seiten des Gehirns am meisten seitlichen Punkt. Setzen Sie diese durch das Gehirn.
- Wählen Sie VOI, VOI-Konvertierung, Alles in binäre Maske. Wählen Sie Dienstprogramme, Bildrechner. Wählen Sie ein oder vom Bediener Drop-Down-Menü und wählen Sie das Gehirn Maske. Wählen Sie "Fördern Zielbildtyp"
- Wählen Sie Algorithmen, Morphologische, Löcher füllen. Überprüfen Sie "Prozess in 2,5D"
- Speichern Sie die binäre Maske als Brain_Model.nii.
- Wählen Sie Algorithmen, Extract Oberfläche (Marching Cubes). Wählen Sie Maskenbild, Speichern unter Brain_Model.ply.
3. Auswahl Scheibe Standorte: Mipav 7.2
- Identifizieren Sie Gewebe von Interesse oder Ausgangsposition. Berechnen Sie die beabsichtigte Brammendicke. In marmoset Gehirn, pro Abschnitt 30 MRI Scheiben zählen und 5 MRI Scheiben pro Messerspalt erzeugt 3 mm Abschnitte mit 0,5 mm Schaufelzwischenräume, was zu ~ 3,5 mm Platten. Das menschliche Gehirn: 20 MRI Scheiben pro Abschnitt und 4 MRI slgefriert pro Messerspalt erzeugt 6 mm Abschnitte mit 1,2 Lücken mm Klinge, in ~ 7,2 mm Platten zur Folge hat.
- An der Stelle des ersten Messerspalt, klicken Sie auf "Feld VOI" und dann eine Box über das Gehirn zu ziehen. Klicken Sie in der Box, um es auszuwählen. Kopieren Sie die Kontur für jede MRT-Scheibe entsprechend dem Messerspalt.
- Direkt vor durch die Anzahl der MRI Scheiben entsprechend der Schnittdicke und kopieren und einfügen, um die Kontur auf die nächste Messerspalt entspricht. Wiederholen Sie diesen Vorgang durch das Gehirn.
- Wählen Sie VOI, VOI-Konvertierung, Alles zu Binary-Maske. Speichern unter Blade_Gaps.nii.
- Wählen Sie Algorithmen, Flächen lösen (Marching Cubes), Maskenbild, Blade_Gaps.ply.
4. Erstellen von MRI-Blade-Karte: Mipav 7.2
- Öffnen Sie die Brain_MRI_Resampled und die Blade_Gaps.nii Bilder.
- Mit dem Blade_Gaps.nii Bild ausgewählt haben , wählen Sie Dienstprogramme, Bild Math. Wählen Sie fördern Bildtyp und Multiplizieren.Geben Sie 10000 als Wert ein.
- Wählen Sie Dienstprogramme, Bildrechner. Wählen Sie Hinzufügen, und wählen Sie dann das Brain_MRI_Resampled Bild aus dem Bild Dropdown - Box. Wählen Sie fördern Zielbildtyp.
- Speichern Sie das Bild als Brain_BladeMap.nii.
- Durch Klicken auf den Triplanar Ansicht, die Orte, an denen das Gehirn in Scheiben geschnitten werden kann, in drei orthogonalen Ansichten zu sehen.
5. Importieren von Gehirn und Blade-Gap Oberflächen: Netfabb Berufs
- Wählen Sie Part, part. Wählen Sie die Dateien Brain_Model.ply und Blade_Gaps.ply
- Wählen Sie das Gehirn und klicken Sie auf Reparaturmodus. Im Reparaturmodus:
- Klicken Sie auf die Shell-Auswahltaste, und klicken Sie dann auf das Gehirn.
- Klicken Sie auf die Toggle-Auswahltaste, um die anderen Maschen wählen. Klicken Sie auf Entfernen Sie die anderen Maschen zu löschen.
- Klicken Sie auf Übernehmen Reparatur, und entfernen Sie den alten Teil.
- Rechtsklick auf das Gehirn. Wählen Sie bewegen. Klicken Sie aufZum Ursprung Taste. Notieren Sie die XYZ-Parameter, die angezeigt werden. Diese Übersetzungsparameter werden benötigt, um die Klinge Positionierung in Mipav einrichten zu halten. Klicken Sie auf Übersetzen. Dann schließen Sie das Fenster. (Klicken Sie nicht mehr als einmal zu übersetzen. Es wird erneut übersetzen, die gleichen Parameter verwenden.)
- Wählen Sie das Blade_Gaps Modell. Rechtsklick auf den Teil, und wählen Sie Verschieben. Geben Sie die XYZ-Werte aufgezeichnet zuvor in den XYZ-Parameter-Boxen. Klicken Sie nun auf das Fenster Übersetzen und zu schließen.
- Wählen Sie das Brain_Model Modell und klicken Sie auf Reparaturmodus. Im Reparaturmodus:
- Klicken Sie auf die Shell-Auswahltaste, und klicken Sie dann auf das Gehirn.
- Rechts klicken und glatte Triangles wählen. Geben Sie 4-5 Wiederholungen. Überprüfen Volumenschrumpfung verhindern. Das menschliche Gehirn: 1-2 Wiederholungen.
- Rechts klicken, wählen Sie reduzieren Triangles. Geben Sie 200000 in das Zieldreieck Zahl und klicken Sie auf Ausführen.
- Klicken Sie auf Automatische Reparatur, Standard-Reparatur. Klicken Sie auf Übernehmen dann Repair
- Rechts klicken, Umbenennen. Benennen Sie die geglättete Gehirn als Smoothed_Brain_Model.
- Wählen Sie Smoothed_Brain_Model. Rechts klicken, Export, STL.
6. Bearbeiten von Gehirn Konturen: Meshmixer
- Importieren Sie die Smoothed_Brain_Model in Meshmixer.
- Verwenden Sie die Bildhauerei und Auswahl-Werkzeuge, um Anpassungen an das Gitter zu machen. Edits sind:
- Verwenden Sie Sculpting-Tools, robust glatt. Glätten Sie die Fläche entsprechend der Linie VOI. Das menschliche Gehirn: diesen Schritt überspringen.
- Glätten Sie die Oberfläche der Hirnrinde, die in der Box unten sein Gesicht.
- Glätten kleine Einschläge, die in der Vernetzung und Bearbeitungsprozess geschaffen worden sein könnte.
- Wählen Sie Analyse, Inspektor, Autoreparatur alle.
- Exportieren Sie die Smoothed_Brain_Model als Smoothed_Edited_Brain_Model.
7. Erstellen des Gehirns Slicer Box: Netfabb Profi
- Wählen Sie einen Teil, hinzufügen Part. Wählen Sie die file Smoothed_Edited_Brain_Model.
- Wählen Sie einen Teil, hinzufügen Part. Dann wählen Sie die STL - Datei Gehirn Slicer Parts_Marmoset und klicken Sie auf. Das menschliche Gehirn: Gehirn - Slicer Parts_Human (Supplemental Code - Dateien).
- Rechtsklick auf den Teil, und wählen Sie Erweitert, Muscheln zu Teilen. Wählen Sie jedes Teil einzeln und rechts klicken, um sie umzubenennen. Umbenennen (Klick auf das Auge neben einem Objekt versteckt es oder macht es sichtbar.) Das große Feld Main, das kleine Feld Sub, und die Abschneidkasten Box_Cutout, die Hexagon Form Blade_Holder_Main, die kleine flache Box Mikrotom Klinge, und die Halb Rohr Objekt Cradle. Das menschliche Gehirn: nein Blade_Holder_Main, Mikrotom Blade, oder Cradle.
- Ausblenden Main, Sub, Blade, Blade_Holder_Main, Mikrotom Blade, und Cradle und verwenden Sie Shift-select alle sechs und die Box_Cutout auszuwählen. Nur die Box_Cutout und die Smoothed_Edited_Brain_Model sollte sichtbar sein, aber die Smoothed_Edited_Brain_Model sollte nicht ausgewählt werden.
- Klicken Sie auf und die ausgewählten Teile ziehen Sie das Feld Position in Bezug auf das Gehirn zu justieren.
- Positionieren Sie das Gehirn in der Mitte der Box. Postion das Gehirn tief genug in der Box fest ergriffen zu werden, aber nicht zu tief Hänge zu schaffen, die richtige Platzierung zu verhindern.
- Nach der Positionierung kann das Gehirn Kasten Kontur für Auskragungen getestet werden. Wählen Sie die Box_Cutout und die Smoothed_Edited_Brain_Model. Wählen Sie Boolesche Betrieb.
- Klicken Sie auf das Smoothed_Edited_Brain_Model es rot.
- Wählen Sie Boolesche Subtraktion, und die Berechnungen gelten.
- Überprüfen Sie die Gehirn-Box für Überhänge, die das Hirngewebe vor einer sicher platziert in den Strafraum verhindern würde. Wenn diese Überhänge vorhanden sind, stellen Sie das Gehirn, so dass es weniger tief in der Box ist. Wenn das Gehirn ist in der gewünschten Tiefe und Auskragungensiehe vorhanden sind, um zusätzlichen Abschnitt 10 für eine Lösung für die Überhänge zu entfernen.
- Wählen Sie das Blade_Gaps Modell. Rechts klicken, wählen Sie Verschieben. Notieren Sie sich die Position Z-Wert, dann das Fenster zu schließen. Dadurch wird die Position der hintersten Klingenspalt sein.
- Wählen Sie den Blade - STL , die aus dem Gehirn Slicer Parts kam. Rechts klicken, wählen Sie Verschieben.
- Absolute Übersetzung auswählen. Geben Sie den Z-Wert aus dem 7.8. Für die X- und Y-Werte, den entsprechenden Wert aus der aktuellen Position Parameterfelder eingeben
- Wählen Sie den Blade - STL. Rechts klicken, Klick Duplikate.
- Überprüfen Sie Teile anordnen. Geben Sie die Gesamtzahl der Blätter in der Gesamtzählung. Geben Sie die gleiche Anzahl in der Z Zählung Box. Geben Sie die Brammendicke in der Z-Lücke. Klicken Sie auf Duplizieren. Wenn die Plattendicke variiert wurde, müssen die Blätter einzeln positioniert werden. Duplizieren jedes neue Klinge aus dem vorherigen (von hinten bewegen anterior)mit dem z Spalt gleich der Schnittdicke für diesen Abschnitt.
- Positionieren Sie die Mikrotom Klingenteile in genau den gleichen Abständen wie die Messer in der Schneidemaschine durch Wiederholen von Schritt 7.9 und 7.10 für die Mikrotom - Klinge. Das menschliche Gehirn: diesen Schritt überspringen.
- Wählen Sie Mikrotom Klinge zusammen mit dem Blade_Holder_Main Teil und wählen Sie Boolesche Betrieb.
- Wählen Sie alle Blätter in Mikrotomklinge ihnen rot markieren. Klicken Sie auf Boolesche Subtraktion, und die Berechnungen gelten.
- Wählen Sie Reparaturmodus. Führen Sie eine erweiterte Reparatur. Wählen Sie gelten die Reparatur, und entfernen Sie den alten Teil.
- Benennen Sie das Teil Klingenhalter. Exportieren Sie das Teil als STL.
- Umschalt-select Smoothed_Edited_Brain_Model, die alle die Blade - Modelle im vorherigen Schritt erstellt, und die Sub und Main. Klicken Sie auf Boolesche Operation.
- Machen Sie alle Teile außer Haupt rot durch selecting ihnen und den Pfeil unter dem grünen Feld klicken, um sie zu rot wechseln.
- Wählen Sie Boolesche Subtraktion und wählen Sie dann Berechnungen anwenden.
- Klicken Sie auf Reparaturmodus. Im Reparaturmodus:
- Markieren Sie das Kästchen für die Auskragungen und scharfe Punkte. Diese können in Netfabb oder Meshmixer geglättet werden.
- Klicken Sie auf Automatische Reparatur erweiterte Reparatur. Wenden Sie dann die Reparatur und entfernen Sie den alten Teil
- Rechtsklick auf das reparierte Gehirn - Box und benennen Sie es Gehirn Slicer Box. Export als STL.
8. Drucken des Gehirns Slicer Box auf der Ultimaker 2
- Marmoset Gehirn: Cura
- Importieren Sie die Gehirn - Slicer Box in Cura.
- Wählen Sie Drehen und ziehen Sie den Kreis, um das Feld zu drehen, so dass es flach auf dem Bett ist.
- Passen Sie die Druckeinstellungen: 0,1 mm Schicht Auflösung, 50% Besatzdichte, Floß.
- Wählen Sie Werkzeugbahn speichern auf SD-Karte. (Druckzeit ~ 12 h.)
- Import Klingenhalter inzu Cura und drehen, so dass die Schlitze an den Seiten sind und das Sechseck Gesicht in der XZ oder YZ-Ebene ist.
- Duplizieren Sie das Objekt.
- Passen Sie die Druckeinstellungen: 0,2 mm Schichtauflösung, 20% Besatzdichte, Krempe.
- Wählen Sie Werkzeugbahn speichern auf SD-Karte. (Druckzeit ~ 3 h)
- Human Brain: Cura
- Importieren Sie die Gehirn - Slicer Box in Cura und drehen , wie in 8.1.2.
- Passen Sie die Druckeinstellungen: 0,2 mm Schichtauflösung, 30-35% Besatzdichte, Floß.
- Wählen Sie Werkzeugbahn speichern auf SD-Karte. (Druckzeit ~ 70 h für einzelne Hemisphäre Box).
- Auf der Ultimaker 2
- Tragen Sie eine dünne Schicht von Klebestift Leim auf Platte zu bauen.
- Legen Sie die SD-Karte. Wählen Sie Drucken und wählen Sie das Teil.
9. Schneiden des Gehirns
- Marmoset Gehirn
- Bereiten Sie eine Workstation mit dem festen Gehirn, das Gehirn Hobel, zwei Messerhalter, Mikrotomklingen, 1 ml fluorierten Öl, flat Pinzette, Schutzhandschuhe und Einbettkassetten.
- Setzen Sie neue Mikrotomklingen in die Schlitze an den Blatthaltern. Sicherstellen, dass die abgeschrägte Kante jeder Klinge in die gleiche Richtung weist. Schutzhandschuhe tragen , wenn Mikrotomklingen Handhabung.
- Entfernen Sie das Gehirn aus Formalin und vorsichtig trocknen.
- Legen Sie das Gehirn in die Schneidemaschine. Einige Tropfen fluorierte Öl kann zum Gehirn und Slicers angewendet werden für die einfache Positionierung zu ermöglichen. Stellen Sie sicher, dass das Gehirn ist fest an seinem Platz.
- Positionieren Sie die Messerhalter mit den Messern in den entsprechenden Klingenschlitze.
- Drücken Sie auf den Messerhalter fest und langsam Balance Druck ausüben durch das Gehirn zu schneiden.
- Entfernen jeder Platte, einer nach dem anderen, von der Vorderseite des Gehirns beginnt. Es hilft, die Mikrotom-Klinge vor einer Platte zu entfernen, bevor die Platte selbst zu entfernen. Achten Sie auf den vorderen / hinteren Ausrichtung jeder Platte.
- Machen Sie Fotos von der vorderen eind hinteren Fläche jeder Platte. Die hinteren Platten wird höchstwahrscheinlich enthalten getrennte Teile, so die Aufmerksamkeit für die Einbettung auf die Orientierung der Teile zu bezahlen. Platzieren Sie jede Platte in eine Einbettungskassette und sie alle in einer 10% igen Formalin-Lösung.
- Menschliches Gehirn
- Sorgfältig prüfen Sie den Sitz des Gehirns in der Box.
- Schneiden Sie das Gehirn von einem Ende ausgehend einen abgewinkelten Schnitt verwenden, langsam aber fest schneiden. Schneiden Sie das Gehirn durch die einzelnen Messerspalt.
- Entfernen Sie jede Platte ein zu einer Zeit, aufmerksam auf die Anzahl und die anterior / posterior Ausrichtung jeder Platte.
- Machen Sie Fotos von den vorderen und hinteren Oberfläche jeder Platte. Legen Platten in verschlossenen 10% Formalin Taschen. Gewebeblöcke werden aus den Platten und platziert in Kassetten für die Einbettung geschnitten werden.
10. Entfernen Überhänge in Brain Box (Zusatzteil)
- Das Herausziehen Scheiben: Meshmixer
- Importieren Sie die Smoothed_Edited_Brain_Model.
- Wählen Sie Bearbeiten, Make-Scheiben. In Make Scheiben:
- Wählen Sie Stacked 3D, Z, geben Sie 1-2 mm Dicke. Klicken Sie auf Compute. Wenn die Scheiben zu laden, klicken Sie auf Annehmen.
- Wählen 1 oder 2 Scheiben mit großen Umfängen in der Nähe der Unterseite des Kortex. Diese Scheiben sollten unter dem Niveau des Sub - Box sein.
- Exportieren Sie jede dieser Scheiben als Brain_Slice_ #.
- Die Ausweitung der Scheiben von Überhängen zu entfernen: Netfabb Berufs
- Importieren Sie die Brain_Slice_ # Scheiben.
- Duplizieren jedes Brain_Slice_ # (Markierung aufheben Teile anordnen , wenn aktiviert).
- Rechtsklick auf eine Kopie jeder Brain_Slice_ # und wählen Sie Skala.
- Deaktivieren Sie die Option Feste Skalierungsverhältnis. Dann skalieren Richtung der Hirnschnitt in der Y so , dass sie die Höhe des Bodens des Teilfeld erreichen.
- Benennen Sie diese Scheiben Brain_Slice_Big_ #.
- Überprüfen Sie die Y-Position of der ursprünglichen Brain_Slice_ # durch einen Rechtsklick auf den Teil und die Auswahl bewegen. Notieren Sie sich die Y - Position für jede der ursprünglichen Brain_Slice_ # Scheiben schneiden.
- Führen Sie die Berechnung: Brain_Slice_ # [y - Position] - (Brain_Slice_Big_ # [y size] - Brain_Slice _ # [y size])
- Wählen Sie jede der Brain_Slice_Big_ # einzeln rechten Maustaste und wählen Sie Verschieben.
- Geben Sie den Wert berechnet aus 10.2.6 in der Y-Übersetzung Parameterfeld. Für die X- und Z-Translationsparameter, geben Sie in der aktuellen Position Parameterfelder befinden Werte. Wählen Sie Absolute Übersetzung. Klicken Sie auf Übersetzen und das Fenster zu schließen.
HINWEIS: Die Brain_Slice_Big_ # Scheiben werden zusammen mit dem Gehirn und Schaufeln abgezogen werden , wenn die Box macht.
- Geben Sie den Wert berechnet aus 10.2.6 in der Y-Übersetzung Parameterfeld. Für die X- und Z-Translationsparameter, geben Sie in der aktuellen Position Parameterfelder befinden Werte. Wählen Sie Absolute Übersetzung. Klicken Sie auf Übersetzen und das Fenster zu schließen.
11. Marmoset Brain MRI Cradle for Weitere Scan
- Erstellen des Brain MRI Cradle
- Stellen Sie sicher , dass die obere Oberfläche des CrADLE ist auf der gleichen Höhe wie die Box_Cutout. Die Tiefe und die Position des Gehirns in der Wiege sollte so eingestellt werden, so wie es für den Slicer ist.
- Umschalt-select die Smoothed_Edited_Brain_Model und Cradle auszuwählen.
- Boolesche Operation auswählen. Wählen Sie das Gehirn es rot zu markieren, und Boolesche Subtraktion auszuwählen. Dann die Berechnungen gelten. (Wählen Sie auch die Brain_Slice_Big_ # Scheiben falls zutreffend.)
- Geben Sie Reparaturmodus keine scharfen Punkte in der Wiege Kontur zu entfernen, wie zuvor für den Slicer getan. Wählen Sie Erweiterte Reparatur. Tragen Sie die Reparatur, und entfernen Sie den alten Teil.
- Rechtsklick auf das Teil MRI Gehirn - Cradle umbenennen. Wählen Sie Export, STL.
- Drucken der Wiege: Cura
- Importieren Sie die MRI Gehirn - Cradle in Cura und drehen Sie es so , dass der flache Teil mit dem Gehirn Ausschnitt Gesicht nach oben.
- Passen Sie die Druckeinstellungen: 0,1 mm Schicht Auflösung, 100% Besatzdichte, Floß.
- Wählen Sie Pfad Tool Speichern auf SD-Karte. (Druckzeit ~ 10 h)
- Drucken auf dem Ultimaker 2, wie in 8.3 beschrieben.
- Der Erwerb hochauflösende T2 * MRT die Wiege mit
- Vorsichtig trocken Formalin von der Oberfläche des Gehirns mit einem Papiertuch.
- Positionieren Sie das Gehirn in der Wiege, wie für die Schneidemaschine beschrieben.
- Schieben Sie das Gehirn und die Wiege in die 50 ml Zentrifugenröhrchen. Füllen Sie es mit fluorierten Öl bis zum Rand.
- Drücken Sie vorsichtig den Schlauch Luftbläschen entweichen aus dem Gehirn zu ermöglichen. Legen Sie die Überwurfeinsatz in den Einschub der Rohrkappe Bildung von Luftblasen dort zu verhindern. Befestigen Sie die Kappe und versiegeln das Rohr mit Paraffin.
- Das Röhrchen wird in die Spule, wie zuvor beschrieben. 3D-T2 * Parameter sind in Tabelle 1 angegeben.
- Öffnen Sie die 18 anatomischen 100 Mikron T2 * -gewichteten Akquisitionen in Mipav und registrieren zum 10. Akquisition. Registrierungsparameter sind die gleichen wie in 1.1.7. Der Mittelwert der registrierten Bilder: Utilities, Bildrechner-Bulk-Bilder, Durchschnitt.
Ergebnisse
Der Arbeitsablauf dieses Verfahrens ist in Figur 1 zusammengefaßt Sobald das Gehirn in Scheiben geschnitten wird, wird ein visueller Vergleich zwischen den MR-Bildern und Bildern der oberflächlichen Oberflächen der Brammen zeigt eine gute Orientierungsspiel über mehrere Platten (Abbildung 2). Nachdem die Platten in Paraffin eingebettet sind, werden sie auf einem Mikrotom und gefärbt geschnitten. Eine gründlichere Vergleich zwischen der hochauflösenden MRT Obduktion und den gefärbten Histologieschnitten zeigt eine genaue und konsistente Übereinstimmung über alle Strukturen des Seidenäffchen Gehirns (Abbildung 3).
In diesem Tiermodell der MS, entwickeln die Tiere Läsionen der weißen Substanz in der gesamten weißen Hirnsubstanz zu verbreiten. Diese Läsionen können nichtinvasiv erfasst werden durch MRI durchgeführt wird. Figur 4 zeigt die Fähigkeit dieser Technik, um die pathologischen Substrat der MRT aufzuklären. Kleine Läsionen in vivo MRI nachgewiesen kannsowohl postmortalen MRT und Histologie werden verfolgt. Wie in den Einschüben gezeigt, ist Demyelinisierung innerhalb der Läsionen eine der Hauptkomponenten des MR-Signaländerung (hyperintenser im Vergleich zu umliegenden Gewebe) zu fahren. Die Histologie und postmortalen MRT kann auch Läsionen zeigen verpasste in vivo MRI (Abbildung 4).
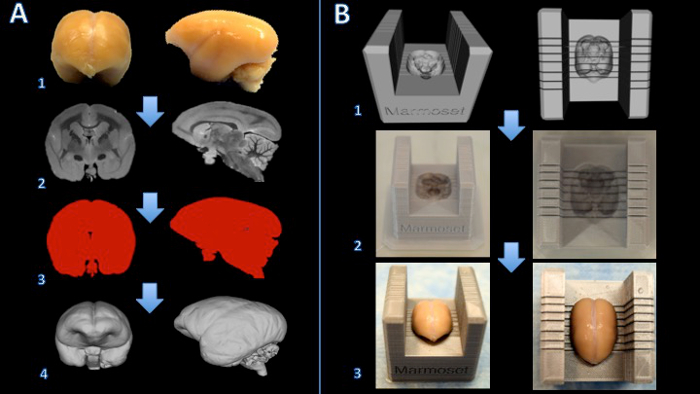
Abbildung 1. Arbeitsablauf für ein Seidenäffchen Gehirn Slicer Feld zu schaffen. Das Gehirn ist mit Formalin (A1) befestigt und ein T2-gewichteten MRT mit isotropen Voxel von 150 & mgr; m pro Kante (A2) erworben. Bilder werden verarbeitet und Schwellwertbildung eine binäre Maske (A3) zu erstellen. Die Oberfläche wird dann in 3D-Modellierungssoftware (A4) wiedergegeben. Eine Boolesche Subtraktion zwischen einem Slicer-Vorlage und dem Gehirn-Modell erstellt ein digitales Modell des Gehirns Slicer (B1). Das Gehirn Slicer-Box ist auf einem 3D-Drucker (B2) gedruckt. Das Gehirn ist dann fest in der Slicer Kasten gelegt fürSchneiden (B3). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
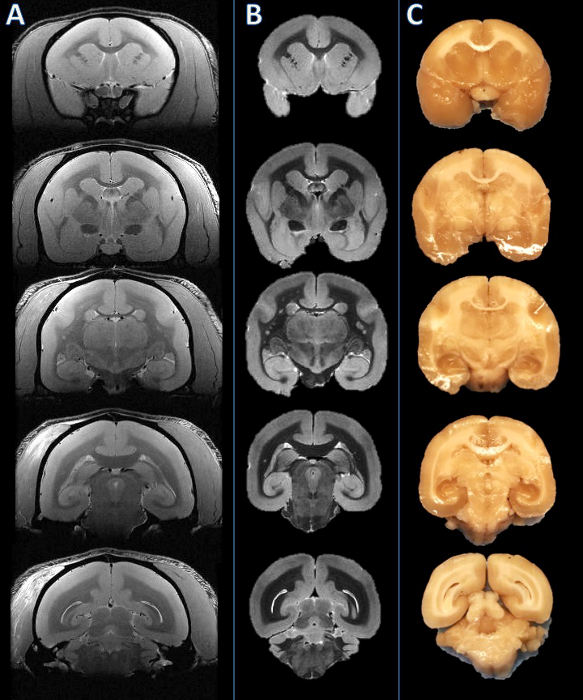
Abbildung 2. Von links nach rechts: In - vivo - MRT, MRI postmortalen und Gewebeplatte Fotografie. Schnittebenen wurden auf der Grundlage der postmortalen MRI (B) festgelegt und visuell im Vergleich zu den entsprechenden in vivo MRI slice (A). Das Gehirn wurde dann geschnitten, und die resultierenden Platten gefunden wurden konsistent (C) zu sein. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
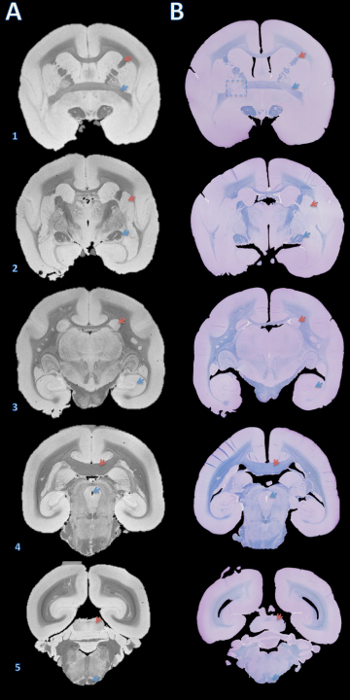
Abbildung 3. Hochauflösende Obduktion MRT und HistologieAbschnitt Anpassung. Brammen wurden in Paraffin eingebettet, geschnitten mit einem Mikrotom in 4 um Abschnitte, und gefärbt mit schnell blau und Kresylviolett (B). Die Schnitte wurden dann visuell mit dem 100 & mgr; m abgestimmt T2 * -gewichteten MRI basiert auf Hirnstrukturen (A). Details zum Erwerb dieses Bild sind in dem zusätzlichen Abschnitt des Protokolls und Tabelle 1. Hirnstrukturen: (1) roter Pfeil = innere Kapsel, blauer Pfeil = Commissura anterior; (2) roter Pfeil = Putamen, blauer Pfeil = Tractus; (3) roter Pfeil = Nucleus caudatus, blauer Pfeil = Hippocampus; (4) roter Pfeil = Corpus callosum, blauer Pfeil = zerebrale Aquädukt; (5) roter Pfeil = Colliculus inferior, blauer Pfeil = Pyramidenbahn. Die gestrichelten Kasten in B1 zeigt eine Scheibe, wo, entweder während der Hirnschneiden oder Paraffineinbettung, ein Fehler eine leichte Drehung um die Y-Achse verursacht wird, auf der linken Seite Mismatch der anterioren Kommissur führt. Bitte klicken Sie hier anzuschauenGrößere Version der Figur.
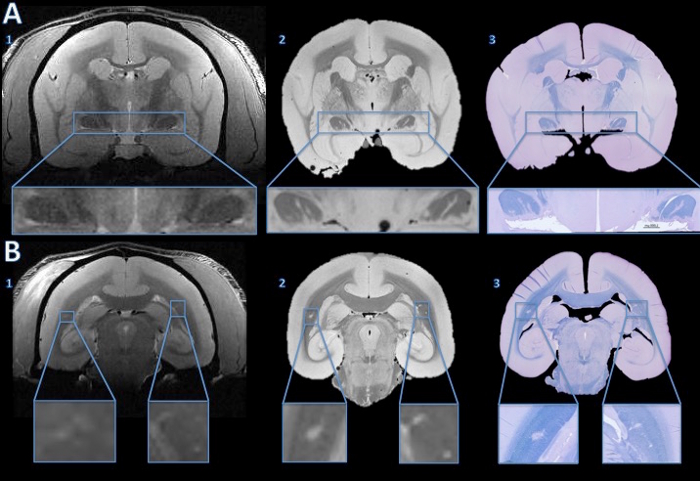
Abbildung 4. Läsionen von in - vivo - MRT Histologie Abschnitt der Spur. Die in - vivo - MRT zeigte keine überzeugenden Hinweise auf eine anomale hyperintenser Signal Läsionen in beiden Tractus (A1) vorzuschlagen. Allerdings zeigt die hochauflösende Obduktion MRI klar hyper intensive Linien in beiden Optik Bahnen (A2). Die schnelle blau / Kresylviolett Fleck eines 4 um Histologie Abschnitt zeigt , dass die hyperintensen Bereiche auf der Ex - vivo - MRT zu sehen sind demyelinierten (A3). In der weißen Hirnsubstanz, zeigt die in vivo MRI subtile hyperintenser bilateral (B1, vergrößerte in den Einsätzen). Die hyperintensen Bereiche sind offensichtlich auf dem hochauflösenden Obduktion MRI (B2). Der LFB Fleck eines 4 um Histologie Abschnitt zeigt, dass diese Bereiche (B3) sind demyelinierten. Nach dem Vergleich mit dem Ausgangswert in vivo MRI und Hämotoxylin-and-Eosin - Färbung wurde die rechte Seite bestimmt eine anatomische Anomalie zu sein, nicht ein demyelinisierten Läsion. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Zusätzliche Codedateien. Brain_Slicer_Parts_Marmoset.stl: Bitte hier klicken , um diese Datei herunterzuladen. Brain_Slicer_Parts_Human.stl: Bitte hier klicken , um diese Datei herunterzuladen. Cap_Insert.stl: Bitte hier klicken , um diese Datei herunterzuladen.
Diskussion
Das Protokoll hier skizzierte ermöglicht einen genauen Vergleich zwischen MRT und Histologie Abschnitte. Das Protokoll wird in einem einheitlichen Format dargestellt, das Gehirn von Menschen oder Kleintieren, wie Nagetieren oder marmosets angewendet werden kann. Bedingte Unterschiede zu groß (Mensch) und kleine (nicht-menschlichen Primaten und Nagetieren) Gehirne markiert sind, und im zugehörigen Video und Figuren zeigen wir die Anwendung in der Seidenäffchen. Obwohl das Konzept einfach ist, erfordert das Verfahren viele Schritte, sowie die Verwendung von mehreren Arten von Software. Darüber hinaus möglicherweise einige Probleme für die Richtigkeit dieser Methode beeinflussen, sind wichtig, zu erwähnen.
Die Bildqualität der in vivo MRI ist ein wichtiger Faktor. Zur Minimierung sollten die Unterschiede in der Bildauflösung zwischen MRI und digitalisierter Histologie Bilder, die kleinste mögliche MRI Voxelgröße verwendet werden. Dieses Konzept gilt auch für die Bildqualität des postmortalen MRI. Während die erhöhteAkquisitionszeit in Obduktion MRT erlaubt viel höhere Bildauflösung, kann das Präparat Bildartefakte einführen, wie fokale Signalausfälle im Zusammenhang mit Luftblasen. Diese Artefakte können Bereiche des Gewebes verdunkeln sowie dessen Kontur beeinflussen. Außerdem sind die Dimensionen des Gewebes auf der postmortalen MRI wahrscheinlich durch die Fixierungsprozess und der Dauer beeinflusst. Während die in vivo zu ex vivo kann MRI Spiel eng durch die Verwendung von anatomischen Landmarken in Schichtgeometrie Setup während der Erfassung angenähert werden, eine nichtlineare Registrierung wäre immer noch notwendig sein , ein höheres Maß an Genauigkeit zu erreichen in diesen beiden MRT - Bilder passen.
Das Design des Gehirns Halters und Slicer ist auch ein entscheidender Schritt. Beim Erzeugen des digitalen Modells des Gehirns wird ein Glättungsalgorithmus angewendet, daß das Modell relativ zur festen Gehirn leicht vergrößert. Dies ermöglicht ein leichtes Einführen des Gehirns in die Halterung und Slicer und reduziert scharfe Kanten im Halter &# 39; s Kontur. Wenn jedoch das Modell zu groß ist (beispielsweise um mehr als 5%), kann das Gehirn während der postmortalen MRI und / oder der Schnitt bewegen. Ein weiterer wichtiger Punkt ist die Gestaltung des Gehirns Modell anzupassen, so dass das Cerebellum richtig in die 3D-gedruckten Objekt platziert wird. Dies kann besonders schwierig sein, wenn das Kleinhirn hat während des Gehirns Extraktion bei der Autopsie beschädigt wurde.
Beim Drucken mit dem Gehirn Slicer und Halter, muss die Art der 3D-Drucker auch sorgfältig ausgewählt werden. Einige Multi-Jet-Drucker benötigen Nachbearbeitung unter Verwendung eines Ofens Trägermaterial zu entfernen. Während diese Drucker Objekte erzeugen können, die fusionierten als Deposition Modelling Desktop wasserdicht und relativ haltbarer sind (FDM) Drucker, um die Erwärmung Stützen entfernen Sie die Box leicht verziehen kann, Schaufelzwischenräume zu schaffen, die zum Gehirn Kontur nicht perfekt senkrecht stehen.
Das Gehirn sectioning Prozess ist ein weiterer entscheidender Schritt. Vor dem Schneiden the ganze Gehirn in Platten, ist es wichtig, sicherzustellen, dass das Gehirn dicht im Inneren des Gehirns Slicer sitzt: Es gibt keine Bewegung sein sollte, wenn leichter Druck auf das Gehirn angewendet wird. Dadurch wird es möglich machen, die Blätter durch das Gehirn an den genauen Ort von den Ermittlern festgelegt zu schneiden. Eine kontinuierliche, ausgeglichene Druck sollte auf beide Blatthalter angewendet werden beim Schneiden. In Abhängigkeit von der Schärfe der Messer und die Steifigkeit des Gewebes, eine leichte Querschnittbewegung könnte für die Aufrechterhaltung der flachen Schnittflächen von Vorteil sein.
Das Paraffin-Einbettungsprozess kann auch eine Quelle der Fehlausrichtung zwischen den MRT und Histologie sein. Wenn die Gewebeplatte ist nicht flach gegen die Kassette während des Einbettungsprozesses sitzt, gibt es eine Neigung zwischen der Schneidebene des Mikrotoms und der Oberfläche anstelle der Platte sein. Dies wird Schneiden unbrauchbar Abschnitte erfordern eine flache Ebene zu finden, bei dem alle Gewebe ausgesetzt ist. Eine Möglichkeit für die Neigung zu korrigierenwird durch den Winkel der Betrachtungsebene auf dem hochauflösenden isotropen postmortale MRI ändern. Dies ist jedoch nahezu unmöglich, auf die in vivo MRI durchzuführen, die normalerweise mit anisotropen Auflösung (typischerweise dicke koronale Schnitte) erfasst wird.
Schließlich kann das Gewebe eine gewisse Deformation während der Periode Formalinfixierung erfahren und Paraffineinbettung (Schrumpfung), sowie bei der Herstellung von Folien (Falten, Risse, Falten). Einige dieser Verformungen kann, indem man die 4-5 & mgr; m Abschnitte in einem Wasserbad korrigiert werden, bevor auf Objektträger übertragen. Andere Verformungen können teilweise durch Ausführen einer verformbaren Bild Koregistrieren der histologischen digitalisierten Bilder auf die postmortale MRT-Bilder gelöst werden. Dennoch ist die Verformungen mit einer sorgfältigen und qualifizierten Praxis minimiert der effektivste Ansatz, um passende MRI Bände Abschnitte Histologie.
Abschließend stellte die Methodik hier ermöglicht investigators, um genau die zugrundeliegende Pathologie der MRT-Befunde zu bewerten. Allgemeiner gesagt, ist es ein vielversprechender Ansatz zur Identifizierung und / oder neuartigen Biomarker für MRI Studien validiert, die bestimmte pathologische Prozesse, wie Entzündungen oder Remyelinisierung zielen.
Offenlegungen
The authors declare that they have no competing financial interests.
Danksagungen
The Intramural Research Program of NINDS supported this study. We thank the NIH Functional Magnetic Resonance Imaging Facility. We thank Jennifer Lefeuvre and Cecil Chern-Chyi Yen for assistance with postmortem MRI acquisition. We thank John Ostuni and the Section on Instrumentation Core Facility for assistance with 3D printing. Figure 1 of this work used snapshots from MeshLab, a tool developed with the support of the 3D-CoForm project.
Materialien
| Name | Company | Catalog Number | Comments |
| 7T/30cm USR AVIII Bruker MRI | Bruker Biospin | ||
| 38 mm Bruker Biospin volume coil | Bruker Biospin | ||
| Fomblin | Solvay Solexis | ||
| 50 ml Falcon Centrifuge Tubes, Polypropylene, Sterile | Corning | 21008-951 | |
| Fisherbrand Gauze Sponges | Fisher Scientrific | 13-761-52 | |
| Parafilm M All-Purpose Laboratory Film | Bemis | ||
| Leica RM2235 rotary microtome | Leica | ||
| Leica Disposable Blades, low profile (819) | Leica | ||
| Cresyl Violet Acetate, 0.1% Aqueous | Electron Microscopy Sciences | 26089-01 | |
| Luxol Fast Blue, 0.1% in 95% Alcohol | Electron Microscopy Sciences | 26056-15 | |
| ETOH | |||
| Ultimaker 2 Extended | Ultimaker | ||
| .75 kg Official Ultimaker Branded PLA Filament, 2.85 mm, Silver Metallic | Ultimaker | ||
| Axio Observer.Z1 | Zeiss | ||
| Zen 2 (Blue Edition) | Zeiss | ||
| Netfabb Professional 5.0.1 | Netfabb | http://www.netfabb.com/professional.php | |
| Meshmixer 10.9.332 | Autodesk | http://www.meshmixer.com/download.html | |
| Mipav 7.2 | NIH CIT | http://mipav.cit.nih.edu | |
| Cura | Ultimaker | https://ultimaker.com/en/products/cura-software |
Referenzen
- Evans, A. C., Frank, J. A., Antel, J., Miller, D. H. The role of MRI in clinical trials of multiple sclerosis: comparison of image processing techniques. Ann Neurol. 41 (1), 125-132 (1997).
- 't Hart, B. A., van Kooyk, Y., Geurts, J. J. G., Gran, B. The primate autoimmune encephalomyelitis model; a bridge between mouse and man. Ann Clin Transl Neurol. 2 (5), 581-593 (2015).
- Ontaneda, D., Hyland, M., Cohen, J. A. Multiple sclerosis: new insights in pathogenesis and novel therapeutics. Annu Rev Med. 63, 389-404 (2012).
- Guy, J. R., Sati, P., Leibovitch, E., Jacobson, S., Silva, A. C., Reich, D. S. Custom fit 3D-printed brain holders for comparison of histology with MRI in marmosets. J Neurosci Methods. 257, 55-63 (2016).
- Breen, M. S., Lazebnik, R. S., Wilson, D. L. Three-dimensional registration of magnetic resonance image data to histological sections with model-based evaluation. Ann Biomed Eng. 33 (8), 1100-1112 (2005).
- Dauguet, J., et al. Three-dimensional reconstruction of stained histological slices and 3D non-linear registration with in-vivo MRI for whole baboon brain. J Neurosci Methods. 164 (1), 191-204 (2007).
- McGrath, D. M., Vlad, R. M., Foltz, W. D., Brock, K. K. Technical note: fiducial markers for correlation of whole-specimen histopathology with MR imaging at 7 tesla. Med Phys. 37, 2321-2328 (2010).
- Schormann, T., Zilles, K. Three-Dimensional linear and nonlinear transformations: An integration of light microscopical and MRI data. Hum Brain Mapp. 6, 339-347 (1998).
- Jiang, L., et al. Combined MR, fluorescence and histology imaging strategy in a human breast tumor xenograft model. NMR Biomed. 26 (3), 285-298 (2013).
- Absinta, M., et al. Postmortem magnetic resonance imaging to guide the pathologic cut: individualized, 3-dimensionally printed cutting boxes for fixed brains. J Neuropathol Exp Neurol. 73 (8), 780-788 (2014).
- Gaitán, M. I., et al. Perivenular brain lesions in a primate multiple sclerosis model at 7-tesla magnetic resonance imaging. Mult Scler. 20 (1), 64-71 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten