Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Herstellung eines UV-Vis und Raman-Spektroskopie Immunoassay-Plattform
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Nanoparticle-based optical probes have been designed as a vehicle for detecting antigens using Raman and UV-Vis spectroscopy. Here we describe a protocol for preparing such probes for a UV-Vis/Raman spectroscopy immunoassay in such a way to incorporate future multiplexing capabilities.
Zusammenfassung
Immunoassays werden verwendet, um Proteine zu detektieren basierend auf dem Vorhandensein von assoziierten Antikörpern. Aufgrund ihrer umfangreichen Einsatz in Forschung und klinischen Umgebungen kann eine große Infrastruktur von Immunoassay Instrumenten und Materialien zu finden. Beispielsweise 96- und 384-Well-Polystyrol-Platten sind kommerziell erhältlich und haben eine Standard-Design-ultraviolett-sichtbare (UV-Vis) Spektroskopie Maschinen verschiedener Hersteller aufzunehmen. Darüber hinaus ist eine Vielzahl von Immunglobulinen, Nachweis-Tags, und Blockierungsmittel für individuelle Immunoassay Designs wie enzyme-linked immunosorbent assays (ELISA) zur Verfügung.
Trotz der bestehenden Infrastruktur, Standard-ELISA-Kits erfüllen nicht alle Forschungsbedarf und erfordert individualisierte Immunoassay-Entwicklung, die teuer und zeitaufwendig sein kann. Zum Beispiel haben ELISA-Kits niedriger Multiplexen (Nachweis von mehr als einem Analyten in einer Zeit) -Funktionen, wie sie üblicherweise auf Fluoreszenz oder col hängenorimetric Methoden für die Erkennung. Kolorimetrische und fluoreszenzbasierte Analysen haben Multiplexierungsfähigkeiten aufgrund breiten spektralen Spitzen begrenzt. Im Gegensatz dazu ist die Raman-Spektroskopie-basierte Methoden haben eine viel größere Fähigkeit zum Multiplexen aufgrund enger Emissionsspitzen. Ein weiterer Vorteil der Raman - Spektroskopie ist , dass Raman - Reporter erfahren deutlich weniger Photobleichens als Fluoreszenzmarkierungen 1. Trotz der Vorteile, die Raman-Reporter haben über fluoreszierende und kolorimetrische Tags, um Protokolle herzustellen Raman-basierte Immunoassays begrenzt sind. Der Zweck dieser Arbeit ist es, ein Protokoll zu schaffen, funktionalisierte Sonden herzustellen in Verbindung mit Polystyrol-Platten für den direkten Nachweis von Analyten durch UV-Vis-Analyse und Raman-Spektroskopie verwendet werden. Dieses Protokoll wird es den Forschern ermöglichen, für die zukünftige Multi-Analyt-Detektion ein Do-it-yourself-Ansatz zu nehmen, während auf bereits etablierte Infrastruktur Kapital.
Einleitung
Typische Sandwich-Immunoassays Erkennung indirekt die Anwesenheit eines Antigens unter Verwendung zweier Antikörper. Der Capture-Antikörper an eine feste Oberfläche gebunden und bildet einen Antikörper-Antigen-Komplexes, wenn sie in der Nähe einer geeigneten Antigen. Ein Detektionsantikörper wird dann eingeführt und bindet an das Antigen. Die Antikörper / Antigen / Antikörper - Komplex bleibt nach dem Waschen, und wird von dem markierten Nachweisantikörper nachgewiesen , wie in 1A gezeigt. Typische Nachweis wird durch einen fluoreszierenden oder kolorimetrischen Detektor durchgeführt, Multiplexing zu 10 Analyten aufgrund breiten spektralen Peaks 2,3 zu begrenzen. Im Gegensatz dazu haben Raman-basierten Systemen viel schmalere Emissionspeaks was zu verbesserten Multiplexierungsfähigkeiten mit Quellen beanspruchen simultanen Nachweis von bis zu 100 Analyte 2,3.
Viele Literaturquellen zur Verfügung , die wichtige Aspekte im Zusammenhang decken Immunoassays 4 - 6 wie Schritt- für -SchrittDetails personalisierte ELISA-Kits zu erstellen. Leider sind diese Protokolle für fluoreszierende oder kolorimetrische Detektion, Multiplexing-Fähigkeit von kundenspezifischen Immunoassays begrenzen. Um diesem Bedarf zu begegnen, stellen wir ein detailliertes Verfahren der UV-Vis / Raman - Immunoassay vorher 7 für einen direkten Immunoassay veröffentlicht herzustellen , wie in 1B dargestellt.
Dieses Protokoll umfasst die Herstellung von funktionalisierten Goldnanopartikel-basierten Sonden, die in 2 dargestellt. Das Verfahren für die Raman / UV-Vis - Sonden zu machen beginnt durch Bindung Raman Reporter an der Oberfläche der Gold - Nanopartikel (AuNPs). Die AuNPs werden dann mit Antikörpern, die mit funktionalisierten Polyethylenglykol (PEG) verbunden sind. Verbleibende Bindungsstellen auf den AuNPs werden durch Bindung Methoxypolyethylenglykol thiol (mPEG-SH) zu AuNPs blockiert nachfolgende unspezifische Bindung während der Analyse zu verhindern. Die hergestellten AuNP Sonden werden durch die Bindung an Antigene getestetwie dargestellt in Figur 1B zu den Vertiefungen einer Polystyrolplatte fixiert. Nach dem Waschen der Platte werden die AuNP Sonden unter Verwendung von UV-Vis-Spektroskopie, während die zugehörigen Raman-Reporter mit Raman-Spektroskopie detektiert werden. Die Kombination von UV-Vis und Raman-Spektraldaten bietet zwei Methoden von Analysen, die Fähigkeiten dieses Immunoassays zu verbessern.
Protokoll
1. Herstellung von Puffern
- Phosphat - gepufferte Saline (PBS)
- Verdünnen Sie 50 ml 10x PBS mit 450 ml HPLC-Wasser, um eine 1x PBS Konzentration machen. Sterilfilter die Lösung mit einem 0,22 um-Filter.
- Shop-Lösung bei Raumtemperatur.
- Herstellung von Tris Buffered Saline + Tween 20 (TBST)
- Verdünnen Sie 50 ml 10x Tris-gepufferte Salzlösung (TBS) mit 450 ml HPLC-Wasser, um eine 1x Konzentration machen. Hinzufügen 250 ul Tween-20 für eine 0,05% (v / v) Tween-20. Sterilfilter die Lösung mit einem 0,22 um-Filter.
- Bei Raumtemperatur lagern.
- Herstellung von humanem Serumalbumin (HSA) Blocking Solution
- Man wiegt 0,45 g HSA in 15 ml steril filtriert 1x PBS mit 3% w / v HSA-Lösung zu machen. Vortex-Lösung bis HSA vollständig aufgelöst ist.
- Store HSA-Lösung bei 4 ° C.
HINWEIS: Rinderserumalbumin(BSA) kann auch als Blockierungslösung verwendet werden.
- Herstellung von PEGylierten Antikörpers (PEG-Ab) Lösung
HINWEIS: Die Antikörperlösung muss frei sein von Träger- oder Stabilisierungs Proteine wie BSA, das mit Konjugationsreaktionen durch Konkurrieren für die n-Hydroxysulfosuccinimid (NHS) Bindungsstellen stören würde. Wenn der Antikörper in einer Lösung, Tris- oder Glycin-Puffer kommt, muss es einen Pufferaustausch zu verhindern, Amine oder Ammoniumsalze von störenden mit dem NHS Konjugationsreaktion unterziehen. Wenn der Antikörper in lyophilisierter Form vorliegt, kann es entsprechend den Empfehlungen des Herstellers in einer Konzentration von 1-10 mg / ml resuspendiert werden.- Für Antikörper in einem Puffer Tris oder Glycin, führen Sie einen Pufferaustausch zu 100 mM Natriumbicarbonat eine Entsalzungs Spalte. Verwenden Sie die 100 mM Puffer, um den pH-Wert auf etwa 8,5 zu erhöhen, die Konjugationsreaktion zu beschleunigen.
- Hydrats ortho-Pyridyldisulfid-PEG-NHS (OPSS-PEG-NHS) mit 100 mM sVerhasstheit Bicarbonat auf ein Volumen von 1 ml bei einer Konzentration von 1 mg / ml oder mehr.
HINWEIS: OPSS-PEG-NHS frisch hergestellt und innerhalb von etwa 20 Minuten verwendet werden soll. Die NHS-Gruppe auf dem OPSS-PEG-NHS hat eine Halbwertszeit von etwa 20 min in einer wässrigen Lösung bei pH 8,5. - Hinzufügen, OPSS-PEG-NHS zu der Antikörperlösung in einem 2: 1-Verhältnis (PEG: Antikörper) conjugation Verhältnis für die Testproben verwendet werden. 1 Konjugation Verhältnis verwendet werden für die Steuerung: In einem separaten Reaktionsgefäß, OPSS-PEG-NHS an das Antigen-Lösung bei einer 2 hinzuzufügen.
HINWEIS: Das Verhältnis 2: 1 wird eine 50% ige Konjugation Effizienz annimmt. Das Ziel ist es, jeden Antikörper mit einer PEG-Kette zu beschriften. In diesem Schritt über Kennzeichnung besser ist als unter Kennzeichnung. Verwenden Sie die folgende Gleichung die entsprechenden Volumina OPSS-PEG-NHS und Antikörper-Lösung zu bestimmen:
wobei V Volumen ist, C - Konzentration in Molekülen oder ein exprimiertestibodies pro ml. Subscripts PEG und Ab sind OPSS-PEG-NHS und Antikörper, respectively. Das Endvolumen sollte ungefähr 250 & mgr; l sein. - Inkubieren PEG-Ab-Lösung bei 4 ° C für 8 Stunden oder über Nacht. Shop-Lösung in Teilmengen von etwa 25 & mgr; l bei -20 ° C arbeiten, um die Gefrier-Auftau-Zyklen zu begrenzen und sicherstellen, dass niedrige Bindungs Rohre zu verwenden.
2. Bereiten Sie UV-Vis / Raman-Sonden
- Bereiten nackten AuNP Lösung
- Bereiten Sie eine 2 ml Lösung von AuNPs mit einer Konzentration von etwa 1 x 10 11 Partikel pro ml.
- Wenn die AuNPs konzentriert werden müssen, füllen geringe Bindungszentrifugenröhrchen mit 2000 ul Lager AuNP und Zentrifuge bei 5000 × g für 20 Minuten oder bis der Überstand klar ist. Entfernen Sie den Überstand mit einer Pipette, wobei darauf geachtet, nicht die AuNP Pellet zu stören.
- Kombinieren Sie die restlichen AuNP Lösungen in ein Rohr und schätzen die Konzentration durch ein UV-Vis-Messung erhalten und Werte zu bekannten Konzentrationen verglichen, da dies eine lineare Beziehung ist.
- Bereiten Sie eine 2 ml Lösung von AuNPs mit einer Konzentration von etwa 1 x 10 11 Partikel pro ml.
- Bestimmen Sie die entsprechenden Raman - Reporter Markierungsverhältnis
- Bereiten Sie eine Arbeitslösung des Reporters Raman in Methanol gelöst. Diese Konzentration wird auf dem Reporter verwendet abhängig sein. In dieser Arbeit vorbereiten 3,3'-diethylthiatricarbocyanine Iodid (DTTC) bei einer Arbeitslösung von 200 & mgr; M.
- Unter der Annahme, auch um ein Endvolumen von 100 & mgr; l für jede fügen genug von der Arbeits reporter Lösung in jede Vertiefung der ersten Reihe einer 96-Well-Platte, so daß der Reporter Raman wird in Konzentrationen von 0,2 uM bis 10 uM reichen. Fügen Sie genug HPLC-Wasser zu jeder Vertiefung, so dass das Volumen 80 ul ist. In 20 ul AuNP in jede Vertiefung ein endgültiges Volumen von 100 & mgr; l für jede Vertiefung zu machen. Ein Beispiel ist in Tabelle 1 bereitgestellt.
- Messen der UV-Vis-Spektren von 400 bis700 nm eine Platte-Lese UV-Vis-Spektralphotometer mit. Die geeignete Konzentration ist die höchste Konzentration mit definierten Peaks für die UV-Vis-Spektren. Wiederholen Sie Schritt 2.2.2 bei Konzentrationen bis zur höchsten Konzentration Verhältnis der Raman-Reportern zu AuNPs gefunden zunimmt.
HINWEIS: Der Farbstoff und die AuNP Form, Grße und Hersteller beeinflussen die geeignete Konzentration. Daher aufgeführten Schritte müssen in Abhängigkeit von den verwendeten Komponenten ausgewertet und verändert werden. Dieses Protokoll beinhaltete die Verwendung eines positiv geladenen Farbstoff. Als solches wurde zwischen dem AuNP und Reporterbindungs verbessert durch negativ geladene AuNPs verwenden. Dies wurde unter Verwendung von Citrat capped AuNPs getan. Siehe die Diskussion Abschnitt für weitere Details.
- Raman - Bindung Reporter und PEG-Ab zu AUNP
- Bereiten zwei 1,5 ml Chargen AuNP und Raman Reporter an der zuvor bestimmten Konzentration, so dass die Raman-Reporter für 30 min bei Raumtemperatur an die AuNPs zu binden.
- Fügen Sie den PEGylierten Antikörper (PEG-Ab) zu einer Charge des AuNP und Raman-Reporter-Lösung mit einer 200 zu erstellen: 1-Verhältnis von Antikörpern gegen Partikel. Diese Lösung wird für die Testproben sein. In einem getrennten Mikrozentrifugenröhrchen, fügen das pegylierte Antigen zur anderen Charge des AuNP und Raman-Reporterlösung bei einem 200: 1-Verhältnis von Antikörper an Partikel als Kontrolle verwendet werden. Inkubieren der Lösung für 30 min bei Raumtemperatur.
ANMERKUNG: Das Verhältnis von Antikörper zu Partikeln werden den AuNPs spezifisch sein und verwendete Farbstoff und sollte für jeden Einzelfall optimiert werden. Das Ziel hier ist das höchste Verhältnis von Antikörpern zu haben, für die AuNP Sonden zu binden, während eine Aggregation der Partikel zu verhindern. Verwenden Sie die folgende Gleichung die entsprechenden Volumina zu bestimmen hinzuzufügen zusammen:
wobei V Volumen ist, C - Konzentration in Teilchen oder Antikörper pro ml ausgedrückt. Die Flosseal Volumen sollte etwa 1,5 ml betragen.
- Block verbleibenden Stellen auf der Oberfläche AuNP mit mPEG-SH.
- Bereiten Sie mPEG-SH durch Auflösen von festem Methoxypolyethylenglykol Thiol zu einem 200 & mgr; M Konzentration mit Wasser. Vortexen der Lösung, bis mPEG-SH vollständig gelöst ist.
- In mPEG-SH bei 40.000: 1-Verhältnis zu dem AuNP-PEG-Ab-Lösung in Schritt 2.3 hergestellt. Inkubieren der Lösung bei Raumtemperatur für 10 min die restlichen Stellen auf der Gold-Nanopartikel, um sicherzustellen, blockiert sind. Verwenden Sie die folgende Gleichung die entsprechenden Volumina zu bestimmen hinzuzufügen zusammen:
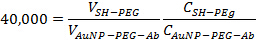
wobei V Volumen ist, C - Konzentration in Teilchen oder Antikörper pro ml ausgedrückt. Das Endvolumen sollte etwa 1,5 ml betragen.
- Recover funktionalisierte Raman - Sonden.
- Centrifuge Partikel bei 5000 × g für 20 min in niedrigen binden centrifuge Rohre oder bis der Überstand klar ist. Entfernen Sie den Überstand durch Pipettieren vorsichtig nicht die AuNPs zu stören.
- Resuspendieren der Partikel mit 1 ml 1x PBS-Lösung, die zuvor hergestellt wurde. Schätzen Sie die AuNP Konzentration durch ein UV-Vis-Messung eines kleinen Volumens von Lösung (3 & mgr; l) unter und vergleichen die Ergebnisse auf Messungen von einer bekannten AuNP Konzentration. Die Lautstärke anpassen , so daß die endgültige Lösung , die mindestens 1 x 10 11 Partikel pro ml ist.
- Store Lösungen bei 4 ° C bis es zur Funktionalisierung des Immunoassays Platte verwendet. Verwenden Sie die Lösungen innerhalb einer Woche.
| Volumes jeder Komponente hinzuzufügen (ml) | |||
| DTTC Endkonzentration (mM) | DTTC Arbeitslösung (200 mM) | AuNP | Wasser |
| 0,2 | 0,1 | 20 | 79.9 |
| 0,6 | 0,3 | 20 | 79,7 |
| 1 | 0,5 | 20 | 79.5 |
| 2 | 1.0 | 20 | 79 |
| 5 | 2.5 | 20 | 77.5 |
| 7 | 3.5 | 20 | 76.5 |
| 10 | 5.0 | 20 | 75 |
Tabelle 1. DTTC Verdünnung Beispiel. Verschiedene Verdünnungen von DTTC und die damit verbundenen Volumina Lager DTTC, Gold - Nanopartikel - Lösung und Wasser.
3. Immunoassay Plattenvorbereitung
- Binden Sie gewünschtes Antigen zu dem Immunoassay - Platte.
- Bereiten Sie genügend verdünnt Antigen (50 ug / ml), um die Polystyrol-Brunnen zu füllen. Vortex die Lösung,und sofort die Lösung für die Plattenvertiefungen hinzufügen. Ermöglichen das Antigen an die Platten für 1 Stunde bei Raumtemperatur zu binden.
- Waschen Sie die ungebundenen Antigene.
- Entfernen Sie die überschüssige Antigen-Lösung durch Lösung in einen Entsorgungsbehälter Dumping und die Platte gegen ein Papiertuch bedeckten Tisch zu schlagen.
- Hinzufügen TBST zu den Vertiefungen um die Oberfläche zu waschen dann die Wäsche in der gleichen Art und Weise zu entfernen, wie zuvor angegeben. Wiederholen Sie diesen Schritt zwei weitere Male.
- Block verbleibenden Bindungsstellen auf der Platte nicht-spezifische Bindung zu verhindern.
- Hinzufügen 70 ul HSA Blockierungslösung in jede Vertiefung der Platte und Inkubation für 30 min bei Raumtemperatur.
- Entfernen und spülen Sie die Platte nach dem gleichen Verfahren wie in Schritt 3.2 festgelegt. Decken Sie die Platte und trocken lagern bei 4 ° C bis zur weiteren Verwendung.
- Funktionalisieren Immunoassay - Platte.
- In 70 ul tSonde er Nanopartikel hergestellt in Abschnitt 2 auf die erste Säule einer 96-Well-Platte und verdünnen nachfolgenden Spalten unter Verwendung eines 1: 2 Verdünnungsreihe. Das Feinblech für mindestens 1 h inkubiert. Ein Beispiel, wie die Immunoassay - Platte herzustellen ist in Abbildung 3 angegeben.
- Waschen Sie die Platte mit TBST fünfmal wie in den Schritten 3.2 detailliert, um sicherzustellen, entsprechend der AuNPs zu entsorgen. Nach dem letzten Waschen, fügen Sie in jede Vertiefung 70 ul 1x PBS und Deckel mit einem Plattendichtung.
HINWEIS: Die Kontrollproben sollte klar sein. Wenn die nichtspezifische Bindung aufgetreten ist, besitzen die Kontrollproben eine ähnliche Farbe wie die Testproben.
- Test Testempfindlichkeit durch UV-Vis - Spektroskopie und Raman.
- Für jeden gut, messen Sie die UV-Vis-Spektren von 400 bis 700 nm im Bereich unter Verwendung eines Plattenlese UV-Vis-Spektralphotometer.
- Unter Verwendung eines invertierten Mikroskops Raman, fokussieren das Objektiv auf die Oberfläche der Vertiefung, die die AuNP Sonden aufweist. ObTain ein Raman-Spektren des Brunnens. Sammeln Sie ein Spektrum von 1.800 cm im Bereich von -1 bis 400 cm -1. Wiederholen Sie diesen Schritt für alle Vertiefungen.
- Unter Verwendung eines geeigneten spektralen Software, führen Sie eine 11 - ter Ordnung Polynom Basislinienkorrektur für die Raman - Spektren und 3. Ordnung Polynom für die UV-Vis - Spektren.
- Unter Verwendung eines geeigneten spektralen Software, normalisieren die Raman- und UV-Vis-Spektren. Stellen Sie den Maximalwert auf 1 und skaliert entsprechend alle anderen Werte. Um die Raman-Spektren normalisieren, wählen Sie eine eindeutige Polystyrol Spitze und setzte es gleich 1 und skaliert entsprechend alle anderen Werte.
- Unter Verwendung eines geeigneten spektralen Software, führen Peak-Integration für jedes Spektrum. Für Raman-Spektren, müssen in einem Bereich fehlt Polystyrol Peaks werden der Peak der Raman reporter darstellt. Um Peak-Integration durchzuführen, geben Sie die Integralgrenzen für die gewünschte Spitze und notieren Sie die gewünschte Spitzenbereich für alle Proben einschließlich der Kontrollen.
- Plot die durchschnittliche Peakfläche von Interesse als eine Funktion des Logarithmus der Konzentration AuNP mit Fehlerbalken für jeden Punkt seiner zugehörigen Standardabweichung angibt. Setzen Sie diese Kalibrierungspunkte auf eine 4-Parameter logistischen Kurve.
- Bestimmen Sie den Mittelwert des Rohlings durch die Fläche des Peaks von Interesse für eine Blindprobe gemittelt werden. Bestimmen Sie die Standardabweichung dieser Gebiete; Dies ist die Standardabweichung des Rohlings.
- Für den gleichen Peak in dem vorherigen Schritt analysiert, finden die Standardabweichung dieser Peakfläche für die niedrigste Konzentration.
- Berechnen Sie die Grenze des Rohlings und untere Nachweisgrenze, wie in der repräsentativen Ergebnisse Abschnitt angegeben. Verwenden Sie diese Werte mit den 4PL Kalibrierungskurven, die untere Nachweisgrenze in Bezug auf AuNP Konzentration zu bestimmen.
Ergebnisse
In dieser Studie wurden 60 nm Goldpartikel wurden für UV-Vis-Spektroskopie verwendet. UV-Vis - Absorptionsspektren von 400 bis 700 nm gesammelt wurden und die Peakflächen für jede AuNP Konzentration Spektralanalyse - Software 8 eine Open - Source wurden ermittelt. Vor Integration zu Peak wurde das gesammelte Spektren Basislinienkorrektur ein Drei-Punkt-Polynomfit verwenden. Peakflächen wurden verwendet , um eine logarithmische Kalibrierungskurve zu erzeugen , wie in Figur 4 gezeigt. Es is...
Diskussion
In dem detaillierten Protokoll gibt es mehrere kritische Punkte abdecken. Ein Problem ist die Wahl der Raman-Reporter und Gold-Nanopartikel. Obwohl das Protokoll für den individuellen Gebrauch angepasst werden geschrieben wurde, wurde das Raman-Reporter DTTC als Beispiel verwendet. DTTC eine positiv geladene Reporter und bindet an negativ geladenen Oberflächen, wie Citrat capped AuNPs. Dieses Protokoll kann mit einer positiven Oberflächenladung durch Verwendung von Gold-Nanopartikeln für negativ geladene Reporter an...
Offenlegungen
The authors declare that they have no competing financial interests.
Danksagungen
This work was supported by a Research Catalyst Award from Utah State University. The authors would like to thank Annelise Dykes, Cameron Zabriskie, and Donald Wooley for their contributions.
Materialien
| Name | Company | Catalog Number | Comments |
| 60 nm Gold Nanoparticle | Ted Pella, Inc. | 15708-6 | These are citrate capped gold nanoparticles. Please see Discussion for relationship between Raman reporter and AuNP surface charge and its imporance to proper selection of AuNP and/or Raman reporter. |
| Sodium Bicarbonate | Fisher Scientific | S233-500 | |
| Methanol | Pharmco-Aaper | 339000000 | |
| Tris Buffered Saline (10x) pH 7.5 | Scy Tek | TBD999 | |
| Bottle Top Filtration Unit | VWR | 97066-202 | |
| Tween 20 (polysorbate 20) | Scy Tek | TWN500 | Used as an emulsifying agent for washing steps. |
| Phosphate Buffered Saline 10x Concentrate, pH 7.4 | Scy Tek | PBD999 | |
| Protein LoBind Tube 2.0 ml | Eppendorf Tubes | 22431102 | LoBind tubes prevent binding of proteins and AuNPs to surfaces of the tubes. |
| Protein LoBind Tube 0.5 ml | Eppendorf Tubes | 22431064 | LoBind tubes prevent binding of proteins and AuNPs to surfaces of the tubes. |
| Microplate Devices UniSeal | GE Healthcare | 7704-0001 | Used for sealing and storing functionalized plates. |
| Assay Plate, With Low Evaporation Lid, 96 Well Flat Bottom | Costar | 3370 | |
| HPLC grade water | Sigma Aldrich | 270733-4L | |
| 3,3′-Diethylthiatricarbocyanine iodide (DTTC) | Sigma Aldrich | 381306-250MG | Raman reporter |
| mPEG-Thiol, MW 5,000 - 1 gram | Laysan Bio, Inc. | MPEG-SH-5000-1g | |
| OPSS-PEG-SVA, MW 5,000 - 1 gram | Laysan Bio, Inc. | OPSS-PEG-SVA-5000-1g | OPSS-PEG-SVA has an NHS end. |
| Mouse IgG, Whole Molecule Control | Thermo Fisher Scientific | 31903 | Antigen |
| Goat anti-Mouse IgG (H+L) Cross Adsorbed Secondary Antibody | Thermo Fisher Scientific | 31164 | Antibody |
| Human Serum Albumin Blocking Solution | Sigma Aldrich | A1887-1G | Bovine serum albumin can be used instead. |
| Mini Centrifuge | Fisher Schientific | 12-006-900 | |
| UV-Vis Spectrophotometer | Thermo Scientific | Nanodrop 2000c | |
| UV-Vis Spectrophotometer | BioTek | Synergy 2 | |
| Desalting Columns | Thermor Scientific | 87766 | |
| In-house built 785 nm inverted Raman microscope unit | N/A | N/A | An inverted Raman microscope is best for proper focusing onto surface of the well plate. Otherwise a very low magnification will be used due to height of the 96-well plate. An in-house built system was used as it was cheaper than buying from a vendor. However, any commercially available inverted Raman microscope system can be used. |
Referenzen
- Israelsen, N. D., Hanson, C., Vargis, E. Nanoparticle properties and synthesis effects on surface-enhanced Raman scattering enhancement factor: an introduction. Sci. World J. , e124582 (2015).
- Wang, Y., Schlücker, S. Rational design and synthesis of SERS labels. Analyst. 138 (8), 2224-2238 (2013).
- Wang, Y., Yan, B., Chen, L. SERS tags: novel optical nanoprobes for bioanalysis. Chem. Rev. 113 (3), 1391-1428 (2013).
- . . The Immunoassay Handbook: Theory and applications of ligand binding, ELISA and related techniques. , (2013).
- Cox, K. L., Devanarayan, V., Kriauciunas, A., Manetta, J., Montrose, C., Sittampalam, S. Immunoassay Methods. Assay Guid. Man. , (2004).
- . . ELISA development guide. , (2016).
- Israelsen, N. D., Wooley, D., Hanson, C., Vargis, E. Rational design of Raman-labeled nanoparticles for a dual-modality, light scattering immunoassay on a polystyrene substrate. J. Biol. Eng. 10, (2016).
- Menges, F. . Spekwin32 - optical spectroscopy software. Version 1.72.1. , (2016).
- Findlay, J. W. A., Dillard, R. F. Appropriate calibration curve fitting in ligand binding assays. AAPS J. 9 (2), E260-E267 (2007).
- Yu, X. Quantifying the Antibody Binding on Protein Microarrays using Microarray Nonlinear Calibration. BioTechniques. 54, 257-264 (2013).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. Clin. Biochem. Rev. 29 (Suppl 1), S49-S52 (2008).
- . . EP17-A2: Evaluation of Detection Capability for Clinical Laboratory Measurement Procedures; Approved Guideline. 32. No 8, (2012).
- Leigh, S. Y., Som, M., Liu, J. T. C. Method for assessing the reliability of molecular diagnostics based on multiplexed SERS-coded nanoparticles. Plos One. 8 (4), e62084 (2013).
- Sinha, L. Quantification of the binding potential of cell-surface receptors in fresh excised specimens via dual-probe modeling of SERS nanoparticles. Sci. Rep. 5, 8582 (2015).
- Shi, W., Paproski, R. J., Moore, R., Zemp, R. Detection of circulating tumor cells using targeted surface-enhanced Raman scattering nanoparticles and magnetic enrichment. J. Biomed. Opt. 19, 056014 (2014).
- Xia, X., Li, W., Zhang, Y., Xia, Y. Silica-coated dimers of silver nanospheres as surface-enhanced Raman scattering tags for imaging cancer cells. Interface Focus. 3 (3), 20120092 (2013).
- McLintock, A., Cunha-Matos, C. A., Zagnoni, M., Millington, O. R., Wark, A. W. Universal surface-enhanced Raman tags: individual nanorods for measurements from the visible to the infrared (514-1064 nm). Acs Nano. 8 (8), 8600-8609 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten