Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Medikamentöse Behandlung und
In diesem Artikel
Zusammenfassung
Small laboratory fish have become popular models for bone research on the mechanisms underlying human bone disorders and for the screening of bone-modulating drugs. In this report, we describe a protocol to assess the effect of alendronate on bone cells in medaka larvae with osteoporotic lesions.
Zusammenfassung
Knochenbildenden Osteoblasten interagieren mit dem Knochen-Osteoklasten, den Umsatz der Knochenmatrix zu koordinieren und zu Skelett Homöostase Kontrolle zu bringen. Medaka und Zebrafisch-Larven sind weit verbreitet zu analysieren das Verhalten von Knochenzellen während der Knochenbildung, Degeneration und Reparatur verwendet werden. Ihre optische Klarheit ermöglicht die Visualisierung von fluoreszierend markierten Knochenzellen und Fluoreszenz auf die mineralisierte Skelett-Matrix gebunden Farbstoffe. Unser Labor hat transgene Medaka Fisch erzeugt, die die Osteoklasten-inducing factor Receptor Activator of Nuclear-Faktor kappaB Ligand (RANKL) unter der Kontrolle eines Hitzeschock-induzierbaren Promotors exprimieren. Ektopische Expression von RANKL ergibt die überschüssige Bildung von aktivierten Osteoklasten, die in reporter Linien mit nlGFP Expression unter der Kontrolle des Cathepsin K (cSt) -Promotor sichtbar gemacht werden kann. RANKL Induktion und ektopische Osteoklastenbildung führt zu schweren Osteoporose-ähnlichen Phänotypen. Verbindung transgenen Medaka lines , die ausdrücken cSt: nlGFP in Osteoklasten sowie mCherry unter der Kontrolle des osterix (OSX) -Promotor in vorzeitigem Osteoblasten, verwendet werden , um die Interaktion beider Zelltypen zu untersuchen. Dies erleichtert die in vivo Beobachtung von Zellverhalten unter den Bedingungen der Knochenabbau und Reparatur. Hier beschreiben wir die Verwendung dieses Systems ein Medikament häufig in menschlichen Osteoporose-Therapie verwendet zu testen und ein Protokoll für Echtzeit-Bildgebung beschrieben. Das Medaka - Modell ergänzt Studien in Zellkultur und Mäuse und bietet ein neuartiges System für die in vivo Analyse der Arzneimittelwirkung im Skelettsystem.
Einleitung
Die wirbel Skelett sorgt für eine strukturelle Unterstützung und Schutz von Organen ermöglicht Mobilität und dient als Quelle für Calcium. Im Laufe des Lebens wird die extrazelluläre Knochenmatrix kontinuierlich gedreht über die Knochenstabilität und Steifigkeit zu erhalten. Dieser Prozess erfordert die eng koordinierte Aktivität und das Zusammenspiel von Knochen bildenden Osteoblasten und Knochen-Osteoklasten. Osteoblasten sind von multipotenten mesenchymalen Vorläuferzellen abgeleitet und produzieren Kollagen die osteoid zu bilden, die proteinartiges Teil der Knochenmatrix 10. Osteoblasten mit Osteoklasten zusammenwirken , eine ausgewogene Aktivität beider Zelltypen zu erreichen, die Knochenhomöostase 7 zu steuern , erforderlich ist. Aufgrund dieser komplizierten regulatorischen Wechselwirkungen, Antworten auf eine medikamentöse Therapie und Knochenhomöostase nicht voll werden kann unter Verwendung von in vitro - Studien untersucht. Daher gibt es eine starke Nachfrage nach Tiermodellen. Im Vergleich zu den Zellkultur - Einstellungen können in - vivo - Modellen liefernwertvolle Einblicke in die vielzelligen Netzwerke innerhalb des Knochenumgebung.
Zahlreiche Mausmodelle existieren für eine Vielzahl von menschlichen Knochenerkrankungen einschließlich Osteoporose 16. Allerdings stellen die Größe und die Zugänglichkeit von Maus-Embryonen erhebliche Einschränkungen für die Live-Darstellung von Skelett Prozesse. Kleinknochenfischen, andererseits dienen als attraktive Alternative für die in vivo Bildgebung. Zebrabärbling (Danio rerio) und Medaka (Oryzias latipes) haben für Skelett-Forschung in den letzten zwei Jahrzehnten beliebte Tiermodelle werden 17, 19, 22, 24. Knochen in Knochenfischen und in Säugetieren ist sehr ähnlich, sowohl auf struktureller und auf physiologischer Ebene und viele der wichtigsten regulatorischen Gene und Signalwege sind 3 konserviert. Wie bei Säugetieren, Knochenfische regulieren sorgfältig die Aktivität von Osteoblasten und Osteoklasten 26 Knochenbildung und Resorption zu balancieren. Am wichtigsten ist, die optische Klarheit der fish Larven ermöglicht die Verwendung von Fluoreszenz - Reportern , Knochenzellen und die verkalkten Skelettmatrix 8, 9, 12, 21, 23, die die Beobachtung von zellulären Prozessen in lebenden Tier erleichtert zu etikettieren. Darüber hinaus hat eine Reihe von genetischen Werkzeugen erzeugt worden biomedizinisch relevante Forschung in den Fischen zu erleichtern. Für Medaka insbesondere Verfahren zur gezielten Genveränderung von CRISPR / Cas9 2, Zell-Linie Tracing 6 und ortsspezifische Transgene 14 haben nun weit verbreitet in Gebrauch 15 vor kurzem gegründet und sind sind.
Kleine Teleost Larven wurden für chemische Bildschirme, die zur Entdeckung von mehreren pharmakologisch relevanten Wirkstoffe 1, 18 eingesetzt.
Fischlarven sind tolerant gegenüber niedrigen Konzentrationen von DMSO und können Verbindungen aus ihren Gewässern zu absorbieren, entweder durch die Haut oder durch den Gastrointestinaltrakt 1, 5. Unser Labor zuvor reported transgenen Medaka Linien, die fluoreszierende Reporter in Knochenzellen unter der Kontrolle von verschiedenen Osteoblasten und Osteoklasten spezifischen Promotoren exprimieren. Dazu gehören vorzeitige Osteoblasten (Kollagen 10a1, Col10a1; osterix, osx) 20, 21, reifen Osteoblasten (Osteocalcin, osc) 27, und Osteoklasten (Cathepsin K, cSt) 24. Wir erzielten auch eine transgene Linie, die die Osteoklasten-inducing factor Receptor Activator von 24 Nuclear-Faktor kappaB Ligand (RANKL) unter der Kontrolle eines Hitzeschock-induzierbaren Promotor exprimiert.
Induktion von RANKL in diesem System ergibt die ektopische Bildung von aktiven Osteoklasten. Dies führt zu erhöhten Knochenresorption und eine schwere Osteoporose-ähnlichen Phänotyp, mit drastisch reduziert Mineralisierung in den Wirbelkörpern. Wir haben kürzlich gezeigt, dass die Aktivität der Osteoklasten in diesem Modell kann durch die Bisphosphonate Etidronat und Alendronat blockiert werden, two Medikamente häufig in menschlichen Osteoporose - Therapie, so Medaka als ein geeignetes Modellsystem für Osteoporose 27 zu validieren.
Aufgrund ihrer großen Brutgröße, schnelle Entwicklung, und die geringe Größe von Embryonen, sind transgene Medaka Larven in einzigartiger Weise geeignet für die groß angelegte Screening von Osteoporose Medikamente und für die in - vivo - Analyse von Knochenzellverhalten. so können Studien in Medaka effizient Experimente in Zellkulturen und bei Mäusen zu ergänzen, die auf die Entdeckung neuer therapeutischer Ziele und neuartige Therapien für menschliche Knochenerkrankungen abzielen.
In der vorliegenden Studie beschreiben wir ein Protokoll Medaka Knochen-Reporter Larven mit dem gemeinsamen Osteoporose-Medikament, Alendronat zu behandeln. Wir beschreiben auch im Detail, wie behandelten Larven montiert sind und für die Live-Darstellung von Knochenmatrix und Knochenzellen hergestellt. Diese Protokolle können leicht auf andere kleine chemische Verbindungen, die entweder Arbeit als Knochen anabole oder antiresorptiv Medikamente angepasst werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Experimente wurden in Übereinstimmung mit genehmigten Institutional Animal Care und Use Committee (IACUC) Protokolle der National University of Singapore (R14-293) durchgeführt.
1. Fischhaltung und die Sammlung von Embryonen
- Heben Sie WT, cSt: nlGFP 24, RANKL: HSE: CFP 24 und osx: mCherry 21 ein- oder Verbindung transgenen Medaka Fisch bei 26 ° C unter einem gesteuerten Lichtzyklus (14 h Licht, 10 h Dunkelheit) zu induzieren Laichen.
- Täglich Laich findet während der ersten 30 Minuten, nachdem das Licht eingeschaltet wird. Die Eier halten zusammen durch Filamente und zu den weiblichen Bauch für mehrere Stunden befestigen. Verwenden Sie ein feinmaschiges Netz mit einer weiblichen Erwachsenen trägt ein Ei-Cluster zu fangen. Lassen Sie die Fische kurz im Netz ruhen und dann sanft massieren den Bauch der Fische vorsichtig die befruchtete Eizelle Cluster aus dem Bauch des Weibchens abzustreifen.
HINWEIS: Eine gesunde Medaka weiblichfür ca. 5 Monate 20 Eier pro Tag - können 10 produzieren. - Legen Sie die Eier in eine 60 mm Petrischale aus Kunststoff. Verwenden einer Plastikpipette die Embryonen mit 5 auszuspülen - 10 ml 0,3x Danieau-Lösung (Fischmedium; 19,3 mM NaCl, 0,23 mM KCl, 0,13 mM MgSO 4, 0,2 mM Ca (NO 3) 2 und 1,7 mM HEPES, pH 7.0). 1 ml einer 0,25% (w / v) Methylenblau-Stammlösung auf 2,5 L Fischmedium Pilzwachstum zu verhindern.
- Sanft Ei Cluster rollen einen Knoten der Befestigung Filamente zu bilden. Verwenden einer Pinzette vorsichtig die Befestigungsfäden aus der befruchteten Eizelle Cluster entfernen , um einzelne Embryonen (1A) erhalten.
- Bühne , um die Embryonen nach Iwamatsu 2004 13.
- Kultur 20 bis 30 Embryonen pro 60 mm Kunststoff-Petrischale in einem 28 ° C Inkubator. Ändern, um die mittlere tägliche normale Entwicklung des Embryos zu gewährleisten.
HINWEIS: Die Zeit um die Schraffur Stufe (8 - 9 d postfertilization,DPF) ist besonders kritisch für das Überleben. Entfernen Chorion frei schwebenden das Medium sauber zu halten und um eine gute Larvenüberlebensraten.
2. Transgene Embryo Screening
- Verwenden Sie ein Stereomikroskop mit einer Quecksilberlampe ausgestattet für Fluoreszenz-Imaging und GFP, RFP und GFP Filter transgenen Embryonen für Fluoreszenzreporter-Expression mit 40-facher Vergrößerung zu screenen.
- Identifizieren Visuell osx: mCherry Embryonen durch mCherry Reporter Ausdruck in der frühen Bildung Schädelknochen, wie die Cleithrum, auf beiden Seiten des hinteren Kopf (Abbildung 1B, Pfeil), und der Parasphenoid, an einer zentralen Stelle im ventralen Schädel ( 1B, Pfeilspitze).
HINWEIS: Reporter Ausdruck beginnt ab 5 DPF ab 21. - Identifizieren cSt: nlGFP Embryonen , die durch starke nlGFP Ausdruck im Kopf (1C, Pfeil) und Schwanz (1C, Pfeilspitze), ausgehend von6 DPF.
HINWEIS: Endogene Osteoklasten erst nach 21 DPF bilden. nlGFP-exprimierenden Zellen in diesem frühen Stadium (6 DPF) nicht Osteoklasten aber auch andere, bisher nicht charakterisierten, CTSK -positiven Zellen 24. - Identifizieren RANKL: HSE: GFP transgenen Embryonen , die durch allgegenwärtige GFP - Expression nach einer kurzen Hitzeschockbehandlung für 20 min bei 39 ° C, bei 2 DPF durchgeführt oder später für Screening - Zwecken.
HINWEIS: Die RANKL und CFP Transgene stehen unter der Kontrolle des gleichen bidirektionalen Heat Shock Element (HSE). GFP - Expression zeigt die erfolgreiche RANKL Induktion 24. - Durchführen einer 1,5-2 h Hitzeschockbehandlung bei 9 DPF oder später eine große Anzahl von ektopischen Osteoklasten im Rumpfbereich zu induzieren, die folglich in einem Osteoporose-ähnlichen Phänotyp führt 24.
HINWEIS: Die transgene Expression RANKL induziert bei 9 DPF führt zu einer ektopischen Aktivierung ruhender Osteoklasten-Vorläuferzellen, die endogen nicht ausgelöst wirdvor 21 DPF. Verwenden Sie ein Wasserbad stabil 39 ° C Bedingungen zu erhalten. Lassen Sie die Petrischale mit Medaka Embryonen auf der Wasseroberfläche schwimmen. Stellen Sie sicher, dass der Deckel der Petrischale ist trocken die Versenkung der Schale zu verhindern. - Bildschirm Embryonen aus Verbindung Linien, wie RANKL: HSE: CFP / cSt: nlGFP doppelt transgenen und osx: mCherry / RANKL: HSE: CFP / cSt: nlGFP triple-transgenen nach dem Expressionsmuster jedes einzelnen Transgen.
HINWEIS: Hemizygote und homozygote wurden transgene Embryonen, die durch unterschiedliche Fluoreszenzwerte des Reporter-Transgen aus. Homozygote Embryonen hatten eine Fluoreszenzintensität, die ungefähr doppelt so im Vergleich zu der hemizygous transgenics. Compound Linien , die für beide RANKL homozygot waren: HSE: CFP und cSt: nlGFP wurden durch wiederholte erhalten mehrere Generationen Einkreuzung über. Für Triple-transgenen osx: mCherry / RANKL: HSE: CFP / cSt: nlGFP Fisch, homozygot RANKL: HSE: CFP / cSt: nlGFP Fische wurden mit homozygot osx gekreuzt: mCherry Träger. Die resultierende heterozygote triple-transgenen Nachkommen wurden angehoben und incrossed Embryonen für RANKL homozygot zu erhalten: HSE: GFP. Die RANKL: HSE: GFP - Transgen muss, um homozygot sein , um die effiziente Induktion der ektopischen Osteoklasten zu erhalten.
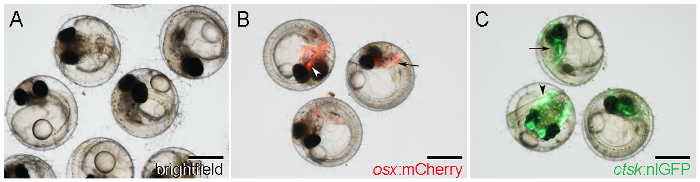
Abbildung 1: WT und transgener Medaka Embryonen bei 7 D Postfertilization (DPF). A. WT Embryonen mit Hellfeldbeleuchtung beobachtet. B. Transgene Embryonen zeigt osx: mCherry Ausdruck um den Cleithrum (Pfeil) und Parasphenoid (Pfeilspitze). C. Transgene Embryonen zeigt cSt: nlGFP Ausdruck im Kopf (Pfeil) und Schwanz (Pfeilspitze). Maßstabsbalken: 500 & mgr; m.025 / 55025fig1large.jpg "target =" _ blank "> Bitte hier klicken, um eine größere Version dieser Figur zu sehen.
3. Bisphosphonat Behandlung von Medaka Larvae
- Herstellung von Lösungen, die verschiedene Konzentrationen an Bisphosphonaten (BPs) zur Dosis-Wirkungs-Studien.
HINWEIS: Die beispielhafte BP in diesem Protokoll verwendet ist Alendronat.- Löse Alendronat in Fisch Medium in einer Konzentration von 100 ug / ml einer Stammlösung herzustellen.
- Mit einem Wirbelmischer eine vollständige Auflösung zu gewährleisten. Bewahren Sie die Stammlösung bei 4 ° C.
- Bereiten verschiedene Arbeitslösungen durch Verdünnen der Stammlösung mit Fisch Medium zu einer Reihe von Konzentrationen (dh, 25, 37,5, 50, 62,5 und 75 & mgr; g / ml).
HINWEIS: Verschiedene Medikamente können unterschiedliche Absorption, Verteilung, Metabolismus haben und Ausscheidung (ADME) Parameter, die während des Tests mit dieser Medaka Larven System berücksichtigt werden müssen. Auch Arzneistofflöslichkeit und Stabilität kann variieren, wennals wässrige Lösung aufgetragen. Weniger wasserlösliche Verbindungen können müssen zunächst in organischen Lösungsmitteln, wie DMSO, gelöst werden. In diesem Fall wird eine Stammlösung in DMSO hergestellt, die weiter wird dann in Fischmedium verdünnt. Beachten Sie, dass die Arbeitslösungen in Wasser (Fisch mittel) kann im Kühlschrank für mehrere wk gespeichert werden. Jedoch DMSO enthaltende Lösungen müssen bei RT gelagert werden, um Kristallisation zu verhindern.
- Transfer Medaka Larven in Sechs-Well-Platten (sechs Larven / Vertiefung) für nachfolgende BP (Alendronat) Behandlung.
- Entfernen Sie den Fisch Medium vorsichtig mit einem sauberen Plastikpipette und fügen Sie ein kleines Volumen (ca.. 0,5 ml) von Alendronat-Lösung in jede Vertiefung.
- Vermeiden Sie übrig gebliebenen Fischmedium, wie der zusätzliche BP Lösung könnte verdünnt werden, was besonders wichtig ist für weniger konzentriert Alendronat-Lösungen.
- Entfernen Sie ein kleines Volumen von Alendronat-Lösung (bis zu 0,5 ml) von jeder Vertiefung mit einem sauberen Plastikpipette und ersetzen Sie es mit einem größeren Volume (4 ml) von Alendronat-Lösung.
- Ändern Medium täglich normalen Embryonen Entwicklung zu gewährleisten.
4. Live-Färbung von mineralisierte Knochenmatrix
- Man löst 0,5 g Alizarin-Komplexon (ALC, Alizarin-3-Methyliminodiessigsäure) oder 0,05 g von Calcein in 50 ml Fischmedium 1% und 0,1% ige Stammlösungen herzustellen, respectively. Mit einem Wirbelmischer eine vollständige Auflösung zu gewährleisten.
HINWEIS: Fisch Medium ohne Zusatz von Methylenblau wird in diesem und in den nachfolgenden Schritten verwendet Autofluoreszenz in den Larven zu reduzieren. - Verwenden Sie eine Spritze und Einwegfilter (0,2 & mgr; m), um die Farblösung zu filtern. Bewahren Sie die gefilterten Lösung im Dunkeln bei RT.
HINWEIS: Die Farbe des gefilterten, klare ALC-Färbelösung ist dunkelgelb bis orange. Die Farbe des gefilterten, klar Calcein Lösung ist leuchtend gelb. Lösungen können für mehrere Monate verwendet werden. - Verdünnen Sie die gefilterten ALC oder Calcein Stammlösung 1:10 in Fisch Mediumund brüten die Medaka Larven für 1,5 bis 2 h (0,1% ALC-Lösung) oder 2-2,5 h (0,01% Calcein-Lösung) in einem 28 ° C Inkubator, wenn Larven zwischen 9 und 17 DPF verwendet werden. Halten Sie die Proben im Dunkeln.
- Übertragen Sie die Larven zu frischem Fisch Medium, das eine saubere Plastikpipette.
- Entfernen Sie den Fisch Medium mit einem sauberen Plastikpipette und fügen Sie frischen Fisch Medium. Wiederholen Sie diesen Schritt für 3 bis 4-mal, bis keine rot- oder gelb-gefärbte Lösung (ALC oder Calcein, beziehungsweise) übrig bleiben. Lassen Sie die Larven in Fisch Medium für 30 - 60 Minuten vor dem sie für die Bildgebung zu vermeiden Epifluoreszenz aus dem Medium Montage.
HINWEIS: 0,1% ALC-Färbelösung ist schädlich für die Medaka Larven für längere Belichtungszeiten. Inkubationszeiten von mehr als 2 h beeinflussen das Überleben der Larven. Konzentration und Färbezeit müssen daher für verschiedene Stufen optimiert werden, um eine optimale Embryo Überleben und Färbung Ergebnisse zu erzielen.
5. Live-Fluoreszenz-Imaging p>
- Anesthetize die Medaka Larven mit 0,01% Tricaine (Ethyl-3-aminobenzoat methansulfonat) in Fisch Medium.
HINWEIS: Narkotisierte Larven immobilisiert werden nach 5 - 10 min in Tricaine Lösung und sind in der Regel liegen entweder auf ihren Seiten oder den Rücken. - Verwenden Sie einen Kunststoff Micro die Larven zu orientieren nach der Region von Interesse. Die Orientierung der Larven in diesem Protokoll verwendet ist lateral.
- Verwenden Sie ein Stereomikroskop mit Fluoreszenzbeleuchtung für die Bildgebung. Verwenden hoher Vergrößerung, wenn die Aufnahme von Bildern, auf verschiedenen Teilen der Larven Fokussierung (Kopf, vordere Rumpf, hintere Rumpf und Schwanz). Sticheinzelbilder zusammen an überlappenden Bereichen unter Verwendung eines geeigneten Bildverarbeitungssoftware (Einfügungen in Figur 3G).
HINWEIS: Dies hilft, die Bildqualität aller relevanten Körperteile in der richtigen Brennebene zu verbessern. - Bringen Sie die Larven zum Fisch Medium für die Erholung nach der Abbildung.
- Anesthetize die Larven mit 0,01% Tricaine in Fisch Medium 5 - 10 min, bis sie immobilisiertem werden.
- Auflösen niedrigschmelzenden Agarose zu 1,5% in Fischmedium durch sie in einem Mikrowellenofen erhitzt wird. Kühle die Lösung auf etwa 30 ° C.
- In 0,5-1 ml Flüssigkeit 1,5% niedrig schmelzenden Agarose in Fischmedium zu einem Glasboden-Petrischale. Übertragen Sie die betäubten Larven in die Lösung eine saubere Plastikpipette.
HINWEIS: Besondere Vorsichtsmaßnahme, dass die Temperatur der Flüssigkeit mit niedrigem Schmelz Agarose niedrig genug ist, nicht die Larven zu schaden. - Bevor die Agarose erstarrt, mit einem Kunststoff Micro die Larven auf den Boden der Petrischale zu schieben und zu orientieren, die Larven nach der Region von Interesse. Die Orientierung der Larven in diesem Protokoll verwendet ist lateral.
HINWEIS: Die Proben sind bereit für die konfokale Live-Bildgebung, nachdem die Agarose vollständig erstarrt. - Verwenden Sie ein konfokales Mikroskop zu ACQuire Bilder.
- Verwenden Sie einen 543-nm-Laserlinie für mCherry und ALC-Färbung analysiert. Verwenden Sie einen 488-nm-Laserlinie für nlGFP und Calcein-Färbung analysiert.
- Nach der Bebilderung Fischmedium auf die Petrischale hinzufügen und ein Paar von feinen Injektionsnadeln (27 G x 1 ½ ") verwenden, um sorgfältig die Larven aus der Agarose zu entfernen. Übertragen Sie die Larven mit residually Agarose gebunden in eine Petrischale mit Fisch Medium zu erholen .
- Verarbeiten Sie die Bilder mit einer Bildanalysesoftware 27 verwendet wird .
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Reichlich Ei Zahlen, sowie die geringe Größe der Larven, machen Medaka ein hervorragendes Modell für Wirkstoff-Screening. Eine einzige Sechs-Well-Platte wurde bis zu 36 Larven zur Kultur verwendet, die statistisch signifikante Daten zu liefern ausreichend war. Ein weiterer großer Vorteil der Verwendung von Fisch für Skelett-Analyse ist die Möglichkeit der Live-Bildgebung zu tun. Die Transparenz von Fischlarven ermöglicht die Verwendung von fluoreszierenden Proteinen Knochenzellen zu kennzeichnen, sowie die Verwen...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Kritische Schritte im Rahmen des Protokolls
Es ist wichtig, dass die Bedingungen für die Hitzeschockbehandlung sind konsistent und stabil, wenn verschiedene Proben zu vergleichen. Stabile Temperaturbedingungen garantieren ähnliche Niveaus von RANKL - Induktion in transgenen Larven und damit vergleichbar Osteoklastenbildung, die durch Screenen auf cSt bestätigt werden kann: nlGFP Ausdruck. Letztendlich führt dies zu einem ähnlichen Grad an induzierter ectopic Knochenresorption und ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass sie keine oder finanzielle Interessen haben.
Danksagungen
Dieses Projekt wurde durch Zuschüsse aus dem Singapur Bildungsministerium (MOE, Grant-Nummer 2013-T2-2-126) und das National Institute of Health, USA finanziert (NIH gewähren Nummer 1R21AT008452-01A1). TY erhielt ein Diplom-Stipendium der NUS Department of Biological Sciences. Wir danken der konfokalen Einheit der NUS Zentrum für Bioimaging Sciences (CBIS) für ihre ständige Unterstützung.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Alendronate | Sigma | A4978 | |
| alizarin-3-methyliminodiacetic acid, Alizarin Complexone | Sigma | A3882 | |
| Calcein | Sigma | C0875 | |
| ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma | A5040 | |
| ImageJ (1.4.3.67) | National Institute of Health (NIH) | https://imagej.nih.gov/ij/ | |
| LSM 510 Meta confocal | Zeiss | ||
| LSM Image Browser (4.2.0.121) | Zeiss | http://www.zeiss.com/microscopy/en_de/downloads/lsm-5-series.html | |
| Micro-loader | Eppendorf | 5242956003 | Eppendorf ep T.I.P.S 20 μL |
| NIS-Elements BR 3.0 software | Nikon | ||
| Photoshop CS6 (13.0.0.0) | Adobe | ||
| SMZ1000 stereomicroscope | Nikon |
Referenzen
- Ablain, J., Zon, L. I. Of fish and men: using zebrafish to fight human diseases. Trends Cell Biol. 23 (12), 584-586 (2013).
- Ansai, S., Kinoshita, M. Targeted mutagenesis using CRISPR/Cas system in medaka. Biol Open. 3 (5), 362-371 (2014).
- Apschner, A., Schulte-Merker, S., Witten, P. E. Not all bones are created equal-using zebrafish and other teleost species in osteogenesis research. Methods Cell Biol. 105, 239-255 (2011).
- Bajoghli, B., Aghaallaei, N., Heimbucher, T., Czerny, T. An artificial promoter construct for heat-inducible misexpression during fish embryogenesis. Dev Biol. 271 (2), 416-430 (2004).
- Barrett, R., Chappell, C., Quick, M., Fleming, A. A rapid, high content, in vivo model of glucocorticoid-induced osteoporosis. Biotechnol J. 1 (6), 651-655 (2006).
- Centanin, L., Ander, J. J., Hoeckendorf, B., Lust, K., Kellner, T., Kraemer, I., Urbany, C., Hasel, E., Harris, W. A., Simons, B. D., et al. Exclusive multipotency and preferential asymmetric divisions in post-embryonic neural stem cells of the fish retina. Development. 141 (18), 3472-3482 (2014).
- Charles, J. F., Aliprantis, A. O. Osteoclasts: more than 'bone eaters. Trends Mol Med. 20 (8), 449-459 (2014).
- DeLaurier, A., Eames, B. F., Blanco-Sanchez, B., Peng, G., He, X., Swartz, M. E., Ullmann, B., Westerfield, M., Kimmel, C. B. Zebrafish sp7:EGFP: a transgenic for studying otic vesicle formation, skeletogenesis, and bone regeneration. Genesis. 48 (8), 505-511 (2010).
- Du, S. J., Frenkel, V., Kindschi, G., Zohar, Y. Visualizing normal and defective bone development in zebrafish embryos using the fluorescent chromophore calcein. Dev Biol. 238 (2), 239-246 (2001).
- Eriksen, E. F. Cellular mechanisms of bone remodeling. Rev Endocr Metab Disord. 11 (4), 219-227 (2010).
- Hockendorf, B., Thumberger, T., Wittbrodt, J. Quantitative analysis of embryogenesis: a perspective for light sheet microscopy. Dev Cell. 23 (6), 1111-1120 (2012).
- Inohaya, K., Takano, Y., Kudo, A. The teleost intervertebral region acts as a growth center of the centrum: in vivo visualization of osteoblasts and their progenitors in transgenic fish. Dev Dyn. 236 (11), 3031-3046 (2007).
- Iwamatsu, T. Stages of normal development in the medaka Oryzias latipes. Mech Dev. 121 (7), 605-618 (2004).
- Kirchmaier, S., Hockendorf, B., Moller, E. K., Bornhorst, D., Spitz, F., Wittbrodt, J. Efficient site-specific transgenesis and enhancer activity tests in medaka using PhiC31 integrase. Development. 140 (20), 4287-4295 (2013).
- Kirchmaier, S., Naruse, K., Wittbrodt, J., Loosli, F. The genomic and genetic toolbox of the teleost medaka (Oryzias latipes). Genetics. 199 (4), 905-918 (2015).
- Komori, T. Animal models for osteoporosis. Eur J Pharmacol. 759, 287-294 (2015).
- Mackay, E. W., Apschner, A., Schulte-Merker, S. A bone to pick with zebrafish. Bonekey Rep. 2, 445(2013).
- MacRae, C. A., Peterson, R. T. Zebrafish as tools for drug discovery. Nat Rev Drug Discov. 14 (10), 721-731 (2015).
- Mitchell, R. E., Huitema, L. F., Skinner, R. E., Brunt, L. H., Severn, C., Schulte-Merker, S., Hammond, C. L. New tools for studying osteoarthritis genetics in zebrafish. Osteoarthritis Cartilage. 21 (2), 269-278 (2013).
- Renn, J., Buttner, A., To, T. T., Chan, S. J., Winkler, C. A col10a1:nlGFP transgenic line displays putative osteoblast precursors at the medaka notochordal sheath prior to mineralization. Dev Biol. 381 (1), 134-143 (2013).
- Renn, J., Winkler, C. Osterix-mCherry transgenic medaka for in vivo imaging of bone formation. Dev Dyn. 238 (1), 241-248 (2009).
- Schilling, T. F., Kimmel, C. B. Segment and cell type lineage restrictions during pharyngeal arch development in the zebrafish embryo. Development. 120 (3), 483-494 (1994).
- Spoorendonk, K. M., Peterson-Maduro, J., Renn, J., Trowe, T., Kranenbarg, S., Winkler, C., Schulte-Merker, S. Retinoic acid and Cyp26b1 are critical regulators of osteogenesis in the axial skeleton. Development. 135 (22), 3765-3774 (2008).
- To, T. T., Witten, P. E., Renn, J., Bhattacharya, D., Huysseune, A., Winkler, C. Rankl-induced osteoclastogenesis leads to loss of mineralization in a medaka osteoporosis model. Development. 139 (1), 141-150 (2012).
- Wakamatsu, Y., Pristyazhnyuk, S., Kinoshita, M., Tanaka, M., Ozato, K. The see-through medaka: a fish model that is transparent throughout life. Proc Natl Acad Sci USA. 98 (18), 10046-10050 (2001).
- Witten, P. E., Huysseune, A. A comparative view on mechanisms and functions of skeletal remodelling in teleost fish, with special emphasis on osteoclasts and their function. Biol Rev Camb Philos Soc. 84 (2), 315-346 (2009).
- Yu, T., Witten, P. E., Huysseune, A., Buettner, A., To, T. T., Winkler, C. Live imaging of osteoclast inhibition by bisphosphonates in a medaka osteoporosis model. Dis Model Mech. 9 (2), 155-163 (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten