Method Article
Ein Hochdurchsatz-Zellarray-Plattform für korrelative Analyse von Zelldifferenzierung und Zugkräften
In diesem Artikel
Zusammenfassung
Die Zelldifferenzierung wird durch eine Vielzahl von Mikroumgebungsfaktoren reguliert, einschließlich der beiden Matrixzusammensetzung und Substratmaterialeigenschaften. Wir beschreiben hier eine Technik unter Verwendung von Zellmicroarrays in Verbindung mit Traktionskraftmikroskopie sowohl Zelldifferenzierung und biomechanischen Zell-Substrat-Wechselwirkungen als Funktion der Mikroumgebungskontext zu bewerten.
Zusammenfassung
Mikrofabrizierten zelluläre Mikroarrays, welche der kontakt gedruckten Kombinationen von Biomolekülen auf einem elastischen Hydrogel Oberfläche bestehen, bieten eine streng kontrolliert, Hochdurchsatz-engineered System für den Einfluss von angeordneten biochemische Signale auf die Zelldifferenzierung zu messen. Die jüngsten Bemühungen der Zelle unter Verwendung von Microarrays haben ihre Nützlichkeit für kombinatorische Studien nachgewiesen, in denen viele Mikroumgebungsfaktoren sind parallel dargestellt. Allerdings haben diese Bemühungen konzentriert sich hauptsächlich auf die Auswirkungen der biochemischen Signale auf Zell-Reaktionen zu untersuchen. Hier präsentieren wir eine Zelle Microarray-Plattform mit abstimmbaren Materialeigenschaften für die Beurteilung sowohl der Zelldifferenzierung durch Immunofluoreszenz und biomechanische Zell-Substrat-Wechselwirkungen, die durch Traktion Kraftmikroskopie. Dazu haben wir zwei verschiedene Formate unter Verwendung von Polyacrylamid-Hydrogele unterschiedlicher Young-Modul entweder auf Mikroskop-Objektträger oder Glasboden-Petrischalen hergestellt entwickelt. Wir bieten BESt Praktiken und Problemlösungen für die Herstellung von Microarrays auf diesen Hydrogel-Substrate, die nachfolgende Zellkultur auf Mikroarrays, und der Übernahme von Daten. Diese Plattform ist gut geeignet für die Verwendung bei Untersuchungen der biologischen Prozesse , bei denen sowohl biochemische (beispielsweise extrazelluläre Matrixzusammensetzung) und biophysikalischen (beispielsweise Substrat Steifigkeit) cues signifikante spielen können, sich schneidende Rollen.
Einleitung
Wechselwirkungen zwischen Zellen und der umgebenden Mikroumgebungsfaktoren vermitteln eine Vielzahl von biologischen Prozessen während der Entwicklung, Homöostase und Pathogenese der Erkrankung 1, 2, 3, 4. Diese Mikroumgebungs Wechselwirkungen umfassen die Lieferung von löslichen Faktoren zu Zellen, Zell-Matrix-Bindung, und die Zell-Zell-Wechselwirkungen über Ligand-Rezeptor-Bindung. Zusätzlich zu den oben genannten biochemischen Überlegungen biophysikalischen Parameter, wie Substrat mechanischen Eigenschaften (beispielsweise Elastizitätsmodul, Porosität) und der Zellform und die damit verbundenen nachgeschalteten Mechanotransduktion zunehmend Anerkennung als wichtige Mediatoren der Zelldifferenzierung gewonnen haben , 5, 6, 7, 8, 9 10. Die Signale aus diesen Mikroumgebungs Wechselwirkungen dienen als Eingänge für Gen-Netzwerke und Signalwege. Darüber hinaus sind diese Zellen intrinsische Komponenten bieten auch Rückmeldung an die Mikroumgebung über sekretierte Faktoren und Matrix-abbauende Enzyme, eine komplexe co-Regelungsschleife zwischen zell intrinsischen genetischen Programme und zell extrinsische Faktoren Mikroumgebungs 5, 11, 12 vervollständigt.
Die Verwendung von technischen Systemen für die kontrollierte Darstellung von Mikroumgebungsfaktoren wurde in einer Reihe verschiedener Kontexte 13, 14, 15 bewährt. Mikrofabrizierte Systeme haben insbesondere präzise räumliche Strukturierung von Proteinen und Zellen erleichtert sowie hoch parallelisierte Analyse über Miniaturisierung 13, 16, 17, 18, 19, 20, 21, 22. Zelle Mikroarrays stellen eine solche mikrofabrizierten System , in dem Kombinationen von Biomolekülen sind berührungs gedruckt auf einem elastischen Polyacrylamidhydrogel Substrat 23, 24, 25. Die Einbeziehung von zellKlebstoffKomponenten (nämlich Matrixproteine) ermöglicht anhalt Zelladhäsion und Kultur auf Mikroarrays, die häufig durch nachgeschaltete Analyse mittels Immunzytochemie und fluoreszierende Reporter folgt. Zellmikroarrays wurden zur Verwirklichung eines verbesserten Verständnis von Leberzell - Phänotyp 23, 26, neurale Vorläufer Differenzierung 27, produktiv gerichtet Mammary Vorläufer Entscheidungen Schicksal 28, embryonalen Stammzellen Wartung / Differenzierung 23, 29, 30, Metastasen Lungenkrebs 31 und therapeutische Reaktion bei Melanom 32. Wir haben vor kurzem gezeigt , die Verwendung von Zell - Microarrays zur Bestimmung der Rolle der extrazellulären Matrix (ECM) Proteinzusammensetzung in Endoderm Spezifikation 33, Lebervorläufer Differentiation 34, 35, und die Zelllungentumorarzneimittelantwort 36. In diesen Arbeiten haben wir uns auf die Erweiterung der kombinatorischen Fähigkeiten der Array-Plattform und die Erkundung der Kreuzungen von Zell-Eigen Signalisierung mit extrazellulären Matrixzusammensetzung und Biomechanik konzentriert. Darüber hinaus haben wir biophysikalischen Ablesungen in diesem Array Plattform implementiert quantitativ Eigenschafte, um die Möglichkeit zu bieten,zeichnen die Rolle der Zellkontraktilität in Differenzierungsprozessen 35. Dazu integriert wir Traktionskraftmikroskopie (TFM) mit Zellmicroarrays Hochdurchsatzbewertung von zell erzeugten Traktion zu ermöglichen. TFM ist ein weithin genutzt Verfahren zell erzeugten Zugkräfte zu messen und hat sich mit der Zusammensetzung und der Biomechanik des lokalen Mikro 37, 38, 39, 40 in Bezug auf die Koordinierung der einzelnen Zelle und Gewebe-Ebene Funktion wichtige Erkenntnisse zur Verfügung gestellt. Somit liefert für Meßtaste, physiologisch relevanten biophysikalischen Parameter eines Hochdurchsatzsystem mit Zellmicroarrays TFM kombiniert.
Die Zelle Microarray-Plattform hier beschriebene besteht aus vier Abschnitten: Herstellung von Polyacrylamid-Substrate, die Herstellung von Arrays, Zellkultur und Assay Auslesung und Analyse von Daten. SehenFigur 1 eine schematische Zusammenfassung der ersten drei Versuchsabschnitte; siehe Abbildung 2 für eine schematische Zusammenfassung des letzten Abschnitts mit einem Schwerpunkt auf der Analyse von Immundaten. Um die Zelle Microarray - Plattform auf Studien von biomechanischen Zell-Substrat - Wechselwirkungen anzupassen, haben wir Polyacrylamid - Substrate von abstimmbaren Elastizitätsmodul , aber ähnliche Porosität, je Wen et al. 41. TFM Messungen der Kräfte, die durch Zellen, die auf ihrem Substrat ausgeübt zu ermöglichen, haben wir ein Glasboden-Petrischale Format neben dem dicken Glasobjektträgern durch andere Gruppen häufig genutzt. Somit ist diese Zelle Microarray Plattform, die parallel Messungen der Zelldifferenzierung mittels Immunofluoreszenz auf Objektträgern und zell erzeugten Kräfte über TFM auf separaten Glasbodenschalen. Wir haben auch eine Reihe von Verbesserungen an den analytischen Ansatz häufig verwendete mit Zellmikroarrays angewendet. specifically statt parametrischer Z-Scoring der gesamten Insel Intensität messen wir Intensität Einzelzelle und Quantil Normalisierung, um für Nicht-Normalverteilungen und genauer Zellverhalten beschreiben zu berücksichtigen gilt. Wir glauben, dass diese Verbesserungen einen besonderen Nutzen auf Untersuchungen von biologischen Prozessen schaffen, bei dem sowohl biochemische und biophysikalische Signale signifikant, sich schneidende Rollen spielen. Ferner ermöglichen es unseren analytischen Verbesserungen, um die Anwendung von Zellmikroarrays Studien einer Reihe von zellulären Funktionen, für die das Verhalten Einzelzelle und Bevölkerungsebene abweichen.
Protokoll
1. Herstellung von Polyacrylamid-Substrate
- Saubere Glassubstrate - entweder Standard-Mikroskop-Objektträger für die Endpunkt Immunofluoreszenz oder Glasboden 35 mm Petrischalen für TFM - um eine optimale Polyacrylamid-Hydrogel-Herstellung und Integrität während der Zellkultur zu gewährleisten. Alternativ Verwendung von Glassubstraten vorgereinigt.
- Tauchen Glassubstrate in 0,25% v / v Triton X-100 in destilliertem Wasser (dH 2 O). Platzieren Substrate auf einem Orbitalschüttler für 30 min.
- Entfernen Triton X-100 - Lösung und spülen Substrate 5 - mal mit dH 2 O Leave Substrate in der letzten Spülung und auf einem Schüttler getaucht 30 min.
- Entfernen dH 2 O und tauchen Substrate in Aceton. Platzieren Substrate auf einem Orbitalschüttler für 30 min.
- Entfernen Aceton und tauchen Substrate in Methanol. Platzieren Substrate auf einem Orbitalschüttler für 30 min.
- Entfernen Methanol und spülen Substrate 5-mal mit dH 2 O. Immerse Substrate in 0,05 N NaOH und auf einem Orbitalschüttler für 1 h.
ACHTUNG: NaOH ist stark ätzend und kann schwere Verätzungen der Haut und Augenschäden verursachen. Schutzhandschuhe tragen, Kleidung und Augenschutz. - Entfernen NaOH - Lösung und spülen Substrate 5 - mal mit dH 2 O Verwenden gefilterte Druckluft Substrate zu trocknen und backen bei 110 ° C auf einer heißen Platte , bis sie trocken (5 bis 15 min). Gereinigte Substrate können auf unbestimmte Zeit bei Raumtemperatur gelagert werden.
- Silanisieren saubere Glassubstrate, um Befestigung des Polyacrylamid-Hydrogel zu gewährleisten.
- Tauchen saubere Glassubstrate in frisch hergestelltem 2% v / v 3- (Trimethoxysilyl) propyl-methacrylat (3-TPM) in Ethanol. Platzieren Substrate auf einem Orbitalschüttler für 30 min.
ACHTUNG: 3-TPM ist eine brennbare Flüssigkeit. Halten Sie weg von Hitze, Funken, offenem Feuer und heißen Oberflächen und verwenden Sie nur in einer chemischen Abzugshaube. - Entfernen Sie 3-TPM-Lösung und tauchen Substrate in ethanicht ich. Platzieren Substrate auf einem Orbitalschüttler für 5 min.
- Verwenden Sie gefilterte Druckluft, die Substrate, um zu trocknen und backen bei 110 ° C auf einer heißen Platte, bis sie trocken (5 bis 15 min). Silanisierte Substrate können für bis zu 1 Monat bei Raumtemperatur gelagert werden.
- Tauchen saubere Glassubstrate in frisch hergestelltem 2% v / v 3- (Trimethoxysilyl) propyl-methacrylat (3-TPM) in Ethanol. Platzieren Substrate auf einem Orbitalschüttler für 30 min.
- Option 1: Fabrizieren Polyacrylamid-Hydrogele auf silanisierte Objektträger für die Endpunkt Immunofluoreszenz.
- Bereiten Sie eine Präpolymerlösung in dH 2 O mit den gewünschten Acrylamid / Bisacrylamid Prozentsatz (w / v) Verhältnis herzustellen Substraten mit Youngsche Moduli von 4 kPa (4% Acrylamid, 0,4% Bisacrylamid), 13 kPa (6% Acrylamid, 0,45 Bisacrylamid%) oder 30 kPa (8% Acrylamid, 0,55% Bisacrylamid) und ähnliche Porosität pro Wen et al. 41. Vortex-Lösung, bis sie klar und Filter mit einer 0,2 & mgr; m Spritze. Pre-Polymer-Lösungen können bei 4 ° C für 3 Monate gelagert werden.
ACHTUNG: Die Exposition gegenüber Acrylamid oder Bisacrylamid kann in der akuten Toxizität führen, neurotoxicity und Irritation. Schutzhandschuhe tragen, Kleidung und Augenschutz. - Vorbereiten eines Photoinitiator-Lösung von 20% w / v Irgacure 2959 in Methanol. Dieser Photoinitiator-Lösung kann nicht gespeichert werden und müssen frisch jedes Mal hergestellt werden.
- Mischen das Vorpolymer und Photoinitiator-Lösungen in einem 9: 1 (Vorpolymer: Photoinitiator) -Verhältnis. Optional entgasen mit einer Vakuumkammer für 15 min Blasen zu entfernen.
- Legen Sie silanisiert gleitet in eine Glastrockenschale und pipettieren 100 & mgr; l von 9: 1 Pre-Polymer: Photoinitiator-Lösung auf jeder Folie. Sanft decken jede Folie mit einem Deckglas 22 × 60 mm, während die Erzeugung von Blasen zu vermeiden. Man beachte, dass das Deckglas Hemmung der Polymerisationsreaktion durch Sauerstoff verhindert.
- Platz Trockenschale in einem UV - Vernetzer und belichten Folien bis 365 nm UV - A für 10 min (4 W / m 2). Optimieren Sie Polymerisationszeit nach Bedarf. Längere Belichtungszeiten Risiko Schwierigkeit das Deckglas aufgrund overpolymerization entfernen. Kürzere Belichtungen risk underpolymerization und niedrige Hydrogel Stabilität.
- Tauchen Hydrogele in dH 2 O für 5 min. Entfernen Sie Deckgläser mit einer Rasierklinge, dabei nicht die polymerisierten Hydrogele zu beschädigen.
- Verlassen Hydrogele in dH 2 O bei Raumtemperatur von 1 bis 3 d, Ändern dH 2 O täglich. Entwässern Hydrogele bei 50 ° C auf einer heißen Platte, bis sie trocken (15 - 30 min) und bei Raumtemperatur lagern bis zu 3 Monaten.
- Bereiten Sie eine Präpolymerlösung in dH 2 O mit den gewünschten Acrylamid / Bisacrylamid Prozentsatz (w / v) Verhältnis herzustellen Substraten mit Youngsche Moduli von 4 kPa (4% Acrylamid, 0,4% Bisacrylamid), 13 kPa (6% Acrylamid, 0,45 Bisacrylamid%) oder 30 kPa (8% Acrylamid, 0,55% Bisacrylamid) und ähnliche Porosität pro Wen et al. 41. Vortex-Lösung, bis sie klar und Filter mit einer 0,2 & mgr; m Spritze. Pre-Polymer-Lösungen können bei 4 ° C für 3 Monate gelagert werden.
- Option 2: Fabrizieren fluoreszierende Kügelchen enthaltende Polyacrylamid-Hydrogele auf silanisierte 35 mm Glasboden-Petrischalen für die Live-Auswertung von Zell-Substrat-Wechselwirkungen TFM verwendet wird.
- Beschallen eine Stammlösung von 1 & mgr; m fluoreszierende Kügelchen für 15 min Aggregate zu dispergieren.
- Bereiten Sie eine Präpolymerlösung in dH 2 O mit den gewünschten Acrylamid / Bisacrylamid Prozentsatz (w / v) Verhältnis herzustellen Substraten mit Youngsche Moduli von 4 kPa (4% Acrylamid, 0,4% Bisacrylamid), 13 kPa (6% Acrylamid, 0.45% Bisacrylamid) oder 30 kPa (8% Acrylamid, 0,55% Bisacrylamid) und ähnliche Porosität pro Wen et al. 41. Vortex-Lösung, bis sie klar und Filter mit einer 0,2 & mgr; m Spritze. Pre-Polymer-Lösungen können bei 4 ° C für 3 Monate gelagert werden.
ACHTUNG: Die Exposition gegenüber Acrylamid oder Bisacrylamid kann in der akuten Toxizität, Neurotoxizität führen, und Irritation. Schutzhandschuhe tragen, Kleidung und Augenschutz. - Hinzufügen fluoreszierenden Kügelchen in die Vorpolymer-Lösung bei einer Endkonzentration von 0,2% v / v und Wirbel zu mischen.
- Vorbereiten eines Photoinitiator-Lösung von 20% w / v Irgacure 2959 in Methanol. Dieser Photoinitiator-Lösung kann nicht gespeichert werden und müssen frisch jedes Mal hergestellt werden.
- Mischen Sie die Vorpolymer / Wulst und Photoinitiator-Lösungen in einem 9: 1 (pre-Polymer / bead: Photoinitiator) -Verhältnis. Optional entgasen mit einer Vakuumkammer für 15 min Blasen zu entfernen.
- Legen Sie silanisiert 35 mm Glasboden-Petrischalen in eine Glastrockenschale und pipet 20 & mgr; l von 9: 1 Pre-Polymer / Kornes: Photoinitiator-Lösung auf die Mitte jedes Gericht. Abdeckung vorsichtig jede Folie mit einem 12 mm großen kreisförmigen Deckglas, während die Erzeugung von Blasen zu vermeiden. Man beachte, dass das Deckglas Hemmung der Polymerisationsreaktion durch Sauerstoff verhindert.
- Um die fluoreszierende Kügelchen an der Oberfläche des Hydrogels zu verteilen, invertieren die Gerichte und 20 min, pro Knoll et al bei Raumtemperatur belassen. 42.
- Während noch umgekehrt wird , belichten Gerichte bis 365 nm UV - A für 10 min (4 W / m 2). Optimieren Sie Polymerisationszeit nach Bedarf. Längere Belichtungszeiten Risiko Schwierigkeit das Deckglas aufgrund overpolymerization entfernen. Kürzere Belichtungen Risiko underpolymerization und niedrige Hydrogel Stabilität.
- Tauchen Hydrogele in 0,1 M 4- (2-Hydroxyethyl) -1-piperazinethansulfonsäure (HEPES) Puffer und bei Raumtemperatur im Dunkeln über Nacht belassen. Entfernen Sie vorsichtig Deckgläser mit einem Rasiermesser, kümmert sich nicht um die polyme zu beschädigentigten Hydrogelen.
- Entwässern Hydrogele bei 50 ° C auf einer heißen Platte, bis sie trocken (15 - 30 min). Hydrogele können bei Raumtemperatur im Dunkeln für 3 Monate gelagert werden.
2. Herstellung von Arrays
- Bereiten Puffer Biomolekülen zu drucken. Verwenden Sie den Druckpuffer entsprechend den Biomolekülen von Interesse. Wachstumsfaktor (GF) Druckpuffer ist allgemein geeignet für andere Klassen von Molekülen, wie Zell-Zell-Liganden.
- Zur Herstellung von 2 × ECM - Protein Druckpuffer, fügen 164 mg Natriumacetat und 37,2 mg (EDTA) bis 6 ml dH 2 O Vortex und Inkubation bei 37 ° C bis gründlich löslich zu machen . 50 & mgr; l vorgewärmtes Triton X-100 und 4 ml Glycerol Nach der Solubilisierung hinzuzufügen. Vortex und inkubiere wieder bei 37 ° C löslich zu machen. In 40 - 80 Für ul Eisessig, titriert den pH-Wert auf 4,8 einzustellen. 2 × ECM-Protein Druckpuffer kann bei 4 gelagert werden° C für 1 Monat.
ACHTUNG: Essigsäure ist leicht entzündlich und ätzend. Schutzhandschuhe tragen, Kleidung und Augenschutz. - Zur Herstellung von 2 × GF Druckpuffer, fügen 105,5 mg Natriumacetat und 37,2 mg EDTA zu 6 ml phosphatgepufferter Kochsalzlösung (PBS). Vortex und bei 37 ° C inkubieren, um gründlich solubilisieren. Nach der Solubilisierung mit 100 mg 3 - [(3-Cholamidopropyl) dimethylammonium] -1-propansulfonat (CHAPS) und 4 ml Glycerin. 2 × GF Protein Druckpuffer kann bei 4 ° C für 1 Monat gelagert werden.
- Zur Herstellung von 2 × ECM - Protein Druckpuffer, fügen 164 mg Natriumacetat und 37,2 mg (EDTA) bis 6 ml dH 2 O Vortex und Inkubation bei 37 ° C bis gründlich löslich zu machen . 50 & mgr; l vorgewärmtes Triton X-100 und 4 ml Glycerol Nach der Solubilisierung hinzuzufügen. Vortex und inkubiere wieder bei 37 ° C löslich zu machen. In 40 - 80 Für ul Eisessig, titriert den pH-Wert auf 4,8 einzustellen. 2 × ECM-Protein Druckpuffer kann bei 4 gelagert werden° C für 1 Monat.
- Bereiten Sie Quellenplatte.
- In einem 384-Well-V-Boden Mikrotiterplatten, kombinieren gleiche Volumen von 2 × Druckpuffer mit jedem Biomolekül-Lösung mit der doppelten Zielkonzentration.
HINWEIS: Eine geeignete Zielkonzentration für die gebräuchlichsten ECM Proteine beträgt 250 & mgr; g / ml, während Zielkonzentrationen für andere Arten von Array-Faktoren auf die Retention in dem Hydrogel und biologische Funktion variieren. Das Gesamtvolumen in jeder Vertiefung sein kannso niedrig wie 5 & mgr; l und brauchen nicht mehr als 15 & mgr; l sein. Zusätzlich zu den Biomolekül Kombinationen von Interesse, gehören gruppierten Fluoreszenzmarker, um nachgeschaltete Bildanalyse zu erleichtern. Verwenden Rhodamin-konjugiertem Dextran (2,5 mg / ml). - Mischen Sie jede Vertiefung gründlich durch Pipettieren, kümmert sich nicht um Blasen zu erzeugen. Zentrifugieren Sie die Quelle Mikrotiterplatten für 1 min bei 100 × g. Fabrizieren Mikroarrays unter Verwendung von Quellenplatten am gleichen Tag hergestellt und bei 4ºC bis zur Mikroarrayherstellung gespeichert.
- In einem 384-Well-V-Boden Mikrotiterplatten, kombinieren gleiche Volumen von 2 × Druckpuffer mit jedem Biomolekül-Lösung mit der doppelten Zielkonzentration.
- Saubere Stifte entsprechend den Anweisungen des Herstellers vor jedem Microarray-Fertigung laufen. Laden sauber Stifte direkt in den Druckkopf des Microarrayers.
- Bereiten Sie Microarrayers und Programm der Software des Herstellers verwendet wird. Obwohl die folgenden Schritte zum Teil speziell für den bestimmten Microarrayers hier verwendet werden, ist der Betrieb der meisten Microarrayer ähnlich.
- Schalten Sie die Befeuchtereinheit, stellen Sie den Sollwert auf 65% RH (nicht-condensing), und warten Sie, bis das Rheometer den Sollwert übereinstimmt. Setzen Sie Source-Platte in den entsprechenden Adapter.
- Entwässern Hydrogel Substrate bei 50 ° C für 15 min und legen Sie in den entsprechenden Adapter. Die Microarrayers hat Adapter für beide Objektträger und Mikrotiterplatten. Für Gruppierungs 35 mm Glasboden-Petrischalen, laden das Geschirr in einer 6-Well-Mikroplatte und legen Sie die Mikrotiterplatte in die Mikrotiterplatten-Adapter auf dem Arrayers.
- Passen Sie die Parameter des Programms genau das Layout der Quellplatte, Array - Design reflektieren und gewünschte Format (zB Mikroskop - Objektträger oder Mikrotiterplatten mit 35 mm Petrischalen). Fügen Waschschritte sowohl Wasser und Dimethylsulfoxid (DMSO) zwischen jeder Bedingung, um mit den Übertrag und eine Kreuzkontamination zu vermeiden.
- Starten Sie Arrayherstellung, die Überprüfung nicht seltener als einmal pro Stunde, die die Luftfeuchtigkeit unter 65% RH (nicht kondensierend) nicht gesunken ist und dass die Stifte nicht verstopft sind. Wenn der humidity wurde unerwartet gesunken, Pause Gruppierungs den Befeuchter zu füllen und klar zugeordnete Röhren der Kondensation. Wenn die Stifte verstopft sind, unterbrechen Sie die Stifte zu reinigen Gruppierungs oder auf andere Weise mit vorgereinigte Stifte ersetzen. Man beachte , dass es möglich ist, Array mehrere Typen von Biomolekülen nacheinander auf den gleichen Substraten ausreichende Trocknungszeit vorgesehen (dh 4 h bis über Nacht).
- Sobald das Programm abgeschlossen ist, legen hergestellt Arrays in einer Dia-Box oder Mikrotiterplatte mit Aluminiumfolie abgedeckt bei Raumtemperatur und 65% relative Luftfeuchtigkeit (nicht kondensierend) über Nacht. Beachten Sie, dass es notwendig sein kann Array Qualität und Retention unter Verwendung der allgemeinen Protein Flecken oder Immunofluoreszenz zu bewerten; siehe Brafman et al. Weitere Einzelheiten 25.
3. Zellkultur und Assay Ablesbarkeit
- Der Tag nach der Herstellung, legen angeordnet Substrate in 4-Kammer-Gerichte (Objektträger) oder 6-Well-Mikroplatten (Petrischalen) und tauchen in 1%v / v Penicillin / Streptomycin in PBS; Verwenden Sie 4 ml für Dias und 3 ml für Gerichte. Expose für 30 min mit UV-C. Austausch Penicillin / Streptomycin-Lösung für Zellkulturmedien.
- Sammeln und Zählen Zellen. Seed auf Arrays bei 500 × Oktober 3-02 × 10 6 Zellen / array bei 4 ml pro Objektträger und 3 ml pro 35 mm Petrischale. Inkubieren Array Kulturen bei 37 ° C und 5% CO 2 für 2 - 24 h , oder bis zur Bildung von gut besiedelten Zellinseln. Passen beide Aussaatdichte und Zeit für Ihre Zellen und bestimmte Anwendung benötigt wird. Underseeding (dh mit geringer Dichte oder Seeding Zeit) in einem schlechten Array Bevölkerung und schiefe biologischen Ergebnissen führen kann. Nachsaat (dh mit hoher Dichte oder Seeding Zeit) in reduzierter Array Integrität führen kann aufgrund Insel Ablösung.
- Nach Berücksichtigung der Bildung von Zellinseln, Array Kulturen zweimal mit vorgewärmten Zellkulturmedien waschen; Verwenden Sie erneut 4 ml für Dias und 3 ml für Gerichte. optionahinzufügen lly geeignete Kontrollen und Behandlungen (zB kleine Molekül - Inhibitoren, Wachstumsfaktoren, etc.) von Interesse für das biologische System. Ändern Sie die Medien der Arrays alle 1 bis 2 d, um die Konzentration von irgendwelchen Behandlungen zu erhalten. Bewerten Zellmarker Expression und Zellfunktion durch Immunofluoreszenz oder Zell-Substrat-Wechselwirkungen von TFM innerhalb von 1 bis 5 d Array Kulturen zu initiieren - siehe Option 1 und Option 2 unten.
- Option 1: Führen Endpunkt Immunofluoreszenz. Man beachte, dass Immunofluoreszenz einiger Proteine strengerer Permeabilisierung Methanol erfordern, Ethanol oder HCl. Aufgrund möglicher Arrays zu Schaden, zu bewerten und jedes Permeabilisierung Protokoll in größerem Maßstab Experimente vor der Verwendung zu optimieren.
- Aspirat Zellkulturmedien von Array Dias in 4-Kammer-Gerichten und 4 ml / Objektträger von frisch hergestelltem 4% v / v Paraformaldehyd (PFA) in PBS. Inkubieren für 15 min bei Raumtemperatur.
ACHTUNG: Messenure zu PFA kann in der akuten Toxizität führen und kann auch Hautkontakt Reizungen auslösen oder korrodieren. Schutzhandschuhe tragen, Kleidung und Augenschutz und verwenden Sie nur in einer chemischen Abzugshaube. - Absaugen PFA-Lösung und Waschen Sie jeden Schieber 3 mal mit 4 ml PBS. An diesem Punkt können fixierte Objektträger bei 4 ° C für 1 Woche gelagert werden. Es ist jedoch ratsam, durch Immunmarkierung und Montage am selben Tag wie die Befestigungs um weiterhin Array Integrität zu gewährleisten.
- Absaugen PBS und 4 ml / Dia von 0,25% v / v Triton X-100 in PBS. Inkubieren für 10 min bei Raumtemperatur.
- Absaugen Triton X-100-Lösung und Waschen Sie jeden Schieber 3 mal mit 4 ml PBS. 4 ml / Objektträger von 5% v / v Serum abgestimmt auf die Art des sekundären Antikörpers (zB Eselserum für Esels sekundäre Antikörper) in PBS und Inkubieren bei Raumtemperatur für 1 h.
- Gründlich entfernen Sie die Blockierungslösung aus jeder Folie. Hinzufügen, 500 & mgr; l / slide des primären Antikörpers, verdünnt in 5% v / v Serum in PBS. Dieses Volumen ist ausreichend Arrays bei Raumtemperatur sowie über Nacht Inkubationen bei 4 ° C für beide 1 h Inkubationen zu decken.
- Waschen jedes Array slide 3-mal mit 4 ml PBS. Gründlich dem letzten Waschen entfernen, und 500 & mgr; l / slide des geeigneten sekundären Antikörper in 5% v / v Serum in PBS verdünnt hinzu.
- Waschen jedes Array slide 3-mal mit 4 ml PBS. Waschen Sie kurz mit dH 2 O vor sorgfältig Array Dias aus der Lösung mit einer Pinzette entfernen. Verwenden Sie ein Labor Gewebe Docht oder trockene Rest dH 2 O.
- Jeweils 100 & mgr; l über die Dia-Lösung mit DAPI Montage während visuell eine vollständige Abdeckung des gesamten Array bestätigt.
- Legen Sie eine 22 × 60 mm Deckglas über den Objektträger zu montieren. Seal die Ränder des Deckglases mit klarem Nagellack. Lagerung im Dunkeln bei 4 ° C bis zur Bildgebung, frühestens am nächsten Tag.
- Bild gesamte Arrays entweder einen Microarray-Scanner oder invertiert Fluoreszenzmikroskop equipp mited mit einem Roboter-Bühne. Microarray - Scanner liefern schneller Auslesung kann aber Cy3- oder Cy5-kompatible Fluorophore und sind oft von begrenzter Auflösung bei der Bestellung von einzelnen Zellen erfordern (dh 1 - 10 & mgr; m). Fluoreszierende Mikroskope bieten die Möglichkeit, eine Vielzahl von Fluoreszenz Kanäle zu nutzen und eine höhere Auflösung (<1 & mgr; m, ~ 100 × Gesamtvergrößerung), aber langsamer Auslese bieten auf die Qualität der Roboterstufe und Vergrößerung / Ziel abhängig.
- Speichern aufgenommenen Bilder ganzer Arrays aus beiden Methoden als TIFF - Dateien , um die Datenkomprimierung oder Verlust mit anderen Dateiformaten (zB JPG) assoziiert zu verhindern.
- Aspirat Zellkulturmedien von Array Dias in 4-Kammer-Gerichten und 4 ml / Objektträger von frisch hergestelltem 4% v / v Paraformaldehyd (PFA) in PBS. Inkubieren für 15 min bei Raumtemperatur.
- Option 2: Führen Sie Live-Auswertung von Zell-Substrat-Wechselwirkungen TFM verwendet wird.
- Bereiten einer Lösung von 1% v / v Rinderserumalbumin (BSA) und 1% v / v Natriumdodecylsulfat (SDS) in PBS-Zellen von Substraten während TFM zu dissoziieren.
- Verschieben 35 mm Petrischalen mit Array Kulturenein inkubiert (37 ° C, 5% CO 2), invertierte Fluoreszenzmikroskop mit einem Roboter - Stufe für TFM - Messungen.
- In einer Schüssel, Phasenkontrastmikroskopie markieren die Positionen (X-Koordinate, Y-Koordinate) und Fokusebenen (Z-Koordinate) einzelner Zell Inseln mit.
- Wechseln Sie zu weit roten Fluoreszenzmikroskopie, um die Perlen zu visualisieren. Zurück zu jeder der Positionen im vorherigen Schritt gespeichert und korrigieren die Z-Koordinate der Fokusebene, so dass nur die erste Schicht aus Perlen unterhalb der Zelle Insel im Fokus ist. Speichern Sie die neuen Koordinaten und fahren Sie mit automatisierten Imaging aller Zell Inseln Prädissoziation Phasenkontrast und weit roten Fluoreszenzbilder zu erfassen.
- Vorsichtig 150 ul BSA / SDS-Lösung in die Schale und warten 5 Minuten für eine vollständige Zelle Dissoziation von dem Substrat zu ermöglichen; Zelldissoziationsmedium unter Verwendung von Phasenkontrastmikroskopie überwachen.
- Nachdem die Zellinseln wurden von dem Substrat dissoziiert wurden, kehren zu ter markiert Positionen und prüfen Sie, dass die erste Schicht der Kügelchen noch im Fokus sind. Wenn diese Kügelchen aufgrund der Verformung durch zell erzeugte Traktion induzierte out-of-Ebene sind, korrigieren Sie die Z-Koordinate der Fokusebene, so dass sie wieder im Mittelpunkt stehen. Speichern Sie die korrigierten Z-Koordinaten und wiederholen automatisierte Abbildung von allen Inseln post-Dissoziation weit rot fluoreszierende Bilder zu erfassen.
- Wiederholen Sie die Schritte 3.5.1 - 3.5.4 für die restlichen Gerichte.
4. Analyse der Daten
- Analyse der Immunfluoreszenz-Daten.
- Prozess erworben Array Bilder. Split - Verbund Array Bilder in Dateien mit einzelnen Kanäle (dh rot, blau oder grün) und Konvertieren in 8-Bit - TIFF - Bilder 43, 44. Bewerben Binning (zB 2 × 2 oder 4 × 4) zu reduzieren Bildgröße ~ 32 Megapixel pro Kanal Speicherbedarf während der Downstream - Einzelzellanalyse von enti zu reduzierenRe-Array Bilder. Siehe Ergänzenden Code Datei mit dem Titel "array_processing.ijm" für eine ImageJ Makro Umsetzung dieser Array Verarbeitungsschritte.
- Beachten Sie die Koordinaten in Pixeln von oben links, unten links und unten rechts Rhodamin-konjugierten Dextran Marker oder angeordnet Bedingungen. Verwenden Sie diese Koordinaten, die die 8-Bit-TIFF-Bilder zu drehen, um perfekt vertikal und später mit Anmerkungen versehen Ausgang von der Einzelzellanalyse mit spezifischen Bedingungen angeordnet. Siehe Ergänzenden Code Dateien Titel "rb_array_rotater.ijm", "rg_array_rotater.ijm", "rgb_array_rotater.ijm" und "array_gridding.ijm" für Implementierungen dieser Anordnung dreht und Schritte Rasterung.
- Führen Sie Einzelzellanalyse von binned, gedreht 8-Bit - TIFF - Bilder in CellProfiler (Version 2.1.1) 45 unter Verwendung der folgenden Module: IdentifyPrimaryObjects, IdentifySecondaryObjects und MeasureObjectIntensity. IdentifyPrimaryObjects identifiziert Kerne, IdentifySecondaryObjects identifiziert immunolabels mit jedem Zellkern verbunden sind, und MeasureObjectIntensity bietet Quantifizierungen für beide Kern Etiketten und immunolabels.
- Ausgabeeinzelzellendaten von allen drei Modulen als CSV-Datei durch den Kanal ExportToSpreadsheet Modul später Downstream-Analyse zu erleichtern. Siehe Ergänzenden Code Dateien Titel "b_array_image_analysis.cppipe", "gb_array_image_analysis.cppipe", "rb_array_image_analysis.cppipe" und "rgb_array_image_analysis.cppipe" für CellProfiler Pipelines diese Schritte für Bildsätze enthalten, rot, grün implementieren oder blauen Kanäle.
- Um die Datentransformation für die experimentelle Variabilität Rechnung zu tragen und nicht-Gauß - Einzelzellverteilungen gelten Quantils Normalisierung durch biologische Replikation 46. Dieser Prozess erzeugt eine gemeinsame Verteilung über Replikate und ermöglicht unvoreingenommene Vergleiche von Veränderungen in immunolabel Intensität. Darüber hinaus unlIKE-Z-scoring und andere parametrische Methoden, Quantil Normalisierung nicht-parametrischer und übernimmt nicht eine bestimmte Verteilung von Daten für mehrere repräsentative ermöglicht Analysen von Einzelzellverhalten in Abhängigkeit von angeordneten Zustand.
- Plot-Daten und zu interpretieren. Je nach dem biologischen System und Hypothese, zu berechnen und eine oder mehrere der folgenden Ensemble Maßnahmen für jedes aufgereiht Zustand Grundstück:
- Berechnen und Zellen pro Insel als kombiniertes Maß der Haftung und das Überleben im Laufe des Experiments plotten.
- Berechnen und zeichnen Sie die Quantil-normalisiert immunolabel Intensität als Maß für das Zellschicksal oder Funktion.
- Berechnen und geplottet den Prozentsatz der Zellen, die positiv für einen immunolabel wie durch Intensität oberhalb einer einheitlichen Schwelle bestimmt, in der Regel 2 SD über dem mittleren Intensität einer negativen Kontrolle.
- Alternativ Grundstück Verteilungen von immunolabel Intensität, um Einzelzellverhalten zu untersuchen und kategorisierenals Funktion der angeordneten Zustand. Diese Verteilungen können weiter charakterisiert werden durch Maßnahmen der zentralen Tendenz (Mittelwert, Median, Modus) und Variation (Varianz, Variationskoeffizient, Fano-Faktor) und Hypothesentestverfahren wie der Kolmogorov-Smirnov-Test.
- Analyse von TFM Daten. Hier ist ein Ansatz , Einbau eines zuvor entwickelten Algorithmus , der von Butler et al. und Wang et al. 40, 47.
- Verwenden Sie ImageJ Batch-Konvertierung der Bilder in 8-Bit-TIFF-Dateien. Anwenden Pixel gemittelten Binning (zB 2 × 2) , um den Rechenaufwand und Zeit nachgeschalteter Analyse zu reduzieren. Als Algorithmen für TFM weitgehend auf Einzelzellanalyse, die große Zell-Substrat - Grenzfläche der Inseln (~ 17,5 × 10 3 & mgr; m 2) im Vergleich zu der Zell-Substrat - Grenzfläche einer Einzelzelle (75 & mgr; m 2) konzentriert necessiTates der Binning-Schritt.
- Geben Sie die erfassten Phasenkontrast und weit rot fluoreszierende Bilder (beide Prädissoziation und post-Dissoziation) in eine wissenschaftliche Programmierumgebung wie MATLAB und Verfahren , die zuvor entwickelten Algorithmen von Butler et al. und Wang et al. 40, 47.
- Wählen Sie drei Regionen weit entfernt von der Zelle Insel. Diese Regionen werden für die Verschiebungen Drift aufgrund Bild oder Muster zu berücksichtigen.
- Geben Sie den Faktor von Pixeln & mgr ; m zu konvertieren (zB 0.454 Pixel / um), der Young - Modul des Substrats ( zum Beispiel 13 kPa) und das Poisson-Verhältnis (zB 0,48 für die Polyacrylamidgelen hier beschrieben).
- Für jede Insel, die eine Grenze um den Umfang zeichnen, um die geometrischen Beschränkungen zu definieren; alle Kräfte außerhalb dieser Grenze werden auf Null gesetzt. Dieses eingeschränkte System ist sinnvoll angesichts der großen distance (dh 450 & mgr; m) zwischen den Inseln.
- Berechnen Sie den root mean square Zugspannung und Kontraktions Moment für jede Insel. Die kontraktilen Moment ist ein Maß für die Restspannung an der Zelle Insel und wurde die Stärke der Zell-Zell - Interaktionen 48 widerzuspiegeln gezeigt. Für jede angeordneten Zustand, durchschnittliche root mean square Werte über mehrere Inseln und biologischen Replikaten und berechnen Varianz für Hypothesentests in Verbindung gebracht. Es ist auch möglich , die Verteilung der Beanspruchungen und Momente über viele Inseln zu mitteln einen repräsentativen Karte beider Maßnahmen als Funktion der Geometrie zu schaffen, zum Beispiel Entfernung von der Mitte der Insel.
Ergebnisse
Mit dieser Plattform haben wir untersucht die Rolle der beiden biochemischen und biophysikalischen Signale in das Schicksal Spezifikation der Leber Vorläufern 34, 35. Protein A / G-konjugierten Notch - Liganden zeigten verbesserte Retention und clustering in dem Polyacrylamid - Hydrogel (3A) und waren weiterhin fähig ist zu einer Gallengang - Zellschicksal Differenzierung der Leber - Vorläufern Antriebs (3B). Verwendung von Einzelzellanalyse quantifiziert wir die Antwort auf die Notch - Liganden für die ECM - Proteine Kollagen I, Kollagen III, Kollagen IV, Fibronectin und Laminin (3C), Feststellung , dass die Antwort von Lebervorläuferzellen an den Liganden auch auf die hängt ECM Kontext. Zuletzt verwendeten wir shRNA Knockdown Leber Vorläufern ohne den Liganden DLL1 und JAG1 zu erzeugen. Die Reaktion auf die aufgereiht Notch-Liganden variiert in Abhängigkeit von der prEsence von beiden Liganden zu bestätigen, dass die Reaktionsfähigkeit der Zell extrinsischen Liganden ist auch eine Funktion des Zell intrinsischen Liganden - Expression (Abbildung 3D). Ferner beobachteten wir eine deutliche Subpopulation von doppelt positiven (ALB + / OPN +) Zellen im DLL1 Knockdown (3D). Zusammen zeigen diese repräsentative Ergebnisse: (1) die kombinatorischen Fähigkeiten des Array-Format, wie es durch die Paarung von mehreren aufgereiht ECM Proteine und Notch-Liganden mit Zuschlags einzelner Liganden veranschaulicht; (2) die Funktionalität nicht nur angeordnet Proteine ECM, sondern auch Zell-Zell-Liganden mittels Protein A / G-vermittelte Konjugation angeordnet sind; und (3) die Umsetzung unserer Single-Cell-Analyse und seine Fähigkeit, einzigartige Subpopulationen zu erkennen.
Wir beobachteten auch , dass die Differenzierung der Leber - Vorläufern ist sowohl abhängig von der Substrat Steifigkeit und der ECM - Zusammensetzung (4A Ong>), ist , dass Kollagen IV speziell der Suche nach nur der Differenzierung unterstützend auf beiden weichen und steifen Substraten während Fibronektin Differenzierung auf steife Substrate (4B) unterstützt. Repräsentative Wärme Karten von TFM Messungen vorgeschlagen , dass nachhaltige Zugspannung bei niedrigen Substratsteifigkeit auf Kollagen IV Differenzierung in den Gallengang - Zellen (4C) gefördert, die Feststellung bestätigt durch das durchschnittliche Effektivwert-Quadrat - Werte (4D). Zusammen zeigen diese repräsentative Ergebnisse: (1) die erfolgreiche Integration von TFM mit Zellmikroarrays auf Substraten mit einer abstimmbaren Steifigkeit sowohl die Zell-Phänotyp und die Traktion Stress zu bewerten; (2) die Koordination der Lebervorläuferzellschicksal sowohl mit der Matrix-Zusammensetzung und dem Substrat Steifigkeit; und (3) die Umsetzung unserer TFM Analyse und typische Traktionsspannungsprofile in Zellmikroarrays.
e 1 "src =" / files / ftp_upload / 55362 / 55362fig1.jpg "/>
Abbildung 1: Schematische Übersicht der ersten drei Versuchsteilen angezeigt. In Abschnitt 1 werden Glassubstrate gereinigt und silanisiert die Herstellung von Polyacrylamid-Hydrogele zu erleichtern. In Abschnitt 2 werden die Biomolekül Kombinationen von Interesse in einer Quelle Mikrotiterplatte mit 384 gut vorbereitet. Ein Roboter-Arrayers wird dann mit sauberen Nadeln, die Quelle Mikrotiterplatte und die Polyacrylamid-Hydrogele und initialisiert, Herstellung Arrays auf den Hydrogelen geladen. In Abschnitt 3 werden die Zellen auf die Array-Domänen ausgesät und haften gelassen, wonach das Kulturprotokoll von Interesse durchgeführt wird. Am Endpunkt werden die Zellen entweder für Immunzytochemie / Immunofluoreszenz analysiert feste oder TFM verwenden. Maßstabsbalken sind 75 & mgr; m. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
innerhalb-page = "1"> 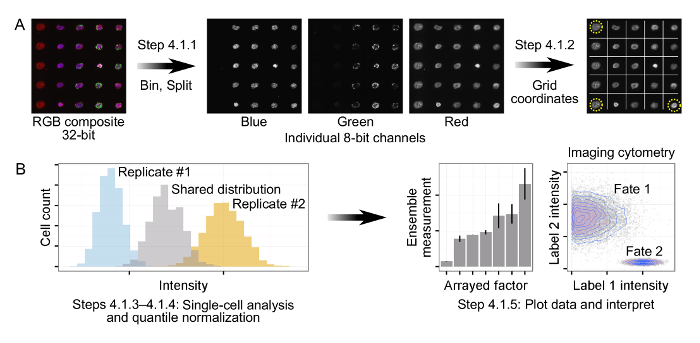
Abbildung 2: Verarbeitung und Analyse von Immunfluoreszenz - Daten aus Arrays. (A) Fliesen-, Composite - 32-Bit - RGB - Bilder werden zuerst binned und dann aufgeteilt in einzelne 8-Bit - Kanäle. Verwendung einer Kombination von angeordneten fluoreszierenden Marker und Zellinseln, drei Ecken des Arrays identifiziert für die automatische Ausrichtung und Rasterung der Arrays zu ermöglichen. (B) Einzelzellendaten für jeden Kanal der eingegebenen Arrays erzeugt. Um für experimentelle Drift zu berücksichtigen, wird Quantils Normalisierung durch biologische Replikation angewendet, um eine Shared Verteilung über alle Replikate zu erzeugen. Quantile normalisierten Daten werden anschließend über die Berechnung der Ensemblemessungen (zB Zellen / Insel, mittlere Intensität, Prozent Zellen positiv für ein Label) oder direkte Analyse von Einzelzellverteilungen aufgetragen und interpretiert.m / files / ftp_upload / 55362 / 55362fig2large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
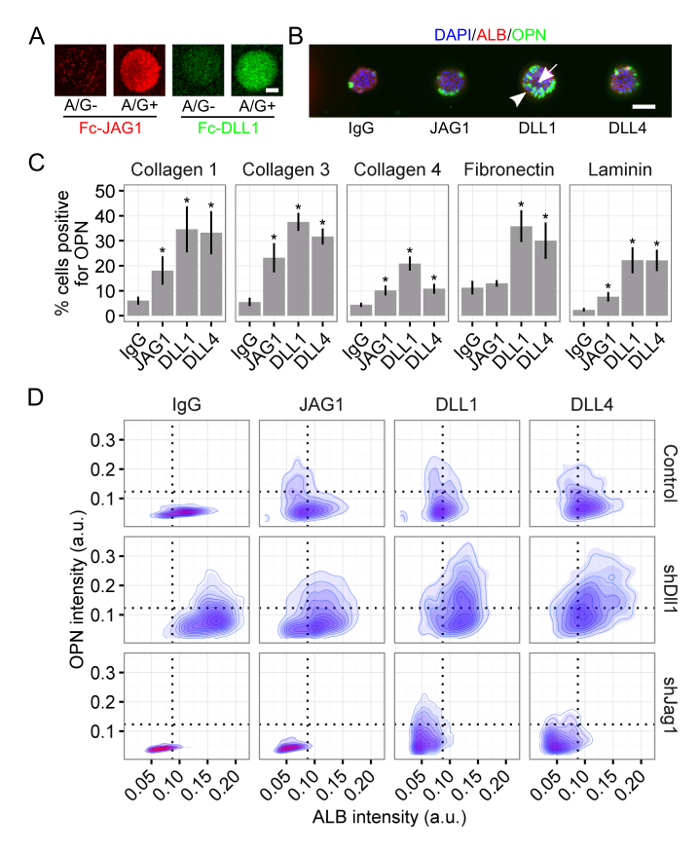
Abbildung 3: Notch - Liganden Präsentation Vermittelt Leber Progenitor Differenzierung. (A) Fc-rekombinante Notch - Liganden Jagged-1 (JAG1) und Delta-like 1 (DLL1) zeigten verbesserte Retention und Clustering , wenn sie mit Protein A / G angeordnet. Maßstabsbalken ist 50 & mgr; m. (B) Leber Vorläufern differenziert in den Gallengang - Zellen bei Vorlage mit Notch - Liganden. 4 ', 6-Diamidino-2-phenylindole (DAPI) ist ein Kern Etikett, Albumin (ALB) ein Leberzellmarker ist, und Osteopontin (OPN) ist eine Gallengang-Zellmarker. Maßstabsbalken ist 150 & mgr; m. (C) Quantifizierung der Prozentsatz der Zellen , die positiv für OPN für den Notch - Liganden JAG1, DLL1 und Delta-like 4 (DLL4) auf die ECM - Proteine Kollagen I, collagen III, Kollagen IV, Fibronectin und Laminin. Student-t -Tests wurden für jede aufgereiht Notch - Liganden gegen Kontroll - IgG durchgeführt innerhalb jedes ECM - Proteins mit P-Werte angegeben für P <0,05 (*). (D) Imaging - Zytometrie von ALB und OPN für Zellen auf Kollagen III mit den Notch - Liganden präsentiert JAG1, DLL1 und DLL4. Leber - Vorläufern ohne die Notch - Liganden DLL1 und JAG1 (dh shDll1 und shJag1) wurden shRNA Knockdown generiert. Daten in (C) dargestellt als Mittelwert ± SEM Diese Zahl wurde von Kaylan et al modifiziert. 34. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
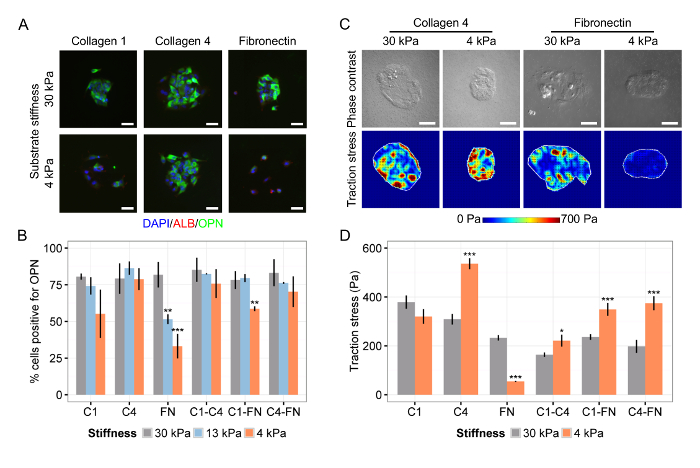
Abbildung 4: Matrixzusammensetzung und Substrat Stiffness Coordinate Leber Progenitor Differenzierung. (A) Differenzierung Liver Vorläuferzellen zu Gallenkanal ist abhängig von sowohl ECM - Zusammensetzung und dem Substrat Steifigkeit. DAPI ist ein Kern Etikett ist ALB eine Leberzellmarker und OPN ist ein Kanalzellmarker Galle. (B) Quantifizierung der Prozentsatz der Zellen positiv für OPN auf Substraten des Elastizitätsmoduls 30 kPa, 13 kPa, und 4 kPa für Kollagen I (C1), Kollagen IV (C4), Fibronectin (FN), und alle Zwei-Wege - Kombinationen diese ECM-Proteine. (C) Zell Zug beansprucht wird sowohl Substrat Steifigkeit und ECM - Zusammensetzung abhängig. (D) Die Quantifizierung der root-mean-square Werte der Zugspannung auf Substraten des Elastizitätsmoduls 30 kPa und 4 kPa für Kollagen I (C1), Kollagen IV (C4), Fibronektin (FN) und alle Zwei-Wege - Kombinationen derjenigen ECM-Proteine. In (B) und (D) wurden die Daten als Mittelwert ± SEM und Student t -Tests präsentiertfür jede ECM Kombination mit P-Werten gegenüber 30 kPa wurden für P <0,05 (*), P <0,01 (**) und P <0,001 (***) angegeben durchgeführt. Maßstabsbalken sind 50 & mgr; m. Diese Zahl wurde von Kourouklis et al modifiziert. 35. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Abschnitt | Problem | Mögliche Ursachen | Lösung |
| 1. Herstellung von Polyacrylamid-Substrat. | Abdeckglas nicht aus Hydrogel entfernt werden. | Overpolymerization. | Reduzieren Polymerisationszeit auf <10 Minuten (4 W / m 2). Überprüfen Sie, dass UV-crosslinker Ausgang ist im erwarteten Bereich. |
| Schlechte Polyacrylamid-Hydrogel-Polymerisation. | Underpolymerization. | Erhöhen Polymerisationszeit auf> 10 Minuten (4 W / m 2). Überprüfen Sie, dass UV-Vernetzer Ausgang innerhalb des erwarteten Bereichs liegt. | |
| Polyacrylamide Hydrogele werden nach Entfernung der beschädigten Abdeckglas. | Soft-Polyacrylamid-Hydrogele sind leicht zu beschädigen. | Wir beobachten Abnahme Hydrogel Fertigungsausbeute (~ 50%) für den weichsten (dh 4 kPa) Hydrogele im Besonderen. Griff Hydrogele sanft und Startnummern erhöhen, um die gewünschte Ausbeute zu erreichen. | |
| 2. Herstellung von Arrays. | Schlechte oder inkonsistent Stelle Morphologie. | Uneinheitliche Luftbefeuchter Funktion. | Überprüfen Sie, dass Luftbefeuchter und Rheometer eine funktionelle während jeder Auflage und 65% relativer Feuchtigkeit halten. |
| Pins stecken in Druckkopf oder Clogged. | Reinigen Sie den Druckkopf kostenlos Stift Bewegung zu ermöglichen. Saubere Stifte gründlich vor oder nach jedem Drucklauf Aggregate von Stiftkanälen zu entfernen. | ||
| 3. Zellkultur und Execution-Assay. | Zellablösung oder Tod auf Arrays nach der ersten Anlage. | Nachsaat und übermäßige Vermehrung. | Reduzieren anfängliche Aussaatdichte und Zeit. Verwenden Sie "Wartung" oder "Differenzierung" Medien während Array Kultur Zellproliferation zu reduzieren. |
| Freisetzung toxischer Acrylamidmonomer aus Hydrogel. | Einweichen Hydrogele in dH 2 O für mindestens 3 d für die Diffusion / Freisetzung von Acrylamidmonomer zu ermöglichen und Zelltoxizität reduzieren. | ||
| Die Zellen nicht legen zu Arrays. | Underseeding. | Erhöhen anfängliche Aussaatdichte und Zeit. Verwenden Sie eine stärker adhärenten Zelltyp. | |
| Schlechte Ablagerung vonMatrix oder Biomolekül Zustand. | Saubere Stifte und Aggregate, Druckparameter bestätigen und zu bewerten Spotten von Fluoreszenzmarkern, zB Rhodamin-konjugierten Dextran. | ||
| Spezifität der Zell-Matrix-Wechselwirkungen. | Verschiedene Zelltypen haften spezifisch an einige, aber nicht andere ECM-Proteine. Testen Sie mehrere verschiedene ECM-Proteine mit Ihren Zellen. | ||
| Suboptimale Array Lagerung nach der Herstellung. | Wir empfehlen über Nacht bei 65% relativer Luftfeuchtigkeit und Raumtemperatur hergestellten Arrays speichern, teilweise Phasenänderungen zu vermeiden, während des Einfrierens. Zelladhäsion ist empfindlich gegenüber sowohl Feuchtigkeit, Temperatur und Lagerzeit; sicherstellen, dass diese Parameter für Ihre Experimente konsistent / optimiert sind. | ||
| Detachment von Hydrogel aus Glassubstrat während der Zellkultur. | Schlechte Rutsche Reinigung und Silanisierung. | Ersetzen Arbeitslösungen für Dia-Reinigung undSilanisierung. | |
| Overdehydrated Hydrogel. | Lassen Sie keine Hydrogele auf einer heißen Platte länger als 15-30 Minuten auszutrocknen. | ||
| 4. Analyse der Daten. | Hohe Variabilität zwischen Replikat Flecken und Dias. | Variability in der Arrayherstellung. | Überprüfen Sie, ob Stifte und Druckkopf sauber sind. Bestätigen Luftbefeuchter Funktion. Visualisieren und zu quantifizieren Stelle und Array Qualität unter Verwendung von fluoreszierenden Markern. Shop-Arrays, wie oben empfohlen. |
Tabelle 1: Fehlerbehebung.
Diskussion
In unseren Experimenten haben wir, dass die häufigste Defekt gefunden auf die Qualität der hergestellten Arrays verbunden sind und schlecht Reaktion im biologischen System von Interesse charakterisiert. Wir verweisen auf die Tabelle 1 für die gemeinsame Ausfallarten in Zellmikroarray - Experimenten und der damit verbundenen Schritte zur Fehlerbehebung. In Bezug auf die Qualität von Arrays insbesondere empfehlen wir folgendes. Bestätigen Sie die technische Qualität und Robustheit der Gruppierungs Programme, Parameter und Puffer fluoreszenzmarkierte Moleküle wie Rhodamin-konjugierten Dextran verwendet wird. Gründlich reinigen Stifte entweder vor oder nach dem Gruppierungs pro Anweisungen des Herstellers und weitere visuell zu überprüfen, dass die Stiftkanäle frei sind von Schutt mit einem Lichtmikroskop. Bestätigen Sie gruppierten Biomolekül Retention unter Verwendung der allgemeinen Protein Flecken oder Immunmarkierung. Man beachte , dass Biomoleküle mit einem Molekulargewicht unter 70 kDa sind, häufig nicht 23 in dem Hydrogel beibehalten sup> 31. Validieren gruppierten Biomolekül Zell-Funktionalität mehrere Zelltypen verwendet. Beachten Sie, dass nur anhaftenden Zellen mit Arrays kompatibel sind; Zusätzlich Haftung auf Arrays hängt sowohl zellspezifischen Eigenschaften (zB Integrin - Expressionsprofil) und den ausgewählten ECM Proteine.
Aufgrund des beschränkten Platz, haben wir nicht eine umfassende Behandlung von Array - Design, Layout und Fertigung hier und verweisen den Leser auf vorherigen 23 vorgesehen funktioniert, 25. Wir verwenden in der Regel 100 Spot Subarrays (150 & mgr; m Fleckdurchmesser, 450 & mgr; m von Mitte zu Mitte Abstand) , bestehend aus 10-20 einzigartigen Biomolekül Bedingungen (dh 5-10 Punkte / Zustand). Die Anzahl der Sub-Arrays in einem Array in Abhängigkeit von der Anzahl der Biomolekül Bedingungen von Interesse abhängig, die bequem bis zu einer 25 × 75 mm Objektträger bis 1.280 skaliert werden kann (~ 6.400 Spots in 64 Subarrays)xref "> 25, 31 Die Parameter weiter oben in Abhängigkeit von der Mustergröße von Interesse variieren;. Stifte in der Lage , Muster von 75 zu erzeugen - 450 & mgr; m sind leicht verfügbar.
Array-Experimente werden am besten durch die Validierung von High-Scoring angeordnet Bedingungen von Interesse unter Verwendung anderer Kulturformate, Assay Ablesungen ergänzt und biologische Modellsysteme. Insbesondere empfehlen wir weitere Effekte von ausgewählten Array - Bedingungen Validierung unter Verwendung von Massenkulturen in Verbindung mit molekularbiologischen Standardtechniken (zB qRT-PCR, Immunoblot) oder Standard - TFM. Genetische Manipulation (zB Zuschlags oder Überexpression) des Faktors von Interesse in einem geeigneten biologischen Modellsystem kann auch dazu dienen , Effekte in Arrays beobachtet , um zu bestätigen. In - vivo - Tiermodelle repräsentieren ein weiteres Mittel zur Validierung und wurden vor kurzem verwendet, zum Beispiel die zentrale Rolle von Galectin-3 und Galectin-8 in bestätigenmetastatischen Nische der Lungenkrebs, wie zunächst über Zellmikroarray identifiziert 31, 49.
Eine Anzahl von anderen Verfahren verwendet wurden Mikroumgebungs Regulation von Zellfunktionen zu untersuchen, einschließlich einer Vielzahl von zweidimensionalen mikrofabrizierten Systeme 18, 50, 51, 52, 53, 54, 55 und dreidimensionale engineered Biomaterial Systeme 56, 57, 58 , 59, 60, 61. Im Vergleich mit anderen Verfahren, beschrieben die besonderen Vorteile der Zelle Microarray-Plattform hier bestehen aus: (1) einem Durchsatz von bis zuHunderte oder Tausende von unterschiedlichen Kombinationen von Faktoren, so dass Analyse Wechselwirkungen; (2) zugänglich, automatisierte Bildgebung und Analyse; (3) Integration der beiden biochemischen und biophysikalischen Ablesungen mit kontrollierter Darstellung von angeordneten Faktoren; (4) die Fähigkeit Substratmaterialeigenschaften zu variieren; und (5) High-Content-Single-Cell-Analyse von Zellschicksal und Funktion.
Zusammenfassend ermöglicht die Kombination von Zell-Mikroarrays mit TFM auf Substraten von abstimmbaren Substrat Steifigkeit gründliche Charakterisierung der beiden biochemischen und biophysikalischen Signale. Wie hier dargestellt, ist diese Plattform generalizable und können leicht an eine Vielzahl von adhärenten Zelltypen und Gewebe Kontexten zu einem verbesserten Verständnis der kombinatorischen Mikroumgebungs Regulation der Zelldifferenzierung und Mechanotransduktion angewendet werden.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
Wir erkennen an Austin Cyphersmith und mayandi Sivaguru (Carl R. Woese Institute for Genomic Biologie, Universität von Illinois in Urbana-Champaign) für die Unterstützung bei der Mikroskopie und für die großzügige Aufnahme von Bildschirm und Video-Capture in der Mikroskopie Kern.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | Pall Corporation | 4433 | Match with appropriately-sized Luer lock plastic syringes. |
| 100 × penicillin–streptomycin solution | Fisher Scientific | SV30010 | |
| 22 × 60 mm coverglasses | Electron Microscopy Sciences | 63765 | |
| 3-(trimethoxysilyl)propyl methacrylate (3-TPM) | Sigma-Aldrich | 440159 | Store under inert gas per manufacturer's instructions. Exposure of 3-TPM to air could compromise silanization of glass substrates. CAUTION: 3-TPM is a combustible liquid. Keep away from heat, sparks, open flames, and hot surfaces and use only in a chemical fume hood. |
| 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate hydrate (CHAPS) | Sigma-Aldrich | C3023 | |
| 35 mm glass-bottom Petri dishes | Cell E&G | GBD00002-200 | 13 mm well consisting of #1.5 coverglass. Enables TFM and live-cell imaging. |
| 384-well polypropylene V-bottom microplate, non-sterile | USA Scientific | 1823-8400 | |
| 6-well polystyrene microplates | Fisher Scientific | 08-772-1B | 35 mm glass-bottom Petri dishes fit into wells of microplate, easing array fabrication. |
| Acetone | Sigma-Aldrich | 179973 | |
| Acrylamide | Sigma-Aldrich | A3553 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Collagen I, rat tail | EMD Millipore | 08-115MI | |
| Collagen III, human | EMD Millipore | CC054 | |
| Collagen IV, human | EMD Millipore | CC076 | |
| Crosslinker, 365 nm | UVP | CL-1000 | |
| Dextran, rhodamine B-conjugated, 70 kDa | ThermoFisher Scientific | D1841 | Used as a marker for array location. |
| Dimethyl sulfoxide | Fisher Scientific | BP231 | |
| Dulbecco's phosphate-buffered saline (PBS) | Fisher Scientific (HyClone) | SH3001302 | |
| Ethyl alcohol | Decon Labs | 2701 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | ED | |
| Fc-recombinant DLL1, mouse | R&D Systems | 5026-DL-050 | |
| Fc-recombinant DLL4, mouse | AdipoGen | AG-40A-0145-C050 | |
| Fc-recombinant JAG1, rat | R&D Systems | 599-JG-100 | |
| Fibronectin, human | Sigma-Aldrich | F2006 | |
| Fluorescent microscope, inverted | Zeiss | Axiovert 200M | Ensure microscope is equipped with a robotic stage for both automated fluorescent imaging and TFM. Environmental control (i.e., 37 °C and 5% CO2) is highly advisable for TFM. |
| Fluoromount G with DAPI | SouthernBiotech | 0100-20 | |
| Glacial acetic acid | Sigma-Aldrich | 695092 | CAUTION: Acetic acid is flammable and corrosive. Wear protective gloves, clothing, and eye protection. |
| Glycerol | Sigma-Aldrich | M6145 | |
| Irgacure 2959 | BASF Corporation | 55047962 | |
| Laminin, mouse | EMD Millipore | CC095 | |
| Methanol | Sigma-Aldrich | 179957 | |
| Microarray scanner | GenePix | 4000B | Fluorophores must be Cy3- or Cy5-compatible. |
| Microarrayer | Digilab | OmniGrid Micro | Other microarrayers of similar or greater capability can readily be substituted. |
| Microscope slides, 25 × 75 mm | Sigma-Aldrich | CLS294775X25 | ~0.9 – 1.1 mm thickness. |
| N,N′-Methylenebisacrylamide (bisacrylamide) | Sigma-Aldrich | M7279 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Paraformaldehyde (PFA), 16% v/v | Electron Microscopy Sciences | RT15710 | Prepare PFA fresh (do not store) for optimal fixation. CAUTION: Exposure to PFA can result in acute toxicity and can also irritate or corrode skin on contact. Wear protective gloves, clothing, and eye protection and use only in a chemical fume hood. |
| Protein A/G, recombinant | ThermoFisher Scientific | 21186 | |
| Pyrex drying tray, 2,000 mL | Fisher Scientific | 15-242B | |
| Rectangular 4-chambered culture dish | Fisher Scientific (Nunc) | 12-565-495 | For cell culture on arrayed microscope slides. |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | 415413 | CAUTION: NaOH is highly caustic and can cause severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Stealth pin for arraying | ArrayIt | SMP3 | Clean pins after each array run using the instructions of the manufacturer. Produces 150 micron domains; purchase other pin sizes (75–450 microns) as suited to your particular application. |
| Triton X-100 | Sigma-Aldrich | X100 |
Referenzen
- Joyce, J. A., Pollard, J. W. Microenvironmental regulation of metastasis. Nat Rev Cancer. 9 (4), 239-252 (2009).
- Hsu, Y. C., Fuchs, E. A family business: stem cell progeny join the niche to regulate homeostasis. Nat Rev Mol Cell Biol. 13 (2), 103-114 (2012).
- Whiteside, T. L. The tumor microenvironment and its role in promoting tumor growth. Oncogene. 27 (45), 5904-5912 (2008).
- Jones, D. L., Wagers, A. J. No place like home: anatomy and function of the stem cell niche. Nat Rev Mol Cell Biol. 9 (1), 11-21 (2008).
- Discher, D. E., Mooney, D. J., Zandstra, P. W. Growth factors, matrices, and forces combine and control stem cells. Science. 324 (5935), 1673-1677 (2009).
- Trappmann, B., et al. Extracellular-matrix tethering regulates stem-cell fate. Nat Mater. 11 (7), 642-649 (2012).
- Ivanovska, I. L., Shin, J. W., Swift, J., Discher, D. E. Stem cell mechanobiology: diverse lessons from bone marrow. Trends Cell Biol. 25 (9), 523-532 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Chaudhuri, O., et al. Hydrogels with tunable stress relaxation regulate stem cell fate and activity. Nat Mater. 15 (3), 326-334 (2016).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev Cell. 6 (4), 483-495 (2004).
- Legate, K. R., Wickstrom, S. A., Fassler, R. Genetic and cell biological analysis of integrin outside-in signaling. Genes Dev. 23 (4), 397-418 (2009).
- Kessenbrock, K., Plaks, V., Werb, Z. Matrix metalloproteinases: regulators of the tumor microenvironment. Cell. 141 (1), 52-67 (2010).
- Underhill, G. H. Stem cell bioengineering at the interface of systems-based models and high-throughput platforms. Wiley Interdiscip Rev Syst Biol Med. 4 (6), 525-545 (2012).
- Underhill, G. H., Galie, P., Chen, C. S., Bhatia, S. N. Bioengineering methods for analysis of cells in vitro. Annu Rev Cell Dev Biol. 28, 385-410 (2012).
- Zorlutuna, P., et al. Microfabricated biomaterials for engineering 3D tissues. Adv Mater. 24 (14), 1782-1804 (2012).
- Ruiz, S. A., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Guillotin, B., Guillemot, F. Cell patterning technologies for organotypic tissue fabrication. Trends Biotechnol. 29 (4), 183-190 (2011).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. J. Cell Sci. 123 (Pt 24), 4201-4213 (2010).
- Ranga, A., Lutolf, M. P. High-throughput approaches for the analysis of extrinsic regulators of stem cell fate. Curr. Opin. Cell Biol. 24 (2), 236-244 (2012).
- Kobel, S., Lutolf, M. High-throughput methods to define complex stem cell niches. Biotechniques. 48 (4), ix-xxii (2010).
- Fernandes, T. G., Diogo, M. M., Clark, D. S., Dordick, J. S., Cabral, J. M. S. High-throughput cellular microarray platforms: applications in drug discovery, toxicology and stem cell research. Trends Biotechnol. 27 (6), 342-349 (2009).
- Montanez-Sauri, S. I., Beebe, D. J., Sung, K. E. Microscale screening systems for 3D cellular microenvironments: platforms, advances, and challenges. Cell Mol Life Sci. 72 (2), 237-249 (2015).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat Methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Flaim, C. J., Bhatia, S. N., Parekkadan, B., Yarmush, M. . Methods in Bioengineering: Stem Cell Bioengineering Artech House Methods in Bioengineering. , 63-73 (2009).
- Brafman, D. A., Chien, S., Willert, K. Arrayed cellular microenvironments for identifying culture and differentiation conditions for stem, primary and rare cell populations. Nat Protoc. 7 (4), 703-717 (2012).
- Brafman, D. A., et al. Investigating the role of the extracellular environment in modulating hepatic stellate cell biology with arrayed combinatorial microenvironments. Integr Biol. 1 (8-9), 513-524 (2009).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol Syst Biol. 2, 37 (2006).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integr Biol. 1 (1), 70-79 (2009).
- Anderson, D. G., Levenberg, S., Langer, R. Nanoliter-scale synthesis of arrayed biomaterials and application to human embryonic stem cells. Nat Biotechnol. 22 (7), 863-866 (2004).
- Brafman, D. A., Shah, K. D., Fellner, T., Chien, S., Willert, K. Defining long-term maintenance conditions of human embryonic stem cells with arrayed cellular microenvironment technology. Stem Cells Dev. 18 (8), 1141-1154 (2009).
- Reticker-Flynn, N. E., et al. A combinatorial extracellular matrix platform identifies cell-extracellular matrix interactions that correlate with metastasis. Nat Commun. 3, 1122 (2012).
- Wood, K. C., et al. MicroSCALE screening reveals genetic modifiers of therapeutic response in melanoma. Sci Signal. 5 (224), rs4 (2012).
- Braga Malta, D. F., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kaylan, K. B., Ermilova, V., Yada, R. C., Underhill, G. H. Combinatorial microenvironmental regulation of liver progenitor differentiation by Notch ligands, TGFbeta, and extracellular matrix. Sci Rep. 6 (23490), 23490 (2016).
- Kourouklis, A. P., Kaylan, K. B., Underhill, G. H. Substrate stiffness and matrix composition coordinately control the differentiation of liver progenitor cells. Biomaterials. 99, 82-94 (2016).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integr. Biol. , (2016).
- Mann, C., Leckband, D. Measuring Traction Forces in Long-Term Cell Cultures. Cellular and Molecular Bioengineering. 3 (1), 40-49 (2010).
- Heisenberg, C. P., Bellaiche, Y. Forces in tissue morphogenesis and patterning. Cell. 153 (5), 948-962 (2013).
- Schwarz, U. S., Soine, J. R. Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta. 1853 (11 Pt B), 3095-3104 (2015).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology-Cell Physiology. 282 (3), C595-C605 (2002).
- Wen, J. H., et al. Interplay of matrix stiffness and protein tethering in stem cell differentiation. Nat Mater. 13 (10), 979-987 (2014).
- Knoll, S. G., Ali, M. Y., Saif, M. T. A novel method for localizing reporter fluorescent beads near the cell culture surface for traction force microscopy. J Vis Exp. (91), e51873 (2014).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. Am J Physiol Cell Physiol. 282 (3), C606-C616 (2002).
- Krishnan, R., et al. Substrate stiffening promotes endothelial monolayer disruption through enhanced physical forces. Am J Physiol Cell Physiol. 300 (1), C146-C154 (2011).
- Reticker-Flynn, N. E., Bhatia, S. N. Aberrant glycosylation promotes lung cancer metastasis through adhesion to galectins in the metastatic niche. Cancer Discov. 5 (2), 168-181 (2015).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Kilian, K. A., Bugarija, B., Lahn, B. T., Mrksich, M. Geometric cues for directing the differentiation of mesenchymal stem cells. Proc Natl Acad Sci USA. 107 (11), 4872 (2010).
- Nelson, C. M., Chen, C. S. Cell-cell signaling by direct contact increases cell proliferation via a PI3K-dependent signal. FEBS Lett. 514 (2-3), 238-242 (2002).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proc. Natl. Acad. Sci. U. S. A. 104 (14), 5722-5726 (2007).
- Lutolf, M. P., Blau, H. M. Artificial stem cell niches. Adv Mater. 21 (32-33), 3255-3268 (2009).
- Gobaa, S., et al. Artificial niche microarrays for probing single stem cell fate in high throughput. Nat. Methods. 8 (11), 949-955 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Nelson, C. M., VanDuijn, M. M., Inman, J. L., Fletcher, D. A., Bissell, M. J. Tissue geometry determines sites of mammary branching morphogenesis in organotypic cultures. Sci. STKE. 314 (5797), 298 (2006).
- Liu Tsang, V., et al. Fabrication of 3D hepatic tissues by additive photopatterning of cellular hydrogels. FASEB J. 21 (3), 790-801 (2007).
- Albrecht, D. R., Underhill, G. H., Mendelson, A., Bhatia, S. N. Multiphase electropatterning of cells and biomaterials. Lab. Chip. 7 (6), 702-709 (2007).
- Chan, V., Zorlutuna, P., Jeong, J. H., Kong, H., Bashir, R. Three-dimensional photopatterning of hydrogels using stereolithography for long-term cell encapsulation. Lab. Chip. 10 (16), 2062-2070 (2010).
- Boghaert, E., et al. Host epithelial geometry regulates breast cancer cell invasiveness. Proc. Natl. Acad. Sci. U. S. A. 109 (48), 19632-19637 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten