Method Article
Bewertung der Kontamination der DNA in RNA-Proben anhand der ribosomalen DNA
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll zur Rückverfolgung genomische DNA (gDNA) Kontamination in RNA-Proben. Die vorgestellte Methode nutzt spezifische Primer für die internen übertragenen Distanzscheibe Region (ITS) der ribosomalen DNA (rDNA) Gene. Die Methode eignet sich für zuverlässige und empfindlichen Nachweis von DNA-Kontaminationen in den meisten Eukaryoten und Prokaryoten.
Zusammenfassung
Eine Methode, die weitgehend für die Quantifizierung von Veränderungen der Genexpression und Transkript Häufigkeiten verwendet ist Reverse Transkription quantitative Echtzeit-PCR (RT-qPCR). Freuen Sie sich auf präzise, sensible, zuverlässige und reproduzierbare Ergebnisse. Mehrere Faktoren können die Sensitivität und Spezifität der RT-qPCR beeinflussen. Verbleibender genomischer DNA (gDNA) RNA-Proben verunreinigt ist einer von ihnen. In gen Expressionsanalyse unspezifische Verstärkung durch gDNA Verschmutzung wird die Fülle der Abschrift Ebenen zu überschätzen und die RT-qPCR-Ergebnisse beeinflussen kann. In der Regel gDNA röntgenologisch qRT-PCR Primer mit Paare, Glühen, intergenetischer Regionen oder ein Intron des Gens von Interesse. Leider sind Intron/Exon Anmerkungen noch nicht für alle Gene von Wirbeltier, Bakterien, protiste, Pilze, Pflanzen und Wirbellosen riffbildende Arten bekannt.
Hier stellen wir Ihnen ein Protokoll für Erkennung von gDNA Kontamination in RNA Proben mithilfe der ribosomalen DNA (rDNA)-basiert Zündkapseln. Die Methode basiert auf der einzigartigen Eigenschaften von rDNA: ihre multigene Natur, hoch konservierte Sequenzen und hoher Frequenz im Genom. Auch als Fallstudie, ein eindeutigen Satz von Primer wurden entwickelt basierend auf der konservierten Region der ribosomalen DNA (rDNA) in der Familie Poaceae. Die Universalität dieser Grundierung Paare wurde von Schmelze Kurve Analyse und Agarose-Gel-Elektrophorese getestet. Obwohl unsere Methode erklärt wie rDNA basiert Zündkapseln für gDNA Kontamination Assay in der Familie der Poaceae angewendet werden können, könnte es leicht auf andere intramolekulare und Eukaryote Arten verwendet werden
Einleitung
Exploring transcriptional Regelung interessanter Gen-Sets oder Signalisierung Netzwerke ist wichtig zu verstehen, die komplexen molekularen Mechanismen in biologische Gefahrenlagen1. QPCR Analyse ist derzeit das am häufigsten verwendete Ansatz für Genexpressionsstudien, die Ziel können entweder (das Genom) DNA oder RNA (das Transkriptom), die Methylome und Transkriptom-Analyse bzw. erlauben. Reversen Transkription (RT) gefolgt von qPCR ist weit verbreitet für die Transkriptom-Analyse, die gen-Ausdruck-Ebenen in verschiedenen Bereichen der biologischen Forschung2messen. Im Vergleich zu anderen Methoden wie die traditionellen nördlichen Hybridisierung, Gewebe spezifische Erkennung mittels in Situ Hybridisierung, Ribonuklease Schutz Assays (RPA) und Semi-RT-PCR, die Genauigkeit, Komfort, Geschwindigkeit und Dynamikbereich von qPCR basierende Assays sind höchst bemerkenswerte3,4. Es gibt mehrere wichtige Faktoren, die für eine zuverlässige Quantifizierung der Boten-RNA (mRNA), darunter die Qualität und Quantität der RNA Ausgangsmaterial berücksichtigt werden müssen. Darüber hinaus sind unspezifische Verstärkung, die Effizienz der RT-qPCR und PCR-Effizienz zu5,6beachten.
Das Vorhandensein von gDNA ist eine inhärente Problem während der RNA-Extraktion zurückzuführen, zum Teil auf ähnlichen physikalischen und chemischen Eigenschaften von DNA und RNA7. Wegen des Sequenz Identität gDNA und komplementären DNA (cDNA) abgeleitet aus der mRNA-Proben kann unspezifische Verstärkung auftreten, beeinflussen die Genauigkeit der RT-qPCR Ergebnisse. Die restlichen gDNA führt zur Überschätzung der Fülle der mRNA in gen Ausdruck Analyse8Ziel.
Grundsätzlich ergibt sich unspezifische Amplikons meist aus Grundierung-Dimer-Bildung oder unspezifischen Hintergrund Verstärkung durch gDNA, beide von denen beurteilt werden können, durch den Einsatz geeigneter Kontrollproben. Solche Proben sind keine Vorlage-Kontrolle (NTC) bzw. keine Reverse-Transkriptase-Kontrolle (NRT). Da unterscheiden sich die Ebenen der gDNA Kontamination in den untersuchten Proben und die Sensibilität gegenüber gDNA unterscheidet sich stark zwischen den Genen analysiert, sind die NRT-Steuerelemente für jede Probe/Test-paar erforderlich. Obwohl dies Kosten und Arbeit in RT-qPCR profiling Studien deutlich erhöht, werden diese Steuerelemente erforderlich7,9.
Alternative Methoden, Umgang mit gDNA Kontamination umfassen die Verwendung von Primer-Paaren Glühen auf intergenetischer Gebiete oder ein Intron des Gens von Interesse10und die Verwendung von Primern, die entweder eine große Intron flankieren oder eine Exon-Exon-Junction, d.h. umfassen die Glühen Seiten fehlen in der Reifen mRNA-Sequenz1,4. Intron/Exon-Anmerkungen für alle Gene aus vielen Wirbeltier, Bakterien, protiste, Pilze, Pflanzen und Wirbellosen riffbildende Arten sind jedoch noch nicht bekannt. Darüber hinaus haben viele Eukaryonten Pseudogene Vervielfältigung Veranstaltungen abgeleitet. Darüber hinaus garantiert besser gekleideteres Design über Introns nicht nicht-Verstärkung des gDNA. Als das Chromatin Erreichbarkeit der genomischen Regionen, DNase ich variiert, es empfiehlt sich, verschiedene Primer-Paaren auf verschiedenen Chromosomen10zu entwerfen.
Die Genome von Eukaryonten können bis zu tausend Kopien der rDNA Gene Kodierung ribosomale Untereinheiten notwendig für Ribosomen Bildung umfassen. Diese rDNA-Gene sind oft in Einzel- oder tandem repeat Arrays11organisiert. Polycistronic rRNAs (Abbildung 1) einschließlich der großen Untereinheit (LSU) und der kleinen Untereinheit (SSU) sind durch die RNA-Polymerase transkribiert ich (RNA-Pol ich). Die daraus resultierende Pre-rRNAs sind durch den Wegfall der beiden internen übertragenen Distanzscheibe Regionen ITS1 und ITS2 weiterverarbeitet. Als Endprodukte, Reifen drei rRNAs, 17-18 s rRNA (SSU), 5.8S und 25-28 s rRNA (LSU) sind generierte12. rDNA Gene sind typische Vertreter einer multigene Familie mit hoch konservierte Sequenzen. Sie kommen mit einer hohen Frequenz im Genom und sind eventuell vorhandene bei mehr als einem chromosomalen Position13. Die Verarbeitung der rRNA und den Abbau der transkribierten Abstandhalter ist ein schneller Prozess in der Nukleolus. Durch die hohe Wiederholbarkeit ist das Verhältnis von genomischen Exemplarzahl und nachweisbar unverarbeitete RNA Premolecules niedriger im Vergleich zu den niedrig-Kopie Intron-Sequenzen und unspliced Vorstufen. Diese Eigenschaften machen rDNA-Gene, die für zuverlässige und hochempfindlichen Nachweis von gDNA Kontamination in den meisten Eukaryoten und Prokaryoten3gut geeignet.
Hier ist ein neuartiges Verfahren zum Nachweis von gDNA Kontamination in RNA-Proben beschrieben. Eine Reihe von universellen Primer basierend auf der konserviert rDNA Sequenz ist für gDNA Assays in verschiedenen Poaceae vorgestellt. Die Spezifität und die Universalität der vorgeschlagenen Primer wurden von Schmelze Kurvenanalyse mit DNA als Vorlage getestet. Unser Protokoll gilt nicht nur für Poaceae, sondern könnte auch leicht auf andere eukaryotischen und prokaryotischen Arten angepasst werden.
Protokoll
Hinweis: Jedes Gewebe kann verwendet werden.
(1) Nukleinsäure-Extraktion
- 100 mg von Gewebeproben in einem 2,0 mL-Tube, zwei 5 mm Edelstahl Perlen und homogenisieren des Gewebes bei 25-30 Hz für 30 s (Homogenisierung Dauer und Häufigkeit je nach Gewebetyp) für RNA und DNA.
- Gesamt-RNS gemäß den Anweisungen des Herstellers zu isolieren.
- Gesamt-DNA nach den Anweisungen des Herstellers zu isolieren.
- Kontrollieren Sie die Reinheit und Menge der RNA-Proben durch Messung der Absorption bei 260 und 280 nm.
- Kontrollieren Sie die Reinheit und Menge von DNA-Proben durch Messung der Absorption bei 260 und 280 nm.
Hinweis: Während Nukleinsäuren absorbieren Licht mit einer Wellenlänge von 260 nm (A260), die Absorption des Lichtes bei der Wellenlänge 280 (A280) kann verwendet werden, um die Menge an Proteinen zu quantifizieren und Phenole zu präsentieren, in der Probe. Daher kann das Verhältnis von A260/A280 nm verwendet werden, um die Reinheit von DNA und RNA extrahiert aus einer Probe zu bewerten. A260/280 Werte im Bereich von ≥1.8 und > 2.0 gelten im Allgemeinen als "pur" für DNA und RNA, beziehungsweise. Unteren A260/280 Werte können darauf hinweisen, dass Verunreinigungen durch Protein oder organische Chemikalien. - Testen Sie die Qualität der DNA durch Ausführen einer 0,7 % Agarose-Gelelektrophorese. Bereiten Sie das Gel und 1 x EDTA-Puffer TRIS-Bor einmünden (TBE: 89 mM Tris, Borsäure 89 mM und 2 mM EDTA) bei 100 V für 30 min. High Qualität gDNA erscheint als eine scharfe, hoch Molekulargewicht (HMW) band mit keine Abstriche im Bereich von niedermolekularen (LMW) Moleküle.
- Isolierten RNA für Menge, Reinheit und Integrität unter denaturierenden Bedingungen durch eine Guanidin Thiocyanat (GTC) Agarose-Gelelektrophorese oder durch Kapillarelektrophorese Chip, gemäß den Anweisungen des Herstellers zu überprüfen.
- Bereiten Sie das GTC-Gel durch Zugabe von 5 mM AGB zu einem standard 1 X TBE 1 % Agarose-Gel nach dem Abkühlen der Agar bis 60 ° C.
Hinweis: GTC ist giftig, also in einer Dampfhaube verzichten und geeigneten persönlichen Schutzausrüstung zu tragen. - Bereiten Sie RNA Denaturierung laden Puffer: 95 % Formamid, 10 mM EDTA pH 8.0, 0,1 % Bromophenol blau, 0,1 % Xylol Cyanole und 10 µL Interkalation Bromid.
Hinweis: Formamid und Interkalation Bromid sind giftig und sollte in einer Dampfhaube verzichtet werden. - Last 1-5 µg von Gesamt-RNS in RNA Denaturierung laden Puffer, Hitze die Mischung für 5 min bei 70 ° C, legen Sie es auf dem Eis vor dem Laden auf ein Gel und trennen sich dann RNA auf AGB Gel bei 100 V für 45 min. Last DNA oder RNA Molekulargewicht Marker serienmäßig neben die RNA-Probe.
- Färben Sie die Gele mit Interkalation Bromid und visualisieren Sie die Bands mit Image Capture-Systeme unter UV-Licht zu. In den Eukaryotes wird intakt Gesamt-RNS laufen bei denaturierenden Bedingungen mindestens zwei scharfe und klare rRNA Bands (28 s und 18 s) mit einem 2:1 Intensitätsverhältnis zeigen.
- Bereiten Sie das GTC-Gel durch Zugabe von 5 mM AGB zu einem standard 1 X TBE 1 % Agarose-Gel nach dem Abkühlen der Agar bis 60 ° C.
- Entfernen Sie Spuren von gDNA durch Behandlung mit DNase (DNase ich RNase-freie). Hinzufügen einer RNase-freies Rohr in 10 µL Gesamtvolumen: 0,1 - 1 µg Gesamt-RNS, eine Einheit der DNase I und 1 µL 10-fach Reaktion mit MgCl2Puffer. Inkubieren Sie die Mischung für 30 min bei 37 ° c Kündigen Sie die Reaktion durch Hinzufügen von 1 µL 50 mM EDTA und bei 65 ° C für 10 min Inkubation.
- Entfernen Sie Spuren von RNA aus gDNA Extrakte mit DNase frei RNase A, laut Protokoll des Herstellers. Fügen Sie 5 µL RNase 10 mg/mL, die Gesamt-DNA und Inkubation bei 37 ° C für 1 h Store RNA und DNA-Extrakte bei-80 ° C.
2. besser gekleideteres Design von rDNA Region für gDNA Assay
Hinweis: Die rDNA Full-length-Sequenz enthält zwei Regionen (ITS1 und ITS2), die in den Reifen rRNA Moleküle durch eine Reihe von Endonucleolytic Spaltung entfernt und dann abgebaut (Abbildung 1).
- Die rDNA Nukleotidsequenz von NCBI (http://www.ncbi.nlm.nih.gov) für die Arten von Interesse abrufen. Das beste Stichwort zum Durchsuchen der Datenbank ist "interne transkribierten Spacer."
- Geben Sie die Ziel Nukleotidsequenz in eine BLASTn Suche für Suche nach internen übertragenen Distanzscheibe Regionen (ITSs), SSU und LSU Regionen erhalten.
- Wählen Sie Primer, dass entweder eine ITSs-Sequenz flankieren oder zu verstärken, dass ITSs-Sequenzen, die nicht in die Reifen rRNA vorhanden sind.
- Gestaltung von Primern flankieren die ITS1 oder ITS2 Sequenzen: richten Sie die konservierten Bereiche von verschiedenen Arten von ClustalW. Entwerfen Sie spezifische Primer für die flankierenden Region nach Taxa spezifische/Cross-Arten Analyse mit AlleleID Software. Die zwei Grundierung Paare verstärkendes SSU-5.8S und 5.8S-LSU Amplifikate können entworfen werden basierend auf den flankierenden Regionen ITS1 und ITS2, beziehungsweise. Weil diese Amplifikate in der ITS-Region erstreckt sich über sind und die Länge der Amplifikate erhöht werden, für mindestens 300 bp in Amplifikate aus gDNA. Dieser Anstieg verringert die Empfindlichkeit.
- Flankierende ITS1: Wählen Sie SSU und 5.8S rRNA Sequenz. Ausgewählten Primer für Poaceae sind: SSU, SF: CGTAACAAGGTTTCCGTAGGTG, R: GGTTCACGGGATTCTGCAAT. Diese Primerpaar (SF: vorwärts und rückwärts R:) verstärkt den Teilbereich des SSU, abendfüllenden ITS1 und den Teilbereich der 5.8S rDNA.
- Flankierende ITS2: Wählen Sie 5.8S und LSU Sequenz. Ausgewählten Primer für Poaceae sind: F: ATTGCAGAATCCCGTGAACC LSU Konsensussequenz, LR: TGCTTAAAYTCAGCGGGTAGYC. Diese Primerpaar verstärkt den Teilbereich der 5.8S, der in voller Länge ITS2 und den Teilbereich der LSU (Abbildung 1).
Hinweis: Im Falle besser gekleideteres Design basierend auf ITSs flankierenden Region, hoch konservierte Bereiche von SSU, wurden 5.8S und LSU identifiziert. Die Forward- und reverse Primer 5.8S rRNA basierend auf ein konserviertes Motiv in Blütenpflanzen14entworfen wurden. Die Forward- und reverse Primer wurden entwickelt, basierend auf SSU und LSU konserviert Regionen in Poaceae, beziehungsweise. Die Divergenz der SSU und LSU Primer für jede Art wird in Tabelle 1angegeben.
- Grundierungen, die Verstärkung einer ITSs-Sequenz: In diesem Protokoll Design ITS1 Primer basierend auf Aeluropus seiner Sequenz (NCBI-USt-Nummer: 110873). Für Grundierungen, verwenden: nach vorne: GGTATGGCGTCAAGGAACACT, Reverse: ATAGCATCGCTGCAAGAGGT. Nach der Amplifikate durch die Grundierung Paare in Silicoerzeugt, die Größe sollte reichen von 60 bis 200 bp. Dies ist auch die empfohlene Größe für qPCR-Analyse.
- Gestaltung von Primern flankieren die ITS1 oder ITS2 Sequenzen: richten Sie die konservierten Bereiche von verschiedenen Arten von ClustalW. Entwerfen Sie spezifische Primer für die flankierenden Region nach Taxa spezifische/Cross-Arten Analyse mit AlleleID Software. Die zwei Grundierung Paare verstärkendes SSU-5.8S und 5.8S-LSU Amplifikate können entworfen werden basierend auf den flankierenden Regionen ITS1 und ITS2, beziehungsweise. Weil diese Amplifikate in der ITS-Region erstreckt sich über sind und die Länge der Amplifikate erhöht werden, für mindestens 300 bp in Amplifikate aus gDNA. Dieser Anstieg verringert die Empfindlichkeit.
- Wählen Sie die Primer unter Berücksichtigung dieser Empfehlungen: GC-Gehalt: 40-60 %, Grundierung Länge: 18-23, PCR Produkt Basislänge: 60-160 bp (speziell für die Grundierung), Schmelzpunkt (Tm): 60 ° C, die endgültige Tm für beide Primer unterscheiden sich nicht mehr als 5 ° C und die Prim Ers sind nicht selbst ergänzen oder partner Primer.
- Grundierung Spezifität und Kopie Schecknummer. In-Silico -Analyse der ausgewählten Primer Sequenz durch Primer-Blast-Programm (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) durchführen.
- Öffnen Sie die Primer-BLAST Vorlage Seite. Geben Sie beiden Primer-Sequenzen in die Primer-Parameter-Abschnitt des Formulars. Der Primer paar Spezifität Parameterabschnitt zu überprüfen geben Sie einen Organismus (oder Organismus Gruppenname) und wählen Sie das Genom Datenbank. Diese Einstellungen geben die Spezifität Informationen über Zielparameter Sequenz und Primer einschließlich Produktlänge, Position auf dem Chromosom, und kopieren Sie Nummer.
3. führen Sie qPCR Schritt für die Validierung der rDNA-basierte Primer mit DNA-Templates
Hinweis: Die Funktionalität der gestalteten Primer sollte überprüft werden, indem Sie durchführen qPCR gDNA als Vorlage verwenden. Um mehrere parallele Reaktionen und Pipettieren Fehler zu vermeiden, empfiehlt sich die Erstellung einer master-Mix. Bereiten Sie eine master-Mix ein Volumen entspricht der Gesamtzahl der Reaktion Mischung plus ca. 10 %.
- Bereiten Sie einen master-Mix durch mischen alle Reaktionskomponenten mit Ausnahme der DNA Schablone in einer PCR-Reaktion-Röhre. Gehobene, wie aus einer Reaktion notwendig, um den Master Mix vorzubereiten: 5 µL SYBR Green (SYBR) Mix (2 X), 0,3 µL Grundierung (jeweils 0,3 µM vorwärts- und Grundierung) zu meistern, und passen Sie das Endvolumen zu 10 µL mit RNase-freies Wasser. Verwenden Sie ca. ≤200 ng in 1 µL Vorlage gDNA für Analyse.
Hinweis: Auftauen, montieren, und halten Sie alle Reagenzien, Komponenten und Reaktionsmischungen auf Eis. - Aliquot den Master-Mix in eine optische 96-Well-Platte. Pipette 1 µL gDNA in jede Vertiefung, und bedecken Sie es mit optischen Platte Abdichten Film. Drehen und legen in Cycler.
- Die qPCR-Assay auf eine Echtzeit-Thermocycler unter folgenden Bedingungen laufen: 10 min bei 95 ° C gefolgt von 40 Zyklen von 95 ° C für 15 s und 60 ° C für 1 min. Perform Datenerfassung bei 60 ° C Glühen/Verlängerung Schritt.
- Unterziehen Sie nach der Verstärkungsprozedur alle PCR Reaktionen auf einem schmelzenden Kurvenanalyse mit kontinuierlichen Fluoreszenzmessung von 55 ° C bis 95 ° C. In der Regel sammeln Sie ein Datenpunkt pro Zyklus durch eine schrittweise Erhöhung der Temperatur um 0,5 ° C pro Zyklus.
Hinweis: Enthalten Sie mindestens 2 nicht-Template-Kontrollen (NTC) für jede Grundierung-paar-master-Mix. Führen Sie alle Tests in mindestens drei Wiederholungen. - Bestätigen Sie die Grundierung Spezifität über die Schmelze Kurvenanalyse. Kurven mit einheitlicher Schwellenwert Zyklus und subtrahierten Kurvenanpassung Methode zu analysieren.
Hinweis: Die Darstellung eines scharfen einzelne Peaks zeigt eine einheitliche individuelle Amplikons. Primer-Dimer-Produkte können als einzelne Peaks bei niedrigeren Temperaturen angezeigt. - Überprüfen Sie die Größe der einzelnen Amplifikate durch Agarose-Gelelektrophorese.
- Bereiten Sie eine 3 % Agarose-Gel durch Mischen von 3 g Agarose mit 100 mL TBE-Puffer (TBE: 89 mM Tris, Borsäure 89 mM und 2 mM EDTA).
- Mischen Sie 5-10 µL des PCR-Produkt mit DNA und 1-2 µL 6 x Puffer laden. Laden Sie PCR-Produkt neben einem DNA-Leiter auf 3 % Agarosegel. Führen Sie eine elektrophoretische Trennung in 1 x TRIS-boric EDTA-Puffer bei 100 V für 45 Minuten.
- Färben Sie die Gele mit Interkalation Bromid oder andere intercalating Agenten und visualisieren Sie die Bands mit Image Capture-Systeme unter UV-Licht zu.
Hinweis: DNA verschuppen Agenten (z.B. Interkalation Bromid) krebserzeugend sind und sollten mit Vorsicht behandelt und gesondert verzichtet. Das Erscheinungsbild einer einzigartigen scharfen Band (in Bezug auf Größe und ohne Grundierung-Dimer oder künstliche Hintergrund Verstärkung) bestätigt die Spezifität der Amplifikate.
4. gDNA Kontamination Testverfahren mit RNA-Vorlagen
Hinweis: Nach der Behandlung mit DNase, die gereinigten RNA wird Probe von rDNA-spezifische Primer getestet. Durch die Bearbeitung der Intron-ähnliche Funktion von ITSs wenn diese Regionen zur Verstärkung verwendet werden sollten keine Verstärkung Signal in DNA-freie RNA-Proben nachgewiesen werden. Auf dieser Grundlage, wenn eine Verstärkung Signal in qPCR erkannt wird oder eine Band in das Agarosegel mit der erwarteten Größe (Schätzung in Silico -Analyse), beobachtete dies aufgrund von gDNA Verunreinigung sein sollte. Die Schritte in diesem Abschnitt sind Abschnitt 3, ähnlich, außer dass cDNA aller Proben als Vorlage statt gDNA verwendet wird.
- Bereiten Sie einen master-Mix durch mischen alle Reaktionskomponenten mit Ausnahme von RNA-Vorlage in einer PCR-Reaktion-Röhre. Mix-Kombination für eine Reaktion zu meistern: 5 µL SYBR Mix (2 X), 0,3 µL Grundierung (jeweils 0,3 µM forward und reverse Primer-Mix) zu meistern, und passen Sie das Endvolumen zu 10 µL mit RNase-freies Wasser. Verwenden Sie ca. 500 ng Vorlage RNA in 1 µL Volumen für die Analyse.
- Aliquot der master-Mix in eine optische 96-Well-Platte. Pipette 1 µL RNA in jede Vertiefung, und decken Sie es durch optische Platte Abdichten Film. Zentrifuge und Platz in der Cycler.
Hinweis: Enthalten Sie mindestens zwei NTC-Steuerungen und zwei positive gDNA Steuerungen für jedes Assay. Führen Sie alle Tests in drei technischen Reproduktionen. - Die qPCR-Assay auf eine Echtzeit-Thermocycler unter folgenden Bedingungen laufen: 10 min bei 95 ° C gefolgt von 40 Zyklen von 95 ° C für 15 s und 60 ° C für 1 min. Perform Datenerfassung bei 60 ° C Glühen/Verlängerung Schritt.
- Unterziehen Sie nach der Verstärkungsprozedur alle PCR Reaktionen auf einem schmelzenden Kurvenanalyse mit kontinuierlichen Fluoreszenzmessung von 55 ° C bis 95 ° C. In der Regel sammeln Sie ein Datenpunkt pro Zyklus durch eine schrittweise Erhöhung der Temperatur um 0,5 ° C pro Zyklus.
- Überprüfen Sie alle PCR-Produkte von laufen auf 3 % Agarose gel Elektrophorese.
Hinweis: Die Darstellung von Band oder Peak in der NTC Reaktion wahrscheinlich Grundierung-Dimer-Bildung bezieht sich auf, die in der Regel bei niedrigen Temperaturen in der schmelzenden Kurve gesehen wird, während das Vorhandensein von Band oder Spitze im RNA-Proben gDNA Kontamination führt. Es empfiehlt sich, zunächst alle RNA-Proben testen von rDNA basiert Zündkapseln und dann kontaminierte Proben-DNA für downstream-Anwendungen wie cDNA Synthese, gen Expressionsanalyse, etc.verwendet werden.
5. Schritt RT-PCR cDNA Synthese und qPCR Analyse
- Tauen Sie DNase-RNA und die cDNA-Synthese-Reagenzien bei Raumtemperatur behandelt. Nach dem Auftauen spin-down der Reagenzien. Fügen Sie 1 µg RNA und 1 µL Oligo (dT) 18 Grundierung in ein Rohr Nuklease-frei. Passen Sie das Gesamtvolumen von 12 µL RNase-freies Wasser, vorsichtig mischen und dann auf Eis lagern.
- Sekundärstrukturen der RNA-Vorlage durch Inkubation der Reaktion bei 65 ° C für 5 min. Spin down schmelzen und das Fläschchen auf Eis abkühlen.
- Bereiten Sie die Reaktion-master-Mix (Endvolumen 20 µL für jede Reaktion) wie folgt: 1 µL Reverse Transkriptase (200 U/µL), 4 µL Reaktion Puffer (5 X), 1 µL RNase -Inhibitor (20 U/µL) und 2 µL dNTP-Mix (10 mM). Vorsichtig mischen und das Fläschchen auf Eis abkühlen. Fügen Sie 19 µL in die vorbereitete Röhrchen mit der RNA.
- Inkubieren Sie die Reaktion für 60 min bei 42 ° C und inkubieren Sie bei 70 ° C für 5 min, Reverse-Transkriptase-Aktivität zu beenden. Legen Sie die RT-Reaktionen auf Eis und fahren Sie mit gen Expressionsanalyse von routinemäßigen qPCR Verfahren (wie in Abschnitt 3 und 4 beschrieben).
Ergebnisse
Wir schlagen vor, die Verwendung von rDNA-basierte Primer, das Fehlen von gDNA Kontamination in RNA-Proben von Blattgewebe zu validieren. Das Flussdiagramm qPCR Analyse und gDNA Kontamination Assay ist in Abbildung 2dargestellt. Im vorliegenden Protokoll dienten zwei ergänzende Strategien für rDNA-basierte besser gekleideteres Design: (1) Spezies-spezifische Primer wurden von ITSs-Sequenzen und (2) universal Primer ITSs flankierenden Regionen ausgewählt wurden. Für Proof-of-Concept entwickelt wir Primer spezifisch für Aeluropus Littoralis und universal Primer basierend auf Poaceae Arten, wie Sie im Protokoll. Die 5.8S vorwärts und rückwärts Primer wurden ausgewählt, basierend auf einem konservierten 14 Basenpaare (bp)-Motiv, das Ähnlichkeit zwischen Blütenpflanzen, Moose und mehrere Aufträge von Algen und Pilzen14zeigt. Die Merkmale des gestalteten Primer sind in Tabelle 2dargestellt. Die Universalität des SSU, 5.8S und die LSU Grundierung wurden durch BLASTn überprüft und Grundierung Homologie Ergebnisse sind in Abbildung 3 als Motiv Logo. Die Liste der Arten enthalten in der Homologie-Analyse sowie die unterschiedlichen Primer für jede Art erhalten Tabelle 1. Grundierung Spezifität betrug Prüfung durch Primer-BLAST. Für Arten, wo die ganze Genomsequenz zur Verfügung steht, wurde die chromosomale Position der rDNA Gene geschätzt. Zum Beispiel liegen in Oryza Sativa und Arabidopsis Thaliana rDNA Gene auf zwei unterschiedlichen Chromosomen und in Zea Mays auf drei unterschiedlichen Chromosomen.
qPCR Validierung der rDNA-basierte Primer erfolgte mit schmelzen Kurvenanalyse der ITS1 und ITS2-Flanke Amplifikate DNA als Vorlage verwenden. Wie im Abbildung 4 und Abbildung 5, Grundierung Spezifität wurde experimentell bestätigt durch die Beobachtung eines einzelnen scharfe Peaks mit keine Grundierung-Dimer-Bildung in verschiedenen Poaceae Arten einschließlich Triticum Aestivum, Hordeum Vulgare, Oryza Sativa, und in der Dicotyledonen Medicago Sativa, Cucumis Sativus, Nicotiana Tabacum, Trifolium Alexandrinum, Vicia Fabaund Arabidopsis Thaliana. Die weitere Prüfung der Amplifikationsprodukte durch elektrophoretische Größe Trennung zeigte eine einzigartige Band. Wie erwartet, die Bands aus Proben von verschiedenen Arten variiert in der Größe (Abb. 6A und 6 b) abgeleitet. Interessant ist, die Nutzung der universellen Primer speziell für die drei Arten der Poaceae sind nicht nur nützlich für andere Poaceae Arten, sondern auch für andere Pflanzenarten wie A. Thaliana und ein endophytischen Pilz viz. Piriformospora Indica.
Die Gültigkeit der entwickelten spezifischen Primer (ITS1) bestätigte auch qPCR in A. Littoralis gDNA als Vorlage verwenden. Eine einzelne Spitze mit keine Grundierung-Dimer-Bildung beobachtet werden. Überraschend, A. Littoralis ITS1 Primer (als spezifische Primer) erzeugt ein einzelnes scharfes Band nicht nur in A. Littoralis , sondern auch für alle anderen Arten mit Ausnahme von Nicotiana Tabacum und Trifolium Alexandrinum getestet die beiden Bands (Abbildung 6) produziert. Die gDNA Kontamination Assay wurde von ITS oder ITS flankierende Primer in allen RNA-Proben durchgeführt. Eine schematische Darstellung der Verstärkung Platte in gDNA Kontamination Assay und der Interpretation der Ergebnisse ist in Abbildung 7dargestellt.
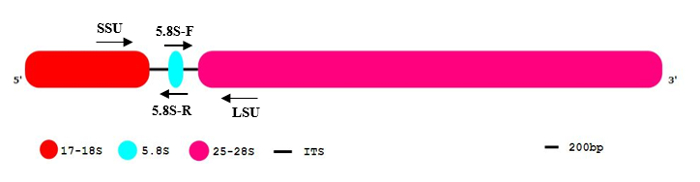
Abbildung 1: Das allgemeine Muster der eukaryotischen rDNA Sequenz Organisation.
Die eukaryotische rDNA-Segment enthält 17-18 (rot), 5.8S (blau) und 25-28 s rRNA (rosa). Die interne transkribierten Abstandhalter (ITS) werden als schwarze Linien angegeben. 5´and 3´ zeigen die Ausrichtung des DNA-Moleküls. Bitte klicken Sie hier für eine größere Version dieser Figur.
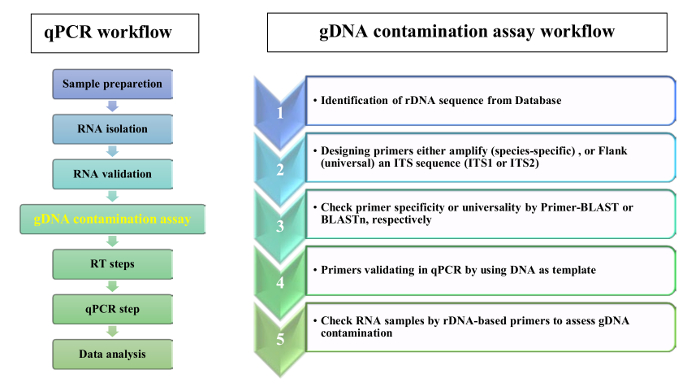
Abbildung 2: Workflow für einen RT-qPCR und gDNA Kontamination Assay. Bitte klicken Sie hier für eine größere Version dieser Figur.
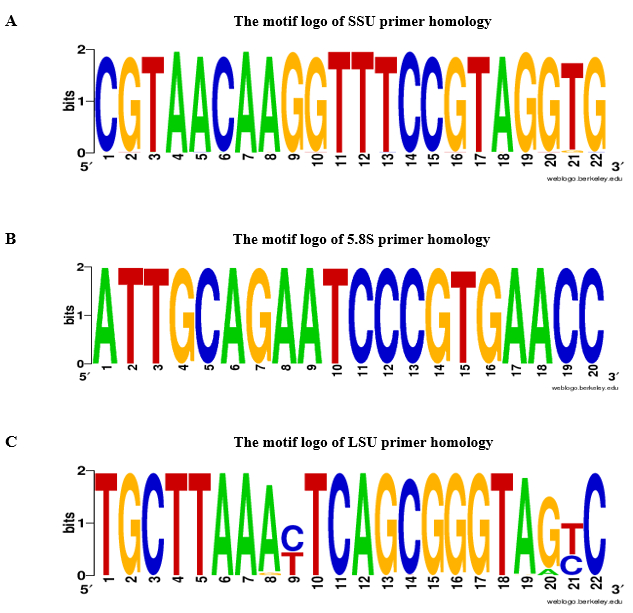
Abbildung 3: Motiv Logo von A. SSU, B. 5.8S und C. LSU Grundierung Homologie. Für SSU, 5.8S, und LSU Zündkapseln, die Motiv-Logo wurde von BLASTn basierend auf 2.000 Grünpflanze Aufzeichnungen gebaut (NCBI-USt-Nummer: 33090) mit einem Cut-off e-Wert ≤10-10. A-Adenin, T-Thymin, G-Guanin, C-Cytosin. Bitte klicken Sie hier für eine größere Version dieser Figur.
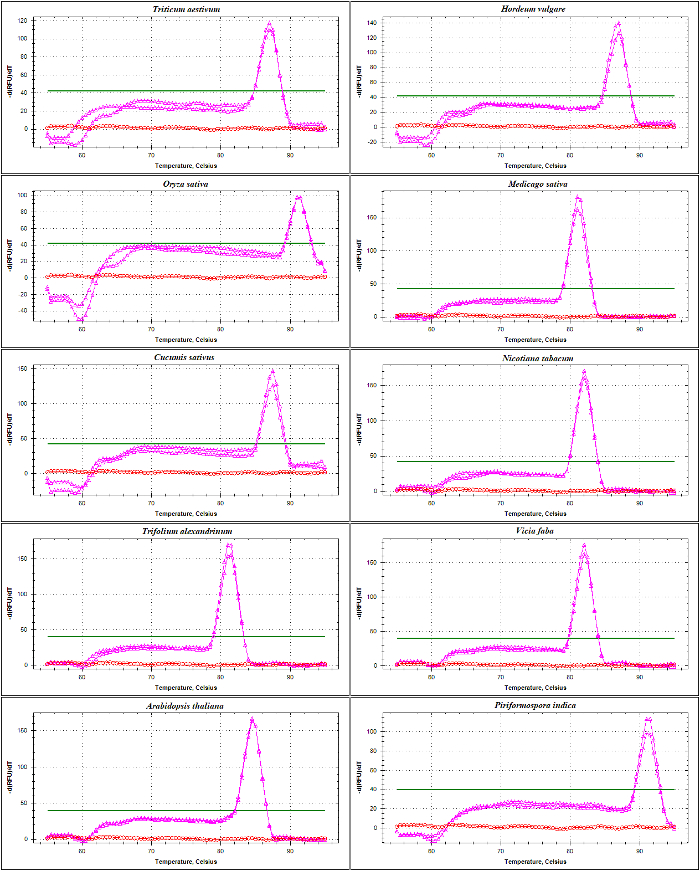
Abbildung 4: Die Schmelze Kurvenanalyse der ITS1 flankierende Amplifikate in verschiedenen Arten.
Diese Amplifikate durch verstärkt SSU und 5.8S-R Primer, enthält einen Teil der Sequenz aus der 17-18-Codierung-Region, die gesamte Sequenz der ITS1 und Teilsequenz von 5.8S. Gezeigt werden die schmelzenden Kurven der Amplifikate generiert (rosa) und NTC (rot) aus Triticum Aestivum, Hordeum Vulgare, Oryza Sativa, Medicago Truncatula, Cucumis Sativus, Nicotiana Tabacum, Trifolium Alexandrinum, Vicia Faba, Arabidopsis Thaliana und Piriformospora Indica. Die flache fette Linie zeigt den Baseline-Grenzwert. Bitte klicken Sie hier für eine größere Version dieser Figur.
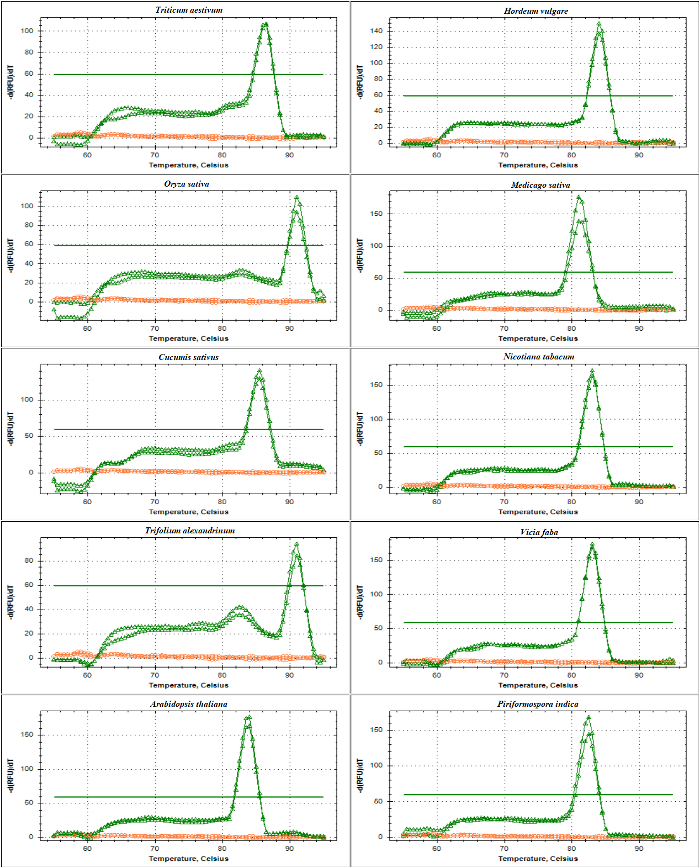
Abbildung 5: Die Schmelze Kurvenanalyse der ITS2 flankierende Amplifikate in verschiedenen Arten.
Diese Amplifikate entsteht durch die Verwendung von 5.8S-F und LSU Primern. Die beschriebenen Amplikons enthält Sequenzen des Teils von 5.8 S, die ganze Sequenz von ITS2 und eine Teilsequenz von 25-28 s. Gezeigt werden die schmelzenden Kurven der Amplifikate (grün) und NTC (rot) erzeugt aus Triticum Aestivum, Hordeum Vulgare, Oryza Sativa, Medicago Truncatula, Cucumis Sativus, Nicotiana Tabacum, Trifolium Alexandrinum, Vicia Faba, Arabidopsis Thaliana und Piriformospora Indica. Bitte klicken Sie hier für eine größere Version dieser Figur.
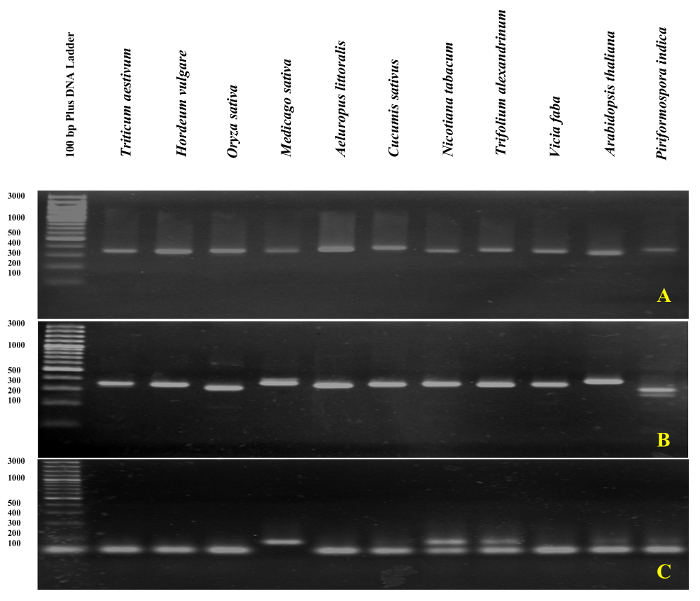
Abbildung 6: Agarose-Gelanalyse der rDNA basierende PCR-Produkt.
Der Amplifikate ITS1-Flanken (A), ITS2-Flanken (B), und ITS1 (C) wurden auf 3 % Agarosegel laufen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 7: Intron-ähnliche Funktionen von ITSs können als Primer zu entwerfen, die gDNA Verschmutzung erkennen kann.
Jeder Gipfel oder Band mit der erwarteten Größe in qPCR Analyse zeigen gDNA Belastung in der RNA-Probe. UNK: unbekannter Proben, pos: Positivkontrolle, NTC: nicht-Template-Kontrolle. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Grundierung | Gattung | USt-ID | Arten | Divergierende Grundierung | ||
| SSU | Arabidopsis | 3701 | Kamchatica, Thaliana und lyrata | - | ||
| Vicia | 3904 | Mannsschild, Americana, Unijuga, Amoenane, Amurensis, Craccamal, Pseudo-Orobus, Multicaulis, Japonica, Ramuliflora und faba | ||||
| Trifolium | 3898 | Alexandrinum, Montanum, Resupinatum und repens | - | |||
| Nicotiana | 4085 | Tabacum, Benthamiana, Otophora, Picilla, Bigelovii, Palmeri, Tomentosiformis, Tomentosa, Digluta, Kawakamii, Clevelandii, Nesophila, Solanifolia, Cordifolia, Debneyi, Arentsii, Thyrsiflora, Wigandioides, Undulata, Glutinosa, Noctiflora, Petunioides, Obtusifolia, Miersii, Pauciflora, dämpfen, Acuminata, Linearis, Alata, Sylvestris, Rustica und Suaveolens | - | |||
| Cucumis | 3655 | Anguria, Melo und sativus | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | Keine Grundierung gefunden | |||
| Medicago | 3877 | Sativa, Lupulina, Pamphylica, Lunata, rostrate, Plicata und truncatula | - | |||
| Oryza | 4597 | Sativa, Glumipatula, Rufipogon Barthii Berichte punctata, Longistaminata, Meridionalis, Nivara, Meridionalis und longistaminata | - | |||
| Triticum | 4564 | Aestivum, Urartu und Kulturgetreide | - | |||
| Hordeum | 4512 | Vulgare, Bulbosum, Marinum, Brevisubulatum und bogdanii | - | |||
| LSU | Arabidopsis | 3701 | Petraea, Thaliana und lyrata | TGCTTAAACTCAGCGGGTAATC | ||
| Vicia | 3904 | Sylvatica, Tetrasperma, Sativa, behaart, Sepium, Parviflora, Cracca, Lathyroides, Orobus, Orobus, Bithynica und faba | TGCTTAAATTCAGCGGGTAGCC | |||
| Trifolium | 3898 | Vorwand, hier, Resupinatum, Occidentale, Subterraneum, Strictum, Ochroleucon, Glomeratum, Squamosum, Ornithopodioides und repens | TGCTTAAATTCAGCGGGTAGCC | |||
| Nicotiana | 4085 | Tabacum, Benthamiana, Otophora, Picilla, Bigelovii, Palmeri, Tomentosiformis, Tomentosa, Digluta, Kawakamii, Clevelandii, Nesophila, Solanifolia, Cordifolia, Debneyi, Arentsii, Thyrsiflora, Wigandioides, Undulata, Glutinosa, Noctiflora, Petunioides, Obtusifolia, Miersii, Pauciflora, dämpfen, Acuminata, Linearis, Alata, Sylvestris und Suaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| Cucumis | 3655 | Melo, Ritchiei und javanica | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | Lagopoide, Pungens und littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| Medicago | 3877 | Ruthenica, Sativa, Lupulina, Arabica, Polymorpha und minima | TGCTTAAATTCAGCGGGTAGCC | |||
| Pamphylica, Lunata, rostrate und plicata | TGCTTAAACTCAGCGGGTAGTC | |||||
| Oryza | 4597 | Sativa, Glumipatula, Rufipogon, Barthiial, Berichte, Australiensis, Officinalis, Australiensis, Ridleyi, Malampuzhaensis, Alta, Nivara, Rufipogon, Meridionalis und longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| Triticum | 4564 | Aestivum, Spelta, Turgidum, Dicoccoides, Petropavlovskyi, Urartu und Kulturgetreide | TGCTTAAACTCAGCGGGTAGTC | |||
| Hordeum | 4512 | Vulgare, Bulbosum, Murinum, Secalinum, Brevisubulatum und bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| Eine degenerierte Primer ist definiert als IUPAC System für Nukleotid-Nomenklatur | ||||||
Tabelle 1: Die Liste der Arten für die Kommissionierung von rDNA-basierte Primer.
SSU Bindungsstelle im Vergleich zu LSU Bindungsstelle zeigten höhere Sequenzhomologie gegeben Gattung.
| Amplifikate Länge | Verstärkung-Bereich | Sequenz | Primer-name | Amplifikate | |
| 332 - 405 bp | Teilsequenz SSU, ganze Abfolge der ITS1 und Teilsequenz von 5.8S | CGTAACAAGGTTTCCGTAGGTG | SSU | ITS1-Flanken | |
| GGTTCACGGGATTCTGCAAT | 5.8S-R | ||||
| 318 - 361 bp | 5.8S, ganze Abfolge von ITS2 und Teilsequenz der LSU Teilsequenz | ATTGCAGAATCCCGTGAACC | 5.8S-F | ITS2-Flanken | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100 - 200 bp | ITS1 | GGTATGGCGTCAAGGAACACT | ITS1-F | ITS1 | |
| ATAGCATCGCTGCAAGAGGT | ITS1-R | ||||
Tabelle 2: Primer Sequenzen.
Diskussion
Gen Expressionsanalyse mittels quantitativer PCR hat in den letzten Jahren weit angewendet worden. Der Hauptvorteil dieser schnelle, kostengünstige und automatisierte Methode ist das genaue Ergebnis. Optimale Nutzen von diesen Vorteilen zu gewinnen erfordert jedoch ein klares Verständnis des Aufbaus der Parameter für das Experiment qPCR verwendet. Um ein zuverlässiges Ergebnis in qPCR Genanalyse Ausdruck zu erhalten, ist es notwendig, die unspezifische Verstärkung zu vermeiden, die aus Grundierung-Dimer oder gDNA Kontamination in der RNA-Probe3,15entsteht. Es wird erwartet, dass die RNA-Transkript-Ebenen unter gDNA Kontamination8überschätzt werden. Hier galt die Besonderheiten eines rDNA-Gens für einen gDNA Kontamination Assay in RNA-Proben.
Grundeigenschaften der rDNA in dieses Protokoll verwendet: Ribosomale Gene bestehen aus zwei ITSs, nämlich ITS1 und ITS2 und die drei rRNA codieren Gene, 17-18, 5.8S und 25-28 s Untereinheit12. Die zwei ITS-Regionen sind nicht Bestandteil der kodierenden Sequenz der ribosomalen Untereinheiten. Sie werden durch mindestens drei enzymatischen Aktivitäten den Vorläufer rRNA Reifen verarbeiten entfernt: eine Endonuklease, Helikase und Exonuclease Tätigkeit. Wie als eine Polycistronic Abschrift der ribosomalen RNA transkribiert wird, ist ein Vorprodukt, enthält die ITSs sicherlich vorhanden. Die Verarbeitung ist sehr schnell und in der Nukleolus stattfindet, und die Menge der nachweisbaren Vorläufer Moleküle mit der ITS ist unterhalb der Nachweisgrenze der qPCR-Methode. Daher als ITS1 oder ITS2 verstärkt durch ITS flankierenden Primern, kann keine Verstärkung in RNA-Proben nachgewiesen werden, es sei denn gDNA Verunreinigungen vorhanden ist. Die Anzahl der rDNA-Gene in das Genom der Eukaryonten wurde geschätzt auf bis zu tausend Exemplaren, die in Einzel- oder Tandem-Arrays auf den Chromosomen11angeordnet sind. In diesem Protokoll schlagen wir eine alternative Möglichkeit, anstelle von NRT, gDNA Verunreinigungen zu erkennen, die in jeder Reaktion/Assay verwendet wird.
Vorteile und Einschränkungen in Bezug auf bestehende Methoden: NRT dient normalerweise zum testen, ob die vorbereitete RNS-Probe sauber oder verschmutzt durch gDNA ist. Da gDNA Kontamination zwischen verschiedenen RNA-Proben nicht gleichmäßig verteilt, und die Ansprechempfindlichkeit, gDNA wesentlich durch die Gene analysiert beeinflusst wird, sind NRT Steuerelemente für jede Probe/Test paar7,15erforderlich. Dies wird im wesentlichen Kosten und Arbeit im Umgang mit viele Proben gleichzeitig3,9. Andere alternativen Methoden in der Literatur dokumentiert umfassen die Verwendung von Intron spezifische Primer für die Erkennung von gDNA oder entwerfen Zündkapseln, die entweder ein Intron flankieren oder eine Exon-Exon-Junction überspannen. Die Grenzen dieser Methoden ergeben sich aus der Nichtverfügbarkeit der Intron Sequenzinformation, unvollständige Beschriftung der Intron/Exon Struktur und das Fehlen von Introns in den Genen oder Pseudogene Interesse1,4,10 . Durch Evolution existieren rDNA Gene als multigene und hoch konservierte Genfamilien. Sie sind sehr reichlich im Genom und auf verschiedenen Chromosomen13vorhanden. Im Vergleich zu anderen Codierung oder Nonconding Gene, zeigen die rDNA-Gene die beste Lösung für die Erkennung von gDNA Kontamination. In vergleichenden transkriptomischen Analysen, die Normalisierung der qPCR Daten durch rRNA Kalibrator empfiehlt sich nicht für einige Probleme, wie z. B. Unterschiede in cDNA Vorbereitung (PolyA Priming vs. random Hexamer Grundierung), große Unterschiede in Hülle und Fülle rRNA und mRNA , und verschiedene Biogenese die irreführende erzeugen kann Ergebnisse10,16. Jedoch sind die Probleme, die wir gerade erwähnt haben einen Vorteil für die gDNA Kontamination Assay. Zum Beispiel in Bezug auf höhere targeting Website Hülle und Fülle in das Genom und Lokalisierung auf verschiedenen Chromosomen verbessern rDNA-basierte Primer erheblich die Nachweisempfindlichkeit von gDNA im Vergleich zu bisherigen Methoden.
Wandlungsfähigkeit der rDNA-basierten auf anderen Organismus: rDNA-Gene sind eine gut untersuchte Genfamilie in den meisten Organismen identifiziert. Die vorgeschlagene rDNA-basierte Methode stellt ein einfaches, hochsensible und wirtschaftliches System für gDNA Kontamination Assays, die anderen eukaryotischen und prokaryotischen Organismen (Protokoll Nr. 2 - 5) leicht angepasst werden können. Als Fallstudie haben wir hier die Nützlichkeit dieser Methode bei einigen Poceae Arten (Abbildung 4 und Abbildung 5) gezeigt. Die verwendeten Primer zeigen eine hohe Rate der Übertragbarkeit auf andere Poceae Arten aufgrund der hoch konservierte Struktur des rDNA Untereinheiten zwischen den Arten. Dieses Problem wird umso wichtiger, wenn ausreichende genomischen Sequenzinformationen nicht für besser gekleideteres Design verfügbar ist. So können ITS flankierende Primer für eine Spezies entwickelt in einer verwandten Art verwendet werden. Auch die 5.8S-F/R Primer abgeholt wurden, basierend auf einem konservierten Motiv, die große Ähnlichkeit in den meisten Blütenpflanzen14zeigt. Obwohl Hochdurchsatz-Sequenzierung Techniken dauerhaft erhöhen Sie die Anzahl der bekannten Genome, die Exon-Intron-Anmerkung der meisten Organismen ist nicht abgeschlossen, und so ist es oft nicht möglich, Primer umfassen eine Exon-Exon-Grenze zu entwerfen. Unsere Methode erklärt, wie rDNA-basierte Primer für gDNA Kontamination Assay qPCR Analyse der Prokaryoten und Eukaryoten mit dem Ziel der Beseitigung teuer NRT-Kontrollen in jedem Assay/Primer-Kombination angewendet werden können.
Offenlegungen
Die Autoren haben keinen finanziellen Interessenkonflikt.
Danksagungen
Diese Forschung wurde durch die genetischen und landwirtschaftliche Biotechnologie Institut von Tabarestan (GABIT), Sari Agrarwissenschaften und Naturressourcen Universität (SANRU) unterstützt. Die Nachwuchsgruppe abiotischen Stress Genomik wurde finanziert von IZN (interdisziplinäres Zentrum für Crop Pflanzenforschung, Halle (Saale), Deutschland. Wir danken Rhonda Meyer für kritische Lektüre des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
Referenzen
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18(2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51(2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51(2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7(2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880(2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten