Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Optische Quantifizierung der intrazellulären pH in Drosophila Melanogaster Malpighian Röhrchen Epithelien mit einem fluoreszierenden genetisch codierte pH Indikator
In diesem Artikel
Zusammenfassung
Zelluläre Ionentransport durch die Überwachung der intrazellulären pH-Wert oft beurteilt werden kann (pHich(). Genetisch bieten codiert pH-Indikatoren (GEpHIs) optische Quantifizierung von intrazellulären pH in intakten Zellen. Dieses Protokoll beschreibt die Quantifizierung von intrazellulären pH durch zelluläre ex Vivo Leben-Bildgebung von Malpighian Tubuli von Drosophila Melanogaster mit pHerry, ein Pseudo-ratiometrischen genetisch codiert pH-Indikator.
Zusammenfassung
Epitheliale Ionentransport unbedingt systemische Ion Homöostase sowie Wartung von wesentliche zelluläre elektrochemischen Gradienten. Intrazelluläre pH-Wert (pHich) wird von vielen Ionen-Transporter beeinflusst und somit pHich Überwachung ist ein nützliches Werkzeug für die Beurteilung der Transporter Aktivität. Moderne genetisch codierte pH-Indikatoren (GEpHIs) bieten optische Quantifizierung der pH-Wertich in intakten Zellen auf zellulärer und subzellulärer Ebene. Dieses Protokoll beschreibt in Echtzeit Quantifizierung der zellulärenich pH-Wertes in Malpighian Tubuli (MTs) von Drosophila Melanogaster durch ex-Vivo Leben-Bildgebung von pHerry, ein Pseudo-ratiometrischen GEpHI mit einem p-Kein gut geeignet zum Nachverfolgen von Änderungen der pH-Wert in der Zellflüssigkeit. Extrahierten Erwachsenen Fliege MTs bestehen aus morphologisch und funktionell unterschiedliche Abschnitte der einzellige Schicht Epithelien und dient als Modell für die Untersuchung von epithelialen Transport zugänglich und genetisch gefügig. GEpHIs bieten mehrere Vorteile gegenüber herkömmlichen pH-Sensitive fluoreszierende Farbstoffe und ionenselektive Elektroden. GEpHIs können unterschiedliche Zellpopulationen beschriften, sofern entsprechende Promotor Elemente verfügbar sind. Diese Kennzeichnung ist besonders nützlich in ex Vivo, in Vivound in Situ Präparate, die von Natur aus heterogen sind. GEpHIs erlauben auch die Quantifizierung der pHi im intakten Gewebe im Laufe der Zeit ohne Notwendigkeit einer Behandlung oder Gewebe Externalisierung wiederholte Farbstoff. Der Hauptnachteil des aktuellen GEpHIs ist die Tendenz zu aggregieren in cytosolischen Einschlüssen in Reaktion auf Gewebeschäden und Überexpression zu konstruieren. Diese Mängel, Lösungen und die Vorteile des GEpHIs werden in diesem Protokoll durch Bewertung der basolateralen (H+) Protonentransport in funktionell unterschiedliche Haupt- und Ganglion Zellen der extrahierten Fliege MTs demonstriert. Die Techniken und Analyse beschrieben sind leicht anpassbar an eine Vielzahl von Wirbeltieren und Wirbellosen Vorbereitungen und die Raffinesse des Assays kann von der Lehre Labs, um komplizierte Bestimmung der Ionen-Fluss über spezifische Transporter skaliert werden.
Einleitung
Das Ziel dieses Protokolls ist es, Quantifizierung von intrazellulären pH (pHi) mit einer genetisch codierte pH-Indikator (GEpHI) zu beschreiben und zeigen, wie diese Methode zur basolateralen H+ Transport in einem Modell Insekt (D. Beurteilung herangezogen werden können Melanogaster) renal Struktur, Malpighian Röhrchen (MT). MTs dienen als die Ausscheidungsorgane der Fruchtfliege und sind funktionell ähnlich der Säugetier-Nephron in einigen wesentlichen Punkten1. MTs sind als 2 Paare der Tubuli (Front- und Seitenzahnbereich) in den Thorax und Abdomen der Fliege angeordnet. Die einzelligen epitheliale Röhre von jeder MT besteht aus metabolisch aktiven wichtigsten Zellen mit deutlichen apikalen (luminalen) sowie basolateralen (Salzhaushalts) Polarität und eingefügten Ganglion Zellen. Vorderen MTs bestehen aus 3 morphologisch, funktionell und entwicklungsgeschichtlich verschiedene Segmente, vor allem die anfängliche geweitet, Segment, Übergangs-Segment und sekretorischen Hauptsegment, verbindet die Harnleiter-2. Auf der zellulären Ebene Trans-epithelialen Ionentransport in das Lumen wird erreicht, indem eine apikale Plasmamembran V-ATPase3 sowie eine Alkali-Metall/H+ -Wärmetauscher und einen basolateralen Na+-K+-ATPase4, nach innen-Gleichrichter-K+ Kanäle5, Na+-getrieben Cl−/HCO3− Wärmetauscher (NDAE1)6und Na+-K+-2 Cl− Cotransporter (NKCC; Ncc69)7, während Ganglion Zellen Cl– vermitteln und Wassertransport8,9. Dieses komplexe, aber zugänglich physiologische System bietet hervorragende Möglichkeiten zur Untersuchung der endogenen Ion Transportmechanismen in Kombination mit den vielfältigen genetischen und Verhaltensstörungen Toolsets von Drosophila.
Die Gründe für dieses Protokoll war ein genetisch formbar System für das Studium der epithelialen Ionentransport mit Potenzial für die Integration von Zelle zu Verhalten und Export von Werkzeugen zu Fremdsystemen Modell zu beschreiben. Ausdruck von pHerry10, erlaubt ein GEpHI abgeleitet aus einer Fusion von grünen pH-Sensitive Super-Ekliptik pHluorin11,12 (SEpH) und rote pH-unempfindliche mCherry13, in MTs Quantifizierung der H+ Transport in MT-Einzelzellen durch die hohe K+/nigericin Kalibrierung Technik14. Wie viele Ionen-Transporter H+ äquivalente bewegen, dient Quantifizierung der intrazellulären pHich als eine funktionelle Darstellung der Ionenbewegung über eine Vielzahl von Transportern. Die Drosophila MT-Modell-System bietet auch genetische Werkzeuge in gewebespezifischen Transgene15 und RNA-Interferenz (RNAi)16 Ausdruck, die zelluläre Bildgebung mit ganz-Orgel-Assays17 kombiniert werden können , 18 , 19 der Röhrchen-Funktion, erstellen Sie eine robusteste Toolset mit vertikalen Integration von Molekülen zu Verhalten. Dies steht im Gegensatz zu vielen anderen Protokollen zur Beurteilung epithelialen Biologie, wie historisch solche Messungen auf komplizierte verlassen haben und erschreckend, Mikro-Dissektion, anspruchsvolle ionenselektive Elektroden20,21, und teure pH-sensitiven Farbstoffen22 mit restriktiven laden Anforderungen und schlechte zelluläre Spezifität in heterogenen Geweben. GEpHIs wurden verwendet, um ausgiebig pHich in einer Vielzahl der Zelle Arten23messen. Frühwerk ausgenutzt die inhärente pH-Empfindlichkeit von Green Fluorescent Protein (GFP) pH-Wertich in kultivierten Epithelzellen24 überwachen, aber zwei Jahrzehnte habe GEpHIs verwendet in Neuronen25, Glia26, Pilze27 , und Pflanzenzellen28. Die Kombination des Potenzials für zelluläre targeting der genetischen Konstrukte durch die GAL4/UAS Ausdruck System15 und die physiologische Zugänglichkeit der Drosophila MT machen dies eine ideale Vorbereitung für Untersuchungen von pHich Regulierung und epithelialen Ionentransport.
pH-Wertesich wurde seit Jahrzehnten untersucht und ist lebenswichtig. Die MT-Vorbereitung bietet ein stabiles Modell zur Physiologie des pH-Wertesich Lehren aber auch anspruchsvolle Untersuchungen von pHi Verordnung ex-Vivo - und in Vivo. Dieses Protokoll beschreibt Quantifizierung der H+ -Bewegung in der basolateralen Membran der Epithelzellen der Drosophila MT mit NH4Cl Puls Säure laden Technik21, aber als pH-Indikator ist genetisch codiert, können diese Methoden und ihre theoretischen Rahmen zur Vorbereitung zu Transgenese und live-Imaging angewendet werden.
Protokoll
Alle Schritte in diesem Protokoll ist von der Mayo Clinic (Rochester, MN) Tiernutzung Richtlinien entsprechen.
1. fliegen Tierhaltung
- Raise fliegt und Satz kreuzt nach standard Tierhaltung29.
Hinweis: Fluoreszierende Reporter Ausdruck durch das GAL4/UAS-System ist proportional zur Temperatur und somit Aufzucht Temperatur einstellbar um Ausdruck Ebene zu ändern. Während hohe Expression Ebenen oft zu einem besseren Signal-Rausch-Verhältnis führen ist diese Bedingung ebenfalls erhöhte cytosolische und Organellen Aggregate zugeordnet, wenn mit GFP Fusion rot fluoreszierende Protein (RFP) wie z. B. pHerry10konstruiert, 30,31. Wenn Aggregation unvermeidbar ist, ist die Quantifizierung durch ausführen noch möglich Kalibrierungen in jedem Experiment und Normalisierung der Daten zeigen, so dass ein Fluoreszenz-Verhältnis von 1,0 pHich 7.0 entspricht (siehe Punkt 7.4 Hinweis auf Kalibrierung unten). - Legen Sie Kreuze der homozygot CapaR-GAL432 Männer homozygot UAS-pHerry10 jungfräulichen Weibchen und homozygot c724-GAL42 Männchen homozygot UAS-pHerry jungfräulichen Weibchen Bildgebung erlauben der pH-Wertich in Haupt und Ganglion Zellen des Mt beziehungsweise. Platz 6 UAS-pHerry Weibchen mit 3 GAL4-Männchen in frisches Fläschchen von Essen und lassen Sie zu Paaren bei 28 ° C.
Hinweis: Larven sollte deutlich in 4D und Erwachsene beginnt zu Eclose ganzen Tag 10. - Frauen fliegen auf Bewegungsapparate sammeln und beiseite stellen und Alter für 10d bei 28 ° C.
Hinweis: Das Timing des Experimentierens kann angepasst werden, um restriktiven Verhaltens Assays (z. B. die Ramsay Sekretion Assay17,19) entsprechen, die intrazellulären pH live Imaging korreliert werden. Männlichen fliegen können verwendet werden, aber Tubuli von Weibchen sind oft größer und robuster.
2. Vorbereitung des Poly-L-Lysin-Folien.
- Zeichnen Sie einen 40 x 20 mm Rahmen mit einem hydrophoben PAP-Stift um die Spitze des standard 75 x 25-mm-Dias und beiseite stellen und für 15 min bei RT. Einsatz große Deckgläsern trocknen, wenn die Folien nicht kompatibel sind mit imaging-Optik.
- 2 mL der Stammlösung 0,01 % Poly-L-Lysin (PLL) auf jeder Folie übertragen und beiseite für 1 h bei RT
- Entfernen Sie überschüssige PLL mit einer Pipette. Speichern Sie die Lösung in ein 50 mL konische Fläschchen für die zukünftige Verwendung. Shop bei 4 ° C.
- Aspirieren Sie jede verbleibende Lösung mit einer Vakuumleitung. Die gesamte Folie Oberfläche, damit keine Lösung auf den Folien bleibt überfahren Sie die Vakuumleitung.
- RT vor dem Gebrauch die Folien beiseite für 1 zusätzliche h festgesetzt. Speichern Sie die Folien trocken bei RT für bis zu 1 Monat in einem Standardfolie Buch.
3. Vorbereitung sezieren Gericht und Glasstäben
- Hinzugeben Sie 0,5 mL Elastomer Härtemittel bis 4,5 mL Elastomer Basis in 35 x 10 mm Polystyrol Petrischale bei RT zu einer Tiefe von 5 mm. Mix mit einem Einweg-Pipettenspitze. Lassen Sie Elastomer, O/N bei RT heilen
Hinweis: Elastomer sollte klar und frei von Luftblasen. Lichtung Blasen kann erleichtert werden, indem man Elastomer-Platten in einem Vakuum Glas für 10-15 min nach dem Gießen. - Halten Sie einen 5 mm Durchmesser Glasstab zwischen den Händen zu und schmelzen Sie die Mitte des Stabes bei einer beleuchteten Bunsenbrenner, und ziehen Sie die Enden auseinander. Als das Glas ziehen schmilzt mehr schnell zu produzieren ein dünn (0,1 mm) und konische Welle (Abbildung 1).
Hinweis: Ein Winkel von 45° auf dem Schaft ist oft hilfreich im Umgang mit Tubuli. Dies kann erreicht werden, indem einerseits zu senken, da der schaft gezogen wird (siehe Abbildung 1). - Brechen Sie die dünne Welle in der Mitte mit der stumpfen Seite einer Rasierklinge einschneidig Kohlenstoffstahl. Inspizieren Sie das dünne Ende des Stabes sezierenden Rahmen um sicherzustellen, dass der Bruch sauber ist.
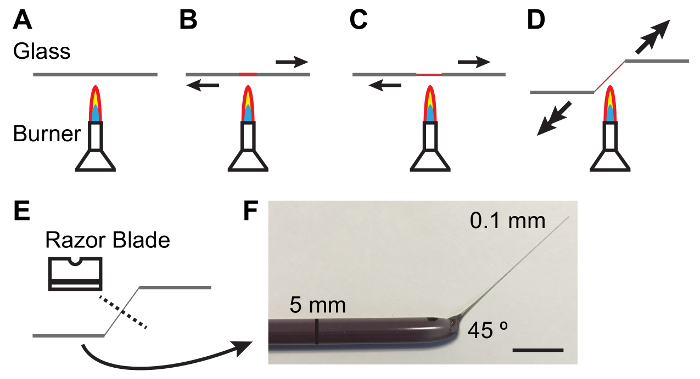
Abbildung 1: Herstellung von Glasstäben für den Umgang mit Malpighian Tubuli.
A - E. Prozess der Erwärmung und ziehen einen Glasstab zu produzieren eine Verjüngung und Winkel geeignet für den Umgang mit MTS Pfeile bezeichnen Richtung und das Ausmaß von Gewalt angewendet werden. F. Foto eines entsprechend gefertigten Glas-Tool. Maßstabsleiste = 10 mm. Bitte klicken Sie hier für eine größere Version dieser Figur.
4. Vorbereitung der Lösungen und Perfusion System
Hinweis: Perfusion Systeme unterscheiden sich von Hersteller. Dieses Protokoll basiert auf eine Schwerkraft-gefütterten 8-Kanal offenen Behälter mit einer Eingabe Durchflussregler Rate und eine Vakuum-driven Abfluss, aber die Methode der Montage, die MTs wie hier beschrieben mit einer Perfusion System arbeiten angepasst werden können.
-
Bereiten Sie die folgenden Lösungen:
- Aliquoten Schneider Medium (40 mL in 50 mL konische Röhrchen) und bei 4 ° C.
- Lösungen (d.h. Insekt Phosphate-Buffered Kochsalzlösung (iPBS) und iPBS mit NH4Cl) bei RT Bedarf gemäß Tabelle 1). Warmen Lösungen für RT vor Gebrauch am Tag des Experiments.
Hinweis: iPBS iPBS mit 40 mM NH4Cl in großen Mengen (1 L oder mehr) vorbereitet und werden bei 4 ° c gelagert - Bereiten Sie 8 Kalibrierlösungen in 500 mL Volumen bei pH = 5.0, 6.0, 6.5, 7.0, 7.3, 7.6, 8.0 und 9.0 gemäß Tabelle 1 und bei 4 ° C. PH-Wert der jeweiligen Lösung durch Titration mit N-Methyl-D-Glucamine (NMDG) und HCl einstellen.
- Am Tag der Experimente warm 5 mL Aliquots der Kalibrierlösungen auf RT und Lager Nigericin Lösung (20 mM Dimethyl Sulfoxid (DMSO)) hinzufügen, um eine Endkonzentration von 10 µM zu produzieren.
Achtung: Behandeln Sie Nigericin mit Handschuhen. Behandeln Sie alle Geräte, der in Kontakt mit Nigericin als Einweg kommt. Nigericin bleibt auf Glas und Kunststoff und gefährden biologische Präparate, wenn Ausrüstung wiederverwendet wird.
-
Perfusion System:
- Grundieren Sie die Perfusion System füllen Sie alle Behälter mit DdH2O (Abbildung 2). Öffnen Sie die Kanäle eins zu einem Zeitpunkt zu allen Linien proximal zu Rate Durchflussregler füllen lassen.
Hinweis: Es möglicherweise notwendig, Luft in den Leitungen zu löschen, um den ins Stocken geratenen Kanal öffnen und mit einem Kolben zu Laufwerk fließen aus dem Reservoir. - 2 Kanäle zu öffnen und DdH2O abtropfen lassen. Sobald der Behälter fast leer sind, füllen Sie den ersten Tank mit iPBS und den zweiten Behälter mit NH4Cl-gepulste iPBS. Durchflussmenge auf maximum mit Rate Durchflussregler und lassen Sie jede Lösung für 1 min um den distalen Linien füllen fließen, dann stoppen Sie die Strömung (Abbildung 2).
- Position 2 Sätze von Löten "helfende Hände" Klemmen auf der bildgebenden Mikroskoptisch. Legen Sie eine Klammer auf jeder Seite der imaging-Plattform.
- Vorsichtig erhitzen die distalen 0,5 Zoll ein Stück Kapillare Glas (innen-ø 1,5 mm, Außendurchmesser 0,86 mm, Länge 100 mm) über dem Bunsenbrenner. Erstellen Sie eine 45 °-Biegung, indem das distale Ende, durch die Schwerkraft zu biegen und das Glas von der Flamme entfernen, sobald der gewünschte Winkel erreicht ist. Wiederholen Sie diesen Vorgang mit einem zweiten Stück der Kapillare Glas.
- Gebogenes Glaskapillaren in der in-Flow Line und Vakuum angeschlossen Abfluss Line bzw. einfügen aus und klebe sie in die "helfende Hände" mit der bildgebenden Bühne des Mikroskops (Abbildung 3) ausrichten.
- Grundieren Sie die Perfusion System füllen Sie alle Behälter mit DdH2O (Abbildung 2). Öffnen Sie die Kanäle eins zu einem Zeitpunkt zu allen Linien proximal zu Rate Durchflussregler füllen lassen.

Abbildung 2: Perfusion System und Imaging-Konfiguration.
Komponenten für die physiologische Beurteilung des MT basolateralen Transportfunktion durch gleichzeitige Leben Fluoreszenz imaging und schnelle Lösung Austausch. Gasleitungen gezeigt sind optional und Ausbau der Experimente zur Bewertung der HCO3– Transport zu ermöglichen. Bitte klicken Sie hier für eine größere Version dieser Figur.
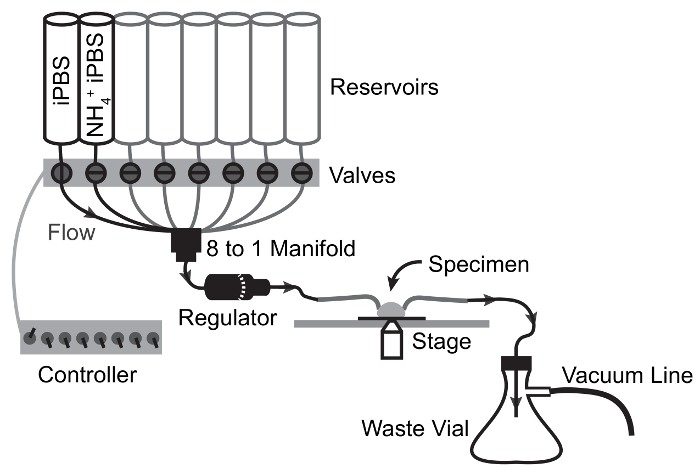
Abbildung 3: Flow schematische Darstellung der Perfusion Apparat für NH4Cl Puls experimentiert.
Pfeile zeigen Fließweg und Ventil Schaltpunkte. Lösung zieht von Reservoir zur Probe durch Schwerkraft und wird durch Vakuum-Saug die Probenkammer in den Abfall Kolben entnommen. Klicken Sie bitte hier, um eine größere Version dieser Figur.
(5) Sezieren von Erwachsenen Drosophila vorderen Malpighian Tubuli.
- Die Dissektion Schüssel sammeln und zog Glasstab aus Abschnitt 3, eine PLL-beschichtete Folie aus Abschnitt 2, eine selbstklebende Perfusion-Brunnen-Teiler, Vakuum Fett, ein 4 x 2" Streifen des Versiegelns Film, 2 Paar #5 feinen Pinzette und 40 mL Aliquote des Mediums und RT iPBS eiskalte Schneider.
- Verbreiten Sie Vakuum Fett auf das Dichtband zu, und drücken Sie den Klebstoffen Perfusion-Brunnen-Teiler auf das Band an die Unterseite mit Fett bestreichen. Abziehen des Klebstoffen Perfusion-Brunnen-Teilers und Fett-Side-Down auf der Oberseite eine PLL beschichtete Folie legen. Entfernen des Perfusion-Brunnen-Teilers um einzelne Probe Brunnen in hydrophoben Fett verfolgt verlassen.
- Platz 200 µL RT iPBS in das Fett umgeben auf der PLL beschichtete Folie gut und bewegen Sie den Schlitten unter das Stereoskop.
- Platz FH-pHerry/CapaR-GAL4 fliegen in einem leeren fliegen Fläschchen und für 10 min auf Eis zu betäuben.
Hinweis: Diese Methode der Anästhesie, im Gegensatz zu CO2, sorgt dafür, dass die fliegen nicht austrocknen. - Gießen Sie eiskalte Schneider Medium in die sezierenden Schüssel und verwenden Sie feine Pinzette, um eine Fliege narkotisierten weibliche in die Schüssel unter einem sezierenden Stereoskop übertragen.
- Halten Sie die Fliege durch den Brustkorb mit einer Zange und die andere um sanft den hinteren des Bauches greifen. Ziehen Sie den hinteren der Fliege mit der Pinzette kurz, bewusste Bewegungen. Sobald der Enddarm sichtbar ist, greifen das distale Ende und den Darm und MTs frei von der zugrunde liegenden Tracheen durch den Enddarm vom Körper durch sich wiederholende, kurze Schlepper ziehen.
Hinweis: Die vordere und hintere MTs werden angezeigt wo sie auf die Kreuzung der Mitteldarm und Enddarm durch den Harnleiter treffen. Das erste paar der MTs zu freien Willen wahrscheinlich werden die hinteren Tubuli werden wie sie den Enddarm umschließen. Diese können werden ignoriert (Abb. 4A). - Kneifen Sie aus der vorderen MTs in den Harnleiter mit feinen Zangen, sobald der zweite Satz von MTs des Bauches ist. Dies wird der vorderen MTs aus dem Bauch heraus zu trennen und schließen Sie den Harnleiter.
- Die kostenlose anterior MTs mit gezogenem Glas-Stab durch die Rute unter den Harnleiter schieben, so dass die Tubuli auf beiden Seiten fallen abholen. Heben Sie die MTs gerade nach oben aus der Lösung.
- Drehen Sie den Glasstab so, dass die MTs und Harnleiter eingehalten an der Unterseite der Rute werden und den Harnleiter gerade nach unten auf die Folie senken. Befestigen Sie den Harnleiter und verschließen Sie die distalen Enden der MTs durch den Harnleiter nach unten drücken, auf der Glas-Folie (Abbildung 4 b). Manipulieren Sie die MTs nicht mehr als nötig. Der MTs sollte sich in der Lösung mit den Harnleiter zur Folie verankert schweben.
- Verwenden Sie das feine Ende der Glasstab jedes Röhrchen vorsichtig über die Oberfläche der Folie zu fegen. Stemmen Sie die Rute gegen die Folie zu vermeiden, brechen die Röhrchen und schieben Sie den Stab über den oberen Teil des Röhrchens, Umzug distal nach proximal, Anfügen die volle Länge der einzelnen Röhrchen an der Oberfläche der PLL-beschichtete Folie (Abbildung 4).
- Ort der Klebstoff Perfusion-Brunnen-Teiler zurück auf der Folie bilden eine kleine flüssigkeitsgefüllte weit über die montierten Röhrchen.
- Legen Sie die Probe auf den Mikroskoptisch. Positionieren Sie den Zufluss und Abfluss Kapillaren über die ein- und Auslassöffnung der Durchblutung gut, beziehungsweise.
Hinweis: Der gut-Teiler kann ggf. eine offene Perfusion Kammer aufgehört werden. In diesem Fall können der Zufluss und Abfluss Kapillaren auf gegenüberliegenden Seiten der eine Bildgebung gut ausgerichtet werden.
6. Überprüfung von Imaging-Protokoll und Röhrchen Gesundheit
Hinweis: Dieses Protokoll wird durchgeführt auf einem invertierten Weitfeld-Epifluorescent Mikroskop mit GFP (SEpH) und RFP (mCherry) Filter-Sets (470/40 nm Anregung (ex), 515 nm Langpass-Emission (Em), 500 nm dichroitischen und 546/10 nm ab 590 nm Langpass-Em, 565 nm dichroitischen), ein 10 X / 0,45 Luft Ziel, eine monochrome Kamera für live-Bild-Aufnahme und imaging-Software. Das Protokoll kann für jeden aufrechten angepasst werden oder Mikroskop mit automatisierter Filter Wechsel zwischen GFP und RFP Optik und Bild Software zur Datenerfassung, invertiert, obwohl optimale Belichtungszeiten, Lichtintensität und binning Parameter variieren. In allen Analyse sollte der Fluoreszenzintensität analysiert werden als mittlere Pixel Intensität in der Region of Interest (ROI), nach Hintergrundabzug in jedem Kanal mit einem ROI mit keine Fluoreszenz angrenzend an das Signal ROI enthält.
- Schalten Sie das Mikroskop, Lichtquelle und imaging-System.
- Offen damit verbundenen imaging-Software.
- Blick durch das Okular und Fokus manuell einzustellen, bis das Lumen der MT unter Durchlicht sichtbar ist.
- Klicken Sie auf Registerkarte "Acquisition" Bildanalyse-Software und wählen Sie "2 x 2" in der "Binning" Pull-Down-Menü im Abschnitt "Acquisition Mode".
- Legen Sie ein 5 %-Graufilter in den Lichtweg Beleuchtung Licht reduzieren und minimieren Immunofluoreszenz.
- Die GFP (SEpH) Kanal im Menü "Programme", klicken Sie auf "Live" das Fluoreszenzsignal über die Kamera zu beobachten.
- Stellen Sie den Schieberegler "Time" um die Belichtungszeit so einstellen, dass die hellsten Pixelwerte im Histogramm Intensität ca. 40 % des maximalen Wertes sind, dann klicken Sie auf "Stopp", um die Beleuchtung zu stoppen.
- Wiederholen Sie Schritte 6,6-6,7 in der Ausschreibung (mCherry) Kanal und bestätigen das Vorhandensein von dilatierten ersten Segment des vorderen MT und das Fehlen der cytosolischen mCherry Aggregate (indikativ für Gewebeschäden oder Überexpression) (Abbildung 4).
Hinweis: Dilatierte Segment sollte klar ersichtlich, da es am proximalen Segment der Röhrchen und der Durchmesser des inneren Lumens dieses Segments ~ 20 µm größer als die der angrenzenden Übergangszeit Segment ist. 2 x 2 Pixel binning ist oft ausreichend, aber kann erhöht werden, um die erforderliche Beleuchtungsstärke weiter zu reduzieren. Typische Belichtungszeiten liegen zwischen 150 und 800 ms/Kanal. Verwenden Sie so wenig Licht wie möglich, um Immunofluoreszenz zu minimieren. Minimierung der Immunofluoreszenz ist entscheidend für die Verwendung von Dual-Fluorophor Indikatoren wie pHerry, wie die zwei Fluorophore unabhängig, Bleichen kann somit ungültig Verhältnis Kalibrierung. - Aktivieren Sie ein Zeitraffer bildgebenden Protokoll durch Klicken auf das Kontrollkästchen "Time Series".
- Passen Sie die "Dauer" im Pulldown-Menü im Abschnitt "Time Series" auf 10 min und den "Intervall" Regler auf 0 um die gesamte Erfassungszeit mit einer maximale Bildrate Erwerb einzustellen. Ein total Erfassungsrate von 0,2 Hz ist oft ausreichend.
- Überprüfen Sie die RFP (mCherry) Felder im Abschnitt "Kanäle" und GLP (SEpH).
- Öffnen Sie die iPBS-Linie des Systems der Perfusion durch Aktivierung der entsprechenden Stellungsregler und starten Sie die Image-Protokoll durch Klicken auf "Start-Experiment". Nach 1 min, wechseln Sie zu NH4Cl-Puls-Lösung für 20 s durch das entsprechende Ventil öffnen und Schließen der iPBS-Linie, dann zurück nach iPBS mit NH4Cl Linie schließen und erneuten Öffnen der iPBS-Ventil. Lassen Sie das volle bildgebende Protokoll vor dem Anhalten der Perfusion System abgeschlossen.
Hinweis: Time-Lapse Analyse sollte zeigen, eine stabile mCherry und ein SEpH Signal erhöht sich im Beisein von NH4Cl, stillt nach Auswaschung und allmählich erholt. - Durchführen Sie eine 2-Punkt-Kalibrierung.
- Gut Teiler Weg von der zugrunde liegenden Folie abziehen und entfernen Sie der kapillaren Durchblutung und Klemmen aus der Bildgebung gut.
- Die Bildgebung mit einer 200 µL Pipette gut 200 µL Kalibrierung iPBS (pH 7.4, 10 µM Nigericin) zuweisen. Die Lösung aus der Bildgebung mit der Pipette entfernen, dann mit einem anderen 200 µL der Kalibrierlösung ersetzen. Wiederholen Sie diesen Vorgang 4 Mal, um Komplettlösung Austausch zu gewährleisten.
- Inkubieren Sie die Vorbereitung in Kalibrierlösung für 30 min vor Bildgebung. Wiederholen Sie die Image-Protokoll mit den gleichen Parametern in Schritten 6,6-6.11 ermittelt, mit der Änderung von nur 1 min Bild zu erfassen.
Hinweis: Der Perfusion System und Kapillaren werden in diesem Schritt nicht benötigt und sollte nicht an die Bildgebung auch zu vermeiden, dass die Kapillaren zu Nigericin. - Die Bildgebung mit einer 200 µL Pipette gut 200 µL Kalibrierung iPBS (pH 9,0, 10 µM Nigericin) hinzufügen. Die Lösung aus der Bildgebung mit der Pipette entfernen, dann mit einem anderen 200 µL der Kalibrierlösung ersetzen. Wiederholen Sie diesen Vorgang 4 Mal, um Komplettlösung Austausch zu gewährleisten.
- Inkubieren Sie die Vorbereitung in die zweite Kalibrierlösung für 10 min vor Bildgebung. Wiederholen Sie die Image-Protokoll wie in Schritt 6.13.3.
- Überprüfen Sie den aufgenommene Bildstapel in Bildanalyse-Software zu bestätigen, dass keine Pixel in jedem Kanal gesättigt sind, indem Sie auf "Meine ROI" und scrollen obwohl Bildstapel mit dem "Frame"-Slider beobachtend, die keine Werte in der Intensität Histogramm berichtet den Maximalwert nachweisbaren zu erreichen. Wenn keine Frames Pixel, die die maximale nachweisbare Intensität erreichen enthalten, reduzieren Sie die Belichtung Zeit oder Beleuchtung Intensität und wiederholten Abschnitt 6.
Hinweis: Sobald hergestellt, bildgebenden Parameter zwischen Experimenten oder Kalibrierung nicht ändern, sofern Punkt Kalibrierungen sind in jeder Vorbereitung verwendet werden (siehe Punkt 8.3).
- Analysieren Sie den Bildstapel um Fluoreszenz Intensität und Fluoreszenz-Verhältnis (SEpH/mCherry) als Funktion der Zeit zu zeichnen.
- Klicken Sie auf "Meine ROI" und wählen Sie das Freiform-Werkzeug. Halten Sie mit der linken Maustaste eine ~ 50 µm Länge des Mt. zu verfolgen Rechts klicken Sie und den ROI zeichnen abzuschließen, dann wiederholen Sie in einem Gebiet neben der MT einen Hintergrund ROI (Abb. 5A) definieren.
- Klicken Sie auf "Mittlere Intensität" unter "Messungen." Erstellen Sie eine Tabelle der Intensitätswerte durch Klicken auf "Export > Datentabelle > erstellen."
- Klicken Sie auf die Konfiguration-Zahnrad-Symbol und wählen Sie deaktivieren alle Parameter mit Ausnahme von "Time" und "Meine Intensität." Mit der rechten Maustaste der Registerkarte für die neu erstellte Tabelle, wählen Sie "Speichern unter", und die Daten als CSV-Datei exportieren.
Hinweis: Ähnliche Messungen können auch mit freier Software wie ImageJ vorgenommen werden. - Öffnen Sie eine Tabelle und importieren Sie die Datentabelle zu, wählen Sie die Registerkarte "Daten" gefolgt von "Vom Text".
- Verwenden Sie Funktionen in der Tabellenkalkulation, um die SEpH Hintergrundintensität von SEpH Signalintensität zu jedem Zeitpunkt zu subtrahieren. Wiederholen Sie diesen Vorgang für das mCherry-Signal.
- Plot-jeder Kanal Intensität als Funktion der Zeit durch die Auswahl der Spalten, in denen die Zeit und Intensitätsdaten Hintergrund korrigiert und dann klicken "Einfügen > Scatter (Charts) > streuen mit geraden Linien" (Abb. 5 b).
- Verwenden Sie Arbeitsblattfunktionen, um SEpH/mCherry Fluoreszenz Verhältnis zu jedem Zeitpunkt zu berechnen.
- Grundstück-Fluoreszenz-Verhältnis als eine Funktion der Zeit durch die Auswahl der Spalten, in denen die Zeit und Verhältnis Daten und klicken dann auf "Einfügen > Scatter (Charts) > streuen mit geraden Linien" (Abbildung 5).
7. vollständige Kalibrierung des pHerry in Malpighian Tubuli Ex Vivo.
- Zerlegen Sie und montieren Sie eine neue Reihe von anterior MTs, wie in Abschnitt 5 beschrieben.
- Tauschen Sie iPBS für Kalibrierung iPBS (pH 7.4, 10 µM Nigericin), wie in Schritt 6.13.2 beschrieben. 30 min inkubieren.
- Suchen Sie die MTs und sammeln Sie Bildpaare SEpH/mCherry wie in 6.1-6.11 beschrieben. Ersetzen Sie die Lösung durch ein weiteres Lager der Kalibrierung iPBS beschriebene in Schritt 6.13.4., 10 min warten und Bild wieder. Wiederholen Sie diesen Vorgang, bis die SEpH/mCherry-Verhältnis in allen Lösungen abgebildet worden ist. Erhalten pH 9,0 Bilder dauern, wie das Exemplar selten erholt sich von hohen pH-Wert.
- Plot-Fluoreszenz Verhältnis von SEpH, mCherry von Kalibrierungen in acht Proben als Funktion der auferlegten pHich wie im Schritt 6.14.9 beschrieben. Passen Sie die Kalibrierdaten mit einer Boltzmann Kurve voll Kalibrierfunktion nach Gleichung 1 (Abbildung 5) zu erhalten. Wenn die Daten nicht übereinstimmen, Plotten Sie Kalibrations-Sets von jeder Probe normalisiert, so dass ein Fluoreszenz-Verhältnis von 1,0 pHi 7.0 entspricht und erneut analysieren Sie (Abb. 5E).
Hinweis: Wenn der letzte Prozess notwendig ist einzelne Experimente benötigen ihren eigenen inneren Punkt Kalibrierungen33 (siehe Quantifizierung folgenden Verfahren (Schritt 8.3)). -
Gleichung 1
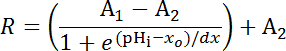
Wo R = SEpH/mCherry-Verhältnis und eine1,2, Xound Dx sind Kurve Formstück-Parameter, minimale Fluoreszenz-Verhältnis, maximale Fluoreszenz-Verhältnis, pKein, und Breite der Funktion darstellt bzw.. Xo = scheinbare pKeine der pHerry, die zwischen 7.1 und 7.4 je nach Zelltyp und genaue Kalibrierung Bedingungen variieren kann.
(8) Quantifizierung der basolateralen Säure Extrusion von Ex Vivo Malpighian Röhrchen Epithelien.
- PHerry exprimierenden stellate Bildzellen und Principal pHerry exprimierenden Zellen gleichzeitig.
- Sezieren Sie vorderen MTs von einer FH-pHerry/CapaR-GAL4 -Fliege zu, wie in Abschnitt 5 beschrieben, aber nicht übertragen Sie MTs von der sezierenden Schneider Medium für die Bildgebung gut.
- Vorderen MTs von einer FH-pHerry/c724-GAL4 -Fliege in der sezierenden Tonschüssel mit dem in Abschnitt 5 beschriebenen Verfahren zu sezieren.
- Übertragen Sie die 2 Sätze von MTs in der gleichen imaging sowie 5,8-5.11 beschrieben.
Hinweis: Wenn die Arme der MTs auf der Folie zu fegen, legen die MTs UAS-pHerry/c724-GAL4 und der FH-pHerry/CapaR-GAL4 -Tubuli nahe beieinander so pHerry exprimierenden Haupt- und Ganglion Zellen im gleichen Feld (visualisiert werden können Abbildung 6A).
- Wenden Sie das NH4Cl Prepulse wie in Schritt 6.12 beschrieben an.
Hinweis: Wenn konsistente Kalibrierung (Abbildung 4 b) nicht erreicht werden konnte, durchführen einer Punkt-Kalibrierung indem pHi auf 7.0 am Ende jedes Experiments mit Kalibrierung iPBS (pH 7,0, 10 µM Nigericin, 30 min Inkubation) nach der Klebstoff Perfusions-Brunnen-Teiler und die Perfusion Ausrüstung wurden entfernt. - Kalibrieren Sie Spuren von Ganglion und wichtigsten Zellen verschiedener MT-Segmente (mit dem absoluten oder normalisierte Verhältnis je nach Bedarf) mit Gleichung 2 und analysieren Sie die Erholungsphase nach NH4Cl Rückzug durch die Anwendung der exponentiellen Zerfall Funktionen mit Software zur statistischen Analyse und Kenntnis nehmend von den Verfall-Konstante (τ) (Abb. 6 b).
Gleichung 2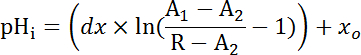
Wo R = SEpH/mCherry-Verhältnis und eine1,2, Xound Dx Kurve Formstück-Parameter durch Kalibrierung in Schritt 7.4 (Gleichung 1) bestimmt sind.- Berechnen Sie sauren Extrusionsgeschwindigkeit (JH +, siehe Gleichung 3) als Funktion der pH-Wertich , um Variationen in ruhenden pHich und Säure zwischen Vorbereitungen34laden zu berücksichtigen. Verwenden der Exponentialfunktionen im Schritt 8.3 abgeleitet, um die Ableitung von pHich zeitlich in jedem Zeitintervall zu berechnen.
Gleichung 3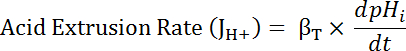
- Berechnen Sie die intrinsische Pufferkapazität (β-i; Gleichung 4) von der Zellflüssigkeit bei der pHich von Anfang an jedes Intervall im Schritt 8.3.1 basierend auf bisherigen Literatur (siehe Gleichung 4).
Hinweis: Bei Drosophila, die gründlichste Charakterisierung von βich stammt aus Larven motorischen Nerv Klemmen35 und diese Daten können davon ausgegangen werden, für MT Zellen in Ermangelung anderer verfügbarer Daten zu halten.
Gleichung 4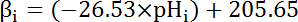
- Berechnen Sie das Produkt von βT (aus Schritt 8.3.2)und DpHich/dt (aus Schritt 8.3.1) um JH + (Gleichung 3) festzustellen.
Hinweis: In nominell Bikarbonat-freien Lösungen wie beschrieben in diesem Protokoll, Bikarbonat-abgeleitete Pufferkapazität (β-b) wird angenommen, dass ~ 0 mM. Insgesamt Pufferkapazität (βT) ist die Summe der βich βbund somit βich = βT in Ermangelung von HCO3–Co236. - Plot JH + in Abhängigkeit von dem pH-Wertich am Anfang jedes Zeitintervall wie in Schritt 6.14.9 beschrieben.
- Bereich für alle Datensätze, die in pHich mittels statistischer Analysesoftware überlappen zuweisen Sie exponentiellen Zerfall Funktionen. Vergleichen Sie die Preise des Wandels der daraus resultierenden Funktionen, sauren Extrusion Preise zwischen Zellen und MT Segmente (Abbildung 6) zu vergleichen.
Hinweis: Die am besten geeignete Funktion für Kurvenanpassung verwendet möglicherweise nicht immer einem exponentiellen. Weitere Funktionen können ersetzt werden, wenn sie die Güte der Anpassung verbessern. - Berechnen Sie Säure flux (siehe Gleichung 5) als Funktion der pH-Wertich , um Variationen in Form und Größe der Zellen zu berücksichtigen.
Gleichung 5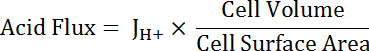
Hinweis: Zellengröße können entweder direkt in Bilder gemessen oder angenähert. Wichtigsten Zellen darstellbar als Hälften ein hohles Rohr mit den folgenden Abmessungen: Innendurchmesser 24 µm; Außendurchmesser 48 µm; Höhe 50 µm. Transitional Ganglion Zellen sind variabel aber als Zylinder mit Höhen von 50 µm und einem Durchmesser von 10 µm etwa dargestellt werden können. Letzten Absatz des Vertreters Ergebnisse unten zu sehen. - Bereich für alle Datensätze, die in pHich mittels statistischer Analysesoftware überlappen zuweisen Sie exponentiellen Zerfall Funktionen. Vergleichen Sie die Preise des Wandels der daraus resultierenden Funktionen, saure Flussmittel zwischen Zellen und MT Segmente (Abbildung 6) zu vergleichen.
- Berechnen Sie sauren Extrusionsgeschwindigkeit (JH +, siehe Gleichung 3) als Funktion der pH-Wertich , um Variationen in ruhenden pHich und Säure zwischen Vorbereitungen34laden zu berücksichtigen. Verwenden der Exponentialfunktionen im Schritt 8.3 abgeleitet, um die Ableitung von pHich zeitlich in jedem Zeitintervall zu berechnen.
Ergebnisse
Gesundes Gewebe und korrekte Kennzeichnung der vorderen MTs sind entscheidend für den Erfolg dieses Protokolls. Während der Dissektion sollte darauf geachtet werden, nicht direkt berühren die MTs und einzige Griff führt sie durch den Harnleiter als greifen die MTs direkt zum Bruch (– Abbildung 4A B). Wenn MTs flach auf der Folie gekehrt werden, die Tubuli darf so wenig wie möglich berührt werden und überflüssige Bew...
Diskussion
Der Erfolg der Quantifizierung der pH-Wertich in Drosophila MTs hängt ganz von der Gesundheit der extrahierten MTs und die Qualität der Montage und Zerlegung (Abbildung A – C). So ist der sorgsame Umgang mit Gewebe wie beschrieben zwingend. Folien, die frisch in PLL beschichtet Folien wesentlich Hilfe MT Montage, wie sie neigen dazu, viel mehr Kleber als die zuvor ausgesetzt waren auf Lösung. Sorgfältige Montage hilft auch bei der Identifizier...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde unterstützt von NIH DK092408 und DK100227, Herst AJR wurde von T32-DK007013 unterstützt. Möchten die Autoren danken Dr. Julian A.T. Dow für die CapaR-GAL4 und c724-GAL4 Drosophila Bestände. Wir danken auch Jacob B. Anderson für die Unterstützung, die Aufrechterhaltung der experimenteller fliegen Kreuzungen.
Materialien
| Name | Company | Catalog Number | Comments |
| Poly-L-Lysine (PLL) Solution | Sigma-Aldrich | P4832 | Store at 4 °C, can be reused. |
| Nigericin Sodium Salt | Sigma-Aldrich | N7143 | CAUTION: Handle with gloves. Store as aliquots of 20 mM stock solution in DMSO at 4 °C. |
| Adhesive Perfusion Chamber Covers, adhesive size 1 mm, chamber diameter × thickness 9 mm × 0.9 mm, ports diameter 1.5 mm | Sigma-Aldrich | GBL622105 | Can be substituted as needed to match perfusion system. |
| Sylgard 184 Silicone Elastomer Kit | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Available from multiple vendors. |
| Helping Hands Soldering Stands | Harbor Freight Tools | 60501 | Available from multiple vendors. |
| Open Gravity-fed Perfusion System with Valve Controller, 8 to 1 Manifold and Reserviors | Bioscience Tools | PS-8S | Any comparable perfusion system can be used. |
| Flow Regulator | Warner Instruments | 64-0221 | Can be substituted as needed to match perfusion system. |
| Schneider's Medium | Fisher Scientific | 21720024 | Store at 4 °C in sterile aliquots. |
| #5 Inox Steel Forceps | Fine Science Tools | 11252-20 | Can be substituted based on experimenter comfort. |
| 35 x 10 mm polystyrene Petri dish | Corning Life Sciences | Fisher Scientific 08-757-100A | Exact brand and size are unimportant. |
| 75 x 25 mm Microscope Slides | Corning Life Sciences | 2949-75X25 | Exact brand and size can vary as long as perfusion wells are compatible. |
| Filimented Borosilicate Capillary Glass, ID 1.5 mm, OD 0.86 mm, thickness 0.32 mm | Warner Instruments | 64-0796 | Filiment not necessary, glass can be substituted to match perfusion tubing and perfusion wells. |
| Tygon Tubing, ID 1/16", OD 1/8", thickness 1/32" | Fisher Scientific | 14-171-129 | Available from multiple vendors, can be substituted to match perfusion system. |
| Vacuum Silicone Grease | Sigma-Aldrich | Z273554 | Available from multiple vendors. |
| Plastic Flow Control Clamp | Fisher Scientific | 05-869 | Available from multiple vendors, sterility not required |
| Glass rods, 5 mm diameter | delphiglass.com | 9198 | Exact size is personal preference, multiple vendors available |
| PAP Hydrophobic Pen | Sigma-Aldrich | Z377821 | Available from multiple vendors. |
| Sealing Film | Sigma-Aldrich | P7668 | Available from multiple vendors. |
| 15 mL Falcon tube | BD Falcon | 352096 | Available from multiple vendors. |
| 50 mL Falcon tube | BD Falcon | 352070 | Available from multiple vendors. |
| HEPES; 4-(2-Hydroxyethyl)piperazine-1-ethanesulfonic acid | Sigma-Aldrich | H3375 | Available from multiple vendors. |
| MES; 4-Morpholineethanesulfonic acid monohydrate | Sigma-Aldrich | 69892 | Available from multiple vendors. |
| TAPS; N-[Tris(hydroxymethyl)methyl]-3-aminopropanesulfonic acid | Sigma-Aldrich | T5130 | Available from multiple vendors. |
| 10X/0.45 Air Objective | Zeiss | 000000-1063-139 | Comparable objectives can be substituted. 40X objectives can be used for single cell imaging. |
| Dissecting Stereoscope | Zeiss | Discovery.V8 | Any dissecting stereoscope can be used. |
| UAS-pHerry transgenic Drosophila melagnogaster | Available from Romero Lab | First published: Citation 10 | |
| capaR-GAL4 driver line Drosophila melagnogaster | Available from Romero Lab | First published: Citation 32 | |
| c724-GAL4 driver line Drosophila melagnogaster | Available from Romero Lab | First published: Citation 2 | |
| Monochromatic High Sensitivity Digital Camera | Zeiss | Axiocam 506 mono | Exact brand and model can vary, can be replaced with any monochromatic high-sensitivity camera suited to live cellular imaging. |
| GFP/FITC filter set, 470/40 nm ex., 515 nm longpass em., 500 nm dichroic | Chroma | CZ909 | Any GFP/FITC filer set can be substituted. |
| RFP/TRITC filter set, 546/10 nm ex., 590 nm longpass em., 565 nm dichroic | Chroma | CZ915 | Any GFP/FITC filer set can be substituted. |
| Inverted Epifluoescent Microscope | Zeiss | Axio Observer Z.1 | Any comparable microscope with motorized filter switching can be used. Upright microscopes can be used with open perfusion baths and water-immersion objectives. |
| Statistical Analysis Software | Microcal | Origin 6.0 | Any software with comparable functionality can be substituted |
| Image Analysis Software | National Institutes of Health | ImageJ 1.50i | Any software with comparable functionality can be substituted |
| Image Acquisition Software | Zeiss | Zen 1.1.2.0 | Any software with comparable functionality can be substituted |
| Single-edged Carbon Steel Razor Blade | Electron Microscopy Sciences | 71960 | Available from multiple vendors. |
| Microscopy Slide Folder | Fisher Scientific | 16-04 | Available from multiple vendors. |
| Bunsen Burner | Fisher Scientific | 50-110-1231 | Available from multiple vendors. |
| Polystrene Drosophila Rearing Vials with Flugs | Genesee Scientific | 32-109BF | Comparable items can be substituted. |
| 2.5 L Laboratory Ice Bucket | Fisher Scientific | 07-210-129 | Available from multiple vendors. |
| NMDG; N-Methyl-D-glucamine | Sigma-Aldrich | M2004 | Available from multiple vendors. |
| 200 uL barrier pipette tips | MidSci | AV200 | Available from multiple vendors. |
| 200 μL variable volume pipette | Gilson Incorporated | PIPETMAN P200 | Available from multiple vendors. |
Referenzen
- Dow, J. A. T., Romero, M. F. Drosophila provides rapid modeling of renal development, function, and disease. Am J Physiol Renal Physiol. 299 (6), F1237-F1244 (2010).
- Sozen, M. A., Armstrong, J. D., Yang, M., Kaiser, K., Dow, J. A. Functional domains are specified to single-cell resolution in a Drosophila epithelium. P Natl Acad Sci USA. 94 (10), 5207-5212 (1997).
- Davies, S. A., et al. Analysis and inactivation of vha55, the gene encoding the vacuolar ATPase B-subunit in Drosophila melanogaster reveals a larval lethal phenotype. J Biol Chem. 271 (48), 30677-30684 (1996).
- Torrie, L. S., et al. Resolution of the insect ouabain paradox. P Natl Acad Sci USA. 101 (37), 13689-13693 (2004).
- Evans, J. M., Allan, A. K., Davies, S. A., Dow, J. A. Sulphonylurea sensitivity and enriched expression implicate inward rectifier K+ channels in Drosophila melanogaster renal function. J Exp Biol. 208 (Pt 19), 3771-3783 (2005).
- Sciortino, C. M., Shrode, L. D., Fletcher, B. R., Harte, P. J., Romero, M. F. Localization of endogenous and recombinant Na(+)-driven anion exchanger protein NDAE1 from Drosophila melanogaster. Am J Physiol Cell Physiol. 281 (2), C449-C463 (2001).
- Ianowski, J. P., O'Donnell, M. J. Basolateral ion transport mechanisms during fluid secretion by Drosophila Malpighian tubules: Na+ recycling, Na+:K+:2Cl- cotransport and Cl- conductance. J Exp Biol. 207 (Pt 15), 2599-2609 (2004).
- O'Donnell, M. J., et al. Hormonally controlled chloride movement across Drosophila tubules is via ion channels in stellate cells. Am J Physiol. 274 (4 Pt 2), R1039-R1049 (1998).
- Cabrero, P., et al. Chloride channels in stellate cells are essential for uniquely high secretion rates in neuropeptide-stimulated Drosophila diuresis. P Natl Acad Sci USA. 111 (39), 14301-14306 (2014).
- Rossano, A. J., Kato, A., Minard, K. I., Romero, M. F., Macleod, G. T. Na+ /H+ -exchange via the Drosophila vesicular glutamate transporter (DVGLUT) mediates activity-induced acid efflux from presynaptic terminals. J Physiol. 595 (3), 805-824 (2017).
- Sankaranarayanan, S., De Angelis, D., Rothman, J. E., Ryan, T. A. The use of pHluorins for optical measurements of presynaptic activity. Biophys J. 79 (4), 2199-2208 (2000).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat biotechnol. 22 (12), 1567-1572 (2004).
- Thomas, J. A., Buchsbaum, R. N., Zimniak, A., Racker, E. Intracellular pH measurements in Ehrlich ascites tumor cells utilizing spectroscopic probes generated in situ. Biochemistry. 18 (11), 2210-2218 (1979).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448 (7150), 151 (2007).
- Dow, J. A., et al. The malpighian tubules of Drosophila melanogaster: a novel phenotype for studies of fluid secretion and its control. J Exp Biol. 197, 421-428 (1994).
- Hirata, T., et al. In vivo Drosophilia genetic model for calcium oxalate nephrolithiasis. Am J Physiol Renal Physiol. 303 (11), F1555-F1562 (2012).
- Schellinger, J. N., Rodan, A. R. Use of the Ramsay Assay to Measure Fluid Secretion and Ion Flux Rates in the Drosophila melanogaster Malpighian Tubule. J Vis Exp. (105), (2015).
- Caldwell, P. C. An investigation of the intracellular pH of crab muscle fibres by means of micro-glass and micro-tungsten electrodes. J Physiol. 126 (1), 169-180 (1954).
- Boron, W. F., De Weer, P. Intracellular pH transients in squid giant axons caused by CO2, NH3, and metabolic inhibitors. J Gen Physiol. 67 (1), 91-112 (1976).
- Rink, T. J., Tsien, R. Y., Pozzan, T. Cytoplasmic pH and free Mg2+ in lymphocytes. J Cell Biol. 95 (1), 189-196 (1982).
- Bizzarri, R., Serresi, M., Luin, S., Beltram, F. Green fluorescent protein based pH indicators for in vivo use: a review. Anal Bioanal Chem. 393 (4), 1107-1122 (2009).
- Kneen, M., Farinas, J., Li, Y., Verkman, A. S. Green fluorescent protein as a noninvasive intracellular pH indicator. Biophys J. 74 (3), 1591-1599 (1998).
- Raimondo, J. V., Irkle, A., Wefelmeyer, W., Newey, S. E., Akerman, C. J. Genetically encoded proton sensors reveal activity-dependent pH changes in neurons. Front Mol Neurosci. 5, 68 (2012).
- Raimondo, J. V., et al. Tight Coupling of Astrocyte pH Dynamics to Epileptiform Activity Revealed by Genetically Encoded pH Sensors. J Neurosci. 36 (26), 7002-7013 (2016).
- Bagar, T., Altenbach, K., Read, N. D., Bencina, M. Live-Cell imaging and measurement of intracellular pH in filamentous fungi using a genetically encoded ratiometric probe. Eukaryot Cell. 8 (5), 703-712 (2009).
- Gjetting, K. S., Ytting, C. K., Schulz, A., Fuglsang, A. T. Live imaging of intra- and extracellular pH in plants using pHusion, a novel genetically encoded biosensor. J Exp Bot. 63 (8), 3207-3218 (2012).
- Greenspan, R. J. . Fly pushing: the theory and practice of Drosophila genetics. , (2004).
- Raimondo, J. V., et al. A genetically-encoded chloride and pH sensor for dissociating ion dynamics in the nervous system. Front Cell Neurosci. 7, 202 (2013).
- Koivusalo, M., et al. Amiloride inhibits macropinocytosis by lowering submembranous pH and preventing Rac1 and Cdc42 signaling. J Cell Biol. 188 (4), 547-563 (2010).
- Terhzaz, S., et al. Mechanism and function of Drosophila capa GPCR: a desiccation stress-responsive receptor with functional homology to human neuromedinU receptor. PloS one. 7 (1), e29897 (2012).
- Boyarsky, G., Ganz, M. B., Sterzel, R. B., Boron, W. F. pH regulation in single glomerular mesangial cells. I. Acid extrusion in absence and presence of HCO3. Am J Physiol. 255 (6 Pt 1), C844-C856 (1988).
- Chesler, M. The regulation and modulation of pH in the nervous system. Prog Neurobiol. 34 (5), 401-427 (1990).
- Rossano, A. J., Chouhan, A. K., Macleod, G. T. Genetically encoded pH-indicators reveal activity-dependent cytosolic acidification of Drosophila motor nerve termini in vivo. J Physiol. 591 (7), 1691-1706 (2013).
- Roos, A., Boron, W. F. Intracellular pH. Physiol Rev. 61 (2), 296-434 (1981).
- Vaughan-Jones, R. D., Wu, M. L. pH dependence of intrinsic H+ buffering power in the sheep cardiac Purkinje fibre. J Physiol. 425, 429-448 (1990).
- Buckler, K. J., Vaughan-Jones, R. D., Peers, C., Nye, P. C. Intracellular pH and its regulation in isolated type I carotid body cells of the neonatal rat. J Physiol. 436, 107-129 (1991).
- Bevensee, M. O., Schwiening, C. J., Boron, W. F. Use of BCECF and propidium iodide to assess membrane integrity of acutely isolated CA1 neurons from rat hippocampus. J Neurosci Methods. 58 (1-2), 61-75 (1995).
- Arosio, D., et al. Simultaneous intracellular chloride and pH measurements using a GFP-based sensor. Nat Methods. 7 (7), 516-518 (2010).
- Wu, Y., Baum, M., Huang, C. L., Rodan, A. R. Two inwardly rectifying potassium channels, Irk1 and Irk2, play redundant roles in Drosophila renal tubule function. Am J Physiol Regul Integr Comp Physiol. 309 (7), R747-R756 (2015).
- Schulte, A., Lorenzen, I., Bottcher, M., Plieth, C. A novel fluorescent pH probe for expression in plants. Plant Methods. 2, 7 (2006).
- Shen, Y., Rosendale, M., Campbell, R. E., Perrais, D. pHuji, a pH-sensitive red fluorescent protein for imaging of exo- and endocytosis. J Cell Biol. 207 (3), 419-432 (2014).
- Johnson, D. E., et al. Red fluorescent protein pH biosensor to detect concentrative nucleoside transport. J Biol Chem. 284 (31), 20499-20511 (2009).
- Mahon, M. J. pHluorin2: an enhanced, ratiometric, pH-sensitive green florescent protein. Adv Biosci Biotechnol. 2 (3), 132-137 (2011).
- Li, Y., Tsien, R. W. pHTomato, a red, genetically encoded indicator that enables multiplex interrogation of synaptic activity. Nat Neurosci. 15 (7), 1047-1053 (2012).
- Tantama, M., Hung, Y. P., Yellen, G. Imaging intracellular pH in live cells with a genetically encoded red fluorescent protein sensor. J Am Chem Soc. 133 (26), 10034-10037 (2011).
- Matlashov, M. E., et al. Fluorescent ratiometric pH indicator SypHer2: Applications in neuroscience and regenerative biology. Biochimica et biophysica acta. 1850 (11), 2318-2328 (2015).
- Kogure, T., et al. A fluorescent variant of a protein from the stony coral Montipora facilitates dual-color single-laser fluorescence cross-correlation spectroscopy. Nat biotechnol. 24 (5), 577-581 (2006).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. P Natl Acad Sci USA. 95 (12), 6803-6808 (1998).
- Poburko, D., Santo-Domingo, J., Demaurex, N. Dynamic regulation of the mitochondrial proton gradient during cytosolic calcium elevations. J Biol Chem. 286 (13), 11672-11684 (2011).
- Stornaiuolo, M., et al. KDEL and KKXX retrieval signals appended to the same reporter protein determine different trafficking between endoplasmic reticulum, intermediate compartment, and Golgi complex. Mol Biol Cell. 14 (3), 889-902 (2003).
- Makkerh, J. P., Dingwall, C., Laskey, R. A. Comparative mutagenesis of nuclear localization signals reveals the importance of neutral and acidic amino acids. Curr Biol. 6 (8), 1025-1027 (1996).
- Zacharias, D. A., Violin, J. D., Newton, A. C., Tsien, R. Y. Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science. 296 (5569), 913-916 (2002).
- McGuire, R. M., Silberg, J. J., Pereira, F. A., Raphael, R. M. Selective cell-surface labeling of the molecular motor protein prestin. Biochem Biophys Res Comm. 410 (1), 134-139 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten