Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Vorhersage des Gen-Silens durch die räumlich-zeitliche Kontrolle der siRNA-Freisetzung von photoreaktiven polymeren Nanocarriern
In diesem Artikel
Zusammenfassung
Wir stellen eine neuartige Methode vor, die photoempfindliche Blockcopolymere für eine effizientere räumlich-zeitliche Kontrolle von Gen-Silencing ohne nachweisbare Off-Target-Effekte verwendet. Zusätzlich können Veränderungen in der Genexpression unter Verwendung einfacher siRNA-Freisetzungsassays und einfacher kinetischer Modellierung vorhergesagt werden.
Zusammenfassung
Neue Materialien und Methoden sind erforderlich, um die Bindung gegen die Freisetzung von Nukleinsäuren für eine breite Palette von Anwendungen, die die genaue Regulation der Genaktivität erfordern, besser zu kontrollieren. Insbesondere würden neuartige stimuli-reaktionsfähige Materialien mit einer verbesserten räumlich-zeitlichen Kontrolle über die Genexpression verschiebbare Plattformen in der Wirkstoffforschung und der regenerativen Medizintechnologie freischalten. Darüber hinaus würde eine verbesserte Fähigkeit, die Nucleinsäure-Freisetzung aus Materialien zu kontrollieren, die Entwicklung von stromlinienförmigen Verfahren ermöglichen, um die Nanokarrier-Wirksamkeit a priori vorherzusagen, was zu einem beschleunigten Screening von Lieferfahrzeugen führt. Hier präsentieren wir ein Protokoll zur Vorhersage von Gen-Silencing-Effizienzen und zur Erzielung einer räumlich-zeitlichen Kontrolle über die Genexpression durch ein modulares photoempfindliches Nanocarrier-System. Kleine interferierende RNA (siRNA) wird mit mPEG- b- Poly (komplexen (5- (3- (Amino) propoxy) -2-nitrobenzylmethacrylat) (mPEG- b- P (APNBMA)) -Polymeren zu foRm stabile Nanocarrier, die mit Licht gesteuert werden können, um einstellbares, on / off siRNA Release zu ermöglichen. Wir skizzieren zwei komplementäre Assays mit Fluoreszenz-Korrelationsspektroskopie und Gelelektrophorese für die genaue Quantifizierung der siRNA-Freisetzung aus Lösungen, die intrazelluläre Umgebungen nachahmen. Informationen, die aus diesen Assays gewonnen wurden, wurden in ein einfaches RNA-Interferenz- (RNAi) -Kinetikmodell eingearbeitet, um die dynamischen Stummschaltungsreaktionen auf verschiedene Photo-Stimulus-Bedingungen vorherzusagen. Im Gegenzug ermöglichten diese optimierten Bestrahlungsbedingungen die Verfeinerung eines neuen Protokolls zur räumlich-zeitlichen Steuerung von Gen-Silencing. Diese Methode kann zelluläre Muster in der Genexpression mit Zell-zu-Zell-Auflösung und keine nachweisbaren Off-Target-Effekte erzeugen. Zusammenfassend bietet unser Ansatz eine einfach zu bedienende Methode zur Vorhersage dynamischer Veränderungen der Genexpression und zur präzisen Steuerung der siRNA-Aktivität in Raum und Zeit. Dieser Satz von Assays kann leicht angepasst werden, um eine breite Vielfalt von ot zu testenIhre stimuli-reaktionsfähigen Systeme, um die wichtigsten Herausforderungen zu adressieren, die für eine Vielzahl von Anwendungen in der biomedizinischen Forschung und Medizin relevant sind.
Einleitung
Kleine interferierende RNAs (siRNAs) vermitteln das posttranskriptionelle Gen-Silencing durch einen katalytischen RNAi-Weg, der hochspezifisch, potent und maßgeschneidert für nahezu jedes Zielgen 1 ist . Diese vielversprechenden Eigenschaften haben es ermöglicht, dass siRNA-Therapeutika in menschlichen klinischen Studien zur Behandlung zahlreicher Krankheiten, einschließlich metastasiertem Melanom und Hämophilie 2 , 3 vorankommen. Allerdings bestehen erhebliche Lieferprobleme, die die Übersetzung behindert haben 4 . Insbesondere müssen Lieferfahrzeuge stabil bleiben und siRNAs vor extrazellulärem Abbau schützen und dennoch die Nutzlast in das Zytoplasma 5 freisetzen. Darüber hinaus erfordern viele RNAi-Anwendungen verbesserte Methoden zur Regulierung der Gen-Silencing in Raum und Zeit 6 , die Nebenwirkungen in siRNA-Therapeutika reduzieren 7 und ermöglichen transformative aDvances in Anwendungen von Zell-Mikroarrays für die Wirkstoffforschung 8 bis hin zur Modulation von Zellreaktionen in regenerativen Gerüsten 9 . Diese Herausforderungen unterstreichen die Notwendigkeit neuer Materialien und Methoden zur besseren Kontrolle der Bindung gegenüber der Freisetzung in siRNA-Nanocarrier.
Eine der vielversprechendsten Strategien zur Kontrolle der siRNA-Freisetzung und zur Verbesserung der räumlich-zeitlichen Regulierung ist der Einsatz von stimuli-reaktionsfähigen Materialien 10 . Beispielsweise wurde eine Vielzahl von Biomaterialien mit einer veränderlichen Nukleinsäure-Bindungsaffinität in Reaktion auf verändertes Redoxpotential oder pH-Wert oder angewandte Magnetfelder, Ultraschall oder Licht 11 entwickelt . Obwohl viele dieser Systeme eine verbesserte Kontrolle über die Nukleinsäureaktivität zeigen, ist die Verwendung von Licht als Trigger besonders vorteilhaft aufgrund seiner augenblicklichen zeitlichen Reaktion, der präzisen räumlichen Auflösung und der Leichtigkeit der Abstimmbarkeit 12. Darüber hinaus wurde das Potenzial von photoempfindlichen Technologien zur Regulierung der Genexpression durch modernste induzierbare Promotor- und optogenetische Regulierungssysteme nachgewiesen; Diese Systeme leiden jedoch unter zahlreichen Herausforderungen, einschließlich begrenzter Kapazitäten zur Regulierung endogener Gene, Sicherheitsbedenken wie Immunogenität und Schwierigkeiten bei der Bereitstellung von Mehrkomponenten-Baugruppen 13 , 14 , 15 . Photo-responsive siRNA-Nanocarrier sind ideal geeignet, um diese Nachteile zu überwinden und bieten einen einfacheren und robusteren Ansatz zur räumlich-modulierenden Genexpression 16 , 17 , 18 . Leider sind Methoden zur genauen Vorhersage der resultierenden Protein-Knockdown-Antwort unklar.
Eine zentrale Herausforderung ist, dass quantitative Auswertungen der siRNA-Freisetzung sindSelten 19 , 20 , und selbst wenn diese Auswertungen durchgeführt werden, wurden sie nicht mit Analysen der siRNA / Protein-Umsatzdynamik gekoppelt. Sowohl die Menge an siRNA freigesetzt als auch ihre Persistenz / Lebensdauer sind wichtige Determinanten der resultierenden Gen-Silencing-Dynamik; Daher ist ein Mangel an solchen Informationen eine große Trennung, die eine genaue Vorhersage der Dosis-Antwort in RNAi 21 ausschließt . Die Auseinandersetzung mit dieser Herausforderung würde die Formulierung der entsprechenden Struktur-Funktions - Beziehungen in Nanocarriern beschleunigen und besser informieren Biomaterial 22 - Design. Darüber hinaus würden solche Ansätze die Entwicklung von effektiveren siRNA-Dosierungsprotokollen ermöglichen. In einem Versuch, die dynamische Stummschaltung zu verstehen, haben mehrere Gruppen mathematische Modelle von RNAi 23 , 24 , 25 untersucht . Diese Rahmenbedingungen warenErfolgreich bei der Bereitstellung von Einsichten in siRNA-vermittelte Veränderungen in der Genexpression und zur Ermittlung der Geschwindigkeitsbegrenzungsschritte 26 . Diese Modelle wurden jedoch nur auf kommerzielle Genabgabesysteme ( z. B. Lipofectamin und Polyethylenimin (PEI)) angewendet, die nicht in der Lage sind, eine siRNA-Freisetzung zu kontrollieren, und die Komplexität der Modelle hat ihre Nützlichkeit stark eingeschränkt 27 . Diese Mängel unterstreichen einen unerfüllten Bedarf an neuen Materialien, die in der Lage sind, eine genau abstimmbare siRNA-Freisetzung zu kombinieren, kombiniert mit stromlinienförmigen und einfach zu bedienenden prädiktiven kinetischen Modellen.
Unsere Methode adressiert alle diese Herausforderungen durch die Integration einer lichtempfindlichen Nanocarrier-Plattform mit gekoppelten Methoden zur Quantifizierung der freien siRNA und Modell-RNAi-Dynamik. Insbesondere wird die präzise kontrollierte siRNA-Freigabe 28 unserer Plattform durch zwei komplementäre Methoden zur genauen Quantifizierung von verkapselten vs. un überwachtGebundene siRNA. Die experimentellen Daten aus diesen Tests werden in einem einfachen kinetischen Modell eingegeben gene silencing Effizienzen a priori 29 vorherzusagen. Schließlich wird die On / Off-Natur der Nanocarrier leicht ausgenutzt, um Zellmuster in der Genexpression mit räumlicher Kontrolle auf der zellulären Längenskala zu erzeugen. Somit bietet dieses Verfahren ein leicht anpassbares Verfahren zur Steuerung und Vorhersage von Gen-Silencing in einer Vielzahl von Anwendungen, die von einer räumlich-zeitlichen Regulierung des Zellverhaltens profitieren würden.
Protokoll
1. Formulierung von siRNA-Nanocarriern
- Vorbereitung von separaten Lösungen von siRNA und mPEG- b -P (APNBMA) mit gleichen Volumina, verdünnt in 20 mM 4- (2-Hydroxyethyl) piperazin-1-ethansulfonsäure (HEPES) -Puffer bei pH 6,0.
- SiRNA in einer Konzentration von 32 μg / ml bis 20 mM HEPES-Lösung zugeben.
HINWEIS: Die siRNA war eine nicht zielgerichtete, universelle negative Kontrollsequenz; Jedoch kann die siRNA entworfen werden, um jedes Gen von Interesse zu zielen. - MPEG- b -P (APNBMA) -Polymere in eine 20 mM HEPES-Lösung auflösen. Fügen Sie eine geeignete Menge an mPEG- b -P (APNBMA) hinzu, um eine 220 μg / ml Lösung herzustellen, so dass das N / P-Verhältnis (N, Amin-Gruppen an mPEG- b -P (APNBMA), P, Phosphatgruppen an siRNA) Ist 4
ANMERKUNG: Das Syntheseprotokoll für die MPEG- b -P (APNBMA) Polymere wird an anderer Stelle 30 angegeben.
- SiRNA in einer Konzentration von 32 μg / ml bis 20 mM HEPES-Lösung zugeben.
- Füge die mPEG- b -P (APNBMA) Lösung tropfenweise zu einem gleichen Volumen hinzuE der siRNA-Lösung beim sanften Mischen auf einer Wirbelmaschine. Weiter, um für 30 s nach der Polymerzugabe zu verwirbeln. Inkubieren Sie die Proben im Dunkeln bei Raumtemperatur für 30 min.
2. Messung der siRNA-Freisetzung mittels Gel-Elektrophorese
- Formulieren Sie die Nanocarrier nach den Schritten 1.1-1.2 und skalieren Sie die Volumina, um die Anzahl der gewünschten Proben zu berücksichtigen.
- Mischen Sie den Nanocarrier mit Natriumdodecylsulfat (SDS).
- Eine 1 mg / ml Lösung von SDS in Wasser vorbereiten. Aliquotierung der Menge an SDS-Lösung, die benötigt wird, um Lösungen mit einem S / P-Verhältnis (S, Sulfatgruppen auf SDS, P, Phosphatgruppen auf siRNA) von 15 zu erzeugen.
HINWEIS: Wenn die Polyplexlösung 1 μg siRNA enthält, müssen 13 μg SDS zugegeben werden, um ein S / P-Verhältnis von 15 zu erreichen. - Fügen Sie die SDS-Lösung jeder Nanocarrier-Lösung tropfenweise hinzu, während Sie sanft auf einer Wirbelmaschine mischen. Weiter, um für 30 s nach der SDS-Addition zu verwirren.
- Die Proben bei 5000 xg für 5 s zentrifugieren. Inkubieren Sie die Proben im Dunkeln bei Raumtemperatur für 30 min.
- Eine 1 mg / ml Lösung von SDS in Wasser vorbereiten. Aliquotierung der Menge an SDS-Lösung, die benötigt wird, um Lösungen mit einem S / P-Verhältnis (S, Sulfatgruppen auf SDS, P, Phosphatgruppen auf siRNA) von 15 zu erzeugen.
- Kalibrieren und setzen Sie einen UV-Laser mit einem 365 nm Filter auf eine Intensität von 200 W / m. Stellen Sie sicher, dass die Lichtintensität von der Stelle gemessen wird, an der der Boden der Probenlösung sitzt.
- Laden Sie die Nanocarrier / SDS-Lösung in eine Glaskammer, die aus Glasscheiben besteht, die durch eine Gummidichtung getrennt sind.
- Vorspülen von Glasrutschen in einer 7: 3 (v / v) Ethanol / Wasser-Lösung in Wasser und trocknen vollständig. Schneiden Sie ein Loch (~ 2 x 3 cm Rechteck) in eine Gummidichtung. Die Gummidichtung auf einen Glasschieber legen.
- Die Nanocarrier / SDS-Lösung auf den Glasschieber im Loch der Gummidichtung pipettieren. Laden Sie einen Überschuss an Lösung (20 μl Überschuss) auf den Glasschieber und vermeiden Sie den Kontakt mit der Gummidichtung.
HINWEIS: Bei den nachfolgenden Schritten wird etwas Flüssigkeit verloren. - Lege das zweite Glas aufAuf die Gleitdichtung aufschieben. Um eine Luftblasenerzeugung zu vermeiden, legen Sie zuerst ein Ende des Schiebers nach unten und senken dann langsam das andere Ende ab.
- Befestigen Sie Binder Clips an jeder Seite der Glaskammer, um es geschlossen zu halten.
- Bestrahlen Sie die Proben für die gewünschte Zeitdauer ( zB 0-60 min) mit dem UV-Laser mit einem 365 nm-Filter bei einer Intensität von 200 W / m. Entfernen Sie die Binder-Clips und öffnen Sie die Kammer.
- 25 μl der bestrahlten Nanocarrier / SDS Proben in ein Mikrozentrifugenröhrchen pipettieren. Inkubieren Sie die Lösungen im Dunkeln bei Raumtemperatur für 30 min.
- Bereiten Sie ein 2 Gew .-% Agarosegel vor, das mit 0,5 μg / ml Ethidiumbromid in Tris / Borat / EDTA (TBE) gepufferter Lösung gemäß Standardprotokollen 31 vorgefärbt wurde. Vorbereiten eines Beladungspuffers aus 3: 7 (v / v) Glycerin / Wasser.
- Füge 5 μl der Ladungspufferlösung zu 25 μl jeder Nanocarrier / SDS Probe hinzu. Inkubieren Sie die Proben im Dunkeln anRaumtemperatur für 10 min.
- Laden Sie 30 μl jeder Nanocarrier / SDS-Probe in das 2% Agarosegel. Führen Sie das Gel im Dunkeln bei 100 V für 30 min. Bild das Gel mit einem Gelbildsystem mit Ethidiumbromidfiltern. Speichern Sie die Gel-Bilddateien und fahren Sie mit Schritt 2.10 für die Bandintensitätsquantifizierung fort. Stellen Sie sicher, dass die Bandintensitäten hell genug sind, um deutlich zu visualisieren, aber nicht zu hell, dass die Signale gesättigt sind.
- Quantifizierung der Bandintensitäten mit öffentlich zugänglichen ImageJ Software 32 .
- Mit dem ROI-Tool von ImageJ bestimmen Sie die Fluoreszenzintensität der freien siRNA-Banden in jeder Spur, indem Sie ein Rechteck um jedes Band ziehen. Zeichnen Sie die Intensitätskurven jeder Spur und integrieren Sie den Bereich unter den Kurven, indem Sie eine horizontale Linie über die Intensitätskurven ziehen und auf den Spurstab in den umschlossenen Bereichen klicken.
- Berechnen Sie die relative Intensität jeder Spur, indem Sie die Fläche unter dem cu teilenRve jeder Probe durch die Fläche unter der Kurve der siRNA-Positivkontrolle (kein mPEG- b -P (APNBMA) hinzugefügt und kein SDS hinzugefügt). Melden Sie den Prozentsatz der siRNA, die als normalisierte Bandintensität jeder Probe freigegeben wurde.
3. Messung der siRNA-Freisetzung mittels Fluoreszenz-Korrelationsspektroskopie (FCS)
- Erhalten Sie siRNA, die mit einem einzigen Fluorophor am 5'-Ende des Sense-Strangs markiert ist.
HINWEIS: Die siRNA kann mit den Etiketten, die an der gewünschten Stelle konjugiert sind, vorgeglüht werden. Der Fluorophor sollte photostabil sein und zwischen 450 und 750 nm absorbieren / emittieren, um UV-Licht-Quenching und Energieübertragung mit mPEG- b- AP (APNBMA) zu vermeiden. - Formulieren Sie die Nanocarrier nach den Schritten 1.1-1.2 unter Verwendung der markierten siRNA. Skalieren Sie die Volumes, um die Anzahl der gewünschten Proben zu berücksichtigen.
- Inkubieren Sie die Lösungen in SDS und bestrahlen Sie für die gewünschte Zeitdauer gemäß den Schritten 2.2-2.6.
- VorbereitungAtion der FCS-Probenkammer.
- Waschen Sie eine Glasrutsche mit einer 7: 3 (v / v) Ethanol / Wasser-Lösung und trocknen Sie das Glas vollständig mit einem Wischtuch und einem Luftstrom.
- Entfernen Sie die Papierstücke von einem doppelseitigen Klebstoff-Abstandshalter, um den doppelseitigen Klebstoff-Abstandshalter freizulegen. Befestigen Sie den Abstandshalter an einem Glasdeckel.
- Die Nanocarrier / SDS-Lösung auf den Deckel in der Mitte des Loches aus dem Klebstoff-Abstandshalter pipettieren.
- Legen Sie den Glasschieber auf das Deckglas. Schieben Sie auf den Glasschieber, um sicherzustellen, dass der Glasschlitten und das Deckglas gut befestigt sind und eine Dichtung bilden.
- Verwenden Sie ein konfokales Mikroskop für FCS Messungen 33 . Verwenden Sie ein 40X Wasser Immersion Apochromat Objektiv mit einer numerischen Apertur von 1,2. Verwenden Sie den entsprechenden Anregungslaser-Kanal (488 nm), um mindestens 30 Messungen von jeweils 10 s pro Probe 34 zu sammeln. Stellen Sie sicher, dass die Laserintensität und die Detektorausrichtung verbleibenDas gleiche für jede Probe.
- Zusätzlich zu den experimentellen Proben, messen Kontrollen einschließlich: eine leere Probe ohne markierte siRNA; Und eine freie siRNA-Probe mit markierter siRNA, aber kein mPEG- b -P (APNBMA).
- Analysieren Sie die Daten mit der FCS-spezifischen Software. Identifizieren Sie die Baseline-Zählrate jeder Probe, indem Sie die stabile Zählrate während einer Zeit bestimmen, in der keine Nanocarrier durch das konfokale Volumen 29 hindurchgehen.
- Subtrahieren Sie die Zählrate der Blindprobe von jedem Sample-Baseline-Zählwertwert. Normalisieren der sich ergebenden Werte auf die freie siRNA Steuerung den Prozentsatz an freiem siRNA 35 zu berechnen.
4. Kinetische Modellierung zur Vorhersage von Gen-Silencing
- Erstellen Sie Skripte in einer mathematischen Programmiersprache mit dem einfachen Satz von gewöhnlichen Differentialgleichungen, um Gen Silencing 2 vorherzusagen9
HINWEIS: Skripte können auf Anfrage zur Verfügung gestellt werden.- Schreiben Sie die Menge der gewöhnlichen Differentialgleichungen als:
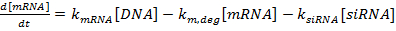 (1)
(1)  (2)
(2) 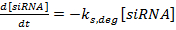 (3)
(3)
HINWEIS: Für die Gleichungen 1-3 sind die Ausdrücke k mRNA , k siRNA und k prot die Geschwindigkeitskonstanten für die Produktion von mRNA, siRNA und Protein. Die Terme k m, deg , k s, deg und k p, deg sind die Geschwindigkeitskonstanten für den Abbau von mRNA, siRNA bzw. Protein. Abbaugeschwindigkeitskonstanten werden auf der Basis der Komponentenhalbwertszeiten berechnet, und die Produktionsratenkonstanten sind geeignet, um sicherzustellen, dass mRNA- und Protein-Steady-State-Werte in Abwesenheit o erreicht werdenF siRNA- Bestimmen Sie die Halbwertszeiten der mRNA und des Proteins für das / die Gen (e) von Interesse, entweder experimentell, wie in Referenz 36 oder aus der Literatur beschrieben (siehe Diskussion ). Bestimmen Sie auch die Verdopplungszeit für die Zelllinie. Geben Sie diese Werte in die entsprechenden Degradationsratenausdrücke ein.
- Tune die Produktionsratenkonstanten, so dass die Genexpressionsniveaus bei einem normalisierten Wert von 100 stabil bleiben, wenn keine siRNA eingeführt wird. Speziell setzen Sie [siRNA] auf Null und variieren die Werte der k mRNA , k siRNA und k prot Produktionsgeschwindigkeitskonstanten, bis [mRNA] und [Protein] innerhalb von 1% des anfänglichen normierten Wertes von 100% für die Dauer von Die Simulation.
- Unter Verwendung der relativen Mengen an siRNA, die aus den zuvor beschriebenen Gelelektrophorese- und FCS-Assays als Schätzungen freigesetzt wurden, die anfängliche relative Konzentration von siRNA in dem Skript anzupassen. Speziell variiere [siRNA] proportional zur relativen Menge der freigesetzten siRNA mit einem Wert von 100, der der maximalen Menge 29 entspricht .
- Schreiben Sie die Menge der gewöhnlichen Differentialgleichungen als:
5. Zellkultur und in vitro siRNA Lieferung
- Kultur NIH / 3T3 murine embryonale Fibroblasten nach den Protokollen des Lieferanten.
- Wachsen Sie die Zellen in Wachstumsmedium (Dulbecco's Modified Eagle Medium (DMEM), ergänzt mit 10% hitzeinaktiviertem fötalem Rinderserum und 1% Penicillin-Streptomycin). Halten Sie die Zellen bei 37 ° C in einer befeuchteten Umgebung mit 5% CO 2 .
- Samen Sie die Zellen in 6-Well-Gewebekultur-behandelten Platten.
- Befolgen Sie das empfohlene Unterkulturverfahren vom Lieferanten. Zähle die Zellen mit einem Hämocytometer. Die Zellen in ergänzten Wachstumsmedien auf eine Konzentration von 75.000 Zellen / ml verdünnen.
- Füge 2 ml Zellsuspension hinzu (75.000 ZelleS / ml) zu jeder Vertiefung der 6-Well-Platte. Lassen Sie die Zellen haften und sich für 24 h im Inkubator erholen.
- Die Zellen für die Transfektion durch Waschen mit phosphatgepufferter Kochsalzlösung (PBS) vorbereiten und 1,5 ml Serum- und Antibiotikum-freies Transfektionsmedium (siehe Tabelle der Materialien ) zu jeder Vertiefung zugeben.
- Formulieren Sie die siRNA-Nanocarrier nach den Schritten 1.1-1.2. Füge 25 μl Nanocarrier-Lösung mit 30 pmol siRNA zu jeder Vertiefung hinzu. Legen Sie das Medium vorsichtig nach oben und unten ab, um es zu mischen. Legen Sie die Zellen 3 Stunden lang in den Inkubator.
- Entfernen Sie die Transfektionsmedien und waschen Sie sich mit PBS. Füge 1 ml ergänztes Wachstumsmedium hinzu und lege die Zellen in den Inkubator, um 30 min zu gewinnen.
- Zur Vorbereitung der Zellen zur Behandlung mit einem Photo-Stimulus, entfernen Sie die ergänzten Wachstumsmedien. Füge 1 ml Transfektionsmedium (ohne Phenolrot) zu jeder Vertiefung hinzu.
HINWEIS: Sicherstellen, dass das Transfektionsmedium kein Phenolrot enthält. - Kalibrieren und setzen Sie einen UV-Laser mit einem 365 nm Filter auf eine Intensität von 200 W / m. Stellen Sie sicher, dass die Lichtintensität von der Stelle gemessen wird, an der die Unterseite der Zellplatte sitzt.
- Legen Sie die Zellen auf eine heiße Platte auf 37 ° C. Entfernen Sie die Plattenabdeckung der Zellen. Bestrahlen Sie die Zellen von der Platte über die gewünschte Zeit (bis zu 20 min) mit dem UV-Laser mit einem 365 nm-Filter bei einer Intensität von 200 W m -2 .
- Entfernen Sie die Transfektionsmedien und fügen Sie 2 ml ergänztes Wachstumsmedium hinzu. In den Inkubator stellen bis zur weiteren Analyse ( zB 24 h für qPCR und 48 h für Western Blotting).
- Messen Sie Veränderungen in der Genexpression unter Verwendung einer Vielzahl von Techniken wie Western Blotting 37 und qPCR. 38 Für Gene mit sichtbaren Signalen wie GFP verwenden Sie die Fluoreszenzmikroskopie 29 .
HINWEIS: Diese Techniken werden aufgrund ihrer Benutzerfreundlichkeit und Genauigkeit vorgeschlagenBei der Quantifizierung der Genexpression
- Messen Sie Veränderungen in der Genexpression unter Verwendung einer Vielzahl von Techniken wie Western Blotting 37 und qPCR. 38 Für Gene mit sichtbaren Signalen wie GFP verwenden Sie die Fluoreszenzmikroskopie 29 .
6. Kontrolle des Gen-Silens in einer räumlich-zeitlichen Manner
- Kultur, Samen und transfizierende Zellen nach den Schritten 5.1-5.7.
- Bereiten Sie eine Photomaske vor, die 365 nm Licht vollständig blockiert und Reflexionen minimiert.
HINWEIS: In diesem Fall wurden 10 x 10 cm Stück Aluminiumfolie und schwarzes Baupapier verwendet, um das Licht zu blockieren und um Reflexionen zu reduzieren. Die Aluminiumfolie und das Baupapier wurden zu einer Einheit zusammengeklebt.- Schneiden / Stanzen / Maschine die gewünschte Form in die Fotomaske. Verwenden Sie zum Beispiel eine scharfkantige Klinge und einen Locher, um ein geradliniges Muster (~ 5 cm lang) und ein kreisförmiges Muster (~ 7 mm Durchmesser) in der Photomaske zu bilden.
- Kleben Sie die Photomaske auf den Boden der 6-Well-Platte, wobei das Muster unter dem Brunnen zentriert ist, der die Zellen mit der Antireflexseite enthält ( z. B. schwarz cAuflagepapier). Achten Sie darauf, dass der Leim nicht in der Nähe der Kante (innerhalb ~ 3 mm) des Musters platziert wird.
- Richten Sie zwei Ringständer ca. 25 cm auseinander und befestigen Sie eine Plattform an jedem Ringständer, so dass die Plattformen gleich groß sind. Hängen Sie die Zellplatte zwischen den beiden Ständern auf, indem Sie die Platte auf die Plattform stellen. Stellen Sie sicher, dass die Platte eben ist.
- Bestrahlen Sie die Zellen unterhalb der Probe für die gewünschte Zeit (bis zu 20 min) mit dem UV-Laser mit einem 365 nm-Filter bei einer Intensität von 200 W / m 2 .
- Entfernen Sie die Transfektionsmedien und fügen Sie 2 ml ergänztes Wachstumsmedium hinzu. In den Inkubator stellen, um mindestens 24 Stunden lang zu erholen. Bild die Zellen mit Fluoreszenzmikroskopie wie beschrieben 29 .
Ergebnisse
Nach der Formulierung der Nanocarrier wurden siRNA-Freisetzungsassays durchgeführt, um die Bestrahlungsbedingungen, die in den in vitro- Transfektionen verwendet werden sollen, zu informieren. Es wurden verschiedene Lichtdosierungen angewendet, um den Prozentsatz der freigesetzten siRNA zu bestimmen. Der erste Assay verwendete die Gelelektrophorese, um die freien siRNA-Moleküle von den noch komplexen / assoziierten siRNA-Molekülen zu trennen. Nanocarriers, die nicht mit Licht...
Diskussion
Es gibt ein paar Schritte in der Methode, die besonders kritisch sind. Bei der Formulierung der Nanocarrier sind die Reihenfolge der Komponentenaddition und der Mischgeschwindigkeit zwei wichtige Einflussfaktoren für die Wirksamkeit 39 . Dieses Protokoll erfordert, dass die kationische Komponente, mPEG- b -P (APNBMA), der anionischen Komponente, siRNA, tropfenweise unter Vortexen zugesetzt wird. Je nach Formulierungsvolumen dauert dieser Mischvorgang 3-6 s. Um zu testen, ob die Nanocarr...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren danken dem Nationalen Institut für Allgemeine Medizinische Wissenschaften der National Institutes of Health (NIH) für die finanzielle Unterstützung durch einen Institutional Development Award (IDeA) unter der Nummer P20GM103541 sowie der Nummer P20GM10344615. Die hierin enthaltenen Aussagen spiegeln nicht die Ansichten des NIH wider. Wir bestätigen auch das Delaware Biotechnology Institute (DBI) und das Delaware Economic Development Office (DEDO) für die finanzielle Unterstützung durch den Bioscience Center for Advanced Technology (Bioscience CAT) Award (12A00448).
Materialien
| Name | Company | Catalog Number | Comments |
| siRNA | Sigma-Aldrich | SIC001 | non-targeted, universal negative control |
| mPEG-b-P(APNBMA) | synthesized in our lab | N/A | photo-responsive polymer |
| HEPES | Fisher Scientific | BP310-100 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 436143 | |
| rubber gasket | McMaster-Carr | 3788T21 | 0.5 mL thick |
| UV laser | Excelitas Technologies | Omnicure S2000 | collimating lens and 365 nm filter used |
| agarose | Fisher Scientific | BP160-100 | |
| ethidium bromide | Fisher Scientific | BP1302-10 | |
| siRNA labelled with Dy547 | GE Healthcare Dharmacon, Inc. | custom order | fluorophore conjugated to 5’ end of sense strand |
| microscope slide | Fisher Scientific | 12-550-A3 | pre-cleaned glass |
| Secure-Seal Spacer | Life Technologies | S24735 | double-sided adhesive |
| LSM 780 | Carl Zeiss | N/A | confocal microscope |
| ZEN 2010 | Carl Zeiss | N/A | FCS analysis software |
| MATLAB | MathWorks | N/A | programming language |
| NIH/3T3 cells | ATCC | ATCC CRL-1658 | |
| DMEM | Mediatech | 10-013-CV | growth media |
| fetal bovine serum | Mediatech | 35-011-CV | heat-inactivated |
| penicillin-streptomycin | Mediatech | 30-002-CI | |
| 6-well plates | Fisher Scientific | 08-772-1B | |
| Opti-MEM | Life Technologies | 11058021 | transfection media |
Referenzen
- Forbes, D. C., Peppas, N. A. Oral delivery of small RNA and DNA. J Control Release. 162 (2), 438-445 (2012).
- Davis, M. E., et al. Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles. Nature. 464 (7291), 1067-1070 (2010).
- Bouchie, A. Companies in footrace to deliver RNAi. Nat Biotechnol. 30 (12), 1154-1157 (2012).
- Burke, P. A., Pun, S. H., Reineke, T. M. Advancing Polymeric Delivery Systems Amidst a Nucleic Acid Therapy Renaissance. ACS Macro Lett. 2 (10), 928-934 (2013).
- Gooding, M., Browne, L. P., Quinteiro, F. M., Selwood, D. L. siRNA Delivery: From Lipids to Cell-penetrating Peptides and Their Mimics. Chem Biol Drug Des. 80 (6), 787-809 (2012).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nat Chem Biol. 10 (3), 196-202 (2014).
- Wang, X., Chen, X., Yang, Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods. 9 (3), 266-269 (2012).
- Ziauddin, J., Sabatini, D. M. Microarrays of cells expressing defined cDNAs. Nature. 411 (6833), 107-110 (2001).
- Saltzman, W. M., Olbricht, W. L. Building drug delivery into tissue engineering. Nat Rev Drug Discov. 1 (3), 177-186 (2002).
- Mura, S., Nicolas, J., Couvreur, P. Stimuli-responsive nanocarriers for drug delivery. Nat Mater. 12 (11), 991-1003 (2013).
- Shim, M. S., Kwon, Y. J. Stimuli-responsive polymers and nanomaterials for gene delivery and imaging applications. Adv Drug Delivery Rev. 64 (11), 1046-1058 (2012).
- Kelley, E. G., Albert, J. N. L., Sullivan, M. O., Epps, T. H. Stimuli-responsive copolymer solution and surface assemblies for biomedical applications. Chem Soc Rev. 42 (17), 7057-7071 (2013).
- Weber, W., Fussenegger, M. Emerging biomedical applications of synthetic biology. Nat Rev Genet. 13 (1), 21-35 (2012).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Shimizu-Sato, S., Huq, E., Tepperman, J. M., Quail, P. H. A light-switchable gene promoter system. Nat Biotechnol. 20 (10), 1041-1044 (2002).
- Huschka, R., et al. Gene Silencing by Gold Nanoshell-Mediated Delivery and Laser-Triggered Release of Antisense Oligonucleotide and siRNA. ACS Nano. 6 (9), 7681-7691 (2012).
- Li, H. -. J., Wang, H. -. X., Sun, C. -. Y., Du, J. -. Z., Wang, J. Shell-detachable nanoparticles based on a light-responsive amphiphile for enhanced siRNA delivery. R Soc Chem Adv. 4 (4), 1961-1964 (2014).
- Braun, G. B., et al. Laser-Activated Gene Silencing via Gold Nanoshell-siRNA Conjugates. ACS Nano. 3 (7), 2007-2015 (2009).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nat Biotechnol. 31 (7), 638-646 (2013).
- Wittrup, A., et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown. Nat Biotechnol. 33 (8), 870-876 (2015).
- Roth, C. M. Quantitative measurements and rational materials design for intracellular delivery of oligonucleotides. Biotechnol Prog. 24 (1), 23-28 (2008).
- Mao, S., et al. Influence of polyethylene glycol chain length on the physicochemical and biological properties of poly(ethylene imine)-graft-poly(ethylene glycol) block copolymer/SiRNA polyplexes. Bioconjugate Chem. 17 (5), 1209-1218 (2006).
- Raab, R. M., Stephanopoulos, G. Dynamics of gene silencing by RNA interference. Biotechnol Bioeng. 88 (1), 121-132 (2004).
- Cuccato, G., et al. Modeling RNA interference in mammalian cells. BMC Systems Biology. 5, 1 (2011).
- Varga, C. M., Hong, K., Lauffenburger, D. A. Quantitative analysis of synthetic gene delivery vector design properties. Mol Ther. 4 (5), 438-446 (2001).
- Chen, H. H., et al. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum Dot-FRET. Mol Ther. 16 (2), 324-332 (2008).
- Bartlett, D. W., Davis, M. E. Insights into the kinetics of siRNA-mediated gene silencing from live-cell and live-animal bioluminescent imaging. Nucleic Acids Res. 34 (1), 322-333 (2006).
- Foster, A. A., Greco, C. T., Green, M. D., Epps, T. H., Sullivan, M. O. Light-Mediated Activation of siRNA Release in Diblock Copolymer Assemblies for Controlled Gene Silencing. Adv Healthc Mater. 4 (5), 760-770 (2015).
- Greco, C. T., Epps, T. H., Sullivan, M. O. Mechanistic Design of Polymer Nanocarriers to Spatiotemporally Control Gene Silencing. ACS Biomater Sci Eng. 2 (9), 1582-1594 (2016).
- Green, M. D., et al. Catch and release: photocleavable cationic diblock copolymers as a potential platform for nucleic acid delivery. Polym Chem. 5 (19), 5535-5541 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Marquer, C., Leveque-Fort, S., Potier, M. C. Determination of Lipid Raft Partitioning of Fluorescently-tagged Probes in Living Cells by Fluorescence Correlation Spectroscopy (FCS). J Vis Exp. (62), (2012).
- Staaf, E., Bagawath-Singh, S., Johansson, S. Molecular Diffusion in Plasma Membranes of Primary Lymphocytes Measured by Fluorescence Correlation Spectroscopy. J Vis Exp. (120), e54756 (2017).
- Buyens, K., et al. Monitoring the disassembly of siRNA polyplexes in serum is crucial for predicting their biological efficacy. J Control Release. 141 (1), 38-41 (2010).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc Natl Acad. Sci U S A. 76 (9), 4350-4354 (1979).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous Fluorescence Monitoring of Rapid Cycle DNA Amplification. Biotechniques. 54 (6), 314-320 (2013).
- Cho, S. K., Dang, C., Wang, X., Ragan, R., Kwon, Y. J. Mixing-sequence-dependent nucleic acid complexation and gene transfer efficiency by polyethylenimine. Biomater Sci. 3 (7), 1124-1133 (2015).
- Liu, Y. M., Reineke, T. M. Poly(glycoamidoamine)s for gene delivery: Stability of polyplexes and efficacy with cardiomyoblast cells. Bioconjugate Chem. 17 (1), 101-108 (2006).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Greco, C. T., Muir, V. G., Epps, T. H., Sullivan, M. O. Efficient tuning of siRNA dose response by combining mixed polymer nanocarriers with simple kinetic modeling. Acta Biomater. 50, 407-416 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten