Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Lichtvermittelte reversible Modulation des mitogenaktivierten Proteinkinase-Pathways während der Zelldifferenzierung und
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine optogenetische Strategie zur Modulation der mitogenaktivierten Proteinkinase (MAPK) Aktivität während der Zelldifferenzierung und der Xenopus embryonalen Entwicklung. Diese Methode ermöglicht die reversible Aktivierung des MAPK-Signalwegs in der Säugerzellkultur und in multizellulären Lebendorganismen wie Xenopus- Embryonen mit hoher räumlicher und zeitlicher Auflösung.
Zusammenfassung
Kinase-Aktivität ist entscheidend für eine Fülle von zellulären Funktionen, einschließlich Zellproliferation, Differenzierung, Migration und Apoptose. Während der frühen embryonalen Entwicklung ist die Kinase-Aktivität hochdynamisch und weit verbreitet im Embryo. Pharmakologische und genetische Ansätze werden häufig verwendet, um Kinaseaktivitäten zu untersuchen. Leider ist es schwierig, mit diesen Strategien eine überlegene räumliche und zeitliche Auflösung zu erreichen. Weiterhin ist es nicht möglich, die Kinaseaktivität reversibel in lebenden Zellen und multizellulären Organismen zu kontrollieren. Eine solche Einschränkung bleibt ein Engpass für ein quantitatives Verständnis der Kinaseaktivität während der Entwicklung und Differenzierung. Diese Arbeit präsentiert eine optogenetische Strategie, die ein bicistronisches System mit photoaktivierbaren Proteinen Arabidopsis thaliana cryptochrome 2 (CRY2) und der N-terminalen Domäne von Cryptochrom-interagierenden Basis-Helix-Loop-Helix (CIBN) nutzt. ReversiDie Aktivierung des mitogen-aktivierten Proteinkinase- (MAPK-) Signalisierungsweges wird durch lichtvermittelte Proteintranslokation in lebenden Zellen erreicht. Dieser Ansatz kann auf Säugetierzellkulturen angewendet werden und lebende Wirbeltierembryonen. Dieses bicistronische System kann verallgemeinert werden, um die Aktivität anderer Kinasen mit ähnlichen Aktivierungsmechanismen zu kontrollieren und kann auf andere Modellsysteme angewendet werden.
Einleitung
Wachstumsfaktoren sind an einem breiten Spektrum von Zellfunktionen beteiligt, darunter Proliferation, Differenzierung, Migration und Apoptose und spielen in vielen biologischen Ereignissen, einschließlich embryonaler Entwicklung, Alterung und Regulierung des mentalen Status 1 , 2 , 3 , 4 , 5 Viele Wachstumsfaktoren signalisieren durch komplexe intrazelluläre Signalkaskaden. Diese Signalisierungsereignisse werden oft durch reversible Proteinphosphorylierung präzise geregelt 6 , 7 betrieben. So ist ein Verständnis der Signalisierungsergebnisse von Proteinkinasen, die für die Proteinphosphorylierung verantwortlich sind, von grundlegender Bedeutung.
Unterschiedliche Wachstumsfaktoren wirken durch ein relativ häufiges intrazelluläres Signalisierungsnetzwerk, obwohl sie dist anregenInct zelluläre Reaktionen 8 , 9 . Häufige intrazelluläre Mediatoren von Rezeptortyrosinkinasen umfassen Ras, Raf, extrazelluläre signalregulierte Kinase (ERK), mitogenaktivierte Proteinkinase (MAPK) / ERK-Kinase (MEK), Phosphoinositid-3-kinase (PI3K), Akt und Phospholipase C gamma (PLC & gamma;) 10 , 11 . Die akkumulierende Evidenz deutet darauf hin, dass die Signalisierungsvielfalt und die Spezifität von der räumlichen und zeitlichen Regulierung der Signalisierungsaktivität abhängen. Zum Beispiel aktiviert bei Ratten-Pheochromozytomzellen (PC12) die epidermale Wachstumsfaktor (EGF) -Stimulation, die zu einer Zellproliferation führt, den ERK-Weg 9 vorübergehend. Auf der anderen Seite aktiviert die Stimulation mit dem Nervenwachstumsfaktor (NGF), die zur Zelldifferenzierung führt, den ERK-Weg nachhaltig 9 , 13 . In kultivierten rBei hippocampalen Neuronen fördert die transiente Signalisierung durch den vom Gehirn abgeleiteten neurotrophischen Faktor (BDNF) das primäre Neuritenwachstum, während die anhaltende Signalisierung zu einer erhöhten Neuritenverzweigung führt. Während der frühen embryonalen Entwicklung ist die phosphorylierte ERK-Aktivität zeitlich dynamisch und weit verbreitet im Embryo 6 . Ein aktueller genetischer Screen während der frühen Xenopus- Embryogenese zeigte, dass ERK- und Akt-Signalkaskaden, zwei nachgeschaltete primäre Wachstumsfaktorwege, bühnenspezifische Aktivierungsprofile zeigen 7 . So verlangt ein Verständnis der Kinase-Signalisierungsergebnisse Werkzeuge, die die räumlichen und zeitlichen Merkmale der Kinaseaktivität mit ausreichender Auflösung untersuchen können.
Konventionelle experimentelle Ansätze, die dynamische Natur der Signaltransduktion während der Entwicklung zu untersuchen, fehlt der wünschenswerten räumlichen und zeitlichen Auflösung. Zum Beispiel verwenden pharmakologische Ansätze kleine chemIcal oder biologischen Molekülen zur Stimulierung oder Unterdrückung der Signaltransduktion in Zellen und Geweben. Die diffuse Natur dieser kleinen Moleküle macht es schwierig, ihre Handlung auf eine bestimmte Region von Interesse zu beschränken 15 . Genetische Ansätze ( zB Transgenese, Cre-Lox-System oder Mutagenese) führen häufig zur irreversiblen Aktivierung oder Repression der Zielgenexpression bzw. Proteinaktivität 16 , 17 , 18 . Das Tet-On / Tet-Off-System 19 bietet eine verbesserte zeitliche Kontrolle der Gentranskription, aber es fehlt eine strikte räumliche Kontrolle, da es auf der Diffusion von Tetracyclin beruht. Jüngste Entwicklungen in der chemisch induzierten Proteindimerisierung 20 oder photo-uncaging 21 , 22 , 23 , 24 haben stark verbessertDie zeitliche Steuerung von Signalisierungsnetzwerken. Die räumliche Kontrolle bleibt jedoch aufgrund der diffusen Beschaffenheit der eingesperrten Chemikalien anspruchsvoll.
Jüngste aufkommende optogenetische Ansätze, die die Kraft des Lichts nutzen, um Protein-Protein-Wechselwirkungen zu kontrollieren, erlauben die Modulation von Signalwegen mit hoher räumlich-zeitlicher Präzision sowie Reversibilität. Kurz nach ihrem anfänglichen Erfolg bei der Kontrolle des neuronalen Brennens 25 , 26 , 27 wurde die Optogenetik erweitert, um andere zelluläre Prozesse wie die Gentranskription, Translation, Zellmigration, Differenzierung und Apoptose 28 , 29 , 30 , 31 , 32 , 33 zu kontrollieren , 34 . Eine Strategie mit dem pHotoaktivierbares Proteinpaar Arabidopsis thaliana cryptochrome 2 (CRY2) -Protein und die N-terminale Domäne von Cryptochrom-interagierenden Basic-Helix-Loop-Helix (CIBN) wurde kürzlich entwickelt, um die Raf1-Kinase-Aktivität in Säugetierzellen und Xenopus- Embryonen 35 zu kontrollieren. CRY2 bindet an CIBN bei Blue-Light-Stimulation, und der CRY2 / CIBN-Proteinkomplex dissoziiert spontan im Dunkeln 34 . Blaues Licht erregt den CRY2-Cofaktor, Flavin-Adenin-Dinukleotid (FAD), was zu einer Konformationsänderung in CRY2 und dessen anschließender Bindung an CIBN führt. Konstitutiv aktive (W374A) und Flavin-defiziente (D387A) -Mutanten von CRY2 können durch Mutationen in der FAD-Bindungstasche produziert werden : Die CRY2 W374A- Mutante bindet an CIBN unabhängig von Licht, während die CRY2 D387A- Mutante nicht an CIBN unter blau bindet Leichte Stimulation 36 , 37 . Das optogenetische System beschrieben iN dieses Protokoll verwendet Wildtyp-CRY2 und CIBN, um die Protein-Translokation-vermittelte Raf1-Aktivierung in lebenden Zellen zu induzieren. Es ist bekannt, dass die Membranrekrutierung von Raf1 seine Aktivität verstärkt. In diesem System ist ein Tandem-CIBN-Modul an der Plasmamembran verankert und CRY2-mCherry ist an den N-Terminus von Raf1 35 fusioniert. In Abwesenheit von blauem Licht bleibt CRY2-mCherry-Raf1 im Zytoplasma und Raf1 ist inaktiv. Die Blaulichtstimulation induziert die CRY2-CIBN-Bindung und rekrutiert Raf1 an die Plasmamembran, wo Raf1 aktiviert wird. Die Raf-Aktivierung stimuliert eine Raf / MEK / ERK-Signalkaskade. Sowohl CRY2- als auch CIBN-Fusionsproteine sind in einem bikistronischen genetischen System codiert. Diese Strategie kann verallgemeinert werden, um andere Kinasen, wie zB Akt, zu kontrollieren, deren Aktivierungszustand auch durch Protein-Translokation in Zellen 39 eingeschaltet werden kann. Diese Arbeit stellt detaillierte Protokolle für die Umsetzung dieser optogenetischen Strategie in Säugetierzellkultur darRes und multizellulären Organismen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Die Tierforschung wurde in Übereinstimmung mit den Richtlinien des Illinois Institutional Animal Care and Use Committee (IACUC) und der University of Illinois Department of Animal Resources (DAR) durchgeführt.
1. Optogenetische Induktion der Protein-Lokalisation in BHK21 Säugetierzellkultur
HINWEIS: Die Schritte 1.1-1.3 stellen eine Methode zur Zusammenstellung einer Zellkulturkammer zur Bildgebung mit hochvergrößerten Objektiven ( zB 63X oder 100X) zur Verfügung, die typischerweise kurze Arbeitsabstände aufweisen. Diese Ziele erfordern ein dünnes Glasdeckglas ( z . B. # 1,5, 170 μm Dicke) als das Abbildungssubstrat. Alternativ kann eine Glasboden-Zellkulturschale / -rutsche verwendet werden. In einem solchen Fall können die Schritte 1.1-1.3 übersprungen werden.
- Reinigung von Glasdeckgläschen
- Legen Sie 30 Glasdeckel in einen Deckglashalter.
- In einem 600 ml Plastikbecher werden 20 g Spülmittel und 400 ml warmes Leitungswasser zugegeben. PlacE das Becherglas auf einem Magnetrührer geben und rühren, bis alles Waschmittel aufgelöst ist. Entfernen Sie den Schaum mit Seidenpapier.
- Verwenden Sie den Boden einer 100 mm x 15 mm Petrischale als Abstandshalter aus dem Rührstab und als Oberfläche für den Deckelhalter. Um eine ausreichende Strömung während des Reinigungsvorgangs zu gewährleisten, 3-4 Entwässerungslöcher in der Petrischale herstellen.
- Um die abtropfenden Löcher zu machen, erwärmen Sie einen Lötkolben und brennen durch den Plastik in einer belüfteten Kapuze.
ACHTUNG: Beruhigen Sie den beheizten Lötkolben nicht mit bloßen Händen. - Legen Sie den Deckglashalter auf die Petrischale in den Becher. Rühre für 2 h über Nacht bei Raumtemperatur.
- Waschen Sie dreimal mit 500 ml DI-Wasser in einem 1000 ml-Becherglas. Die Waschmittellösung kann wiederverwendet werden.
- Mit Pinzetten einzelne Deckgläser aufnehmen. Tauchen Sie die Deckgläser in 190 ° C (95%) Ethanol für 1 min ein.
- Trocknen Sie die Deckgläser in einer sterilen Kapuze. Lagern Sie die Deckgläser in einem sterilen Plastikbehälter bis zum Gebrauch.
- Poly-L-Lysin (PLL) -beschichtete Deckgläser herstellen
- Mischen Sie 0,1 M Boratpuffer durch Lösen von 1,24 g Borsäure und 1,9 g Dinatriumtetraborat in 400 ml Wasser. Den pH-Wert auf 8,5 mit 10 M NaOH einstellen.
- PLL im Boratpuffer bis zu einer Endkonzentration von 1 mg / ml auflösen. Aliquotieren Sie die PLL-Lösung in 50-ml-sterilen Zentrifugenröhrchen.
- Füge 50 ml PLL-Beschichtungslösung zu einer 10-cm-Gewebekulturschale hinzu. Legen Sie die Schale in einen 37 ° C Inkubator, um die PLL-Lösung aufzuwärmen.
- Öffnen Sie den Deckglasbehälter (Schritt 1.1.8) in einer sterilen Kapuze.
- Tauchen Sie die gereinigten Deckgläser einzeln in die vorgewärmte PLL-Lösung in die Gewebekulturschale ein. Vermeiden Sie Blasenbildung, um alle Oberflächen eintauchen zu lassen.
- Legen Sie die Schale mit einem Deckel in einen 37 ° C CO 2 Inkubator über Nacht. Nehmen Sie jedes Deckglas heraus und legen Sie es in einen sterilen Deckglashalter. Spülen Sie dreimal mit autoklaviertem Reinstwasser (18,2 MΩ & #183, cm-Widerstand) in einem 1.000-ml-Becherglas in einer sterilen Kapuze.
- Trocknen Sie die Deckgläser im Deckglashalter in der Sterilhaube. Lagern Sie die Deckgläser in einem sterilen Plastikbehälter bis zum Gebrauch.
- Zusammenbau von Polydimethylsiloxan (PDMS) Zellkulturkammern
- In einem Plastikbehälter wiegen 40 g PDMS-Base und fügen 4 g Härtungsmittel hinzu, um ein 10: 1 W / W-Verhältnis von PDMS und Härtungsmittel zu erreichen. Mischen Sie das PDMS / Härtungsmittel durch Rühren mit einer Pipettenspitze für 2 min. Alternativ einen Zentrifugalmischer verwenden.
- Verwenden Sie einen Becher mit einem 4 Zoll Durchmesser als Schablone, um einen Halter mit einem Stück Aluminiumfolie zu machen. Legen Sie den Aluminiumfolienhalter in eine 15 cm Petrischale. Legen Sie einen 4-Zoll-Silizium-Wafer in diesen Aluminium-Folienhalter.
- Gießen Sie die gemischte PDMS-Lösung in den Halter. Verwenden Sie einen dünnen Glasstab, um den Rand des Wafers sanft zu berühren. Entfernen Sie alle Luftblasen, die unter dem Wafer gefangen sind.
- Setzen Sie die 15 cm Petrischale in eine Vakuum-Chambe einR, um die PDMS-Lösung zu entgasen. Fortsetzen Sie die Entgasung für ca. 20 min, bis keine Blasen erzeugt werden. In der Zwischenzeit einen Konvektionsofen auf 65 ° C vorheizen.
- Setzen Sie die 15 cm Petrischale vorsichtig in den Ofen. Inkubieren für 2 h.
- Entfernen Sie die Platte aus dem Ofen. Verwenden Sie einen Glasstab, um die Kante des PDMS vorsichtig zu berühren und eine vollständige Vernetzung zu gewährleisten. Wenn nicht, inkubieren Sie länger, bis das PDMS fest und nicht klebrig ist.
- Entfernen Sie die Aluminiumfolie von der Platte und schälen Sie sie vom Siliziumwafer ab. Starten Sie von der Kante vorsichtig das gehärtete PDMS vom Siliziumwafer ab.
ACHTUNG: Vermeiden Sie es, zu viel Kraft zu setzen, da es den Wafer brechen kann. - Trimmen Sie das PDMS in 20 mm x 30 mm rechteckige Stücke, um das 24 mm x 40 mm Deckglas mit einer Rasierklinge zu passen.
- Verwenden Sie eine meißelförmige Klinge, um eine rechteckige Öffnung von 10 mm x 20 mm von der Mitte jedes PDMS-Stückes zu schneiden.
- Reinigen Sie die PDMS-Kammern mit dem in Schritt 1.1 beschriebenen Waschmittelprotokoll.
- Die PDMS-Kammern in einer sauberen Kapuze trocknen. Legen Sie die trockenen Kammern in einen autoklavierbaren Behälter, der mit Aluminiumfolie bedeckt ist.
- Autoklavieren Sie den Behälter mit der Schwerkraft (121 ° C, 15 psi) für 30 min und lagern Sie den Behälter bei Raumtemperatur.
- BHK21 Zellkultivierung und Transfektion
- Mischen Sie 500 ml Medium für die BHK21-Zellkultur durch Mischen von 445 ml DMEM mit 50 ml FBS und 5 ml 100 × Penn-Strep-Glutamin (10 000 U / ml Penicillin, 10 mg / ml Streptomycin und 29,2 mg / ml L -Glutamin). Stellen Sie sicher, dass die Endkonzentration der FBS 10% beträgt.
- Aliquot 10 ml Nicht-HEPES-CO 2 -unabhängiges Medium für die Live-Zell-Bildgebung.
HINWEIS: CO 2 -unabhängiges Medium unterstützt das Zellwachstum ohne CO 2 -Inkubator und eignet sich ideal für die Bildgebung von Zellen unter atmosphärischen Bedingungen. Weitere Informationen finden Sie in der Materialliste. Zusätzlich zur BHK21-Zelllinie sollten auch andere Arten von Säugetierzellen gebenÄhnliche Ergebnisse. - Montieren Sie eine Zellkulturvorrichtung, indem Sie eine autoklavierte, sterile PDMS-Kammer (Schritt 1.3) auf ein steriles PLL-beschichtetes Deckglas (Schritt 1.2) in einer sterilen Kapuze legen.
- Legen Sie jedes Gerät in eine sterile 60 mm Petrischale.
- Verwenden Sie 0,5 ml 0,25% Trypsin, um die Zellen aus einer Vertiefung einer 12-Well-Gewebekulturplatte zu lösen. Zählen Sie die Zelldichte mit einem Hämocytometer. Plate 20.000 BHK21 Zellen in der Kammer (ca. 10.000 Zellen / cm 2 ) mit 200 μl Zellkulturmedium.
- Bereiten Sie DNA-Plasmid mit einem Plasmid-Präparationskit vor (siehe Materialliste).
HINWEIS: Das Design und die Funktionalität des CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX sind in Abbildung 1A - 1B dargestellt. - Vierundzwanzig (24) h nach der Zellplattierung, transfizieren die Zellen mit 50-100 ng CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX-Plasmid nach dem Protokoll des Herstellers.
- Drei (3) Stunden nach der Transfektion, ändern Sie die mEdium zu 200 μl frischem Kulturmedium (Schritt 1.4.1) und lassen die Zellen über Nacht durch Inkubieren der Kultur in einem 37 ° C, 5% CO 2 -Inkubator nachvollziehen.
- Fluoreszenz-Live-Zell-Bildgebung
- Schalten Sie einen Computer, eine Lichtquelle, ein Mikroskop und eine Kamera ein. Verwenden Sie für den Rest des Protokolls ein konfokales Fluoreszenzmikroskop (ausgestattet mit einem 100fachen Öl-Immersionsobjektiv).
HINWEIS: Ein invertiertes Epifluoreszenzmikroskop kann auch verwendet werden. - Ersetzen Sie das Zellkulturmedium durch 200 μl CO 2 -unabhängiges Medium.
- Richten Sie das Datenerfassungsprotokoll ein, bevor Sie die Zellen auf das Mikroskop stellen. Verwenden Sie 488-nm-Anregung FITC-Kanal für optogenetische Stimulation. Verwenden Sie 561 nm Anregungs-TRITC-Kanal, um transfizierte Zellen zu lokalisieren und die zelluläre Lokalisierung von mCherry-markiertem Protein zu verfolgen.
- Stellen Sie die Verstärkung der FITC- und TRITC-Kanäle als 120 bzw. 200 ein. Verwenden Sie eine Pixel-Verweilzeit von 2,21; s und Größe von 512 x 512 Pixeln.
- Messen Sie die Leistung des 488-nm-Lichts, indem Sie einen Leistungsmesser in die Nähe des Objektivfensters legen. Eine Gesamtleistung von 2 μW (ca. 10.000 W / cm 2 im Fokus) reicht aus, um die CIBN-CRY2PHR-Assoziation zu induzieren.
- Richten Sie eine Zeitstempelerfassung mit einem Intervall von 5 s und einer gesamten Erfassungszeit von 2 min ein.
- Auf das Objektivfenster ein entsprechendes Index-Matching-Material (Immersionsöl) auftragen. Verwenden Sie den Phasenkontrast-Modus, um sich auf die Zellen auf der Deckglasoberfläche zu konzentrieren.
- Finden Sie eine transfizierte Zelle unter 561 nm Licht, indem Sie die Mikroskopstufe bewegen.
ACHTUNG: Vermeiden Sie die Verwendung von blauem Licht während dieses Schrittes, da es die Assoziation zwischen photoaktivierbaren Proteinen aktiviert. - Sobald eine transfizierte Zelle lokalisiert ist, initiieren Sie die Datenerfassung.
HINWEIS: Erfolgreich transfizierte Zellen sollten sowohl Fluoreszenz in beiden FITC (von 2CIBN-GFP-CaaX) als auch TRITC (aus CRY2-mCherry-Raf1) Kanälen zeigen (Abbildung 1C-D ). - Notieren Sie eine Reihe von zeitgestempelten Bildern sowohl im FITC- als auch im TRITC-Kanal (Abbildung 1D ).
- Um die spontane Dissoziation des CRY2-CIBN-Proteinkomplexes zu untersuchen, notieren Sie einen weiteren Zeitstempel mit dem Txred-Kanal alleine für 20 min mit einem 30 s-Intervall.
- Sparen Sie das Zeitstempel für die Datenanalyse.
- Schalten Sie einen Computer, eine Lichtquelle, ein Mikroskop und eine Kamera ein. Verwenden Sie für den Rest des Protokolls ein konfokales Fluoreszenzmikroskop (ausgestattet mit einem 100fachen Öl-Immersionsobjektiv).
- Bildanalyse
- Öffnen Sie die Bilder mit einer Bildanalyse-Software, die die Intensität aus einem Bild extrahieren kann 40 .
- Wählen Sie zwei repräsentative Schnappschüsse aus: eine vor der blauen Belichtung und die andere Nach-Blau-Belichtung.
- Zeichnen Sie eine Linie über die Zelle, die den Hintergrund, die Plasmamembran und das Zytoplasma überspannt. Projekt die Intensität entlang dieser Linie. Speichern Sie die Werte und zeichnen Sie sie aus, um den Unterschied vor und nach der Belichtung zu vergleichen (Abbildung 1D ).
- Zur Analyse der Kinetik der Protein-Assoziation, selecT eine repräsentative Region von Interesse (ROI) in der Plasmamembran, ein ROI im Zytoplasma und ein ROI im Hintergrund.
- Erfassen Sie die mittleren Intensitäten der Plasmamembran (I PM ), des Zytoplasmas (I CYT ) und des Hintergrunds (I BKD ) für den Bildstapel.
- Berechnen Sie das Verhältnis der Membran / Zytosol-Intensität für jedes Bild unter Verwendung der folgenden Gleichung:
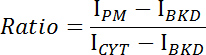
- Zeichnen Sie das Verhältnis über die Zeit und bestimmen Sie die Bindungs- oder Dissoziationskinetik von CRY2-CIBN (Abbildung 1E- F ).
2. Aufbau eines LED-Arrays für Langzeit-Lichtstimulation in einem CO 2 -Inkubator
HINWEIS: Das Gesamtschema des experimentellen Aufbaus ist in Abbildung 2A dargestellt.
- Machen Sie das LED-Array, indem Sie 12 blaue LEDs in zwei Brotkarren einfügenDs und verbindet Strombegrenzungswiderstände.
HINWEIS: Bei einer Stromversorgung von 30 V können vier LEDs in Serie geschaltet werden und ihre Helligkeit kann durch einen Strombegrenzungswiderstand gesteuert werden. - Legen Sie die Breadboards in eine Aluminiumbox.
HINWEIS: Die Höhe des Aluminiumkastens sollte 2 Zoll sein. Diese Höhe ist optimal für die Beleuchtung einer 12-Well-Gewebekulturplatte, da die Größe des divergierenden Lichtflecks die gleiche ist wie die eines einzigen Brunnens. - Verwenden Sie zwei Metalldrähte, um an die Stromversorgung anzuschließen. Vergewissern Sie sich, dass die Länge des Drahtes ausreichend ist, wenn der Lichtkasten in einen CO 2 -Inkubator gestellt wird.
- Verwenden Sie einen transparenten Lichtdiffusor als Deckel des Lichtkastens (Abbildung 2C ).
- Kalibrieren Sie die Leistungsabgabe jeder LED an einem Bereich von Spannungseingängen. Verwenden Sie eine Leistung von 0,2 mW / cm 2 für den 24 h PC12-Zelldifferenzierungsassay. Verwenden Sie eine Leistung von 5 mW / cm 2 für Live- Xenopus- Embryonen oder ExplantateAssays
3. Optogenetische Induktion der PC12-Zelldifferenzierung
- Zellkultivierung und Transfektion
- Mischen Sie 500 ml Medium für eine Ratten-Pheochromozytom (PC12) Zellkultur durch Mischen von 407,5 ml F12K mit 75 ml Pferdeserum + 12,5 ml FBS + 5 ml 100 × Penn-Strep-Glutamin (Endkonzentrationen von Pferdeserum und FBS : 15% bzw. 2,5%). Machen Sie niedrig-Serum-Medium durch Mischen 1 Volumen des vollen Mediums in 99 Volumina F12K Medium.
- Platte PC12-Zellen in einer 12-Well-Platte mit einer Dichte von 300.000 Zellen / Well oder 75.000 Zellen / cm 2 .
- Transformieren Sie die Zellen mit CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 h nach der Zellplattierung (ähnlich wie bei Schritt 1.4.7). Verwenden Sie für jede Vertiefung 1,2 μg DNA. Lassen Sie die Zellen über Nacht in Kulturmedium in einem 37 ° C-Inkubator, ergänzt mit 5% CO 2, zurückgewinnen. Überprüfen Sie die Transfektionseffizienz 16 h nach der Transfektion.
HINWEIS: Eine 30-50% Transfektion effUm eine ausreichende Zellzahl zu gewährleisten. - Ändern Sie das Medium bis zum niedrigen Serummedium 24 h nach der Transfektion. Verwenden Sie 1 ml Medium pro Well einer 12-Well-Platte.
- Lichtinduzierte PC12-Zelldifferenzierung
- Legen Sie das LED-Array in einen CO 2 -Inkubator. Verbinden Sie es mit einem Netzkabel mit der Stromversorgung. Stellen Sie die Leistung der LED auf 0,2 mW / cm 2 ein . Legen Sie die 12-Well-Platte mit transfizierten PC12-Zellen (Schritt 3.1.3) auf das Fenster des LED-Arrays. Kontinuierliche Lichtbeleuchtung für 24 h in einem 37 ° C Inkubator, ergänzt mit 5% CO 2, auftragen.
- Fluoreszenz-Live-Zell-Bildgebung
- Befolgen Sie die Schritte 1.5.1-1.5.4 für das Mikroskop und die Probenvorbereitung.
- Einrichten der Einzel-Schnappschuss-Datenerfassung. Benutze 200 ms für den GFP- und TXred-Kanal.
- Erfassen von Bildern der transfizierten Zellen sowohl im GFP- als auch im TXred-Kanal. Rekord etwa 200 celLs für jede Bedingung. Speichern Sie die Dateien für die Datenanalyse.
- Bildanalyse
- Zähle den Prozentsatz der differenzierten Zellen über die Gesamtzahl der transfizierten Zellen.
- Verwenden Sie ein beliebiges Zelle-Modul in der Bildanalyse-Software, um die Zellen zu zählen.
- Die differenzierten Zellen manuell zählen.
HINWEIS: Differenzierte Zellen sind definiert als solche, bei denen mindestens ein Neurit deutlich länger als der Zellkörper ist ( Abbildung 3A- 3B ). - Wiederholen Sie Schritt 3.4.3 mit undifferenzierten Zellen.
- Berechnen Sie das Differenzierungsverhältnis mit folgender Gleichung:
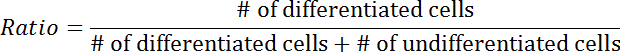
4. Optogenetische Kontrolle der Kinase-Aktivität in Xenopus- Embryonen
- Vorbereitung der Puffer
- Vorbereitung 1 L von 1x Marc's Modified Ringer (MMR): 100 mMNaCl, 2 mM KCl, 1 mM MgCl 2 , 2 mM CaCl 2 und 5 mM HEPES; PH 7,5
- Herstellung von mRNA
- Digest die CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX-Plasmid-DNA (2 μg) mit 1 μl ApaI (50 Einheiten) bei 37 ° C für 2 h.
- Niederscheidung linearisierte DNA in 60 μl 100% Ethanol. Spin bei 12.000 × g für 5 min bei Raumtemperatur, um die DNA zu pelletieren. Den Überstand vorsichtig entfernen und das Pellet erneut mit 60 μl 70% igem Ethanol waschen. Spin bei 12.000 × g für weitere 5 min bei Raumtemperatur. Den Überstand vorsichtig entfernen und das DNA-Pellet in 6 μl RNase-freiem H 2 O resuspendieren.
- In-vitro- Transkription durch Inkubieren von 1 & mgr; g linearisierter DNA mit 2 & mgr; l zugeführter SP6-RNA-Polymerase, einer Ribonukleotidmischung (10 mM ATP, CTP und UTP, 2 mM GTP und 8 mM Cap-Analog) und 20 & mgr; l Nuklease- Freies Wasser bei Raumtemperatur für 1 Stunde.
- Entfernen Sie die DNA-Matrize durch DNase I-Verdau bei 37 ° C für mindestens 15 min und Reinigen der synthetisierten RNA unter Verwendung einer Siliciumdioxid-Membran-Spin-Säule.
- Stoppen Sie die Reaktion und präzipitieren die RNA durch Zugabe von 30 & mgr; l nukleasefreiem Wasser und 30 & mgr; l LiCl-Präzipitationslösung. Mischen Sie gründlich und kühlen Sie für 30 min bei -20 ° C ab.
- Bei 4 ° C für 15 min bei 12.000 × g zentrifugieren, um die RNA zu pelletieren.
- Den Überstand vorsichtig entfernen. Die RNA einmal mit 1 ml 70% igem Ethanol waschen und in 40 μl nukleasefreies Wasser resuspendieren.
- Laden Sie 40 μl RNA in eine Spinsäule. Zentrifugieren bei 4 ° C für 1 min bei 12.000 × g, um die nicht eingebauten Nukleotide und Kappen zu entfernen.
- Ernte Xenopus Embryos
- Folgen Sie dem von Sive et al. 41 , um die Xenopus-Embryonen zu erhalten.
- MRNA-Mikroinjektion
- FabrizierenDie Nadeln für die Mikroinjektion durch Ziehen von Glaskapillaren mit einem Kapillarzieher. Setzen Sie das Zugprogramm auf: Hitze = 355, ziehen Sie Stärke = 80, Geschwindigkeit = 50 und Zeit = 100.
- Den Geleemantel von den Embryonen entfernen, indem man sie mit 3% Cystein behandelt (verdünnt in 0,2x MMR).
- Übertragen Sie die Embryonen auf 3% Polysucrose und 0,5x MMR Lösung für Mikroinjektion. Inpizieren Sie 500 pg auf 1 ng CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX-RNA in jeden Embryo.
HINWEIS: Die Injektion einer Dosis höher als 1 ng führt zu einer blauen lichtunabhängigen Aktivierung der MAPK-Signalisierung.
- Optogenetische Stimulation der Kinaseaktivität
- Kultur mikroinjizierte Embryonen in 3% Polysucrose, 0,5x MMR-Lösung bei Raumtemperatur bis zum Mittel-Gastrula-Stadium (Stadium 12). Dann Kultur die Embryonen in 0,2x MMR-Lösung.
- Übertragen Sie die Embryonen oder Explantate auf eine 12-Well-Platte. Legen Sie die 12-Well-Platte auf das hausgebaute LED-Array (Schritt 2.5) für Blue-LighT (475 nm) Behandlung.
- Legen Sie einen Spiegel auf die Oberseite der 12-Well-Platte, um die volle blaue Lichtbeleuchtung der Embryonen oder Explantate zu gewährleisten.
- Tune die Macht des blauen Lichts auf 5 mW / cm 2 .
HINWEIS: Die Blue-Light-Behandlung kann zu jeder gewünschten Zeit durchgeführt werden, entweder in 3% Polysucrose, 0,5x MMR-Lösung oder 0,2x MMR-Lösung. - Ernte die Embryos zu jeder gewünschten Zeit. Verwenden Sie sie für histologische, Western-Blot- oder Gen-Expressions-Analyse.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ratiometrische Expression von photoaktivierbaren Proteinpaaren: Abbildung 1A zeigt den Entwurf eines bicistronischen optogenetischen Konstrukts, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (bezeichnet als CRY2-2A-2CIBN), basierend auf dem Schweine-Teschovirum- 1 2A (P2A) -Peptid, das die höchste Ribosomen-überspringende Effizienz unter den Säugerzelllinien 42 zeigt . In früheren Arbeiten wurde festgestellt, dass das optimale Verhält...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Beim Bau des Lichtkastens sollte die Leistung einzelner LEDs gemessen werden. Basierend auf bisherigen Erfahrungen kann die Leistung zwischen einzelnen LEDs aufgrund der Fertigungsvarianz variieren. Wählen Sie einen Satz von LEDs, die eine Leistungsabgabe innerhalb von 10% voneinander haben. Die Anzahl der LEDs, der Strombegrenzungswiderstand und die Leistungsaufnahme können für verschiedene Arten von Zellkulturbehältern ( zB eine 6-Well- oder 24-Well-Platte) modifiziert werden. Eine 24 h Lichtbeleuchtung b...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Diese Arbeit wurde von der University of Illinois bei Urbana-Champaign (UIUC) und den National Institutes of Health (NIGMS R01GM111816) unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
Referenzen
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975(2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469(2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. Early Development of Xenopus laevis: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556(2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917(2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256(2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057(2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738(2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten