Method Article
Korrelative Höchstauflösung und Elektronenmikroskopie Protein Lokalisierung im Zebrafisch Netzhaut zu lösen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die notwendigen Schritte zur subzellulären Protein Lokalisierung auf Zebrafisch Netzhaut Ergebnisse korrelieren Höchstauflösung Lichtmikroskopie und Elektronenmikroskopie Bilder scannen.
Zusammenfassung
Wir präsentieren Ihnen eine Methode, um die subzelluläre Protein-Lokalisierung in der Larven Zebrafisch Netzhaut zu untersuchen, durch die Kombination Höchstauflösung Lichtmikroskopie und Rasterelektronenmikroskopie. Die Sub-Beugung begrenzt Auflösung Fähigkeiten von Lichtmikroskopen Höchstauflösung zulassen Verbesserung der Genauigkeit der korrelierten Daten Kurz gesagt, 110 Nanometer Dicke Cryo-Abschnitte werden auf einem Silizium-Wafer übertragen und nach Immunfluoreszenz-Färbung, durch Lichtmikroskopie Höchstauflösung abgebildet sind. Anschließend sind die Abschnitte in Methylcellulose und Platin beschattet vor Bildgebung in einem Rasterelektronenmikroskop (REM) erhalten. Die Bilder von diesen zwei Mikroskopie-Modalitäten sind leicht zusammengeführt mit Gewebe Wahrzeichen mit open-Source-Software. Hier beschreiben wir die angepasste Methode für die Larven Zebrafisch-Netzhaut. Diese Methode ist jedoch auch für andere Arten von Geweben und Organismen. Wir zeigen, dass die ergänzende Informationen durch diese Korrelation in der Lage, den Ausdruck der mitochondrialen Proteine im Zusammenhang mit den Membranen und Cristae der Mitochondrien sowie zu anderen Abteilungen der Zelle zu lösen ist.
Einleitung
Methoden zur Bestimmung der subzellulären Lokalisierung von Proteinen und deren Beziehung zu verschiedenen Kompartimenten der Zelle sind wesentliche Instrumente, um ihre Funktionen und möglichen Wechselwirkungen zu verstehen. Super-Auflösung Mikroskopie in Kombination mit Elektronenmikroskopie bietet solche Informationen1. Grundzustand Erschöpfung Mikroskopie, gefolgt von einzelnen Molekül Rückkehr (GSDIM) ist eine super-Resolution-Mikroskopie-Technologie kompatibel mit einer Vielzahl von organischen und genetisch codierte Fluorophore2 und erreicht eine laterale Auflösung bis zu 20 nm 3. die Einbeziehung von Methoden mit höherer Auflösung als standard Beugung begrenzte Mikroskopie verbessert die Genauigkeit der Korrelation4,5,6. Um die beste Korrelation der Proteinexpression mit einem spezifischen subzelluläre Fach zu erreichen und die Unsicherheit7 die Verwendung der gleichen ultradünnen Sektion für Licht- und Elektronenmikroskopie reduzieren wird empfohlen. Unter den verschiedenen speziellen Methoden Tokuyasu Cryo-Abschnitt Protokoll erfordert keine Austrocknung oder Harz einbetten und, darüber hinaus bewahrt die Antigenität von vielen Epitopen und bietet gute Gewebe Ultrastruktur8. Mehrere Methoden haben gezeigt, dass die Anwendbarkeit dieser Abschnitte in korrelative Licht- und Elektronenmikroskopie (CLEM)4,5,9,10.
Die Zebrafisch Netzhaut ist ein wertvolles Modell, visuelle Entwicklung und menschliche Krankheitsmechanismen angesichts seiner hoch konservierte Struktur und Funktion in Wirbeltieren zu studieren. In bestimmten, retinalen Photorezeptoren Display bestehend aus die gleiche Architektur als Säugetier Photorezeptoren mit einer basalen Synapse, ein Apico-basaler länglichen Kern, clustering von Mitochondrien in dem mehr apikalen innere und eine äußere Segment Membran Festplatten in den meisten apikale Position11. Protein-Lokalisierung, die vielfältigen zellulare Fächer ist zwischen Zebrafish konserviert und menschlich, so dass die Untersuchung der biologischen Funktion des menschlichen krankheitsrelevanten Proteinen12,13.
Hier präsentieren wir ein Protokoll, um Larven Zebrafisch Vorbereitung Netzhaut Proben, die Lokalisierung der mitochondrialen Außenmembran Protein Tom20 korrelative Höchstauflösung Licht- und Elektronenmikroskopie zu lösen. Die Methode basiert auf das Sammeln von Cryo-Abschnitte auf Silizium-Wafer und Erlangung Kontrast durch topographische Informationen nach dem Auftragen einer dünnen Schicht aus Platin hergestellt. Diese Schritte sind klare technische Verbesserungen in Bezug auf Benutzerfreundlichkeit, Reproduzierbarkeit und Zeit für Experimente. Wir haben vor kurzem die Anwendbarkeit der Methode zur Erkennung von Kernporen und mitochondrialen Proteine in Maus Gewebe14gezeigt.
Protokoll
alle Experimente wurden gemäß der ARVO-Erklärung für die Verwendung von Tieren in Ophthalmic und Vision Research durchgeführt und von den örtlichen Behörden genehmigt wurden.
1. Vorbereitung der ultradünnen Abschnitte auf Silizium-Wafer
- Probe Fixierung
- Fixativ Lösung mit 0,1 % Glutaraldehyd, 4 % Formaldehyd in 0,1 M Cacodylate Puffer zubereiten.
Hinweis: Vorsicht! Vorsicht beim Arbeiten mit Glutaraldehyd, Formaldehyd und Cacodylate Puffer, tragen geeigneten persönlichen Schutzausrüstung und arbeiten in einer Dampfhaube. - Einschläfern 5 Tage post Düngung (5 Dpf) Zebrafischlarven mit Tricaine (Ethyl-3-Aminobenzoate-Methanesulfonate, 0,4 % w/V mit PBS-Puffer (Phosphat-Puffer Kochsalzlösung, pH 7)) wie zuvor beschriebenen 15.
- Fixieren die Larven durch Eintauchen in vorgekühlt Fixativ Lösung auf Eis. Entfernen Sie den Kopf und Inkubation über Nacht bei 4 ° C mit sanftes Schaukeln.
- Sezieren die Augen durch das Gewebe um das Auge mit feinen Pinzette und Skalpell Mikrodissektion (unter einem Feldstecher) in gekühlten Fixativ Lösung auf einem 1 % Agarose-Bett Teller trimmen. Übertragen Sie die Augen zu einem Schlauch mit frisch vorgekühlt Fixiermittel.
- Waschen zweimal mit PBS-Puffer (Phosphat-Puffer Kochsalzlösung, pH 7,4) für 5 min jedem Waschgang bei Raumtemperatur (RT).
- Fixativ Lösung mit 0,1 % Glutaraldehyd, 4 % Formaldehyd in 0,1 M Cacodylate Puffer zubereiten.
- Gelatine Infiltration und Montage
- Warm up 15 mL lokales Essen Marke Gelatine 12 % w/V in PB (Phosphatpuffer, 0,1 M pH 7.4) bei 40 ° c entfernen der PBS und Gelatine-Lösung in das Röhrchen mit den Augen. Tippen Sie auf das Rohr sanft um die Gelatine Infiltrate die Probe und 10-30 min bei 40 ° C in einem Thermoblock mit sanft schütteln oder im Wasserbad inkubieren.
- Füllen Sie 12 x 5 x 3 mm Silikon oder Polyethylen flache Einbettung Formen mit warmen Gelatine im Wasserbad 40 ° C. Fügen Sie zwei Augen pro Form mit einer Pipette, unter ein Fernglas mit einer Dissektion Nadel richtig ausrichten und lassen Sie die Gelatine bei Raumtemperatur für 1 min abkühlen und Aushärten bei 4 ° C für 20 min.
- Nachtrimmen den Gelatine Block unter das Fernglas ein Auge pro Block mit einer Rasierklinge passen.
- Übertragen die Gelatine eingebettet-Augen auf 2,3 M Saccharose in PB auf Eis. Über Nacht bei 4 ° C inkubieren.
- Austausch neue 2,3 M Saccharoselösung und Store bei 4 ° C oder-20 ° C; nach diesem Schritt Proben sind bereit für schneiden oder kann mehrere Wochen bis Monate bei-20 ° C gelagert werden.
- Nachtrimmen den Gelatine Block auf fast die Größe des Auges vor der Übertragung an eine Cryo-Pin. Einfrieren in flüssigem Stickstoff und Transfer zum Cryo-regungsloses.
- Kryoschneiden
- Cut 110 nm dick-Abschnitte bei-120 ° C mit einem Diamant-Messer in der Cryo-regungsloses.
- Wählen Sie die Abschnitte mit einer kabelgebundenen Schleife mit einem Tröpfchen von 2 % Methylcellulose (im Wasser) und 2,3 M Zuckerlösung (1:1). Übertragen Sie Abschnitte auf einem 7 x 7 mm-Silizium-Wafer. Abschnitten bei 4 ° C bis zur Weiterverarbeitung lagern.
2. Immunolabelling
- Wash Wafer mit PBS bei 0 ° C für 20 min auf vier Tropfen indem die Wafer kopfüber auf die Tropfen. Waschen mit PBS-Puffer 2 x 2 min bei Raumtemperatur.
- Inkubieren Sie 3 Mal in 0,15 % Glycin in PBS, für 1 Minute. 3 X 1 min. Waschen/Spülen mit PBS. Pre-incubate mit PBG (PBS mit 0,5 % Rinderserumalbumin BSA und 0,2 % Gelatine Typ B) für 5 min.
- Inkubation mit Kaninchen Anti-Tom20 (siehe die Tabelle der Materialien) in PBG (4 µg/mL) für 30 min bei Raumtemperatur.
- Waschen 6 X für 1 min/Waschgang in PBG. Pre-incubate mit PBG für 5 min bei RT inkubieren mit Anti-Kaninchen-Alexa-647-F(ab')-2 (siehe Tabelle of Materials) in PBG (7,5 µg/mL) für 30 min.
- Waschen 6 X für 1 min/Waschgang in PBG. Waschen Sie 3 x 2 min in PBS. Inkubieren mit DAPI (4 µg/mL) mit PBS-Puffer für 10 s. Waschen 2 x 2 min in PBS.
3. Super-Auflösung Mikroskopie
- Inkubation der Wafer kurz (10 s) durch Inverkehrbringen ein Tropfen einer 1:1 Lösung von Glycerin (80 %) und imaging-Puffer mit einem Sauerstoff Aufräumvorgang System (200 mM Phosphatpuffer mit 10 % Glukose, 0,5 mg/mL Glucoseoxidase, 40 µg/mL Katalase, 15 mM Beta-Mercaptoethylamine-Hydrochlorid (MEA HCL), pH 8,0).
- Übertragen die Wafer (Abschnitt nach unten) auf einer Petrischale von Glas unten (Dicke 170 ± 5 µm) auf einen frischen Tropfen der 1:1 Mischung von Glycerin (80 %) und imaging-Puffer (wie in Schritt 3.1).
- Entfernen von allen Seiten mit einer Pipette, die meisten der Flüssigkeit unter der Wafer. Silikon-Streifen verwenden, um den Wafer auf den Boden der Petrischale beheben.
Hinweis: Silikon-Streifen bestehen aus zwei-Komponenten-Silikon-Kleber (3 x 12 mm). - Bild Abschnitte auf einem inversen Mikroskop mit hoher numerischer Apertur (z. B. 160 X / NA 1,43; siehe Tabelle of Materials) Öl eintauchen Superresolution engagierte Ziel. Vor der Bildgebung, lassen Sie die Probe auf Mikroskop Temperatur Reduzierung minimieren/laterale und axiale Drift equilibrate.
- Zentrieren Sie den gewünschten Bereich und erste Weitfeld Epifluoreszenz Referenzbilder zu erwerben. Ändern Sie in die Betriebsart super Auflösung. Passen Sie die Belichtungszeit der Kamera auf 15 ms und setzen das Elektron Multiplikation (EM) zu gewinnen, um das Maximum von 300.
- Leuchten Proben mit den 642 nm kontinuierliche Welle Laser auf maximale Laserleistung (entsprechend ~2.8 kW/cm 2) in Epifluoreszenz-Modus. Sobald die einzelnen Moleküls blinkt sind gut in jedem Frame getrennt, so dass die Wahrscheinlichkeit gering ist, dass die einzelnen Signale überlappen, legen Sie die Laserleistung auf ~0.7 kW/cm 2. Notieren Sie die raw-Bild in Epifluoreszenz-Modus durch den Erwerb von mindestens 30.000 Bilder.
Hinweis: Diese Parameter kann variieren, abhängig von verschiedenen Proben und Kennzeichnung dichten. - Aus den Rohdaten erzeugen eine Wiederaufbau-Event-Liste (Lokalisierungen von jedem Einzelmolekül blinzeln in die raw-Bild) mit einer Nachweisgrenze von 30 Photonen (muss angepasst werden, entsprechend der Probe) durch Klicken auf " Evaluate " in der ' t-Serie Analyse ' unter ' Tools '.
- Visualisieren die super Auflösung durch "glockenförmig" passende 16 Anwendung eine Rendering-Pixel-Größe von 4 nm durch Klicken auf " Create Image " in der ' Event Liste Verarbeitung Panel ' unter ' Werkzeuge. '
Hinweis: Für die Erzeugung von super gelöst Bild waren die integrierten Software-Tools des Systems in dieser Studie verwendeten beschäftigt. Jedoch die beschriebenen Superresolution Bildgebung auf anderen Einzelmolekül Lokalisierung basierten Systemen durchgeführt werden konnte und der Datenverarbeitung konnte mit open-Source-Software-Tools wie Gewitter 17.
4. Platin Shadowing
- Silikon-Streifen zu entfernen und einen Tropfen PBS nah an den Rändern des Wafers, lift it up aus der Petrischale. Die Wafer 2 x 2 min in PBS, waschen, dann Post fix it mit 0,1 % Glutaraldehyd mit PBS-Puffer für 5 min. Waschen 2 X 2 min/waschen in PBS.
- Inkubieren den Wafer zweimal für 5 min/Inkubation in 1 Tropfen von Methylcellulose 2 % in Wasser auf dem Eis. Legen Sie die Wafer in einem Zentrifugenröhrchen und Zentrifugieren bei 14.100 x g 90 S. Mount es auf ein SEM-Aluminium-Stub mit der Durchführung von Kohlenstoff Zement.
- Fügen Sie eine Schicht von 2 bis 10 nm von Platin/Kohlenstoff auf der Probe von rotary Beschattung bei 8°, mit einem Elektron Lichtstrahl Verdunstung Geräteeinstellungen: 1,55 kV 55 mA, bei 0,3 nm/s und einem Winkel von 8°, Rotationsebene 4.
5. Rasterelektronenmikroskopie
- Bildausschnitten mit einem scanning Electron mIcroscope bei 1,5 kV, 2 mm arbeiten Abstand und mit einer In Objektiv Sekundär-Elektronen-Detektor.
6. Ausrichtung der Licht- und Elektronenmikroskopie Bilder
- Öffnen beide Arten von Bildern mit Fidschi 18 klicken Sie " Datei | Offene ". Leinwandgröße anpassen, indem Sie auf " Bild | Anpassen | Leinwand Größe " und beide Bilder zu einem Stapel zu bringen, indem Sie auf " Bild | Stapel | Bilder stapeln ". Speichern Sie den Stapel als TIFF-Datei-Typ.
- In Fidschi, öffnen eine neue TrackEM2- 19-Schnittstelle, indem " Datei | neu | TrackEM2 (neu) ". Importieren Sie den Stapel mit den beiden Bildern durch Rechtsklick auf das schwarze Fenster und Auswahl " Import Stack ".
- Align Licht Mikroskopie Bild Elektronenmikroskopie Bild manuell mit Sehenswürdigkeiten, indem mit Hilfe der rechten Maustaste auf das Bild und " ausrichten | Richten Sie Layer manuell mit Wahrzeichen ".
- Pick Select Tool (schwarzer Pfeil) Sehenswürdigkeiten hinzufügen. Verwenden Sie die Form der Kerne, die gleichen Kanten in beiden Bildern (ergänzende Abbildung1) wählen Sie als Referenz. Fügen Sie mehrere Punkte (mindestens drei Punkte müssen ausgewählt werden). Ausrichtung mit einem affinen Modell anwenden, indem Rechte Maustaste klicken und auswählen " wenden Transformation | Affine Modell ".
- Layer-Transparenz zu ändern (siehe ergänzende Abbildung1) zur Bewertung der Qualität der Achse.
Ergebnisse
Die Expression des Proteins Tom20, eine Untereinheit der Translocase mitochondrialen Außenmembran Komplex20, war entschlossen, in Dünnschliffen Larven Zebrafisch Netzhaut, durch Höchstauflösung Lichtmikroskopie (Abbildung 1) und diese Information war ergänzt mit dem topographischen Signal durch Rasterelektronenmikroskopie nach Platin Schattierung von die gleiche Abschnitte erhalten. Diese korrelativen Daten bestätigen die Lokalisation eines Proteins in Verbindung mit einem bestimmten Fach, der mitochondrialen Außenmembran und darüber hinaus Auskunft über das Verhältnis des Proteins mit anderen Organellen der Zelle.
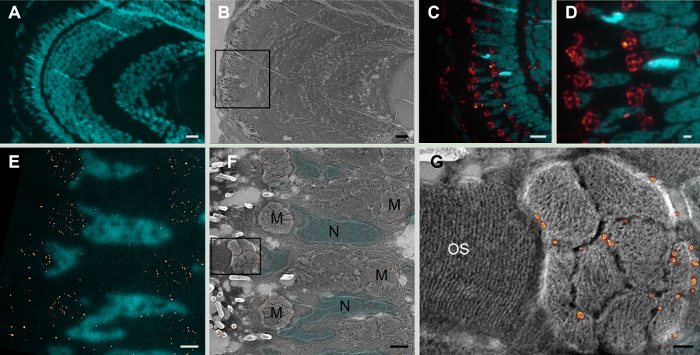
Abbildung 1: CLEM auf Zebrafisch Netzhaut. A. geringer Vergrößerung Weitfeld Bild eines 5 Dpf Zebrafisch retinalen Abschnitts, Kerne gebeizt mit DAPI (Cyan). B. Rasterelektronenmikroskopie des gleichen Gebietes. C. höhere Vergrößerung Weitfeld-Bild des Rahmens in B. Kerne gebeizt mit DAPI (Cyan) und Tom20 mitochondriale Färbung wird in rot angezeigt. D. Weitfeld Bild des gleichen Abschnitts bei höherer Vergrößerung. Das Muster der Tom20 Ausdruck ist in den Clustern von Mitochondrien. E. Ausdruck von Tom20 (rote Punkte) von GSDIM Mikroskopie erkannt. Kerne gebeizt mit DAPI (Cyan). F. gleichen Abschnitt als E Kombination korrelative Höchstauflösung und Rasterelektronenmikroskopie. Tom20 Färbung (rote Punkte) erscheint am mitochondriale Cluster (M) an der äußeren Membranen von Mitochondrien. DAPI Fluoreszenzsignal in den Kernen (N) entspricht mit der Topographie des SEM Bild. G. hohe Vergrößerung Bild des Frames in F. Das scanning Electron Microscopy Bild bietet Zusammenhang GSDIM (rote Punkte). Mitochondrien Cristae sind deutlich sichtbar und die Tom20 Färbung an den äußeren Membranen der Mitochondrien lokalisiert ist. Die Membranen der äußeren Segment die Photorezeptoren (OS) eindeutig gelöst werden. Bild Pixel Größe 5 nm. Skalieren Sie Bars: A, B und C: 10 µm; D: 2 µm; E und F: 1 µm und G: 0,2 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergänzende Abbildung1: Ausrichtung der Licht- und Elektronenmikroskopie Bilder. A. Screenshot von TrackEM2 Schnittstelle mit SEM Bild und nummerierten Sehenswürdigkeiten entlang verschiedenen Kernen (gelb). B. Screenshot von TrackEM2 Schnittstelle mit Fluoreszenzbild und nummerierten Sehenswürdigkeiten (gelb) entlang verschiedenen DAPI gefärbt Kerne. Layer-Transparenz ändern können die Schieberegler auf der linken oberen Teil des Menüs verwendet werden. Maßstab: 1 µm. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Diese Methode verbindet super gelöst Protein Lokalisierung mit Kontextinformationen zu bestimmen, die genaue Position der Proteine in einer Organelle. Wir zeigen hier die Beendigung des Experiments, die Expression von Tom20 in der äußeren Membran der Mitochondrien und seine Beziehung zu anderen Organellen wie Kerne oder Außensegmenten der Sehzellen in der Netzhaut Larven Zebrafisch zu visualisieren.
Tokuyasu Cryo-Schneiden erfordert einige Übung, gut erhaltene Abschnitte zu erwerben. Dies ist jedoch eine Methode, die in vielen Laboratorien mit Erfolg21eingesetzt. Übertragung der Abschnitte auf dem Silizium-Wafer ist sehr einfach und benötigt keine besonderen Überlegungen. Die Verwendung von Glycerin in der bildgebenden Puffer ist ein sehr wichtiger Schritt um zu vermeiden, Trocknen der Abschnitte. Für Super-Resolution Imaging sind die besten Ergebnisse erzielt, wenn der Wafer in unmittelbarer Nähe der Glasboden der Petrischale befindet. Die Silikon-Streifen helfen Aufrechterhaltung des Wafers in dieser Position. Besonderer Sorgfalt ist geboten beim Entfernen der Streifen um die Abschnitte Schäden zu vermeiden.
Die Dicke der Cryo-Abschnitte, rund 100 nm, dünner als die optische Auflösung in der Z-Dimension, erleichtert zusätzlich die Genauigkeit dieser korrelativen Methode, wie die Super-Resolution-Mikroskopie-Signal nur diese dünne Schicht stammt und die Raster-Elektronenmikroskop Signal zeigt die Topografie der Probe. Diese Methode könnte auch mit mehrfarbigen Bildgebung kombiniert werden. Allerdings muss besondere Vorsicht geboten, dass die Probe unter gleichen Bedingungen (z. B. bildgebende Puffer) abgebildet kann und Übersprechen verhindert werden sollte.
Eine Einschränkung der Methode ist die zweidimensionale nähern, da nur eine begrenzte Anzahl von Schnittserien (ca. 3 bis 7 pro Wafer) gesammelt werden kann. Projekte zum Thema Volumen Analyse wäre so nicht ideal. Es ist jedoch eine Methode, die angewendet werden kann, um Protein-Expression in jede Art von Gewebe zu erkennen, durch einfaches Schneiden der Probe. Wir bieten eine Methode ohne Einsatz von klassischen Kontrastmittel wie Uranyl-Acetat oder Blei-Citrat. Der Kontrast von Platin Schattierung bietet sehr informativ topographischen Kontrast, aber in einigen Fällen Membranen sind schwer zu lösen. Dies kann Probleme für Projekte darstellen, die, zum Beispiel die Expression von Proteinen in kleinen Vesikeln zu bestimmen müssen.
Unser Protokoll basierend auf Tokuyasu Cryo-Abschnitte, nutzt Serienausstattung Höchstauflösung und Rasterelektronenmikroskope korrelative Ergebnisse erhalten. Die Sammlung von Abschnitten auf Silizium-Wafern und die Verwendung von Platin für Kontrast sind einfache Schritte, um Stabilität und Reproduzierbarkeit für die Probenvorbereitung.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Finanzierung von Ionen und RGB: Swiss National Science Foundation Ambizione-SCORE zu gewähren, PZ00P3 142404/1 und PZ00P3 163979.
Materialien
| Name | Company | Catalog Number | Comments |
| Paraformaldehyde | Sigma-Aldrich | #158127 | |
| Glutaraldehyde EM Grade | EMS, USA | #16220 | |
| Cacodylate | Merck | #8.20670 | |
| Tricaine | Sigma-Aldrich | #886-86-2 | |
| Agarose, peqGOLD Universal | VWR International GmbH | 35-1020 | |
| Flat embedding molds | BEEM Flat | ||
| Local food brand gelatin | Dr.Oetker | Extra Gold | |
| Sucrose | Merck | #1.07687 | |
| Methylcellulose | Sigma | #M-6385 | |
| Glycine | Sigma | #G-7126 | |
| Gelatin type B | Sigma | #G-6650 | |
| BSA | Applichem | #A6588.0050 | |
| Silicon wafer | Si-Mat Silicon Materials | Type: P/Boron; Orientation <111> ON; Growth method: CZ; Resistivity:1-30 ohm/cm; Surface: polished; Laser cut at 7 x7 mm | |
| Cryo-pin | Baltic Preparation | #16701950 | |
| Wired loop "Perfect loop" | |||
| Silicone stripes | Picodent | Twinsil 22 | |
| Glucose | Sigma-Aldrich | #G8270 | |
| glucoseoxidase | Sigma-Aldrich | #G7141 | |
| catalase | Sigma-Aldrich | #C40 | |
| beta-Mercaptoethylamine hydrochloride | Sigma-Aldrich | #M6500 | |
| anti-Tom20 | Santa Cruz Biotechnology | #sc - 11415 | |
| AlexaFluor 647 AffiniPure F(ab')2 fragment donkey anti rabbit IgG | Jackson Immuno-Research | #711-606-152 | |
| DAPI | ROCHE, Switzerland | #10236276001 | |
| Glycerol solution | Sigma-Aldrich | #49782 | |
| Glass bottom petri dish | Ibidi, Germany | u-Dish 35mm, high glass bottom, #81158 | |
| SEM aluminium stub | Agar Scientific | #G301F | |
| Conducting carbon cement Leit-C | Plano, Germany | AG3300 | |
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| Diamond Knife (Cryo Immuno) | Diatome | DCIMM3520 | |
| Cryo-ultramicrotome | Leica Microsystems | Leica EM FCS | |
| Widefield-TIRF microscope GSD Leica SR GSD 3D | Leica Microsystems | ||
| 160x 1.43 TIRF objective | Leica Microsystems | 11523048 | |
| SEM - Zeiss Supra 50 VP | Zeiss | Supra 50 VP | |
| FIB-SEM - Zeiss Auriga 40 | Zeiss | Auriga 40 |
Referenzen
- Hauser, M., Wojcik, M., Kim, D., Mahmoudi, M., Li, W., Xu, K. Correlative Super-Resolution Microscopy: New Dimensions and New Opportunities. Chem Rev. , (2017).
- Dempsey, G. T., Vaughan, J. C., Chen, K. H., Bates, M., Zhuang, X. Evaluation of fluorophores for optimal performance in localization-based super-resolution imaging. Nat Methods. 8 (12), 1027-1036 (2011).
- Fölling, J., Bossi, M., et al. Fluorescence nanoscopy by ground-state depletion and single-molecule return. Nat Methods. 5 (11), 943-945 (2008).
- Betzig, E., Patterson, G. H., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science (New York, N.Y.). 313 (5793), 1642-1645 (2006).
- Kopek, B. G., Shtengel, G., Grimm, J. B., Clayton, D. A., Hess, H. F. Correlative photoactivated localization and scanning electron microscopy. PLOS one. 8 (10), e77209 (2013).
- Paez-Segala, M. G., Sun, M. G., et al. Fixation-resistant photoactivatable fluorescent proteins for CLEM. Nat Methods. 12 (3), 215-218 (2015).
- Narayan, K., Subramaniam, S. Focused ion beams in biology. Nat Methods. 12 (11), 1021-1031 (2015).
- Tokuyasu, K. T. Immunochemistry on ultrathin frozen sections. Histochem J. 12 (4), 381-403 (1980).
- Suleiman, H., Zhang, L., et al. Nanoscale protein architecture of the kidney glomerular basement membrane. eLife. 2, e01149 (2013).
- Kopek, B. G., Shtengel, G., Xu, C. S., Clayton, D. A., Hess, H. F. Correlative 3D superresolution fluorescence and electron microscopy reveal the relationship of mitochondrial nucleoids to membranes. Proc Nat Acad Sci U S A. 109 (16), 6136-6141 (2012).
- Avanesov, A., Malicki, J. Analysis of the retina in the zebrafish model. Methods Cell Biol. 100, 153-204 (2010).
- Bachmann-Gagescu, R., Phelps, I. G., et al. The ciliopathy gene cc2d2a controls zebrafish photoreceptor outer segment development through a role in Rab8-dependent vesicle trafficking. Hum Mol Gen. 20 (20), 4041-4055 (2011).
- Bachmann-Gagescu, R., Dona, M., et al. The Ciliopathy Protein CC2D2A Associates with NINL and Functions in RAB8-MICAL3-Regulated Vesicle Trafficking. PLOS Gen. 11 (10), e1005575 (2015).
- Mateos, J. M., Guhl, B., et al. Topographic contrast of ultrathin cryo-sections for correlative super-resolution light and electron microscopy. Sci Rep. 6, 34062 (2016).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82 (5), 2775-2783 (2002).
- Ovesný, M., Křížek, P., Borkovec, J., Svindrych, Z., Hagen, G. M. ThunderSTORM: a comprehensive ImageJ plug-in for PALM and STORM data analysis and super-resolution imaging. Bioinformatics. 30 (16), 2389-2390 (2014).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Cardona, A., Saalfeld, S., et al. TrakEM2 software for neural circuit reconstruction. PLOS one. 7 (6), e38011 (2012).
- Wurm, C. A., Neumann, D., et al. Nanoscale distribution of mitochondrial import receptor Tom20 is adjusted to cellular conditions and exhibits an inner-cellular gradient. Proc Nat Acad Sci U S A. 108 (33), 13546-13551 (2011).
- Slot, J. W., Geuze, H. J. Cryosectioning and immunolabeling. Nat Prot. 2 (10), 2480-2491 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten