Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hochdurchsatz-parallele Sequenzierung Maßnahme Fitness Leptospira Interrogans Transposon Insertion Mutanten während Golden syrischen Hamster Infektion
In diesem Artikel
Zusammenfassung
Wir beschreiben hier eine Technik, die verbindet Transposon Mutagenese mit Hochdurchsatz-Sequenzierung zu identifizieren und quantifizieren Transposon Leptospiral Mutanten im Gewebe nach einem Zweikampf von Hamstern. Dieses Protokoll kann verwendet werden, um Bildschirm Mutanten für überleben und Verbreitung bei Tieren und kann auch auf in-vitro- Studien angewendet werden.
Zusammenfassung
In diesem Manuskript beschreiben wir ein Transposon Sequenzierung (Tn-Seq) Technik zu identifizieren und quantifizieren Leptospira Interrogans Mutanten in Fitness während der Infektion von syrischen Goldhamster verändert. TN-Seq kombiniert mit der Kraft der Hochdurchsatz-Sequenzierungstechnologie zufällige Transposon Mutagenese. Tiere sind gefordert, mit einem Pool von Transposon Mutanten (Eingang Schwimmbad), gefolgt von der Gewinnung von Blut und Gewebe ein paar Tage später zu identifizieren und zu quantifizieren, die Anzahl der Mutanten in jedes Organ (Ausgabe Pools). Die Ausgabe-Pools sind gegenüber den Eingang Pool, der in Vivo -Fitness jeden Mutante zu bewerten. Dieser Ansatz ermöglicht Screening von einem großen Pool von Mutanten in einer begrenzten Anzahl von Tieren. Mit geringfügigen Änderungen kann dieses Protokoll mit jeder Tiermodell der Leptospirose, Reservoir Host wie Ratten und akute Infektionsmodelle wie Hamster, sowie in-vitro- Studien durchgeführt werden. TN-Seq ist ein leistungsstarkes Tool zum Bildschirm für Mutanten mit in Vivo und in Vitro Fitness Mängel.
Einleitung
Identifizierung von Genen Virulenz für manche Bakterien, wie z. B. Leptospira spp., ist schwierig, wegen der begrenzten Zahl der genetischen Werkzeuge zur Verfügung. Eine häufig verwendete Methode ist die Erstellung einer Sammlung von Mutanten durch zufällige Transposon Mutagenese, gefolgt von der Identifikation der Insertionsstelle in jeder Mutant und Virulenz von einzelnen Transposon Mutanten in einem Tiermodell testen. Dieser Ansatz ist zeitaufwendig, teuer und erfordert eine große Anzahl von Tieren.
Als zufällige Mutagenese zunächst für den Erreger Leptospira Interrogansentwickelt wurde, wurden an Virulenz beteiligten Gene identifiziert, durch die Prüfung einzelner Mutanten in einem Tiermodell1. Mutanten wurden basierend auf Kriterien wie ihre mögliche Rolle bei der Signalisierung oder Motilität oder ihrer vorhergesagten Außenmembran oder Oberfläche Speicherort ausgewählt. Da die Mehrzahl der Leptospiral Gene kodieren hypothetischen Proteine der unbekannten Funktion2, Auswahl durch Mutation entstehende Variationen anhand dieser Kriterien Grenzen die Möglichkeit, neuartige Leptospiral Virulenz Gene zu entdecken.
Pools von L. Interrogans Transposon Mutanten wurden in jüngerer Zeit, für die Infektiosität in der Hamster und Maus Modelle3gezeigt. Jedes Tier wurde mit einem Pool von bis zu 10 Mutanten in Frage gestellt. Infektiosität einer Mutante erzielte er als positiv, wenn sie durch PCR Kulturen gewonnenen Blut und Niere festgestellt worden. PCR-Tests war mühsam, weil es eine individuelle PCR-Reaktion für jede Mutante im Pool erforderlich. Da die Frequenz jeder Mutant in den Kulturen nicht quantifiziert wurde, war der Ansatz zur Identifizierung von stark abgeschwächte Mutanten voreingenommen.
Wir beschreiben ein Transposon Sequenzierung (Tn-Seq) Technik, als eine Strategie, um effizienter Bildschirm für Virulenz Gene. TN-Seq besteht aus der Erstellung einer Bibliothek von Mutanten durch Transposon Mutagenese gefolgt von massiven parallelen Sequenzierung4,5,6. Transposon Mutanten sind kurz, gebündelt, in die Tiere geimpft und später erholte sich von verschiedenen Organen (Ausgabe Pools). Die DNA von den Ausgabe-Pools wird extrahiert und mit Restriktionsenzymen verdaut oder durch Ultraschallbehandlung geschert. Zwei Runden von PCR gezielt die Kreuzungen der Transposon Einfügung Seiten erfolgen. Dieser Schritt ermöglicht die Zugabe von den Adaptern für die Sequenzierung erforderlich. Die daraus resultierende PCR-Produkte werden von Hochdurchsatz-Sequenzierung Transposon Insertionsstelle jede Mutante des Pools zusammen mit ihrer relativen Fülle, zu identifizieren, die im Vergleich zu der ursprünglichen Zusammensetzung der Pool der Mutant analysiert.
Der Hauptvorteil dieses Ansatzes ist die Fähigkeit, gleichzeitig eine große Anzahl von Mutanten mit einer kleinen Anzahl von Tieren auf den Bildschirm. TN-Seq erfordert keine Vorkenntnisse der Transposon Einfügung Websites die erhöht die Chancen der Entdeckung neuer Leptospiren-spezifischen Genen in Virulenz mit weniger Zeit und mehr Effizienz. Weil Leptospiral Belastung im Gewebe relativ hoch in Nagetier-Modelle anfällig für tödliche Infektion (in der Regel 104 108 Bakterien/g Gewebe)7,8,9 ebenso wie Reservoirwirten 10,11, Gewebe direkt analysiert werden können, ohne die Notwendigkeit, Kultur, Verringerung der Verzerrungen durch in-vitro- Wachstum.
In Tn-Seq-Studien mit den meisten bakteriellen Erregern, die bisher beschrieben erlaubt die hohe Frequenz der insertional Mutagenese Infektion mit großen Pools mit Mutanten, die gemeinsam mit mehreren eng beieinander liegender Transposon Einfügungen innerhalb jedes Gen4 ,12,13,14. TN-Seq wurde auch für ein Bakterium entwickelt für die Mutagenese-Frequenz viel niedriger6 ist. Mit Leptospirakann eine Bibliothek von Transposon Mutanten erzeugt werden, durch die Einführung des Transposons auf ein konduktiven Plasmid durch Konjugation wie von Slamti Et Al15beschrieben. Die Häufigkeit der Transposon Mutagenese von L. Interrogans ist jedoch gering. Wenn das Transposon Himar1 auf ein konjugative Plasmid eingeführt wurde, wurde die Transconjugant Häufigkeit berichtet, nur 8,5 x 10-8 pro Empfänger mit der Lai-Belastung von L. Interrogans16 Zelle und wird voraussichtlich ähnlich schlecht mit den meisten anderen Sorten von L. Interrogans. Das hier beschriebene Protokoll ist im Teil auf der Grundlage, die für Borrelia Burgdorferi, entwickelt in denen die Häufigkeit von Transposon insertional Mutagenese ist auch niedrig6.
Für unsere Pilotversuch mit der Protokoll-17führten wir Transposon Mutagenese mit L. Interrogans Serovar Manilae Stamm L495 aufgrund des Erfolges der anderen Gruppen isolieren Transposon Einfügung Mutanten in den Stamm zusammen mit seinem tief LD50 (letale Dosis) für Virulenz1. Wir 42 Mutanten durch Tn-Seq abgeschirmt und identifiziert mehrere mutierte Kandidaten in Virulenz, darunter zwei mit Einfügungen in einem Kandidaten-Adenylat-Cyclase-gen defekt. Einzelprüfung der beiden Mutanten in Hamster bestätigt, dass sie einen Mangel an Virulenz17waren.
Protokoll
Achtung: Pathogene Stämmen von Leptospira SPP müssen unter Biosafety Level 2 (BSL-2) Containment Verfahren behandelt werden. Geeigneter persönlicher Schutzausrüstung (PSA) muss getragen werden. Ein Klasse II Biosicherheit Kabinett zwingend erforderlich alle Manipulationen von pathogenen Leptospira spp.
1. Erstellung der das Transposon mutierten Bibliothek15
-
Transfer von das Transposon in Leptospira Spp. durch Konjugation (Abbildung 1()
- Impfen Sie ein Volumen von exponentielle Phase Kultur der Pathogene Leptospira spp. in 10 mL Ellinghausen-McCullough-Johnson-Harris mittlerer (EMJH)18,19, 107 Zellen entspricht. Inkubation bei 30 ° C mit 150 u/min schütteln bis die Dichte 2-8 x 108 Zellen/mL erreicht.
Hinweis: Die Verdopplungszeit pathogener Leptospiren ist 12 bis 24 h, abhängig von der Sorte. - Impfen Sie 50 µL der Spender Escherichia coli Stamm β216320 der konduktiven Transposon Plasmid (pCjTKS2)16 in 5 mL von Luria Brühe (LB) ergänzt mit 0,3 mM 2,6-Diaminopimelicacid (DAP), 50 µg/mL zu tragen Kanamycin (Km) und 50 µg/mL Spectinomycin (Spc) und über Nacht in einem 37 ° C Inkubator bei 255 u/min statt.
- Impfen Sie 60 µL von E. Coli Zellen in 3 mL EMJH mit 0,3 mM der DAP (EMJH + DAP) ergänzt. Inkubation bei 37 ° C bei 255 u/min Agitation für 3-4 h bis zu einer OD600nm≈ 0,3.
- Um die Filtergruppe (Abbildung 1) zu montieren, stellen Sie den Fuß auf einen 125 mL Seitenarm Erlenmeyerkolben, positionieren Sie einen Acetat-Zellulose-Filter (Porengröße 0,1 mm; Durchmesser 25 mm) auf der Basis und Klemme den Trichter auf die Basis. Verbinden Sie die Filteranlage mit einer Vakuumanlage.
- Fügen Sie 5 mL Leptospira Spp. Kultur und 0,5 mL der E. Coli -Kultur in den Trichter. Saugen Sie die Flüssigkeit durch den Filter.
- Übertragen Sie den Filter mit der Oberfläche der Bakterien nach oben auf eine EMJH + DAPplate. Inkubation bei 30 ° C über Nacht mit dem Filter nach oben.
- Setzen Sie den Filter in einem 15 mL Röhrchen mit 1 mL EMJH und Vortex für 10 s, die Bakterien in den Medien zu veröffentlichen. 200 µL der Suspension auf 5 EMJH Platescontaining 50 µg/mL Km mit 10-15 1 mm sterile Glasperlen oder einer sterilen Einweg-Streuer zu verbreiten. Wickeln Sie die Platten mit Parafilm und 3 bis 4 Wochen, bis die Kolonien sichtbar werden kopfüber bei 30 ° C inkubieren.
- Übertragen Sie Kolonien einzeln in 3 mL EMJH mit 50 µg/mL Km (EMJH + Km) bei 30° C unter 150 u/min Agitation für 7 bis 10 Tage, bis die Kultur eine Dichte von ≈ 10 erreicht8/mL.
Hinweis: Kulturen können bei-80 ° C oder in flüssigem Stickstoff (mit 4 % Glycerin) gespeichert werden.
- Impfen Sie ein Volumen von exponentielle Phase Kultur der Pathogene Leptospira spp. in 10 mL Ellinghausen-McCullough-Johnson-Harris mittlerer (EMJH)18,19, 107 Zellen entspricht. Inkubation bei 30 ° C mit 150 u/min schütteln bis die Dichte 2-8 x 108 Zellen/mL erreicht.
-
Identifikation der Insertionsstelle Transposon von verschachtelten PCR (Abbildung 2( )
- Lösen Sie 50 µL jeder Transposon-Mutante in PCR Schläuche durch Inkubation bei 95 ° C für 15 Minuten.
Hinweis: DNA kann stattdessen mit einem DNA-Extraktion Kit gereinigt werden. - Vorbereiten des PCR-Mixes mit Deg1 und Tnk1 Primern (Tabelle 3) gemäß Tabelle 1. Transfer 23,7 µL des Mixes zu jedem PCR-Röhrchen und 1.3 µL lysierten Zellen hinzufügen. Führen Sie das Programm: 95° C für 5 min; 40 Zyklen: 95 ° C für 15 s, 40 ° C für 1 min, 72 ° C für 2 min; 72 ° C für 10 Minuten.
- Stellen des PCR-Mixes mit Tag und TnkN1 Primern (Tabelle 3) nach Tabelle 2. Übertragen Sie 24,2 µL des Mixes zu jedem PCR-Röhrchen und fügen Sie 0,8 µL des PCR-#1-Reaktion. Führen Sie das Programm: 95 ° C für 5 min; 35 Zyklen: 95 ° C für 15 s, 55 ° C für 30 s, 72 ° C für 2 min; 72 ° C für 10 Minuten.
- Laufen Sie 3 µL des PCR-Produkte auf einem 1 % Agarose-Gel mit 1 X Tris-Acetat-EDTA (TAE) Puffer mit 10-15 V/cm (Abb. 2 b).
- PCR-Produkte der positiven Proben mit einem PCR-Reinigung-Kit zu reinigen. Eluieren Sie DNA mit dem niedrigsten Volumen erlaubt das Kit um die Konzentration von DNA erholt zu maximieren.
- Senden Sie gereinigten PCR-Produkte für die Sanger-Sequenzierung mit dem TnkN1 Primer (Tabelle 3).
- Vergleicht man die resultierende Sequenz mit der elterlichen Belastung Genomsequenz von BLASTN Analyse (http://blast.ncbi.nlm.nih.gov/) oder über die SpiroScope Datenbank (http://www.genoscope.cns.fr/agc/mage)21identifizieren Sie Einfügung.
- Bestätigen Sie die Einstichstelle das Transposon durch PCR Zündkapseln Tempern zu flankierenden Sequenzen Host verwenden.
Hinweis: Das Transposon erhöht die Größe der Wildtyp Sequenz von ≈ 2 kb.
- Lösen Sie 50 µL jeder Transposon-Mutante in PCR Schläuche durch Inkubation bei 95 ° C für 15 Minuten.
(2) Tierversuch (Abbildung 3)
-
Kultur von Leptospiren Mutanten
- Wachsen Sie individuell jedem ausgewählten Transposon-Mutante in 10 mL EMJH + Km bei 30 ° C bei 150 u/min Agitation zu einer Dichte von 107-108 Leptospiren/mL.
- Rechnen Sie die Leptospiren durch Dunkelfeldmikroskopie mit einem Petroff-Hausser-Zähler oder wie beschrieben von Miller23.
- Jede Kultur in EMJH auf die gleiche Dichte, zum Beispiel 106 Zellen/mL zu verdünnen.
- Zur Montage am Eingang Pools vermischen Sie die verdünnten Kulturen in gleichen Volumina.
Hinweis: Enthalten Sie Steuerelemente im Eingang Pool durch Zugabe von Mutanten mit bekannten Fitness Mängel wie z. B. loa2217,24 und Mutanten mit unveränderten Fitness wie LigB17,25, bzw.. Pools von Mutanten können mit 4 % Glycerin bei-80 ° C oder in flüssigem Stickstoff gelagert werden.
-
Herausforderung
Hinweis: Die hier beschriebenen Methoden wurden von den Veterans Affairs größere Los Angeles institutionelle Animal Care und Use Committee (Protokoll #09018-14) genehmigt.- Intraperitoneal 1 mL des Eingabe-Pools für jedes Tier mit einer Insulin-Spritze U-100 mit 26 x ½" Nadel zu injizieren.
Hinweis: In den Pilotversuch wurden 8 Tiere mit 1 mL der Eingabe Pool, d. h. 106 Bakterien insgesamt17herausgefordert. Die Infektion war, durfte für 4 Tage vor der Euthanasie zu gehen. - Sammeln Sie 10 mL des Eingabe-Pools und spin für 20 min bei 3.220 x g. Entfernen Sie vorsichtig den Überstand ohne zu stören das Pellet. Speichern Sie die Zelle Pellet am - 80˚C bis zur Verwendung (Schritt 3.1.3).
- Beobachten Sie die Tiere täglich gekündigt am vorher festgelegten Endpunkt. Die Hamster täglich wiegen und Endpunkt Kriterien suchen: Appetitlosigkeit, Gang oder atmende Schwierigkeit, Niederwerfungen, gekräuselte Fell oder Verlust von > 10 % des maximalen Gewichts erreicht.
- Intraperitoneal 1 mL des Eingabe-Pools für jedes Tier mit einer Insulin-Spritze U-100 mit 26 x ½" Nadel zu injizieren.
-
In-vitro- experiment
- Am Tag der Herausforderung impfen Sie 3 Flaschen von 25 mL EMJH + Km mit 5 mL des Eingabe-Pools. Wachsen Sie Kulturen bei 30 ° C unter 150 u/min rühren.
- Zählen Sie die Leptospiren täglich mithilfe einer der Methoden der Abschnitt 2.1.2. Erreicht die Dichte ≈ 1 x 10-8/mL, spin-down jede Aussetzung für 20 min bei 3.220 x g.
- Speichern Sie die Zelle Pellets bei-80 ° C bis zur Verwendung.
-
Ernte und Lagerung der Gewebe
- Einschläfern Sie die Tiere durch Isofluran Inhalation gefolgt von bilateralen Thorakotomie26.
- Sammeln Sie 1 bis 2 mL Blut sofort durch Herzpunktion mit einer 3 mL Spritze und Nadel 25 X 5/8". Übertragen Sie das Blut in Röhrchen mit EDTA. 5 bis 6 Mal durch Inversion, mischen.
- Sammeln Sie eine Niere und ⅓ bis ½ der ventrale Median Leberlappen in Cryoröhrchen.
- Gewebe bei-80 ° C bis zu seiner Verwendung zu speichern.
3. Aufbau der genomischen Bibliotheken für Hochdurchsatz-Sequenzierung (Abbildung 4)
-
DNA-Extraktion
- DNA-Extraktion aus Blut.
- Übertragen Sie 100 µL Blut von EDTA-Röhrchen auf einem Microcentrifuge Schlauch.
- DNA mit Hilfe einen DNA-Extraktion Kit zu reinigen. Folgen Sie den Anweisungen des Herstellers.
- DNA-Extraktion aus den Geweben.
- Mit Skalpell und Schere, zwischen 50 bis 80 mg von jedem Organ in kleine Stücke (1 x 1 mm) in Würfel schneiden und in eine sterile Schraubverschluss Trockenrohr überführen. Messen Sie das Gewicht des Gewebes mit einer Präzisionswaage.
- Fügen Sie 500 µL steriler PBS in die Röhre.
- Homogenisieren von Proben mit den Disruptor für 1 min 5 Bewegungen pro Sekunde.
- Berechnen Sie das Volumen entspricht 25 mg des Gewebes unter Verwendung der folgenden Gleichung:
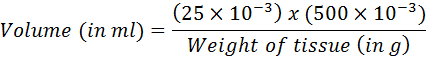
- Übertragen Sie das berechnete Volumen in der DNA-Extraktion-Kit. Gehen Sie mit DNA-Reinigung nach den Anweisungen des Herstellers.
- DNA-Extraktion aus der Eingabe Pool und in-vitro- Kulturen.
- Bakterielle Pellets bei Raumtemperatur für ca. 5-10 min Auftauen.
- Gehen Sie mit DNA-Extraktion, folgen Sie den Anweisungen begleiten das Kit.
- Shop-DNA bei-80 ° C bis zur Verwendung.
- DNA-Extraktion aus Blut.
-
Scheren der DNS (Abbildung 5( )
- Übertragen Sie 50 µL der extrahierten DNA auf einem 1,5 mL Microcentrifuge Schlauch.
- Legen Sie die Rohre in das Gestell des Sonikator Cup Horn gefüllt mit kaltem Wasser (4 ° C).
- Führen Sie die Sonikator für 3 min bei 80 % Intensität mit 10 s auf Puls und 5 s aus Puls.
Achtung: Tragen Sie Gehörschutz oder Ohrstöpsel, Gehörschutz. - 2.5 µL der geschert DNA auf einem 2 % Agarosegel zu bestätigen, dass ein Großteil der DNA ist < 600 bp in der Größe.
-
Ergänzung der C-Rute
- DNA-Konzentration zu messen, mit einem kleinen Volumen-Spektrophotometer.
- Berechnen Sie Volumen entspricht 500 ng DNA mithilfe der folgenden Gleichung:
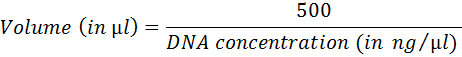
- Bereiten Sie Tailing Reaktion (Tabelle 4). Inkubieren Sie 1 h bei 37 ° C; bei 75 ° C für 20 Minuten inaktivieren.
Hinweis: Für Proben, die Anpassung an ein Volumen von mehr als 14,5 µL erfordern, der letzte Band der Reaktion auf 40 µL zu erhöhen und die restlichen Komponenten entsprechend skalieren. - Reinigen Sie die Proben mit einem PCR-Reinigung-Kit. Eluieren Sie die DNA mit 12 µL Elution Buffer.
-
Nested PCR
- PCR #1
- Bereiten Sie PCR Mischungen gemäß Tabelle 3 und 5. Übertragen Sie 22 µL des Bibliothek-Mix zu jedem PCR-Röhrchen und fügen Sie 3 µL gereinigte DNA. Gehen Sie ebenso mit dem Steuerelement-Mix.
Hinweis: Primer TnkN317 ist spezifisch für das Transposon und Grundierung olj3766 ist spezifisch für das C-Tail. Der Steuerelement-Mix fehlt die TnkN3 Grundierung, die richtet sich speziell an das Transposon. - Führen Sie das folgende Programm: 95 ° C für 2 min; 24-Zyklen: 95 ° C für 30 s, 60 ° C für 30 s, 72 ° C für 2 min; 72 ° C für 2 min.
- Bereiten Sie PCR Mischungen gemäß Tabelle 3 und 5. Übertragen Sie 22 µL des Bibliothek-Mix zu jedem PCR-Röhrchen und fügen Sie 3 µL gereinigte DNA. Gehen Sie ebenso mit dem Steuerelement-Mix.
- PCR #2.
- Bereiten Sie die zweite PCR Mischungen nach Tabellen 3 und 6. Übertragen Sie 49 µL des Bibliothek-Mix zu jedem PCR-Röhrchen und fügen Sie 1 µL des PCR #1 Bibliothek Reaktion. Übertragen Sie 24,5 µL des Kontrolle-Mix zu jedem PCR-Röhrchen und fügen Sie 0,5 µL des PCR #1 Kontrollreaktion.
Hinweis: Primer pMargent2 ist spezifisch für das Transposon und IP-Primer6 enthalten sechs Basenpaare Barcode und spezifische Sequenzen von Next Generation Sequencing-Plattform erkannt. - Führen Sie das folgende Programm: 95 ° C für 2 min; 18 Zyklen: 95 ° C für 30 s, 60 ° C für 30 s, 72 ° C für 2 min; 72 ° C für 2 min.
- Bereiten Sie die zweite PCR Mischungen nach Tabellen 3 und 6. Übertragen Sie 49 µL des Bibliothek-Mix zu jedem PCR-Röhrchen und fügen Sie 1 µL des PCR #1 Bibliothek Reaktion. Übertragen Sie 24,5 µL des Kontrolle-Mix zu jedem PCR-Röhrchen und fügen Sie 0,5 µL des PCR #1 Kontrollreaktion.
- Führen Sie 3 µL auf ein 2 % Agarose-Gel. Die Bibliothek sollte einen Abstrich mit der Mehrheit des Signals zwischen 200 bis 600 zeigen bp (Abbildung 6) und keine Verstärkung für die der Kontrollreaktion.
- PCR #1
-
Reinigung der PCR-Produkte.
Hinweis: Reinigen Sie die genomischen Bibliotheken mit einem PCR-Reinigung-Kit nach den Anweisungen des Herstellers. Eluieren Sie die DNA mit 30 µL Elution Buffer. - Messen Sie die DNA-Konzentration mit einem Fluorometer. Durchschnittlich 2 bis 3 mal gelesen.
- Berechnen Sie das Volumen jeder Bibliothek gleichbedeutend mit 15 oder 20 ng unter Verwendung der folgenden Gleichung:
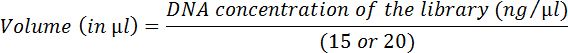
-
Mischen alle Bibliotheken zusammen nach den bisherigen Berechnungen. Bestimmen Sie die molare Konzentration der DNA mit der folgenden Website Gleichung:
http://www.MOLBIOL.edu.ru/eng/Scripts/01_07.HTML
Achtung: DNA-Konzentration und Volumen Anforderungen richten sich nach der Sequenzierung Plattform.
4. Hochdurchsatz-Sequenzierung und Analyse von Daten
- Sequenzierung
- Sequenz-Bibliotheken als 64 bp Einend-liest mit benutzerdefinierten Sequenzierung Primer pMargent3 und die standard kommerzielle Sequenzierung Grundierung. Die Sequenzierung Plattform bieten Ihnen FastQ-Dateien mit allen Sequenzierung liest.
- Analyse mit Galaxy-software
- Die Genom-Sequenz-Datei herunterladen
- In der SpiroScope-Datenbank-Homepage (siehe Punkt 1.2.7 für Link), wählen Sie den Organismus für das Experiment verwendet und klicken Sie auf "LOAD in Genom-BROWSER".
- Wählen Sie in der Symbolleiste (im oberen Bereich der Homepage), "Suche/Export > Daten herunterladen", klicken Sie in der Zeile "Sequence (Fasta)", "Genom" um die Sequenz zu laden.
- Öffnen Sie die Datei mit Notepad (PC) oder TextEdit (Mac) und benennen Sie das Chromosom (z. B. "ChrI"). Das Fasta-Format zu erhalten.
- Befolgen Sie die gleichen Schritte aus, um die Sequenz des Chromosoms II herunterladen. Kombinieren Sie beide Chromosom-Sequenzen in einer einzigen .txt-Datei durch Kopieren und einfügen.
Hinweis: Chromosom Sequenzen können auch von der NCBI-Website (https://www.ncbi.nlm.nih.gov) heruntergeladen und kombiniert in einer einzigen .txt-Datei.
- Hochladen von Dateien auf den Server von Galaxy
Hinweis: Galaxy ist ein open Source, Web-basierte Plattform für die Verwaltung der Daten intensive Bioinformatik Workflows27,28,29,30 und abrufbar unter https://usegalaxy.org/.- Wählen Sie im Menü "Extras", "Daten > Datei von Ihrem Computer hochladen". Ziehen Sie und legen Sie die .fastq Datei(en) erzeugt durch die Sequenzierung Plattform in das Fenster und klicken Sie auf "Start".
- Die gleichen Schritte um die Leptospiren Genom-Sequenz .txt Datei hochzuladen.
- Bräutigam liest
- Wählen Sie "NGS: QC und Manipulation > FASTQ Groomer" aus dem Menü "Extras".
- Neben Datei zu pflegen wählen Sie die Bibliotheken in Schritt 4.2.2 hochgeladen. Eingabe FASTQ Qualität Noten Typ wählen Sie das entsprechende Sequenzierung System. Verlassen Sie für erweiterte Optionen ausblenden Advanced Büro ausgewählt.
- Klicken Sie auf "Ausführen".
- Sequenzierung Artefakte zu entfernen
- Wählen Sie "NGS: QC und Manipulation > Sequenzierung Artefakte zu entfernen". Neben Bibliothek zu filtern, wählen die präparierten Dateien in Schritt 4.2.3 erzeugt. Klicken Sie auf "Ausführen".
- C-Tail-Sequenzen zu entfernen
Hinweis: Wiederholen Sie diese Schritte einmal oder zweimal darauf alle C-Tails entfernt werden.- Wählen Sie "NGS: QC und Manipulation > Clip Adapter Sequenzen".
- Wählen Sie oder geben Sie Folgendes ein:
Bibliothek zum Clip: Wählen Sie die Dateien in Schritt 4.2.4 erzeugt.
Minimale Sequenz Länge: 15
Quelle: Geben Sie eine benutzerdefinierte
Geben Sie eine benutzerdefinierte Clipping: CCCCCCC
Geben Sie Null-Wert zu halten die Adapter-Sequenzen und X-Basen, die ihm folgen: 0
Sequenzen mit unbekannt (N) Basen zu verwerfen: Ja
Ausgabeoptionen: Ausgabe abgeschnittene und nicht abgeschnitten-Sequenzen - Klicken Sie auf "Ausführen".
- Entfernen Sie Adapter Sequenzen
- Wählen Sie "NGS: QC und Manipulation > Clip Adapter Sequenzen".
- Wählen Sie oder geben Sie Folgendes ein:
- Bibliothek zum Clip: Wählen Sie im Schritt 4.2.5 erzeugte Dateien.
- Minimale Sequenz Länge: 15
- Quelle: Geben Sie eine benutzerdefinierte
- Geben Sie eine benutzerdefinierte Clip: CGTATGCCGTCTTCTGCTTG
- Geben Sie Null-Wert zu halten die Adapter-Sequenzen und X-Basen, die ihm folgen: 0
- Sequenzen mit unbekannt (N) Basen zu verwerfen: Ja
- Ausgabeoptionen: Ausgabe abgeschnittene und nicht abgeschnitten-Sequenzen
- Klicken Sie auf "Ausführen".
- Filter liest auf ihre Qualität
- Wählen Sie "NGS: QC und Manipulation >" Filtern nach "Qualität".
Hinweis: Dieses Tool wählt anhand von Qualitätskennzahlen liest. - Wählen Sie Folgendes aus:
Bibliothek zu filtern: Wählen Sie die Dateien in Schritt 4.2.6 erzeugt.
Qualität-Grenzwert: 20
Prozent der Basen in Reihenfolge, die Qualität muss gleich zu/höher als Cut-off-Wert: 95 - Klicken Sie auf "Ausführen".
Hinweis: Mit diesen Einstellungen werden Lesevorgänge kürzer als 20 Nukleotide oder mit einem Qualitätsfaktor von 20 oder weniger für 95 % der Zyklen verworfen. Passen Sie strenge Einstellungen zu Ihrem Experiment.
- Wählen Sie "NGS: QC und Manipulation >" Filtern nach "Qualität".
- Karte liest32
- Wählen Sie "NGS: Mapping > Bowtie2"32.
- Wählen Sie Folgendes in die Felder im Hauptfenster:
Ist dieser einzelne oder gekoppelte Bibliothek: Einend-
FASTQ Datei: Wählen Sie die Bibliothek für Qualität aus Schritt 4.2.7 gefiltert.
Schreiben (im Fastq Format) ausgerichteten liest um Dateien zu trennen: keine
Schreiben (im Fastq Format) ausgerichteten liest um Dateien zu trennen: keine
Sie wählen Sie eine Referenz-Genom aus Ihrem Verlauf oder verwenden Sie einen integrierten Index?: verwenden ein Genoms aus der Geschichte und bauen Index
Wählen Sie die Referenz-Genom: Wählen Sie die Leptospiren genome.txt Datei in Schritt 4.2.2 hochgeladen.
Gruppe Gruppen Informationen gelesen?: nicht festgelegt
Wählen Sie Analysemodus: 1: Standardeinstellung nur
Möchten Sie Presets benutzen?: Nein, nur die Standardeinstellungen verwenden
Speichern Sie die bowtie2 Zuordnung Statistiken zur Geschichte: Nein
Job-Ressourcenparameter: Job Ressource Standardparameter verwenden - Klicken Sie auf "Ausführen" um Lesezugriffe auf das Genom auszurichten.
- Dateien konvertieren
- Wählen Sie "NGS: SAMtools > BAM-SAM SAM BAM umwandeln".
- Wählen Sie Folgendes aus:
BAM-Datei zu konvertieren: Wählen Sie Bibliothek aus Schritt 4.2.8 zugeordnet.
Kopfzeilen-Optionen: Header beinhalten, in der SAM-Ausgabe (-h) - Klicken Sie auf "Ausführen".
- Dateien konvertieren
- Wählen Sie "NGS: SAMtools > Intervall SAM umwandeln".
- Wählen Sie Folgendes aus:
Dataset konvertieren auswählen: Wählen Sie die SAM-zugeordnete Bibliothek-Datei in Schritt 4.2.9 erzeugt.
Drucken Sie alle?: Ja - Klicken Sie auf "Ausführen".
- Art mal gelesen
- Wählen Sie "Filtern und sortieren > Daten in aufsteigender oder absteigender Reihenfolge sortieren".
- Wählen Sie Folgendes aus:
Art-Dataset: Wählen Sie die Intervall-Datei in Schritt 4.2.10 erzeugt.
Spalte: 2
mit Geschmack: Lfd.
alles in: aufsteigend - Klicken Sie auf "Ausführen".
- Wählen Sie passende Chromosom liest ich
- Wählen Sie "Filtern und sortieren > Wählen Sie Zeilen, die einen Ausdruck entsprechen".
- Wählen Sie Folgendes aus:
Wählen Sie Zeilen aus: Wählen Sie Datei mit sortierten liest im Schritt 4.2.11 erzeugt.
: Matching
das Muster: Geben Sie den Namen des Chromosoms, die ich als entschlossen im Schritt 4.2.1.3 (z. B. "ChrI"). - Klicken Sie auf "Ausführen".
- Wählen Sie liest übereinstimmenden Chromosom II
4.2.12 Schritt folgen. - Gruppe, die nach den Standorten der Einfügungen in Chromosom ich liest
- Wählen Sie ", subtrahieren und Gruppe beitreten > Daten nach einer Spalte zu gruppieren und aggregieren Vorgang für andere Spalten".
- Wählen Sie Folgendes aus:
Daten auswählen: Wählen Sie die resultierende Datei aus Schritt 4.2.12.
Gruppieren nach Spalte: 2
Ignorieren und Gruppierung?: Nein
Zeilen beginnen mit diesen Zeichen zu ignorieren: Ø
Betrieb > + Einfügevorgang
Typ: Graf
Spalte: 2
Ergebnis auf die nächste ganze Zahl runden: Nein - Klicken Sie auf "Ausführen".
- Gruppe nach den Standorten der Einfügung in Chromosom II liest
4.2.14 Schritt folgen. - Art-Einfügung sites auf Chromosom ich
- Wählen Sie "Filtern und sortieren > Daten in aufsteigender oder absteigender Reihenfolge sortieren".
- Wählen Sie Folgendes aus:
Art-Dataset: Wählen Sie Datei aus Schritt 4.2.14.
Spalte: 1
mit Geschmack: Lfd.
alles in: aufsteigend - Klicken Sie auf "Ausführen".
- Sortieren Sie Einfügung Websites auf Chromosom II
4.2.16 Schritt folgen.
- Die Genom-Sequenz-Datei herunterladen
- Statistische Analyse
- Die Datenübertragung Galaxy in Tabellenkalkulationsdateien durch Kopieren und einfügen die zwei Spalten von Schritten 4.2.16. und 4.2.17. in einer Excel-Datei.
Hinweis: Die erste Spalte ist die Nukleotid-Koordinate der Insertionsstelle Transposon, und die zweite Spalte ist die Anzahl der Lesevorgänge an jedem Standort einsetzen. - Das Gen beherbergen das Transposon mit der Nukleotid-Koordinate in der Tabelle enthaltenen zu identifizieren.
Hinweis: zum Beispiel ein Transposon eingefügt an Nukleotid #30718 des Chromosoms I ist befindet sich im LIC10024 -gen, die Nukleotide 29263-31539 in Chromosom überspannt ich. - Berechnen Sie relative Häufigkeiten (F) für jede Mutant in jedem Gewebe und in den Eingang Pool nach untenstehender Formel:
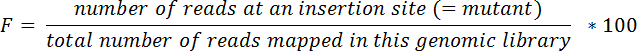
- Berechnen Sie Output/Input-Verhältnis (R) für jede Mutante und Gewebe mit Hilfe der folgenden Gleichung:
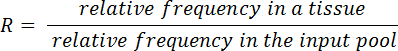
- Wilcoxon-unterzeichnet-Rank-test
- Normalisieren Sie alle Kennzahlen der Ausgang/Eingang durch das mittlere Verhältnis für jedes Gewebe in jedes Tier auf 1,0 festlegen. Ein Verhältnis von 1,0 ist neutral, > 1.0 ist nachteilig, und < 1.0 ist vorteilhaft33.
- Ausgang/Eingang Verhältnisse bis 1.0 (neutrale Fitness) zu vergleichen mit dem Wilcoxon-Rang-Test mit P-Werten < 0,05 als statistisch signifikant angesehen.
- Die Datenübertragung Galaxy in Tabellenkalkulationsdateien durch Kopieren und einfügen die zwei Spalten von Schritten 4.2.16. und 4.2.17. in einer Excel-Datei.
Ergebnisse
Aufbau einer Bibliothek von Transposon Mutanten in L. Interrogans durch Konjugation erfordert eine Filteranlage, wie in Abbildung 1dargestellt. Von jeder Paarung erholt wir 100-200 Transconjugants.
Transposon Insertionsstelle in jeder Mutant durch Sequenzierung das PCR-Produkt erzeugt durch semi-random PCR, die zum Jahresende das Transposon Ziel identifiziert und angrenzenden Host-Sequenzen...
Diskussion
Zwar ergibt sich aus unseren Pilotversuch für Hamster mit 42 L. Interrogans Mutanten intraperitoneal herausgefordert17präsentiert werden, erwarten wir, dass größere Pools von Mutanten durch Tn-FF abgeschirmt werden können Da die Frequenz der Transconjugants niedrig ist (100-200 Transconjugants/Paarungen), mehrere Paarungen sind notwendig, um eine ausreichende Anzahl von Mutanten für große Tn-Seq-Experimente zu erzeugen. Eine große Anzahl von Mutanten in flüssigen Kulturen pflegen...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde unterstützt durch eine Veterans Affairs Merit Award (für D.A.H.) und eine National Institute of Health gewähren R01 AI 034431 (D.A.H.).
Materialien
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
Referenzen
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten