Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bewertung von In-vitro- DNA-Schäden mit Comet-Assay
In diesem Artikel
Zusammenfassung
Die Comet-Assay ist eine effiziente Methode um zu erkennen, dass DNA-Schäden einschließlich Einzel- und Doppelstrang-DNA bricht. Wir beschreiben basische und neutrale Comet Assays zur Messung der DNA-Schäden in den Krebszellen, die therapeutische Wirkung der Chemotherapie zu bewerten.
Zusammenfassung
DNA-Schäden ist ein weit verbreitetes Phänomen für jede Zelle während seiner Lebensdauer und ist definiert als eine Veränderung der chemischen Struktur von genomischer DNA. Krebs-Therapien, wie z. B. Strahlen- und Chemotherapie, stellen enorme zusätzliche DNA-Schäden, was zu Zellzyklus Festnahme und Apoptose zu Krebsentwicklung zu begrenzen. Quantitative Bewertung von DNA-Schäden während experimentelle Krebstherapie ist ein wichtiger Schritt um die Wirksamkeit eines genotoxischen Agenten zu rechtfertigen. In dieser Studie konzentrieren wir uns auf eine einzelne Zelle Elektrophorese Assay, auch bekannt als die Comet-Assay, die einzigen quantifizieren kann und Doppel-Strang DNA bricht in Vitro. Die Comet-Assay ist ein DNA-Schäden Quantifizierungsmethode effizient und einfach durchzuführen, und hat geringen Zeitbudget/Ansprüche und hohe Reproduzierbarkeit. Hier heben wir das Dienstprogramm des Comet-Assays für einer präklinischen Studie durch die Auswertung der gentoxischen Wirkung von Olaparib/Temozolomid Kombinationstherapie U251 Gliom Zellen.
Einleitung
Die Comet-Assay wurde zuerst von Ostling und Johanson im Jahr 1984 durch den Nachweis, dass die Migration der DNA Fragmente von Kernen in einem neutralen Zustand1entwickelt. Die Technik wurde später von Singh Et Al., zeigen, dass eine alkalische Zustand wesentlich die Spezifität und Reproduzierbarkeit der Assay-2 erhöhtentwickelt. Seitdem die neutrale Comet-Assay ist meist verwendet, um doppelsträngige DNA-Brüchen, zu erkennen, während die alkalischen Comet-Assay empfindlicher für kleinere Mengen von DNA-Schäden ist, darunter Einzel- und Doppelstrang DNA, Alkali labil Websites, DNA-DNA bricht oder Vernetzung der DNA-Protein und DNA-Single-Strangbrüchen verbunden mit unvollständiger Exzision reparieren Websites3,4. Beide Assays ermöglichen die Visualisierung von fragmentierten DNA und bieten eine einfache Möglichkeit, DNA-Schäden quantitativ zu bewerten. Die Comet-Assay gilt als eine empfindliche Methode für in Vitro und in Vivo genetische toxikologischen Studien und ist anwendbar auf verschiedenen Forschungsbereichen wie frühe Arzneimittelkandidat Selektion, Umweltüberwachung, human-Biomonitoring, und grundlegende Forschung auf DNA-Schädigung und Reparatur5.
Das Prinzip des Tests ist, dass unter ein elektrisches Feld, fragmentierte DNA wandert aus dem Nucleoid Körper (auch bekannt als "Komet Kopf") und bildet einen DNA-Fleck in der Agarosegel (auch bekannt als der "Kometenschweif"). Mit Nukleotid Färbung, kann das Ausmaß der DNA-Schädigung durch Analyse "Kometen" durch diese einzelne Zelle Elektrophorese gebildet quantifiziert werden. Berechnung des Augenblicks Tail kann weiterhelfen, um DNA-Schäden unter verschiedenen experimentellen Gruppen zu vergleichen. Verglichen mit traditionellen Methoden der DNA-Schäden-Erkennung, ist der Comet-Assay direkter, sensibler, kostengünstig und relativ einfach.
Strahlentherapie und Chemotherapie sind gemeinsame Strategien zur Behandlung von Krebs durch die Generierung von Einzelstrang und Doppelstrang DNA Brüche in Chromosomen6. Der aktuelle Fortschritt in der DNA-Reparatur-Inhibitoren ermöglicht eine effektivere genotoxische Wirkung von Kombinations-Chemotherapie und daher potentiell reduziert systemischen Nebenwirkungen wie Anämie, Infektionen und Knochenmark Unterdrückung7, 8. in dieser Studie zeigten wir die Untersuchung von einem Poly (ADP-Ribose) Polymerase (PARP) Inhibitor Olaparib (Ola)9. PARP ist ein reichlich nuklearen Protein und ist verantwortlich für DNA-base Excision Repair durch die Bildung einer Poly (ADP-Ribose) Polymer-10. Temozolomid (TMZ) ist ein Oral verfügbaren Alkylierungsmittel und weithin für Gliom Patientenbehandlung. Durch die Verwendung der Comet-Assay, um DNA-Schäden zu quantifizieren, zeigen wir, dass die Kombination von Olaparib mit Temozolomid zutiefst DNA-Schäden in den Zellen Gliom, erhöht, was darauf, dass Olaparib/Temozolomid Kombinationstherapie ist eine wirksame Strategie zur Gliom, zu behandeln hindeutet, als im Vergleich mit Temozolomid allein11.
Protokoll
1. bereiten Reagenzien
- 1 X PBS
- 100 mL 10 X PBS mit 900 mL dH 2 O verdünnen und anpassen den pH-Wert 7,4 mit pH-Meter. Bei Raumtemperatur lagern.
- Lyse Lösung (LS)
- bereiten 2,5 M NaCl, 100 mM Binatrium-EDTA, 10 mM Tris-Base und 200 mM NaOH in 900 mL dH 2 O; häufig dauert es etwa 20 min zu der Mischung vollständig auflösen lassen. Stellen Sie den pH-Wert bis 10 mit dem pH-Meter. Fügen Sie 1 % Natrium Natriumlaurylsulfat Sarcosinate und 1 % Triton x-100, und die letzte Lautstärke bis 1.000 mL. Cool, 4 ° C für mindestens 30 min vor Gebrauch.
- Elektrophorese alkalische Lösung (AES), pH > 13
- bereiten 200 mM NaOH und 1 mM Binatrium EDTA in 800 mL dH 2 O. Stellen Sie den pH-Wert und stellen Sie sicher, dass es pH ist > 13. Die letzte Lautstärke bis 1.000 mL. Vor Gebrauch frisch machen und auf 4 ° C für mindestens 30 min vor Gebrauch abkühlen lassen.
- Neutral Elektrophorese Sollution (NES)
- bereiten Sie 1.000 mL neutral Elektrophorese Puffer durch das Mischen von 100 mM Tris-Base und 300 mM Natriumacetat auf 1.000 mL dH 2 O. Stellen Sie des pH-Werts auf 9,0 mit Eisessig. Cool, 4 ° C für mindestens 30 min vor Gebrauch.
- DNA Niederschlag Lösung (DPS)
- Vorbereitung von 10 mL 7,5 M Ammonium Acetat Brühe. Mischen Sie für 50 mL DNA Niederschlag Lösung 6,7 mL 7,5 M Ammoniumacetat mit 43,3 mL 95 % igem Ethanol. Bei Raumtemperatur lagern.
- Retikuläres Lösung
- Add 1 µL 10.000 x grün fluoreszierenden Nukleinsäure-Fleck (z. B. SYBR Green) in 30 mL Tris-EDTA Puffer (10 mM Tris-HCl, 1 mM Binatrium-EDTA, pH 7,4) und Store bei 4 ° C. Protect aus Licht.
- 1 % niedrig schmelzende Agarose
- 1 % niedrig schmelzenden Punkt Agarose (1 g in 100 mL dH 2 O) in der Mikrowelle schmelzen. Schwenken Sie die Agarose alle 15-20 s, um sicherzustellen, dass die Agarose vollständig geschmolzen ist. Legen Sie die Agarose in 37 ° C Wasserbad für mindestens 20 Minuten vor dem Gebrauch.
- Pre-warme Pipettenspitzen
- schneiden Sie die schmalen Enden der P200 Pipettenspitzen von 3 mm und bei 37 ° C warmen vor Pipettieren Agarose.
2. Bereiten Sie Comet gleitet
- Slide Beschichtung
- Schmelze 1 % Agarose (1 g in 100 mL dH 2 O) in der Mikrowelle für 2 – 3 min. oder bis die Agarose vollständig geschmolzen ist. Tauchen Sie die Glas-Objektträger in der Agarose und einer Seite der Folie mit einem fusselfreien Tuch abwischen.
- Legen Sie die Folien auf einer flachen Oberfläche an der Luft trocknen oder Erhitzen bei 50 ° C für schnelleres Trocknen, ein transparente Agarose-Film sollte nach dem Trocknen gebildet werden. Legen Sie die beschichteten Folien in 37 ° C vor dem Gebrauch.
- Vorbereitung der einzelnen Zellsuspensionen
- Kultur und Genuss der Gliom-Zelle
- die U251 MG Kulturzellen in DMEM-Ham f-12 Medium ergänzt mit 10 % FBS, 100 U/mL Penicillin und 10 µg/mL Streptomycin bei 37 & # 176; C mit 5 % CO 2.
- Der Zellen mit 1 mL Trypsin für 3 min zu verdauen und Trypsin mit DMEM-Ham f-12 Medium mit FBS neutralisieren. Sammeln in 15 mL-Tube mit 300 X g für 4 min drehen, aspirieren Sie das Medium und Sperren von Zellen bei 2 x 10 5 Zellen/mL in 1 X PBS.
Hinweis: Die Zellprobe sollte unmittelbar vor Beginn des Tests vorbereitet sein und alle Proben in einem dunklen oder abgeblendet Umfeld zur Vermeidung von DNA-Schäden aus Licht behandelt werden. - Kombinieren die Zellsuspension mit 1 % geschmolzenen niedrigen Schmelzpunktes Agarose (bei 37 ° C) im Verhältnis 01:10 (V/V), von oben und unten Pipettieren vorsichtig mischen und sofort pipette 30 µL in eine Folie. Verwenden Sie die Seite von der Pipettenspitze verbreiten die Agarose/Zelle Mischung um die Bildung einer dünnen Schicht gewährleisten.
- Legen Sie die Folie flach bei 4 ° C im Dunkeln für 10 min. erhöht die Vergelung Zeit bis 30 min Einhaltung von Proben in Umgebungen mit hoher Luftfeuchtigkeit verbessert.
- Tauchen die Folie in 4 ° C im Dunkeln für 1 h über Nacht LS.
- Kultur und Genuss der Gliom-Zelle
3. Einzelne Zelle Elektrophorese
- weiter zu alkalisch (Schritt 3.2) oder Neutral (Schritt 3.3) Comet assay
- für alkalischen Comet Assay
- Entfernen Sie vorsichtig Folien aus dem LS, abtropfen überschüssigen Puffer und sanft Tauchen in AES für 1 h bei 4 ° C erlauben DNA abwickeln. Halten Sie die Folien in der Dunkelheit.
- Add vorgekühlt AES in der Elektrophorese Diahalter nicht mehr als 0,5 cm über die Folien (Dies hängt von der Größe der Elektrophorese Einheiten), legen Sie die Folien innerhalb und mit einer Kappe bedecken. Die Versorgungsspannung auf 1 V/cm (die Länge zwischen den Elektroden) und laufen für 30 min bei 4 ° c
- Abfluss überschüssiger Elektrophorese Lösung aus Folie. Vorsichtig Tauchen Folien zweimal in dH 2 O 5 min bei Raumtemperatur.
- Tauchen sanft Folien in 70 % igem Ethanol für 5 min bei Raumtemperatur. Fahren Sie mit Schritt 4.
- Für neutrale Comet Assay
- Entfernen Sie vorsichtig die Folien aus dem LS, abtropfen überschüssigen Puffer und sanft Tauchen in NES für 30 min bei 4 ° c halten Sie die Folie im Dunkeln.
- Add vorgekühlt neutral Elektrophorese Puffer in der Elektrophorese Diahalter nicht mehr als 0,5 cm über Folien (Dies hängt von der Größe der Elektrophorese Einheiten), legen Sie die Folien innerhalb und mit einer Kappe bedecken. Die Versorgungsspannung auf 1 V/cm (die Länge zwischen den Elektroden) und laufen für 45 min bei 4 ° c
- Abfluss überschüssigen Puffer von den Folien. Vorsichtig Tauchen Folien in DPS für 30 min bei Raumtemperatur.
- Tauchen sanft Folien in 70 % igem Ethanol für 30 min bei Raumtemperatur. Fahren Sie mit Schritt 4.
4. Fleck Comet Folien
- Folien bei 37 ° C für 10-15 min im Dunkeln trocknen.
- Platz 50-100 µL grün fluoreszierenden Nukleinsäure Färbelösung auf jede getrocknete Agarose und Fleck für 15 min bei Raumtemperatur im Dunkeln.
- Folien in dH 2 O kurz abspülen und trocknen komplett bei 37 ° C im Dunkeln. Weiter zur Bildaufnahme und -Analyse.
5. Bild Erfassung und Analyse
Hinweis: die Visualisierung und Quantifizierung von DNA-Brüchen basieren auf Epifluoreszenz Mikroskopie und die Comet-Assay-Software (siehe Tabelle der Materialien) 12 .
- Legen Sie die Folien auf das Mikroskop mit einem Diahalter. Stellen Sie sicher, dass das Agarosegel auf das Objektiv gerichtet ist. Nach dem Zufallsprinzip die Aufnahmen von den gefärbten Komet-Folien mit dem Fluoreszenzmikroskop mit einem 10 X-Objektiv. Die Kanten und die Gebiete rund um Luftblasen zu vermeiden.
- Stellen Sie sicher jeder Kometenschweif horizontal verteilt wird. Komet Köpfe sollten stammen aus der linken Seite und das Heck von der rechten Seite.
- Speichern Sie jedes Bild in einem binären TIF-Format mit hellen Fleck DNA und dunklem Hintergrund. Laden Sie Bilder in die Software mit der " wählen Sie Dateien zu analysieren " Taste, die auf der linken Seite der Symbolleiste befindet. Fenster ein Bild sollte angezeigt werden ( Abbildung 1).
- Ein Messrahmen auf den Bildschirm zeichnen und seine Größe im Einklang mit der Komet der Zelle anpassen. Klicken Sie auf die " Adjust " Taste, um die Schwelle des Kopfes, Kometen und Rute nach dem Bild einrichten, und klicken Sie dann auf die " Messungen beginnen " Taste ( Abbildung 1).
- Wählen Sie eine Zelle mit dem Rahmen und die Messung durch Anklicken mit der Maus aktivieren die " die Comet Assay " Taste; eine Intensität Bild zeigt sich auf der " Profile " Fenster mit der ausgewählten Messparameter. Die Ergebnisse können gespeichert werden, indem Sie auf das " Ergebnis speichern " Taste ( Abbildung 1).
Hinweis: Die Software berechnet die Parameter einschließlich der Länge der Kometenschweif, der Prozentsatz der tailed DNA, die Rute Moment (TM) und der Olive Schweif Moment (OTM). Die Rute Momente werden berechnet, indem die Formeln wie folgt:

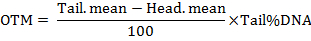
- mindestens 50 Zellen zu analysieren pro Behandlung.
Ergebnisse
Dieses Protokoll beschreibt Schritt für Schritt Workflow für die Comet Assay Ausführung und Datenanalyse (Abbildung 1). Ergebnisse aus der basischen und neutralen Comet-Assays zeigten, dass der Kometenschweif von Doxorubicin behandelt U251 Zellen (1 µM, 20 h) länger war und hatte eine höhere Intensität der DNA, schlägt eine beträchtliche Ansammlung von fragmentierten DNA aufgrund von Chemotherapie (Abbildung 2).
Diskussion
Die Comet-Assay ist ein effizientes Werkzeug, einzelne- und Doppel-Strang DNA-Brüchen auf zellulärer Ebene zu messen. Der Test wurde weithin als "golden Standard" in Studien zur Genotoxizität und Biomonitoring13, angefangen von base Läsionen, DNA-Querverbindungen, Arzneimittelentwicklung und Alkali-empfindlichen Stellen angewendet. In der vorliegenden Studie zeigten wir zwei unterschiedliche schrittweise Protokolle für alkalische und neutrale Comet Assays, beziehungsweise. Einzelne Zelle Elek...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Forschung wurde durch das Intramurale Forschungsprogramm des NIH, NCI und CCR unterstützt. Alle Autoren erhielt Intramural Research Grant von NIH, NCI und CCR.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
Referenzen
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment?. Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157 (2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten