Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Einzellige Quantifizierung der Protein-Abbauraten von Time-Lapse Fluoreszenzmikroskopie in adhärente Zellkultur
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine Methode zum Bestimmen von Protein in einzelne lebende adhärente Zellen, Halbwertszeiten mit Puls Kennzeichnung und Zeitraffer Fluoreszenz-Bildgebung von Fusionsproteinen SNAP-Tag.
Zusammenfassung
Proteine sind in einem dynamischen Zustand der Synthese und Degradation und ihre Halbwertszeiten können unter verschiedenen Umständen angepasst werden. Jedoch am häufigsten verwendeten Ansätze um zu ermitteln Protein Halbwertszeit sind entweder beschränkt auf Bevölkerung Durchschnitte aus lysierten Zellen oder erfordern den Einsatz von Protein-Synthese-Hemmer. Dieses Protokoll beschreibt ein Verfahren zur Messung von Protein-Halbwertszeiten in einzelne lebende adhärente Zellen mit SNAP-Tag Fusionsproteine in Kombination mit Zeitraffer-Fluoreszenzmikroskopie. Jedes Protein des Interesses verschmolzen zu einem Kinderspiel-Tag kann kovalent an ein, Zelle durchlässig Fluoreszenzfarbstoff, der gekoppelt ist ein Benzylguanine Derivat gebunden, und der Zerfall der beschrifteten Protein Bevölkerung nach Auswaschung der restliche Farbstoff überwacht werden kann. Nachfolgende Zelle tracking und Quantifizierung der integrierten Fluoreszenzintensität über Zeit Ergebnisse in einer exponentiellen Zerfall Kurve für jede überwachte Zelle, so dass für die Bestimmung von Protein Abbauraten in Einzelzellen durch Kurvenanpassung. Diese Methode liefert eine Schätzung für die Heterogenität der Halbwertszeiten in einer Population von kultivierten Zellen, die leicht durch andere Methoden bewertet werden kann. Der hier vorgestellte Ansatz gilt für jede Art von kultivierten adhärente Zellen ein Protein des Interesses verschmolzen zu einem Kinderspiel-Tag zum Ausdruck zu bringen. Hier verwenden wir Mauszellen embryonaler Stammzellen (ES) auf E-Cadherin-beschichtete Kultur Zellplatten angebaut, wie einzelne Zelle Abbau illustrieren Preise von Proteinen mit einer breiten Palette von Halbwertszeiten ermittelt werden können.
Einleitung
Es ist bekannt, dass zelluläre Proteine umfangreiche Umsatz mit Synthese und Abbau spezifisch für jedes Protein und einer physiologischen Regulierung zu unterziehen. Traditionell wurden Protein Abbauraten mit Bulk-Methoden wie radioaktive Puls Chase Analyse, oder an denen Protein-Synthese-Hemmer wie Cycloheximide1gemessen. Vor kurzem wurde stabiler Isotope Etikettierung mit Aminosäuren in der Zellkultur (SILAC) in Kombination mit der Massenspektrometrie gegründet, um Protein-Umsatz auf einer globalen Skala2zu quantifizieren. Aber diese Methoden sind begrenzt, indem der Durchschnitt der Bevölkerung, und Informationen über Zell-Zell-Variabilität geht also verloren. Darüber hinaus können vorübergehende Änderungen im Proteinabbau, die in der Zellenbevölkerung unsynchronized sind identifiziert werden.
Protein-Halbwertszeiten können alternativ auch durch Fluoreszenz-basierte Ansätze, die haben oft den Vorteil, dass einzellige Auflösung ermittelt werden. Zum Beispiel wurde ein Photoactivatable grün fluoreszierendes Protein (PaGFP) zur Oct4 Halbwertszeit in den frühen Säugetieren Embryo3zu bestimmen. Eine weitere Methode zur Protein-Zerfall in lebenden Zellen zu überwachen ist die Verwendung eines SNAP-Tags in Kombination mit Zeitraffer-Fluoreszenz-Bildgebung. Der SNAP-Tag ist eine mutierte Version von der DNA-Reparatur Enzym O6- Alkylguanine DNA-Alkyltransferase (AGT), die speziell mit Benzylguanine (BG) Derivate, reagiert die molekularen Sonden4,5, gekoppelt werden können 6. daher SNAP-Tag-Fusionsprotein kann irreversibel mit einer, Zelle durchlässig Leuchtstofffärbung beschriftet werden. Puls Kennzeichnung eines Proteins des Interesses mit der SNAP-Tag, gefolgt von Auswaschung der restliche Farbstoff verschmolzen ermöglicht für die Überwachung des Abbaus der beschrifteten Protein Bevölkerung und damit für die Bestimmung von Protein-Halbwertszeit. SNAP-Tags haben erfolgreich eingesetzt für Puls-Jagd Kennzeichnung von Proteinen und für die Bestimmung von Protein Halbwertszeiten im Anhänger Zell-Kultur und in Vivo5,7,8,9. Eine Vielzahl von SNAP-Tag Substrate für gebräuchliche fluoreszierende Spektren sind im Handel erhältlich, so dass die Auswahl der optimalen Farbstoff für jede spezifische Anwendung. SNAP-Tags können somit auch für mehrfarbige Bildgebung in Kombination mit anderen fluoreszierende Fusionsproteine oder Farbstoffe verwendet werden. Zelle-undurchlässig Farbstoffe eignen sich für die Kennzeichnung von Membran-angebunden Proteine während Zelle durchlässig Farbstoffe für die Überwachung der intrazellulären und Membrane-springen Proteine anwendbar sind. Darüber hinaus einige der diese Sonden fast keine basale Fluoreszenz ausstellen und erst emittierende eine starke Fluoreszenzsignal nach Bindung an ein SNAP-Tag10.
Dieses Protokoll beschreibt, wie die Abbauraten verschiedener Proteine des Interesses an einzelne Zellen mit einem SNAP-Tag zu messen. Hier wenden wir diese Methode auf embryonale Stammzellen (ES) Mauszellen kultivierten auf E-Cadherin, aber es sollte möglich sein, es mit jeden anhaftende kultivierte Zelltyp verwenden. Wir zeigen, dass Puls Kennzeichnung von Fusionsproteinen SNAP-Tag gefolgt von Time-Lapse Fluoreszenz-Bildgebung ermöglicht die Bestimmung der Einzelzelle Halbwertszeiten von Interesse verschiedener Proteine und eine Schätzung für die Zell-Zell-Variabilität der Halbwertszeiten in liefert eine Bevölkerung von kultivierten Zellen.
Protokoll
Hinweis: In dieser Studie wurde die E14 ES-Zell-Linie verwendet. Dieses Protokoll ist jedoch auf andere Maus-ES Zell-Linie mit dem Ausdruck ein Protein des Interesses verschmolzen zu einem SNAP-Tag, entweder durch das körpereigene Protein tagging oder mittels Überexpression unmittelbar anwendbar. Für die Beispiele in den Abschnitt "Ergebnisse" wurden Doxycyclin-induzierbaren SNAP-Tag-Fusion-Zelllinien verwendet (SNAP-Tag verschmolzen, die folgende Proteine: Nanog, Oct4, Srsf11, oder die fluoreszierende Proteine mOrange2 und SfGFP und setzen Sie unter der Kontrolle der ein Doxycyclin-induzierbaren Promoter. Die Plasmide verwendet für die Generation der Doxycyclin-induzierbaren SNAP-Tag-Fusion-Zell-Linien11 für weitere Informationen siehe). Doxycyclin-induzierbaren Systems kann besonders nützlich sein, da es erlaubt für streng zu kontrollieren, den Zeitpunkt und die Intensität des Ausdrucks des Proteins des Interesses. C-terminale Positionierung des SNAP-Tag wird empfohlen, wie das Ändern der N-terminalen Aminosäure-Sequenz ist wahrscheinlicher, die Halbwertszeit des Zielproteins (N-Seite Regel12) zu ändern.
1. E-Cadherin-Beschichtung und Zelle seeding
- Generieren Sie eine ES-Zell-Linie mit dem Ausdruck ein Protein des Interesses verschmolzen zu SNAP-Tag4.
- Mantel 100 mm Zelle Kultur Gerichte mit 4 mL 0,1 % Gelatine (verdünnt in Phosphat gepufferte Kochsalzlösung (PBS) ohne Ca2 +/Mg2 +) für 1 h Gelatine zu entfernen und sofort für 1 h Nutzung trocknen lassen oder die beschichteten Speisen für bis zu 2 Monate zu speichern.
- Wachsen Sie die Zellinie interessiert ES Zellkulturmedium (Glasgow Minimum wesentliche Mittel, mit 10 % ES Zelle qualifiziert fötalen Rinderserum, 2 mM L-Glutamin, 100 µM 2 - 2 mM Natrium Pyruvat, 1 % nicht-essentiellen Aminosäuren, 1 % Penicillin/Streptomycin ergänzt Mercaptoethanol, Leukämie hemmenden Faktor (LIF), 3 µM CHIR99021 und 0,8 µM PD184352) auf Gelatine beschichtet Gerichte bei 37 ° C und 5 % CO2 für 2 Tage bis zum Erreichen eines Zusammenfluss von 10-20 Mio Zellen pro Schale.
Hinweis: Hier produzierte LIF transiente Transfektion von HEK 293T Zellen, gefolgt von überstand Sammlung und filtern. Jede Charge von LIF wurde für sein Potenzial zur Aufrechterhaltung der Pluripotenz getestet, aber Konzentrationen wurden nicht bestimmt. Jedoch rekombinante LIF ist auch im Handel erhältlich und die Konzentration häufig verwendet für Maus-ES Zellkultur 1000 Einheiten/mL13. - Mantel einer 96-Well-Platte geeignet für imaging mit 30 µL des rekombinanten Maus E-Cadherin Fc Chimäre Protein (E-Cad-Fc) pro Well (5 ng/µL, verdünnt in PBS mit Ca2 +/Mg2 +. Verwenden Sie Lager Konzentration von 100 ng/µL, bei-80 ° C gelagert). Vermeiden Sie umfangreiche pipettieren, wie E-Cad-Fc sehr zerbrechlich ist. Inkubation bei 37 ° C für 1,5 h.
Hinweis: Je nach dem Mikroskop könnte andere Formate verwendet werden. - Aspirieren Sie E-Cad-Fc, einmal mit 100 µL PBS mit Ca2 +/Mg2 +waschen Sie und fügen Sie 100 µL vorgewärmten ES Zellkulturmedium hinzu.
- Waschen Sie die Zellen des Interesses mit 5 mL PBS ohne Ca2 +/Mg2 +. Aspirieren Sie und 2 mL Trypsin-EDTA 0,25 %. 4 min bei 37 ° c inkubieren 4 mL ES Zellkulturmedium hinzufügen, Aufschwemmen und spin-down bei 1000 X g für 4 min. Aspirat überstand und das Pellet in 2 mL frisch ES Zellkulturmedium aufzuwirbeln.
Hinweis: PBS ohne Ca2 +/Mg2 + sollte verwendet werden für das Waschen der Zellen vor Trypsinization um Ca2 + -Ionen, die für die Zelladhäsion erforderlich sind zu entfernen. Im Gegensatz dazu für die E-Cad-Fc Verdünnung und Beschichtung (Schritt 1.4), PBS mit Ca2 +/Mg2 +verwenden, die Haftung von ES-Zellen in E-Cad-Fc beschichtete Gerichte ist seit Ca2 +-abhängigen14. - Verdünnen Sie die resuspendierte Zellen 01:10 in 1 mL ES Zellkulturmedium und Auszählung, mit einer Zählkammer (Last 10 µL für eine Kammer von 0,1 mm Tiefe).
- Samen 30.000 Zellen/cm2 in E-Cad-Fc beschichtet Bildplatte. Füllen Sie bis zu 200 µL pro Bohrloch mit ES Zellkulturmedium. Für Doxycyclin-induzierbaren Zell-Linien, mit einer angemessenen Dosis von Doxycyclin zu induzieren (Dosis und Zeitpunkt der Induktion im Voraus zu optimieren. Die hier verwendeten Zelllinien wurden mit 500 ng/mL Doxycyclin 24 h vor Bildgebung behandelt). Inkubation bei 37 ° C und 5 % CO2 und fahren Sie mit dem Puls Kennzeichnung und imaging-24 h nach Aussaat der Zelle.
2. Puls Kennzeichnung des SNAP-tag
Hinweis: Für Protein Verfall Experimente ist es entscheidend, eine angemessene SNAP Farbstoff Konzentration zu verwenden. Die Konzentration sollte hoch genug sein, ergeben eine hellere Signal am Anfang von den Zeitraffer, wie die Fluoreszenz im Laufe der Zeit sinkt. Jedoch möglicherweise zu hoch Farbstoff Konzentrationen mit residual Farbstoff wird links im Medium oder in den Zellen auch nach dem Waschen. Der freie Farbstoff könnte anschließend an neuproduzierten SNAP-Tag-Moleküle im Laufe des Films, binden, die Verfall-Kurve verzerren. Beobachteten Fluoreszenzsignal hängt von den Eigenschaften der Farbstoff, der Zell-Linie verwendet, als auch die Expression des entsprechenden Proteins. Daher ist es entscheidend, die Farbstoff-Konzentration zu optimieren, indem Sie testen verschiedene Verdünnungen, ausgehend von der Verdünnung, die vom Hersteller für das live Cell Imaging vorgeschlagen. Für diese Studie war eine dunkelrote fluoreszierende Substrat verwendet. Eine optimale Konzentration von 12 nM für Doxycyclin-induzierbaren Überexpression Zelllinien festgelegt wurde.
- Den SNAP-Farbstoff auf die entsprechende Konzentration in vorgewärmten ES Zellkulturmedium verdünnt. Verwenden Sie eine Pipette, um entfernen vorsichtig das Medium aus den Zellen in den 96-Well-Platte zuvor gesät, und fügen Sie 50 µL verdünnter SNAP Farbstoff pro Bohrloch. 30 min bei 37 ° c inkubieren
- Abzusaugen Sie die verdünnte Farbe mit einer Pipette und waschen Sie 3 X mit 200 µL vorgewärmten PBS (ohne Ca2 +/Mg2 +). Fügen Sie 200 µL ES Zellkulturmedium und 15 min bei 37 ° c inkubieren
- Wiederholen Sie den vorherigen waschen zweimal mehr.
Hinweis: Umfangreiche waschen ist wichtig, um alle ungebundenen Farbstoff zu entfernen. Die wiederholte Waschschritten mit PBS entfernen den restlichen extrazellulären Farbstoff in das Medium, während 15 min Inkubationen robuste Kennzeichnung von SNAP-Tag-Fusionsproteinen sorgen vor Beginn der imaging Experiments. Allerdings führen Sie die Waschschritte durch sanfte pipettieren und verwenden Sie keine automatische Absauganlage, da Zellen ausgesät auf E-Cad-Fc tendenziell leicht lösen. - Fügen Sie 200 µL von imaging-Medium. Zur Reduzierung der Fluoreszenz-Hintergrund, Phenol rot freie Mittel (ergänzt mit 10 % ES Zelle qualifiziert fötalen Rinderserum, 2 mM Natrium Pyruvat, 1 % nicht-essentiellen Aminosäuren, 1 % Penicillin/Streptomycin, 2 mM L-Glutamin, 100 µM 2-Mercaptoethanol, LIF, 3 µM CHIR99021 und 0,8 µM PD184352).
3. Time-Lapse-Mikroskopie
- Verwenden Sie geeignet für live Aufnahmen, so dass kontrollierter Temperatur und CO2Mikroskop. Stellen Sie die Temperatur auf 37 ° C und die CO2 bis 5 % verwenden und lassen Sie für 1-2 h equilibrate vor.
- Legen Sie die Platte in das Mikroskop und finden Sie Flecken geeignet für Bildgebung zu. Wählen Sie Orte, wo die Zellen sind gleichmäßig verteilt, aber nicht zu dicht, da dies die Analyse erleichtern wird. Passen Sie die Anzahl der ausgewählten Orte, die Anzahl der Zellen, die für die Analyse erforderlich. In dieser Studie wurden 3-5 Flecken pro Zelle Zeile erfasst.
- Wählen Sie das Ziel. Ein 20 X Objektiv eignet sich für die Größe von ES-Zellen.
- Wählen Sie die Beleuchtung-Einstellungen für die fluoreszierenden Kanäle aufgezeichnet werden. Passen Sie die Laserleistung und Belichtung entsprechend der Intensität der Fluoreszenzsignal aus den Zellen von Interesse. Achten Sie auf ein starkes erstes Signal zu erhalten, da wird es im Laufe des Films zu verringern, aber vermeiden Sie Laserleistungen höher als 30 % um Phototoxizität und Immunofluoreszenz zu minimieren. Für diese Studie, eine Erregung Filter von 632/22 nm (Wellenlänge/Bandpass), Emission Filter 679/34 nm (Wellenlänge/Bandpass), Laserleistung von 10 % und die Belichtung Zeit von 100 ms verwendet wurden.
- Wählen Sie die bildgebenden Intervalle und Dauer des Time-Lapse-Experiments. Wählen Sie für Proteine mit erwarteten Halbwertszeiten von 2-20 h Erfassungszeiten von 12-24 h mit zeitlichen Abständen von 15 Minuten. Reduzieren Sie für kurzlebigere Proteine die Intervalle und Erfassungszeiten langlebigeren Proteine Erhöhung entsprechend.
- Starten Sie imaging.
4. Verarbeitung und Analyse von Bildern
- Sammeln Sie die aufgenommenen Bilder als Stapel im TIFF-Format. Verwenden Sie zur Weiterverarbeitung und Analyse Fidschi Software15. Wenn die Time-Lapse Daten nicht als Stapel von der Mikroskop-Software gesammelt werden, öffnen Sie alle Frames des Time-Lapse-Experiments in der Software (entweder durch Klicken auf Datei | Öffnen... oder per Drag & Drop die entsprechenden Dateien in der Symbolleiste) und klicken Sie auf Bild | Stacks | Bilder zu stapeln.
- Die Funktion der subtrahieren Hintergrund Hintergrund aus allen Bildern im Stapel zu entfernen. Dazu klicken Sie auf Prozess | Hintergrund zu subtrahieren. Wählen Sie aus des rollenden Kugel Radius in das Popup-Fenster. Verwenden Sie einen Radius, der mindestens die Größe der Zellen abgebildet ist. Um die Hintergrundabzug auf alle Zellen im Stack anzuwenden, wählen Sie Ja in den Prozess-Stack? Popup-Fenster.
- Wählen Sie eine Zelle von Interesse und zeichnen Sie eine Region of Interest (ROI) um sie über die Symbolleiste. Verwenden Sie für nukleare Signale den Oval -Auswahl vom ersten Klick auf die entsprechende Registerkarte in der Symbolleiste klicken Sie auf den Kern des Interesses und Anpassung das Oval um ihn herum. Für zytoplasmatischen Signale oder sehr dichten Regionen der Freihandauswahl verwenden, um die Zelle Umrisse verfolgen können. Vermeiden Sie zu große Regionen des Hintergrunds, obwohl es nicht notwendig, die Zelle Umrisse genau durch die anschließende Subtraktion alle Hintergrundpixel (Schritt 4,8-4,9) zu folgen ist.
- Die Region des Interesses an den ROI-Manager hinzufügen, indem analysieren | Werkzeuge | ROI-Manager | Fügen Sie, oder T auf der Tastatur drücken.
- Gehen Sie zum nächsten Bild durch Verschieben der Registerkarte im Fenster "Stapel" oder durch Drücken der Umschalttaste + < auf der Tastatur, dann wiederholen Sie die vorherigen Aktionen. Folgen Sie der Zelle von Interesse im Laufe des Films und fügen Sie den ROI des einzelnen Frames an den ROI-Manager hinzu. Speichern den letzten ROI set für jede überwachte Zelle durch Klicken mehr | Speichern... in der ROI-Manager.
Hinweis: Nach Zellteilungen, verfolgen beide Tochterzellen getrennt und die Summe ihrer integrierten Intensitäten nach Hintergrundabzug (siehe Schritte 4,6-4,9), um Protein-Verdünnung während der Zellteilung zu berücksichtigen. Speichern Sie die ROI-Sets für beide Tochterzellen. - Sobald die Zelle von Interesse im Laufe des Films verfolgt wird, klicken Sie auf messen, um Werte für die mittlere Intensität und den Bereich des ROIs zu erhalten. Die erhaltenen Werte in eine elektronische Tabellenkalkulations-Programm kopieren und die rohe integrierte Intensität der Zelle für jeden Zeitpunkt wie folgt zu berechnen:

wo die mittlerecist die mittlere Intensität undBereichCBereich der entsprechenden ROI. - Um den lokalen Hintergrund abschätzen zu können, fügen Sie einen ROI in der Nähe der Zelle von Interesse für jeden Zeitrahmen. Vermeiden Sie alle zellulären Fluoreszenzsignal. Verwenden Sie einen kreisförmigen ROI, die etwa die Größe einer Zelle und verschieben Sie oder verkleinern Sie es entsprechend zu, bei Bedarf, z.B. wenn benachbarte Zellen stören. Gehen Sie wie zuvor mit den zellulären gestellten ROI gesetzt, um einen Hintergrund ROI zu erzielen und kopieren Sie die gemessenen Intensitätswerte in das Arbeitsblatt.
- Um einen Hintergrund-korrigierte Wert für die integrierte Intensität der Zelle zu erhalten, berechnen Sie zunächst die integrierte Intensität des Hintergrunds für jeden Zeitpunkt:

wo MeineBG die mittlere Intensität der Hintergrundsignal und BereichC ist, ist der Bereich des ROI rings um die Zelle. Verwenden Sie die Fläche des Hintergrunds ROI, nicht, es sei denn, beide Bereiche die gleiche Größe haben. - Die endgültige Hintergrund subtrahiert integrierte Intensität der Zelle für jeden Zeitpunkt zu berechnen:

- Um die einzelne Zelle Verfall Kurven zu normalisieren, teilen Sie den Intensitätswert des jeden Zeitpunkt durch den Intensitätswert der ersten Zeit-Punkt.
Hinweis: Die Kurvenanpassung (siehe Schritt 4.11) kann entweder auf jeder einzelnen Zelle oder auf die durchschnittliche Bevölkerung durchgeführt werden. Eine Normalisierung ist erforderlich, wenn ein Bevölkerungsdurchschnitt berechnet wird, um Verzerrungen aus verschiedenen Fluoreszenz-Intensitäten zwischen Zellen zu vermeiden. Die Normalisierung gewährleistet, dass jede Zelle mit dem gleichen Gewicht zur endgültigen Verfall Kurve trägt. Darüber hinaus kann die Normalisierung, die einzelne Zelle zerfällt unabhängig von ihrer absoluten Fluoreszenz-Intensitäten zu visualisieren (siehe Abbildungen 3A-3 C) nützlich sein. Jedoch, wenn nur die einzelligen Halbwertszeiten ermittelt werden und die Daten nicht gemittelt, Normalisierung Schritt kann ausgelassen werden und die Kurvenanpassung kann direkt auf die raw-Daten durchgeführt werden. - Verwenden Sie für die Schätzung der Protein-Halbwertszeit eine Kurve Montagewerkzeug. In dieser Studie wurde die MATLAB Kurvenanpassung Toolbox 3.4.1 verwendet, die befindet sich standardmäßig im Bereich Apps der MATLAB-Benutzeroberfläche. Importieren der Fluoreszenzintensität und Zeitwerte aus der elektronischen Tabellenkalkulation in MATLAB durch Klicken auf die Registerkarte " Daten importieren " die Kurvenanpassung Toolbox öffnen und wählen Sie die Zeitpunkte und Fluoreszenz-Verfall-Daten in der X-Daten und Y Daten tab. Wählen Sie Benutzerdefinierte Gleichung in der Kurvenanpassung Registerkarte, und geben Sie die Gleichung für eine exponentielle Zerfall:
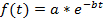
Wenn f(t) die Fluoreszenzintensität zu einem bestimmten Zeitpunkt ist eine der Anfangsintensität und b die Zerfallsrate. In den Optionen passen... Registerkarte, wählen Sie 0 für die untere Grenze von a und b. Die geschätzten Werte für a und b werden dann im Ergebnisfenster angezeigt. Berechnen Sie die Halbwertszeit wie folgt:
Ergebnisse
Das beschriebene Protokoll stellt eine Schätzung der Zell-Zell-Variabilität in Half-Life für jeden gegebenen Proteins verschmolzen zu einem Kinderspiel-Tag. Die Verwendung von rekombinanten E-Cadherin-Fc für die Beschichtung von die Speicherfolie ermöglicht einzelne Zelle Auflösung in ES-Zellen, die sonst in den Kolonien wachsen. Einzelne Zellen können separat im Laufe des Films ( Abbildung 1A) verfolgt werden.
Um die Protein-Halbwertszeit für jede einzeln...
Diskussion
Der wichtigste Schritt bei der Verwendung eines SNAP-Tags Protein Verfall zu überwachen ist, um sicherzustellen, dass kein passives ungebundenen Farbstoff im Medium oder in den Zellen nach dem Waschen, da sonst es links neu binden könnte produziert SNAP-Tag Moleküle später im Verlauf des Experiments und ther Eby Kompromiss der Verfall-Kurve. Dies ist einerseits durch sorgfältig ausführen der beschriebenen Waschschritte erreicht. Auf der anderen Seite sollte die Farbstoff-Konzentration werden so gering wie möglich,...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Time-Lapse Mikroskopie Experimente wurden an der biomolekularen Screening Facility (BSF), EPFL durchgeführt. Wir danken Marc Delachaux (Service Audiovisuel, EPFL) für die Videografie und Bearbeitung des Films.
Materialien
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
Referenzen
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745 (2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156 (2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15 (2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten