Method Article
Optimierte 3D zerebelläre Differenzierung Protokoll mit Optional 2D Modifikation
In diesem Artikel
Zusammenfassung
Wir beschreiben eine vereinfachte 3D Differenzierung Protokoll für hPSCs, mit definierten Medium und Wachstumsfaktoren, geeignet zur Erzeugung Zelle Aggregate mit frühen neuroepithelialer Strukturen und positiv für zerebelläre-assoziierten Marker, sowie eine optionale reduziert 2D Modifikation zur Differenzierung von Zellen als eine Monolage, funktionelle Neuronen zu generieren.
Zusammenfassung
Reduzierung von Komplexität und Kosten der Differenzierung Protokolle ist wichtig für die Forscher. Dieses Interesse passt mit Sorgen über mögliche unerwünschte Wirkungen, dass extrinsische Musterung Faktoren in menschlichen pluripotenten Stammzelle (hPSC) Modelle der Entwicklung des Gehirns oder Pathophysiologie, wie Maskierung Krankheit Phänotyp führen könnten. Hier präsentieren wir Ihnen zwei zerebelläre Differenzierung Protokolle für hPSCs, ausgelegt mit einfacher Startmethode, weniger Musterung Faktoren und weniger Materialbedarf als frühere Protokolle. Vor kurzem haben wir Kultur Verfahren, die frei schwebenden 3-dimensionale (3D) Produkte mit anderen Gehirn "organoide" Protokolle, einschließlich Morphologien relevant zur Entwicklung des Gehirns wie Sub/ventrikuläre Zone - Modellierung und rhombischen generieren Lippe-ähnlichen Strukturen. Die zweite verwendet ein anhaftende, 2D Monolayer-Verfahren, um vollständige Differenzierung, die funktionelle zerebelläre Neuronen erzeugen, wie Produkte für zerebelläre-assoziierten Marker positiv sind, und Neuron-wie Kalzium Zustrom weisen angezeigt wird. Zusammen bieten diese Protokolle Wissenschaftler eine Auswahl von Optionen, geeignet für verschiedene Forschungszwecke sowie ein Grundmodell für Tests andere Arten von optimierte neuronale Differenzierungen.
Einleitung
In-vitro- Protokolle zur Differenzierung von hPSCs in Richtung Kleinhirn Linien zunächst betrieben auf dem Prinzip der Nachahmung in Vivo zerebelläre Entwicklung1,2,3,4. So verlangten sie eine Reihe von Faktoren, die zu bestimmten Zeiten Pro-zerebelläre Musterung und Reifung fahren eingeführt. Das wichtigste davon WNT, wurden Knochen morphogenetische Proteine (BMPs) und Fibroblasten-Wachstumsfaktoren (FGFs) mit bekannten Rollen in der Mitte Hinterhirn Entwicklung und Bildung der Isthmische Veranstalter5,6,7. Natürlich, jeder weitere Schritt und Faktor bedeutet einen Anstieg der arbeitsintensiven Manipulationen und größeren Aufwand für den Forscher, und so entwickeln einfacher Protokolle in der Lage, die gleichen Ergebnisse zu erzielen ist von Interesse. Dieser praktischen Frage knüpft sehr schön die hypothetische Frage, ob Zellen solche engen, externe Kontrolle über ihre Entwicklung in Vitro benötigen.
Zerebelläre Differenzierung adressiert ein Protokoll veröffentlicht im Jahr 2015 die Notwendigkeit der Verwendung einer umfangreichen Reihe von Wachstumsfaktoren mit nur FGF2, FGF19 und Stromazellen Cell-derived Factor 1 (SDF1) für Zwecke8-Strukturierung. Diese Studie unterschieden sich auch aus vorherigen zerebelläre Protokolle mit einem frei schwebenden 3D Culture-System. Neben der Produktion von Zellen positiv für zerebelläre Markierungen, zeigte die Gehirn "Organellen" durch ihre Technik erzeugt relevanten Morphologie, nicht verfügbar in traditionelle 2D Monolayer-Kulturen wie rhombischen Lippe-ähnliche Strukturen aufweisen. Obwohl weniger komplex und kostspielig im Hinblick auf Wachstumsfaktoren, weitere Funktionen wie z. B. Bildung von einheitlichen Embryoid Körper (EBs) und Kultur in 96-Well-Platten (96WPs), machte es verfahrensrechtlich komplexe während der ersten Schritte. Ein weiteres 3D Protokoll veröffentlichte im gleichen Jahr, erfolgreiche Differenzierung zu neuronalen Linien mit gemeinsamen und kostengünstige Zelle Kultur Techniken9berichtet. Obwohl diese Gruppe kortikalen anstatt zerebelläre Differenzierung untersucht wurde, konnte Anwendung ihres Konzepts für zerebelläre Differenzierung nicht vernachlässigt werden.
Wir berichteten vor kurzem ein 3D zerebelläre Differenzierung-Protokoll mit einer reduzierten Anzahl von Musterung Faktoren (nämlich FGF2, 4 und 8), sowie ein vereinfachtes Setup indem man Zellen in 6-Well-Platten (6WPs) durchgehend bis mittleren Anforderungen10zu minimieren. Zur Herstellung von Granulat-Zellen zu unterstützen, wurde geglättet Agonist (SAG) während die endgültige Reifung Schritt verwendet. SAG ist eine weniger teure chemische Alternative zu sonic Hedgehog (SHH), die in früheren zerebelläre Protokolle, wegen seiner Rolle bei der Förderung des Wachstums von Granulat Zelle Vorläufer (GCPs) in Vivo1,2, benutzt worden 11,12,13. Differenzierung-Produkte wurden mit denen von anderen 3D Protokolle, einschließlich des Kleinhirn-assoziierten Marker in morphologisch relevanten Strukturen8,9. Solche Ergebnisse stärken die frühere Nachricht, Mimikry in die Tiefe, des in Vivo Umgebung möglicherweise nicht für komplexe 3D in-vitro- Differenzierung Protokolle erforderlich.
Neben dem 3D Protokoll der vorliegende Bericht beschreibt ein 2D Protokoll entwickelt, mit der gleichen Schnelleinrichtung, Grundstoffen, und reduzierte Anzahl von Wachstumsfaktoren. Es ist in der Lage, Zellen aus menschlichen embryonalen Stammzellen (HES) oder induzierte pluripotente Stammzellen (HiPSCs), positiv für Markierungen im Zusammenhang mit frühen neuronalen, zerebelläre, und Granulat Zelle Identitäten. Zusätzlich zeigt Kalzium Bildgebung das Vorhandensein von funktionalen menschlichen Neuronen. Die Möglichkeit zu wählen zwischen Protokollen, fügt ein Maß an Flexibilität für Forscher, für Interessenten an entweder: (1) Erzeugung von bestimmten Zelle eingibt, (2) Modellierung menschlichen Gehirnentwicklung und die damit verbundenen Strukturen, (3) Analyse in monomolekularen Film optimiert Einstellungen (z.B., Patch-Clamp-Aufnahmen), oder (4) Zell-Zell-Interaktionen in neuronalen Mischkulturen. Ihre einfache und kostengünstige Art macht sie zugänglich für Forscher, die sind neu auf dem Gebiet der hPSC oder base hPSC Verfahren aus, um weitere Differenzierung Optionen zu erkunden.
Protokoll
1. Vorbereitungen
Hinweis: Alle Schritte finden Sie unter Tabelle der Materialien nach bestimmten Elementen.
-
500 mL definiert hPSC Kulturmedium für hPSC Kultur vorbereiten
Hinweis: Verwenden Sie Medium für Schritte 2.1-2.6.- HPSC mittlere Ergänzung über Nacht (o/n) bei 4 ° c Auftauen Mittlere Flasche 12,5 mL hPSC Basis Medium entfernen, dann fügen Sie 10 mL Ergänzung und 2,5 mL (macht 100 U/L) Penicillin/Streptomycin (Pen/Strep) bis zur Flasche.
- Bei 4 ° C lagern und innerhalb von 2 Wochen.
Hinweis: Das hPSC Medium kann Zellen Einfrieren durch Zugabe von 10 µM ROCK-Inhibitor (RI) und 10 % DMSO eingesetzt werden.
-
1 L neuronale Wartung Medium (NMM) für Differenzierung Kultur vorbereiten
Hinweis: Verwenden Sie Medium für Schritte 3.1-4.4.- Mix-Glutamin angereichert DMEM/F12 und neuronale Basismedium (Verhältnis 1:1) in eine 1 L Flasche, dann Ergänzung mit N2-Ergänzung (1 X), (1 X) B27 ergänzen, 5 µg/mL Insulin, 1,5 mM L-Glutamin, 100 µM nicht-essentiellen Aminosäuren (NEAA), 100 U/L Pen/Strep und 10 µM Beta-Mercaptoethanol.
- Medium bei 4 ° C lagern und innerhalb von 3 Wochen.
Hinweis: Bevor Sie gemischte basal Medium Ergänzungen hinzufügen, entfernen Sie das entsprechende Volume, um die erforderliche Menge des hinzugefügten Komponenten basierend auf Lager Konzentrationen anzupassen.
-
Passagierung hPSCs bereiten Sie 500 mL 0,5 mM EDTA Lösung vor
Hinweis: Verwenden Sie Medium für Schritte 2.4 und 2.5.- Unter einem Fluss-Haube 49 mL von Phosphat gepufferte Kochsalzlösung (PBS) aus einer 500 mL Flasche für sterile PBS zum Übertragen einer 50 mL-Tubus Die 50 mL-Tube 0,5 mL 0,5 M EDTA und 0,9 g NaCl hinzufügen. Die Mischung sanft aufzulösen.
- Filter-sterilisieren Lösung mit einem 0,22 µm Filter und Transfer um die 500 mL-Flasche für sterile PBS. Lagerung bei Raumtemperatur (RT).
-
Vorbereiten der hPSC Kultur Platten hPSC Kultur
Hinweis: Verwenden Sie Platten für Schritte 2.1-2.6.- 50 x Arbeitslösung hPSC angemessen haftenden Beschichtung (PAAC) zu machen: ein Fläschchen PAAC o/n bei 4 ° c Auftauen Verdünnen Sie PAAC im Verhältnis 1:1 mit DMEM/F12 und Übertragung als 400 µL-Aliquots in 1,5 mL Röhrchen. Speicher 50 x Arbeitslösung PAAC bei-80 ° C.
Hinweis: (wichtig) PAAC erstarrt schnell bei RT, daher ist es notwendig, dass alle Komponenten (DMEM, Rohre, etc.) auf dem Eis (oder bei 4 ° C) gehalten werden. - Rohr 50 x Arbeitslösung PAAC bei 4 ° C Auftauen, dann 50 X kalt DMEM/F12 zu verdünnen. Ein 6WP 750 µL/Well des verdünnten PAAC hinzufügen. Inkubieren Sie Platte für mindestens 1 h bei 37 ° c
Hinweis: PAAC Platten können für 1 Woche bei 4 ° C gelagert werden, indem man die Platte nach 1 h Inkubationszeit. Warme Platte auf 37 ° C vor dem Gebrauch.
- 50 x Arbeitslösung hPSC angemessen haftenden Beschichtung (PAAC) zu machen: ein Fläschchen PAAC o/n bei 4 ° c Auftauen Verdünnen Sie PAAC im Verhältnis 1:1 mit DMEM/F12 und Übertragung als 400 µL-Aliquots in 1,5 mL Röhrchen. Speicher 50 x Arbeitslösung PAAC bei-80 ° C.
-
Anti-Adhäsiv (AA) Platten für Differenzierung Kultur vorbereiten
Hinweis: Verwenden Sie Platten für Schritte 3.1-4.1.- 5 mg/mL machen Poly (2-Hydroxyethyl Methacrylat) (Poly-HEMA) Lösung in 95 % Ethanol. O/n bei 37 ° C zu schütteln, bis eine klare Lösung entsteht. Shop bei RT
Hinweis: Durch einen 0,22 µm-Filter filtern kann unaufgelöste Poly-HEMA entfernen. - Fügen Sie Poly-HEMA auf die Kultur-Platte hinzu, so dass es am Ende jedes gut bedeckt. 2 Tage inkubieren Sie die Platte bei 37 ° C, und überprüfen Sie die Platte, um vollständige Verdampfung der Flüssigkeit/Uniform Beschichtung von Brunnen zu gewährleisten. AA Platten kann gewickelt und bei RT gespeichert werden
- 5 mg/mL machen Poly (2-Hydroxyethyl Methacrylat) (Poly-HEMA) Lösung in 95 % Ethanol. O/n bei 37 ° C zu schütteln, bis eine klare Lösung entsteht. Shop bei RT
-
Poly-L-Ornithin/Laminin (PLO/LAM) Platten für Differenzierung Kultur vorbereiten
Hinweis: Verwenden Sie Platten für Schritte 4.2-4.4.- Die Fläche von Wells, mit 20 µg/mL PLO aufgelöst in sterilen PBS zu beschichten. Inkubieren Sie die Platte o/n bei 37 ° c Aspirieren Sie PLO und spülen Sie 3 Mal mit PBS ab.
Hinweis: (Optional) Incubated Platte mit PLO kann gewickelt und bei 4 ° C bis benötigt gespeichert werden. - Die Flächen der PLO-beschichtete Brunnen zu beschichten, mit 10 µg/mL LAM in sterilen PBS aufgelöst. Mindestens 2 h bei 37 ° C oder o/n bei 4 ° c inkubieren LAM zu entfernen und waschen Brunnen 2-3x mit PBS, fügen Sie dann sofort in anderer geeigneter Form und/oder Zellen.
Hinweis: (Optional) LAM entfernt Lösung kann bei 4 ° C gelagert und bis zu 2 mal wiederverwendet werden. (Wichtig) Lassen Sie LAM-beschichtete Oberflächen trocknen nicht zu; um zu verhindern, dass dies sofort hinzufügen PBS oder in anderer geeigneter Form.
- Die Fläche von Wells, mit 20 µg/mL PLO aufgelöst in sterilen PBS zu beschichten. Inkubieren Sie die Platte o/n bei 37 ° c Aspirieren Sie PLO und spülen Sie 3 Mal mit PBS ab.
2. Protokoll 1: Feeder-freie hPSC Kultur
Hinweis: hESC stammen aus einer nichtkommerziellen Organisation (H01-Linie, siehe Tabelle of Materials). Drei HiPSC Steuerleitungen (hvs51, 60 und 88) wurden durch Umprogrammierung Fibroblasten aus drei gesunden menschlichen Patienten (Fibroblasten wurden von anonymen, nicht identifizierbaren Spendern abgeleitet und daher von der IRB Genehmigung befreit) erzeugt10, 17.
- HPSCs in der Feeder-freie Kultur pflegen
- Nach dem Auftauen und Galvanik hPSCs auf PAAC Teller in hPSC Medium (siehe Punkt 2.2), hPSCs bei 37 ° C mit 5 % CO2zu erhalten. Aktualisieren Sie hPSC Medium täglich (siehe Punkt 2.3), außer am Tag nach dem Auftauen oder Passagierung, und Zellen unter dem Mikroskop zu untersuchen (Ziele: 2.5x/0.06, 5 x / 0,12 Ph0, 10 x / 0,25 Ph1) zu beobachten das Wachstum bewerten und mögliche Bereiche der Differenzierung (Abbildung 3 , obere linke Tafel zeigt Beispiel der Differenzierung).
- Durchgang hPSCs alle 3-4 Tage, oder wenn Kultur erreicht > 80 % Zusammenfluss. Wenn weniger als 5 % der Zellen Ausstellung Differenzierung, Verwendung normaler hPSC Passagierung Methode (siehe Schritt 2.4), ansonsten verwenden Sie die sanfte Methode (siehe Punkt 2.5). Wenn nicht mehr in Kultur benötigt, können hPSCs für langfristige Lagerung eingefroren werden (siehe Punkt 2.6).
- HPSCs Tauwetter in hPSC medium
- Das erforderliche Volumen der hPSC Medium zu übertragen, zu sterilen Röhrchen für das Auftauen (9 mL/Kryo Röhrchen) und auf die vorbereiteten PAAC Platte aufgetaute Zellen zu erhalten. Ergänzen Sie das Medium in beiden Röhren mit 10 µM RI.
- Rufen Sie die Cryotube aus LN2 Lagerung und Ort direkt in ein Wasserbad (37 ° C). Wenn nur ein kleines Eis Kristall bleibt, aus dem Wasserbad nehmen und den Inhalt der kryogenen Röhre auf das Rohr zum Auftauen Prozess übertragen (Gesamtmenge 10 mL). Zentrifugieren Sie das Rohr auf 290 X g für 5 min bei RT
- Eine serologische Pipette verwenden, um die PAAC entfernen Lösung aus den Brunnen der PAAC Platte (siehe Punkt 1.4) sollen Zellen zu erhalten, und fügen Sie hPSC Medium mit 10 µM RI.
Hinweis: (wichtig) nicht Aspirat PAAC Lösung mit einer Saug-Nadel, oder es kann zu festigen und verstopfen Leitungen zur Vakuumpumpe. - Entfernen Sie den Überstand aus dem Rohr zu und Aufschwemmen Sie Zellen in hPSC Medium mit 10 µM RI. Verteilen Sie die Zellen an der Ziel-Platte im Verhältnis von 1 Kryo Röhrchen/Brunnen des 6WP. Inkubation bei 37 ° C mit 5 % CO2, und aktualisieren Sie Medium nicht für 1 Tag.
Hinweis: Zellen um 5 % O2 Inbetriebnahme kann überleben der Zellen erhöht.
- HPSC Medium aktualisieren
- Das benötigte Volumen der hPSC Medium in ein steriles Röhrchen bei RT oder in einem Wasserbad warm; 2 mL/Brunnen des 6WP wird empfohlen.
Hinweis: (Optional): Wenn Sie einen zusätzlichen Betrag von hPSC Medium hinzufügen, kann hPSCs bleiben einen zusätzlichen Tag ohne zu aktualisieren; jedoch können Sie dies nicht mehr als einmal in der Woche. - Aspirieren Sie das Medium aus Brunnen mit hPSCs und fügen Sie frischen hPSC Medium hinzu.
- Kultur der hPSCs in einem Inkubator bei 37 ° C und 5 % CO2.
- Das benötigte Volumen der hPSC Medium in ein steriles Röhrchen bei RT oder in einem Wasserbad warm; 2 mL/Brunnen des 6WP wird empfohlen.
- Durchgang hPSCs in hPSC medium
- Übertragen Sie das erforderliche Volumen der hPSC Medium auf sterilen Röhrchen, Passagierung Prozess und Vorbereitung der PAAC Platte Zellen passagiert erhalten. Ergänzen Sie das Medium für die Ziel-Platte mit 10 µM RI. Erwärmen Sie die mittlere Rohre bei RT oder im Wasserbad.
Hinweis: Vorbereitung und Handhabung werden abweichen, wenn mit einer alternativen Beschichtungsmaterial als in Tabelle Materialienaufgeführt. - Eine serologische Pipette verwenden, um die PAAC entfernen Lösung aus den Brunnen der PAAC Platte (siehe Punkt 1.4) sollen Zellen zu erhalten, und fügen Sie hPSC Medium mit 10 µM RI.
Hinweis: (wichtig) nicht Aspirat PAAC Lösung mit einer Absaugung Nadel, verfestigen und verstopfen Leitungen zur Vakuumpumpe. - Aspirieren Sie das Medium aus Brunnen mit hPSCs passagiert werden, waschen Sie Zellen zweimal mit 0,5 mM EDTA, dann fügen Sie 0,5 mM EDTA und 2-5 min bei 37 ° c inkubieren
Hinweis: 1 mL/Brunnen des 6WP ist ein ausreichendes Volumen an EDTA zum Waschen und Inkubation. - Überprüfen Sie die Brunnen unter dem Mikroskop (Ziele: 2.5x/0.06, 5 x / 0,12 Ph0, 10 x / 0,25 Ph1). Wenn Zellen zu lösen beginnen, aspirieren Sie EDTA Lösung und bündig Zellen frei mit hPSC Medium.
Hinweis: (wichtig) kümmern sich nicht um ganze hPSC Kolonien zu entfernen, beim Absaugen von EDTA (nicht warten, bis die ganze Kolonien trennen sind). Nicht spülen Sie Zellen mehr als 5mal da dies hPSCs Schaden und Pluripotenz beeinflussen. Lassen Sie auch nicht Zellen an hPSC Medium mit RI stehen vor leeren Zellen aus Brunnen, wie sie die Platte wieder einhalten können. - Basierend auf empirischen Bestimmung (in der Regel im Zusammenhang mit Zusammenfluss, Größe der Kolonien und Wachstumsrate), transfer hPSCs zum Brunnen der Ziel-Platte mit ein splitting-Verhältnis von 1:4-1:16 (d.h., 1 gut von der Originalplatte, 4 Brunnen des Ziels Platte). Inkubation bei 37 ° C mit 5 % CO2, und aktualisieren Sie Medium nicht für 1 Tag.
Hinweis: Splitting Verhältnisse so hoch wie 01:16-01:20 möglich sind, um zu verhindern, Verdrängung und Aussehen der Kolonien zu verbessern.
- Übertragen Sie das erforderliche Volumen der hPSC Medium auf sterilen Röhrchen, Passagierung Prozess und Vorbereitung der PAAC Platte Zellen passagiert erhalten. Ergänzen Sie das Medium für die Ziel-Platte mit 10 µM RI. Erwärmen Sie die mittlere Rohre bei RT oder im Wasserbad.
- Durchgang hPSCs mit der sanften Methode (G-Methode)
- Übertragen Sie das erforderliche Volumen der hPSC Medium zum sterilen Röhrchen, für den passaging Prozess und zur Vorbereitung der PAAC Platte Zellen passagiert erhalten Ergänzen Sie das Medium für Ziel-Platte mit 10 µM RI. Warme mittlere Rohre bei RT oder im Wasserbad bei 37 ° C.
- Verwendung serologische Pipette entfernen PAAC Lösung aus Brunnen PAAC Platte sollen (siehe Punkt 1.4) Zellen zu erhalten, und fügen Sie hPSC Medium mit 10 µM RI.
Hinweis: (wichtig) nicht PAAC Absaugen mit einer Saug-Nadel, oder es kann zu festigen und verstopfen Leitungen zur Vakuumpumpe. - Aspirieren Sie Medium aus den Brunnen mit hPSCs, passagiert werden, und waschen Sie die Zellen, die zweimal mit 0,5 mM EDTA. Auf dem zweiten waschen, warten 30 s vor dem Absaugen von EDTA, dann fügen Sie 1 mL PBS und 4-9 min bei 37 ° c inkubieren Während des Wartens, bereiten Sie ein steriles Röhrchen mit 4 mL PBS.
Hinweis: 1 mL/Brunnen des 6WP ist ausreichendes Volumen an EDTA zum Waschen. - Überprüfen Sie die Brunnen unter Mikroskop (Ziele: 2.5x/0.06, 5 x / 0,12 Ph0, 10 x / 0,25 Ph1). Wenn Zellen beginnen zu trennen, tippen Sie sorgfältig auf den Seiten der Platte freie Kolonien zu helfen. Wenn > 50 % der Kolonien sind frei schwimmende, verwenden Sie eine serologische 5-mL-Pipette, um Kolonien in 1 mL PBS auf das Rohr mit 4 mL PBS zu übertragen (nicht genannte).
Hinweis: (wichtig) da hPSC Kultur bereinigen soll, befestigt prüfen um festzustellen, ob differenzierte Zellen bleiben bis auf den Teller. Auch ist es nicht notwendig, alle Kolonien in einer gut mit dabei, so dass Kolonien, die sich nicht wegziehen können zurückgelassen werden passage. - Warten Sie ca. 5-10 min bei RT für Zellen sich in der Röhre (nicht zentrifugieren). Aspirieren Sie PBS aus der Tube, kümmert sich nicht um ständige hPSCs zu entfernen. Sorgfältig Aufschwemmen der Zellen in hPSC Medium (nicht genannte), und übertragen Sie die Zellen an der Ziel-Platte mit ein splitting-Verhältnis von 1:4-1:16. Inkubation bei 37 ° C mit 5 % CO2, und aktualisieren Sie Medium nicht für 1 Tag.
- Unten hPSCs frieren
- Je nach dem Zusammenfluss verwenden Sie 1 gut von hPSCs, ca. 2-3 Tage (maximal) in Kultur, um 1-2 Kryokonservierung Fläschchen für die Lagerung in LN2auszufüllen.
- Am Ende des 2.5 Passagierung (Schritt), verwenden Sie 500 µL oder 1 mL der hPSC Medium übertragen Zellen aus gut 6WP 1 zu 1 oder 2 Kryokonservierung Fläschchen, bzw. (500 µL/Vial). Jedes Rohr fügen Sie 500 µL 2 x Einfrieren Mittel enthaltenden hPSC Mittel, 20 µM RI und 20 % DMSO hinzu.
Hinweis: (Optional) Zellen können direkt im 1 x Einfrieren Medium bei 1 mL/Vial übertragen werden. - Legen Sie die kryogene Rohre in einem kryogenen Behälter (mit Isopropanol) vorgekühlt auf 4 ° C und laden sofort bei-80 ° C.
- Am nächsten Tag übertragen Sie die kryogene Rohre auf eine LN2 Tank für die Langzeitspeicherung.
3. Protokoll 2: 3D "organoide" Differenzierung
-
Setup der Differenzierung mit modifizierten G-Methode der Passagierung von hPSCs
- Übertragen Sie das erforderliche Volumen des NMM für die Anzahl der Ziel-Brunnen in ein steriles Röhrchen. Ergänzen Sie mit 4 ng/mL FGF2 und 10 µM RI. Das mittlere Rohr bei RT oder im Wasserbad bei 37 ° c warm
Hinweis: Je nach Zusammenfluss der Entstehung Brunnen, sind hPSCs bei der Verteilung auf die Ziel-Platte mit einem 2:1 oder 3:1 Verhältnis (d.h. 2 Brunnen von der Originalplatte zu 1 auch der Ziel-Platte), mit einem Ende Volumen von 2,5 mL/gut konzentriert der 6WP. - Aspirieren Sie das Medium aus Brunnen mit hPSCs zu unterscheiden, und waschen Sie die Zellen, die zweimal mit 0,5 mM EDTA. Auf dem zweiten waschen, warten 30 s vor dem Absaugen von EDTA, dann fügen Sie 1 mL PBS und 4-9 min bei 37 ° c inkubieren Während des Wartens, bereiten Sie ein steriles Röhrchen mit 4 mL PBS.
Hinweis: 1 mL/Brunnen des 6WP ist ausreichendes Volumen an EDTA zum Waschen. (Wichtig) Es empfiehlt sich, hPSCs zu verwenden, die nach den letzten Durchgang und mindestens 1-2 Passagen nach dem Auftauen nicht länger als 3 Tage in Kultur waren. - Überprüfen Sie Brunnen unter dem Mikroskop (Ziele: 2.5x/0.06, 5 x / 0,12 Ph0, 10 x / 0,25 Ph1). Wenn Zellen beginnen zu lösen, die Zellen durch sanftes Klopfen auf den Seiten der Platte frei. Übertragen Sie die Zellen auf ein Rohr mit 4 mL PBS mit einer 5-mL-Pipette.
Hinweis: (wichtig) Light Spülung und Zerreibung Kolonien aufbrechen und sammeln Sie lose Zellen, sondern ignorieren Sie Zellen, die die Platte eingehalten bleiben darf. - Lassen Sie den Schlauch für 10 min bei RT, für Schwerkrafttrennung sitzen. Zentrifugieren Sie optional die Zellen leicht (nicht größer als 200 X g bei RT, 5 min) bei Bedarf.
- Absaugen der PBS aus der Tube, kümmert sich nicht um ausgeglichene hPSCs entfernen Aufschwemmen der Zellen in NMM mit 4 ng/mL FGF2 und 10 µM RI, und verteilen Sie dann auf die AA-beschichtete Platte in ein Verhältnis 2:1 oder 3:1. Inkubation bei 37 ° C mit 5 % CO2, und aktualisieren Sie das Medium nicht für 3 Tage, sofern nicht erforderlich (siehe Punkt 3.2.1).
Hinweis: (Optional) für die Bequemlichkeit der Übertragung von Zellen, Brunnen der Ziel-Platte vor der Verteilung einen Teil des Kulturmediums hinzu, und Aufschwemmen der Zellen in ein kleineres Volumen. Darüber hinaus können hPSCs eingefärbt und gezählt werden, um die genaue Start Zelldichte definieren. Allerdings sollte das Endvolumen in der Ziel-Platte 2,5 mL/Brunnen des 6WP.
- Übertragen Sie das erforderliche Volumen des NMM für die Anzahl der Ziel-Brunnen in ein steriles Röhrchen. Ergänzen Sie mit 4 ng/mL FGF2 und 10 µM RI. Das mittlere Rohr bei RT oder im Wasserbad bei 37 ° c warm
-
Erhalten Sie freischwebende Differenzierung Kultur bei 37 ° C (5 % CO2)
- Überprüfen Sie die Platten jeden Tag für Veränderungen im Medium Farbe, Anhäufung von abgestorbenen Zellen, verklumpen und Termintreue Böden gut.
- Optional: Unabhängig von den mittleren Änderungsplan aktualisieren (einschließlich der ersten 3 Tage ohne erfrischende) das Medium wenn es geworden ist gelb, und befolgen Sie die Anweisungen für die mittlere Änderung/Aktualisierung (Schritt 3.3). Wenn der Großteil der Zellen tot erscheinen, folgen Sie den Anweisungen für die mittlere Änderung/Aktualisierung mit Schwerkraftabscheidung (Schritt 3.4).
Hinweis: Bildung von EBs und Wachstum in großzellige Aggregate werden erwartet, aber Zellen und Zellen Aggregate können in großen Massen nicht aufgrund einzelner Wachstum/Verbreitung verklumpen. Wenn dies beachtet wird, ist Licht Verreibung Massen aufzubrechen zulässig. Wenn Zellen anfangen, an der AA-Plattenoberfläche halten, kann den verbleibenden Inhalt des Brunnen schwimmende direkt zum neuen Brunnen oder während des Prozesses der mittlere Änderung/Aktualisierung übertragen. Versuchen Sie nicht, die Zellen zu übertragen, die die Platte eingehalten haben.
- Optional: Unabhängig von den mittleren Änderungsplan aktualisieren (einschließlich der ersten 3 Tage ohne erfrischende) das Medium wenn es geworden ist gelb, und befolgen Sie die Anweisungen für die mittlere Änderung/Aktualisierung (Schritt 3.3). Wenn der Großteil der Zellen tot erscheinen, folgen Sie den Anweisungen für die mittlere Änderung/Aktualisierung mit Schwerkraftabscheidung (Schritt 3.4).
- Am 3. Tag ändern Sie das Medium in NMM mit 4 ng/mL FGF2. Aktualisieren Sie das Medium jeden zweiten Tag.
- Am 7. Tag, ändern Sie das Medium in NMM mit 1 µM Retinoic Säure (RA), 100 ng/mL FGF8B und 4 ng/mL FGF2. Aktualisieren Sie das Medium jeden zweiten Tag.
Hinweis: (wichtig) RA ist lichtempfindlich. Schützen Sie RA-Kultur-Proben vor Licht. - Am 14. Tag ändern Sie das Medium in NMM mit 100 ng/mL FGF8B, 100 ng/mL FGF4 und 20 ng/mL FGF2. Aktualisieren Sie das Medium jeden zweiten Tag.
- Am Tag 17 ändern Sie das Medium in NMM mit 100 ng/mL FGF8B. Aktualisieren Sie das Medium jeden zweiten Tag.
- Am Tag 21 ändern Sie das Medium in NMM mit 100 ng/mL Gehirn abgeleiteten Neurotrophic Factor (BDNF) und 10 ng/mL Glia abgeleiteten Neurotrophic Factor (GDNF). Aktualisieren Sie das Medium jeden zweiten Tag.
- Am 28. Tag ändern Sie das Medium in NMM mit 100 ng/mL BDNF und 10 ng/mL GDNF, 3 ng/mL SAG, 100 ng/mL Neurotrophic Factor 3 (NT3) und 25 mM KCl Refresh das Medium jeden zweiten Tag.
- Am Tag 35 sammeln Sie die 3D Organellen für die Analyse.
- Überprüfen Sie die Platten jeden Tag für Veränderungen im Medium Farbe, Anhäufung von abgestorbenen Zellen, verklumpen und Termintreue Böden gut.
-
Änderung/Aktualisierung Differenzierung Medium für 3D culture
- Übertragen Sie das erforderliche Volumen des NFB mit den entsprechenden Komponenten (siehe Schritte 3.2.2-3.2.7 für Komponenten-Bauteilliste) in ein steriles Röhrchen. In einem Wasserbad bei 37 ° c warmen
- Kippen Sie die Platte und schütteln Sie leicht, bis die Zellen an den unteren Rändern der Brunnen zu begleichen. Sorgfältig entfernen Sie 2 mL des alten Mediums mit einer serologischen Pipette, Vermeidung von Entfernung der Zellen, dann fügen Sie 2 mL frisches Medium. Inkubation bei 37 ° C mit 5 % CO2.
Hinweis: (wichtig) Ende Laufwerk sollte 2,5 mL/Brunnen des 6WP. Wenn Verdampfung auftritt, entfernen Sie nicht 2 mL der alten Medium als dies die Zellkulturen weiter austrocknen würde; Stattdessen fügen Sie zusätzliche Mittel.
-
Änderung/Aktualisierung Differenzierung Medium mit Schwerkrafttrennung
- Übertragen Sie das erforderliche Volumen des NFB mit den entsprechenden Komponenten in ein steriles Röhrchen. In einem Wasserbad bei 37 ° c warmen
- Den Inhalt der Brunnen in ein steriles Röhrchen, und das Rohr für 10 min bei RT, für Schwerkrafttrennung sitzen.
- Verwenden Sie eine Pipette, um altes Medium aus dem Rohr zu entfernen, die Pflege nicht zu entfernen ließ sich Zellen, mit entsprechenden Komponenten in NMM Aufschwemmen und an die neue AA-beschichteten Vertiefungen verteilen. Inkubation bei 37 ° C mit 5 % CO2.
Hinweis: (Optional) für Bequemlichkeit, eine Portion Kulturmedium dürfen, zum Ziel zugesetzt werden Brunnen vor der Verteilung mit Differenzierung der Zellen in einer geringeren Lautstärke Nukleinsäuretablette. Das Ende-Laufwerk sollte 2,5 mL/Brunnen des 6WP.
4. Protokoll 3: Alternative 2D Differenzierung Kultur
-
Starten und pflegen mit den Schritten gemäß Abschnitt 3 für 3D Protokoll durch Tag 12
- Befolgen Sie Schritte 3.1-3.2.3 und ändern/aktualisieren Medium gemäß Schritt 3.3 und 3.4.
-
Zu wechseln und als 2D Monolayer Kultur pflegen
- Am Tag 13 der Differenzierung, befolgen Sie die Anweisungen zur Änderung/Aktualisierung Medium mit Schwerkraftabscheidung (Schritt 3.4), nur die Zellen/Aggregate, PLO/LAM beschichtete Platte zu verteilen (siehe Schritt 1.6) mit einem Ende Volumen von 2,5 mL/auch von 6WP.
Hinweis: Das Medium kann während der ersten Beschichtung, Termintreue und das Überleben der Zellen zu helfen mit 10 µM RI ergänzt werden. (Wichtig) Es ist wünschenswert, die Zellen in den Brunnen, um geringer Dichte oder Gedränge auf den Tellern zu vermeiden und gegebenenfalls passage (siehe Punkt 4.4) gleichmäßig zu verteilen. Die bevorzugte Größe des PLO/LAM beschichtete Platten (d. h., 6WP, 12WP, etc.) muss empirisch ermittelt werden, anhand der Proliferationsrate für die Zell-Linie und der Zweck für das Produkt. Anweisungen geben Volumen für 6WP und konvertiert werden können, durch die Halbierung für jede Verdoppelung der gut Anzahl (z.B.2 mL/gut für 6WP, 1 mL/gut für 12WP, etc.) - Am 14. Tag ändern Sie das Medium in NMM mit 100 ng/mL FGF8B, 100 ng/mL FGF4 und 20 ng/mL FGF2. Aktualisieren Sie das Medium täglich wie in Schritt 4.3 beschrieben.
- Am Tag 17 ändern Sie das Medium in NMM mit 100 ng/mL FGF8B. Aktualisieren Sie das Medium täglich wie in Schritt 4.3 beschrieben.
- Am Tag 21 ändern Sie Mittel-bis NMM mit 100 ng/mL BDNF und 10 ng/mL GDNF. Aktualisieren Sie das Medium täglich wie in Schritt 4.3 beschrieben.
- Am 28. Tag ändern Sie das Medium in NMM mit 100 ng/mL BDNF und 10 ng/mL GDNF, 3 ng/mL SAG 100 ng/mL NT3 und 25 mM KCl Refresh des Mediums täglich wie in Schritt 4.3 beschrieben.
- Am Tag 35 Zellen für die Analyse zu sammeln, oder pflegen in dem gleichen Medium als Schritt 4.2.5 für erweiterte Kultur (mögliche Grenze nicht getestet).
- Am Tag 13 der Differenzierung, befolgen Sie die Anweisungen zur Änderung/Aktualisierung Medium mit Schwerkraftabscheidung (Schritt 3.4), nur die Zellen/Aggregate, PLO/LAM beschichtete Platte zu verteilen (siehe Schritt 1.6) mit einem Ende Volumen von 2,5 mL/auch von 6WP.
-
Änderung/Aktualisierung Differenzierung Medium für 2D Kultur
- Übertragen Sie das erforderliche Volumen des NFB mit entsprechenden Komponenten (siehe Schritte 4.2.2-4.2.5 für Komponenten-Bauteilliste) in ein steriles Röhrchen. In einem Wasserbad bei 37 ° c warmen
- Aspirieren Sie das Medium aus Brunnen, dann fügen Sie 2 mL neues Medium. Inkubation bei 37 ° C mit 5 % CO2.
Hinweis: (Optional) Ende Volumen kann gehalten werden, bei 2,5 mL/auch von 6WP, mit einer Pipette, um 2 mL des alten Mediums zu entfernen und Hinzufügen von 2 mL frisches Medium. Reservieren einen Teil der alten kann Medium in den Brunnen, und verhindern, dass Zellen von Luftkontakt, Schock für die Zellen während Änderungsschritte vermindern.
-
Durchgang 2D Differenzierung Kultur
- Übertragen Sie die gewünschte Lautstärke des NFB mit den entsprechenden Komponenten (siehe Schritte 4.2.2-4.2.5 für Komponenten-Bauteilliste) auf sterilen Röhrchen für Passagierung Prozess und separat, um PLO/LAM vorzubereiten beschichtete Platte für den Empfang von Zellen passagiert. Ergänzen Sie das Medium der Ziel-Platte mit 10 µM RI. Die mittlere Rohre bei RT oder in einem Wasserbad bei 37 ° c warm Um Komponenten zu sparen, ist es möglich, NMM allein verwenden Sie zum Waschen von Zellen während des passaging Prozesses.
Hinweis: (wichtig) wenn Produkte in der Kalzium-Bildgebung verwendet werden experimentiert, Durchgang Zellen zwischen 2 bis 6 Tage vor dem Ende der Differenzierung zu gewährleisten. - Aspirieren Sie das Medium aus Brunnen, passagiert werden. 300 µL/Well von Trypsin-basierte Dissoziation Agent hinzufügen (siehe Tabelle der Materialien), Wirbel Platte Brunnen zu decken, dann entfernen sofort die Dissoziation-Agent.
- Lassen Sie die Platte für 2 min bei RT sitzen, dann lösen Sie die Zellen durch Tippen auf Seiten der Platte. Fügen Sie 600 µL/Well definierten Trypsin-Inhibitor (DTI) und übertragen Sie die Zellen in DTI auf ein steriles Röhrchen mit 5 mL NMM.
- Zentrifugieren Sie das Rohr auf 290 X g für 15 min bei RT Aspirat das Medium und eine weitere 5 mL NMM am Rohr.
- Zentrifugieren Sie das Rohr auf 290 X g für 15 min bei RT Aspirat Medium und Aufschwemmen der Zellen in das entsprechende Medium mit RI.
- Verteilen der Zellen auf der PLO/LAM-Platte mit ein splitting-Verhältnis von 1:1-1:12, je nach der anfänglichen Zusammenfluss, Proliferationsrate und Größe differential im Ursprung auf die Ziel-Platte, und bei 37 ° C mit 5 % CO2zu erhalten.
- Übertragen Sie die gewünschte Lautstärke des NFB mit den entsprechenden Komponenten (siehe Schritte 4.2.2-4.2.5 für Komponenten-Bauteilliste) auf sterilen Röhrchen für Passagierung Prozess und separat, um PLO/LAM vorzubereiten beschichtete Platte für den Empfang von Zellen passagiert. Ergänzen Sie das Medium der Ziel-Platte mit 10 µM RI. Die mittlere Rohre bei RT oder in einem Wasserbad bei 37 ° c warm Um Komponenten zu sparen, ist es möglich, NMM allein verwenden Sie zum Waschen von Zellen während des passaging Prozesses.
Ergebnisse
Visuelle Übersicht über verringertes Wachstum Faktor 2D und 3D zerebelläre Differenzierung Protokolle
Abbildung 1 zeigt den Gesamtzeitplan für die 2D und 3D zerebelläre Differenzierung Protokolle, Identifizierung von äußeren Faktoren und Zeit der Beschichtung. Die typische Fortschritt für hPSCs in 3D zerebelläre Differenzierung ist in Abbildung 2dargestellt: mit hESC Linie H01 ab als Kolonien Feeder-freie Kultur am Tag 0 (Abbildung oben links); in der EB Bildung von Tag 2 (oben Mitte); wachsen zu größeren Aggregaten der Zelle mit scheinbaren Lumen nach neuronalen Induktion mit RA und FGF8 am Tag 14 (oben rechts); Bildung von Aggregaten von unterschiedlicher Größe und Form am 28. Tag (links); Entwicklung komplexer mit unterschiedlichen Strukturen von einem einzigen Aggregat angegeben am Tag 28 (Mitte unten); und mit morphologische Veränderungen in den gleichen Strukturen am Tag 35 (unten rechts). Die typische Fortschritt für hPSCs in 2D zerebelläre Differenzierung ist in Abbildung 3dargestellt: eine Linie mit hESC H01 als Kolonien im Feeder-freie Kultur am Tag 0 (oben links der Abbildung, mit Angabe der Fläche von differenzierten Zellen unter hESC Kolonien Kreis) ; in der EB Bildung von Tag 2 (oben Mitte); wachsen zu größeren Aggregaten der Zelle mit scheinbaren Lumen nach neuronalen Induktion mit RA und FGF8 am 13.Tag (oben rechts); wuchernden als adhärente Zellen nach Beschichtung am 14. Tag (links); und dann als eine Monolage von Zellen mit mehr komplexe/ältere Morphologie am Tag 35 unter Low (Mitte unten) und hoher Vergrößerung (unten rechts).
3D Produkte ausstellen, Marker und Strukturen der frühen Neuroepithelium
Abbildung 2 (Bild unten links) und Abbildung 4 zeigt die Heterogenität der 3D aggregierte Morphologie im ganzen Kultivierung, aufgrund der unterschiedlichen Wachstum und/oder Reifung, sowie das stochastische zusammenführen oder Auseinanderbrechen der Aggregate. Trotz Heterogenität produziert jede Differenzierung Aggregate ausstellen frühe neuronale und neuronale Marker, einschließlich zerebelläre Granulat Marker ZIC1, wie durch Immunocytochemistry (ICC) Färbung in Abbildung 5dargestellt. Noch wichtiger ist, Abbildung 5 und Abbildung 6 deuten darauf hin, dass eine einfache 3D Culture mit reduzierten Wachstumsfaktoren, geeignet zur Erzeugung von Aggregaten mit komplexen Strukturen, die im Zusammenhang mit der Entwicklung des Gehirns wie die frühen Neuroepithelium und rhombischen Lippe ist.
2D Produkte ausstellen, zerebelläre Marker und funktioneller neuronaler Aktivität
Während 2D Kulturen komplexe 3D-Strukturen nicht reproduzieren können, sind sie geeignet zur Erzeugung Zellen ausstellenden frühe neuronale und neuronale Marker, einschließlich zerebelläre Granulat Zellmarkierung ZIC1, wie durch ICC Färbung in Abbildung 7gezeigt. Gen Expressionsanalyse über RT-PCR, unterstützt wie in Abbildung 8zu sehen ICC Färbung Ergebnisse, obwohl die Anwesenheit von frühen Granulat Zellmarkierung ATOH1 Variable zwischen Experimenten und Linien ist. Calcium Imaging wird leichter in 2D Kultur behandelt. Wie in Abbildung 9, ergänzende Video 1und ergänzenden Video 2gesehen, zeigen elektrisch stimulierte Zellen Kalzium Zuflüsse, die typisch für neuronaler Impulsmuster vorschlagen Generation von funktionelle Neuronen sind.
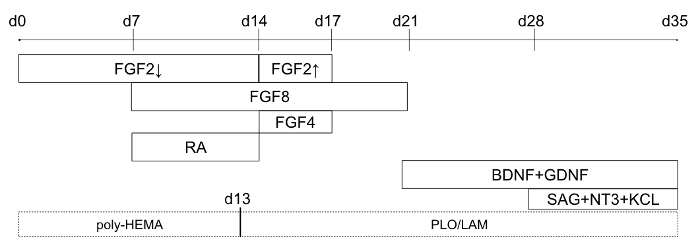
Abbildung 1: Chronologie der Differenzierung Protokoll (ab Tag 0 der Differenzierung). Durchgezogene Linie Kästchen zeigen an, wenn bestimmte Faktoren das Kulturmedium hinzugefügt sind und gepunktete Linie Kästchen zeigen an Teller-Beschichtung für optionale 2D Modifikation. Für FGF2 der Pfeil nach unten bezieht sich auf niedrigeren Konzentrationen (4 ng/mL), und der Pfeil nach oben auf höhere Konzentrationen (20 ng/mL). Diese Zahl wurde von Holmes und Heine10geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
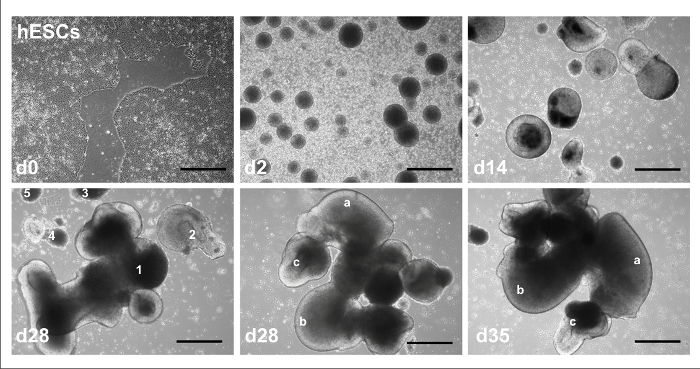
Abbildung 2: Vertreter Brightfield Bilder 3D Protokolls. hESC Kolonien an d0, EBs bei d2, aggregiert nach Induktion bei d14, Aggregate unterschiedlicher Größe und Morphologie (nummeriert 1-5) bei d28, ein einziges Aggregat mit einzigartigen, identifizierbare Merkmale (gekennzeichnet durch Buchstaben einen–c) bei d28, und sichtbare Veränderungen in die gleichen Funktionen bei d35. Maßstabsleiste = 100 µm. Diese Zahl wurde von Holmes und Heine10geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Vertreter Brightfield Bilder von 2D Protokoll. hESC Kolonien Aggregate ein d0, EBs bei d2, nach Induktion bei d13, nach Beschichtung Aggregate bei d14, nach der Reifung bei d35, gesehen bei 5 x Vergrößerung und 20 X Vergrößerung. Der weiße Kreis in der oberen linken Seite zeigt den Bereich der differenzierten Zellen unter hESC Kolonien. Maßstabsleiste = 50 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
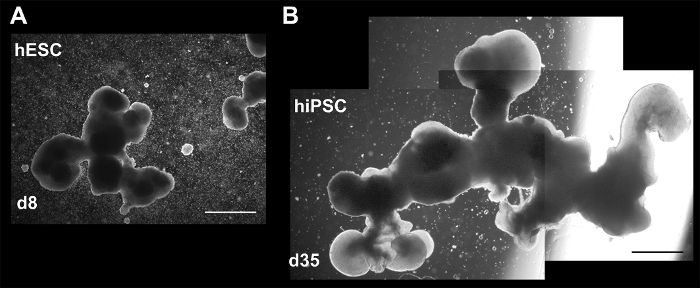
Abbildung 4: Hellfeld Bilder unterschiedlicher Größe und Komplexität in 3D Culture. Aggregate (A) Tag 8 (hESC) und (B) d35 (HiPSCs). Das letztere Bild besteht aus drei separate Bilder um das gesamte Aggregat zeigen. Beide Aggregate können durch die Zusammenlegung von kleineren Aggregaten oder Verlust (Abbruch) Strukturen betroffen. Maßstabsleiste = 200 µm. Diese Zahl ist von Holmes und Heine10neu aufgelegt. Bitte klicken Sie hier für eine größere Version dieser Figur.
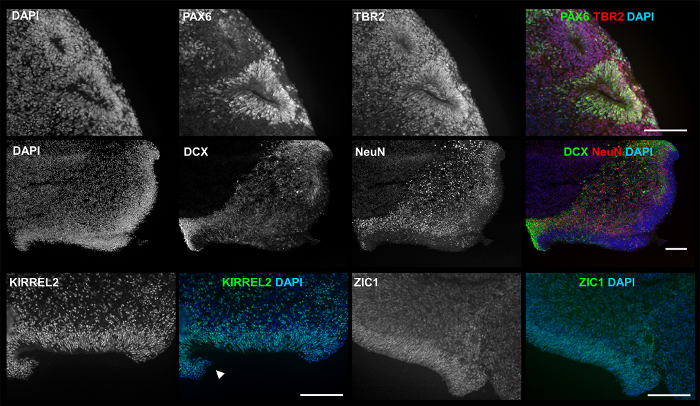
Abbildung 5: ICC Bilder 3D Produkte zeigen relevante Marker und Strukturen. Auf Kultur d35, 3D Produkte ausstellen: PAX6 (grün) und TBR2 (rot) von Lumen der neuronalen Rosette-wie Anordnung (erste Reihe); DCX (grün) und NeuN (rot) Verbreitung von Außenkante ventrikuläre Zone (VZ)-wie Struktur (zweite Zeile); KIRREL2, eine Markierung zugeordneten zerebelläre Neuroepithelium (dritte Reihe, links); und ZIC1 eine Markierung im Zusammenhang mit zerebelläre Granulat-Zellen (dritte Zeile, rechts). Das Experiment wurde durchgeführt, mehrere Male mit verschiedenen hPSC vierzeilig: hESC Linie H01 (n = 5), und iPSC Linien hvs88 (n = 4), hvs60 (n = 3), und hvs51 (n = 1). Pfeile zeigen auf rhombischen Lippe (RL)-wie Struktur. Maßstabsleiste = 100 µm. Diese Zahl ist von Holmes und Heine10neu aufgelegt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6: groß angelegte ventrikuläre Zone-artige Struktur in 3D Produkt. Bei Kultur d35 ist ein Aggregat hESC abgeleitet positiv für PAX6 (grün) und TBR2 (rot) häufig im Zusammenhang mit frühen Neuronen gefunden in ventrikuläre Zonen (VZs) und subventricular Zonen (SVZs) in Vivo. (Oben) Ein Sternchen (*) markiert die apikale Seite einer VZ-ähnliche Region laufen entlang der Kante des Aggregats, mit Klammern zeigt Tiefe/Aufteilung der VZ / SVZs. (Mitte) zusammengeführten Signale zeigen zerstreuten Abschnitte von PAX6 + / TBR2-Zellen immer größer in Richtung der oberen rechten Ende der VZ. (Unten) Höhere Vergrößerung Bild des Abschnitts durch ein Rechteck in der mittleren Spalte angezeigt. Maßstabsleiste = 100 µm. Diese Zahl wurde von Holmes und Heine10geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
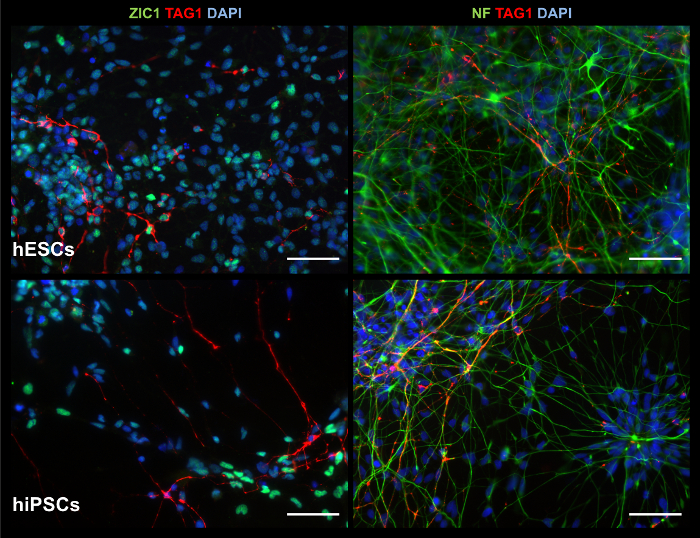
Abbildung 7: ICC 2D Produktbilder zeigen relevante Marker. Bei Kultur d35, 2D Produkte Ausstellung von Zellkulturen (obere Reihe) und HiPSCs (untere Zeile), positiv für Zellen: Granulat Zellmarkierung ZIC1 und wandernden zerebelläre Neuron Marker TAG1 (linke Spalte); und neuronale Marker Neurofilamente (NF) und TAG1 (rechte Spalte). Maßstabsleiste = 50 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
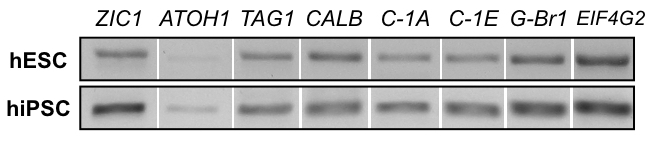
Abbildung 8: RT-PCR von 2D Produkten. mRNA Expressionsanalyse hESC Linie H01 (obere Reihe) und HiPSC Linie hvs60 (untere Zeile) am Ende der 2D Protokoll zeigt Produkte mit Gelelektrophorese für: Granulat Zellmarkierung ZIC1, Granulat Zellmarkierung ATOH1, wandernde zerebelläre Neuron Marker TAG1, Purkinje Zellmarkierung Calbindin (CALB), spannungsabhängige Calcium-Kanal CACNA1A (C-1A), CACNA1E (C-1E), Gamma - Aminobuttersäure (GABA)-B-Rezeptor 1 (G-Br1) und Hauswirtschaft gen EIF4G2).
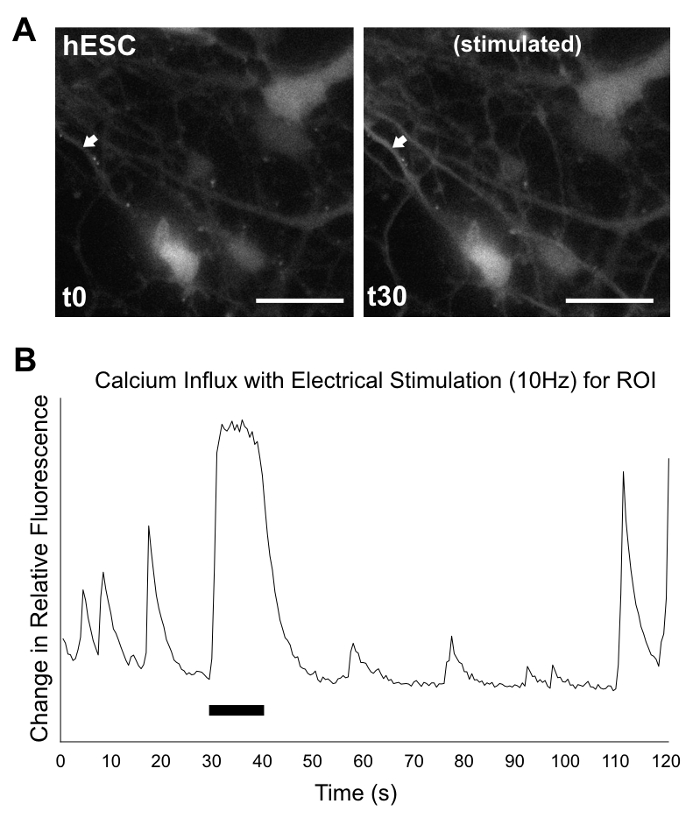
Abbildung 9: Kalzium Imaging/Analyse der 2D Produkte. Bei Kultur d35 verzeichneten hPSC Differenzierung Produkte für 2 min unter dem Mikroskop nach Inkubation mit fluor5 Farbstoff. 30 s, Zellen wurden elektrisch angeregt, für 10 s bei 10 Hz. (A) Standbilder anzeigen hESC bei 0 s (links) und nach dem Beginn der elektrischen Stimulation um 30 s (rechts). Pfeile zeigen Region of Interest (ROI) für die Analyse von Kalzium Zustrom. (B) grafische Auswertung zeigt Veränderung der relativen Fluoreszenz im Vergleich zur Zeit für ROI (Änderung der Fluoreszenz = (F-F0) / f0, wo F0 = (∑F1-n) / n), mit Neuron-wie Spikes vor, während und nach dem Auftritt die Stimulation. Schwarzer Balken gibt die Länge der elektrischen Stimulation. Skala bar (weiß) = 50 µm. vollständige Aufnahme hESC (wie hier zu sehen) und eine HiPSC Linie (nicht dargestellt) sind erhältlich in den ergänzenden Video 1 und ergänzende Video 2, beziehungsweise. Aufnahmen werden im AVI-Format, mit 4 X Geschwindigkeit. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergänzende Video 1: Kalzium imaging-Video von 2D Produkt aus hESC-Linie H01. Bei Kultur d35 färben Differenzierung Produkte hESC Linie H01 verzeichneten für 2 min unter dem Mikroskop nach Inkubation mit fluor5. 30 s, Zellen wurden elektrisch angeregt, für 10 s bei 10 Hz. Aufnahmen wurden auf 2 Bilder/s und verarbeitet in AVI-Video bei ~ 7 Bilder/s, produzieren eine Video dauert ~ 30 s, ~ 4 X Geschwindigkeit. Bitte klicken Sie hier, um diese Datei herunterladen.
Ergänzende Video 2: Kalzium imaging-Video von 2D Produkt aus HiPSC Linie hvs51. Bei Kultur d35 verzeichneten Differenzierung Produkte aus HiPSC Linie hvs51 für 2 min unter dem Mikroskop nach Inkubation mit fluor5 Farbstoff. 30 s, Zellen wurden elektrisch angeregt, für 10 s bei 10 Hz. Aufnahmen wurden auf 2 Bilder/s und verarbeitet in AVI-Video bei ~ 7 Bilder/s, produzieren eine Video dauert ~ 30 s, ~ 4 X Geschwindigkeit. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Komplexität und Kosten sind ausschlaggebend für Stammzellforscher bei der Wahl oder Differenzierung Protokolle zu entwickeln. Dies gilt vor allem, wie es eine offene Frage ist des wieviel externe Steuerung erforderlich ist, um die gewünschte Zelle Arten erzeugen oder – um es anders darstellen — wie zuständigen hPSCs sind bei der Herstellung von eigenen Entwicklungsumgebung, wenn sich selbst überlassen, mit ausreichend Nährstoffe. Einführung von äußeren Faktoren in Vitro kann sehr gut gewünschte Zelle Produkte produzieren, aber sie könnte auch stören mit der inneren Entwicklungen Kapazitäten würden Zellen in Vivoausgestellt haben. Solche Überlegungen sind wichtig, insbesondere dann, wenn das Ziel des Patienten abgeleitet iPSCs für Krankheit Modellierung nützt. Umfangreiche Verwendung von Mustern und/oder Wachstumsfaktoren könnte Krankheit Phänotypen verbergen. Die in diesem Bericht detaillierte Protokolle folgen dem Trend der früheren Studien zur Verringerung von Komplexität, Kosten und/oder Verwendung von extrinsischen Musterung Faktoren8,9.
Basierend auf Ergebnissen von Muguruma Et Al., und unsere eigene Studie berichtet, scheint es, dass es möglich ist, die Differenzierung gegenüber zerebelläre Schicksale ohne konzertierte Anstrengungen, in Vivo Bedingungen zu reproduzieren zu erreichen, wie frühere Studien gemacht haben 1 , 2 , 3 , 4 , 8 , 10. das faszinierende ist, dass die beiden Studien verschiedene Sätze von Wachstumsfaktoren verwendet, was darauf hindeutet, dass weder Satz notwendig waren, obwohl beide FGF2 verwendet. Wir wurden zusätzliche Tests, wo FGFs waren selektiv aus dem Protokoll ausgeschlossen, und zeigten, dass Zellen erzeugen die gleichen Produkte ohne extrinsische FGFs10. Unterschiede zwischen unseren Studien wurden durch die Tatsache, dass wir verschiedene hPSC Linien und Kulturmethoden verwendet, neuronale Differenzierung mit RA induzierte und Komponenten zur Unterstützung von Granulat Zelle überleben und Reifung (BDNF GDNF, SAG und KCL) enthalten qualifiziert11 –14. Darüber hinaus wurde eine weniger komplexe Startup-Methode im Vergleich zu Muguruma Et Al., beschäftigt. Ihr Protokoll begann durch die Generierung von einheitlichen EBs in 96WPs, die sie physikalisch und chemisch voneinander isoliert. Das Protokoll hier hatten alle PSCs relativ engstem in 6WPs bei EB Bildung, die ihnen erlaubt, frei zu interagieren. Wie dies differentiell die physikalische und chemische Umgebung der EBs und späteren Organellen (einschließlich intrinsische Produktion der Signalisierung Verbindungen) betroffen sind ist unbekannt, und erforscht werden konnte. Auch, während wir Expression von zeigen Genen zugeordnet – und so suggestiv von — zerebelläre Herkunft, befindet sich innerhalb der Strukturen morphologisch ähnlich denen von Muguruma Et Al., berichtet, können wir nicht ausschließen Generation von neuronalen-ähnliche Strukturen, die sind nicht-zerebelläre Identität. Zukünftige Studien berichtet mit einem großen Panel von Antikörpern wie jene von Muguruma Et al. (d.h., ATOH1, CALB, etc.) solche Zuordnungen und Vergleich zwischen den Produkten der beiden Protokolle, aussagekräftiger machen würde.
Innerhalb des 3D-Protokolls es ist wichtig, mit zu beginnen und erhalten eine ausreichende Anzahl von Zellen in Kultur in ausreichender Zahl der Endprodukte für die Analyse zu gewährleisten. Bedeutende Absterben früh in das Protokoll gegeben, empfehlen wir, beginnend mit mehr als 500 EBs/Brunnen während der ersten 3 Tage in Kultur (Abbildung 1). Dies sollte nicht schwierig sein, bestimmte Kolonie Größen für hPSCs im Feeder-freie Kultur zu erreichen, aber möglicherweise nicht so einfach, für diejenigen die immer noch mit Feeder-abhängige Methoden. Angesichts der großen Zahl von Zellen, ist es wichtig, für Farbwechsel in Medium (d. h. pH-Wert-Änderungen) und Anhäufung von abgestorbenen Zellen zu beobachten. Beide müssen korrigiert werden, um die Kultur Zusammenbruch zu verhindern. Darüber hinaus kann Verklumpen von Zellen und Aggregate in massiven Strukturen. Obwohl es noch Aggregate führen kann, die analysiert werden können, wird Produktmenge stark reduziert werden, so brechen sie in kleinere Aggregate mit sanften Verreibung nützlich sein kann. Allerdings vermeiden störende normale Aggregate, die selbst für große Größen (Abbildung 4) wachsen können. Wenn Aggregate zu spärlich werden, empfiehlt es sich, Brunnen zu kombinieren, so dass Aggregate nicht vollständig isoliert sind. Produkt-Variabilität (in Anzahl, Größe und Morphologie) ist ein bekanntes Problem in 3D Zellkultur, einschließlich der für diese Protokolle beginnend mit isolierten, einheitliche EB Bildung Schritten darauf hindeutet, dass eine weniger komplexe Startprozedur (wie das hier beschriebene Protokoll ) wäre praktischer8,15. Während diese Heterogenität ist etwas, was Forscher im Auge zu behalten, besonders während der Analyse müssen, generiert der gemeldeten Protokoll Produkte entsprechen denen in anderen 3D Protokolle8,9,15. Je nach Größe und Morphologie, fallen sie im Bereich der neuronalen Rosette zerebralen organoide, wie in den letzten Beitrag von Kelava und Lancaster15, mit den meisten passend die Klassifizierung der Sphäroid beschrieben. Besonders bemerkenswert sind das Vorhandensein von 3D-Strukturen suggestive der neuronalen Rosetten mit Lumen (Unterzonen) ventrikuläre und rhombischen-Lippe wie Funktionen (Abb. 5 und Abbildung 6) als durch andere Gruppen8 , 15 , 16 , 17. da jedes Experiment mindestens produziert Aggregat mit vermeintlichen VZ/SVZs und Kleinhirn-assoziierten Marker (ZIC1, KIRREL2), das sind nützliche Kriterien für den Erfolg einer 3D Differenzierung RL-wie unser Protokoll mit Funktionen, die zusätzliche Unterstützung. Verlängerung der Länge der Kultur seit 35 Tagen wurde nicht getestet, aber könnte verfolgt werden, um das maximale Ausmaß des Wachstums, Komplexität und Reife, die durch diese Technik erlaubt bestimmen.
Das 2D-Protokoll verwendet die gleichen nicht-anhaftende EB Bildung und neurale Induktion-Prozess als 3D-Protokoll und also die obigen Bemerkungen gelten auch. Einmal überzogen, eine andere Gruppe von Überlegungen zu berücksichtigen. Die EBs sollte schnell Zellen zu vermehren nach außen auf die Platte halten. Wenn es Probleme mit der Einhaltung, Zugabe von RI (wenn nicht bereits verwendet wird), reduziert Volumen des Mediums oder experimentelle Veränderungen in PLO/LAM Konzentration angewandt werden. Es ist wichtig, dass Zellen wachsen zu dicht oder spärlich (vorzugsweise zwischen 20-80 % Konfluenz angebaut) in den Brunnen; tägliche Kontrolle und rechtzeitige Passagierung gilt, über-Konfluenz oder schwimmende Zellen zu vermeiden. Im Gegensatz zu den 3D-Protokoll sollte es nicht signifikante Absterben während Kultur, obwohl möglicherweise schlechte Wachstumsfelder oder eine Verlangsamung der Verbreitung Preise. Passagierung betrifft die Reifung Zustand der Zellen (z. B. Zellprozesse und entwickelten Netzwerke zwischen den Zellen zu entfernen) und sollte im Verstand gehalten werden, wenn Punkte nähern, wo Zellen gesammelt oder in irgendeiner Weise analysiert werden. Beispielsweise ist für Kalzium imaging es sehr wichtig, um Durchgang Zellen zwischen 2 bis 6 Tage vor der Analyse. Zu nah an Passagierung bedeuten Analyse Zellen hatte noch keine Zeit, eine Verbindung herzustellen und/oder Reifen und zu weit führen in den Zellen Überbelegung, Bildgebung erschwert. Obwohl Variabilität zwischen Experimente vorhanden sind, sind die Ergebnisse stimmen mit denen im ersten 2D zerebelläre Protokolle1,2gemeldet. ICC Färbung und gen Expressionsanalyse bestätigen das Vorhandensein von Zellen positiv für Granulat Zellmarkierung ZIC1, während auch die Ermittlung Marker, die mit anderen neuronalen und zerebelläre Identitäten (Abbildung 7 und Abbildung 8). Kalzium-Bildgebung, die elektrischen Stimulation der Zellen inkubiert mit fluor5 Farbstoff beinhaltet, angegeben funktionellen neuronalen Aktivität (Abbildung 9, ergänzende Abbildung1und ergänzende Abbildung2), obwohl es nicht bestätigt, ist wenn diese Granulat-Zellen waren. Es ist fraglich, dass von Zellen zu Reifen durch Verlängerung der Länge der Kultur vergangenen 35 Tage mehr Zeit geben, die funktionelle neuronale Aktivität erhöhen sollte. Dieses Potenzial könnte in der Zukunft untersucht werden.
Zusätzlich zu den Linien der Forschung oben vorgeschlagen wäre es von Interesse, um Unterschiede in Produktidentitäten (Quantität und Qualität) bestimmen zwischen 2D und 3D-Protokolle. Die Bedeutung der extrinsischen FGFs wurde nicht in das 2D Protokoll getestet, und es wäre nützlich zu wissen, ob der 3D-Struktur nach Beschichtung fehlt, und so die damit verbundenen Signalwege würde 2D Kulturen mehr oder weniger abhängig machen diejenigen früh Musterung Verbindungen. Weitere abgespeckte Protokolle (z. B.kein RA, BDNF, SAG) sind gleichermaßen plausibel Linien zur weiteren Untersuchung. Schließlich könnte zukünftige Studien profitieren Sie von neuen Recherche-Tools besser zu charakterisieren (und Generation Effizienz zu beurteilen) Mensch-spezifische Kleinhirn neuronale Subtypen.
Mit dem bestimmten Vorbehalte berichteten beide Protokolle für zerebelläre Differenzierungen mit Produkten für verschiedene Zwecke geeignet verwendet werden können. Sie können als praktische Ansatzpunkte für Forscher Pilotstudien, Durchführung von Tests Lebensfähigkeit von Zelllinien für solche Differenzierungen oder als ein einfaches Modell für andere Arten von gezielte neuronale Differenzierung dienen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir sind dankbar, Gerbren Jacobs und Jurjen Broeke für ihre kompetente technische Hilfe zu Prisca Leferink zur Verfügung gestellt, die Generierung und Charakterisierung von zwei Steuerleitungen iPSC und Lisa Gasparotto zur Demonstration unserer Verfahren.
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM/F12+Glutamax | Gibco | 31331-028 | glutamine fortified DMEM/F12 |
| Neurobasal medium | Gibco | 21103-049 | neural basic medium |
| N2 supplement | Gibco | 17502-048 | |
| B27 supplement | Gibco | 17504001 | |
| Insulin | Imgen | PT468-B | |
| L-glutamine | Gibco | 25030-024 | |
| Non-essential Amino Acids (NEAA) | Gibco | 11140-035 | |
| beta-mercaptoethanol | Gibco | 21985-023 | |
| Poly-L-Ornithine | Sigma | P3655 | |
| Poly (2-hydroxyethyl methacrylate) | Sigma | P3932 | aka Poly-Hema |
| Laminin | Sigma | L2020 | |
| E8 medium and supplement | Gibco | A1517001 | hPSC medium and supplement |
| Penicillin/Streptomycin | Gibco | 15140-122 | |
| Sodium Chloride | Sigma | S-5886 | |
| y-27632 (ROCK inhibitor) | SelleckChem | S1049-10mg | |
| DMSO | Sigma | D-2650 | |
| Geltrex | Gibco | A1413302 | hPSC-appropriate adherent coating (PAAC) |
| 0,5M EDTA | Gibco | 15575-020 | |
| 0.2 um filter | VWR | 28145-77 | |
| 1.5 mL Eppendorf tube | VWR | 525-0130 | |
| DMEM/F12 | Gibco | 21331-020 | |
| Ethanol | VWR | 83804360 | |
| Parafilm | Sigma | PM996 | wrap for culture plates |
| cryotubes | ThermoFisher | 368632 | |
| TrypLE | Gibco | 12563-029 | trypsin-based dissociation agent |
| Defined Trypsin Inhibitor (DTI) | Gibco | R-007-100 | |
| FGF-2 | Peprotech | 100-18B | |
| FGF-4 | R&D Systems | 100-31 | |
| FGF-8B | Peprotech | 100-25 | |
| Retinoic Acid | Sigma | R2625 | |
| Brain Derived Neurotrophic Factor | Peprotech | 450-02 | |
| Glial Derived Neurotrophic Factor | Peprotech | 450-10 | |
| Potassium Chloride | Sigma | P5405 | |
| Neurotrophic Factor 3 | Peprotech | 450-03 | |
| Smoothened Agonist (SAG) | Cayman | 11914 | CAS 912545-86-9 |
| Axiovert 40C microscope | Zeiss | Brightfield imaging microscope | |
| Axiocam | Zeiss | Brightfield imaging - image aquisition | |
| Eppendorf Centrifuge 5810 | Eppendorf | 521-0996 | centrifuge for cell culture |
| PBS (gebufferde natrium oplossing) | Braun Medical | 3623140 | |
| 5 ml Serological pipets | VWR | 612-4950 | |
| 10 ml Serological pipets | VWR | 612-4951 | |
| 6-wells culture plates | VWR | 734-2323 | |
| 12-wells culture plates | VWR | 734-2324 | |
| hESCs | WiCELL | line H01 |
Referenzen
- Erceg, S., et al. Efficient differentiation of human embryonic stem cells into functional cerebellar-like cells. Stem Cells Dev. 19, 1745-1756 (2010).
- Erceg, S., Lukovic, D., Moreno-Manzano, V., Stojkovic, M., Bhattacharya, S. S. Derivation of cerebellar neurons from human pluripotent stem cells. Curr Protoc Stem Cell Biol. , (2012).
- Su, H. L., Muguruma, K., Matsuo-Takasaki, M., Kengaku, M., Watanabe, K., Sasai, Y. Generation of cerebellar neuron precursors from embryonic stem cells. Dev Biol. 290, 287-296 (2006).
- Salero, E., Hatten, M. E. Differentiation of ES cells into cerebellar neurons. Proc Natl Acad Sci U S A. 104, 2997-3002 (2007).
- Joyner, A. L. Engrailed, Wnt and Pax genes regulate midbrain--hindbrain development. Trends Genet. 12, 15-20 (1996).
- Joyner, A. L., Liu, A., Millet, S. Otx2, Gbx2 and Fgf8 interact to position and maintain a mid-hindbrain organizer. Curr Opin Cell Biol. 12, 736-741 (2000).
- Tam, E. W. Y., Benders, M. J. N. L., Heine, V. M. Cerebellar Development-The Impact of Preterm Birth and Comorbidities. Fetal and Neonatal Physiology. , (2017).
- Muguruma, K., Nishiyama, A., Kawakami, H., Hashimoto, K., Sasai, Y. Self-organization of polarized cerebellar tissue in 3D culture of human pluripotent stem cells. Cell Rep. 10, 537-550 (2015).
- Pasca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12, 671-678 (2015).
- Holmes, D. B., Heine, V. M. Simplified 3D protocol capable of generating early cortical neuroepithelium. Biology Open. 6, 402-406 (2017).
- Chen, J. K., Taipale, J., Cooper, M. K., Beachy, P. A. Inhibition of Hedgehog signaling by direct binding of cyclopamine to Smoothened. Genes Dev. 16, 2743-2748 (2002).
- Chen, J. K., Taipale, J., Young, K. E., Maiti, T., Beachy, P. A. Small molecule modulation of Smoothened activity. Proc Natl Acad Sci U S A. 99, 14071-14076 (2002).
- Dahmane, N., Ruiz-i-Altaba, A. Sonic hedgehog regulates the growth and patterning of the cerebellum. Development. 126, 3089-3100 (1999).
- Borghesani, P. R., et al. BDNF stimulates migration of cerebellar granule cells. Development. 129, 1435-1442 (2002).
- Kelava, I., Lancaster, M. A. Stem Cell Models of Human Brain Development. Cell Stem Cell. 18, 736-748 (2016).
- Kadoshima, T., et al. Self-organization of axial polarity, inside-out layer pattern, and species-specific progenitor dynamics in human ES cell-derived neocortex. Proc Natl Acad Sci U S A. 110, 20284-20289 (2013).
- Englund, C., et al. Pax6, Tbr2, and Tbr1 are expressed sequentially by radial glia, intermediate progenitor cells, and postmitotic neurons in developing neocortex. J Neurosci. 25, 247-251 (2005).
- Warlich, E., et al. Lentiviral vector design and imaging approaches to visualize the early stages of cellular reprogramming. Mol Ther. 19, 782-789 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten