Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Mikrosonde Capillary Electrophoresis Mass Spectrometry für einzellige Metabolomik in Live-Frosch (Xenopus Laevis) Embryonen
In diesem Artikel
Zusammenfassung
Wir beschreiben Schritte, mit denen schnell in Situ Probenahme von einen kleinen Teil einer einzelnen Zelle mit hoher Präzision und minimalen Invasion mit Kapillar-basierten Mikro-Probenahme, chemische Charakterisierung einer Momentaufnahme der metabolischen Aktivität in zu erleichtern Embryonen mit einer speziell angefertigten Einzelzelle Kapillarelektrophorese und Massenspektrometrie-Plattform zu leben.
Zusammenfassung
Die Quantifizierung von kleinen Molekülen in Einzelzellen wirft neue Potenziale für ein besseres Verständnis der grundlegenden Prozesse, die Embryonalentwicklung zugrunde liegen. Aktivieren einzellige Untersuchungen direkt in live Embryonen, neue analytische Ansätze erforderlich sind, insbesondere die sensiblen, selektive quantitative, robust und skalierbar zu verschiedenen Zellgrößen. Hier präsentieren wir eine Protokoll, die die in Situ -Analyse des Stoffwechsels in einzelnen Zellen bei der Entwicklung von Embryonen von der südafrikanischen krallenbewehrten Frosch (Xenopus Laevis), ein leistungsfähiges Modell in Zell- und Entwicklungsbiologie frei ermöglicht. Dieser Ansatz nutzt eine Kapillare Mikrosonde um einen definierten Teil aus einzelnen identifizierten Zellen des Embryos, die benachbarte Zellen intakt für die spätere Analyse Aspirieren. Die gesammelten Zellinhalt wird durch eine Microscale Kapillarelektrophorese Electrospray Ionisierung (CE-ESI) Schnittstelle gekoppelt mit einem hochauflösenden Tandem-Massenspektrometer analysiert. Dieser Ansatz ist skalierbar, um verschiedenen Zellgrößen und kompatibel mit den komplexen dreidimensionalen Struktur des sich entwickelnden Embryos. Als Beispiel zeigen wir, dass Mikrosonde einzellige CE-ESI-MS die Aufklärung der metabolischen Zelle Heterogenität, die entfaltet sich ermöglicht wie eine Vorfahr Zelle Nachkommen während der Entwicklung des Embryos entstehen. Neben Zell- und Entwicklungsbiologie sind einzellige Analyseprotokolle hier beschriebenen anderen Zellengrößen, Zelltypen oder Tiermodellen zugänglich.
Einleitung
Ein umfassendes Verständnis der embryonalen Entwicklung erfordert Charakterisierung aller molekularen Veränderungen, die in jeder Zelle von den sich entwickelnden Organismus zu entfalten. Während Next-Generation Sequenzierung mit molekulare Verstärkung ermöglicht ist tief Messung von einzelligen Transkriptom1 entwickelnde Systeme2,3, deutlich weniger über die Suite von kleineren Molekülen bekannt. produziert in einzelnen embryonalen Zellen, einschließlich Proteine und insbesondere Metaboliten (molekulare Masse < ~ 1.500 Da). Mit einer schnellen und dynamischen Reaktion auf intrinsische und extrinsische Veranstaltungen dient das Metabolom als eine mächtige Deskriptor der molekularen Zustand einer Zelle. Das einzellige Metabolom wirft daher das Potenzial, die räumliche und zeitliche Entwicklung der Zelle Heterogenität im frühen Embryo zu verfolgen und um neue Moleküle für funktionelle Studien zu identifizieren. Erkennung von das Metabolom verlangt jedoch ohne molekulare Verstärkung für diese Moleküle, außergewöhnlichen Sensibilität mit Massenspektrometrie (MS), die die Technologie der Wahl für Metabolit-Analyse.
Einzellige MS ist eine Sammlung von Technologien mit ausreichender Empfindlichkeit zu Metaboliten in Einzelzellen zu messen (siehe Bewertungen 4,5,6,7,8,9 ,10,11,12,13,14,15). Reproduzierbare Entnahme von Zellen und effiziente Gewinnung von Metaboliten sind entscheidend für den erfolgreichen Nachweis von Metaboliten in einzelnen Zellen. Ganze Zelle Dissektion der identifizierten Zellen aus Embryonen Xenopus ermöglichte die Charakterisierung von kleinen Molekülen und Peptide16. Andere Ansätze beschäftigen Mikropipetten, gefolgt von Erkennung mit Electrospray Ionisierung (ESI) MS live Einzelzellen zu probieren. Beispielsweise wurden Metaboliten in Anlage oder Säugerzellen durch einzellige video MS17, Druck Sonde18, einzelne Sonde19und fluidische Kraft-Mikroskopie20, unter anderen Techniken21gemessen, 22,23,24. Darüber hinaus vereinfacht die Einbeziehung der chemische Trennung vor der Ionisierung in den einzelligen MS Workflow effizient das Metabolom somit Linderung mögliche Störungen während Ion Generation vor Erkennung. Wichtig ist, informiert Trennung auch substanzspezifischen in molekularen Identifikationen zu unterstützen. Kapillarelektrophorese (CE) wurde zur Metaboliten in einzelnen seziert25,26 oder Microsampled27Neuronen, Erfassung von kleinen Molekül Unterschiede zwischen Neuron Phänotypen erkennen. Wir haben vor kurzem CE an ESI Tandem MS die Ablaufverfolgungsebene Erkennung von Hunderten von Metaboliten in einzelnen Zellen aktivieren, die von den frühen Embryos von Xenopus Laevis16,28seziert wurden angepasst. Diese Studien zeigten sich überraschend metabolische Unterschiede zwischen embryonalen Zellen in einem frühen Stadium der Entwicklung und führte zur Entdeckung von Metaboliten mit bisher unbekannten entwicklungspolitischen Auswirkungen16.
Hier bieten wir eine Protokoll, die den Nachweis von Metaboliten in einzelnen Zellen direkt in einem live vertebrate Embryos mit Mikrosonde einzellige CE-ESI-MS29,30aktiviert. Der Modellorganismus ausgewählt ist die 8-32-Zell laevis Embryo, obwohl der Ansatz auch zu späteren Phasen der Entwicklung und andere Arten von Modellorganismen anwendbar ist. Dieses Protokoll verwendet geschärfte Kapillaren mit mehrachsigen translational Control unter Anleitung von einem hochauflösenden bildgebenden System, ein ~ 10 nL Teil der identifizierten Zellen in Situ die morphologisch Komplex sich entwickelnden Embryos Aspirieren. Diese Mikrosonde ist skalierbar, um kleinere Zellen und betreibt innerhalb von Sekunden, das ist schnell genug, um Zelle Linien im Embryo zu verfolgen. Nach dem Extrahieren von polar oder apolaren kleine Moleküle, wie z. B. Stoffwechselprodukte und Peptide aus der erhobenen Stichprobe in ~ 4-5 µL Extraktionslösung, ~ 10 nL von der daraus resultierenden Extrakt wird in einer speziell angefertigten CE-Plattform mit Bindestrich, ein ESI-Massenspektrometer analysiert. Bau und Betrieb von der CE-ESI-MS-Plattform baut auf Protokolle an anderer Stelle beschrieben. 31 , 32 die co-axial CE-ESI-Schnittstelle ist aufgebaut, wie an anderer Stelle beschrieben. 31 diese Plattform ist in der Kegel-Jet Spritzen Regime Tracelevel Empfindlichkeit mit einer Fähigkeit zur Quantifizierung über einen 4-5 Log-Bestellung Dynamikbereich (relative28,29,30 erreichen gepflegt. oder absolute16). Die CE-ESI-MS-Plattform bietet eine Untergrenze von 60-Amol mit 8 % relative Standardabweichung (RSD) in Quantifizierung über einen geprüften Bereich von 10 nM bis 1 µM für kleine Moleküle16, die ausreichen, um endogene Metaboliten in X zu charakterisieren. Laevis Zellen. Microprobed Zellen weiter teilen im Laufe des Embryo durch Entwicklung30, zulassend zeitlich und räumlich aufgelöste Analyse des zellulären Stoffwechsels. In der Tat können einzellige CE-ESI-MS verwendet werden, um metabolische Unterschiede zwischen den Zellen zu finden, die dorsal-Ventral16,29, Tier-pflanzlichen16, und rechts-links-28 Entwicklungsstörungen Achsen sowie Zellen zu besetzen die Nervengewebe fated dorsale Abstammung von einem gemeinsamen Stammvater Zelle in laevis30bilden. Neben der Abfrage metabolische Unterschiede zwischen einzelnen embryonalen Zellen in unterschiedlichen Entwicklungsstadien des Embryos laevis 30, wir erwarten, dass die hier beschriebenen Protokolle auf ein breites Spektrum von Biomolekülen anwendbar sind und einzelne Zellen Microsampled von verschiedenen Stadien der Embryonalentwicklung sowie andere Arten von Zellen und Modellorganismen. Darüber hinaus könnte die Mikrosonde für Microsampling verwendet werden, während eine andere Plattform kompatibel mit winzigen Proben für Trennung und/oder Charakterisierung von Biomolekülen verwendet werden könnten.
Protokoll
Alle Protokolle im Zusammenhang mit der Wartung und Handhabung von Xenopus Laevis wurden durch die institutionelle Animal Care and Use Committee an der George Washington University genehmigt (IACUC keine. A311).
1. Vorbereitung der Probenahme Instrumente, Medien, Lösungsmittel und Gerichte
- 1 x Steinbergs Lösung (SS) durch Auflösen der folgenden Salze in Reinstwasser (~18.2 MΩ.cm bei 25 ° C) in der folgenden Reihenfolge vorzubereiten und in den angegebenen Konzentrationen nach einem Standard Protokoll33: Natriumchlorid (58,2 mM), Kaliumchlorid ( 0,67 mM), Calciumnitrat (0,34 mM), Magnesiumsulfat (0,83 mM), Tris-Hydrochlorid (4,19 mM) und Tris-Base (0,66 mM). Machen 0,5 x SS durch zweifache und 0,1 x SS durch Zehnfache Verdünnung des 1 x SS mit Reinstwasser.
- Bereiten Sie Probenahme Gerichte vom ersten machen 2 % Agarose in 1 x SS Autoklaven bei 120 ° C für 20 min, aufzulösen. Während noch flüssig, streichen Sie die Unterseite der 60 mm Petrischalen mit der Lösung. Sobald das Agarosegel abgekühlt und verfestigt hat, zum Jahresende eine Sechszoll Pasteurpipette Flamme bis es eine Kugel bildet, und berühren Sie leicht erwärmten Ende um 5 bis 10 Brunnen, ~ 1 mm tief, in der Agarose Impressum.
Hinweis: Diese Brunnen werden verwendet, um die Embryonen während der Probenahme zu immobilisieren. - Bereiten Sie das Stoffwechselprodukt Extraktion Lösungsmittel. Passen Sie die physikalisch-chemischen Eigenschaften des Lösungsmittels (z.B., Polarität und pH) in die Klassen von Molekülen, die Interesse an der Studie sind.
Hinweis: zum Beispiel verwenden wir 40 % Methanol und 40 % Acetonitril in LC-MS-Grade Wasser als Entdeckung Ansatz, vor allem polaren Metaboliten und ein paar apolaren Metaboliten und Peptide28Zielen. - Stellen Sie Haare Schleifen mit sauberem Haar und einer Pasteurpipette wie33 für leicht bewegliche Embryonen in die Petrischalen mit minimaler Störung an anderer Stelle beschrieben.
- Konische Spitze Mikropipetten zu fabrizieren, wie in Figur 1adargestellt.
- Ziehen Sie zuerst, Borosilikat Kapillaren (1.000/500 µm äußeren/inneren Durchmesser) in einem flammenden-Brown-Typ Kapillare Abzieher mit den folgenden Einstellungen: Wärme = 355; Pull = 65, Geschwindigkeit = 80; Zeit = 150.
- Als nächstes Abbruch der Spitze der gezogenen Mikropipette mit einer feinen scharfen Zange zu einer Kapillare Tipp Außendurchmesser von ~ 20 µm. führen Sie diesen Schritt unter einem Stereomikroskop, Präzision und Reproduzierbarkeit zu helfen.
Hinweis: Kapillaren mit ein kleines Trinkgeld sind anfällig für Verstopfung während der Aspiration von viskosen Zytoplasma. Während Kapillaren mit einer größeren Spitze sicherlich helfen Verstopfung zu vermeiden und mehr zelluläre Inhalt abzusaugen, können großkalibrigen Kapillaren Herausforderungen während der Probenahme von kleineren Zellen und möglicherweise beschädigen die Zelle für die anschließende Probenahme. Sorgfältige Einstellung des Drucks und die Uhrzeit der Aspiration können teilweise diese Herausforderungen vermindern. Wir finden Mikropipetten mit ~ 20 µm Außendurchmesser ideal für die Arbeit, die hier vorgestellt.
(2) Microsampling einzelne Zellen und Metabolit Extraktion
- Embryonen (befruchtete Eizellen) durch Gonadotropin-induzierte natürliche Paarung von Erwachsenen Xenopus Laevis zu erhalten oder über in-vitro- Befruchtung wie unter Protokolle an anderer Stelle33,34.
Hinweis: Natürliche Paarung sorgt dafür, dass die embryonalen Entwicklungsstadien gestaffelt sind, während durch die in-vitro- Befruchtung Embryonen zuverlässiger in der Lieferung sind. In-vitro- Befruchtung erfordert jedoch Einbußen bei der erwachsenen männlichen Frosch. - Frisch bereiten Sie 2 % Cystein dejellying Lösung von 4 g Cystein in 200 mL Reinstwasser auflösen zu und tropfenweise passen Sie die Lösung auf pH 8 mit 10 N Natronlauge.
- Die Gelee-Mäntel, rund um die Embryonen als sie anfangen, in der 2-Zell-Stadium zu Spalten wie folgt entfernen: lassen Sie Embryonen in die dejellying Lösung für 2 min. ruhen, dann sanft schwenken sie für eine zusätzliche 2 min zu verhindern, dass Embryonen haften an der Oberfläche der Sammlung Gericht.
- Sanft Gericht Inhalt in ein sauberes Becherglas gießen und schnell die dejellying Lösung aus dem Becherglas abgießen. Sofort decken die Eiern mit 0,1 X SS abzuspülen, die restlichen dejellying Lösung, sanft schwenken und dann die Lösung zu dekantieren. Wiederholen Sie diesen Schritt viermal die Embryonen gründlich zu waschen.
Hinweis: Maximal Exposition der Embryonen, die dejellying Lösung für 4 min um Rentabilität zu gewährleisten. Umfangreiche Protokolle, die Gelee-Mäntel zu entfernen gibt es an anderer Stelle33. - Dejellied Embryonen zu übertragen, in 1 x SS in einer Petrischale. Legen Sie zur Minimierung von Verdrängung innerhalb der Platten, ~ 100 Embryonen pro 100 mm Schale33.
Hinweis: Speisen mit Embryonen können gespeichert werden, zwischen 14-18 ° C zu verlangsamen, Entwicklung und Embryonen in gestaffelten Entwicklungsstadien von den gleichen Eltern erhalten. Weitere Leitlinien für die Temperaturabhängigkeit des Wachstums und der Entwicklung werden auf Xenbase und an anderer Stelle veröffentlicht33,35,36,37. - Art Spaltung Embryonen im 2-Zell-Stadium in eine separate Schüssel in welche Stereotypen Pigmentierung selbstbewusst markiert die dorsal-Ventral-Achse, mit Bezug auf etablierten Zelle Schicksal Karten38,39,40.
- Identifizieren Sie richtig anhaftenden Embryonen indem sichergestellt wird, die die erste Spaltung Furche, die das Flugzeug Midsagittal demarks, halbiert die dunkel (ventralen) und leicht (dorsale) pigmentiert sein Tier Pole, so dass die beiden Hälften Spiegelbilder41 sind.
- Eine vorgefertigte Mikropipette auf eine mehrachsige Mikromanipulator (manuelle oder ferngesteuerte) montieren. Die Mikropipette an einen Microinjector anschließen.
- Verwenden Sie ein Kunststoff Transferpipette Aspirieren ~ 5 der 8-Zellen-Embryonen und übertragen Sie sie in die Probenahme-Schale mit 0,5 x SS.
Hinweis: Die Zellen basierend auf Pigmentierung und Position des Embryos, mit Bezug auf Zelle Schicksal Karten38,39,40zu identifizieren.
Hinweis: zum Beispiel wir routinemäßig Aspirieren ~ 10-15 nL Volumen aus der Zelle durch die Kapillare ~ 3 Impulse von-30 Psi zuweisen. Diese ganze Schritt dauert ~ 5 s für streben30.
- Schließen Sie dicht das Fläschchen um Verdunstung zu verhindern und setzen Sie das Fläschchen wieder in den Eiskübel 4 ° C bis Probenahme abgeschlossen ist. Entsorgen Sie die gebrauchte Mikropipette in Sharps Container, eine Gefahr für die Injektionsnadeln zu verhindern.
Hinweis: Um die Menge der angesaugten Zelleninhalt bestimmen, Spritzen Sie das Aspirat in Mineralöl, wo es eine sphärische Form erhält. Der Durchmesser dieser Sphäre kann unter Verwendung eines Mikroskops gemessen werden. Berechnen Sie das abgesaugte Volumen: V = 4/3 π R3, wo V ist das Volumen, und r ist der Radius der Kugel.
(3) CE-ESI-MS-Messung
- Erarbeitung von Standards und Lösungen für CE-ESI-MS
- Bereiten Sie die Hintergrund-Elektrolyt (BGE) bestehend aus 1 % Ameisensäure in LC-MS Grad Wasser.
- Bereiten Sie die Scheide-Lösung 50 % Methanol in LC-MS Grad Wasser und 0,1 % Ameisensäure enthalten.
- 50 nM Acetylcholin-Lösung in der Scheide-Lösung für tägliche Bewertung der Leistung des CE-ESI-MS Systems vorzubereiten.
- Bereiten Sie 150 mM Natriumchlorid-Lösung als Masse-Kalibrierung Standard für den niedrigen m/Z -Bereich in den positiven Ionen-Modus. Eine Masse (m/Z) Genauigkeit der < 10 ppm empfohlen. Nach den Anweisungen des Herstellers Massenspektrometer Sie diesen Schritt durchführen.
Hinweis: Alternativ andere Standards mit bekannten m/Z -Werten lässt sich das Massenspektrometer Masse zu kalibrieren.
- Bau der CE-ESI-Plattform
- Konstruieren Sie eine CE-Injektion-Plattform in der Lage, schnelle vertikale Übersetzung einer Phase hält das BGE-Fläschchen und der Probe laden Microvial. Bau und Betrieb der Plattform finden Sie unter Details im Referenz-31.
- Die CE-ESI-Schnittstelle (Abbildung 1 c) wie folgt zusammenbauen. Einbauen des Elektrospray-Metall-Emitters (130/260 µm innerer/äußerer Durchmesser und ~ 35 mm Länge) in einer 3-Port T-Union. Feed die Trennung CE Kapillare (40/105 µm innerer/äußerer Durchmesser und ca. 100 cm Länge) durch Electrospray Emitter Adressengruppe vorstehen ~ 40-100 µm jenseits der Spitze des Senders. Arbeiten Sie unter einem Stereomikroskop um Genauigkeit zu unterstützen.
- Schließen Sie die Scheide Lösung Kapillare (75/360 µm innerer/äußerer Durchmesser und ca. 100 cm Länge) an den verbleibenden Port Elektrospray-Lösung zu liefern. Verwenden Sie geeignete Ärmel und Finger ziehen Sie Verbindungen für leckagefreier Betrieb der CE-ESI-Schnittstelle. Beziehen sich auf frühere Protokolle31,32 für Details über die Montage und Fehlersuche von dieser Schnittstelle.
Hinweis: Kapillare Maße Einfluss auf das Signal-Rausch-Verhältnis (S/N) und die Dauer der Trennung. Schmal-Bohrung und kurzen Kapillaren erleichtern beispielsweise schnelle Trennungen mit höheren Trennung Spannungen42,43. Darüber hinaus können je nach Art der Moleküle, die in einer Studie interessieren, Trennung Kapillaren zu um minimieren/vermeiden unerwünschte Molekül-Kapillare Wand Interaktionen44beschichtet werden.
- Schließen Sie die Scheide Lösung Kapillare (75/360 µm innerer/äußerer Durchmesser und ca. 100 cm Länge) an den verbleibenden Port Elektrospray-Lösung zu liefern. Verwenden Sie geeignete Ärmel und Finger ziehen Sie Verbindungen für leckagefreier Betrieb der CE-ESI-Schnittstelle. Beziehen sich auf frühere Protokolle31,32 für Details über die Montage und Fehlersuche von dieser Schnittstelle.
- Platte-Halterung befestigen, montieren Sie die CE-ESI-Schnittstelle auf einem dreiachsigen Verschiebetisch und positionieren Sie die Elektrospray-Emitter-Spitze ~ 2 mm von der Massenspektrometer-Öffnung (Abb. 1 c).
- Spülen Sie sauber Komponenten der Benutzeroberfläche indem die Elektrospray Scheide Lösung durch das Elektrospray-Emitter mit 1 µL/min und das BGE durch die Kapillare CE-Trennung. Verwenden Sie Spritzenpumpen, um die Lösungsmittel mit einer konstanten Rate zu ernähren.
- Spülen Sie die CE-Trennung-Kapillare vor jeder Messung durch den Anschluss einer Spritze an der Kapillare Meeresarm-Ende. Verwenden Sie ausreichend große Spritzen um nachfüllen und Grundierung der Lösungsmittel Versorgungsleitungen zu minimieren.
Hinweis: Experimente verwenden in der Regel gasdichte Spritzen 1 mL, um das Elektrospray-Mantel-Lösungsmittel und einer 500 µL Spritze zum Spülen der Trennung Kapillare zu liefern.
- Validierung von CE-ESI-MS-Plattform und Messung von Metaboliten
Hinweis: Das Ziel dieses Schrittes ist die analytische Empfindlichkeit des Instruments CE-ESI-MS bestätigen täglich vor der Analyse einzellige Extrakte.
- Nach dem Abspülen der Trennung Kapillare für ~ 5 min übertragen Sie den Einlass in die BGE-Lösung befindet sich in einer Durchstechflasche aus rostfreiem Stahl.
- Positionieren Sie die Elektrospray-Emitter-Spitze ~ 2 mm von der Massenspektrometer-Öffnung und Feineinstellung dieser Entfernung mit einem Verschiebetisch Elektrospray in das stabile Kegel-Jet-Regime zu generieren, bei der Überwachung des Sprays mit einem Stereomikroskop (siehe Referenzen 31,45). Überwachen Sie die Stabilität des gesamten Ionenstrom (TIC) für ~ 30-45 min zum stabilen Betrieb zu gewährleisten.
- Anwenden ~ 20 kV bis das BGE-Fläschchen durch schrittweise Hochfahren das Potenzial mehr als ~ 15 s, in der Regel generieren ~7.5 µA aktuelle über die Trennung-Kapillare mit 1 % Ameisensäure als das BGE. Gewährleisten Sie vor jeder Messung Systemstabilität zu, durch die Überwachung der TIC-Profil für ~ 5-10 min, und dann schrittweise senken Sie die Trennung (CE)-Potenzial auf 0 V (Masse).
Hinweis: Semi-automatisiert diesen Prozess, verwenden wir eine individuell geschriebenen Software CE Hochspannung Stromversorgung31fernsteuern. Wenn die CE-ESI-MS-Plattform instabil ist, sorgfältig bewerten die Quelle der Instabilität die CE-ESI-MS-Plattform zunächst in ESI-only-Modus testen und dann in die CE-ESI-Betriebsmodus als empfehlenswert an anderer Stelle31. Kurz, um die Plattform in der ESI-only-Modus testen, schalten Sie die CE-Hochspannung und überwachen Sie TIC für ~ 30 min in der Kegel-Jet-Spritzen-Regime zu. Wenn nötig, gehen Sie folgendermaßen vor um Adresse Fehler: (i) inspizieren Anschlüsse auf Dichtheit; (Ii) den Elektrospray-Emitter mit Methanol, Wasser, Isopropanol und Wasser zu reinigen; (Iii) de-Gas Lösungsmittel; (iv) Spülen Sie den Emitter für ~ 25 min vor der Prüfung wieder. Wenn die CE-ESI-Plattform sich im ESI-only-Modus stabil findet aber während CE Trennung instabil wird, untersuchen Sie das System auf Joule Heizung und/oder Elektrolyse: (i) die Trennung Kapillare mit BGE für ~ 25 min spülen und wiederholen Sie den Versuch; (Ii) Verwendung niedriger Trennung Potenziale zu einer linearen ohmschen Reaktion (d.h., aktuelle vs. Trennung Spannungskurve lineare CE); (Iii) die CE-Kapillare für mögliche Schäden, wie Risse, zu überprüfen und ggf. die Kapillare. - Analysieren von ~ 6 nL des Beispiels wie folgt:
- Pipette 1 µL der Acetylcholin-Standardlösung in der Injektion Durchstechflasche.
- Übertragen der Trennung Kapillare aus dem BGE-Fläschchen in der Injektion Durchstechflasche.
- Heben Sie die CE-Injektion-Stufe 15 cm in 1 s.
- Halten die Bühne erhöht für 60 s ~ 6 hydrodynamisch injizieren nL der Probe in die Kapillare Trennung.
- Anschließend übersetzen Sie die Bühne wieder zu Ebenen (im Einklang mit der Kapillare Austritt) ab.
- Sanft bewegen die Kapillare Meeresarm-Ende in das BGE.
- Sofort nach dem Ramp-up die CE-Spannung elektrophoretische Trennung zu starten.
- Start MS Datenerfassung.
Hinweis: Systemleistung kann mit jedem Standard-chemische charakterisiert werden. Die einzelligen CE-ESI-MS liefert niedrigere Nachweisgrenzen bei ~ 10 nM (~ 60 Amol) für Acetylcholin, Methionin und Histidin16. Das Volumen injiziert (VInj), hängt in der Kapillare in nL, der Höhenunterschied (H, cm) während der Injektion, Dichte (ρ, g cm-3) und Viskosität (η, kg m-1s-1) von der BGE, Länge (L, m) und Radius (R, µm) der CE- Kapillare und Einspritzdauer (tInj, s). Diese Beziehung wird durch folgende Formel ausgedrückt wo befindet sich die Erdbeschleunigung g (m s-2):
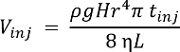
- Sobald der Standard erkannt wurde, stoppen Sie Datenerfassung zu, senken Sie die Trennung Spannung schrittweise auf 0 V (Masse), und rufen Sie dann des Emitters bis 2 cm von der Öffnung. Spülen Sie die Trennung Kapillare für 5 min vor der Analyse des Zelle-Extraktes.
- Maßnahme 10 nL von der einzelnen Zelle extrahieren durch Wiederholen der Schritte 3.3.1-3.3.5 mit 90 s hydrodynamisch Probe injizieren.
Hinweis: Das Ziel der Datenverarbeitung ist zur Identifizierung und Quantifizierung von Verbindungen zwischen den einzelnen Zellen. Das einzellige CE-ESI-MS Protokoll generiert schmale Electropherographic Gipfel mit typischen Basis breiten von wenigen Sekunden. Durch Datenanalyse Semi-manuell durchführen, ist es möglich, molekulare Merkmale (einzigartige m/Z -Werte mit einzigartigen Zugzeiten) zu finden mit den folgenden Schritten. Repräsentative Trennung wird für ausgewählte identifizierten Metaboliten in Abbildung 2aangezeigt.
- Masse-kalibrieren Rohdatendateien post Datenerfassung.
Hinweis: Wir verwenden Signale aus Natrium Formiat Clustern, die generiert werden, während der Trennung von reichlich Natrium-Ionen aus der Probe, die nativ in den Zellen oder extrahiert aus der Embryo Nährmedien sind. Das Ziel dieses Schrittes soll Metabolit Identifikation in späteren Schritten erhöhen, die eine hohe Masse (m/Z) Genauigkeit, vorzugsweise < 5 mDa oder < 10 ppm zwischen m/Z 50-1, 000. Hier, Post-Daten-Übernahme-Kalibrierung ermöglicht es, routinemäßig Masse Genauigkeiten zu erreichen < 1 mDa oder < 2 ppm für m/Z 50-500. - Verwenden ein verarbeitende Skript, Suche nach molekularen Funktionen über die erkannten Massenbereich. Durchschnittliche Massenspektren über jede Spitze bestimmen die genaue Masse, und notieren sich ihre entsprechenden Zugzeiten. Für die Identifizierung von massearmen Metaboliten im Bereich von m/Z 50-500, verwenden Sie ein 500 mDa Schritt Fenster molekulare Merkmale mit S/N überwachen > 3.
- Integrieren Sie die Peak-unter-der-Kurve Arbeitsbereich für jede molekulare Funktion manuell oder automatisch. Die Ergebniswerte Bereich dienen als eine Maßnahme der Metabolit Fülle.
- Molekulare Merkmale zu identifizieren, mit hohem Vertrauen wie folgt (Siehe Abb.
- Zunächst vergleichen Sie die genaue Masse der molekularen Eigenschaften gegen ein Metabolit-Datenbank (z.B., Metlin46 und Morbiditätsmuster47) mit 10 ppm Genauigkeit erhalten Sie eine Liste der vermeintlichen Masse Spiele.
- Als nächstes, bewerten diese Masse Spiele durch den Vergleich ihrer Tandem-Massenspektrum gewonnen aus der Zelle Extrakten mit Daten in der Metabolit Datenbanken oder Tandem-Massenspektrum für die entsprechende chemische standard gemessen.
- Schließlich bestätigen diese Zuordnungen durch den Vergleich der Migration der Zeit der molekularen Eigenschaften in Zellextrakte aufgenommen mit entsprechenden chemischen Standards analysiert, indem das gleiche Instrument CE-ESI-MS.
Hinweis: Um experimentelle Durchsatz zu erhöhen, finden wir in der Regel nur molekulare Merkmale, die statistisch signifikant zwischen experimentellen Bedingungen oder Zelltypen unterscheiden. Repräsentative Identifikationen sind in Abbildung 2dargestellt. Um Metaboliten mit hohen Massengenauigkeit zu identifizieren, empfehlen wir extern kalibrieren das Massenspektrometer täglich, in Echtzeit Rekalibrierung bei jeder Messung unter Verwendung eines internen Standards und/oder extern Masse-Kalibrierung jeder Messung Datei nach Datenerfassung (z.B.für Natrium Formiat Cluster hier) werden empfohlen.
Hinweis: In unseren Experimenten, online-Software-Plattformen47 wurden verwendet, um alle Schritte der nachfolgenden Datenanalyse, einschließlich der folgenden Schritte ausführen: ich) Filtern der molekularen Eigenschaften mit Vorkommen in mindestens 50 % jedes Sample-Set (z.B., Zelle Typ); (II) Normalisierung der Daten; (III) statistische (z.B., t-test) und multivariate Datenanalyse, wie z. B. Hauptkomponentenanalyse (PCA) und hierarchische Clusteranalyse (HCA). Wir verwenden p < 0,05 (Schülers t-test) markieren statistischen Signifikanz und Falten-Änderung ≥ 1,5 biologischen Bedeutung festzustellen.
Ergebnisse
Wir Beschäftigten vor kurzem Mikrosonde einzellige CE-ESI-MS Metaboliten in einzelnen identifizierten Zellen bei der Entwicklung von Xenopus Laevis Embryonen29,30frei zu charakterisieren. Die Mikrosonde ermöglicht die schnelle (~ 5 sec/Zelle), in Situ Aspiration von ~ 10 nL aus einer einzelnen Zelle, mehrere Bestrebungen der gleichen Zelle oder mehrere verschiedene Zellen in den gleichen oder späteren Stadien ...
Diskussion
Microprobe CE-ESI-MS ermöglicht die direkte Charakterisierung der Metaboliten in Einzelzellen Leben, frei zu Embryonen entwickeln. Das Herzstück des Konzepts sind zwei technische Unterkomponenten, nämlich in Situ Kapillare Microsampling und hoher Empfindlichkeit CE-ESI-MS. im Vergleich zum gesamten Zelle Dissektion, Kapillare Microsampling hat den Vorteil der schnellen Betrieb (einige Sekunden vs. 5 min / Zelle durch Dissektion), Kompatibilität mit der komplexen dreidimensionalen Morphologie von Embryonen un...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde unterstützt durch nationale Institute der Gesundheit Stipendien GM114854 (, P.N.) und CA211635 (um P.N.), Arnold und Mabel Beckman Stiftung Beckman Young Investigator gewähren (P.N.), die DuPont junge Professor Award (P.N.), der American Society for Mass Massenspektrometrie-Forschungspreis (, P.N.) und COSMOS Club Foundation Fellowships (zur R.M.O. und E.P.P.). Die Meinungen und Schlussfolgerungen in dieser Publikation sind ausschließlich diejenigen der Autoren und repräsentieren nicht unbedingt die offizielle Meinung der Finanzierungsquellen.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents for Embryo Culture Media | |||
| Potasium chloride | Fisher Scientific | BP 366-1 | |
| Magnesium sulfate | Fisher Scientific | M 65-3 | |
| Calcium nitrate | Sigma Aldrich | C1396 | |
| Cysteine | MP Biomedicals | 101444 | |
| Trizma hydrochloride | Sigma Aldrich | T3253 | |
| Trizma base | Sigma Aldrich | T1503 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Name | Company | Catalog Number | Comments |
| Metabolite Extraction Solvents | |||
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Name | Company | Catalog Number | Comments |
| Solvents and Standards for CE-ESI-MS | |||
| Formic acid (LC-MS-grade) | Fisher Scientific | A11710X1-AMP | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Acetylcholine chloride | Acros Organics | 159170050 | |
| Name | Company | Catalog Number | Comments |
| Microprobe Fabrication Setup | |||
| Micropippette puller | Sutter Instrument Co. | P-1000 | |
| Borosilicate capillaries | Sutter Instrument Co. | B100-50-10 | |
| Fine sharp forceps: Dumont #5, Biologie/Dumoxel | Fine Science Tools (USA) Inc | 11252-30 | Corrosion resitant and autoclavable. |
| Name | Company | Catalog Number | Comments |
| Microprobe Sampling Setup | |||
| Micromanipulator | Eppendorf, Hauppauge, NY | TransferMan 4r | |
| Stereomicroscope | Nikon | SMZ18 | Should be vibrationally isolated. |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Glass Pasteur Pipets ( Borosilicate, disposable) | Fisher Scientific | 13-678-20A | |
| Centrifuge | Thermo Scientific | Sorvall Legend X1R | |
| Name | Company | Catalog Number | Comments |
| CE-ESI-MS Setup | |||
| High voltage power supply | Spellman | CZE1000R | The HVPS may be controlled remotely using a low-voltage program generated by a personal computer. Caution: High voltage presents electrical shock hazard; all connective parts must be grounded or carefully shielded to prevent users from accidental exposure. |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Stereomicroscope | Amscope | SM-3BZZ | Stereomicroscope capable of 4.5× magnification, equipped with an illuminator to monitor the spraying mode of the CE-ESI interface. |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-built | This platform is capable of loading nanoliter-amounts of sample into the separation capillary via hydrodynamic injection and supplying the BGE for CE. Both interfaces described in this work were able to inject 6–10 nL of sample within 1 min into a 1 m separation capillary | |
| Stainless steel sample vials | Custom-built | ||
| Stainless steel BGE vial | Custom-built | ||
| Fused silica capillary (40 µm/105 µm ID/OD; 100 cm) | Polymicro technologies | TSP040105 | |
| Fused silica capillary (75 µm/360 µm ID/OD; 100 cm) | Polymicro technologies | TSP075375 | |
| Stainless steel emitter with blunt tips (130/260 µm ID/OD) | Hamilton Co. | 21031A | For better performance, laser-cleave and fine-polish the emitter tip. |
| Syringes (gas-tight): 500 - 1000 µL | Hamilton Co. | 1750TTL | |
| Digital multimeter | Fluke | Fluke 117 | |
| High-resolution Mass Spectrometer | Bruker Daltonics | Maxis Impact HD | High-resolution tandem mass spectrometer equipped with an atmospheric-pressure interface configured for ESI |
| Tunning mixture for mass spectrometer calibration | Agilent technologies | ESI-L G1969-85000 | |
| Data Analysis ver. 4.3 software | Bruker Daltonics | ||
| Name | Company | Catalog Number | Comments |
| Ancillary Equipment | |||
| Vacuum concentrator capable of operation at 4–10°C | Labconco | 7310022 | |
| Analytical microbalance (XSE105DU) | Fisher Scientific | 01911005 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Fisher Scientific | 88300ASP | |
| Refrigerated Incubator | Fisher Scientific | 11475126 | |
| Vortex-mixer | Benchmark | BS-VM-1000 |
Referenzen
- Tang, F. C., Lao, K. Q., Surani, M. A. Development and applications of single-cell transcriptome analysis. Nat. Methods. 8 (4), S6-S11 (2011).
- Veselovska, L., et al. Deep sequencing and de novo assembly of the mouse oocyte transcriptome define the contribution of transcription to the DNA methylation landscape. Genome Biol. 16 (209), (2015).
- Tran, D. A., Bai, A. Y., Singh, P., Wu, X. W., Szabo, P. E. Characterization of the imprinting signature of mouse embryo fibroblasts by RNA deep sequencing. Nucleic Acids Res. 42 (3), 1772-1783 (2014).
- Wang, D. J., Bodovitz, S. Single cell analysis: the new frontier in 'omics'. Trends Biotechnol. 28 (6), 281-290 (2010).
- Svatos, A. Single-cell metabolomics comes of age: new developments in mass spectrometry profiling and imaging. Anal. Chem. 83 (13), 5037-5044 (2011).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nat. Methods. 8 (4), S20-S29 (2011).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat. Biotechnol. 30 (9), 858-889 (2012).
- Rubakhin, S. S., Lanni, E. J., Sweedler, J. V. Progress toward single cell metabolomics. Curr. Opin. Biotechnol. 24 (1), 95-104 (2013).
- Kleparnik, K., Foret, F. Recent advances in the development of single cell analysis: A review. Anal. Chim. Acta. 800, 12-21 (2013).
- Zenobi, R. Single-cell metabolomics: Analytical and biological perspectives. Science. 342 (6163), 1243259 (2013).
- Gholipour, Y., Erra-Balsells, R., Nonami, H. In situ pressure probe sampling and UV-MALDI MS for profiling metabolites in living single cells. Mass Spectrom (Tokyo). 1 (1), A0003 (2012).
- Comi, T. J., Do, T. D., Rubakhin, S. S., Sweedler, J. V. Categorizing cells on the basis of their chemical profiles: progress in single-cell mass spectrometry. J. Am. Chem. Soc. 139 (11), 3920-3929 (2017).
- Lombard-Banek, C., Portero, E. P., Onjiko, R. M., Nemes, P. New-generation mass spectrometry expands the toolbox of cell and developmental biology. Genesis. 55, e23012 (2017).
- Yang, Y. Y., et al. Single-cell analysis by ambient mass spectrometry. Trac-Trends Anal. Chem. 90, 14-26 (2017).
- Lanni, E. J., Rubakhin, S. S., Sweedler, J. V. Mass spectrometry imaging and profiling of single cells. J. Proteomics. 75 (16), 5036-5051 (2012).
- Onjiko, R. M., Moody, S. A., Nemes, P. Single-cell mass spectrometry reveals small molecules that affect cell fates in the 16-cell embryo. Proc. Natl. Acad. Sci. U. S. A. 112 (21), 6545-6550 (2015).
- Mizuno, H., Tsuyama, N., Harada, T., Masujima, T. Live single-cell video-mass spectrometry for cellular and subcellular molecular detection and cell classification. J. Mass Spectrom. 43 (12), 1692-1700 (2008).
- Nakashima, T., et al. Single-cell metabolite profiling of stalk and glandular cells of intact trichomes with internal electrode capillary pressure probe electrospray ionization mass spectrometry. Anal. Chem. 88 (6), 3049-3057 (2016).
- Pan, N., et al. The single-probe: A miniaturized multifunctional device for single cell mass spectrometry analysis. Anal. Chem. 86 (19), 9376-9380 (2014).
- Guillaume-Gentil, O., et al. Single-cell mass spectrometry of metabolites extracted from live cells by fluidic force microscopy. Anal. Chem. 89 (9), 5017-5023 (2017).
- Saha-Shah, A., Green, C. M., Abraham, D. H., Baker, L. A. Segmented flow sampling with push-pull theta pipettes. Analyst. 141 (6), 1958-1965 (2016).
- Hu, J., et al. Synchronized polarization induced electrospray: Comprehensively profiling biomolecules in single cells by combining both positive-ion and negative-ion mass spectra. Anal. Chem. 88 (14), 7245-7251 (2016).
- Zhang, L. W., Vertes, A. Energy charge, redox state, and metabolite turnover in single human hepatocytes revealed by capillary microsampling mass spectrometry. Anal. Chem. 87 (20), 10397-10405 (2015).
- Zhang, L. W., et al. In Situ metabolic analysis of single plant cells by capillary microsampling and electrospray ionization mass spectrometry with ion mobility separation. Analyst. 139 (20), 5079-5085 (2014).
- Lapainis, T., Rubakhin, S. S., Sweedler, J. V. Capillary electrophoresis with electrospray ionization mass spectrometric detection for single-cell metabolomics. Anal. Chem. 81 (14), 5858-5864 (2009).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis electrospray ionization mass spectrometry. Anal. Chem. 83 (17), 6810-6817 (2011).
- Aerts, J. T., et al. Patch clamp electrophysiology and capillary electrophoresis mass spectrometry metabolomics for single cell characterization. Anal. Chem. 86 (6), 3203-3208 (2014).
- Onjiko, R. M., Morris, S. E., Moody, S. A., Nemes, P. Single-cell mass spectrometry with multi-solvent extraction identifies metabolic differences between left and right blastomeres in the 8-cell frog (Xenopus) embryo. Analyst. 141 (12), 3648-3656 (2016).
- Onjiko, R. M., Plotnick, D. O., Moody, S. A., Nemes, P. Metabolic comparison of dorsal versus ventral cells directly in the live 8-cell frog embryo by microprobe single-cell CE-ESI-MS. Anal. Methods. , (2017).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: Metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Anal. Chem. 89, 7069-7076 (2017).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nat. Protoc. 8 (4), 783-799 (2013).
- Knolhoff, A. M., Nemes, P., Rubakhin, S. S., Sweedler, J. V., Wevers, R., Lutz, N., Sweedler, J. V. . Methodologies for Metabolomics. , 119-139 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: a laboratory manual. , (2000).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods Mol Biol. 135, 331-347 (2000).
- Bowes, J. B., et al. Xenbase: a Xenopus biology and genomics resource. Nucleic Acids Res. 36, D761-D767 (2008).
- Karpinka, J. B., et al. Xenbase, the Xenopus model organism database; new virtualized system, data types and genomes. Nucleic Acids Res. 43 (D1), D756-D763 (2015).
- James-Zorn, C., et al. Xenbase: expansion and updates of the Xenopus model organism database. Nucleic Acids Res. 41 (D1), D865-D870 (2013).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Dev. Biol. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell-stage Xenopus embryo. Dev. Biol. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Dev. Biol. 120 (1), 299-304 (1987).
- Rollman, C. M., Moini, M. Ultrafast capillary electrophoresis/mass spectrometry of controlled substances with optical isomer separation in about a minute. Rapid Commun. Mass Spectrom. 30 (18), 2070-2076 (2016).
- Moini, M., Martinez, B. Ultrafast capillary electrophoresis/mass spectrometry with adjustable porous tip for a rapid analysis of protein digest in about a minute. Rapid Commun. Mass Spectrom. 28 (3), 305-310 (2014).
- Huhn, C., Ramautar, R., Wuhrer, M., Somsen, G. W. Relevance and use of capillary coatings in capillary electrophoresis-mass spectrometry. Anal. Bioanal. Chem. 396 (1), 297-314 (2010).
- Nemes, P., Marginean, I., Vertes, A. Spraying mode effect on droplet formation and ion chemistry in electrosprays. Anal. Chem. 79 (8), 3105-3116 (2007).
- Zhu, Z. J., et al. Liquid chromatography quadrupole time-of-flight mass spectrometry characterization of metabolites guided by the METLIN database. Nat. Protoc. 8 (3), 451-460 (2013).
- Wishart, D. S., et al. HMDB 3.0 The Human Metabolome Database in 2013. Nucleic Acids Res. 41 (D1), D801-D807 (2013).
- Liu, J. X., Aerts, J. T., Rubakhin, S. S., Zhang, X. X., Sweedler, J. V. Analysis of endogenous nucleotides by single cell capillary electrophoresis-mass spectrometry. Analyst. 139 (22), 5835-5842 (2014).
- Hubrecht, L., Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin). A systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1967).
- Grant, P. A., Herold, M. B., Moody, S. A. Blastomere explants to test for cell fate commitment during embryonic development. J. Vis. Exp. (71), (2013).
- Sellick, C. A., Hansen, R., Stephens, G. M., Goodacre, R., Dickson, A. J. Metabolite extraction from suspension-cultured mammalian cells for global metabolite profiling. Nat. Protoc. 6 (8), 1241-1249 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten