Method Article
Eine Blut-basierte Test zum Nachweis von ROS1 und RET Fusion Transkripte von zirkulierenden Ribonukleinsäure mit digitalen Polymerase-Kettenreaktion
In diesem Artikel
Zusammenfassung
Nachweis von zirkulierenden Ribonukleinsäure (cRNA) aus Blut besteht ein ungedeckter Bedarf in der klinischen Diagnostik. Hier beschreiben wir Methoden, die cRNA von nicht-kleinzelligem Lungenkrebs-Patienten mit empfindlichen und spezifischen digitalen Polymerase-Kettenreaktion zu charakterisieren. Die Tests treffen Entwurfsanforderungen zu Fusion Varianten innerhalb von 72 Stunden zu erkennen.
Zusammenfassung
Wir haben neue Methoden zur Isolierung und Charakterisierung von Tumor-abgeleitete zirkulierenden Ribonukleinsäure (cRNA) für Blut-basierten flüssigen Biopsie entwickelt. Robuste Erkennung von cRNA erholte sich von Blut stellt eine Lösung für ein kritischer ungedeckter Bedarf in der klinischen Diagnostik. Der Test beginnt mit der Sammlung von Vollblut in Blutentnahmeröhrchen, enthält Konservierungsstoffe, die cRNA stabilisieren. Zellfreie, Exosomal und Thrombozyten-assoziierter RNA ist in diesem Testsystem aus dem Plasma isoliert. Die cRNA ist umgekehrt zu komplementären DNA (cDNA) transkribiert und verstärkt mit digitalen Polymerase-Kettenreaktion (dPCR). Proben werden für die Ziel-Biomarker sowie ein Steuerelement gen ausgewertet. Testvalidierung enthalten maximal Erkennung, Genauigkeit und Robustheitsstudien mit analytischen Proben. Als Ergebnis dieser Studien reproduzierbar entwickelte Methode erkennen mehrere Fusion Varianten für ROS1 (C-Ros protoonkogenproduktes 1; 8 Varianten) und RET (neu geordnet, während Transfektion protoonkogenproduktes; 8 Varianten). Die Beispiel-Verarbeitung-Workflow wurde optimiert, damit Testergebnisse konsequent innerhalb von 72 Stunden nach Probeneingang generiert werden können.
Einleitung
Bis zu 25 % der nicht-kleinzelligem Lungenkrebs (NSCLC) können Patienten nicht ausreichend Gewebe für den Test zum Zeitpunkt der Diagnose haben. Selbst in Fällen, in denen Gewebe verfügbar ist, kann es nicht genügend Quantität oder Qualität durchführen molekularbiologische Tests1,2empfohlen werden. In Fällen wo es genügend Gewebe aus einer Biopsie für die molekulare Profilierung, Patienten müssen mehrere Wochen oder länger auf Ergebnisse warten, oder beginnen Sie die Behandlung ohne Molekulare Ergebnisse3,4. Allerdings ist es wichtig, dass informative molekulargenetische Diagnostik zur Verfügung gegeben das Aufkommen von mehrere gezielte Behandlungsmöglichkeiten für Patienten mit NSCLC. Testen der zirkulierenden zellfreie DNA (Blut) aus flüssigen Biopsie ist eine Lösung für die Herausforderungen der traditionellen Gewebe testen4,5,6. Aktuelle Testoptionen für umsetzbare Mutationen in NSCLC mit Blut und eine ähnliche dPCR-basierten Workflow für schnelles Ergebnis Generation gehören die epidermalen Wachstumsfaktor-Rezeptor (EGFR) Sensibilisierung der Mutationen ΔE746-A750 und L858R, EGFR Widerstand Mutation T790M , KRAS protoonkogenproduktes (KRAS) Varianten und B-Raf protoonkogenproduktes (BRAF) Variante V600E. Obwohl nicht so weit vom Feld verabschiedet, bieten im Umlauf Tumor-abgeleitete Boten-RNA (mRNA) isoliert aus flüssigen Biopsie auch wichtige klinische Informationen7,8,9. Wir haben zuvor entwickelt und berichtete über Multiplex Nachweismethoden der Echinodermen Mikrotubuli Associated Protein wie 4-anaplastische Lymphom-Rezeptor-Tyrosin-Kinase (EML4-ALK) Fusion Varianten von Blutplasma10. In dieser Studie haben wir diese Methoden, um höherer Ordnung gemultiplexten RNA ROS1 und RET, für acht Fusion Varianten innerhalb jedes Assay gehören erweitert. Ziel war es, eine schnelle, sensitive, spezifische und reproduzierbare Technik zum Nachweis dieser Fusion-Varianten aus dem Plasma von zuvor diagnostizierten Patienten mit NSCLC zu entwickeln.
Der Test wird in einer Arztpraxis mit RNA stabilisiert Blut Sammlung Röhren11eingeleitet. Diese Rohre enthalten eine Zelle Konservierungsmittel sowie RNase-Hemmer. Proben sind gelieferte Priorität über Nacht auf die zentralisierte College der amerikanischen Pathologen CAP akkreditiert/klinische Labor Verbesserung Änderungsanträge (CLIA)-zertifizierten Labor (Clinical Laboratory) für die Verarbeitung durch kompetentes Personal. Sobald das klinische Labor erhalten, wird jeder Schritt der Verarbeitung unter zugelassenen Standard Operating Procedures (SOP) durchgeführt. Vollblut wird zentrifugiert, um Plasma, zu erholen, die dann verwendet wird, zu isolieren, zirkulierenden RNA, die entweder frei im Blut oder in Kapseln Moieties, z. B. exosomen und Blutplättchen7,8,9. Zur Isolierung von RNA aus diesen Fächern wählten wir das System für die RNA-Wiederherstellung basiert auf dem Vergleich der verschiedenen Extraktionsmethoden. Die isolierte RNA ist konzentriert und umgekehrt in cDNA umgeschrieben. Verschiedene Enzyme Reverse Transkriptase und gen-spezifische Primer wurden bei der Optimierung der cDNA Synthese Methode, um zu maximieren ROS1 und RET Ziel Transkript Konvertierung10bewertet. Dies ist wichtig für niedrige Fülle Protokolle, wie z. B. Tumor-abgeleitete Fusion Varianten im Umlauf. Zu guter Letzt haben wir optimiert dPCR Primer und Sonde Konzentrationen für Multiplex Erkennung von RET oder ROS1 Fusion-Varianten und das Steuerelement gen, Glucuronidase-β (GUSB) ermöglichen. Vor der Durchführung der analytische Validierungsstudien in diesem Bericht beschriebenen wird dann die besten Bedingungen von jedem der Optimierungsstudien zu gesperrten Schlussprotokolls zusammengefasst. Dieses Protokoll und diese Ergebnisse bilden die Grundlage für eine rasche und sensible Workflow für den routinemäßigen Nachweis von seltenen Fusion Varianten im Umlauf.
Protokoll
Den Anweisungen des Herstellers Folgen für die unten aufgeführten, Reagenzien, sofern nicht anders beschrieben. Die PCR-Assays sind im Handel erhältliche Produkte entwickelt, um ROS1 und RET Fusionen zu erkennen.
1. Arbeiten mit RNA in Vorbereitung auf Reverse Transkription (RT)-dPCR: beste Laborpraxis
- Erstellen Sie eine RNase-freie Umgebung, beim Arbeiten mit RNA.
- Verwenden Sie handelsübliche Sprays kontaminierenden RNases inaktivieren sollen.
- Verwendung zertifizierter RNase-freie Reagenzien, Tipps und Röhren. Verwenden Sie Barriere Tipps für Nadeln, um Einführung von RNases oder Kreuz-Kontamination der Proben zu verhindern.
- Tragen Sie immer einen Laborkittel verhindern, dass Partikel von Kleidung in Ihrer Probe fallen. Benennen Sie eine Labor Mantel spezifische mit RNA-Verarbeitung verwenden.
- Tragen Sie Handschuhe um Probe Verunreinigungen von RNases in der Haut zu verhindern. Ändern Sie häufig Handschuhe.
Hinweis: Davon ausgehen Sie, dass Labor Oberflächen mit RNase verschmutzt sind, da sie die Umwelt ausgesetzt sind. Handschuhe, die Berührung der Haut, Haare, Türklinken, Gefrierschrank Griffe, Stifte/Marker, etc. werden als nicht mehr RNase-freie angesehen. - Nadeln, Arbeitsplatte, Zentrifugen und andere Arbeitsflächen mit einem RNase Inaktivierung Spray vor dem Gebrauch zu dekontaminieren.
- Wenn möglich, pflegen Sie eine Reihe von Geräten für den Einsatz mit RNA nur.
- Minimieren Sie Unterbrechung des Luftstroms in den Bereichen Labor, beim Arbeiten mit RNA-Proben, Partikel fallen in Proben oder Verunreinigung des Arbeitsbereichs zu verhindern.
- Store gereinigt RNA bei-80 ° c.
- Vermeiden Sie mehrere Einfrieren-Taut der RNA-Proben, da dies Verschlechterung führen kann.
2. Generation der analytischen RNA-Material für die Positivkontrollen
- Entwerfen Sie synthetischen DNA veröffentlichten mRNA-Sequenzen für Fusion Varianten von Interesse10verwenden.
- Wählen Sie für einen gegebenen Fusion-Variante eine mRNA-Fusion-Sequenz, die die Fusion Website plus ausreichend lang auf jeder Seite zur Deckung der PCR-Amplifikate flankierende enthält.
- Wählen Sie Nukleotidsequenzen zwischen 50-250 nt, die Größe des zirkulierenden RNA mit Thrombozyten angereichertem Plasma erfasst zu imitieren.
- Hinzufügen eine T7-Promotor-Sequenz (5'-CAGAGATGCATAATACGACTCACTATAGGGAGA-3 "), 5'-Ende der Zielsequenz.
- Bestellen Sie synthetische Sequenzen, wie doppelte gestrandete Desoxyribonukleinsäure (DNA) Fragmente.
- Bereiten Sie synthetischen DNA in Tris-EDTA (TE)-Puffer auf eine Endkonzentration von 10 ng/µL.
- Konvertieren Sie 60 ng synthetische DNA in RNA mit in-vitro- Transkription.
- RNA-Transkripte mit Phenol/Guanidin-basierte Reagenz12zu reinigen.
- Enthalten DNase I, RNase-freie, restliche Vorlage DNA zu entfernen.
- Konzentration des gereinigten in Vitro RNA mit einem handelsüblichen Fluorometer mit RNA-spezifische Farbstoffe und Standards zu messen. Sicherstellen Sie, dass die RNA innerhalb des Bereichs für den gewählten Standards akzeptabel ist. Verdünnung kann erforderlich sein.
- Bestätigen Sie erfolgreiche Transkription durch Gelelektrophorese mit ein 2 % Agarose-Gel gemischt mit RNA Gel Fleck und eine hohe Reichweite RNA Leiter einschließlich 50-250 nt Größenbereich.
- Belastung 500 ng des einzelnen in-vitro- RNA auf ein Gel.
- Führen Sie das Gel mit 5 V/cm.
- Visualisieren Sie einzelne Bands mit Beleuchtung zu und dokumentieren Sie die Ergebnisse.
- Bestätigen Sie die erwartete Transkript Größe für jede der Fusion-Varianten (basierend auf Design im Schritt 2.1.2).
- Erkennung von jedes in-vitro- RNA durch RT-dPCR mit aufeinander abgestimmten Variante-spezifische PCR Assay zu bestätigen (siehe Schritte 5 bis 8 des Protokolls).
- Optional: Bereiten Sie eine äquimolaren Mischung aus in-vitro- RNA, die jeweils von der Fusion-Varianten und die Kontrolle gen GUSB enthält.
- Wenn Schritt 2,9 durchgeführt wird: Erkennung von jeder der Fusion Varianten enthalten in der Steuerelement-Mischung von dPCR mit Variante-spezifische PCR-Assays (siehe Schritte 5 bis 8 dieses Protokolls) zu bestätigen.
- Ermitteln Sie gewünschte Eingabe Konzentration für analytische Positivkontrollen durch Konzentrationen von 0,25 bis 2,5 fg10Tests. Wählen Sie anhand der gewünschten Nummer Ausgabe Konzentration.
- Bereiten Sie nach der Bestätigung 10 µL Einweg-Aliquote analytische RNA für die Verwendung in der Positivkontrolle (Schritt 4.4) und bei-80 ° c vor.
(3) Spender Exemplare
- Sammeln Sie 10 mL menschlichen Vollblut-Proben in 10 mL Blutentnahmeröhrchen (BCT) mit Konservierungsstoff zellfreie RNA.
Hinweis: Alle menschliche Spendern werden Einwilligung zur Nutzung zu erforschen und keine Spender-spezifischen identifizierenden Informationen werden gesammelt oder verwendet während des Tests. - Vollblut-Proben vom BCT Hersteller angegebenen Zeitrahmen zu verarbeiten.
- Gepoolte normales Humanplasma kann eine kommerzielle Quelle für die Verwendung innerhalb der analytischen Positivkontrolle erhältlich. Bereiten Sie Einweg-, 1 mL Aliquots der gepoolten normales Humanplasma und bei-80 ° c für den Einsatz mit der Positivkontrolle (Schritt 4.4).
4. Wiederherstellung des zirkulierenden RNA aus Plasma
Hinweis: Es ist wichtig, schnell zu arbeiten, während dieses Vorgangs.
- Zentrifugieren Sie Vollblut-Rohre bei 200 X g für 20 min.
- Sammeln Sie bis zu 4 mL Plasma aus zentrifugiert Blut Sammelrohr mit einer serologischen Pipette. Achten Sie darauf, nicht zu stören oder zu Aspirieren die buffy Coat-Schicht.
- Isolieren Sie zirkulierende RNA mit einem kommerziell erhältlichen Kit, die exosomen, Thrombozyten und zellfreie RNA aus Plasma erfassen können. Isolieren Sie RNA von positive Kontrollprobe neben jeder Charge.
-
Bereiten Sie die positiv-Kontrolle für jede Charge von klinischen Proben wie folgt:
- Tauen Sie 1 mL gebündelt normales Humanplasma aliquoten (Schritt 3.3).
- Tauen Sie 10 µL analytische RNA aliquoten (Schritt 2,12).
- Bereiten Sie positive Kontrolle durch Zugabe von 10 µL analytische RNA in die normalen menschlichen Plasma-Probe, sobald das Plasma lysate Äthanol hinzugefügt worden.
- Eluieren Sie Proben mit 100 µL Nuklease-freies Wasser. Gehen Sie sofort Aufräumen mit RNA und Konzentration.
- Proben können auf nassem Eis gespeichert und für bis zu eine Stunde abgedeckt werden.
- RNA mit Spalte basierend Methode zu konzentrieren und eluieren in 9 µL RNase-freies Wasser.
- Gehen Sie sofort zu Schritt 5, oder halten Sie Proben auf nassem Eis bis zu einer Stunde.
(5) reverse Transkription von RNA in cDNA
- Konvertieren Sie konzentrierten zirkulierenden RNS-Probe in cDNA mit einem handelsüblichen reversen Transkription Reaktion Kit, einschließlich zufällige Primer (siehe Tabelle 1 für Komponenten).
Hinweis: Gen-spezifische Primer sind optional und können für Testvarianten ausgelegt werden. Primer sind anhand der Ziel-RNA-Sequenz ausgelegt. Verwenden Sie Fusion Variante Sequenzen aus Schritt 2.1.- Enthalten keine Reverse-Transkriptase-Kontrollprobe und keine RNA-Kontrollprobe (siehe Tabelle 1).
- CDNA aus reverse Transkription Reaktion unter Verwendung einer handelsüblichen DNA-Konzentrator Spin Spalte zu isolieren.
Hinweis: Dieser Schritt erleichtert die Entfernung von Enzymen, Primer und kostenlose Deoxynucleotide Triphosphate (dNTPs). - CDNA in PCR-Reaktionen sofort verwenden oder bei-80 ° c lagern.
6. digitale PCR
Hinweis: Diese PCR ist spezifisch für Tröpfchen digitale PCR (siehe Tabelle der Materialien).
-
PCR Mischung Vorsichtsmaßnahmen.
- Tragen Sie ein Einweg-Labor Mantel und Nitril-Handschuhe.
- Verwendung PCR mix Reagenzien in einem dedizierten Reagenz-Vorbereitungsbereich. Umgehen Sie cDNA in der nur-Reagenz-Vorbereitungsbereich nicht.
- Decken Sie Sonden beim Arbeiten, um sie vor Licht zu schützen. Übermäßig viel Licht kann Foto bleichen die fluoreszierenden Farbstoff an der Sonde befestigt.
- Mixe, abgedeckt und vor Licht geschützt, in einem separaten Bereich der Vorverstärkung vor cDNA ist hinzugefügt werden.
- Fügen Sie cDNA zu prüfenden PCR Mischung in einer PCR cleane Motorhaube in Vorverstärkung Gegend gelegen.
- Eine abschließende Reaktionsvolumen von 20 µL nach Tabelle 2PCR Mischungen vorbereiten.
- Verteilen Sie PCR Mischungen + cDNA, PCR-Platten.
Hinweis: Verwenden der Layoutbreite Platte wie ein Reiseführer empfohlen wird. - Decken Sie die Platte mit einer abnehmbaren Platte Versiegelung ab.
- Zentrifugieren Sie Platten kurz zum Proben an der Unterseite der Brunnen zu sammeln.
- Mix auf Platte Shaker auf eine niedrige Einstellung für 10 s.
- Zentrifugieren Sie Platten kurz zur Probe an der Unterseite des Brunnens zu sammeln.
- Entfernen Sie die Platte Sealer. Durchführen Sie Tröpfchen-Generation für PCR cDNA-Mix mit entweder eine manuelle oder automatisierte Droplet-Generation-System.
- Übertragen Sie für manuelle Tröpfchen Generation 20 µL PCR Mischung zum Beispiel Brunnen auf das Droplet Generation Patrone. 70 µL Tröpfchen Generation Öl hinzufügen. Deckel mit Kautschuk Dichtung und Transfer Patrone, manuelle tropfengenerators, Tröpfchen Generation zu initiieren. Übertragen Sie nach Tröpfchen Generation Tröpfchen auf ein frisches PCR-Platte mit Tipps vom Hersteller empfohlenen. Aspirieren und verzichten die Tröpfchen langsam, über 5 bis 6 s jeweils ohne die Öffnung der Spitze an den Tröpfchen Patrone oder Platte.
- Für automatisierte Droplet-Generation die Dichtplatte mit folienversiegelung und Übertragung auf das Droplet-Generator. Alle Tipps, Patronen, zu gewährleisten und Platten sind vor Beginn der Tröpfchen-Generation.
- Folgenden Tröpfchen Erzeugung und Übertragung von Tröpfchen mit einer frischen PCR Platte Siegel mit Folie Platte Siegler und thermischen Zyklus Platten mit den Einstellungen in Tabelle 3.
- Nach der Thermocycler läuft komplett, die Platte mit einem Tröpfchen-Reader zu lesen. Erstellen Sie eine Platte Layout für Reader-Software, die die Position der Steuerelemente, Proben, etc., identifiziert und Last in Software zu lesen beginnen.
7. Daten Analyse und Überprüfung und Generierung von Ergebnissen
- Analysieren Sie Platte lesen Ergebnisse mit im Handel erhältlichen Software.
- Navigieren Sie zum Menü "Analyze" zweidimensionale (2D) Amplitude Grundstücke anzeigen.
- Bewerten Sie die Qualität der Daten anhand der Tröpfchen-Daten.
- Werten Sie die Daten für insgesamt akzeptierten Ereignis-IDs über Menü "Veranstaltungen". Wenn es weniger als 10.000 Ereignisse pro Bohrloch, prüfen Sie sorgfältig die Daten für zusätzliche Probleme.
- Überprüfen Sie Daten für aberrante Fluoreszenz Amplituden. Bedeutende Amplitude und Konzentrationsunterschiede zwischen replizieren Proben zeigen schlechte Verarbeitung oder Vermischung der Proben.
- Machen Sie sich Notizen von Tröpfchen Clustern mit Spritzbilder auf einer 45-Grad-Achse, was bezeichnend für schlechte Qualität Tröpfchen oder problematische Proben.
- Untersuchen Sie zunächst Positivkontrolle, No Reverse Transkriptase (No RT) und No-RNA-Kontrolle (NRC) Daten. Wählen Sie alle Kontrollproben und untersuchen Sie Cluster Qualität von 2D Grundstück. Für richtige Schnittstellenüberwachung sollte eine klare Trennung zwischen den Clustern Tröpfchen ersichtlich sein.
- Für jede Probe setzen Variante, Sie den Schwellenwert basierend auf Kontroll-Vertiefungen.
- Festgelegten Schwellenwerte auf 2D Plots mit dem Fadenkreuz-Werkzeug, um die doppelte Verneinung Tröpfchen Bevölkerung aus der Kontrollgruppe gen (beschriftet mit 5'-Hexachloro-Fluorescein-CE phosphoramidit-Sonde), y-Achse und variant gen Bevölkerung zu trennen wenn präsentieren) beschriftet mit Fluorescein Amidite OR 6-Carboxyfluorescein Sonde), x-Achse.
- Summe aus jedes Replicate gut für ein einzelnes Sample kopiert.
- Express-Testergebnisse als die Anzahl der Varianten Kopien erkannt.
Hinweis: Den analytischen cutoff Wert für den Aufruf einer positiven oder negativen Probe bestimmen ein normales laufen gesunden Spenders Probe (mindestens 10 Einzelproben) über das abgeschlossene Verfahren festgelegt und etablieren die Cutoff über nachweisbaren Hintergrundsignal für die Mutation von Interesse. Darüber hinaus legen Sie die Anzahl der Kontrolle gen Kopien benötigt, um ein positives oder negatives Ergebnis nennen. Kontrolle gen Grenzwertes fungiert als eine interne Qualitätskontrolle (QC) zu bewerten, die Quantität und Qualität jeder einzelnen RNA-Probe, die verarbeitet wird.
8. Überprüfung der RT-dPCR Reaktionsbedingungen mit Zelllinien (Optional)
- Zur Erkennung von Fusion Varianten zu überprüfen, verwenden handelsübliche Zelllinien mit dem Ausdruck der ROS1 oder RET Fusion mRNA von Interesse. Gehen Sie folgendermaßen vor:
- Blitz-gefrorenen Zellen in einer Lyse Guanidinium-basierte Lösung direkt aus dem gefrorenen Zustand zu homogenisieren. Sogar kurze Auftauen vor der Homogenisierung kann dazu führen, dass RNA-Abbau und Verlust.
- Isolieren Sie RNA mit Silikon-Membran Spin Spalten für RNA konzipiert.
- Messen Sie die Konzentration von RNA-Proben mit einem Fluorometer mit RNA-spezifischen Reagenzien und Standards.
- Verdünnen Sie isolierten RNA in einen Hintergrund der Wildtyp RNA aus Plasma oder eine andere kommerzielle Quelle.
- Führen Sie die Schritte aus Reverse Transkription von RNA in cDNA, digitale PCR und Datenanalyse und Überprüfung und Generation der Ergebnisse aufgelistet in diesem Protokoll Erkennung der gewünschten Variante zu bestätigen.
Ergebnisse
Dieses Protokoll beschreibt ein Testsystem zum Nachweis von RNA-Fusion-Varianten für den Einsatz bei der Messung der Fahrer Mutationen innerhalb des Plasmas von NSCLC-Patienten (Abb. 1A) entwickelt. Fusion-mRNA, die Produkte aus dem Ausdruck der häufigsten RET und ROS1 Umstellungen in der NSCLC Bevölkerung wurden identifiziert,13,14,15,16,17. Multiplex PCR-Assays wurden dann entwickelt, um die acht am häufigsten Transkript Varianten für jedes Ziel in NSCLC innerhalb einer einzelnen Reaktion zu erkennen. Die am häufigsten verwendeten Translokationen bei ROS1 Locus generieren Assoziationen an die 5' Teile der CD74, SDC4, SLC34A2, EZR oder TPM3 Gene (Abbildung 1B). Die am häufigsten verwendeten Translokationen bei der RET -Lokus führen zu Gegenüberstellung mit KIF5B, wofür deckt der Test sechs Exon Kreuzungen. Weitere RET -Partner, die behandelt werden sind diejenigen mit CCDC6 und TRIM33 (Abbildung 1C). Insgesamt decken die Assays ca. 88 % der ROS1 und 99 % der RET-Änderungen in der NSCLC Patientenpopulation17verbreitet.
Die Spezifität des Assays Komponenten war zunächst mit acht einzelnen in-vitro- RNAs, die die mRNA-Sequenz enthalten, für die Fusion Protokolle fallenden ROS1 oder RET Assays gemultiplext ausgewertet. Jede RNA-Spezies wurde gegen jede einzelne Variante Assay getestet, die die gebündelte Version umfasst. Gab es keine Kreuzreaktivität diese Assays, bewiesen damit 100 % analytische Spezifität innerhalb des gestalteten gemultiplext Assays (Daten nicht gezeigt). Um die untere Grenze der Erkennung von Prüfprotokoll zu ermitteln, wurden Gesamt-RNS abgeleitet von Zell-Linien mit dem Ausdruck einer Fusion-Variante in der Probe enthalten in einen Hintergrund des normalen RNA bei 5 %, 1 %, 0,2 % und 0,04 % Konzentrationen gemischt. Die Multiplex RET und ROS1 Variante PCR-Assays weniger als 0,2 % Fusion Variante (Abbildung 2A-B) erkannt. Darüber hinaus eine Vorbereitung von 5 % Ziel Zelllinie abgeleiteten RNA (Ausdruck eine EML4-ALK-Fusion-Transkript) nicht mit der gemultiplexten ROS1 und RET Assays erkannt wurde weiter demonstrieren Spezifität (Abbildung 2A-B).
Präzision des RT-dPCR Prozesses Tests wurden für beide ROS1 und RET Analytic Kontrollmaterial bestehend aus äquimolaren in-vitro- RNAs in drei Konzentrationen (hoch, Mittel und niedrig) durch reverse Transkription und dPCR auf drei verarbeitet wurde verschiedene Anlässe innerhalb des gleichen Tages (Intra-Day), an drei aufeinanderfolgenden Tagen (Inter-Tag) und mit zwei Operatoren (Inter-Operator). Ergebnisse aus Präzision Tests zeigten präzise Erkennung von der Fusion Abschrift von Interesse sowie eine Kontrolle gen, GUSB, die als eine interne QC-Metrik (Abbildung 2-D) enthalten ist.
Neben der internen Steuerung GUSB wurde jede Charge von klinischen Proben mit einem Satz von Steuerelementen Batch ausgeführt. Positivkontrolle wurde aus einer Mischung von analytischen in Vitro RNA, die jede der Fusion-Varianten getestet in RT-dPCR vertreten, sowie analytische in Vitro RNA für entwickelt GUSB. Diese RNA in normalen menschlichen Plasma lysate während der RNA-Extraktion gespickt war und neben der klinischen Proben im gesamten Protokoll verarbeitet wurde. Die keine Reverse Transkriptase (No RT) Kontrolle wurde eine Negativkontrolle zu bestätigen das Fehlen von kontaminierenden Material in der RNA-Extraktion-Workflow und demonstrieren die Spezifität der Primer für RNA. Das No RT-Steuerelement generiert wurde, mit dem gleichen Material wie die positive Kontrolle, aber Enzym innerhalb der cDNA Synthese Reaktion ist nicht enthalten. Der keine RNA-Steuerelement (NRC) ist ein negativ, das Fehlen einer Kontaminierung Transkripte in der reversen Transkription reaktionskomponenten zu bestätigen. Dieses Steuerelement in den Workflow bei der cDNA Synthese Schritt eingeführt wurde, und Wasser war in der Reaktion statt einer RNA-Vorlage hinzugefügt. Nr. RT und NRC Steuerelemente müssen negativ sein, in beiden Kanälen, wenn genaue Ergebnisse geliefert werden sollen. In Tabelle 1 sind die reverse Transkription reaktionskomponenten für jedes Steuerelement. Beispiele für die 2D Grundstücke für jedes dieser Steuerelemente sind für die ROS1 (Abbildung 3 A-C) und RET (Abbildung 3 E-G) Multiplex-Assays. Fusion-Varianten wurden mittels Fluorescein Amidite (FAM) Sonde erkannt und werden entlang der y-Achse dargestellt, während das Steuerelement gen, GUSB, mittels 5'-Hexachloro-Fluorescein-CE phosphoramidit (HEX) Sonde erkannt wurde und ist auf der x-Achse. Diese Batch-Steuerelemente wurden im Laufe der 21 Tage, um festzustellen, Assay Robustheit bewertet. Fusion positive Tröpfchen und GUSB Kontrolle gen Tropfen wurden ROS1 und RET in allen 21 Läufen ausgeführt im Laufe der Studie (Abbildung 3D, H) beobachtet. Alle Negativkontrollen (No RT und NRC) hat negative Ergebnisse ergeben, über die ganzen 21 Tage (Daten nicht gezeigt).
Die Fähigkeit zu beheben ist ein wichtiger Bestandteil jeder Test-Protokoll in der klinischen Laborumgebung ausgeführt werden. Hier bieten wir praktische Beispiele von Sub-optimale Ergebnisse mit dem RT-dPCR Protokoll. Die erste ist eine Beispiel-2D-Handlung zeugt von der Bedeutung des keine Reverse-Transkriptase-Steuerelements (Abb. 4A). In diesem Beispiel waren mutierte positive Tröpfchen anwesend, obwohl gab es keine cDNA-Konvertierung aufgrund des Fehlens von Enzym. Dieses Ergebnis war wahrscheinlich wegen dPCR Primer Ziel genomischen DNA verstärken. In diesem Fall wird Design ein Intron-spanning Assay Amplifikation genomischer DNA verhindern. Alternativ ein RNase-freie DNase-Enzym kann verwendet werden, um die kontaminierende DNA zu beseitigen, aber dies wird nicht empfohlen für die Erkennung von seltenen Ziele, wie einige RNA-Abbau während der Inkubation mit dem Enzym auftreten. Das nächste Beispiel 2D Grundstück war ein NRC mit positiven Tröpfchen in beiden Kanälen (Abbildung 4B). Dies Kontamination zu einem bestimmten Zeitpunkt in der RT-dPCR Einrichtung. In diesem Fall empfiehlt es sich, alle potentiell kontaminierten Reagenzien in der Prüfung zu verwerfen, gründlich alle Geräte zu dekontaminieren und Testwiederholungen mit frischen reaktionskomponenten. Das dritte Beispiel 2D Grundstück als ein Spray von Tröpfchen entlang einer 45°-Linie (Abb. 4C) dargestellt. Dies wird oft durch Schneiden und Zusammenfügen von Tröpfchen verursacht. Sorgfältige Handhabung vor Temperaturzyklen Tröpfchen ist wichtig, da Tröpfchen anfällig für Beschädigungen sind. Wir empfehlen die Verwendung von automatisierten Droplet-Generation, wenn verfügbar. Wenn Tröpfchen Übertragung manuell generiert werden, seien Sie sicher, wählen die empfohlene weite Bohrung Tipps und vorsichtig pipettieren Technik zu beschäftigen. Tröpfchen-Übertragung erfordert langsam Aspiration und Dosieren, mit jeder statt mehr als 5-6 Sekunden, und es ist wichtig, dass die Pipette Tipp Öffnung nicht die Tröpfchen-Patrone berühren oder gut. Bei der Abgabe, halten Sie die Pipettenspitze auf den Flüssigkeitsstand und erhöhen Sie es langsam zu, wie Tröpfchen verzichtet (Demo video ansehen). Das endgültige 2D Plot-Beispiel zeigt einen Mangel an Trennung zwischen den positiven und negativen Droplet-Populationen (Abbildung 4). Dies kann mehrere Ursachen haben. Strong PCR-Inhibitoren, wie z. B. Waschmittel in Lyse Puffer und ein Übermaß an stark geschädigter DNA, können der Trennung verloren gehen. In diesem Fall erwägen Sie, einen Bereinigung Schritt zwischen cDNA Synthese und dPCR (wie in Schritt 5 dieses Protokolls beschrieben). Zu guter Letzt kann fehlende Trennung auch sub-optimale Verstärkung bedingt, und Optimierung der PCR Schritt sollten ebenfalls berücksichtigt werden.
Daten in Abbildung 5 sind 984 realen Patienten probieren Sie Turn-around-Zeiten und zeigt die schnelle Natur dieses Test-Workflows. Ergebnisse wurden an den behandelnden Arzt so früh als innerhalb von 48 Stunden (79 % der Fälle) von Probeneingang und in 95 % der Fälle innerhalb von 72 Stunden berichtet. Zusammenfassend kann die Verwendung von bieten stabilisiert zirkulierenden RNA Blutentnahmeröhrchen, optimierte RNA-Extraktionsverfahren aus Blut und RT-dPCR laufen nach ein optimiertes Protokoll mit den entsprechenden internen und Batch-Steuerung, ein Schnelltest-System für die präzise Erkennung von Fusion RNA Varianten bei NSCLC.
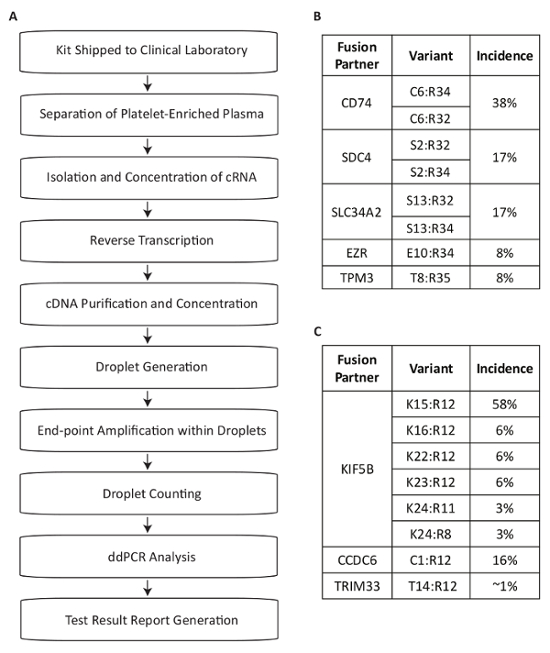
Abbildung 1 : Überblick über Blut Verarbeitung Beispielschritte für Fusion Variante Erkennung bei NSCLC spezifisch Assays für die am meisten verbreiteten RET und ROS1 Varianten. (A) Stichproben wird eingeleitet, wenn Vollblut gezogen und ein BCT ist innerhalb der Probe Sammlung Kit im klinischen Labor versandt. Zirkulierenden RNA wird aus mehreren Quellen innerhalb der Thrombozyten angereichertem Plasma gewonnen, Rückseite mit Gen spezifische Priming transkribiert und für den Einsatz in dPCR gereinigt. Proben werden verarbeitet, mit einem im Handel erhältlichen System bestehend aus Tröpfchen Generation (Emulsion), Verstärkung und Tropfen zu zählen. Daten werden unter Verwendung handelsüblichen Software analysiert. Die Testergebnisse sind dann dokumentiert und berichtet an den Arzt Test anfordern. Der Prozess soll innerhalb eines Zeitraums von 72 Stunden nach Probeneingang Ergebnis Version arbeiten. Acht Varianten für ROS1 (B) und (C) RET sind innerhalb der Multiplex-Assays gedeckt. Adaptiert von Biodesix Website mit Erlaubnis. Bitte klicken Sie hier für eine größere Version dieser Figur.
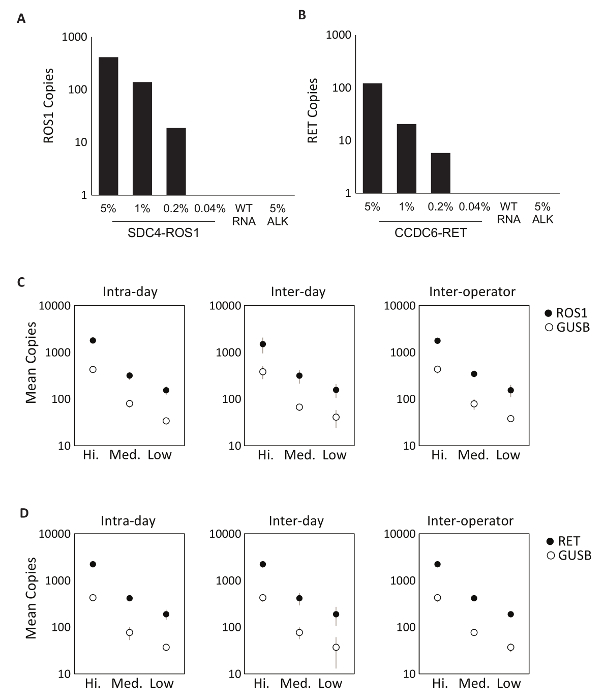
Abbildung 2 : Analytische Validierung. Zelllinien mit dem Ausdruck (A) SDC4 ROS1 Fusion und (B) CCDC6-RET Fusion wurden in einem Hintergrund der gesamten menschlichen Wildtyp RNA (WT-RNA) verdünnt. Mit jeder Fusion-Variante wurde der Nachweisgrenze bei 0,2 % Variante Frequenz mit vordefinierten Kriterien für jede Variante Assay gegründet. Alle Proben oberhalb dieser Schwelle enthielt auch mindestens 21 Kopien des Gens Kontrolle. 5 % EML4-ALK (ALK) Standard in einem Hintergrund von Wildtyp RNA wurde getestet, um die Spezifität des Assays, unter Beweis zu stellen, die durch ein negatives Ergebnis bestätigt wurde. Analytische gemultiplexten RNA-Standards wurden gemessen am hohen, mittleren und niedrige Konzentrationen für ROS1 (C) und (D) i.r. Präzision wurde über drei Testläufe am selben Tag (Intra-Day), drei Runs an drei aufeinanderfolgenden Tagen (Inter-Tag) und mit ausgewertet zwei unabhängige Marktbeteiligte (Inter-Operator). Die Mittel der Kopienzahl und Standardabweichungen werden angezeigt. Adaptiert von Biodesix Website mit Erlaubnis. Bitte klicken Sie hier für eine größere Version dieser Figur.
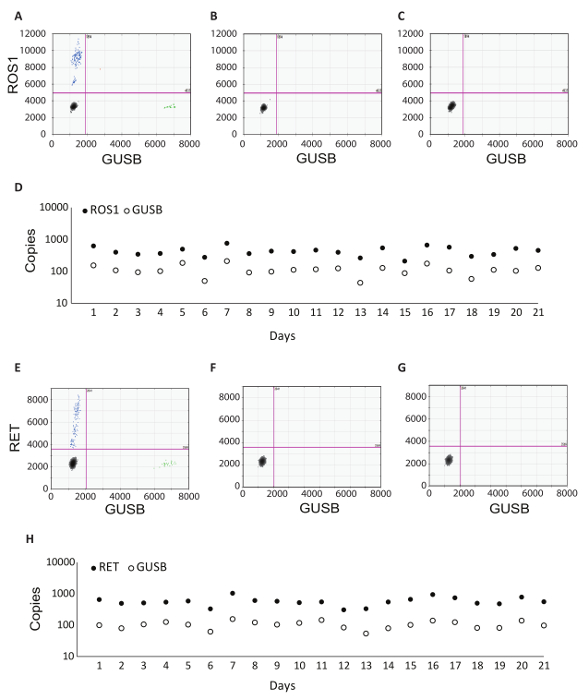
Abbildung 3 : Batch-Verarbeitung Kontrolle Beispiele und Robustheit Daten. 2D Grundstück ROS1 gemultiplext Assay dPCR ergibt sich für positive Kontrolle (A), (B) keine Reverse-Transkriptase-Steuerelement, und (C) keine RNA-Vorlage-Kontrolle. (D) Kontrollen wurden an 21 aufeinanderfolgenden Tagen (außer an Wochenenden und Feiertagen) ausgeführt. Bedeuten Sie Exemplarzahl +/-Standardabweichung für positive Kontrolle wurden 439 + / 141 ROS1. Kein Reverse-Transkriptase und keine Vorlagensteuerelemente wurden auch jeden Tag laufen, und diese waren alle negativ (Daten nicht gezeigt). 2D Grundstück von RET gemultiplext Assay dPCR ergibt sich für Positivkontrolle (E), (F) keine Reverse-Transkriptase-Kontrolle und (G) keine RNA-Vorlage-Kontrolle. (H) Kontrollen wurden an 21 aufeinanderfolgenden Tagen (außer an Wochenenden und Feiertagen) ausgeführt. Bedeuten Sie Kopien + / Standardabweichungen für positive Kontrolle wurden 586 + 182 / RET. Nicht gezeigt werden keine Reverse-Transkriptase und keine Vorlagensteuerelemente, die waren auch jeden Tag laufen und waren alle negativ. Adaptiert von Biodesix Website mit Erlaubnis. Bitte klicken Sie hier für eine größere Version dieser Figur.
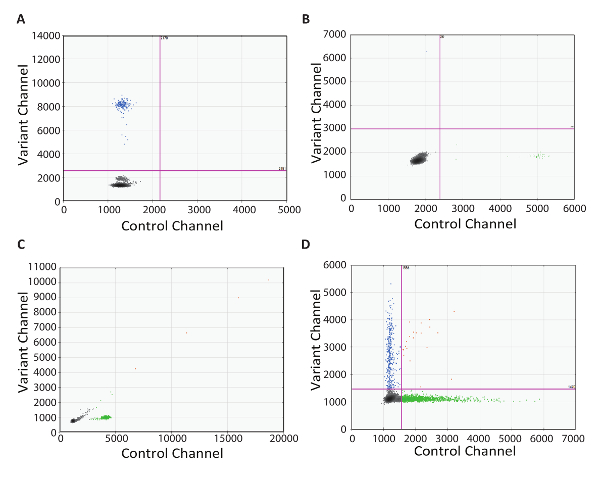
Abbildung 4 : RT dPCR Fehlerbehebung. 2D plots vertreten suboptimalen dPCR Ergebnisse erzielt, wenn (A) innerhalb der keine reverse-Transkriptase-Kontrolle, (B) Kontamination innerhalb der keine RNA-Kontrolle, (C Kontamination) schneiden und Zusammenfügen von Tröpfchen und (D ) schlecht optimiert PCR-Zustände oder PCR-Hemmung. Bitte klicken Sie hier für eine größere Version dieser Figur.
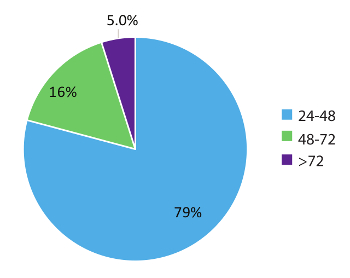
Abbildung 5 : Turnaround-Zeit (TAT). TAT (in Stunden) für Tests anfordern eine RNA-Variante zusammengestellt wurde (n = 984). Daten ausschließt, Wochenenden, Feiertagen und Proben für statt > 24 h aufgrund von unvollständigen klinischen Informationen über Labor Testformen anfordern. Bitte klicken Sie hier für eine größere Version dieser Figur.
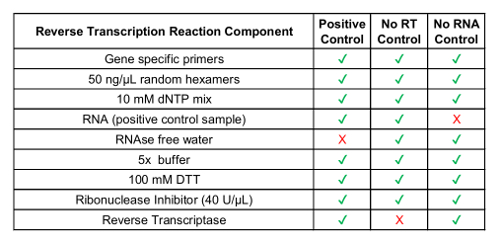
Tabelle 1: Vorbereitung der reversen Transkription Reagenzien für Prozess-Steuerungen.
| Komponente | Volumen |
| 2 x dPCR Supermix für Sonden (keine 2' Deoxyuridine 5'-Triphosphat) | 10 ΜL |
| 20 X Variante Ziel Primer/Sonden eingestellt (450 Nmol/L Primer, 250 Nmol/L FAM Sonde) | 1 ΜL |
| 20 x Kontrolle Zielsatz Primer/Sonden (450 Nmol/L Primer, 250 Nmol/L HEX Sonde) | 1 ΜL |
| Nuklease-freies Wasser | 1 ΜL |
| cDNA | 7 ΜL |
Tabelle 2: Vorbereitung der master-Mix für dPCR.
| Radfahren Schritt | Temperatur | Zeit | # Zyklen | Ramp-Rate |
| Enzym-Aktivierung | 95 ° C | 10 min | 1 | ~ 2 oC/s |
| Denaturierung | 94 ° C | 30 s | 40 | |
| Glühen/Verlängerung | 55 ° C | 1 min | ||
| Enzym-Deaktivierung | 98 ° C | 10 min | 1 | |
| Halten (optional) | 4 ° C | unendliche | 1 | ~ 1 oC/s |
Tabelle 3: Radsport Thermik.
Diskussion
RET und ROS1 Rearrangements bilden zusammen ~ 3 % der Mutationen innerhalb der NSCLC Bevölkerung18Fahrer. Obwohl selten, ist die Erkennung dieser genetischen Veränderungen entscheidend. NSCLC-Patienten mit diesen Änderungen können profitieren von zielgerichteten Therapeutika, die speziell die aberrante Kinase-Aktivität hemmen, die ergibt sich aus der Onko-Protein-13. Einige solcher Therapien sind bereits zugelassen von der FDA für den Einsatz in ROS1 positiven NSCLC, während andere nachgewiesen wirksam gegen RET in klinischen Studien19sein.
PCR-Digitaltechnik bietet Sensibilität, die ideal für flüssigbiopsie Anwendungen20ist. Es gab erhebliche Verbreitung dieser Technologie für den Einsatz mit zirkulierenden zellfreie DNA für die Messung von Tumor-Mutationen bei Patienten mit NSCLC4,6,21,22,23 . Neben Blut entwickelten wir ein Protokoll für robuste Messung der am weitesten verbreitete Fusion Varianten bei Patienten mit NSCLC von zirkulierenden Tumor RNA (Abb. 1A)10.
Unsere etablierte Protokoll ermöglicht analytischen Grenzen der Erkennung auf 0,2 % (Abbildung 2). Während der RT-dPCR sehr spezifisch und sensitiv ist, beschränken sich die Assays auf das Panel von bekannten Fusion-Varianten, die ausgewählt und gemultiplext zur Erkennung in der PCR-Test. Fusionen in Multiplex-Assays aufgenommen werden müssen so sorgfältig ausgewählt werden, um eine ordnungsgemäße Abdeckung innerhalb der Population von Patienten mit NSCLC gewährleisten. Wir haben erfolgreich Assays für RET und ROS1, die gleichzeitig acht Fusion Varianten Resultante von Umstellungen der RET oder ROS1 Loci zu erkennen und decken 99 % und 88 % der RET und ROS1 positiven Bevölkerung, bzw. entwickelt (Abbildung 1B-C )17.
Der Abschlusstest Workflow wie in dieser Studie beschrieben umfasst Batch Kontrollen zur Gewährleistung der Konsistenz der Ergebnisse. Diese Steuerelemente umfassen einen positiven analytische Standard sowie zwei negative Kontrollen, die gemeinsam dafür, dass es keine Kontamination oder PCR-Hemmung sorgen, die innerhalb der Charge (Abbildung 3). Um die Robustheit des Tests zu gewährleisten, wurde eine Studie durchgeführt mit der Batch-Steuerung über einen Zeitraum von 21 Tagen (Abbildung 3D, H). Diese Daten zeigen die Konsistenz des RNA-Prozesses, da innerhalb dieses Protokolls eingerichtet.
Gute Laborpraxis und angemessenen Umgang mit RNA sind Schlüsselkomponenten robuste und exakte Ergebnisse zu gewährleisten. Laborfläche und spezielle Ausrüstung mit RNA, Reinigung der Geräte nach jedem Gebrauch, mit RNase-freie Reagenzien und Verbrauchsmaterialien und eine RNase-Inaktivierung-Spray auf der Arbeitsoberfläche, die alle dazu beitragen, kontaminierenden RNases anzuwenden. Gewissenhafter Umgang mit RNA-Proben von Technikern, einschließlich einen dedizierten Laborkittel, häufige Handschuh, schnell durch die RNA-Extraktion-Verfahren arbeiten und halten Proben auf Eis sind von größter Bedeutung für die Erhaltung der Integrität der Probe. Sobald RNA transkribiert, cDNA umgekehrte gewesen ist, wird die Probe in stabiler Form, die weniger anfällig für Abbau. Neben der Praktiken, die Integrität der RNA zu unterstützen, werden PCR-Komponenten und Proben aufrechterhalten in getrennten Bereichen, um Kreuzkontaminationen zu vermeiden, die zu falsch positiven Ergebnissen führen könnte. Lager PCR Reagenzien und Vorbereitung der PCR master Mix sollte getrennt von PCR Vorlagen gehalten werden und großer Sorgfalt ergriffen werden aus allen vorverstärkt Materialien einschließlich Reagenzien, RNA und DNA-Proben, verstärkte Vorlage (Post-PCR) zu trennen. Zu guter Letzt ist korrekte Generierung und Abwicklung von emulgierten PCR Mischungen vor Verstärkung von zentraler Bedeutung zur Erhaltung Tröpfchen Integrität und optimale dPCR Bedingungen. Während der Ausführung dieses Protokolls, konsistente und genaue Ergebnisse zu erzielen sind Vorsichtsmaßnahmen wie diese kritisch. Alle Daten sollten sicher sein durch geschultes Personal vor der Veröffentlichung der Ergebnisse untersucht werden, traf alle QC-Metriken. Bei suboptimalen Ergebnissen (Abbildung 4) die Charge überprüft werden, durch technisches Personal und der Laborleiter und erneute Verarbeitung erfordern.
RT-dPCR Ergebnisse können bereits 24 Stunden nach Probeneingang und 95 % der Ergebnisse in das Testset in dieser Studie verwendeten hergestellt werden (n = 984) wurde berichtet, der Bestellung Arzt in weniger als 72 Stunden ab dem Zeitpunkt des Eingangs (Abbildung 5). Dieser Turn-around-Zeit bietet Ärzten mit viel benötigt molekularen Informationen in einem Zeitrahmen, die Einleitung einer entsprechenden Therapie ermöglicht. Diese Ergebnisse stehen in der Regel früher als jene, die mit einem herkömmlichen Gewebeentnahme. Weitere Biomarker für NSCLC und anderen Krebsarten mit ähnlichen zirkulierenden RNA-basierten Ansätzen entwickelt werden könnte und würde profitieren von den gleichen schnellen Zeit-zu-Ergebnissen. Messung der programmierten Tod Liganden 1 (PD-L1) mRNA Abschrift mit RT-dPCR konnten beispielsweise Ärzte über Immuntherapie Optionen informieren. Außerdem gibt es ein wachsendes Interesse an den Nutzen des flüssigen Biopsie und dPCR bei der Überwachung der therapeutischen Wirksamkeit. Früheren Angaben von Wiederauftreten des Tumors mit genomischen Tests für bestimmte Varianten könnte Ärzten erlauben, Behandlungsschemata anpassen, bevor Patienten vom Standard der Pflegemaßnahmen wie imaging24symptomatisch sind. Protokolle wie berichtet in dieser Studie sind ideal für Überwachung aufgrund ihrer nicht-Invasivität, Empfindlichkeit, schnelle Turn-around-Zeit und Preis-/Leistungsverhältnis. Die hier beschriebene Test liefert Ergebnisse innerhalb von 72 Stunden nach Probeneingang, mit minimalen falsch-Positive Erkennungsraten, die rasche Behandlungsentscheidungen erleichtert und umgeht einige Einschränkungen mit Gewebe-basierten Test4erlebt.
Unser Protokoll und Daten zeigen eine robuste Testsystem zur Identifizierung von niedrigen Fülle RNA Varianten sowie das Potenzial für blutbasierte Mutation Tests in der klinischen Praxis. Für jene Patienten, die nicht über eine nachvollziehbare Fahrer nähert Mutation identifiziert durch schnelle gezielte flüssigbiopsie wie dieser, die Zugabe von umfassender Genom und Proteom aus Gewebe und Blut testen noch breiteren klinische Informationen liefern kann Behandlungsplanung zu unterstützen.
Offenlegungen
H.M, l.j., k.a. und g.a.p. sind Mitarbeiter und Aktien in Biodesix, Inc. H.M, L.J und g.a.p. sind Co-Erfinder auf eine Patentanmeldung durch Biodesix, für ein diagnostisches Testsystem zum Nachweis von zirkulierenden genetische Varianten in nicht-kleinzelligem Lungenkrebs.
Danksagungen
Wir bedanken uns bei unseren Mitarbeitern, Stephen Jones, Nia Charrington, Dr. Dianna Maar und Dr. Samantha Cooper aus der Digital-Biologiezentrum (Bio-Rad Inc. CA) für ihre Test-Design zu unterstützen; Nezar Rghei und Dr. Moemen Abdalla (Norgen Biotek, Kanada) für kritische Beratung bei der Optimierung der RNA-Extraktion-Protokoll; und Shannon Campbell, Scott Thurston, Jeff Fensterer, Shannon Martello und Joellyn Enos Unterstützung bei testen Sie Anforderungen und kommerziellen Überwachung.
Materialien
| Name | Company | Catalog Number | Comments |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1604071 |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1809353 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1604071 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1606077 |
| Phosphate Buffered Saline 1X, Sterile | Amresco | K812-500mL | 1446C189 |
| Phosphate Buffered Saline 1X, Sterile – 500 mL | Invitrogen | 10010023 | 1916C092 |
| RNase Zap (Life Tech) (250 mL) | Ambion | AM9780 | 353952 |
| Beta-Mercaptoethanol (BME) (250 mL) | CalbioChem | 6050 | W105B |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56054611 |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56238638 |
| Isopropyl Alcohol | VWR | 0918-4L | 2116C416 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 403648 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 288461 |
| DNase I | Thermo | K0441 | 371299 |
| QIAzol Lysis Reagent | Qiagen | 79306 | 54809699 |
| 20x TE buffer pH 8.0 | Alfa Aesar | J62388 | R13C548 |
| UltraPure Agarose | Invitrogen | 16500-100 | 552730 |
| 10x TBE buffer | Invitrogen | AM9863 | 353065 |
| Cell-Free RNA BCT | Streck | 218976 | 60110327 |
| Cell-Free RNA BCT | Streck | 218976 | 61900327 |
| Cell-Free RNA BCT | Streck | 218976 | 61480327 |
| Cell-Free RNA BCT | Streck | 218976 | 62320327 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 585849 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 588308 |
| Lysis Buffer | Norgen | 21205 | A5F61E |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 585848 |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 588309 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC186976 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188077 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188413 |
| Collection Tubes 500 pack | Zymo | C1001-500 | N/A |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 391657 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 392504 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 448001 |
| SuperScript IV Reverse Transcriptase | Life Technologies | 18090200 | 451702 |
| Qubit HS RNA Assay Kit (500) | Life Technologies | Q32854 | 1745264 |
| Qubit assay tubes (500) | Life Technologies | Q32856 | 13416Q311 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64031651 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64063941 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065740 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065741 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64079083 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64025320 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64052358 |
| gBlock KIF5B-RET K15:R12 | IDT | 151004172 | 4-Oct-16 |
| gBlock KIF5B-RET K16:R12 | IDT | 151004173 | 4-Oct-16 |
| gBlock KIF5B-RET K22:R12 | IDT | 151004174 | 4-Oct-16 |
| gBlock KIF5B-RET K23:R12 | IDT | 151004175 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R11 | IDT | 151004176 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R8 | IDT | 151004177 | 4-Oct-16 |
| gBlock CCDC6-RET C1:R12 | IDT | 151004178 | 4-Oct-16 |
| gBlock TRIM33-RET T14:R12 | IDT | 151004179 | 4-Oct-16 |
| RET exon 8 RT Gene Specific Primer | IDT | 150554385 | 28-Sep-16 |
| 5’-CTCCACTCACACCTG-3’ | IDT | 150554385 | 28-Sep-16 |
| RET exon 11 RT Gene Specific Primer | IDT | 150554384 | 28-Sep-16 |
| 5’-GCAAACTTGTGGTAGCAG-3’ | IDT | 150554384 | 28-Sep-16 |
| RET exon 12 RT Gene Specific Primer | IDT | 150554383 | 28-Sep-16 |
| 5’-CTGCCTTTCAGATGGAAG-3’ | IDT | 150554383 | 28-Sep-16 |
| gBlock CD74-ROS1 C6:R34 | IDT | 152324366 | 15-Nov-16 |
| gBlock CD74-ROS1 C6:R32 | IDT | 152324367 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R32 | IDT | 152324368 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R34 | IDT | 152324369 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R32 | IDT | 152324370 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R34 | IDT | 152324371 | 15-Nov-16 |
| gBlock EZR-ROS1 E10:R34 | IDT | 152324372 | 15-Nov-16 |
| gBlock TPM3-ROS1 T8:R35 | IDT | 152324373 | 15-Nov-16 |
| ROS1 exon 34 RT Gene Specific Primer | IDT | 152704983 | 21-Nov-16 |
| 5’-CCTTCCTTGGCACTTT-3’ | IDT | 152704983 | 21-Nov-16 |
| ROS1 exon 35 RT Gene Specific Primer | IDT | 152704985 | 21-Nov-16 |
| 5’-CTCTTGGGTTGGAAGAGTATG-3’ | IDT | 152704985 | 21-Nov-16 |
| ALK Gene Specific Primer | IDT | 140035422 | 26-Aug-16 |
| 5’-CAGTAGTTGGGGTTGTAGTCG-3’ | IDT | 140035422 | 26-Aug-16 |
| EML4-ALK Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| SLC34A2-ROS1 Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| CCDC6-RET Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| Human Brain Total RNA | Ambion | AM7962 | 1703548 |
| PrimePCR ddPCR Expert Design Assay: K15:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K16:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K22:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K23:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R11 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R8 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C1:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: T14:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: E10:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: T8:R35 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| (version 2) | Bio-Rad | 12003909 | 213939881 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 13-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 20170112v3.2 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851151 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 207383915 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 195995635 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851152 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 213949301 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 20160914 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 211383227 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 1065C220 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052953 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052358 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 1065C320 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64052952 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64064127 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000065883 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084276 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000079928 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084395 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084634 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20160627 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161107 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161206 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161216 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20170125 |
| Pipet Tips for Automated Droplet Generator | Bio-Rad | 1864120 | PR125340 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206894 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206893 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 1409850 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 100402 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 145851 |
| Microseal 'B' seals | Bio-Rad | MSB1001 | BR00428490 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64039089 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049253 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049255 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64081870 |
| DNA Lo Bind Tube 0.5 mL | Eppendorf | 22431005 | E1629620 |
| DNA Lo Bind Tube 1.5 mL | Eppendorf | 22431021 | F16698K |
| DNA Lo Bind Tube 2 mL | Eppendorf | 22431048 | E160610I |
| 50 mL Conicals, Polypropylene (25) | Thermo | 339652 | G5ZF5W8118 |
| TempAssure PCR 8-Strips, Optical Caps, Natural, polypropylene (120) | USA Scientific | 1402-4700 | 16202 |
| For Rainin LTS Pipettors 0.5-20 µL tips | Pipette.com | LF-20 | 40155-642C4-642C |
| For Rainin LTS Pipettors 5-200 µL tips | Pipette.com | LF-250 | 40154-642C4-642B |
| Tips LTS 200 ul Filter 960/10 RT-L200F (10 boxes) | Rainin | 17002927 | 1635 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F1175551-1108 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F118054L-1720 |
| Pipet Tips, 100 ul TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1180-1740 | 0014961Q-2501 |
| Pipet Tips, 200 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1180-8710 | E116684P-1540 |
| Pipet Tips, 1000 ul XL TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1182-1730 | F118815P |
| 5 mL Standard Racked Gilson-fit Reference Tips | Scientific Specialties | 4411-00 | 14312 |
| Combitips advanced, 0.1 mL Biopur | Eppendorf | 003 008 9618 | F165414H |
| Combitips advanced, 0.2 mL Biopur | Eppendorf | 0030 089.626 | F166689J |
| Combitips advanced, 5 mL Biopur | Eppendorf | 0030.089 669 | F166054J |
| Combitips advanced, 50 mL Biopur | Eppendorf | 003.008.9693 | F166055I |
| Reagent Reservoir | VWR | 89094-680 | 141500 |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165029I |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165028G |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Green | Eppendorf | 951020346 | F166183K |
| Equipment Type | Equipment ID | ||
| Analytical Balance | EQP0125 | ||
| Cryogenic Freezer 1, -80oC | EQP0095 | ||
| Refrigerator 6.1 cu ft GP06W1AREF | EQP0139 | ||
| -20oC Freezer | EQP0140 | ||
| Beckman Coulter Microfuge 22R | EQP0025 | ||
| Beckman Coulter Microfuge 22R | EQP0124 | ||
| Thermo Scientific Hereaus Megafuge 8 | EQP0104 | ||
| Mini Centrifuge | EQP0131 | ||
| Mini Centrifuge | EQP0136 | ||
| Mini Centrifuge | EQP0134 | ||
| Mini Centrifuge | EQP0235 | ||
| Mini Centrifuge | EQP0216 | ||
| Thermo Scientific HeraTherm Incubator | EQP0105 | ||
| Pipette 0.1 - 2.5 μL | EQP0182 | ||
| Pipette 0.1 - 2.5 μL | EQP0072 | ||
| Pipette 0.1 - 2.5 μL | EQP0070 | ||
| Pipette 0.5-10 μL | EQP0218 | ||
| Pipette 0.5-10 μL | EQP0075 | ||
| Pipette 0.5-10 μL | EQP0169 | ||
| Pipette 0.5-10 μL | EQP0074 | ||
| Pipette 0.5-10 μL | EQP0147 | ||
| Pipette 2 - 20 μL | EQP0128 | ||
| Pipette 2 - 20 μL | EQP0160 | ||
| Pipette 2 - 20 μL | EQP0018 | ||
| Pipette 2 - 20 μL | EQP0146 | ||
| Pipette 10 - 100 μL | EQP0079 | ||
| Pipette 10 - 100 μL | EQP0181 | ||
| Pipette 10 - 100 μL | EQP0085 | ||
| Pipette 10 - 100 μL | EQP0077 | ||
| Pipette 20 - 200 μL | EQP0088 | ||
| Pipette 20 - 200 μL | EQP0087 | ||
| Pipette 20 - 200 μL | EQP0231 | ||
| Pipette 100 - 1000 μL | EQP0050 | ||
| Pipette 100 - 1000 μL | EQP0158 | ||
| Pipette 100 - 1000 μL | EQP0217 | ||
| Pipette 100 - 1000 μL | EQP0082 | ||
| Pipette 100 - 1000 μL | EQP0183 | ||
| Pipette 100 - 1000 μL | EQP0083 | ||
| Pipette 5 mL | EQP0153 | ||
| Timer | S/N 140623950 | ||
| Hamilton SafeAire VAV Fume Hood | EQP0206 | ||
| Biosafety Cabinet | EQP0205 | ||
| Biosafety Cabinet | EQP0204 | ||
| Qubit 3.0 | EQP0102 | ||
| Benchmark Digital Heat Block | EQP0108 | ||
| Benchmark Digital Heat Block | EQP0231 | ||
| Polaroid Z2300 Instant Print Digital Gel Camera with WiFi and 16GB SDHC memory card | EQP0111 | ||
| Electrophoresis Power Unit | EQP0113 | ||
| Electrophoresis Small Gel Box | EQP0116 | ||
| Maestro Transilluminator | EQP0118 | ||
| Microwave | EQP0215 | ||
| Multichannel 8-well Pipette 2 - 20 μL | EQP0207 | ||
| Multichannel 8-well Pipette 10 - 100 μL | EQP0090 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0094 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0161 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0162 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0163 | ||
| Vortex Genie 2 | EQP0052 | ||
| Vortex Genie 2 | EQP0007 | ||
| Vortex Genie 2 | EQP0132 | ||
| Vortex Genie 2 | EQP0137 | ||
| Vortex Genie 2 | EQP0135 | ||
| Air Clean PCR Workstation | EQP0203 | ||
| Air Clean PCR Workstation | EQP0096 | ||
| Air Clean PCR Workstation | EQP0148 | ||
| Air Clean PCR Workstation | EQP0097 | ||
| QX200 Droplet Generator | EQP0202 | ||
| QX200 Droplet Generator | EQP0121 | ||
| Automated Droplet Generator | EQP0179 | ||
| PX1 PCR Plate Sealer | EQP0123 | ||
| PX1 PCR Plate Sealer | EQP0186 | ||
| C1000 Touch Cycler w/96W FS RM | EQP0120 | ||
| S1000 Cycler w/96W FS RM | EQP0174 | ||
| S1000 Cycler w/96W FS RM | EQP0173 | ||
| T100 Thermal Cycler | EQP0180 | ||
| T100 Thermal Cycler | EQP0175 | ||
| QX200 Droplet Reader | EQP0194 | ||
| QX200 Droplet Reader | EQP0122 |
Referenzen
- Ignatiadis, M., Lee, M., Jeffrey, S. S. Circulating Tumor Cells and Circulating Tumor DNA: Challenges and Opportunities on the Path to Clinical Utility. Clin Cancer Res. 21 (21), 4786-4800 (2015).
- Alix-Panabieres, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discov. , (2016).
- Paxton, A. Is Molecular AP testing in sync with guidelines. CAP Today. , (2014).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA Oncol. , (2016).
- Sozzi, G., et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol. 21 (21), 3902-3908 (2003).
- Oxnard, G. R., et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res. 20 (6), 1698-1705 (2014).
- Best, M. G., et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics. Cancer Cell. 28 (5), 666-676 (2015).
- Rodriguez, M., et al. Different exosome cargo from plasma/bronchoalveolar lavage in non-small-cell lung cancer. Genes Chromosomes Cancer. 53 (9), 713-724 (2014).
- Kalluri, R. The biology and function of exosomes in cancer. J Clin Invest. 126 (4), 1208-1215 (2016).
- Mellert, H., et al. Development and Clinical Utility of a Blood-Based Test Service for the Rapid Identification of Actionable Mutations in Non-Small Cell Lung Carcinoma. J Mol Diagn. 19 (3), 404-416 (2017).
- Qin, J., Williams, T. L., Fernando, M. R. A novel blood collection device stabilizes cell-free RNA in blood during sample shipping and storage. BMC Res Notes. 6, 380 (2013).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques. 15 (3), 532-537 (1993).
- Kohno, T., et al. Beyond ALK-RET, ROS1 and other oncogene fusions in lung cancer. Transl Lung Cancer Res. 4 (2), 156-164 (2015).
- Takeuchi, K., et al. ROS1 and ALK fusions in lung cancer. Nat Med. 18 (3), 378-381 (2012).
- Rimkunas, V. M., et al. Analysis of receptor tyrosine kinase ROS1-positive tumors in non-small cell lung cancer: identification of a FIG-ROS1 fusion. Clin Cancer Res. 18 (16), 4449-4457 (2012).
- Tsuta, K., et al. RET-rearranged non-small-cell lung carcinoma: a clinicopathological and molecular analysis. Br J Cancer. 110 (6), 1571-1578 (2014).
- Forbes, S. A., et al. COSMIC: somatic cancer genetics at high-resolution. Nucleic Acids Res. 45 (1), 777-783 (2017).
- Salgia, R. Diagnostic challenges in non-small-cell lung cancer: an integrated medicine approach. Future Oncol. 11 (3), 489-500 (2015).
- Cagle, P. T., Raparia, K., Portier, B. P. Emerging Biomarkers in Personalized Therapy of Lung Cancer. Adv Exp Med Biol. 890, 25-36 (2016).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci U S A. 96 (16), 9236-9241 (1999).
- Oxnard, G. R., et al. Association Between Plasma Genotyping and Outcomes of Treatment With Osimertinib (AZD9291) in Advanced Non-Small-Cell Lung Cancer. J Clin Oncol. 34 (28), 3375-3382 (2016).
- Reckamp, K. L., et al. A Highly Sensitive and Quantitative Test Platform for Detection of NSCLC EGFR Mutations in Urine and Plasma. J Thorac Oncol. 11 (10), 1690-1700 (2016).
- Yanagita, M., et al. A prospective evaluation of circulating tumor cells and cell-free DNA in EGFR mutant non-small cell lung cancer patients treated with erlotinib on a phase II trial. Clin Cancer Res. , (2016).
- Abbosh, C., et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545 (7655), 446-451 (2017).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-534 (1993).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten