Method Article
Optimierung eines Multiplex-RNA-basierten Ausdruck-Assays mit Brust-Krebs-Archivmaterial
In diesem Artikel
Zusammenfassung
Nukleinsäure-Abbau im Archiv Gewebe, Tumor Heterogenität und einen Mangel an frischen gefrorene Gewebeproben kann negative Auswirkungen auf Krebs-Diagnose-Services in Pathologie Laboren weltweit. Dieses Manuskript beschreibt die Optimierung eines Panels von Biomarkern, die mit einen multiplex magnetischer Wulst-Assay, um Brustkrebs zu klassifizieren.
Zusammenfassung
Nukleinsäure-Abbau im Archiv Gewebe, Tumor Heterogenität und einen Mangel an frischen gefrorene Gewebeproben kann negative Auswirkungen auf Krebs-Diagnose-Services in Pathologie Laboren weltweit. Gen-Amplifikation und Ausdruck diagnostischen Tests mit archivalischen Material oder Material, das Transport, Laboratorien, Wartung erfordert braucht einen robuste und genauen Test angepasst an aktuellen klinischen Arbeitsabläufe. Unser Research-Team optimiert den Einsatz von Invitrogen™ QuantiGene™-Plex-Assay (Thermo Fisher Scientific), RNA Archivmaterial mit verzweigten-DNA (bDNA) Technologie auf Luminex xMAP® magnetische Beads zu quantifizieren. Die Gen-Ausdruck-Assay beschrieben in diesem Manuskript ist ein Roman, schnelle und Multiplex-Methode, die genau Brustkrebs in der molekularen Subtypen, weglassen der Subjektivität der Auslegung innewohnt, bildgebende Verfahren einordnen kann. Darüber hinaus können aufgrund der low-Input des benötigten Materials, heterogenen Tumoren Laser-Microdissected mit Hämatoxylin und Eosin (H & E) gebeizt Abschnitte. Diese Methode hat eine breite Palette von möglichen Anwendungen einschließlich Tumor-Klassifizierung diagnostische Potenzial und Messung von Biomarkern in flüssigen Biopsien, die besseres Patientenmanagement und Überwachung von Krankheiten ermöglichen würde. Darüber hinaus eignet sich die quantitative Messung von Biomarkern in Archivalien in der Onkologie-Forschung Zugang zu Bibliotheken von klinisch kommentierte Material, in der Retrospektive Studien potenzielle Biomarker und ihre klinischen Ergebnisse überprüfen können Korrelation.
Einleitung
Optimierung von RNA-basierten Assays mit Formalin fixierten Paraffin-eingebetteten (FFPE) Archivmaterial ist schwierig wegen der Variabilität in der chirurgischen Gewebe Verarbeitung und Abbau der RNA durch Formalin verwendet für Gewebe Integrität Erhaltung1verursacht, 2. Um die Begrenzung der Durchführung genau genexpressionsstudien aus Archivmaterial zu überwinden, verwendet unsere Fraktion bDNA Multiplex-magnetic Bead Assays. Statt enzymatische Verstärkung einer Ziel-Vorlage nutzt die bDNA Technologie Hybridisierung von spezifischen Sonden und Verstärkung von einem Reporter Signal3. Die kurze Erkennungssequenzen der Erfassung und Erkennung Sonden sollen für kurze Fragmente der Ziel-RNA4hybridisieren. Darüber hinaus überwindet die Verwendung von Gewebe Homogenates als direkte Ausgangsmaterial in diesem Assay den unvermeidlichen Verlust der RNA, die Ergebnisse aus Tests erfordern vorherige RNA-Extraktion und Reinigung. Signalverstärkung, die Verwendung von kurzen Erkennungssequenzen und den Ausschluss von einem aufbereitungsschritt beitragen zu reduzieren in technische Variante des Assays. Die Technologie bietet die Möglichkeit, multiplex-Assays (bis zu 80 RNA Ziele) und Messen Sie den Ausdruck eines Panels von Zielen von geringen Materialeinsatz. Dieses Protokoll beschreibt die Vorbereitung und Färbung von Gewebeproben für Laser-Mikrodissektion. Flecken auf der Membran-Folien erleichtern die Bildgebung der Tumor und der histologischen Architektur, sorgfältige Auswahl bieten und Profilierung der: (1) der Tumor und normalen Kanäle im Brustgewebe und (2) die maligne Zelle Klont in heterogenen Tumoren.
Molekulare Klassifikation von Brustkrebs ist ein Prozess, der molekulare Marker um Patienten Tumoren in drei Molekulare Klassen, d. h., luminalen, menschlichen epidermalen Wachstumsfaktor-Rezeptor 2 (HER2) kategorisieren verhört-angereichert und basalen Subtyp. Die HER2-angereicherten Verlaufsform ist gut definiert, mit hohen Ausdruck des HER2-Rezeptors durch die ERBB2-gen-Verstärkung, kombiniert mit niedrig oder abwesend Östrogen-Rezeptor (ER) und Progesteron-Rezeptor (PgR). Der luminalen Untertyp ist generell positiv, denn ER und der basalen Untertyp im allgemeinen negativ für die drei Rezeptoren (HER2, ER, PgR) und deutlich Überschneidungen mit der Dreifach negative Brust Krebs (TNBC) diagnostische Subtyp5,6. Andere Markierungen dienen zur epithelialen und mesenchymalen Merkmale zu bestimmen. Fibronektin (FN1) ist ein Hauptbestandteil der Brust Gewebe mesenchymalen Fach. Erhöhte FN1 Ausdruck wird begleitet von hohen Ki67 Färbung, und zeigt eine Signatur für ein invasiver Tumor7,8 und Metastasierung9zugeordnet ist. Interessanterweise fand FN1 Microvesicles aus den Tumorzellen, die Aktivierung der mitogenen Signale im Empfänger Fibroblasten10induzierte vorhanden sein. Daher kursieren Microvesicles wie exosomen potenzieller Marker für die Früherkennung oder Metastasen Rückfall11.
Tumor-Bereichsauswahl für Brust Krebs transkriptionelle Subtypisierung wurde immer wieder von Macrodissection12,13durchgeführt. Um Gewebe Heterogenität zu überwinden und Empfindlichkeit zu erhöhen, haben wir zuverlässig klassische Gewebe Färbungen mit Multiplex-Molekulare profiling-Methoden kombiniert. Als Beweis des Prinzips wurden zwei unterschiedliche Brust-Krebs-Klone durch ihre epitheliale Mesenchymale Signatur und metastatische Potential definiert. Der Workflow des beschriebenen Protokolls kann leicht nach der aktuellen klinischen Einrichtung übersetzt und verwendet, um selektiv zu isolieren und zu charakterisieren Gewebe Subtypen durch gezielte mRNA profiling.
Protokoll
Ethischen einsetzbar Brust Gewebematerial in dieser Studie wurde Zustimmung der von Universität Forschung Ethics Committee (UREC) von der University of Malta (Ref: 22/2012).
(1) Gewebe-Vorbereitung
- Mit entsprechenden H & E Verfahren14, bereiten Sie gefärbten Referenz Gewebeschnitte und Digital Scannen Sie die Dias als Referenz während Mikrodissektion.
- Schneiden Sie ein Mikrotom 20 µm Gewebe Abschnitte auf eine RNAse-freie Wasserbad bei 40 ° C. Die Abschnitte auf Membran-Folien für Laser-Mikrodissektion mit sauberen, RNase dekontaminiert Ausrüstung und Materialien zu sammeln.
- Trocknen Sie die Membran-Folien bei 37 ° C in einem trocknen Inkubator über Nacht. Fleck mit entsprechenden H & E Verfahren15 mit Molekularbiologie Lösungen und Erhöhung der Färbung Zeit von Hämatoxylin, 6 min. Falls erforderlich, Färben Sie die Membran-Folien mit den entsprechenden immunhistochemische Protokolle16.
2. Laser-Mikrodissektion
- Die Folie Grenzen setzen und die Tischbewegung zu kalibrieren.
- Verschieben Sie die Laser-Ansicht auf einen leeren Bereich auf einer Membran Folie. Kalibrieren des Laserfokus durch das Abfeuern eines Lasers Fokus Intervalle (5 %) zu erhöhen, bis der Laser beginnt zu durchdringen die Membran beschossen. Auslosung und Microdissect beliebiger Form und Finetune Laserfokus, die Laser-Effizienz während der Laser-Präparation zu maximieren.
- Erhöht die Bewegungsgeschwindigkeit der Bühne bis zu einem Punkt, wo die Laser Dissektion Effizienz nicht beeinträchtigt wird. Kalibrieren des Lasers bei der ausgewählten Objektivlinse und neu zu kalibrieren, wenn Ziele (Laser-Geschwindigkeit von 1 – 15 %), Schwerpunkt bei 70 – 75 % und macht bei 90 – 100 % ändern, wenn Sie 4 X Objektiv verwenden.
- Scannen Sie die gefärbten Membran-Folien mit 4 X Objektiv.
- Wählen Sie und umschließen Sie die Zielbereiche für Mikrodissektion auf der Membran Folie. Laser zu sezieren eine Mindestfläche von 42 mm2. Rekord seziert der Bereich um zu bestimmen, das Volumen der Probe homogenisierende Puffer verwendet werden.
- Mithilfe der GAP Hebemechanismus sezierten Abschnitt auf die Diffusor-Kappen der beschrifteten Röhrchen angebracht, die entsprechende Anhängsel abrufen. Wenn sezierte Abschnitt auf der Kappe nicht abgerufen wird, wiederholen Sie die Laser-Dissektion des Gebiets.
(3) Gewebe Lyse
Hinweis: Mehrere Lösungen und Materialien werden zusammen mit Kits erwähnt in der Tabelle der Materialiengeliefert.
- Lyse der Probe mit homogenisierenden Lösung und Proteinase K in einem 60:1 Verhältnis bereiten Sie das erforderliche Volumen für homogenisierende Mischung vor.
- Pipette 2,4 µL Homogenisieren Lösung in jedem Röhrchen für jeweils 1 mm2 Gewebebereich, Wirbel für 10 s auf die maximale Geschwindigkeit und Zentrifuge bei Raumtemperatur für 5 s bei 2.500 x g.
- In einem Heizblock bei 65 ° C für 12 – 18 h unter Schütteln bei 600 u/min inkubieren.
- Zentrifugieren Sie Lysates 21.000 x g für 5 min bei Raumtemperatur.
- Mit einer Pipette, vorsichtig Aspirieren des klare Überstands und in einer beschrifteten frische Rohr zu verzichten. Vermeiden Sie die Gewebe/Membran Fragmente absaugen, da dies die Qualität der Ergebnisse verringern kann.
- Den klare überstand in einem-80 ° C Gefrierschrank aufbewahren.
(4) Hybridisierung basierende Assays
- Führen Sie die folgenden Vorbereitungen.
- Vorwärmen der Lyse-Mischung durch die Platzierung der reagenzienflasche im Brutschrank bei 37 ° C für 30 min und durch Umkehrung vorsichtig mischen.
- Wenn das Gewebe Homogenates eingefroren sind, bei Raumtemperatur tauen Sie auf und inkubieren Sie die Proben mit der maximalen Geschwindigkeit nach der Inkubation bei 37 ° C für 30 min. Vortex.
- Den schütteln Inkubator bei 54 ° C eingestellt und legen Sie die Sonde des Temperatur-Validierung-Kit in einen leeren Brunnen einer 96-Well-Platte. Überprüfen Sie nach 2 h die Temperatur auf dem digitalen Thermometer und die Delta-Temperatur der Erschütterung Inkubator für die Temperaturdifferenz zu korrigieren.
- Tauwetter und Wirbel der Sonde eingestellt und blockiert Reagenz für 10 s, Zentrifuge bei Raumtemperatur für 5 s bei 2.500 x g, und halten Sie auf dem Eis.
- Halten Sie das Proteinase K-Fläschchen auf dem Eis während des Gebrauchs.
- Beschallen Sie die Capture-Perlen bei 46 KHz für 3 min, dann Wirbel mit der maximalen Geschwindigkeit für weitere 3 Minuten.
- Bereiten Sie eine 25 µL arbeiten Perle Mischung pro Bohrloch durch die folgenden Reagenzien entsprechend skalieren: 4,25 µL Nuklease-freies Wasser, 16.65 µL Lyse Mischung, 1,00 µL Reagenz, 0,10 µL Proteinase K, 0,50 µL Erfassung Perlen, 2,50 µL der Sonde Satz zu blockieren.
- Wirbel der Arbeiten Perle Mischung für 10 s mit maximaler Geschwindigkeit, dann pipette 25 µL/Well in die magnetische Trennung-Platte.
- Pipette 25 µL Gewebe Homogenat eine magnetische Trennung 96-Well-Platte und 25 µL Lösung 3 Brunnen als Rohlinge zu homogenisieren.
- Dichtplatte mit einem durchsichtigen Kunststoff dichtschott Magnetische Trennung und platzieren Sie die Platte in den Inkubator an 54 ± 1 ° C für 18-22 h unter Schütteln bei 600 Umdrehungen pro Minute.
- Bereiten Sie 1 X Puffer Waschlösung für 24 Vertiefungen durch Mischen von 31,5 mL RNase-freies Wasser mit 0,1 mL waschen waschen Puffer Komponente 2 Komponente 1 und 1,67 mL Puffer vor.
- Entfernen Sie die Platte aus Inkubator. Stellen Sie die Temperatur des Inkubators schütteln 50 ± 1 ° C.
- Montieren Sie die magnetische Trennung-Platte auf dem Handheld Magnetplatte Unterlegscheibe und verriegeln. Entfernen Sie die Druckdichtung und warten Sie 1 Minute für die magnetische Beads zu begleichen.
- Waschschritt: Invertieren Sie die magnetische Trennung Platte auf die Platte über der Spüle und Blot sanft auf einem Papiertuch, verbleibende Lösung zu entfernen. Mit einer Mehrkanal-Pipette 100 µL 1 X waschen Pufferlösung in jede Vertiefung und warten 15 s. Wiederholen Sie diesen Schritt auf die Platte 3 Mal waschen.
- Der Vorverstärker-Reagenz in jede Vertiefung 50 µL Pipette. Die Dichtplatte mit einer Aluminium-Platte-Dichtung und schütteln Sie die Platte bei 800 u/min für 1 min bei Raumtemperatur. 1 h bei 50 ± 1 ° C unter Schütteln bei 600 u/min inkubieren.
- Wiederholen Sie Schritt 4,9 (Waschschritt).
- Die Verstärker-Reagenz in jede Vertiefung 50 µL Pipette. Die Dichtplatte mit einer Aluminium-Platte-Dichtung und schütteln Sie die Platte wie in Schritt 4.10.
- Wiederholen Sie Schritt 4,9 (Waschschritt).
- Wirbel der Label-Sonde für 10 s mit maximaler Geschwindigkeit. Jede Vertiefung 50 µL des Prüfpunkts Label hinzufügen. Verschließen Sie und schütteln Sie die Platte (Schritt 4.10).
- Wiederholen Sie Schritt 4,9 (Waschen Schritt).
- Vortex Streptavidin Phycoerythrin (SAPE) für 10 s mit maximaler Geschwindigkeit. Jede Vertiefung 50 µL SAPE hinzufügen. Verschließen Sie und schütteln Sie die Platte (Schritt 4.10).
- Wiederholen Sie Schritt 4,9 (Waschschritt) außer Gebrauch der SAPE Waschpuffer anstelle der Pufferlösung waschen.
- Jeder Brunnen und Dichtung Platte mit einer Aluminium-Platte-Dichtung 130 µL Waschpuffer SAPE hinzufügen. Schütteln Sie die Platte bei 800 u/min bei Raumtemperatur 3 Minuten lang.
- Inbetriebnahme der magnetischen Wulst-Analyzer. Führen Sie die Kalibrierung und Verifizierung Routine durch Anweisungen des Herstellers.
- Richten Sie ein Protokoll über den magnetischen Wulst-Analyzer mit den folgenden Parametern: Sample-Größe: 100 µL; DD-Gate: 5.000-25.000; Timeout: 90 s; Perle-Event/Wulst-Region: 100; und klicken Sie auf Weiter. Wählen Sie die jeweiligen Wulst-Nummern im Panel und definieren Sie die Wulst Regionen mit den jeweiligen Zielgenen wie vom Hersteller angegeben.
- Fügen Sie eine neue Analyse durch Auswahl "Batch erstellen mithilfe einer vorhandenen Protokolls" der Registerkarte " Chargen hinzu ". In der Platte Layout bzw. bezeichnen Sie der Brunnen als unbekannt oder leer, und bieten Sie eine Bezeichnung für jede Probe im Bedienfeld " Proben ".
- Entfernen Sie die Aluminium-Platte-Dichtung von der Reaktion der 96-Well-Platte. Werfen Sie der Platte Tablett durch Klicken auf die Schaltfläche " auswerfen aus ", laden Sie die Platte in der magnetischen Wulst-Analyzer. Klicken Sie auf einfahren| Batch ausführen , den Analysator einzuleiten. Nach der Lektüre, auswerfen und 96-Well-Platte zu entfernen. Reinigen Sie und Herunterfahren Sie der magnetischen Wulst-Analyzer, wie vom Hersteller empfohlen.
(5) Datenanalyse
- Im Batch| Registerkarte " Ergebnisse ", wählen Sie die entsprechende Analysedatei und klicken Sie auf die Schaltfläche export to.csv . Offene the.csv Datei mit einem Tabellenkalkulations-Software.
- Beobachten Sie der Wulst zählt zu und lassen Sie keine Ergebnisse mit < 40 Perlen/Region.
- Durchschnitt der Werte der leeren Brunnen und erarbeiten die Standardabweichung (SD) und Limit of Detection (LOD) (Durchschnitt leer + (3xSD)) pro Zielgen. Legen Sie die Werte niedriger als das LOD als unentdeckt.
Hinweis: Wenn die Normalisierung Ausdruckswerte unentdeckt sind, betrachten Sie die Beispieldaten als unzureichend. Betrachten Sie Ausdruckswerte über 20.000 als über den oberen Empfindlichkeitsgrenze. Die Lysates dieser Proben sollten verdünnt und erneut analysiert werden. - Subtrahieren Sie die leere Durchschnitt aus den Rohdaten pro gen.
- Das geometrische Mittel der ausgewählten normalisierenden Gene pro Probe zu berechnen. Einzelnen Sample-Daten zu den jeweiligen geometrischen Mittelwert zu normalisieren.
- Analysieren Sie die Daten auf Daten-Wissenschaft-Plattformen oder mit definierten Algorithmen.
- Importieren Sie die Daten in die Datenplattform für die Wissenschaft, indem Sie auf Daten hinzufügen. Ziehen Sie die Datendatei in der Prozess-Arbeitsbereich und dann eine Verbindung zu einem Betreiber [Attributen auswählen] zum einen Principal Bauteilanalyse (PCA) Operator, und dann an den Frühjahrsputz-t-Anschluss.
- Wählen Sie im Attribut auswählen -Operator die Teilmenge der normalisierten gen Daten und eine nominale Variable z. B. Brust-Krebs-Subtyp. Führen Sie den Prozess, indem Sie auf zu spielen. Wählen Sie im Reiter " Diagramme " den "3D Farbe streuen" in der Chart-Vorlage und die nominale Variable im Feld Farbe . Die PCA-Daten-Variabilität auf einem 3-dimensionalen Grundstück zu bewerten.
Ergebnisse
Das beschriebene Verfahren für die gleichzeitige Messung von 40 Transkripte in H & E gefärbt (Abbildung 1) angewendet wurde, Microdissected (Abbildung 2) stark degradiert FFPE-Material. Mit dieser Methode zeigen wir die genaue Charakterisierung der Rezeptor-Status (Abbildung 3A), Klassifizierung von Tumoren in der luminalen und basalen molekularen Subtypen17und differentielle Expression der mesenchymalen Marker, FN1, Tumor im Vergleich und aufeinander abgestimmten kontrollgewebe (Abb. 3 b), in der verschiedene Rezeptor-positiven und negativen Subtypen.

Abbildung 1: Genexpression mit H & E gefärbt Material. Korrelation zwischen einem Expressionsprofil abgeleitet aus ungefärbten Tumor im Vergleich zu einem gefärbten Tumor-Sektion. [Pearson Korrelation p-Wert = 5.34E-26]. Reproduziert mit Erlaubnis17. Bitte klicken Sie hier für eine größere Version dieser Figur.
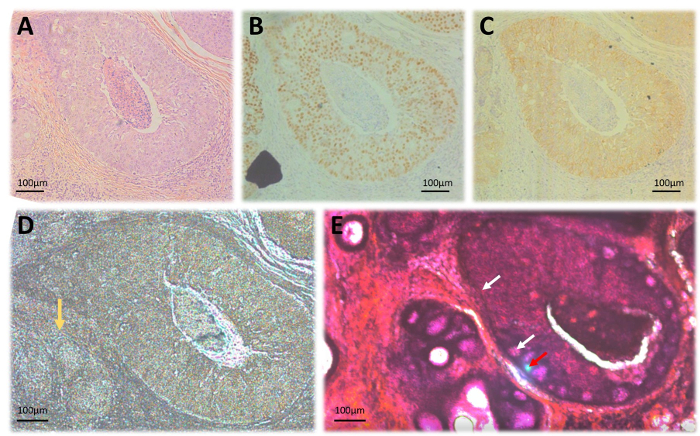
Abbildung 2: Laser Mikrodissektion FFPE Gewebe. (A) H & E gefärbten Objektträger; (B) immunhistochemische Färbung für ER Ausdruck; (C) HER2 immunhistochemische Färbung; (D) ungefärbten 20 µm-Bereich auf Laser Mikrodissektion Membran Folien. Der gelbe Pfeil zeigt eine Fläche von invasiven Tumor, der aufgrund mangelnder Färbung nicht deutlich abgegrenzt ist. (E) A 20 µm Bereich gebeizt mit H & E für bessere Abgrenzung der Gebiete von Interesse. Weiße Pfeile zeigen Laser Dissektion Trail, während der rote Pfeil die Laserfokus während der Präparation zeigt. Alle Abbildungen wurden bei 10 X Vergrößerung gefangen genommen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Ausdruck von (A) Rezeptor-Status und (B) mesenchymalen Marker FN1, in der brusttumor im Vergleich zu normalen abgestimmt. Die klassische Diagnose Brustkrebs Krebs Subtypen gelten gemäß diagnostische Ergebnis mit Immunohistochemistry und Fluoreszenz in Situ Hybridisierung (FISH) für HER2 zweideutige immunhistochemische Färbung. Normalisierte Ausdruck (A) HER2 und ER, (B) FN1 durch Hybridisierung basierende Assays gemessen wird veranschaulicht, in HER2 positiv, ER positiv und TNBC Fälle im Vergleich zu Patienten normalen Brustgewebe abgestimmt. Kruskal-Wallis-Teststatistik (K-W χ2) zeigt, dass die Expression von HER2 signifikant ist (p < 0,05) höher in das Tumorgewebe im Gegensatz zu der übereinstimmenden normalgewebe in der HER2-positive-Kohorte und deutlich in der TNBC-Kohorte. ER Ausdruck nicht signifikant höher im Tumorgewebe im Gegensatz zu der übereinstimmenden Normal in die ER positiven und TNBC Kohorten gefunden. Fn1 ist deutlich höher in das Tumorgewebe in den HER2 und ER positive Subtypen und zeigt einen Trend zu sein im Tumorgewebe auch in der TNBC Kohorte deutlich erhöht. Bitte klicken Sie hier für eine größere Version dieser Figur.
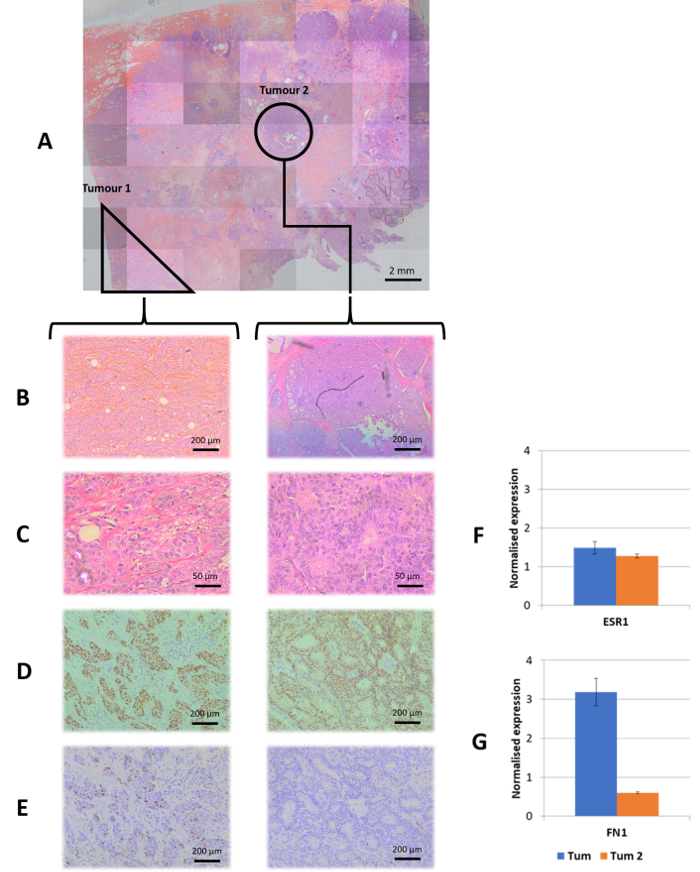
Abbildung 4: Fallstudie: Tumor Heterogenität. Morphologisch unterschiedliche Tumoren waren Microdissected und als unterschiedliche Proben behandelt. (A) der Master Scan des Abschnitts H & E. (B, C) Ein 10 X und 40 X Vergrößerung bzw. für jeden Tumor Morphologie identifiziert. (D) immunhistochemische Färbung für ER bei 10 X Vergrößerung. (E) immunhistochemische Färbung für Ki67 bei 10facher Vergrößerung zeigt eine erhöhte mitotische Aktivität im Tumor 1. (F) normalisiert Ausdruck Niveaus für das ESR1-gen in jeder Tumor relativ hoch und gleich Ausdruck zwischen Tumoren zeigen, wie aus dem immunhistochemischen Ergebnis erwartet. (G) normalisiert Ausdruck Niveaus der FN1, mesenchymalen Marker, wo erhöhte FN1 Ausdruck von hohen Ki67 begleitet ist Färbung zeigt eine Signatur für ein invasiver Tumor. Die Inverse ist im Tumor 2, beobachtet, die scheint, ein langsamer wuchernden Tumor mit niedriger malignen Potential durch reduzierte FN1 Ausdruck dargestellt werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Eine Perle-basierte multiplex bDNA-Assay wurde optimiert, um gen-Expression auf degradierten RNA aus FFPE Krebs Brustgewebe und normale Brust Kanäle abgeleitet zu quantifizieren. Optimierung der Assay, Normalisierung beteiligten entwickeln eines Algorithmus zum Klassifizieren von Brustkrebstumoren in luminalen und basale Subtypen mit 8 bekannten Biomarker und 5 mögliche der Gene. Datennormalisierung erfolgte mit Permutationen der Normalisierung Gene. Die Auswahl der Normalisierung Gene stützte sich auf die beste Vorhersage von Rezeptor-Status mit Luminal/basale Klassifikator Gene. Um Luminal/basale Subtypen aus FFPE Gewebe zu klassifizieren, wurden die Normalisierung Gene ausgewählt Beta-Actin (ACTB), Glyceraldehyde-3-phosphat-Dehydrogenase (GAPDH) und Hypoxanthin-Phosphoribosyltransferase-1 (HPRT1).
Die Methode kann für den Gebrauch in anderen Diagnostik und Forschungsbereiche nach angemessene Auswahl der eingestellten normalisierenden gen angepasst werden. Eine wichtige Anwendung dieser Methode im Bereich Forschung ist die Messung von Biomarkern in Archivalien, die gut mit klinischen Resultaten versehen ist. Dies könnte potentielle prädiktive Marker in retrospektiven Studien zu validieren, schnell und präzise, und langfristige prospektive Studien warten auf krankheitsfreies Überleben und insgesamt Überlebensdaten vermeiden. Unsere Gruppe untersucht derzeit die Verwendung des Assays, Hormonrezeptor-positivem exosomen, zu erkennen, die die Entwicklung eines neuen Algorithmus mit alternativen Normalisierung der Gene für die Datennormalisierung erfordert. Die Verwendung von flüssigen Biopsien und robuste Gen Ausdruck Assays können Hochdurchsatz Multiplex-Assays für die Patienten während der Behandlung angepasst und bieten eine Möglichkeit, die Wirksamkeit der Behandlung, mögliche Rückfall wegen Widerstand gegen die Therapie, zu folgen und die metastasierendem Kapazität des Tumors.
Auch diese Methode hat eine breite Palette von Anwendungsmöglichkeiten in der Diagnose von Tumoren und ist angepasst an die aktuellen diagnostischen Workflow. Die wichtigsten Vorteile dieser Methode im diagnostischen Bereich gehören: (1) Durchführung von Hochdurchsatz-Assays, (2) ohne Subjektivität und fragwürdige Ergebnisse aus Image-basierten Messungen (3) präzise Erkennung von mehreren Zielen gleichzeitig die Genauigkeit verbessern und minimieren die Verwendung von kostbaren Patientenproben und (4) keine Anforderung für hoch spezialisierte Einrichtungen und Human Resources. Die optimale Blutentnahme zusammen mit den low-Input der Materialbedarf für Bead-basierte Multiplex-Assays, ermöglicht weitere Untersuchung des Tumors Heterogenität; mit Laser-Mikrodissektion um mehrere Brennpunkte des malignen Gewebes aus dem gleichen Patienten Abschnitt genau zu trennen, ist es möglich, mehrere Genexpression zwischen ihnen sowie mit aufeinander abgestimmten normalgewebe (Abbildung 4) zu vergleichen. Geringer Materialeinsatz ist lebenswichtig für diagnostische Anwendung auf Tumor Biopsien, die begrenzte Tumorgewebe zu bieten. Die Kapazität des Assays Genexpression von degradierten RNA-Proben messen ermöglicht einfachen Transport der Proben für die Analyse innerhalb einer Einrichtung oder Wartung Laboratorien. Ganzer Abschnitt Analyse war darüber hinaus auch möglich mit H & E gefärbt Material (Abbildung 1).
Für den Erfolg dieses Protokolls ist es zwingend notwendig, um: (1) ordnungsgemäße Probenahme der Tumor Website/s, die für den Assay lysiert werden und (2) entwickeln sich gut optimiert und validiert Daten Normalisierung Algorithmen für jedes Gen Ausdruck Panel und/oder individuelle prognostische zu gewährleisten oder prädiktive Biomarker. Erstere hängt von der technischen Erfahrung der Techniker/Wissenschaftler die Probenahme durchführen. Es wird empfohlen, eine zusätzliche Kern und bereiten ein Tissue Microarray (TMA) in das gleiche Format des Multiplex-magnetic Bead Assays (96-Well-Format). Dieses versieht ein Archiv von Tumor Websites als eine Replik von Proben für die RNA-basierten Assay verwendet. TMAs können auch mit anderen Techniken für Follow-up-Forschung oder Validierung von Ergebnissen beurteilt werden. Die Entwicklung der Normalisierung Algorithmen hängt das Material untersucht und die Normalisierung Gene ausgewählt für die Normalisierung. Verschiedenen Panels der Normalisierung Gene werden ausgewählt, basierend auf das Niveau und die Variabilität des Ausdrucks in der Probe analysiert und diese variiert zwischen Krebsgewebe unterschiedlicher Herkunft, exosomen von Plasma oder zirkulierenden Tumorzellen. Validierung des Tests beinhaltet Probe Verarbeitung, da verschiedene Präparate auch zu verschiedenen Normalisierung Algorithmen führt.
Zusammenfassend, die Verwendung von bDNA Technologie in Kombination mit magnetic Bead-Technologie und der Wahl der richtigen Kassette Zielgene, bieten den zusätzlichen Vorteil der Genexpression direkt im Gewebe Lysates abgeleitet von kleinen Mengen an Messen Patientenmaterial, einschließlich Microdissected Material, exosome und zirkulierenden Tumorzellen. Neben der Erkennung von Tumor Heterogenität hat die ordnungsgemäße Verwendung der Platten das Potenzial, Tumor abgeleitet exosomen für Früherkennung und frühzeitige Erkennung von Rückfällen zu erkennen. Da gibt es keine Notwendigkeit für eine Nukleinsäure-Amplifikation Schritt, die Signalverstärkung bDNA Technologie, kombiniert mit der Wulst-basierte Multiplex misst mehrere Genexpression in klinisch kommentiert Archivmaterial und bieten eine Ressource für Biomarker-Validierung.
Offenlegungen
Die Invitrogen™ QuantiGene™ Plex-Assay ist proprietäre von Thermo Fisher Scientific.
Danksagungen
Die Arbeit wurde von (1) eine Brust Krebs Projektstipendium (2014-2016) gefördert durch die Aktion für Breast Cancer Foundation und ALIVE 2013 durch Forschung, Innovation & Entwicklung Vertrauen (RIDT) von der University of Malta, (2) die medizinische Fakultät unterstützt. & Chirurgie, Universität von Malta und (3) Projekt ACT finanziert durch die Malta-Rat für Wissenschaft und Technologie durch FUSION: The R & I Technology Development Programm 2016. Die Veröffentlichung dieses Manuskriptes wird unterstützt durch die Jupiter-Luminex-Zuschuss.
Materialien
| Name | Company | Catalog Number | Comments |
| Microtome | Leica | RM2235 | |
| Heamatoxylin Mayer's | Sigma | MHS16-500mL | |
| Eosin Y Aquaeous solution | Sigma | HT110216-500mL | |
| Normal Rabbit Serum | Monosan | MONX10963 | Working dilution: 1/40 |
| Biotinylated Rabbit anti-mouse | Dako | E0354 | Working dilution: 1/200 |
| ER antibody (6F11) | Vector Laboratories | VPE614 | Working dilution: 1/45 |
| HER2 antibody (CB11) | Novocastra | CB11-L-CE | Working dilution: 1/325 |
| Ki67 antibody (MIB-1) | Dako | M7240 | Working dilution: 1/500 |
| Avidin Biotin Complex kit | Vector Laboratories | PK-6100 | |
| Nikon Eclipse Ti-E Inverted microscope | Nikon | Ti-E | 4x, 10x, 20x and 40x objectives |
| Laser Microdissection membranes | Molecular Machines &Industries | S0103 | |
| mmi CellCamera 1.4 | Molecular Machines &Industries | MX4285c-ACK07 | |
| mmi Cellcut Plus | Molecular Machines &Industries | ||
| Diffuser caps | Molecular Machines &Industries | 50210 | |
| mmi Celltools Software v.4.01rcl | Molecular Machines &Industries | ||
| Eppendorf Thermomixer comfort | Eppendorf | 5355000038 | |
| 1.5mL heating block for Eppendorf Thermomixer | Eppendorf | 22670522 | |
| 96-well plate heating block for Eppendorf Thermomixer | Eppendorf | 22670565 | |
| Labnet Vortemp 56 Shaking incubator | Labnet | 52056A-220 | |
| LX200 100/200 | Luminex | Magnetic bead analyser | |
| Invitrogen QuantiGene Sample Processing Kit - FFPE Tissues | ThermoFisher Scientific | QS0109 | |
| Invitrogen QuantiGene Plex 2.0 Assay Kit (Magnetic Separation) | ThermoFisher Scientific | QP1011 | |
| Thermaseal RTS Sealing Film | Thermaseal | 765246 | |
| Hand-Held Magnetic Plate Washer | ThermoFisher Scientific | QP1011 | |
| Invitrogen QuantiGene Incubator Temperature Validation Kit | Affymetrix/Panomics | QS0517 | |
| Proteinase K (50µg/µL) | ThermoFisher Scientific | 14622 | |
| Invitrogen QuantiGene Plex 2.0 Sets | ThermoFisher Scientific | Various | |
| Multi Speed Vortex | Kisker Biotech | MSV-3500 | |
| Sonicator | Silvercrest | ||
| RNASEZAP | Sigma | R2020-250ML | |
| Aluminium 96-well plate seal | Sigma | Z721549-100EA | |
| Temperature Validation Kit | ThermoFisher Scientific | QS0517 | |
| RapidMiner Studio Community 7.1.001 | RapidMiner | Data Science Platform | |
| Hybridisation oven | Hybaid (Thermo Scientific) |
Referenzen
- Abrahamsen, H. N., Steiniche, T., Nexo, E., Hamilton-Dutoit, S. J., Sorensen, B. S. Towards quantitative mRNA analysis in paraffin-embedded tissues using real-time reverse transcriptase-polymerase chain reaction: a methodological study on lymph nodes from melanoma patients. J Mol Diagn. 5 (1), 34-41 (2003).
- Macabeo-Ong, M., et al. Effect of duration of fixation on quantitative reverse transcription polymerase chain reaction analyses. Mod Pathol. 15 (9), 979-987 (2002).
- Yang, W., et al. Direct quantification of gene expression in homogenates of formalin-fixed, paraffin-embedded tissues. Biotechniques. 40 (4), 481-486 (2006).
- Flagella, M., et al. A multiplex branched DNA assay for parallel quantitative gene expression profiling. Anal Biochem. 352 (1), 50-60 (2006).
- Goldhirsch, A., et al. Strategies for subtypes--dealing with the diversity of breast cancer: highlights of the St. Gallen International Expert Consensus on the Primary Therapy of Early Breast Cancer 2011. Ann Oncol. 22 (8), 1736-1747 (2011).
- Schnitt, S. J. Classification and prognosis of invasive breast cancer: from morphology to molecular taxonomy. Mod Pathol. 23, S60-S64 (2010).
- Bae, Y. K., et al. Fibronectin expression in carcinoma cells correlates with tumor aggressiveness and poor clinical outcome in patients with invasive breast cancer. Hum Pathol. 44 (10), 2028-2037 (2013).
- Park, J., Schwarzbauer, J. E. Mammary epithelial cell interactions with fibronectin stimulate epithelial-mesenchymal transition. Oncogene. 33 (13), 1649-1657 (2014).
- Zhou, Z., et al. MRI detection of breast cancer micrometastases with a fibronectin-targeting contrast agent. Nat Commun. 6, 7984 (2015).
- Antonyak, M. A., et al. Cancer cell-derived microvesicles induce transformation by transferring tissue transglutaminase and fibronectin to recipient cells. Proc Natl Acad Sci U S A. 108 (12), 4852-4857 (2011).
- Moon, P. G., et al. Fibronectin on circulating extracellular vesicles as a liquid biopsy to detect breast cancer. Oncotarget. 7 (26), 40189-40199 (2016).
- Parker, J. S., et al. Supervised risk predictor of breast cancer based on intrinsic subtypes. J Clin Oncol. 27, (2009).
- Nielsen, T., et al. Analytical validation of the PAM50-based Prosigna Breast Cancer Prognostic Gene Signature Assay and nCounter Analysis System using formalin-fixed paraffin-embedded breast tumor specimens. BMC Cancer. 14, 177-177 (2014).
- Fischer, A. H., Jacobson, K. A., Rose, J., Zeller, R. Hematoxylin and Eosin Staining of Tissue and Cell Sections. Cold Spring Harbor Protocols. (5), (2008).
- Lin, F., Prichard, J. . Handbook of Practical Immunohistochemistry: Frequently asked questions. , (2011).
- Grech, G., et al. Molecular Classification of Breast Cancer Patients Using Formalin-fixed Paraffin-embedded Derived RNA Samples. Journal of Molecular Biomarkers & Diagnosis. 7 (S8), (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten