Method Article
Visualisierung der DNA Verdichtung in Cyanobakterien durch Hochspannungs-Cryo-Elektron Tomographie
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt, wie die vorübergehende DNA-Verdichtung in Cyanobakterien zu visualisieren. Synchrone Anbau, Überwachung durch die Fluoreszenz-Mikroskopie, schnelles Gefrieren und hoher Spannung Cryo-Elektron Tomographie verwendet. Ein Protokoll für diese Methoden vorgestellt, und zukünftige Anwendungen und Entwicklungen diskutiert.
Zusammenfassung
Dieses Protokoll beschreibt, wie die vorübergehende DNA-Verdichtung in Cyanobakterien zu visualisieren. DNA-Verdichtung ist eine dramatische zytoplasmatischen Ereignis fand vor kurzem in einigen Cyanobakterien vor der Zellteilung auftreten. Jedoch aufgrund der großen Zellengröße und der vorübergehende Charakter ist es schwierig, die Struktur im Detail zu untersuchen. Um die Schwierigkeiten zu überwinden, ist zunächst DNA Verdichtung reproduzierbar in cyanobakteriums vorkommenden Elongatus PCC 7942 durch synchrone Kultur mit 12 h jedes Hell/Dunkel-Zyklus produziert. Zweitens ist die DNA Verdichtung durch Fluoreszenzmikroskopie überwacht und aufgezeichnet durch schnelles gefrieren. Drittens: die detaillierte Struktur der DNA Zellen verdichtet wird in drei Dimensionen (3D) durch Hochspannung Cryo-Elektron Tomographie visualisiert. Dieser Satz von Methoden ist überall anwendbar, transiente Strukturen in Bakterien, z. B. die Zellteilung, Chromosom Abtrennung, Phagen Infektion etc.zu untersuchen., welche durch Fluoreszenzmikroskopie überwacht und direkt visualisiert, indem Cryo-Elektron Tomographie zu geeigneten Zeitpunkten.
Einleitung
DNA-Verdichtung ist eine dramatische zytoplasmatischen Ereignis, das in einigen Cyanobakterien identifiziert wurde. Wenn vorkommenden Elongatus kultiviert wurde unter 12 h Zyklus hell/dunkel, erschien Ende der lichtperiode, unterscheidet sich deutlich von seinen Auftritt auf das andere Mal war kondensierten DNA Punkte1. Es wurde vermutet, dass dieser Prozess durch eine innere Uhr, die basierend auf dem Kai Proteine2gesteuert wird. Seki Et Al. haben berichtet, dass die DNA mit Hoechst 33342 befleckt war in S. Elongatus Zellen gegen Ende der lichtperiode verdichtet und zeigte eine wellige Stab-Form unter dem Fluoreszenzmikroskop. Die verdichtete DNA dann getrennt in zwei in der Mitte der Stange wie die Zelle geteilt, und schließlich zurück zu einem normalen gleichmäßige Verteilung in jede tochterzelle3. Jedoch behindert seine Vergänglichkeit und große Größe für Elektronenmikroskopie Strukturanalyse. Murata Et Al. kombiniert mehrere Methoden, einschließlich der synchronen Kultur, Fluoreszenz-Mikroskopie, schnelles Gefrieren und hoher Spannung Elektron Cryo-Tomographie (Cryo-HVET), und erfolgreich bei der Ermittlung der Struktur des vorübergehenden DNA Verdichtung einschließlich der Kinetics von Polyphosphat Körper (PPBs)4. Das Manuskript enthält bildliche Erklärung des Bildmaterials schwer im Detail durch die Kombination der experimentellen Verfahren.
S. Elongatus hat eine Kapsel Form, eine Länge von 2 bis 5 µm, einer Breite von über 0,5 µm und die perfekte DNA-Verdichtung erscheint in lebenden Zellen nur für sehr kurze Zeit. Daher waren die strukturellen Veränderungen in der Cyanobakterien DNA Verdichtung im Detail unbekannt. Um diese Strukturen durch Elektronenmikroskopie untersuchen, ist es notwendig, zwei technische Probleme zu überwinden. Ist die Beobachtung einer dicken Probe des ganzen Bakteriums bei in der Nähe von einheimischen Bedingungen, und die andere ist die schnelle Fixierung einer dynamischen Struktur. Für das erste Problem hängt die unelastische mittlere freie Weglänge (iMFP) der Elektronen die Beschleunigungsspannung der Elektronen-Mikroskop5. In einem Transmissionselektronenmikroskop (TEM) von 300 kV, es ist weniger als 350 nm. Zum Beispiel, wenn ein Eis eingebettet cyanobakteriums (Probe Dicke ≈ 600 nm) wird bei 200kV TEM beobachtet (iMFP ≈ 250 nm), die Strukturen im Inneren der Zelle sind schwer zu beobachten. Durch Kontrast, 1 MV TEM (iMFP ≈ 500 nm) geben kann und Bild der cytoplasmatische Struktur in der Zelle (Abbildung 1). In diesem Protokoll, als ein Teil der Lösung, eine Hochspannungs-Elektronen-Mikroskop (HVEM) bei einer Beschleunigungsspannung von 1 MV war beschäftigt. Allerdings sind Einrichtungen, die HVEM implementieren weltweit begrenzt. Mögliche alternative Lösungen sind auch in die Diskussion Abschnitt diskutiert. Das zweite Problem wurde durch Kryo-Elektronenmikroskopie (Cryo-EM) gelöst. Dies ist ein leistungsfähiges Werkzeug zur Visualisierung von dynamischen Strukturen auf in der Nähe von einheimischen Bedingungen, wo die Probe ist in flüssigem Ethan mit einem schnellen Einfrieren Gerät schnell eingefroren, und der eingefrorene Moment wird direkt mit einem Minimum an Modifikationen6beobachtet. Kombination mit Computertomographie, kann eine Momentaufnahme der dreidimensionalen (3D) Strukturen aus der Neigung der Serie7rekonstruiert werden. In diesem Experiment DNA Verdichtung wurde in S. Elongatus mit synchronen Kultur unter 12 h Zyklus jeder hell/dunkel wiedergegeben, und der Zeitplan für das Einfrieren der Probe wurde durch Überwachung unter dem Fluoreszenzmikroskop bestimmt.
Die hier beschriebenen Vorgehensweisen gelten allgemein für die Untersuchung von dynamischen Strukturen in Bakterienzellen, z. B. die Zellteilung, Chromosom Segregation und Phagen-Infektion, und haben ein Potenzial, neue Wege in der mikrobiologischen Forschung zu öffnen.
Protokoll
(1) synchrone Kultur der Cyanobakterien
- Kultur S. Elongatus PCC 7942 auf sterilisierte BG 11 Teller (in einer 9 cm steril Kunststoff Petrischale), 1,5 % (w/V) Agar und 0,3 % (w/V) Natriumthiosulfat8enthalten.
- Legen Sie die Platten in einer Kammer Wachstum bei 23 ° C mit einer Intensität von 50 µE/m2/s und je nach 12 h Licht/12 h dunkel-Zyklen.
- Übertragen Sie die Zellen auf frischer BG11 Agarplatten einmal pro Woche.
Hinweis: Die Kulturen auf Agar erscheinen als grüne Bänder der aktiv proliferierender Zellen nach einer Woche unter dieser Bedingung Kultur. - Nehmen Sie grüne Klumpen von Zellen mit einer Flamme sterilisiert Drahtschlinge und Streifen die Zellen auf ein frischer BG11 nährbodenplatte. Tun Sie dies auf einer sauberen Werkbank.
2. Überwachung durch Fluoreszenz-Mikroskopie
- Verwenden Sie Zellen auf der Agarplatte für 6 Tage kultiviert, um DNA Verdichtung zu beobachten. Sammeln Sie Zellen von der Platte am Ende die lichtperiode durch Gießen 1 mL 0,2 M Saccharose-Lösung über die Zellen. Wiederholen Sie, Gießen die Lösung auf die Zellen, so dass die meisten Zellen gesammelt werden. Übertragen Sie die Lösung suspendiert-Zelle in eine Mikro-Schlauch für DNA-Färbung.
- Fügen Sie DNA Farbstoff (z.B. Hoechst 33342) Färbelösung zu 500 µL der hängenden Zelle Lösung in ein Mikro-Schlauch, eine Endkonzentration von 1 µg/mL. Dann halten Sie das Rohr in der Dunkelheit für 10 Minuten.
- Zentrifuge für 1 min bei 2.000 X g zu sedimentieren die Zellen. Verwerfen Sie den überstand und fügen Sie 10 µL 0,2 M Saccharoselösung um einen dichten Zellsuspension zu erhalten.
- 1 µL der Lösung mit gefärbten Zellen zu einem Dia-Glas zu übertragen, setzen ein Deckglas und beobachten mit einem Fluoreszenzmikroskop mit einem UV-Filter mit einem Objektiv mit einer Vergrößerung von 100 X und Immersionsöl ausgestattet.
- Bestätigen Sie, dass die DNA-Verdichtung an dieser Stelle in den meisten Zellen beobachtet wird, dann Einfrieren als nächstes bereiten Sie die Probe vor.
3. Probe einfrieren für Cryo-HVET
- Ein Sprung-Einfrieren-Gerät einrichten. Füllen Sie den Tank mit flüssigem Stickstoff und starten Sie die Cryo-Kühlkammer nach dem Anschluss des Tanks und der Kammer mit einem Teflonschlauch.
- Füllen Sie flüssiges Ethan in einen kleinen Cupper Topf im Inneren der Kammer nach dem Abkühlen der Kammer auf Temperatur von flüssigem Stickstoff. Trage eine Brille während des Betriebes, denn flüssiges Ethan explosiv ist.
- Glow discharge die Kohlenstoff-Seite von einer löchrigen Kohlenstoff-beschichtete EM Grid (löchrige Netz) für 30 s bei 50 mA mit einem Plasma-Ionen Bombarder.
- 1 µL der BSA gold Tracer gelten (15 nm) an das löchrige Netz als treuhändische Marker.
- Ein Aliquot 2,5 µL Zellen in der DNA Verdichtung Stufe auf den löchrigen Raster anwenden Tupfen Sie die überschüssige Lösung mit einem Filterpapier. Tauchen Sie das Raster in flüssigem Ethan mit einem Kolben im Sprung Einfrieren Gerät sofort.
- Speichern Sie die gefrorenen Gitter in flüssigem Stickstoff Lagerung, bis sie untersucht werden.
(4) Cryo-HVET
- Richten Sie die HVEM bei einer hohen Spannung von 1 MV.
- Montieren Sie das gefrorene Raster in einem Cryo-Probenhalter für HVEM vorgekühlt bis-150 ° C mit flüssigem Stickstoff innerhalb der Cryo-Workstation und laden Sie sie in die HVEM. Achten Sie darauf, zur Vermeidung von Kontaminationen durch Eis.
- Wählen Sie einen imaging-Bereich bei niedriger Vergrößerung 1, 000 X. Die euzentrische z-Höhe einstellen.
- Kippen Sie den Objekttisch bis-60 ° und entfernen Sie die Gegenreaktion der kippbaren Rotation zu.
- Stellen Sie den Fokus in der Nähe von den Zielort bei einer Vergrößerung von 10, 000 X. Legen Sie eine unter Fokus von 6 bis 10 µm durch Abweichung von den scharfen Bild. Für Bildgebung, setzen Sie die Dosis auf 2 e–/Å-2 oder weniger auf die Probe vorher.
- Messen Sie die Elektron-Dosis durch die Stromdichte auf dem Bildschirm in HVEM. Nehmen Sie ein Bild auf ein Elektron Film oder Digitalkamera mit der gleichen Vergrößerung wie bei der Fokussierung Prozess.
- Kassiere Tilt Bilder manuell nach dem gleichen Verfahren wie in (4.5)-60 ° bis + 60 ° bei einer Neigung Winkel Inkrement von 2°, 4°.
Hinweis: In vielen modernen Elektronenmikroskopen, ist der Erwerb der Tilt-Serie durch die Kombination von einer digitalen Kamera automatisiert. In diesem Fall folgen Sie der Anleitung. Für negativ-Filme, die Filme für 12 min. bei 20° C in einem entwicklertank mit voller Stärke Entwickler entwickeln und fix in ein Fixiermittel für 10 min. digitalisieren die Filme mit einer Auflösung von 4.000 dpi (0.635 nm/Pixel im Bild) mit einem Flachbettscanner. Bei einer Digitalkamera vorbehaltlich der gesammelten Bilder direkt zum nächsten Bild Verarbeitungsschritt. Bei Bedarf verkleinern Sie Bild von einem Median-Filter mit zwei bis vier binning (Größe Verkürzungsfaktor) und entsprechender Software (z.B. ImageJ).
5. tomographische Rekonstruktion
- Machen Sie eine Stapel-Image-Datei aus den einzelnen Bilder mit dem Befehl "tif2mrc" oder "Newstack" in IMOD Software9zu kippen.
- Starten Sie eTomo GUI Software in IMOD und Bildparameter gesetzt: Pixelgröße, treuhändische Durchmesser, Bilddrehung, etc.. Dann erstellen Sie Skripts.
- Individuelle Programme nach den aufgeführten Tabellen von Materialien, Software ausführen wo die Tilt-Serie mit kugelmarker (mit einem mittleren Restfehler weniger als 0,5) ausgerichtet ist. Zu guter Letzt ein 3D Tomogramm mit der SIRT-Algorithmus in IMOD zu rekonstruieren.
- Die Tomogramm eine Region of Interest (ROI) entziehen und denoise mithilfe eines Filters Denoise: ein anisotroper Diffusion in IMOD, eine bilaterale Filter EMAN10oder eine mathematische Morphologie filter11, etc., mit entsprechenden Parametern um den Kontrast zu verbessern.
6. Segmentierung der Funktion von Interesse
Hinweis: Die unten beschriebene Verfahren ist spezifisch für die Software verwendet (siehe Materialtabelle) aber andere Softwarepakete können stattdessen verwendet werden. Lesen Sie in ihrer Bedienungsanleitung.
- Im 3D Viewer-Fenster können Tomogramm auf Amira Software öffnen und eine OrthoSlice zu generieren.
- Erstellen Sie im Editorfenster Segmentierung eine Segmentierung Datei indem Sie ein neues "Label-Feld" auswählen.
- Nachzeichnen Sie die Begrenzung der Funktion von Interesse (FOI) manuell. Folgen Sie der FOI durch alle Tomogramm Scheiben. Erstellen Sie für die zweite FOI ein neues "Material" und wiederholen Sie den Vorgang.
- Generieren Sie eine Surface Rendering durch Auswahl im Menü "SurfaceGen". Um die segmentierten Volumen sichtbar zu machen, wählen Sie Menü "SurfaceView". Zum Verschieben, drehen und Zoomen in das 3D-Volumen, verwenden Sie die Tools in der 3D Viewer-Fenster.
- Verwenden Sie für automatische Segmentierung der Magic-Wand-Tool. Klicken Sie auf ein Objekt, und stellen Sie den Regler bei Anzeige und Maskierung den Wertebereich abzudecken, so dass das Objekt vollständig von seinen Funktionen ausgewählt ist.
Ergebnisse
In einer präzise synchrone Kultur unter 12 h Zyklus jeder hell/dunkel, zeigt beschriftet mit Hoechst DNA eine normale gleichmäßige Verteilung in den dunklen Zustand (Abbildung 2a). Jedoch schrittweise innerhalb der Zelle während der lichtperiode verdichtet, und erscheint als eine stabförmige Wellenstruktur (Abb. 2 b) am Ende der lichtperiode. Schließlich teilt des Stabs in der Mitte (Pfeile in Abbildung 2 c) und zwei Teile sind verteilt auf die Tochterzellen (Abb. 2d). Nach der Zellteilung die verdichtete DNA verschwindet sofort, und die DNA wieder in eine normale gleichmäßige Verteilung.
Wenn ein Aliquot der Zellen, in denen die Zellen in der letzten Phase der DNA Verdichtung wurden sofort übertragen, auf einem löchrigen Raster und schnelle Einfrieren in flüssigem Ethan, und gefrorenen Raster wurde beobachtet, von 1 MV Cryo-HVEM, die internen Strukturen der Cyanobakterien einschließlich DNA, Thylakoidmembran Membran Schichten und Zellwände PPBs, erschien wie eine Momentaufnahme zum Zeitpunkt des Einfrierens (Abb. 3a). Viele Zellen könnte zeigte deutliche Verdichtung der DNA in den Zellen (weiße Pfeile in Abbildung 3a), und leicht zu unterscheiden von normalen Zellen (weiße Pfeilspitzen in Abb. 3 b). Einige ausgestellt einer Einschnürung in der Mitte der Zellen, wie vor der Zellteilung (gelber Pfeil in Abbildung 3a) erwartet.
In 3D Schichtbilder könnte große Organellen der Zelle segmentiert werden; Zellwand, Thylakoidmembran Membran Schichten, DNA und PPBs werden konnte (Abbildung 4) zu unterscheiden. Insbesondere war die verdichtete DNA durch eine deutliche Lücke im Zytoplasma getrennt, wo die DNA von geringer Dichte Material umgeben war und die Thylakoidmembran Membran Schichten auf dem welligen Stab der verdichteten DNA verzerrt waren. Dynamisches Verhalten des PPBs wurde neu beobachtet: in DNA verdichtet Zellen, viele kleine PPBs galten, DNA, einzuhalten, während sie groß sind und weniger in den normalen Zellen. Darüber hinaus ein Großteil der PPBs erschien als Paare, und einige DNA schien sich in einem Prozess der Trennung von der PPBs. Dies suggeriert, dass PPBs selbst als Anbieter von Phosphat für DNA-Synthese durch DNA-Vervielfältigung und Funktion in zwei aufgeteilt werden.
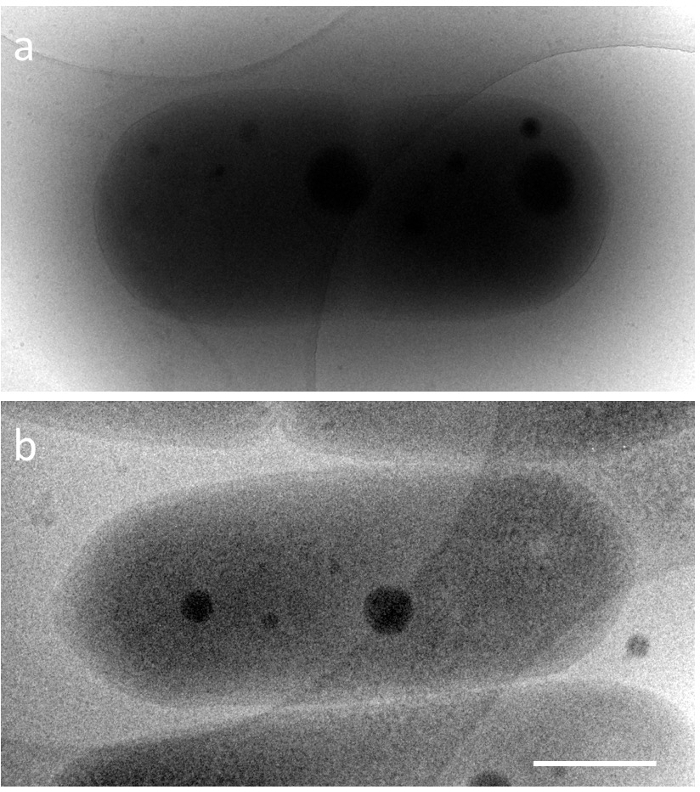
Abbildung 1. Cryo-EM Bilder von Eis eingebettet normalen Cyanobakterien, S. Elongatus PCC 7942 bei verschiedenen zunehmende Spannungen. (a) 200kV und (b) 1000kV. Maßstabsleiste = 500 nm. Bitte klicken Sie hier für eine größere Version dieser Figur.
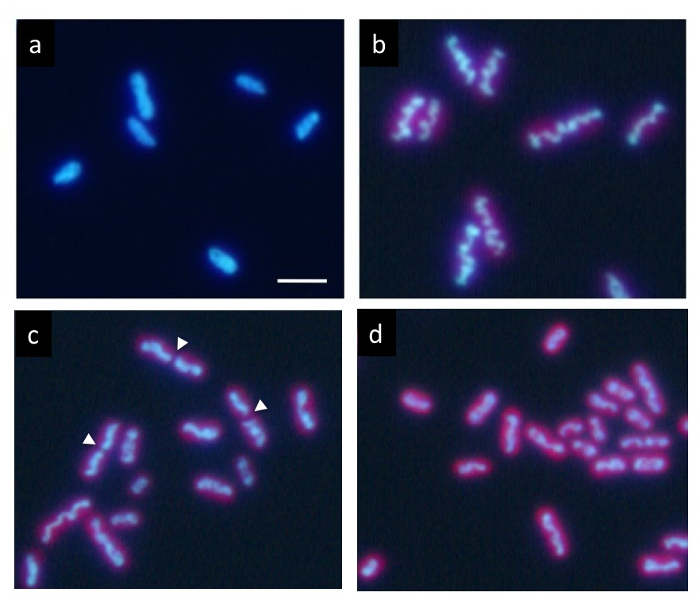
Abbildung 2: Fluoreszenz-Mikroskopie des cyanobakteriums S. Elongatus Zellen. Nach dem synchronen Kultur unter 12 Stunden jede hell/dunkel-Zyklen, wurden Zellen mit Hoechst 33342 gebeizt. (a) Zellen nach 2 h Auftreten von den dunklen Zustand zeigen einheitlich DNA Kennzeichnung. Die DNA in vielen Zellen verdichtet sich allmählich während der lichtperiode. Es bildete letztlich einen dicken Stab wellig (b) typisch für DNA-Verdichtung. Nach dieser Phase, die verdichteten DNA-Strukturen schnell in ihren Zentren (Pfeilspitzen) während der Zellteilung (c), und die zwei Fragmente aufgeteilt in die Tochterzellen getrennt (d). Die Tochterzellen wieder mehr einheitliche DNA Kennzeichnung wieder in die dunkle Zeit. Maßstabsleiste = 2 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
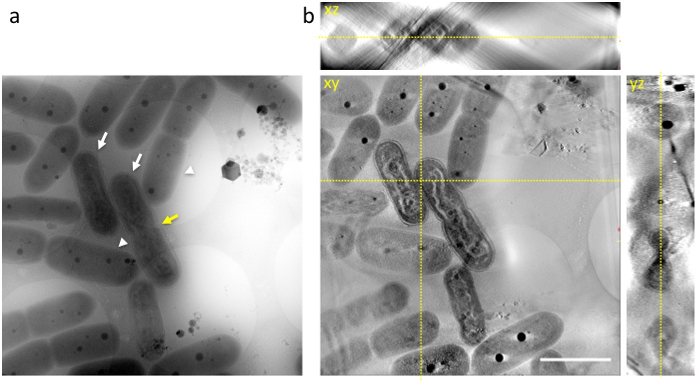
Abbildung 3: Cryo-HVEM Bild von Cyanobakterien eingebettet in amorphen Ice (a) zeigt ein raw-Bild bei 0° Neigung. Bilder wurden am Ende der lichtperiode. Einige Zellen zeigen wellig stabförmige DNA Körper ähnlich wie eukaryotischen kondensierten Chromosomen (weisse Pfeile). In normalen Zellen weisen Cyanobakterien eine erkennbare DNA-Struktur innerhalb des Zytoplasmas (weiße Pfeilspitzen). Einige Weisen einer Einschnürung in der Mitte der Zellen erwartungsgemäß vor der Zellteilung (gelber Pfeil). (b) Xy, Xz, Yz-Scheiben von einem 3D Tomogramm. Gelber gepunkteten Linien zeigen Schnittpunkte der Scheiben. Maßstabsleiste = 2 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
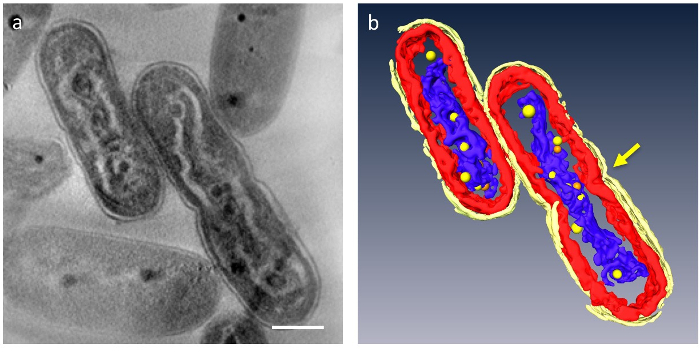
Abbildung 4: Ultrastruktur der Zellen, die DNA verdichtet. Die Hauptkomponenten sind segmentiert: Zellwand (hellgelb), Thylakoidmembran Membranen (rot) und DNA (blau). (a) zeigt eine Z-Scheibe von einem 3D Tomogramm. (b) alle Segmente. Die Einschnürung Zellseparation Angabe erscheint in der Mitte der Zelle (Pfeil). PPBs werden als gelbe oder orangefarbene Kugeln modelliert; jede orange Kugel stellt das Gegenstück zu den engsten gelbe Kugel. Maßstabsleiste = 500 nm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Wir haben eine Reihe von Protokollen zur Visualisierung von Transienten DNA Verdichtung in Cyanobakterien vorgestellt. Das grundlegende Konzept ist ähnlich dem Korrelative Licht-und Elektronenmikroskopie (CLEM)12. Darüber hinaus in dieser Methode waren live Cyanobakterien überwacht durch Fluoreszenz-Mikroskopie, schnell eingefroren auf EM Gittern und direkt mit der Hochspannung Cryo-Elektron Tomographie visualisiert. Als eine erste Anwendung, die detaillierte Struktur der DNA Bakterienzellen verdichtet wurde erfolgreich in 3D visualisiert. Derzeit, dieses Verfahren ist speziell für dieses Thema, aber intensiver, mit modifizierten Methodik in einigen Fällen angewendet wird. Hier werden die Vorteile, die Einschränkungen und die zukünftigen Möglichkeiten dieser Methode diskutiert.
Ein Vorteil dieser Methode ist die 3D Visualisierung der gesamten Zelle. 1 MV HVEM erfolgreich visualisiert die dynamische Struktur der subzellularen Organellen in der DNA Zellen verdichtet. Allerdings könnte die feine Struktur in normalen Zellen nicht aufgrund geringer Bildkontrast unterschieden werden. Erhöhung der unelastisch und Mehrfachstreuung bei dicken Exemplaren verwischt das Bild13. NULL- und die meisten wahrscheinliche Verlust Bildfilterung durch einen Energie-Filter kann durch die Reduzierung inelastische Streuung14,15Bildkontrast verbessern, aber es funktioniert nicht für Proben, die dicker als iMFP. Die NULL- und die meisten wahrscheinliche Verlust Gipfel verringern drastisch mit Dicke der Probe. Es ist besonders schwierig, einen ausreichenden Signal-Rausch-Verhältnis für die Elektron sensible Eis eingebettete Proben zu erhalten. Murata Et Al. haben gezeigt, dass 1MV Scan Übertragung Mikroskopie (STEM) gibt höheren Kontrast als ein Hellfeld-Bild in Kunststoff eingebettet Hefezellen mit 5 µm Dicke, wo den Kontrast des Bildes in erster Linie von Amplitude Kontrast13 gegeben . Allerdings wird erwartet, dass die Wirkung von Domino Schaden auf höhere beschleunigten Elektronen eine weitere Einschränkung auf die Bestrahlungsdosis für Schaden-Sensitive Cryo-Proben16schafft. Die Anwendung der Volta und Zernike Phase Platten17,18 für HVEM möglicherweise nachteilige Schäden verringern, indem die Gesamtdosis in der Zukunft. Eine weitere Einschränkung mit HVEM bei dicken Proben kommt von der Tatsache, dass die Benutzer Einrichtungen, die HVEM weltweit knapp sind.
Verwenden eine alternative Methode, um Dicke Proben, Cryo-STEM-Tomographie bei 300 zu beobachten hat kV kontrastreiche Bilder auf gefrorene hydratisiert Proben mit einer Dicke von mehr als einige hundert Nanometer19gezeigt. Phasenkontrast in Cryo-STEM abzurufen, hat Pthychographic Elektronenmikroskopie auch eingeführt, in denen transponiert der Phase-Platte im Kondensator Objektiv eine Phase moduliert Beugung eine Pixelige 2D Detektor20. Die Bilder werden durch Berechnung aus mehreren Talfüllung abgerufen. Für direkte und schnelle 3D Cryo-Imaging von großen Native gefrorene Proben, Cryo-FIB-SEM kann auch gebrauchte21, wo seriellen Schnitt mit einem fokussierten Ionenstrahl und Gesicht Bildgebung blockieren ist für Bildgebung vollständig gefrorene Proben hydratisiert aufgetragen. Obwohl diese Technologien die Sichtweite des biologischen Proben erweitern, es ist schwer zu finden, den Zielspeicherort der Bakterien, z.B. Bakterien, bezeichnet, weil das Ziel vollständig unter dem Eis steht und nicht vor dem Trimmen identifiziert werden.
DNA-Verdichtung erzeugt eine klare Struktur in Cyanobakterien. DNA verdichtet Zellen unterscheiden sich leicht auch ohne wird durch eine große Dichte Tendenz innerhalb der Zellen, die nicht in normalen Zellen ist befleckt. Jedoch um weitere lokalen Veranstaltungen innerhalb der Zelle sichtbar zu machen, ist es notwendig den eindringmittel beschrifteten ROI in das Elektronenmikroskop zu übertragen. Für Korrelative Licht- und Elektronenmikroskopie (CLEM) sind in der Regel Lichtmikroskop und Elektronenmikroskop Bilder korreliert mit fluoreszierenden Latex-Kügelchen oder Quantenpunkte auf EM Finder Raster12. Die Kennzeichnung Partikel muss hohe Elektronendichte neben Fluoreszenz. Sie können genau und zuverlässig die Positionen zwischen den beiden Bildern korrelieren. Darüber hinaus kann durch die Bestätigung des markierten Bereich mit Cryo-Licht-Mikroskopie, vollständige Überlappung der ROI zwischen zwei Mikroskopen erreicht werden. Wenn genauere strukturelle Ereignisse in DNA Verdichtung zu charakterisieren, werden diese Partikel und Cryo-Licht Mikroskop ein unverzichtbares Werkzeug für eine robuste und präzise Korrelation in der Zukunft.
Dieser Artikel zeigt, wie die vorübergehende Struktur der DNA Verdichtung in Cyanobakterien durch eine Kombination von synchronen Kultur, Fluoreszenz-Mikroskopie und Hochspannung Cryo-Elektron Tomographie zu charakterisieren. Dieses Protokoll konzentriert sich auf die Beobachtung der verdichteten DNA. Durch die Kombination dieser Methode mit anderen neuen Technologien erwähnt, es wird möglich sein, den Prozess der DNA Verdichtung näher untersuchen und entsprechend modifizierte Methoden sind allgemein anwendbar auf anderen dynamischen strukturellen Ereignisse in Bakterien.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Die Autoren danken Tammo Reisewitz für kritische Lesart der Handschrift, und Mako Hayashi und Sayuri Hagiwara für sorgfältige Pflege und Beobachtung von Cyanobakterien, Yoshitaka Kimori für die Bildverarbeitung, Chihong Song, Naoyuki Miyazaki und Miyoko Nagayoshi für die Hilfe bei Segmentierung der Strukturen. Diese Arbeit wurde durch die Collaborative Study Program des National Institute für physiologische Wissenschaften (NIPS), Y.K unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Hoechst 33342 solution | Dojindo | 346-07951 | 1mg/mL in H2O |
| Agar Powder (for plant culture) | Wako | 016-11875 | |
| Boric acid | Wako | 027-02192 | 99.5% |
| Manganese chloride tetrahydrate | Wako | 139-00722 | 99% |
| Zinc sulfate heptahydrate | Wako | 264-00405 | 99.5% |
| Sodium molybdate | Wako | 196-02472 | 99% |
| Copper sulfate pentahydrate | Wako | 039-04412 | 99.5% |
| Cobalt nitrate hexahydrate | Wako | 031-03752 | 99.5% |
| Sodium nitrate | Wako | 191-02542 | 99% |
| Magnesium sulfate heptahydrate | Wako | 131-00405 | 99.5% |
| Calcium chloride dehydrate | Wako | 031-00435 | 99.5% |
| Citric acid | Wako | 036-05522 | 98% |
| EDTA-2Na | Dojindo | 343-01861 | 99.5% |
| Sodium carbonate | Wako | 197-01581 | 99.8% |
| Potassium phosphate dibasic | Wako | 164-04295 | 99% |
| TES (Good’s buffer) | Dojindo | 344-02653 | 99% |
| Ferric ammonium citrate | Wako | 092-00802 | 1st Grade |
| Sodium thiosulfate pentahydrate | Wako | 197-03585 | 99% |
| BSA gold tracer 15nm | Aurion | 215.133 | |
| Quantifoil EM grid | Quantifoil MicroTools | R3.5/1 Copper grid | |
| Electron films | Kodak | SO-163 | |
| Developer | Kodak | D19 | |
| Fixer | Kodak | Rapid fixer | Solution |
| Filter paper | Whatman | Grade 1 | |
| Growth chamber | NKsystem | LH-100SP | |
| Fluorescent microscope | Nikon | ECLIPSE 50i | |
| High voltage TEM | HItachi | H1250M | |
| Cryo-specimen holder for HVEM | Gatan | ||
| plunge-freezing device | Leica | EM CPC | |
| Plasma Ion bombarder | Vacuum device | PIB-10 | |
| Liquid nitrogen storage | Taylor-Wharton | 25LDB | |
| Developing tank | Dosaka EM | TB-3-75 | |
| flatbed scanner | Nikon | Coolscan 9000ED | |
| Segmentation software | FEI | Amira | https://www.fei.com/software/amira |
| Tomographic Reconstruction software | eTOMO | http://bio3d.colorado.edu/imod |
Referenzen
- Smith, R. M., Williams, S. B. Circadian rhythms in gene transcription imparted by chromosome compaction in the cyanobacterium Synechococcus elongatus. Proc. Nat. Acad. Sci. USA. 103 (22), 8564-8569 (2006).
- Kondo, T. A cyanobacterial circadian clock based on the kai oscillator. CSHS Quant. Biol. 72, 47-55 (2007).
- Seki, Y., Nitta, K., Kaneko, Y. Observation of polyphosphate bodies and DNA during the cell division cycle of Synechococcus elongatus PCC 7942. Plant biol. 16 (1), Stuttgart, Germany. 258-263 (2014).
- Murata, K., Hagiwara, S., Kimori, Y., Kaneko, Y. Ultrastructure of compacted DNA in cyanobacteria by high-voltage cryo-electron tomography. Sci. Rep. 6, 34934(2016).
- Koster, A. J., et al. Perspectives of molecular and cellular electron tomography. J. struct. boil. 120 (3), 276-308 (1997).
- Lučić, V., Rigort, A., Baumeister, W. Cryo-electron tomography: The challenge of doing structural biology in situ. J. Cell Biol. 202 (3), 407-419 (2013).
- Murata, K., et al. Visualizing Adsorption of Cyanophage P-SSP7 onto Marine Prochlorococcus. Sci. Rep. 7, 44176(2017).
- Rippka, R., Deruelles, J., Waterbury, J. B., Herdman, M., Stanier, R. Y. Generic assignments, strain histories and properties of pure cultures of Cyanobacteria. J. Gen. Microbiol. 111, 1-61 (1979).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J. struct. boil. 116, 71-76 (1996).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. J. Struct. Biol. 144, 114-122 (2003).
- Kimori, Y. Morphological image processing for quantitative shape analysis of biomedical structures: effective contrast enhancement. J. Sync. Rad. 20, 848-853 (2013).
- Jun, S., Zhao, G., Ning, J., Ga Gibson,, Watkins, S. C., Zhang, P. Correlative microscopy for 3D structural analysis of dynamic interactions. J. Visual. Exper. 76, e50386(2013).
- Murata, K., Esaki, M., Ogura, T., Arai, S., Yamamoto, Y., Tanaka, N. Whole-cell imaging of the budding yeast Saccharomyces cerevisiae by high-voltage scanning transmission electron tomography. Ultramicros. 146, 39-45 (2014).
- Kortje, K. H., Paulus, U., Ibsch, M., Rahmann, H. Imaging of thick sections of nervous tissue with energy-filtering transmission electron microscopy. J. Microsc. 183, 89-101 (1996).
- Bouwer, J. C., et al. Automated most-probable loss tomography of thick selectively stained biological specimens with quantitative measurement of resolution improvement. J. Struct. Biol. 148 (3), 297-306 (2004).
- Egerton, R. F., Li, P., Malac, M. Radiation damage in the TEM and SEM. Micron. 35 (6), 399-409 (2004).
- Danev, R., Buijsse, B., Khoshouei, M., Plitzko, J. M., Baumeister, W. Volta potential phase plate for in-focus phase contrast transmission electron microscopy. Proc. Nat. Acad. Sci. USA. 111 (44), 15635-15640 (2014).
- Murata, K., et al. Zernike phase contrast cryo-electron microscopy and tomography for structure determination at nanometer and subnanometer resolutions. Structure. 18 (8), 903-912 (2010).
- Wolf, S. G., Houben, L., Elbaum, M. Cryo-scanning transmission electron tomography of biological cells. Nat. Methods. 11 (4), 423-428 (2014).
- Ophus, C., et al. Efficient linear phase contrast in scanning transmission electron microscopy with matched illumination and detector interferometry. Nat. Comm. 7, 1-7 (2016).
- Schertel, A., et al. Cryo FIB-SEM: Volume imaging of cellular ultrastructure in native frozen specimens. J. Struct. Biol. 184 (2), 355-360 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten