Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Replikation von bestellt, Nonredundant Bibliothek von Pseudomonas Aeruginosa Stamm PA14 Transposon Insertion Mutanten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Pseudomonas Aeruginosa Infektion verursacht erheblichen Morbidität in gefährdeten Gastgeber. Die nonredundant Transposon Einfügung mutierten Bibliothek von p. Aeruginosa Belastungen PA14, als PA14NR festgelegt, vereinfacht die Analyse der Gen-Funktionalität in zahlreichen Prozessen. Hier ist ein Protokoll, qualitativ hochwertige Kopien der PA14NR Set mutierten Bibliothek zu generieren.
Zusammenfassung
Pseudomonas Aeruginosa ist ein phänotypisch und genotypisiert vielfältig und anpassbar Gramnegative Bakterium in der menschlichen Umgebung allgegenwärtig. P. Aeruginosa ist in der Lage, Biofilme bilden, Antibiotika-Resistenz zu entwickeln, produzieren Virulenzfaktoren und schnell entwickeln im Verlauf einer chronischen Infektion. So können p. Aeruginosa führen, wodurch erhebliche Morbidität in bestimmten Patientengruppen sowohl akute und chronische, zur Behandlung von Infektionen, schwierig. P. Aeruginosa Sorte PA14 ist eine menschliche klinische Isolat mit einer erhaltenen Genom-Struktur, die eine Vielzahl von Säugetieren und nonvertebrate Gastgeber PA14 eine attraktive Belastung für das Studium dieser Erreger infiziert. Im Jahr 2006 wurde eine nonredundant Transposon Einfügung mutierten Bibliothek mit 5.459 Mutanten entsprechend 4.596 vorhergesagten PA14 Gene generiert. Seitdem hat die Verteilung der PA14 Bibliothek die Forschungsgemeinschaft zum besseren Verständnis der Funktion einzelner Gene und komplexe Pfade von p. Aeruginosaerlaubt. Erhaltung der Integrität der Bibliothek durch den Replikationsprozess erfordert ordnungsgemäße Handhabung und präzise Techniken. Zu diesem Zweck stellt diese Handschrift Protokolle, die im Detail beschreiben die Schritte in Bibliothek Replikation, Bibliothek-Qualitätskontrolle und ordnungsgemäße Lagerung der einzelnen Mutanten beteiligt.
Einleitung
Pseudomonas Aeruginosa ist ein phänotypisch und genotypisiert vielfältig und anpassbar gramnegatives Bakterium in Boden, Wasser, sowie die meisten menschlichen Umgebungen und Mikroflora der Haut vorhanden. Im Vergleich zu vielen Bakterienarten, hat p. Aeruginosa eine relativ große Genom von 5,5-7 Mbp mit hohen G + C-Gehalt (65-67 %). Darüber hinaus ein erheblicher Teil seiner Gene metabolische Anpassungsfähigkeit beteiligt sind und sind Teil des regulatorischen Netzwerken ermöglicht große Flexibilität bei der Reaktion auf Umweltstress1. P. Aeruginosa drückt eine Fülle von Virulenzfaktoren, Neigung, Form Biofilme Exponate besitzt die Fähigkeit, Antworten durch mehrere Quorum-sensing-Wege zu koordinieren und zeigt eine bemerkenswerte Fähigkeit, Antibiotika-Resistenz zu entwickeln und Toleranz2,3,4,5,6,7,8. Diese Attribute stellen große Herausforderungen für die Behandlung von Infektionen durch p. Aeruginosa.
Chronische p. Aeruginosa Infektion können bei vielen Krankheitszuständen auftreten. Cystische Fibrose (CF), eine genetische Krankheit, die durch Mutation des Gens Cystic Fibrosis Transmembrane Leitwert Regulator (CFTR) verursacht wiederum eingedickten, infizierte Sekrete in den Atemwegen, progressive Bronchiektasen und letztlich zum Tod respiratorische Insuffizienz9. Erwachsenenalter sind die Mehrzahl der Patienten mit CF chronisch mit p. Aeruginosa, infiziert, eine Schlüsselrolle in der Morbidität und Mortalität dieser Krankheit10zugeordnet spielt. Darüber hinaus sind Patienten mit schweren Verbrennungen Verletzungen11, Tracheotomien12, Gelenkersatz13oder indwelling Katheter14 für p. Aeruginosa Infektion im Zusammenhang mit den Bakterien die Fähigkeit Form gefährdet Biofilme und Flucht zu Host Entzündungsreaktionen15. Kolonisierung weiter, auftritt ohne Konkurrenz, nachdem eine Multi-Antibiotika resistente oder tolerantere Bevölkerung durch Breitspektrum, sequentielle antimikrobielle Behandlung12,16,17 ausgewählt ist , 18. besseres Verständnis der Pathogenese von p. Aeruginosa haben erhebliche Auswirkungen auf zahlreiche Krankheitszustände.
Mehrere p. Aeruginosa klinische Isolaten, einschließlich Belastungen PAO1, PA103, PA14 und PAK, haben ausgiebig untersucht, um verschiedene Funktionen von p. Aeruginosa Pathogenese zu untersuchen. Stamm PA14 ist eine klinische Isolate, die gehört zu den häufigsten klonalen Gruppen weltweit19,20 und nicht ausgiebig im Labor passagiert. PA14is hoch virulenten in vertebrate Modelle der Infektion, mit einer bemerkenswerten Endotoxin Profil21, Pili Struktur22, Pathogenität Inseln23, Typ III-Sekretion System (SSTT), Zytotoxizität gegenüber Säugetieren Zellen24 und Profile in antibiotische Resistenz und Dauerhaftigkeit25. Darüber hinaus PA14 ist auch in zahlreichen Wirt-Pathogen-Modellsystemen hoch virulenten, einschließlich pflanzenblattes Infiltration Modelle26,27,Caenorhabditis Elegans Infektion Modelle28, 29, Insekt Modelle30,31, sowie Maus Lungenentzündung Modelle32,33 und Hautverbrennungen Modelle34.
Genomweite mutierte Bibliotheken sind Sammlungen von isogener Mutanten in nicht benötigten Gene, die sehr leistungsfähige Werkzeuge um die Biologie des Organismus zu verstehen, ermöglicht die Analyse der Genfunktion auf genomischer Ebene darstellen. Zwei in der Nähe von Sättigung Transposon Einfügung mutierten Bibliotheken gebaut in p. Aeruginosa gibt es derzeit für den Vertrieb. Die Einfügung Sehenswürdigkeiten der Transposons sind für beide Bibliotheken ermittelt worden. Diese sogenannten nonredundant Bibliotheken erleichtern genomweite Studien von Bakterienstämmen verringernd, erheblich Zeit und Kosten beteiligt Screening fußgelenkes zufällige Transposon Mutanten. P. Aeruginosa PAO1 Transposon mutierte Bibliothek, gebaut in der MPAO1 von Belastungen PAO1 mit Transposons ISPhoA/ hah zu isolieren und istLacZ/ hah35, wird kuratiert von Manoil Labor, Universität von Washington. Die Bibliothek besteht aus einer Sequenz verifiziert Sammlung von 9.437 Transposon-Mutanten, die große Genom deckt und enthält zwei Mutanten für die meisten Gene36. Informationen über die p. Aeruginosa PAO1 Transposon mutierten Bibliothek steht in der Öffentlichkeit, Internet zugänglichen Manoil Lab-Website unter http://www.gs.washington.edu/labs/manoil/libraryindex.htm zur Verfügung. P. Aeruginosa Stamm PA14 nonredundant Transposon Einfügung mutierten Bibliothek (PA14NR eingestellt) Baujahr Belastung PA14 mit Transposons MAR2xT7 und TnPhoA37 derzeit Department of Pediatrics verteilt wird am Massachusetts General Hospital. Das PA14NR-Set umfasst eine Sammlung von mehr als 5.800 Mutanten mit einzelnen Transposon Einfügungen in nicht benötigten Gene37. Details über den Bau des PA14NR-Sets sind in der Öffentlichkeit, Internet zugänglichen Website http://pa14.mgh.harvard.edu/cgi-bin/pa14/home.cgi?section=NR_LIB beschrieben, die enthält auch eine Vielzahl von Online-Such-Tools zur Erleichterung der Nutzung des PA14NR Satz.
Das ursprüngliche PA14NR Set umfasste 5.459 Mutanten, ausgewählt aus einer umfangreichen Bibliothek von ca. 34.000 zufällige Transposon Einfügung Mutanten, die 4.596 vorhergesagten PA14 Gene entspricht 77 % aller vorhergesagten PA14 Gene37entsprechen. Seit dem Bau der Bibliothek im Jahr 2006 neue Mutanten hinzugefügt wurden, und derzeit das PA14NR Set beinhaltet mehr als 5.800 Mutanten-38 , die etwa 4.600 PA14 Gene darstellen. Die Mehrheit der PA14 Transposon Mutanten wurden in der Wildtyp Hintergrund37generiert. Einzelheiten über jedes Mitglied der mutierten Bibliothek, einschließlich genetischen Hintergrund gibt es entweder durch die Online-Datenbank zu suchen, oder durch das Herunterladen der Nonredundant Bibliothek-Tabellenkalkulation, beide Funktionen auf der Website PA14 (http:// pa14.MGH.Harvard.edu/cgi-bin/pa14/Home.cgi). Die Mehrheit der Mutanten entstanden mit Hilfe der MAR2xT7 (MrT7) Transposon, mit einer kleinen Gruppe mit der TnPhoA (PhoA) Transposon37erstellt. Jede Transposon hat eine Antibiotika-Resistenz-Kassette, die mutierten Auswahl mit Gentamicin (MrT7) oder Kanamycin (PhoA) ermöglicht. Die PA14NR Gruppe von Mutanten in 63 96-Well-Platten gespeichert und enthält zwei zusätzliche 96-Well-Steuerelement, die Platten, die aus Wildtyp PA14 beimpft und nicht inokulierten Brunnen Zwischenspiele in einem vorgegebenen Muster. Die 96-Well-Plattenformat gepaart mit der Online-Recherche-Tools erheblich erleichtert die kundenspezifische Entwicklung von screening-Tests, mit denen Benutzer auf einfache Weise Gene mutierten Phänotypen zugeordnet. Die Online-Such-Tools erleichtern auch die Suche und Auswahl von zusätzlichen relevanten Mutanten für weitere Studien erforderlich.
Die PA14 und PAO1 Transposon mutierten Bibliotheken sind sehr wichtige globale Ressourcen für die wissenschaftliche Gemeinschaft, und sie ergänzen einander in überprüfen die Funktion der unbekannte Gene und Wege von dieser bakteriellen Erreger. Zufälligerweise hat seit dem Bau der PAO1 und PA14 Transposon Mutation Bibliotheken, voll-DNA-Sequenzierung Genomanalyse viele p. Aeruginosa Isolate gezeigt, dass PAO1 und PA14 zu verschiedenen wichtigen Subclades von P. Aeruginosa gehören Phylogenie7,39,40,41. Da klinische p. Aeruginosa Isolate werden gefunden verteilt die Stammesgeschichte, die Tatsache, dass PAO1 und PA14 zu verschiedenen p. Aeruginosa gehören Untergruppen steigert den Wert der beiden Transposon Mutation Bibliotheken für vergleichende Studien.
Publikationen, beschreibt die Konstruktion und screening von bakteriellen mutierten Bibliotheken, einschließlich p. Aeruginosa Bibliotheken35,sind37,42, leicht zugänglich in der Literatur. Sind jedoch nach bestem Wissen und gewissen, keine veröffentlichten Protokolle beschreibt Einzelheiten der Verfahren und Techniken, die für die Replikation, Wartung und Validierung von bakteriellen mutierten Bibliotheken verfügbar.
Die in dieser Publikation beschriebenen Methodik beschreibt eine Reihe von drei Protokollen, die die Nutzung zu erleichtern und Wartung des PA14NR-Sets. Das erste Protokoll beschreibt Replikation der Bibliothek als Empfänger des PA14NR-Sets empfohlen. Das zweite Protokoll enthält Richtlinien für den Streifen, Anbau und Lagerung von einzelnen durch Mutation entstehende Variationen über das PA14NR-Set identifiziert. Das dritte Protokoll beschreibt qualitätskontrolltechniken, einschließlich PCR Verstärkung der Fragmente von Transposon Mutanten und anschließende Sequenzierung, mutierte Identität zu bestätigen. Dieser Satz von Protokollen kann auch für die Replikation und Wartung anderer Bakterien mutierten Bibliotheken oder Sammlungen angepasst werden. Die Replikation von bakteriellen mutierten Bibliotheken oder Sammlungen wird dringend empfohlen, zur Erhaltung der Integrität der "master-Kopie" (Originalversion erhalten). Replikation von mehreren Kopien der PA14NR-Set für die Routinelabor minimiert die Wahrscheinlichkeit der interwell Kontamination von der master-Kopie.
Protokoll
Vorsicht: Verwenden Sie standard BSL-2 Sicherheitsmaßnahmen beim Umgang mit p. Aeruginosa, ein menschlicher Erreger. Wenn Sie ein immungeschwächten Individuum sind oder irgendwelche medizinischen Zustand, der Ihre Anfälligkeit für bakterielle Infektionen erhöht, besondere Vorsicht bei der Arbeit mit P. Aeruginosa. Das Biosafety-Büro in Ihrer Institution zu konsultieren und Einholen der Genehmigung von Ihrem Arzt vor dem Arbeiten mit der PA14 NR eingestellt oder mutierten Bibliotheken von bakteriellen Krankheitserregern.

Abbildung 1: Übersicht über Protokoll I: Replikation von den PA14NR festgelegt. Tag 1: Gefrorene mutierte Kulturen aus "master-Kopie" des PA14NR-Sets in LB-Agar-Medien zu replizieren und wachsen durch Mutation entstehende Variationen über Nacht bei 37 ° C. 2.Tag: Transfer mutierte Wachstum von LB-Agar-Medien zum tiefen auch Blöcke mit LB flüssige Brühe, wachsen über Nacht bei 37 ° C mit schütteln bei 950 u/min. 3.Tag: Über Nacht LB Kulturen mit Glycerin mischen, dann transfer zum Reiseziel 96-Well Platten für die langfristige Lagerung. Platz 96-Well-Platten flach in-80 ° C Gefrierschrank. Bitte klicken Sie hier für eine größere Version dieser Figur.
Abbildung 2: Empfohlene Setup. Sterilität und reibungslosen Arbeitsablauf sollte durch den Einsatz von geeigneten Vorsichtsmaßnahmen einzuhalten. Bitte klicken Sie hier für eine größere Version dieser Figur.
1. Protokoll I: Replikation von den PA14NR festgelegt
Hinweis: Replikation der Bibliothek kann erreicht werden, indem man das PA14NR-Set in vier Untergruppen von sechzehn Platten jede, die in vier aufeinander folgenden Wochen bearbeitet werden können. Erstellung von 1 bis 6 Kopien hält sich für den wöchentlichen Workflow in Tabelle 1 beschrieben, während der Erstellung von mehr als 6 Kopien den wöchentlichen Workflow in Tabelle 2 aufgeführten folgt. Um 12 Kopien des PA14NR-Sets zu generieren, impfen die gleiche PA14NR Teilmenge in Flüssigmedien LB am 2. Tag und wieder am Tag3 (aus dem gleichen Satz von Agarplatten repliziert an Tag1) und Fahrt über Nacht mutierte Kulturen in Kopie Platten am Tag3 und Tag 4 bzw..
| Tag 0 | Tag1 | Tag2 | Tag3 |
| Prep Tag | Wachstum von PA14NR Set Mutanten auf LB-Agar-Medien | Wachstum von PA14NR Set Mutanten in Flüssigmedien LB | Übertragung von PA14NR Set mutierten Kulturen zum Ziel Platten |
Tabelle 1: Zeitplan für die Replikation von 1-6 Kopien des PA14NR-Sets. Replikation von einer kleinen Anzahl von Kopien kann eine wöchentliche Workflow einhalten.
| Tag 0 | Tag1 | Tag2 | Tag3 | Tag 4 |
| Prep Tag | Wachstum von PA14NR Set Mutanten auf LB-agar | PA14NR Set Mutanten in LB Flüssigmedien wachsen | Übertragung von Tag3 mutierte Kulturen, 1. Satz 6 Kopien des PA14NR-Sets zu generieren | Übertragung von Tag 4 mutierten Kulturen, 2. Satz von 6 Kopien des PA14NR-Sets zu generieren |
| Wachstum von PA14NR Set Mutanten in Flüssigmedien LB |
Tabelle 2: Zeitplan für die Replikation von bis zu 12 Kopien des PA14NR-Sets. Replikation von einer größeren Anzahl von Kopien benötigen Schichtung innerhalb eines wöchentlichen Workflows.
- Tag 1: Wachstum PA14NR Set Master-Kopie auf LB-Agar-Platten
- Tragen Sie Handschuhe, Kittel und Maske, die das PA14NR-Set zu behandeln.
- Klar, Bank und wischen Sie die Oberfläche mit 70 % Ethanol.
- LB-Agar-Medien-43 vorzubereiten und Sterilisation im Autoklaven für kalte Medien 25 min. 55 ° c in Wasser Baden und fügen entweder 15 µg/mL Gentamicin für Mutanten mit MAR2xT7 Transposon Insertionen oder 200 µg/mL Kanamycin für Mutanten mit TnPhoA Einfügungen.
- Gießen Sie geschmolzenes LB-Agar in rechteckigen Platten mit ca. 60 mL Medien pro Platte. Trockenplatten in eine sterile Haube für ca. 1 h vor dem verwenden. Stellen Sie sicher, es gibt kein Kondenswasser auf der Oberfläche des Nährbodens. Platten bei 4 ° C zu speichern, falls erforderlich.
- Einen sterilen Bereich mit dem Bunsenbrenner auf Ethanol-wischte Benchtop erstellen und Einrichten von Containern mit geeigneten Lösungen für Replikator Pin Sterilisation (siehe Schritt 1.1.9). Anwendung öffnen Kunststoffbehälter (5,25" L x 4,25" W x 1,75 "H ungefähre Größe) für Replikator Pin Sterilisation. Autoklaven Kunststoffbehälter vor Gebrauch.
Hinweis: Die Hitze der Flamme Bunsenbrenner erstellt eine Konvektionsströmung, die den Raum über der Flamme erhitzt und hebt alle Partikel in der Luft oben und Weg von der kühlere Luft unter den Arbeitsbereich steril zu halten. - Entfernen Sie maximal vier PA14NR Set master Platten aus dem-80 ° C Gefrierschrank (zur Vermeidung von unnötigen Auftauen) zu, und legen Sie sie auf Trockeneis in 4 L Eis Pfanne.
- Treffen Sie die Meister 96-Well Platte aus Trockeneis repliziert werden und legen Sie es auf der Bank erlauben kurze Auftauen, die nur die Zeit nehmen für die Sterilisation der Replikator Stifte (ca. 3-4 min) (siehe Schritt 1.1.8). Markieren Sie die Position der A1 Koordinate auf der Agarplatte vor dem Stanzen. Richten Sie master Platte und Agarplatte mit der A1-Koordinate der beiden Platten in der oberen linken Ecke.
- Sterilisieren Sie den Replikator "Pins" anhand der nachfolgend beschriebenen Schritte. Tippen Sie auf Replikator leicht nach jedem Schritt, überschüssige Flüssigkeit zu entfernen.
- Tauchen Stifte in 250 mL von 0,3-0,5 % Natriumhypochlorit (10 % haushaltsbleiche) für 30 Sekunden. Minimieren Sie Kontakt mit Natriumhypochlorit-Lösung, wie es um zu der Pins zu Schäden führen kann. Stifte in 250 mL sterile DdH2O Tauchen oder sterilen Reinstwasser für 10 Sekunden, dann in 250 mL 70 % igem Ethanol für 30 Sekunden, dann in 250 mL 95 % igem Ethanol für 2 min.
- Flamme sterilisieren Replikator Stifte indem Replikator senkrecht zu den Bunsenbrenner-Flamme, Flamme langsam nähern, bis Ethanol zündet, dann sofort Flamme entziehen. Die Flamme wird einmal alle Ethanol brennt ab. Behalten Sie einen Deckel oder ähnliche Behälter Nähe brennenden Ethanol zu ersticken, wenn nötig.
Hinweis: Extreme Vorsicht beim Arbeiten mit Ethanol in der Nähe einer Flamme. Halten Sie Replikator nicht direkt über die Flamme. - Coole Stifte durch Festhalten auf einen unbenutzten sterilen rechteckigen Platte mit Agar LB Medien für 30 Sekunden.
- Kurz warm Aluminium Dichtplatte aus PA14NR Set Master mit der Hand vor dem abziehen. Tun Sie dies bei der Kühlung Replikator Stifte. Entfernen Sie Aluminium-Dichtung sorgfältig, um zu verhindern, dass die Dichtung Retusche der Plattenrandes. Benutzen Sie eine Pinzette, alle Reste von Aluminium Dichtung abziehen.
- Master-Platte, sanft drücken und schwingenden Replikator in alle vier Richtungen zu gewährleisten, dass Stifte Replikator Pins stecken Kontakt mit gefrorenen Bakterienkulturen in jedem der 96-Brunnen. Achten Sie besonders auf Brunnen befindet sich in der Außenkante der Platte durch Druck Replikator Stifte gegen gefrorenen Kulturen in den Vertiefungen am Rand der Platte.
- Sanft lege Replikator Stifte auf der Agarplatte. Eine leichte Kreisbewegung zu Form Mini-Rasen von ca. 4-5 mm für jede Mutante Replikator einziehen. Vermeiden Sie die Möglichkeit, die Mini-Rasen überlappen können, um Kreuzkontaminationen zu vermeiden.
- Dichtplatte PA14NR Set Meister mit einem neuen sterilen Aluminium-Siegel. Berühren Sie nicht die klebende Seite der Alu-Dichtung an jedem Punkt zur Vermeidung von Kontaminationen. Stellen Sie sicher, dass jede Vertiefung und die Kanten der Platte vollständig abgedichtet sind, mit Hilfe einer Platte Walze. Rückkehr-96-Well-Platte zu Trockeneis.
- Wiederholen Sie den Vorgang für jede master Platte.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach dem Umgang mit dem PA14NR Set.
- Übertragung auf 37 ° C Inkubator Agarplatten repliziert und inkubieren Sie über Nacht.
- Tag 2: Wachstum der PA14NR Set Master-Kopie in LB flüssige Brühe
- Bereiten Sie LB flüssige Brühe43 mit entweder 15 µg/mL Gentamicin für Mutanten mit MAR2xT7 Transposon Insertionen oder 200 µg/mL Kanamycin für Mutanten, die TnPhoA Einfügungen enthalten.
- Deaktivieren Sie Laminar-Flow-Haube der unnötige Geräte und schalten Sie Haube Gebläse für mindestens 10 Minuten vor Beginn der arbeiten. Wischen Sie Haube Oberflächen und Gegenstände, die in die Haube mit 70 % igem Ethanol gelegt.
- Füllen Sie 2 mL Tiefbrunnen Blöcke mit 525 µL LB flüssige Brühe mit geeigneten Antibiotika in Laminar-Flow-Haube mit einem elektronischen 50-1200 µL 12-Kanal-Pipette. Übertragen Sie anschließend Medien gefüllt Tiefbrunnen Blöcke auf Ethanol-wischte Benchtop. Verwenden Sie Tipps, solange sterile Bedingungen eingehalten werden.
- Tragen Sie Handschuhe, Kittel und Maske zu handhaben PA14NR festgelegt.
- Klar, Bank und wischen Sie die Oberfläche mit 70 % Ethanol.
- Bringen Sie Agarplatten mit über Nacht gewachsenen mutierte Stämme zum Tisch.
- Einen sterilen Bereich mit dem Bunsenbrenner auf Ethanol-wischte Benchtop erstellen und Einrichten von Containern mit geeigneten Lösungen für Replikator Pin Sterilisation (siehe nächster Schritt).
- Sterilisieren der Replikator "Pins" nach den unten beschriebenen Schritten. Tippen Sie auf Replikator leicht nach jedem Eintauchen, überschüssige Flüssigkeit zu entfernen.
- Tauchen Stifte in 250 mL von 0,3-0,5 % Natriumhypochlorit für 30 Sekunden. Minimieren Sie Replikator Stiftkontakt mit Natriumhypochlorit-Lösung, wie es um zu der Pins zu Schäden führen kann. Dann tauchen Stifte in 250 mL sterile DdH2O oder Reinstwasser für 10 Sekunden, dann in 250 mL 70 % igem Ethanol für 30 Sekunden, dann in 250 mL 95 % Ethanol 2 min. lang.
- Flamme sterilisieren Replikator Stifte, dann cool Stifte durch Festhalten in ungenutzten rechteckigen Platte mit Agar LB Medien für 30 Sekunden.
Hinweis: Verwenden Sie äußerste Vorsicht beim Arbeiten mit Ethanol in der Nähe einer Flamme (siehe 1.1.8).
- Sanft Replikator Stifte auf Agarplatte mit mutierten Wachstum und kontrollieren, ob Stifte in Kontakt mit allen 96 Mutanten auf Agarplatte, dann tauchen Stifte in Tiefbrunnen mit LB flüssige Brühe. Berühren Sie die Seiten der Brunnen mit den Stiften.
- Siegel der Tiefbrunnen Block mit einer sterilen atmungsaktive Abdichtung Membran. Verwenden Sie eine Platte Walze, um sicherzustellen, dass jeder einzelne richtig gut abgedichtet ist.
- Wiederholen Sie den Vorgang für jede nährbodenplatte.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach dem Umgang mit dem PA14NR Set.
- Wachsen Sie beimpfte flüssige Kulturen für 15-16 h bei 37° C bei 950 u/min mit einem high-Speed-Shaker, falls vorhanden.
Hinweis: Wenn ein high-Speed-Shaker nicht verfügbar ist, ist Inkubation von Tiefbrunnen Blöcke im Shaker bei 250-300 u/min möglich. Allerdings gibt es eine größere Chance auf kleine Kolonie Varianten (WBFS)25 unter niedrigen sauerstoffhaushalt entstehen. Daher empfiehlt es sich sehr zu Inkubation Zeiten unter 15-16 Uhr, beim Anbau von Kulturen in Tiefbrunnen Blöcke mit niedrigeren Geschwindigkeiten Shaker. Unerwünschte Verbreitung von WBFS in mutierten Brunnen kann Mutante Phänotypen ändern, wenn die Bibliothek verwenden, um genetische Bildschirme durchführen.
Bestimmte Brunnen im PA14NR-Set enthalten langsam wachsende/nicht-wachsende Mutanten, Klonen, fehlt oder nicht inokulierten Medien enthalten. Die Lage dieser Bohrlöcher zu verzeichnen und finden Sie in der Datei PA14NR gesetzt Wells Zusatzinformationen mit dieser Veröffentlichung.
- 3. Tag: Transfer von PA14NR eingestellt über Nacht Kulturen in Ziel Platten
- Laminar-Flow-Haube klar und schalten Haube Gebläse für ein Minimum von 10 min vor Beginn der arbeiten. Wischen Sie Haube Oberflächen und Gegenstände, die in die Haube mit 70 % igem Ethanol gelegt.
- Wasserfeste Aufkleber zu drucken (siehe ergänzende PA14NR Set Etiketten-Datei für Vorlage). Entfernen Sie 96-Well-Platten aus Kunststoff wickeln in einer Laminar-Flow-Kapuze. Peel-off Platte beschriften und legen Sie sie entlang der Plattenrand, A1, H1 Brunnen der Ziel-Platte am nächsten. Leicht heben Sie den Deckel von der Ziel-Platte und platzieren Sie die Beschriftung am unteren Rand, Beschriftung anzuzeigen, wenn Teller mit Deckel bedeckt ist.
- Bereiten Sie 3,5 L 60 % Glycerin (V/V) und Sterilisieren Sie 20 min im Autoklaven zu.
- Tragen Sie Handschuhe, Kittel und Maske, die das PA14NR-Set zu behandeln. Entfernen Sie Tiefbrunnen Blöcke aus high-Speed-Shaker oder regelmäßige Shaker.
- Transfer Tiefbrunnen Blöcke zum sterilen Laminar-Flow-Haube und atmungsaktive Abdichtung Membrane entnehmen. Mit Alu-Dichtung ersetzen. Spin-Down die Tiefbrunnen-Blöcke bei sehr niedriger Drehzahl um Kondensation zu sammeln (30 Sekunden bei 50-150 X g, dann verlangsamen quicky mit Zentrifuge Bremse auf).
- Rücktransfer Tiefbrunnen Blöcke zum sterilen Kapuze und mit einer elektronischen 50-1200 µL 12-Kanal-Pipette und sterile gefilterte Tipps hinzufügen 525 µL Glycerin/LB flüssige Brühe Mix (gleiche Teile LB flüssige Brühe und 60 % Glycerin-Lösung) in jede Vertiefung. Mischen Sie, indem Sie sanft pipettieren 300 µL oben und unten 3 Mal mit elektronische Pipette. Berühren Sie Tipps auf Seite der Brunnen vor dem Auswerfen Tipps zum Nachtropfen zu verhindern. Werfen Sie Tipps zu, und fahren Sie mit der nächsten Zeile bis mit ganzen Tiefbrunnen Block fertig.
- Mit dem 12-Kanal elektronische repetitive Pipette und gefilterten Tipps interwell Kontamination während der Aliquotierung zu verhindern, ziehen Sie 900 µL der mutierten Kultur und verzichten Sie 150 µL in jedes Ziel 96-Well Platten 6 Kopien der Bibliothek Platte generieren zu. Verhindern Sie tropft durch Berühren der Wand der Brunnen mit Tipps vor Beginn der Übertragung der Kultur in Ziel-Platten. Wenn keine Wiederholung Pipette zur Verfügung steht, verwenden einer Mehrkanal-Pipette auf um 150 µL in jedem der sechs 96-Well-Platten, mit Hilfe der Technik um Nachtropfen zu verhindern die oben beschriebenen zu verzichten.
- Verwenden Sie sterile Aluminium Dichtungen die Abdeckplatten und verwenden eine Platte Walze komplett Dichtung Plattenkanten und alle Brunnen. Achten Sie darauf, keine Label-ID mit dem Aluminium-Siegel zu decken. Schütteln Sie die Platten nicht, wie Kultur auf Seiten der Brunnen oder Aluminium Dichtung Spritzen kann.
- Entfernen Sie versiegelte Platten von Kapuze und Ort Platten auf eine flache, gleichmäßige Oberfläche in den-80 ° C Gefrierschrank.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach dem Umgang mit der Bibliothek.
- Durchführen Sie Qualitätskontrollen, nach der Bibliothek Replikation und nach der Benutzung der Bibliothek, genetische Bildschirme durchzuführen (siehe Protokoll III).
2. Protokoll II: Handhabung und Lagerung von einzelnen Mutanten aus dem PA14NR-Satz
-
Tag 1: Streak Mutante von Interesse
- Geben Sie den Standort des mutierten des Interesses durch PA14NR Set Link http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS oder mit Hilfe der Nonredundant Library.xls-Datei aus dem Link http:// heruntergeladen pa14.MGH.Harvard.edu/cgi-bin/pa14/Downloads.cgi). Notieren Sie sich das Antibiotikum musste jeder bestimmten Mutante (Gentamicin oder Kanamycin) auswählen.
- LB-Agar-Medien vorbereiten, Sterilisation im Autoklaven für 20-25 min und auf 55 ° C im Wasserbad abkühlen lassen. Fügen Sie 15 µg/mL Gentamicin für Mutanten mit MAR2xT7 Transposon Einfügungen und 200 µg/mL Kanamycin für Mutanten, die TnPhoA Transposon Einfügungen enthalten. Gießen Sie LB-Agar-Medien auf Platten (Runde oder rechteckige Platten sind ausreichend). Trockenplatten in steriler Haube für ca. 30 min bis 1 h vor dem verwenden.
- Autoklav alle Kunststoffprodukte und unsteril Lieferungen vor Gebrauch.
- Tragen Sie Handschuhe, Kittel und Maske, die das PA14NR-Set zu behandeln. Klar, Bank und wischen Sie die Oberfläche mit 70 % Ethanol vor der Arbeit mit dem PA14NR Set.
- Erstellen Sie einen sterilen Bereich mit dem Bunsenbrenner.
- Entfernen PA14NR Set 96-Well-Platte mit Mutant Interesse von-80 ° C Gefrierschrank legen Sie es auf Trockeneis, Trockeneis-Container auf der Bank nehmen und kurz Platz 96-Well-Platte auf Bank ermöglichen leichte Auftauen (ca. 1-2 min.).
Hinweis: Notieren Sie sich alle PA14NR Set 96-Well-Platten zugegriffen, um einzelne Streifen Mutanten, wie mehr Zugang zu Bibliothek Platten mit ein größeres Risiko für interwell Kontamination korreliert ist. - Warmen Aluminium Siegel mit der Hand vor dem abziehen, vorsichtig zu verhindern, dass die Dichtung Retusche der Plattenrandes. Benutzen Sie eine Pinzette, alle Reste von Aluminium Dichtung abziehen.
- Suchen Sie die Mutante von Interesse auf der 96-Well-Platte. Verwenden Sie entweder sterile Holzstab oder sterilen PIPETTENSPITZE, wählen Sie eine kleine Menge der gefrorenen Kultur von der Person, die gut mit der Mutante von Interesse.
- Streak eingefroren Kultur auf Agarplatte für einzelne Mutante Kolonien wie folgt: sanft verteilt die Bakterien über einen Teil der Platte erstelle ich Streifen 1, mit einer frischen, sterilen hölzernen Stock oder Pipette Spitze, ziehen Sie durch Streifen 1 und ein zweiter Abschnitt von den Bakterien verteilt die Platte, um Streifen 2 zu erstellen. Mit einer dritten sterile hölzernen Stock oder Pipette Spitze, ziehen Sie durch Streifen 2 und der letzte Abschnitt der Platte, Ader 3 erstellen die Bakterien verteilt.
- Dichtplatte Quelle mit einem neuen sterilen Aluminium-Siegel. Berühren Sie nicht die klebende Seite der Alu-Dichtung an jedem Punkt zur Vermeidung von Kontaminationen. Stellen Sie sicher, dass jeder Brunnen und die Kanten der Platte vollständig abgedichtet sind, mit Hilfe einer Platte Walze. Rückkehr-96-Well-Platte Trockeneis und dann die-80 ° C Gefrierschrank.
- Über Nacht inkubieren Sie nährbodenplatte im Inkubator 37 ° C.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach dem Umgang mit der Bibliothek.
-
Tag 2: Wachstum der Mutante von Interesse in LB flüssige Brühe
- Bereiten Sie flüssige LB-Brühe mit 15 µg/mL Gentamicin oder 200 µg/mL Kanamycin nach Transposon einsetzen.
- Tragen Sie Handschuhe, Kittel und Maske, p. Aeruginosazu behandeln.
- Klar, Bank und wischen Sie die Oberfläche mit 70 % Ethanol. Erstellen Sie einen sterilen Bereich mit dem Bunsenbrenner.
- Übertragen Sie 3-5 mL LB-Brühe mit entsprechenden Antibiotikum in sterilen Kultur Rohr mit Kappe.
- Mit einem sterilen Applikator oder einer sterilen PIPETTENSPITZE, wählen Sie eine einzelne Kolonie der Mutante und in LB-Medium zu impfen.
- Über Nacht inkubieren Sie LB flüssige Kulturen bei 37 ° C in einem Shaker bei 225-250 u/min.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach dem Umgang mit p. Aeruginosa.
-
3. Tag: Store-Mutante von Interesse an-80 ° C Gefrierschrank
- Beschriften Sie Cryoröhrchen mit mutierten Namen, Antibiotika, LB Brühe und Datum der Speicherung hinzugefügt.
- Bereiten Sie 500 mL von 50 % (V/V) Glycerin und im Autoklaven sterilisieren.
- Tragen Sie Handschuhe, Kittel und Maske, p. Aeruginosazu behandeln.
- Klar, Bank und/oder laminar Flut Haube und wischen Sie die Oberfläche mit 70 % Ethanol.
- Röhrchen mit mutierten Kultur von Shaker zu entfernen.
- Bereiten Sie einen kleinen Behälter mit Trockeneis.
- Verwenden einer Laminar-Flow-Haube oder erstellen Sie einen sterilen Bereich auf Ethanol-wischte Benchtop mit Bunsenbrenner.
- Die beschrifteten Cryoröhrchen mit sterilen Bedingungen gleiche Mengen von Bakterienkultur und 50 % Glycerin hinzu, und vorsichtig mischen mit Pipette (Endvolumen 1-2 mL/Fläschchen abhängig von der Größe der Cryovials verwendet). Trockeneis, quick-freeze setzen Sie Cryoröhrchen auf.
- Beschriftete Feld bei-80 ° C Gefrierschrank entgegenbringen Sie Cryoröhrchen.
- Wischen Sie alle Arbeitsflächen mit 70 % Ethanol nach der Handhabung p. Aeruginosa.
3. Protokoll III: Qualitätskontrolle von PA14NR Set
- Wählen Sie zufällige Gruppe von Mutanten aus neu replizierten Platten zur Erkennung möglichen interwell Kontamination (Prüfung von 30-40 Mutanten wird empfohlen).
Hinweis: In Fällen, wo es notwendig ist, um die Identität des eine Mutante, die für die Charakterisierung eines bestimmten Gens verwendet wird bestätigen, es wird empfohlen, durchführen PCR Verstärkung mit Gen-spezifische Primer entwickelt für die bekannten Sequenz des Gens, das enthält die Transposon einsetzen. Obwohl schwieriger, die Vorteile der Verwendung von willkürlichen gehören PCR Primer statt gen-spezifische PCR Primer bei Amplyfing DNA von Transposon Mutanten Fragmente einfache groß angelegte mutierten Bestätigung und die Fähigkeit, das Vorhandensein von Potenzial erkennen Verunreinigungen. Zur Qualitätskontrolle ist es nicht notwendig, hohe Datenqualität PCR Sequenzierung für alle nach dem Zufallsprinzip durchstreifen Mutanten zu erhalten, solange eine ausreichende Anzahl von Mutanten untersucht werden, um die Fehlerquote zu beurteilen. - Folgen Sie "Protokoll II" zu Streifen und mutierte Stämme wachsen.
- Autoklav alle Kunststoffprodukte und unsteril Lieferungen vor Gebrauch.
- Isolieren Sie genomischen DNA, indem Sie bevorzugte Methode. Für die Analyse in dieser Arbeit beschrieben wurde ein genomischer DNA-Isolierung-Kit des Herstellers-Protokolle verwendet. Mehrere mutierte Stämme können gleichzeitig analysiert werden.
- Messen Sie genomische DNA-Konzentration mit einer Microvolume Spektralphotometer. Passen Sie die genomische DNA-Konzentration um ca. 100 ng/µL.
- Verwenden Sie genomischen DNA als Vorlage, "PCR1 Reaktion" laufen, wie in Schritt 1 der Tabelle3, Generierung von Arb1 PCR-Fragmente (Abbildung 3) beschrieben. Primer für diesen Schritt sind in Tabelle 4 aufgeführt.
- Hinzufügen 0,5 µL 10 x Ladepuffer bis 5 µL der PCR1 Reaktion, laden zu einem 1,5-2 % Agarose-Gel und das Gel bei 80-150 V laufen.
Hinweis: Ein bestimmtes Fragment-Länge oder ein bestimmtes Fragment-Bereich ist nicht zu erwarten, und mehr als eine Band möglicherweise vorhanden, da die willkürliche Grundierung mit mehreren Speicherorten binden kann. - Verwenden Sie DNA von PCR1 Reaktion als Vorlage "PCR2 Reaktion" laufen, wie in Schritt 2 von Tabelle3, Generierung von Arb2 PCR-Fragmente (Abbildung 3) beschrieben. Primer für diesen Schritt sind in Tabelle 4 aufgeführt.
- Hinzufügen 0,5 µL 10 x Ladepuffer bis 5 µL PCR2 Reaktion, laden zu einem 1,5-2 % Agarose-Gel und das Gel bei 80-150 V laufen.
Hinweis: Ein bestimmtes Fragment-Länge oder ein bestimmtes Fragment-Bereich ist nicht zu erwarten, und mehr als eine Band möglicherweise vorhanden, da die willkürliche Grundierung mit mehreren Speicherorten binden kann. - Senden Sie PCR2 Reaktion zusammen mit entsprechenden Transposon-spezifische Primer zur Sequenzierung.
- Analize Sequenzierung Ergebnisse ByBLASTing gegen das gesamte PA14 Genom über den BLAST-Link auf der Homepage der PA14NR Library (http://pa14.mgh.harvard.edu/cgi-bin/pa14/blast.cgi) oder Strahlen sie direkt gegen die spezifischen gen-Sequenz des zur Verfügung die Mutante von Interesse.
Hinweis: Informationen zu ausgewählten Mutanten finden Sie bei der Suche der Website PA14NR gesetzt (Suche http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS oder Download Nonredundant Library.xls Datei aus dem Link http:/ / pa14.mgh.harvard.edu/cgi-bin/pa14/downloads.cgi).
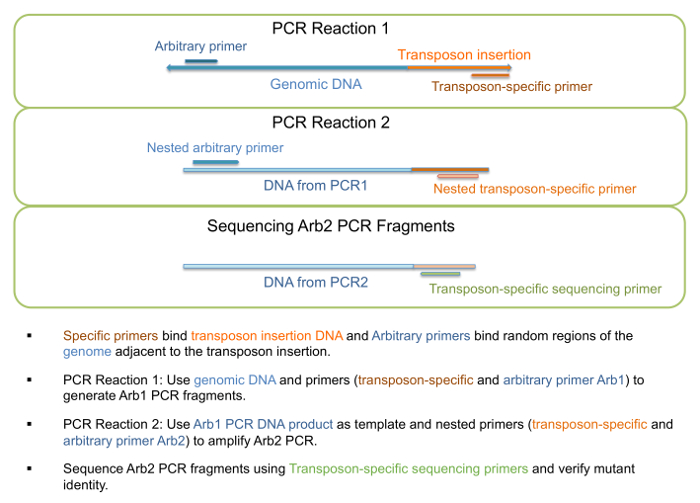
Abbildung 3: PCR Amplifikation und Sequenzierung von Transposon Einfügung Mutanten. Schematische Darstellung der Schritte in PCR-Amplifikation und Sequenzierung für mutierte Identitätsprüfung. Bitte klicken Sie hier für eine größere Version dieser Figur.
| PCR-Reaktion-Aufbau | |
| Schritt 1 | Schritt 2 |
| PCR1 Reaktion: | PCR2 Reaktion: |
| 23.25 µL Wasser (Molekulare Biologie Klasse) | 19.15 Uhr µL Wasser (Molekulare Biologie Klasse) |
| 3µL 10 X Taq Polymerase Puffer | 5 µL 10 x Taq Polymerase-Puffer |
| 0.5µL Taq Polymerase | 0,6 µL Taq Polymerase |
| 0,625 µL 20 µM Primer Arb1D (Tabelle 4) | 0,625 µL 20 µM Primer Arb2A (Tabelle 4) |
| 0,625 µL 20 µM Transposon-spezifische Primer (PMFLGM. GB-3a oder Tn5Ext) (Tabelle 4) | 0,625 mL 20 µM Transposon-spezifische Primer (PMFLGM. GB-2a oder Tn5Int2) (Tabelle 4) |
| 1 µL 10 mM dNTPs | 1 µL 10 mM dNTPs |
| 1 µL genomischer DNA, 100 ng | 5 µL PCR1 Reaktion |
| Endgültige Reaktionsvolumen 30 µL | Endgültige Reaktionsvolumen 30 µL |
| PCR-Reaktion-Einstellungen | |
| PCR1 Thermocycler Bedingungen: | PCR2 Thermocycler Bedingungen: |
| 95 ° C – 2 min | 95 ° C – 2 min |
| Wiederholen Sie 5 Zyklen: | Wiederholen Sie 30 Zyklen: |
| 95 ° C-30 s | 95 ° C-30 s |
| 30 ° C – 1 min | 54 ° C – 30 s |
| 72 ° C – 1 min | 72 ° C – 1,5 min |
| Wiederholen Sie 30 Zyklen: | 72 ° C – 10 min |
| 95 ° C-30 s | 4 ° C – Hold |
| 45 ° C – 30 s | |
| 72 ° C – 1 min | |
| 72 ° C – 10 min | |
| 4° C – Hold |
Tabelle 3: PCR Setup und Thermocycler Reaktionsbedingungen für willkürliche PCR verwendet. Willkürliche PCR-Reaktionen werden nacheinander ausgeführt, und Fragmente generiert während PCR1 Reaktion dienen als Vorlage PCR2 Reaktion. Spezifische Thermocycler Einstellungen werden für jeden Satz von Reaktionen verwendet.
| Primer-Name | Primer Sequenz |
| MAR2xT7 Transposon-spezifische Primer | |
| PMFLGM. GB-3a | TACAGTTTACGAACCGAACAGGC |
| PMFLGM. GB-2a | TGTCAACTGGGTTCGTGCCTTCATCCG |
| MAR2xT7 Transposon Sequenzierung Primer | |
| PMFLGM. GB-4a | GACCGAGATAGGGTTGAGTG |
| TNPhoA Transposon-spezifische Primer | |
| Tn5Ext | GAACGTTACCATGTTAGGAGGTC |
| Tn5Int2 | GGAGGTCACATGGAAGTCAGATCCTGG |
| TNPhoA Transposon Sequenzierung Primer | |
| Tn5Int | CGGGAAAGGTTCCGTTCAGGACGC |
| Willkürliche Primer | |
| ARB1D | GGCCAGGCCTGCAGATGATGNNNNNNNNNNGTAT |
| ARB2A | GGCCAGGCCTGCAGATGATG |
Tabelle 4: Liste der Primer in der Qualitätskontrolle verwendet Experimente. Primer verwendet für PCR-Amplifikation und Sequenzierung von Transposon Einfügung Mutanten, mutierte Identität zu bestätigen.
Ergebnisse
Zwölf neue Kopien des PA14NR-Sets wurden wiederholt mit Protokoll ich und eine Qualitätskontrolle Beurteilung der neuen Kopien erzeugt wurde mit Protokoll III durchgeführt.
PA14NR Set mutierte Platten zusammen mit Steuerplatten, bestehen aus Wildtyp PA14 beimpft und nicht inokulierten Brunnen in einem vorgegebenen Muster (Abbildung 4A) eingelagert, wurden wiederholt nach Methology beschrieben im ...
Diskussion
P. aeruginosa PA14NR ist eine wertvolle Ressource für die wissenschaftliche Gemeinschaft. Nach dem März 2017 Dataset aus Clarivate Analytics Essential Science Indicators Datenbank, Liberati Et al. (2006) 37, beschreibt den Bau des PA14NR-Sets, rangiert in den Top 1 % der Mikrobiologie Publikationen. Google Scholar berichtet über 600 Zitate von Liberati Et al. (2006) original-Manuskript ab August 2017. Die Bibliothek hat eine wichtige Rolle bei der Aufklärung...
Offenlegungen
Die Autoren Berichten keine finanziellen Interessenkonflikte. Eliana Drenkard und Frederick Ausubel teilgenommen bei der Erstellung die PA14 nonredundant Transposon mutierte Bibliothek. Bryan Hurley und Lael Yonker derzeit Haus und verteilen die mutierte Bibliothek als Teil der Abteilung für Pädiatrie am Massachusetts General Hospital.
Danksagungen
Wir möchten danken Lisa Philpotts der MGH Treadwell virtuelle Bibliothek für ihre Führung in der Datenbanksuche. Diese Arbeit wurde unterstützt von der Cystic Fibrosis Foundation (YONKER16G0 und HURLEY16G0) und NIH NIAID (BPH und ADE: R01 A1095338).
Materialien
| Name | Company | Catalog Number | Comments |
| Materials for Library Replication | |||
| Sterile 96-well Tissue-culture treated, case of 50 | Corning Life Sciences | 353072 | via Fisher Scientific |
| Sterile 96 Well Clear V-Bottom 2000μL Deep Well Plates, case of 25 | Corning Life Sciences | 3960 | via Fisher Scientific |
| Nunc OmniTray (rectangular plates), case of 60 | Thermo Scientific Rochester | 242811 | via Fisher Scientific |
| Rectangular Ice Pan, Midi (4L) | Corning Life Sciences | 432104 | via Fisher Scientific |
| Secure-Gard Cone Mask, case of 300 | Cardinal Health | AT7509 | via Fisher Scientific |
| AluminaSeal, pack of 100 | Diversified Biotech | ALUM-100 | via Fisher Scientific |
| Breathe-Easy membrane, pack of 100 | Diversified Biotech | BEM-1 | via Sigma-Aldrich |
| Sterile, individually wrapped, 50mL Solution Trough/Reagent Reservoir, case of 100 | Sorenson | S50100 | via Westnet Incorporated |
| Plate roller | VWR | 60941-118 | via VWR |
| Cryo Laser Labels - CRYOLAZRTAG 2.64" x 0.277", pack of 16 sheets | GA International | RCL-11T1-WH | via Labtag.com (template for printing also available from Labtag.com) |
| 96-well replicator | V & P Scientific, Inc. | Custom 407C, 3.18mm pin diameter, 57mm long | via V & P Scientific, Inc. |
| Multitron Pro, 3mm Shaking incubator | Infors HT | l10003P | via Infors HT |
| Picus 12 Channel 50-1200μL Electronic Pipette | Sartorius | 735491PR | via Sartorius |
| Filter Tips 50-1200μL, pack of 960 | Biohit | 14-559-512 | via Fisher Scientific; use electronic multichannel-compatible tips |
| Dry Ice | User-specific vendor | ||
| Materials for Individual Mutant Storage | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Materials for Quality Control PCR | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| NanoDrop | Thermo Scientific | ND-2000 | via ThermoFisher |
| PCR Thermocycler | |||
| Omnistrips PCR Tubes with domed lids | Thermo Scientific | AB0404 | via Fisher Scientific |
| ART Barrier low-retention pipette tips (10 uL, 100 uL, 1000 uL) | Molecular BioProducts, Inc. | Z676543 (10 uL), Z676713 (100 uL), Z676802 (1000 uL) | via Sigma-Aldrich |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| MasterPure DNA Purification Kit | Epicentre | MCD85201 | via Epicentre Technologies Corp |
| GeneRuler 1 kb Plus DNA Ladder, ready-to-use | Thermo Scientific | SM1333 | via ThermoFisher |
| RediLoad Loading Buffer | Invitrogen | 750026 | via ThermoFisher |
| Chemicals | |||
| Chemicals for Library and Individual Mutant Storage | |||
| Glycerol MB Grade, 1L | Sigma Aldrich | G5516 | via Sigma-Aldrich |
| LB Broth | Per 1L dH2O: 10g tryptone, 5g yeast extract, 5g NaCl, 1ml 1N NaOH (Current Protocols in Molecular Biology. Wiley, 1994.) | ||
| Tryptone | Sigma Aldrich | T7293 | via Sigma-Aldrich |
| Yeast Extract | Sigma Aldrich | Y1625 | via Sigma-Aldrich |
| Sodium Chloride | Sigma Aldrich | S7653 | via Sigma-Aldrich |
| Sodium Hydroxide | Sigma Aldrich | S8045 | via Sigma-Aldrich |
| LB agar | See preparation above, add 15g Bacto Agar | ||
| Bacto Agar | Sigma Aldrich | A5306 | via Sigma-Aldrich |
| Gentamicin sulfate, 10g | BioReagent | 1405-41-0 | via Sigma-Aldrich |
| Kanamycin sulfate | Gibco | 11815024 | via ThermoFisher |
| Ethanol, 190 proof | Decon | 04-355-221 | via Fisher Scientific |
| Chemicals for Quality Control PCR | |||
| Primers | User-preferred vendor | See primers listed in Table 3 | |
| Corning cellgro Molecular Biology Grade Water | Corning | 46000CV | via Fisher Scientific |
| Taq Polymerase Buffer | Invitrogen | 10342020 | via ThermoFisher |
| Taq DNA Polymerase, recombinant | Invitrogen | 10342020 | via ThermoFisher |
| dNTPs | Invitrogen | 10297018 | via ThermoFisher |
| Agarose | Sigma | A9539 | via Sigma-Aldrich |
Referenzen
- Moradali, M. F., Ghods, S., Rehm, B. H. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Front Cell Infect Microbiol. 7, 39 (2017).
- Bleves, S., et al. Protein secretion systems in Pseudomonas aeruginosa: A wealth of pathogenic weapons. Int J Med Microbiol. 300 (8), 534-543 (2010).
- Breidenstein, E. B., de la Fuente-Nunez, C., Hancock, R. E. Pseudomonas aeruginosa: all roads lead to resistance. Trends Microbiol. 19 (8), 419-426 (2011).
- Flynn, K. M., et al. Evolution of ecological diversity in biofilms of Pseudomonas aeruginosa by altered cyclic diguanylate signaling. J Bacteriol. 198 (19), 2608-2618 (2016).
- Hazan, R., Maura, D., Que, Y. A., Rahme, L. G. Assessing Pseudomonas aeruginosa persister/antibiotic tolerant cells. Methods Mol Biol. 1149, 699-707 (2014).
- Klockgether, J., et al. Genome diversity of Pseudomonas aeruginosa PAO1 laboratory strains. J Bacteriol. 192 (4), 1113-1121 (2010).
- Mathee, K., et al. Dynamics of Pseudomonas aeruginosa genome evolution. Proc Natl Acad Sci U S A. 105 (8), 3100-3105 (2008).
- Taylor, P. K., Yeung, A. T., Hancock, R. E. Antibiotic resistance in Pseudomonas aeruginosa biofilms: towards the development of novel anti-biofilm therapies. J Biotechnol. 191, 121-130 (2014).
- Flume, P. A., Van Devanter, D. R. State of progress in treating cystic fibrosis respiratory disease. BMC Med. 10, 88 (2012).
- . Cystic Fibrosis Foundation Patient Registry 2015 Annual Data Report Available from: https://www.cff.org/Our-Research/CF-Patient-Registry/2015-Patient-Registry-Annual-Data-Report.pdf (2016)
- Church, D., Elsayed, S., Reid, O., Winston, B., Lindsay, R. Burn wound infections. Clin Microbiol Rev. 19 (2), 403-434 (2006).
- Sadikot, R. T., Blackwell, T. S., Christman, J., Prince, A. Pathogen-host interactions in Pseudomonas aeruginosa pneumonia. Am J Respir Crit Care Med. 171 (11), 1209-1223 (2005).
- Song, Z., et al. Prosthesis infections after orthopedic joint replacement: the possible role of bacterial biofilms. Orthop Rev (Pavia). 5 (2), 65-71 (2013).
- National Nosocomial Infections Surveillance, S. National Nosocomial Infections Surveillance (NNIS) System Report, data summary from January 1992 through June 2004, issued October 2004. Am J Infect Control. 32 (8), 470-485 (2004).
- Cohen, T. S., Parker, D., Prince, A. . Pseudomonas aeruginosa Host Immune Evasion. 7, 3-23 (2014).
- Fernandes, A., Dias, M. The microbiological profiles of infected prosthetic implants with an emphasis on the organisms which form biofilms. J Clin Diagn Res. 7 (2), 219-223 (2013).
- Khosravi, A. D., Ahmadi, F., Salmanzadeh, S., Dashtbozorg, A., Montazeri, E. A. Study of Bacteria Isolated from Orthopedic Implant Infections and their Antimicrobial Susceptibility Pattern. Res J of Microbiol. 4 (4), 6 (2009).
- Roemhild, R., Barbosa, C., Beardmore, R. E., Jansen, G., Schulenburg, H. Temporal variation in antibiotic environments slows down resistance evolution in pathogenic Pseudomonas aeruginosa. Evol Appl. 8 (10), 945-955 (2015).
- Fischer, S., et al. Intraclonal genome diversity of the major Pseudomonas aeruginosa clones C and PA14. Environ Microbiol Rep. 8 (2), 227-234 (2016).
- Wiehlmann, L., et al. Population structure of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 104 (19), 8101-8106 (2007).
- Lam, J. S., Taylor, V. L., Islam, S. T., Hao, Y., Kocincova, D. Genetic and functional diversity of Pseudomonas aeruginosa lipopolysaccharide. Front Microbiol. 2, 118 (2011).
- Choi, J. Y., et al. Identification of virulence genes in a pathogenic strain of Pseudomonas aeruginosa by representational difference analysis. J Bacteriol. 184 (4), 952-961 (2002).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proc Natl Acad Sci U S A. 101 (8), 2530-2535 (2004).
- Mikkelsen, H., McMullan, R., Filloux, A. The Pseudomonas aeruginosa reference strain PA14 displays increased virulence due to a mutation in ladS. PLoS One. 6 (12), e29113 (2011).
- Drenkard, E., Ausubel, F. M. Pseudomonas biofilm formation and antibiotic resistance are linked to phenotypic variation. Nature. 416 (6882), 740-743 (2002).
- Rahme, L. G., et al. Common virulence factors for bacterial pathogenicity in plants and animals. Science. 268 (5219), 1899-1902 (1995).
- Rahme, L. G., et al. Use of model plant hosts to identify Pseudomonas aeruginosa virulence factors. Proc Natl Acad Sci U S A. 94 (24), 13245-13250 (1997).
- Kirienko, N. V., Cezairliyan, B. O., Ausubel, F. M., Powell, J. R. Pseudomonas aeruginosa PA14 pathogenesis in Caenorhabditis elegans. Methods Mol Biol. 1149, 653-669 (2014).
- Mahajan-Miklos, S., Tan, M. W., Rahme, L. G., Ausubel, F. M. Molecular mechanisms of bacterial virulence elucidated using a Pseudomonas aeruginosa-Caenorhabditis elegans pathogenesis model. Cell. 96 (1), 47-56 (1999).
- Limmer, S., et al. Pseudomonas aeruginosa RhlR is required to neutralize the cellular immune response in a Drosophila melanogaster oral infection model. Proc Natl Acad Sci U S A. 108 (42), 17378-17383 (2011).
- Miyata, S., Casey, M., Frank, D. W., Ausubel, F. M., Drenkard, E. Use of the Galleria mellonella caterpillar as a model host to study the role of the type III secretion system in Pseudomonas aeruginosa pathogenesis. Infect Immun. 71 (5), 2404-2413 (2003).
- Coleman, F. T., et al. Hypersusceptibility of cystic fibrosis mice to chronic Pseudomonas aeruginosa oropharyngeal colonization and lung infection. Proc Natl Acad Sci U S A. 100 (4), 1949-1954 (2003).
- Pazos, M. A., et al. Pseudomonas aeruginosa ExoU augments neutrophil transepithelial migration. PLoS Pathog. 13 (8), e1006548 (2017).
- Maura, D., Hazan, R., Kitao, T., Ballok, A. E., Rahme, L. G. Evidence for direct control of virulence and defense gene circuits by the Pseudomonas aeruginosa quorum sensing regulator, MvfR. Sci Rep. 6, 34083 (2016).
- Jacobs, M. A., et al. Comprehensive transposon mutant library of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 100 (24), 14339-14344 (2003).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomonas aeruginosa PAO1. J Bacteriol. 194 (23), 6387-6389 (2012).
- Liberati, N. T., et al. An ordered, nonredundant library of Pseudomonas aeruginosa strain PA14 transposon insertion mutants. Proc Natl Acad Sci U S A. 103 (8), 2833-2838 (2006).
- Feinbaum, R. L., et al. Genome-wide identification of Pseudomonas aeruginosa virulence-related genes using a Caenorhabditis elegans infection model. PLoS Pathog. 8 (7), e1002813 (2012).
- Stewart, L., et al. Draft genomes of 12 host-adapted and environmental isolates of Pseudomonas aeruginosa and their positions in the core genome phylogeny. Pathog Dis. 71 (1), 20-25 (2014).
- Thrane, S. W., et al. The widespread multidrug-resistant serotype O12 Pseudomonas aeruginosa clone emerged through concomitant horizontal transfer of serotype antigen and antibiotic resistance gene clusters. MBio. 6 (5), e01396-e01315 (2015).
- van Belkum, A., et al. Phylogenetic Distribution of CRISPR-Cas Systems in Antibiotic-Resistant Pseudomonas aeruginosa. MBio. 6 (6), e01796-e01715 (2015).
- Lewenza, S., et al. Construction of a mini-Tn5-luxCDABE mutant library in Pseudomonas aeruginosa PAO1: a tool for identifying differentially regulated genes. Genome Res. 15 (4), 583-589 (2005).
- . . Current Protocols in Molecular Biology. , (1994).
- Breidenstein, E. B., Khaira, B. K., Wiegand, I., Overhage, J., Hancock, R. E. Complex ciprofloxacin resistome revealed by screening a Pseudomonas aeruginosa mutant library for altered susceptibility. Antimicrob Agents Chemother. 52 (12), 4486-4491 (2008).
- Musken, M., Di Fiore, S., Dotsch, A., Fischer, R., Haussler, S. Genetic determinants of Pseudomonas aeruginosa biofilm establishment. Microbiology. 156 (Pt 2), 431-441 (2010).
- Schurek, K. N., et al. Novel genetic determinants of low-level aminoglycoside resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother. 52 (12), 4213-4219 (2008).
- Oumeraci, T., et al. Comprehensive MALDI-TOF biotyping of the non-redundant Harvard Pseudomonas aeruginosa PA14 transposon insertion mutant library. PLoS One. 10 (2), e0117144 (2015).
- Yeung, A. T., et al. Swarming of Pseudomonas aeruginosa is controlled by a broad spectrum of transcriptional regulators, including MetR. J Bacteriol. 191 (18), 5592-5602 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten