Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Auswahl mehrerer Biomarker Teilmengen mit ebenso wirksame binäre Klassifikation Aufführungen
In diesem Artikel
Zusammenfassung
Vorhandene Algorithmen erzeugen eine Lösung für ein Biomarker-Erkennung-Dataset. Dieses Protokoll zeigt die Existenz von mehreren ähnlich effektive Lösungen und stellt eine benutzerfreundliche Software zur biomedizinische Forscher untersuchen ihre Datensätze für die vorgeschlagene Herausforderung helfen. Informatiker können auch dieses Feature in ihren Biomarker Erkennungsalgorithmen vorsehen.
Zusammenfassung
Biomarker-Erkennung ist eines der wichtigeren biomedizinische Fragen für High-Throughput "Omics" Forscher, und fast alle bestehenden Biomarker Erkennungsalgorithmen erzeugen ein Biomarker Teilmenge mit optimierter Performance-Messung für einen bestimmten Datensatz . Eine kürzlich durchgeführte Studie zeigte jedoch, die Existenz von mehreren Biomarker Teilmengen mit ähnlich wirksam oder sogar identische Klassifizierung Aufführungen. Dieses Protokoll stellt eine einfache und unkomplizierte Methode zur Erkennung von Biomarker-Teilmengen mit binären Klassifikation Aufführungen, besser als eine Benutzer-definierten Grenzwert. Das Protokoll besteht aus Datenaufbereitung und laden, Baseline Informationen Verdichtung, tuning-Parameter, Biomarker Screening, Ergebnis-Visualisierung und Interpretation, Biomarker gen Anmerkungen und Ergebnis und Visualisierung Ausfuhr an Publikationsqualität. Die vorgeschlagenen Biomarker screening-Strategie ist intuitiv und zeigt eine allgemeine Regel für Biomarker Erkennungsalgorithmen zu entwickeln. Eine benutzerfreundliche grafische Benutzeroberfläche (GUI) wurde entwickelt, mit Hilfe der Programmiersprache Python, Biomediziner direkten Zugriff auf ihre Ergebnisse zu ermöglichen. Den Quellcode und Handbuch des kSolutionVis können von http://www.healthinformaticslab.org/supp/resources.php heruntergeladen werden.
Einleitung
Binäre Klassifikation, einer der am häufigsten untersuchte und anspruchsvolle Data-mining-Probleme im biomedizinischen Bereich wird verwendet, um ein Klassifizierungsmodell ausgebildet auf zwei Gruppen von Proben mit der genauesten Diskriminierung Power1, bauen 2 , 3 , 4 , 5 , 6 , 7. big Data im biomedizinischen Bereich generiert hat jedoch die inhärente "große kleine PN" Paradigma, mit der Anzahl der Funktionen in der Regel viel größer als die Anzahl der Proben6,8,9. Biomediziner müssen daher die Funktion Dimension vor Nutzung der Klassifizierungsalgorithmen zur Vermeidung von overfitting Problem8,9zu reduzieren. Diagnose-Biomarker sind definiert als eine Teilmenge der erkannten Merkmale von gesunden Proben10,11Patienten einer bestimmten Krankheit zu trennen. Patienten sind in der Regel definiert als die positive Proben und die gesunden Kontrollpersonen sind definiert als der negative Proben12.
Jüngste Studien haben vorgeschlagen, daß es mehr als eine Lösung mit identisch oder ähnlich effektiv Einstufung Leistungen für eine biomedizinische Dataset-5. Fast alle Feature-Auswahl-Algorithmen sind deterministische Algorithmen, produzieren nur eine Lösung für das gleiche Dataset. Genetische Algorithmen können gleichzeitig mehrere Lösungen mit ähnlichen Leistungen erzeugen, aber sie noch versuchen, eine Lösung mit der besten Fitness-Funktion als die Ausgabe für einen bestimmten Datensatz13,14auswählen.
Feature-Auswahl-Algorithmen können etwa als Filter oder Wrapper12gruppiert werden. Ein Filter-Algorithmus wählt Top -k Funktionen geordnet nach ihrer einzelnen signifikante Assoziation mit der binären Klassenbezeichner basiert auf der Annahme, die Funktionen sind unabhängig von einander15,16,17 . Obwohl diese Annahme nicht für fast alle realen Datasets wahr hält, führt die heuristische Filterregel in vielen Fällen zum Beispiel die mRMR (minimale Redundanz und maximale Relevanz) Algorithmus, der Wilcoxon Test basierte Funktion Filtern (WRank) Algorithmus und die ROC (Empfänger Regelkennlinie) Handlung basiert (ROCRank)-Filterung Algorithmus. mRMR, ist eine effiziente Filter-Algorithmus, weil es nähert sich der kombinatorischen Schätzung-Problem mit einer Reihe von viel kleineren Problemen, im Vergleich zu der maximal-Abhängigkeit Feature Auswahlalgorithmus, von denen jede nur zwei Variablen beinhaltet, und Daher nutzt paarweise Joint Wahrscheinlichkeiten sind robuster18,19. Jedoch kann mRMR die Nützlichkeit der einige Funktionen unterschätzen, da es nicht die Interaktionen zwischen Objekten misst die Relevanz steigern können, und so vermisst einige Merkmalskombinationen, die einzeln nutzlos, aber eignen sich nur in Kombination. Der WRank Algorithmus berechnet eine nicht-parametrische Punktzahl wie diskriminierende eine Funktion zwischen zwei Klassen von Proben und ist bekannt für seine Robustheit für Ausreißer20,21. Darüber hinaus wertet der ROCRank-Algorithmus, wie bedeutend die Fläche unter der ROC-Kurve (AUC) eines bestimmten Merkmals für die untersuchten binäre Klassifikation Leistung22,23ist.
Auf der anderen Seite ein Wrapper wertet der vordefinierten Sichter Leistung einer gegebenen Funktion Teilmenge, iterativ durch heuristische Regel erzeugt und schafft die Funktion Teilmenge mit der besten Performance Messung24. Ein Wrapper in der Regel einen Filter in die Klassifizierung Leistung übertrifft aber läuft langsamer25. Beispielsweise verwendet die regularisiert Random Forest (RRF)26,27 Algorithmus gierige in der Regel durch die Auswertung der Features auf eine Teilmenge der Trainingsdaten an jedem zufälligen Wald Knoten, deren Funktion Bedeutung Partituren von der Gini-Index beurteilt werden . Die Wahl eines neuen Features werden benachteiligt, wenn die Informationsgewinnung, die der ausgewählten Features nicht bessert. Darüber hinaus die Vorhersage-Analyse für Microarrays (PAM)28,29 -Algorithmus auch eine Wrapper-Algorithmus berechnet einen Schwerpunkt für jeden der die Klassenbezeichner und wählt dann Eigenschaften gen Zentroide gegenüber der gesamten schrumpfen Klasse-Schwerpunkt. PAM ist robust für vorgelagerten Funktionen.
Mehrere Lösungen mit die höchste Klassifizierung Leistung können für alle angegebenen Dataset erforderlich sein. Erstens das Optimierungsziel eines deterministischen Algorithmus zeichnet sich durch eine mathematische Formel, z.B., minimalen Fehler Rate30, das ist nicht unbedingt ideal für biologische Proben. Zweitens kann ein Dataset mehrere, deutlich verschiedene Lösungen mit ähnlichen wirksam oder sogar identische Leistungen haben. Fast alle vorhandenen Feature Auswahl Algorithmen werden eine der folgenden Lösungen als die Ausgabe31zufällig auswählen.
Diese Studie wird ein Informatik analytische Protokoll für die Erzeugung von mehreren Feature Auswahl Lösungen mit ähnlichen Vorstellungen für jede gegebene binäre Klassifikation Dataset einführen. Wenn man bedenkt, dass die meisten Biomediziner mit informatischen Techniken oder Computer-Codierung nicht auskennen, wurde eine benutzerfreundliche grafische Benutzeroberfläche (GUI) entwickelt, um die schnelle Analyse von biomedizinischen binäre Klassifikation Datasets zu erleichtern. Das analytische Protokoll besteht aus Daten laden und zusammenfassen, tuning-Parameter Pipeline Ausführung und Ergebnis-Interpretationen. Mit einem einfachen Klick kann der Forscher die Biomarker Teilmengen und Publikationsqualität Visualisierung Grundstücke zu generieren. Das Protokoll wurde mit der Transkriptom zwei binäre Klassifikation Datensätze der akute lymphoblastische Leukämie (ALL), d. h., ALL1 und ALL212getestet. Die Datensätze der ALL1 und ALL2 wurden vom Broad Institute Genom Analyse Rechenzentrum, erhältlich bei http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi heruntergeladen. ALL1 enthält 128 Samples mit 12.625 Features. Von diesen Proben sind 95 B-Zelle alle und 33 sind T-Zell ALL. ALL2 umfasst 100 Proben mit 12.625 Funktionen sowie. Von diesen Proben gibt es 65 Patienten, die Rückfall erlitten und 35 Patienten, die nicht der Fall war. ALL1 war eine einfache binäre Klassifikation Dataset mit einer minimalen Genauigkeit von vier Filter und vier Wrapper, 96,7 % und 6 der 8 Feature Auswahl Algorithmen erreichen 100 %12. Während ALL2 ein schwieriger Dataset mit den oben genannten 8 Feature Auswahl Algorithmen nicht besser als 83,7 % Genauigkeit12zu erreichen war. Diese beste Genauigkeit wurde mit 56 Funktionen erkannt durch den Wrapper-Algorithmus, Korrelation basierende Funktion Auswahl (CFS) erreicht.
Protokoll
Hinweis: Das folgende Protokoll beschreibt die Details des analytischen Verfahrens Informatik und Pseudo-Codes der wichtigsten Module. Die automatische Analyse-System wurde mit Python-Version 3.6.0 und die Python-Module-Pandas, Abc, Numpy, Scipy, Sklearn, Sys, PyQt5, Sys, mRMR, Mathematik und Matplotlib entwickelt. In dieser Studie verwendeten Materialien sind in der Tabelle der Materialienaufgeführt.
1. Vorbereiten der Data-Matrix-Klasse Etiketten und
- Bereiten Sie die Datendatei Matrix als Matrix Registerkarte oder Komma-Trennzeichen getrennte Datei wie in Figur 1Adargestellt.
Hinweis: Jede Zeile hat die Werte einer Funktion, und das erste Element ist der Funktionsname. Ein Feature ist ein Probeset-ID für die Microarray-basierte Transkriptom-Dataset oder möglicherweise einen anderen Wert-ID wie ein Cystein-Rückstand mit seinem Methylierung-Wert in einem Methylomic-Dataset. Jede Spalte gibt die Funktionswerte einer bestimmten Probe mit dem ersten Element wird die Probenname. Eine Zeile wird in Spalten durch einen Tabulator (Abbildung 1 b) oder ein Komma (Abbildung 1) getrennt. Eine tabulatorgetrennte Datei ist die Datei-Erweiterung haben.TSV anerkannt, und eine durch Trennzeichen getrennte Datei hat die Erweiterung CSV. Diese Datei kann generiert werden, indem Sie einer Matrix als entweder haben.TSV oder CSV-Format von Software wie Microsoft Excel speichern. Datamatrix kann auch durch Computer-Codierung erzeugt werden. - Bereiten Sie die Klassendatei Label als eine Registerkarte oder Komma-getrennte Matrix Datei (Abbildung 1), ähnlich wie bei der Matrix-Datei.
Hinweis: Die erste Spalte gibt die probennamen und Klasse Label von jeder Probe wird in der Spalte mit dem Titel Klassegegeben. Maximale Kompatibilität gilt das Codierungsverfahren, so dass zusätzliche Spalten hinzugefügt werden können. Die Klassendatei Label kann als haben.TSV oder CSV-Datei formatiert werden. Die Namen in der Spalte Klasse möglicherweise alle Begriffe, und möglicherweise gibt es mehr als zwei Klassen von Proben. Der Benutzer kann zwei Klassen für die folgende Analyse wählen.
2. Legen Sie die Data-Matrix-Klasse Etiketten und
- Laden Sie die Datenbeschriftungen Matrix und Klasse in die Software. Klicken Sie auf Load Data-Matrix die benutzerspezifischen Daten Matrix Datei auswählen. Klicken Sie auf Last Klassenbezeichner die entsprechende Klasse Label-Datei auswählen.
Hinweis: Nachdem beide Dateien geladen sind, wird kSolutionVis einen Routine Bildschirm die Kompatibilität zwischen den beiden Dateien durchführen. - Die Funktionen und Beispiele aus der Datendatei Matrix zusammenfassen. Schätzen Sie die Größe der Datendatei Matrix.
- Fassen Sie die Proben und Klassen von der Klasse Label-Datei. Schätzen Sie die Größe der Beschriftung Klassendatei.
- Testen Sie, ob jede Probe aus der Datenmatrix ein Klasse-Label hat. Fassen Sie die Nummern der Proben mit der Klassenbezeichner.
(3) zusammenfassen und die Baseline-Statistik des Datasets anzeigen
- Klicken Sie den Button zusammenfassen, ohne jede angegebene Schlüsselwort eingeben und die Software zeigt 20 indizierte Eigenschaften und die entsprechenden Funktionen Namen.
Hinweis: Benutzer müssen den Featurenamen angeben, die, den Sie, um seine Grundlinie Statistiken und entsprechenden Wertverteilung unter alle eingabesamples zu sehen finden möchten. - Geben Sie ein Stichwort, z.B. "1000_at" in das Textfeld ein Feature zu einer bestimmten Funktion zusammengefasst werden. Klicken Sie auf zusammenfassen , die Baseline-Statistiken für diese bestimmten Funktion bekommen.
Hinweis: Das Schlüsselwort kann überall in den Zielnamen Feature erleichtert die Suche für Benutzer angezeigt. - Klicken Sie den Button zusammenfassen , mehr als ein Objekt mit dem angegebenen Schlüsselwort zu finden, und geben Sie dann die Besonderheit-ID zum Fortsetzen des oben genannten Schrittdes fasst ein besonderes Merkmal.
4. bestimmen Sie die Klassenbezeichner und die Anzahl der Top-Rankings Features
- Wählen Sie die Namen der positiv ("P (33)") und negativ ("N (95)") Klassen in den Auswahlboxen Klasse Positive und Negative Klasse, wie in Abbildung 2 dargestellt (Mitte).
Hinweis: Es wird vorgeschlagen, eine ausgeglichene binäre Klassifikation Dataset, d. h.die Differenz zwischen der Anzahl von positiven und negativen Proben ist minimal zu wählen. Die Anzahl der Proben ist auch in Klammern hinter dem Namen des jede Klasse Beschriftung in den zwei Dropdown-Boxen angezeigt. - Wählen Sie 10 als die Anzahl der Top-Rankings Merkmale (Parameter pTopX) in der Dropdown-Box Top_X (?) für eine umfassende Bildschirm der Feature-Teilmenge.
Hinweis: Die Software ordnet automatisch alle Funktionen von der P-Wert berechnet, indem ein t-Test für jede Funktion, die die positiven und negativen Klassen zu vergleichen. Ein Feature mit einem kleineren P-Wert hat eine besser unterscheidende Kraft zwischen den beiden Klassen von Proben. Das umfassende Screening Modul ist rechenintensiv. Der Parameter pTopX ist standardmäßig 10. Benutzer können diese Parameter im Bereich von 10 bis 50 ändern, bis sie befriedigend finden Teilmengen mit gute Einstufung Leistungen verfügen.
5. Melodie Systemparameter für verschiedene Leistungen
- Wählen Sie die Performance-Messung (pMeasurement) Genauigkeit (Acc) in der Dropdown-Box Acc/bAcc (?) für die ausgewählten Klassifikator Extreme Learning Machine (ELM). Eine weitere Möglichkeit dieses Parameters ist die Messung ausgeglichen Genauigkeit (bAcc).
Hinweis: Lassen Sie TP, FN, TN, und FP werden die Nummern der wahren positiven, falsche Negative, richtig negativ und Fehlalarme, beziehungsweise. Die Messung Acc ist definiert als (TP+TN)/(TP+FN+TN+FP), das funktioniert am besten bei einer ausgewogenen Dataset-6. Aber ein Klassifikator optimiert für Acc tendenziell alle Proben die negative Klasse zuordnen, wenn die Anzahl der negativen Proben viel größer als die positiven ist. Die bAcc ist definiert als (Sn + Sp) / 2, wo Sn = TP/(TP+FN) und Sp = TN/(TN+FP) sind die richtig vorhergesagten Preise für Positive und negative Proben, beziehungsweise. Daher bAcc normalisiert die Vorhersage Aufführungen über die beiden Klassen und zu einer ausgewogenen Vorhersage Leistung über zwei unsymmetrische Klassen führen kann. ACC ist die Standardauswahl von pMeasurement. Die Software nutzt der Sichter ELM standardmäßig die Einstufung Leistungen zu berechnen. Der Benutzer kann auch eine Klassifizierung von SVM (Support Vector Machine), KNN (k nächsten Nachbarn), Decision Tree oder Naïve Bayes wählen. - Wählen Sie den cutoff Wert 0,70 (Parameter pCutoff) für die angegebene Performance-Messung im Eingabefeld pCutoff:.
Hinweis: Sowohl Acc und bAcc im Bereich zwischen 0 und 1, und der Benutzer kann einen Wert pCutoff angeben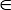 [0, 1] als der Cut-off um die aufeinander abgestimmten Lösungen anzuzeigen. Die Software führt eine umfassende Feature-Teilmenge screening und eine geeignete Wahl der pCutoff machen die 3D-Visualisierung, intuitiver und explizite. Der Standardwert für pCutoff ist 0.70.
[0, 1] als der Cut-off um die aufeinander abgestimmten Lösungen anzuzeigen. Die Software führt eine umfassende Feature-Teilmenge screening und eine geeignete Wahl der pCutoff machen die 3D-Visualisierung, intuitiver und explizite. Der Standardwert für pCutoff ist 0.70.
6. führen Sie die Pipeline und die interaktive VISUALISIERTE Ergebnisse
- Klicken Sie auf die Schaltfläche Analyze Ausführen der Pipelines und die Visualisierung Grundstücke zu erzeugen, wie in Abbildung 2 (unten) dargestellt.
Hinweis: Die linke Tabelle gibt die Funktion Teilmengen und ihre pMeasurement durch die 10-divisibel Kreuzvalidierung Strategie des Klassifikators ELM, berechnet, wie zuvor beschrieben5. Zwei 3D Streudiagrammen und zweizeilige Grundstücke werden für die Feature-Teilmenge Screening-Verfahren mit den aktuellen Parametereinstellungen generiert. - Wählen Sie 0,70 als der Standardwert der pMeasurement cutoff (Parameter PiCutoff, Eingabefeld Wert) und 10 als die standardmäßige Anzahl der besten Feature Teilmengen (Parameter PiFSNum).
Hinweis: Die Pipeline erfolgt mit dem Parameter pTopX, pMeasurement, und pCutoff. Die ermittelte Funktion, die Teilmengen weiter sein können gezeigt mit der cutoff PiCutoff, kann nicht jedoch PiCutoff kleiner als pCutoffsein. Daher PiCutoff als pCutoff initialisiert wird und nur die Funktion Teilmengen mit Performance Messung ≥ PiCutoff visualisiert werden. Der Standardwert für PiCutoff ist pCutoff. Manchmal kSolutionVis erkennt viele Lösungen, und nur die besten PiFSNum (Standard: 10) Feature Teilmengen visualisiert werden. Wenn die Anzahl der Teilmengen der Funktion von der Software erkannt kleiner als PiFSNumist, werden die Funktion Teilmengen visualisiert. - Erfassen Sie und interpretieren Sie die Funktionen von der Software erkannt, wie in Abbildung 3dargestellt.
Hinweis: Die Tabelle in das linke Feld zeigt erkannten Funktion Teilmengen und ihre Performance-Messungen. Die Namen der ersten drei Spalten sind "F1", "F2" und "F3". Die drei Funktionen in jeder Funktion Teilmenge sind in ihrer Rangfolge in einer Zeile angegeben (F1 < F2 < F3). Die letzte Spalte gibt die Performance-Messung (Acc oder bAcc) der einzelnen Feature-Untergruppen, und die Spaltennamen (Acc oder bAcc) ist der Wert der pMeasurement.
(7) interpretieren Sie die 3D Scatter Plots visualisieren und interpretieren Sie die Funktion Teilmengen mit ähnlich effektiv binäre Klassifikation Performances mit 3D Streudiagrammen
- Klicken Sie die Schaltfläche Analyze , 3D Streudiagramm der Top 10-Feature Teilmengen mit Klassifizierung Bestleistungen (Acc oder bAcc) von der Software erkannt, wie in Abbildung 3 (mittlere Feld) zu generieren. Die drei Funktionen in einer Feature-Teilmenge in aufsteigender Reihenfolge von ihren Rängen zu sortieren und verwenden Sie die Reihen der drei Funktionen als die F1/F2/F3-Achsen, d.h.F1 < F2 < F3.
Hinweis: Die Farbe eines Punktes repräsentiert die binäre Klassifikation Performance der entsprechenden Funktion Teilmenge. Ein Dataset kann mehrere Feature Teilmengen mit ebenso effektive Performance-Messungen haben. Daher ist eine interaktive und vereinfachte Streudiagramm notwendig. - Ändern Sie den Wert bis 0.70 in das Eingabefeld pCutoff: und klicken Sie die Schaltfläche Analyze 3D Streudiagramm der Funktion Teilmengen mit Performance Messung ≥ PiCutoffgenerieren wie in Abbildung 3 (rechts Feld) zu sehen. Klicken Sie auf 3D tuning , öffnet ein neues Fenster zum Betrachtungswinkel von 3D Streudiagramm manuell einstellen.
Hinweis: Jede Teilmenge Funktion ist durch einen Punkt in der gleichen Weise wie oben dargestellt. Die 3D Punktwolke wurde in den Standard-Winkel erzeugt. Zur Erleichterung der 3D-Visualisierung und tuning, ein separates Fenster werden geöffnet mit dem Button 3D tuning. - Klicken Sie auf verkleinern , die Redundanz der erkannten Funktion Teilmengen zu reduzieren.
Hinweis: Wenn Benutzer wünschen weiter wählen die Funktion Drillinge und minimieren die Redundanz der Funktion Teilmengen, bietet die Software auch diese Funktion mit dem mRMR Feature Auswahlalgorithmus. Nach einem Klick auf die Schaltfläche " verkleinern ", kSolutionVis entfernen diese redundante Funktionen in der Feature-Triolen und regenerieren in der Tabelle und streuen die beiden oben genannten Grundstücke. Entfernte Features der Funktion Drillinge werden durch das Schlüsselwort in der Tabelle ersetzt werden. Die Werte None in der F1/F2/F3-Achse werden als der Wert des PiFSNum bezeichnet werden (der Bereich des normalen Wertes der F1/F2/F3 ist [1, Top_x]). Daher erscheinen die Punkte, die keinen Wert enthalten werden "Ausreißer" Punkte im 3D-plots. Die manuell einstellbaren 3D-Plots finden Sie in "Handabstimmung der 3D Punkt plottet" in das ergänzende Material.
8. finden Sie gen Anmerkungen und ihre Verbände mit Krankheiten des Menschen
Hinweis: Schritte 8 bis 10 werden ein Gen aus der Sequenz Ebene von DNA und von Protein Anmerkungen zu erläutern. Erstens die Gen-Symbol der Biomarker-ID aus den oben genannten Schritten wird aus der Datenbank DAVID32abgerufen werden, und dann zwei repräsentativen Web-Server werden verwendet, um dieses Gen Symbol aus den Ebenen von DNA und Proteinen, bzw. zu analysieren. Der Server GeneCard bietet eine umfassende funktionale Annotation eines bestimmten Gens-Symbols, und die Online Mendelian Erbschaft im Mann-Datenbank (OMIM) bietet die umfassendste Kuration der Krankheit-gen Verbände. Der Server UniProtKB ist eines der umfassendsten Proteindatenbank und Server Gruppenbasierte Vorhersage System (GPS) prognostiziert die Signalisierung Phosphorylierung des für eine sehr große Liste der Kinasen.
- Kopieren und Einfügen des Weblinks der Datenbank DAVID in einem Web-Browser und öffnen Sie die Web-Seite dieser Datenbank. Klicken Sie den Link Gen ID Konvertierung in Abbildung 4A gesehen und geben Sie die Funktion IDs 38319_at/38147_at/33238_at der ersten Biomarker Teilmenge des Datasets ALL1 (Abbildung 4 b). Klicken Sie auf den Link Gen Liste und klicken Sie auf Liste einreichen , wie in Abbildung 4 bdargestellt. Rufen Sie die Anmerkungen von Interesse ab und klicken Sie auf Liste anzeigen gen (Abbildung 4). Abrufen der Liste der Gen-Symbole (Abbildung 4).
Hinweis: Der Gen-Symbole, die hier abgerufen werden für weitere funktionale Anmerkungen in den nächsten Schritten verwendet werden. - Kopieren und Einfügen des Weblinks der Datenbank Gene Karten in einem Web-Browser und öffnen Sie die Web-Seite dieser Datenbank. Suchen Sie ein Gen namens CD3D in der Datenbank-Abfrage-Eingabefeld und finden Sie die Anmerkungen dieses Gens von Gene Karten33,34, wie in Tabelle 1 und Abb. 5Agezeigt.
Hinweis: Gen-Karten ist eine umfassende gen Knowledgebase, Nomenklatur, Genomik, Proteomik, subzelluläre Lokalisation und Beteiligten Bahnen und anderen Funktionsmodule. Darüber hinaus externe Links zu verschiedenen anderen biomedizinischen Datenbanken wie PDB/PDB_REDO35, Entrez Gene36, OMIM37und UniProtKB38. Wenn der Funktionsname kein standard-gen-Symbol ist, verwenden Sie die Datenbank ENSEMBL39umwandeln. CD3D ist der Name des Gens T-Zell-Rezeptor-T3-Delta-Kette. - Kopieren und Einfügen des Weblinks der Datenbank OMIM in einem Web-Browser und öffnen Sie die Web-Seite dieser Datenbank. Suchen Sie ein Gen namens CD3D und finden Sie die Anmerkungen dieses Gens aus der Datenbank OMIM37, wie in Tabelle 1 und Abbildung 5 bgezeigt.
Hinweis: OMIM dient heute als eines der umfassendsten und maßgeblichen Quellen des menschlichen Gens Verbindungen mit vererbbaren Krankheiten. OMIM wurde initiiert von Dr. Victor A. McKusick, der krankheitsassoziierten genetischen Mutationen40zu katalogisieren. OMIM erstreckt sich jetzt über 15.000 Gene des Menschen und mehr als 8.500 Phänotypen, ab Dezember 1St 2017.
(9) kommentieren Sie die kodierten Proteine und die Post-translationalen Modifikationen
- Kopieren und Einfügen des Weblinks der Datenbank UniProtKB in einem Web-Browser und öffnen Sie die Web-Seite dieser Datenbank. Ein Gen namens CD3D in das Eingabefeld der Abfrage des UniProtKB finden Sie und die Anmerkungen dieses Gens aus der Datenbank38, wie in Tabelle 1 und Abbildung 5gezeigt.
Hinweis: UniProtKB sammelt eine reiche Quelle von Anmerkungen für Proteine, einschließlich der Nomenklatur und funktionelle Informationen. Diese Datenbank bietet auch externe Links zu anderen weit verbreiteten Datenbanken, einschließlich PDB/PDB_REDO35, OMIM37und Pfam41. - Kopieren und Einfügen des Weblinks des Webservers GPS in einem Web-Browser und öffnen Sie die Web-Seite dieses Webservers. Rufen Sie die Proteinsequenz codiert durch die Biomarker-gen CD3D von UniProtKB Datenbank38 und Vorhersagen Sie des Proteins Post-translationale Modifikation (PTM) Rückstände mit dem online-Tool GPS, wie in Tabelle 1 und Abbildung 5gezeigt.
Hinweis: Ein biologisches System ist dynamisch und kompliziert, und die vorhandenen Datenbanken sammeln nur bekannten Informationen. Daher können biomedizinische Vorhersage online-Tools sowie offline Programme sinnvoll ergänzend einen hypothetischen Mechanismus nachweisen. GPS wurde weiterentwickelt und verbessert für über 12 Jahre7,42 und kann verwendet werden, um ein Protein PTM Rückstände in ein bestimmtes Peptid Sequenz43,44vorherzusagen. Werkzeuge sind auch für verschiedene Forschungsthemen, einschließlich der Vorhersage von subzelluläre Lage45 und Transkription Faktor verbindliche Motive 46 unter anderem ein Protein zur Verfügung.
10. mit Anmerkungen versehen Sie, Protein-Protein-Wechselwirkungen und ihre angereicherten Funktionsmodule
- Kopieren und Einfügen des Weblinks des Webservers Zeichenfolge in einen Webbrowser und öffnen Sie die Web-Seite dieses Webservers. Durchsuchen Sie die Liste für die Gene CD3D und P53, Immobilien und finden Sie ihre orchestrierten Verwendung der Datenbank String47. Das gleiche Verfahren kann mit einem anderen Webserver, DAVID32durchgeführt werden.
Hinweis: Neben den oben genannten Anmerkungen für einzelne Gene gibt es viele große Informatik-Tools zur Verfügung, um die Eigenschaften einer Gruppe von Genen zu untersuchen. Eine kürzlich durchgeführte Studie zeigte, dass individuell schlecht Markergene eine deutlich verbesserte gen Set5darstellen könnte. Daher lohnt es sich der Rechenaufwand zum Bildschirm für kompliziertere Biomarker. Die Datenbank String kann die bekannte oder vorhergesagten Interaktion Verbindungen visualisieren und der David-Server erkennt möglicherweise die Funktionsmodulen mit erheblichen Phänotyp-Verbände in der abgefragten Gene47,32. Verschiedenen anderen großen Informatik-Analyse-Tools zur Verfügung stehen.
11. export generierten Biomarker Teilmengen und die Visualisierung Grundstücke
- Exportieren Sie die erkannten Biomarker Teilmengen als haben.TSV oder CSV Textdatei zur weiteren Analyse. Klicken Sie auf exportieren Sie die Tabelle unter dem Tisch der alle erkannten Biomarker Teilmengen und wählen Sie die Text-Format Speichern als.
- Die Visualisierung Grundstücke als Image-Datei zu exportieren. Klicken Sie auf die Schaltfläche Speichern unter jeder Parzelle und wählen Sie welches Bildformat zu speichern.
Hinweis: Die Software unterstützt das Pixel Format .png und Vektor Format .svg. Die Pixelbilder sind gut für die Anzeige auf dem Bildschirm, während der Vektor-Bilder in jede erforderliche Auflösung für Publikationszwecke Journal konvertiert werden können.
Ergebnisse
Das Ziel dieses Workflows (Abbildung 6) ist, mehrere Biomarker Teilmengen mit ähnlichen Effizienzen für eine binäre Klassifikation Dataset zu erkennen. Der gesamte Prozess wird durch zwei Beispiel-Datasets ALL1 und ALL2 extrahiert aus einem kürzlich erschienenen Biomarker-Erkennung12,48Studie veranschaulicht. Ein Benutzer kann kSolutionVis installieren, indem Sie die Anweisungen in den ergänzende...
Diskussion
Diese Studie bietet eine einfach zu befolgende Multi-Lösung Biomarker Erkennung und Charakterisierung Protokoll für eine benutzerspezifische binäre Klassifikation Dataset. Die Software setzt Schwerpunkt auf Benutzerfreundlichkeit und flexible Import-/Export-Schnittstellen für verschiedene Datei-Formate, so dass biomedizinische Forscher, ihre Dataset einfach über die Benutzeroberfläche der Software zu untersuchen. Dieser Studie betont auch, dass mehr als eine Lösung mit ähnlich effektiv Modellierung Aufführungen,...
Offenlegungen
Wir haben keine Interessenkonflikte im Zusammenhang mit diesem Bericht.
Danksagungen
Diese Arbeit wurde durch die strategische Priorität Forschungsprogramm von der chinesischen Akademie der Wissenschaften (XDB13040400) und die Start-Zuschuss von Jilin-Universität unterstützt. Anonymen Gutachtern und biomedizinische Tests Benutzer wurden für ihre konstruktive Kritik zur Verbesserung der Benutzerfreundlichkeit und Funktionalität des kSolutionVis geschätzt.
Materialien
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Referenzen
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten