Method Article
Dissektion der Enhancer-Funktion mit Multiplex CRISPR-basierte Enhancer Einmischung in Zell-Linien
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die erforderlichen Schritte zum Entwerfen und ausführen gemultiplexten Ausrichtung der Enhancer mit deaktivieren Fusionsproteins SID4X-dCas9-KRABBE, auch bekannt als Enhancer Störungen (Enhancer-i). Dieses Protokoll ermöglicht die Identifizierung von Enhancer, die Genexpression regulieren und erleichtert das Sezieren von Beziehungen zwischen Enhancer Regulierung ein gemeinsames Zielgen.
Zusammenfassung
Mehreren Enhancer häufig Regeln eines bestimmten Gens, doch für die meisten Gene, es bleibt unklar, welche Enhancer für die Genexpression notwendig sind und wie diese Enhancer kombinieren, um eine transcriptional Antwort zu produzieren. Da Millionen von Enhancer identifiziert wurden, sind Hochdurchsatz-Werkzeuge notwendig, Enhancer-Funktion auf einer genomweiten Skala bestimmen. Aktuelle Methoden zur Untersuchung von Enhancer-Funktion auch die Abgabe von genetischen Deletionen mit Nuklease-kompetente Cas9, aber es ist schwierig, die kombinatorische Effekte von mehreren Enhancer mit dieser Technik zu studieren, wie mehrere aufeinander folgende klonalen Zelllinien werden muss generiert. Hier präsentieren wir Ihnen Enhancer-i, eine CRISPR Interferenz basierende Methode, die für funktionelle Verhör von mehreren Enhancer gleichzeitig ihre endogenen Loci ermöglicht. Enhancer-i nutzt zwei repressiven Domänen mit Nuklease-defizienten verschmolzen Cas9, SID und KRABBE, Enhancer Deaktivierung über Histon Deacetylation an gezielte Loci zu erreichen. Dieses Protokoll nutzt Transiente Transfektion von Guide RNAs, vorübergehende Inaktivierung von Zielregionen zu ermöglichen und ist besonders effektiv bei der induzierbare transkriptionelle Reaktionen auf Reize in Gewebekultur Einstellungen blockieren. Enhancer-i ist hochspezifisch, sowohl in seiner genomischen targeting und seine Auswirkungen auf die globalen Genexpression. Aus diesem Protokoll erzielten Ergebnisse helfen um zu verstehen, ob ein Enhancer ist zur Genexpression, das Ausmaß des Beitrags und wie der Beitrag von anderen nahe gelegenen Enhancer betroffen ist.
Einleitung
Groß angelegte Projekte wie ENCODE1, Fahrplan Epigenomics2und FANTOM-Sequenzierung3 haben festgestellt, dass Millionen von vermeintlichen Enhancer innerhalb des menschlichen Genoms über Hunderte von Zelltypen. Schätzungen gehen davon aus, dass jeder Veranstalter mit durchschnittlich 4,9 Enhancer Associates und jede Enhancer durchschnittlich 2,4 Gene3 Kontakte, darauf hindeutet, dass gen-Expression oft das Ergebnis der Integration von mehreren verteilten regulatorischer Wechselwirkungen ist. Eine verbleibende Herausforderung ist zu definieren, nicht nur wie die einzelnen Enhancer zur Genexpression, beitragen, sondern wie sie kombinieren, um den Ausdruck beeinflussen. Gentechnische Ansätze werden häufig verwendet, um Beziehungen zwischen Enhancer in Modellorganismen Drosophila4 Mäuse5identifizieren. Diese Experimente sind jedoch zeitaufwendig und niedrigen Durchsatz für die Untersuchung von mehreren Enhancer an mehreren Genen.
Ein Ansatz für das Studium Enhancer-Funktion auf einer großen Skala umfasst massiv parallelen Reporter Assays. Diese Tests ermöglichen die gleichzeitige Vorführung von Tausenden von DNA-Sequenzen für ihre Fähigkeit, den Ausdruck von einem Reporter-gen6fahren. Während dieser Tests gezeigt haben, dass DNA-Sequenz allein ausreichen kann, um gen Verordnung Informationen7vermitteln, kommen sie mit den Einschränkungen von außerhalb des Kontexts native Chromatin durchgeführt wird und mit einem heterologen Promotor. Darüber hinaus ist die Größe der DNA-Sequenz in massiv parallelen Reporter Assays analysiert in der Regel weniger als 200 Basenpaaren, die gewünschte umliegenden Sequenz ausschließen können. Wichtig ist, wie Reporter-Assays nur die Aktivität einer Sequenz zu einem Zeitpunkt Messen, nehmen nicht berücksichtigt die komplexen Beziehungen sie, die zwischen Enhancer bestehen können. So massiv parallelen Reporter Assays informativ über die intrinsischen Aktivität einer DNA-Sequenz können zwar sein, sie nicht unbedingt der Funktion dieser DNA-Sequenz im Rahmen des Genoms mitteilen.
Vor kurzem haben entwickelte CRISPR/Cas9 Werkzeuge8 erleichtert das Studium der Genregulation wie sie für die Löschung der Enhancer an die endogenen Locus ermöglichen. Jedoch mehrere Enhancer gleichzeitig löschen, genomische Instabilität führen kann, und es ist zeitaufwendig, zu aufeinander folgenden Enhancer Löschungen in einer einzelnen Zelle Linie zu erzeugen. Darüber hinaus neue genomischen Sequenz entsteht an der Stelle der Löschung nach Reparatur, und diese Sequenz kann regulatorischen Funktion gewinnen. Eine alternative Version des Cas9 wurde entwickelt, speziell für modulierende Genexpression, unter Berufung auf Verschmelzungen von Aktivierung9,10 oder11,12 Domänen der Nuklease-defizienten Form des Cas9 zu unterdrücken (dCas9). Diese Schmelzverfahren Proteine sind ideal für das Studium mehrere Loci gleichzeitig, da sie nicht physisch die DNA-Sequenz verändern, und stattdessen Epigenetik modulieren um eine regulatorische Region zu befragen. Die am weitesten verbreitete repressive Fusion ist KRABBE, die Rekruten den KAP1 Co Repressor Komplex, Förderung der Ablagerung von Repression-assoziierten Histone H3 Lysin 9 Trimethylation (H3K9me3)13. dCas9-KRABBE, auch bekannt als CRISPR Störungen14, wurde für ihren Beitrag zur Gen Ausdruck15,16, Ziel- und Bildschirm einzelne Enhancer verwendet. jedoch wurde es nicht für mehrere Bereiche gleichzeitig gezielt optimiert. Eine Version von multiplex CRISPR-Störungen für Enhancer, Mosaik-Seq17, verwendet einzelne Zelle RNA-Seq als ein Auslesen, aber diese Technologie ist teuer und nur geeignet für die Untersuchung von stark exprimierten Genen aufgrund der geringen Sensitivität der Einzelzelle RNA-Seq.
Wir wollten eine CRISPR Interferenz basierende Methode für kombinatorische Enhancer-Funktion im Rahmen einer transcriptional Antwort auf Östrogen sezieren zu entwickeln. Etwa die Hälfte der Östrogen-responsive Gene enthalten mindestens 2 Enhancer durch Östrogen-Rezeptor Alpha (ER) gebunden, in der Nähe18, was darauf hindeutet, dass mehrere Enhancer können an die Östrogen-Antwort Teilnahme, und das Verständnis die rechtliche Logik erfordern würde Ausrichtung auf gleichzeitig mehrere Enhancer. Wie ausgangsstudien mit CRISPR Störungen bei Promotoren vorgeschlagen, dass nicht alle Veranstalter gleichermaßen auf KRAB-vermittelten Repression19reagieren, begründete wir, dass die Zugabe von einer deutlichen repressiven Domäne dCas9 die Deaktivierung des erleichtern können diverse Enhancer. Wir entschieden uns für die Sin3a Interaktion Domain von Mad1 (SID)20 führt auf die Rekrutierung von Histon Deacetylases21, die Acetyl-Gruppen auf Histone zu entfernen, die transkriptionelle Aktivität zugeordnet sind. Wichtig ist, die SID der Domäne war effektiv bei der Reduzierung der Genexpression bei dCas922 und Geschichten23verschmolzen, und Sin3a hat gezeigt, dass ein potenter repressiven Co-Faktor in einer Vielzahl von Enhancer Sequenz Kontexten24werden. Wir verwendet, SID4x-dCas9-KRABBE (Enhancer-i) um 10 verschiedene Enhancer, an der ER gebunden und ER Bindungsstellen (Stabilisierungen), die notwendig sind für die Östrogen transcriptional Antwort bei 4 Genen18zu identifizieren. Wir gezielt auch die Kombinationen der Enhancer, die Standorte zu identifizieren, die in der Produktion von Östrogen transcriptional Antwort kooperieren. Wir fanden, dass bis zu 50 Seiten möglicherweise gleichzeitig mit Veränderungen der nachweisbaren Genexpression ausgerichtet werden können. Mit ChIP-Seq und RNA-Seq, haben wir bewiesen, dass Enhancer-i eine hochspezifische Technik für mehrere Enhancer gleichzeitig zu studieren.
In diesem Protokoll beschreiben wir die Schritte bei der Durchführung von Enhancer-i, eine flexible Technik, die die funktionelle Studie von mehreren Enhancer gleichzeitig in einer Gewebekultur-Umgebung ermöglicht. Enhancer-i ist hoch korreliert mit genetischen löschen sondern bietet vorübergehende Deaktivierung, das Histon Deacetylases (HDACs) abhängig ist. Durch die Bereitstellung von Guide RNAs über Transiente Transfektion im Gegensatz zu stabilen Integration über virale Vektoren, vermeidet dieses Protokolls Ablagerung und potentielle Verbreitung von H3K9me3. Dieses Protokoll Details Guide RNA-Design und Klonen über Gibson Versammlung, die Transfektion von guide RNAs mit Lipofektion, und die Analyse der resultierenden Genexpression von qPCR ändert. Wir berücksichtigen auch die Methoden zur Bewertung der Spezifität des Zielens Enhancer-i auf der Ebene des Genoms und Transkriptom. Während diese Technik entwickelt wurde, um zu studieren Genregulation durch ER Enhancer in menschlichen Krebszelllinien gebunden, es gilt für die Zerlegung von jedem Säugetier Enhancer.
Protokoll
1. Generation der Zelle Linien stabil mit dem Ausdruck SID4X-dCas9-KRABBE
Hinweis: Die Transfektion und Drogen-Konzentrationen, die hier vorgestellt wurden für Ishikawa Zellen ein Endometriumkarzinom Zelllinie optimiert in RPMI 1640 Medien ergänzt mit 10 % FBS und 1 % Penicillin/Streptomycin (komplette RPMI) angebaut. Anderen Zelllinien benötigen unterschiedliche Transfection Bedingungen und Wirkstoff-Konzentrationen. Benutzer können auch Transiente Transfektion Experimente in Wildtyp Zellen, anstatt zu generieren eine stabile Zelllinie mit ein Plasmid mit dem Ausdruck SID4X-dCas9-KRABBE zusammen mit Guide RNA mit dem Ausdruck Plasmide ausführen; Ergebnisse aus transiente Transfektionen möglicherweise schwer zu reproduzieren, da SID4X-dCas9-KRAB Ebenen durch Transfektion variieren können.
- Ishikawa Zellen in mindestens 2 Brunnen von einer 6-Well-Platte mit 30-50 % Zusammenfluss (ca. 300.000 Ishikawa-Zellen) in 3 mL komplette RPMI-Platte.
- Aspirieren Sie die Medien aus den Zellen. Waschen Sie die Zellen einmal mit 1 X PBS (pH 7,4). Aspirieren der PBS und Trypsin (4 mL für eine 10 cm Teller oder 5 mL für eine T-75 Flasche) hinzufügen.
- Inkubieren Sie die Zellen für ~ 5 min bei 37 ° C, Kontrolle alle 2 min für freistehende Zellen und das Schiff sanft schütteln.
- Sobald Zellen getrennt haben, trypsiniert Pipette Zellen nach oben und unten ein paar Mal und Pipette vorsichtig an der Seite des Schiffes, alle angeschlossenen Zellen freizugeben.
- Die Zellen auf eine 15 mL konische Rohr übertragen und die Zellen spin-down für 5 min bei 250 X g.
- Aspirieren der Trypsin und Aufschwemmen der Zellen in 5-10 mL der Medien. Verwenden Sie eine Pipette P1000 Zelle Klumpen bei Bedarf zu trennen.
- Zählen der Zellen und bestimmen die Lautstärke ~ 300.000 Zellen pro Bohrloch in einem Gesamtvolumen von 3 mL Platte musste. Die Zellen in 2 separaten Vertiefungen ein 6-Well-Platte hinzufügen. Füllen Sie jeweils gut bis 3 mL mit kompletten RPMI.
- Schütteln der Platte alle 5 min. in den ersten 15 min nach der Beschichtung zu gewährleisten, dass die Zellen gleichmäßig auf der Platte verteilt sind. Verwenden Sie ein Mikroskop, um sicherzustellen, dass die Zellen aus der Mitte des Brunnens zerstreut haben.
- Innerhalb von 24 Stunden nach der Beschichtung führen Sie die folgenden Transfektionen mit einer entsprechenden Transfection Reagens für die Zell-Linie von Interesse. Gehen Sie für Ishikawa Zellen vor wie nachfolgend beschrieben.
Hinweis: Dieses Protokoll nimmt die Verwendung von Kationischen Liposomen-basiertes Transfektion Reagenzien. Electroporation bietet eine alternative Methode für Zelltypen, die sehr empfindlich auf diese Reagenzien oder das haben niedrige Transfektion Wirkungsgrad mit Lipofektion. Transfektion Bedingungen müssen für die Zell-Linie von Interesse vor dem Versuch Enhancer-i Experimente optimiert werden.- In eine 1,7 mL Eppendorf Tube, verdünnen Sie 2,5 μg des SID4X-dCas9-KRAB Plasmid und 800 ng Plasmid ein fluoreszierendes Protein in serumfreien Medien auszudrücken, so dass das Endvolumen in der Röhre 155 µL ist und die Endkonzentration von Plasmid 0,020 µg/µL.
- Verdünnen Sie in einem anderen Gefäß 3,3 µg ein Plasmid, das enthält keine Neomycin Widerstand Kassette, wie pCMV-GFP, in serumfreien Medien so, dass das Endvolumen im Rohr 155 µL und die Endkonzentration von Plasmid 0,020 µg/µL.
- Vortex jedes Rohr kurz und spin-down mit einem Microfuge.
- Jedes Rohr 9,9 µL des Transfection Reagens (Table of Materials) hinzufügen. Mischen Sie durch Vortexen kurz bei niedriger Drehzahl. Spin-down der Röhren mit einem Microfuge.
- Inkubieren Sie die Rohre bei Raumtemperatur für mindestens 5 Minuten, aber nicht mehr als 20 min.
- Fügen Sie in der Biosicherheit Kabinett 150 µL des vorbereiteten DNA: Reagenz Mix tropfenweise einen Brunnen auf der 6-Well-Platte hinzu. Wiederholen Sie für die zweite Röhre des vorbereiteten DNA: Reagenz-Mix. Mischen Sie die Platten durch Umschwenken vorsichtig und wieder die Platte in den Inkubator.
- Am 2. Tag Post Transfektion wechseln Sie das Medium und ergänzen mit G418, eine Endkonzentration von 600 ng/μl. Diese Konzentration kann für den Zelltyp optimiert werden müssen.
- Komplette RPMI Medium wechseln und mit G418 täglich für 2-4 Wochen bis die transfizierten Kontrollzellen tot sind und die Brunnen mit SID4X-dCas9-KRABBE konfluierende werden ergänzen. Der genaue Betrag der Zeitaufwand für die Zellen erholen hängt die Verdopplungszeit der Zellen.
- Wenn die Zellen werden konfluierende, Durchgang zu einem t-25 oder T-75 Schiff in RPMI samt eine niedrigere Dosierung des G418 (300 ng/μl für Ishikawa-Zellen). Während dieser Passage machen Sie 2 Aliquote ~ 100.000 Zellen bzw. jedes (etwa 1/10th einer 6-Well-Platte) in 2 separaten 1,7 mL Eppendorf Tubes für RNA und DNA-Isolation. Drehen Sie diese Rohre nach unten (5 min, 250 X g), entfernen Sie Trypsin durch pipettieren und frieren Sie der Rohre bei-20 ° C für eine spätere Verwendung ein.
- Genomischen DNA mit handelsüblichen Kits zu isolieren und PCR unter Verwendung der "pAC95_PCR" oder "SID4X_PCR" Primer (Tabelle 1) um das Vorhandensein des Fusionsproteins innerhalb überprüfen die Zell-Linie führen. Verwenden Sie genomische DNA extrahiert aus der elterlichen Linie als Negativkontrolle und SID4x-dCas9-KRAB Plasmid DNA als Positivkontrolle. Verwenden Sie einen High Fidelity Polymerase-master-Mix mit 50-100 ng von genomischer DNA und die folgenden Bedingungen, Radsport: 98 ° C für 30 s, 25 Zyklen (98 ° C für 10 s, 58 ° C für 30 s, 72 ° C für 2 min), 72 ° C für 5 min , bei 4 ° c zu halten
- Um den Ausdruck des Fusionsproteins auf RNA-Ebene zu überprüfen, führen Sie qPCR mit RNA aus der Zell-Linie mit handelsüblichen Kits extrahiert. Verwenden Sie die "dCas9_qPCR" Primer (Tabelle 1) und das einstufige qPCR-Protokoll in Schritt 6.3 dieses Protokolls zur Verfügung gestellt.
- Um die Protein-Ebene Ausdruck der Verschmelzung zu überprüfen, führen Sie einen Western Blot auf Lysaten aus der Zell-Linie. Verwenden Sie Anti-FLAG oder Anti-HA Antikörper, die Schmelzverfahren Protein zu erkennen.
(2) Guide RNA Design
Hinweis: Dieses Protokoll ist für den Einsatz mit der U6 Guide RNA klonen Vektor von der Kirche Lab erstellt und verfügbar auf Addgene (Addgene 41824) ausgelegt. Erstellen Sie eine Version dieses Vektors mit Puromycin Widerstand, der für die gleiche Klonen Strategie als 41824 erlaubt, zogen wir die Site mit mehreren Klonen von dieser Vektor in den pGL3-U6-SgRNA-PGK-Puromycin-Vektor (Addgene 51133). Addgene 41824 oder unsere Version mit Puromycin (Addgene 106404) sind kompatibel mit der unten beschriebenen Klonen-Strategie.
- 600-900 Basenpaaren der DNA-Sequenz für jede regulatorischen Region von Interesse zu erhalten. Verwenden Sie Transkription Faktor Bindungsstellen und/oder Chromatin Zugänglichkeit für Hinweise, wo man die Region von Interesse (Abbildung 2A) zu definieren.
Hinweis: Während das Beispiel in Abbildung 2A vor- und nachgelagerten Enhancer verfügt, ist es auch möglich, regulatorische Elemente im Umkreis von Introns. - Legen Sie alle Sequenzen in einer Textdatei im FASTA-Format erhalten.
- Identifizieren Sie mindestens eine Negativkontrolle Region, die voraussichtlich nicht über experimentellen Bedingungen, wie ein Promotor eines Gens zu ändern, die nicht in die Zell-Linie von Interesse zum Ausdruck kommt. DNA-Sequenz für diese Region zu erhalten und der Textdatei im FASTA-Format hinzufügen.
Hinweis: Wir verwenden Guide RNAs targeting IL1RN Veranstalter25 als ein negativ-Kontrolle für alle Regionen, in denen, die wir gezielt. Benutzer können auch intergenetischer Sequenz in der Nähe des Interessenbereichs auswählen, die keine Transkription Faktor Bindungsstellen als Negativkontrolle enthält. Wenn mehrere Loci gleichzeitig angesprochen werden, vereinfacht eine einzelne negative Kontrollregion jedoch, Versuchsplanung und Interpretation der Ergebnisse. Wenn die gezielte Enhancer intronischen ist, kann es sinnvoll zum Ziel sein, auf den gleichen Locus, der kein vermeintlichen regulatorisches Element als eine zusätzliche negative Kontrolle, als die dCas9 Fusion enthält eine intronischen Region Transkription stören kann. - Positivkontrolle Regionen, z. B. die Promotoren, die den vermeintlichen Ziele der regulatorischen Regionen von Interesse sind oder Promotoren von Genen, die hoch in die Zell-Linie von Interesse transkribiert sind zu identifizieren. DNA-Sequenz für diese Regionen erhalten und der Textdatei im FASTA-Format hinzufügen.
- Verwenden Sie ein Programm wie z. B. e-knackig26 (http://www.e-crisp.org/E-CRISP/) auf den DNA-Sequenzen generiert, um Guide RNAs mit niedrigen off-Ziele (im Idealfall 0-3) zu finden. Guide-RNAs bestehen aus 20 Nukleotide stromaufwärts von ein Protospacer angrenzenden Motiv (PAM), die die Form "NGG" für die dCas9 von S. Pyogenes.
- Wählen Sie auf der Website von e-knackig den Organismus mit der Drop-Down-Menü. Die Genom-Versammlung erscheint rechts neben der Artname.
- Aktivieren Sie das Optionsfeld Eingabe ist FASTA-Sequenz . Kopieren Sie die FASTA-Sequenzen von oben und fügen Sie sie in das Dialogfeld. Sicherstellen Sie, dass ein FASTA-Header für jede Sequenz enthalten ist.
Hinweis: Bis zu 50 Sequenzen können gleichzeitig abgefragt werden. - Wählen Sie das Optionsfeld " Medium " und einzelne Design in der Drop-Down-Menü.
- Klicken Sie auf SgRNA Suche starten. Ein neuen Browser-Tab öffnet, und Ergebnisse werden angezeigt. Download der Kandidaten-Sequenzen durch Anklicken des Buttons herunterladen einen Excel formatiert tabellarischen Bericht für alle Abfrage-Sequenzen zusammen.
- Öffnen Sie den tabellarischen Bericht mit Excel oder ein Textbearbeitungsprogramm.
- Verwenden der UCSC Genom Browser BLAT Kandidat in voller Länge gRNA Sequenzen (23 Basenpaaren), das Genom.
- Navigieren Sie in einem Browser zu der UCSC Genom-Browser-Website (http://genome.ucsc.edu). Suchen Sie unter dem Abschnitt unserer Werkzeugedas Wort BLAT und klicken Sie darauf. Das BLAT-Such-Tool öffnet sich.
- Verwenden Sie die Dropdown-Menüs befindet sich unter den BLAT Suche Genom -Text, um den Organismus und Genom Montage von Interesse auszuwählen.
- Kopieren Sie der Guide RNA-Sequenzen aus dem tabellarischen Bericht generiert von e-Crisp und fügen Sie sie in das Dialogfeld. Stellen Sie sicher, dass jede Sequenz hat einen einzigartige FASTA-Header senden am unteren Rand des Dialogfelds klicken.
Hinweis: Bis zu 25 Sequenzen können gleichzeitig geprüft werden. Auf der Seite BLAT Suchergebnisse erscheint Ausrichtungen von jeder Guide RNA-Sequenz, mit jeder Linie, die eine Achse darstellt. Im Idealfall sollte eine Achse für jede Anleitung RNA, zeigt die Einzigartigkeit dieses Leitfadens RNA. - Vermeiden Sie die Führer, die mit mehreren Speicherorten im Genom richten Sie wenn möglich.
- Um Guide RNA Lokalisation und Verteilung in der Region von Interesse zu untersuchen, klicken Sie auf den Browser -Link unter dem Abschnitt Maßnahmen für eines der abgefragten Guide-RNAs. Die Genom-Browser erscheint und wird auf den ausgewählten Guide RNA zentriert. Verwenden Sie die Schaltflächen verkleinern, am oberen Rand der Seite, um die Verteilung der anderen Führer zu visualisieren, RNAs von e-Crisp in der Region von Interesse identifiziert.
- Wählen Sie aus 4 vorzugsweise nicht überlappende Guide RNA-Sequenzen, die in der gesamten Region von Interesse (Abb. 2 b) verteilt werden. Überschreitet die Region von Interesse 600 bp, erwägen, 1-2 zusätzliche Führungen. Vermeiden Sie Guide RNAs mit Homopolymeric Dehnungen und extreme GC-Gehalt, wie diese Features Guide RNA Klonen Prozess behindern und Guide RNA Ausrichtung auf Effizienz verringern können.
- Sobald die Hilfslinien ausgewählt haben, erstellen Sie eine Datei mit den vollständigen Guide RNA-Sequenz (23 Nukleotide) für jede gewünschte Anleitung, und entfernen Sie dann die 5' Nukleotid sowie die PAM (NGG) aus dem 3'-Ende. Dieser Schritt erleichtert die Bestellung von Oligo.
- Die folgende Sequenz 5'-Ende der Oligonukleotid Sequenz hinzufügen: GTGGAAAGGACGAAACACCG.
- Fügen Sie die folgende Sequenz am 3' Ende der Oligonukleotid-Sequenz: GTTTTAGAGCTAGAAATAGC.
Hinweis: Die Schluss-Sequenz sollte sein 59 Nukleotide lang und schauen Sie wie folgt: GTGGAAAGGACGAAACACCG-Ziel (19 nt)-GTTTTAGAGCTAGAAATAGC. - Sicherstellen Sie, dass jeder regulatorisches Element mit Enhancer-i ausgerichtet werden mindestens 4 einzigartige Oligonukleotide, die für sie entworfen hat. Bestellen Sie diese Sequenzen zusammen mit der "U6_internal" Primer in Tabelle 1aufgeführt.
3. führen Sie RNA Klonen
Hinweis: Guide RNA Klonen über Gibson Montage erweist sich als äußerst effizient in unseren Händen, wodurch hunderte von Kolonien pro Platte, mit vorhanden in der Vektorkontrolle nur wenige oder gar keine Kolonien. Diese Effizienz ist entscheidend für die Aufrechterhaltung der Komplexität beim gepoolten Klonen. Ein weiterer Vorteil der Gibson Versammlung Klonen ist, dass Benutzer nicht über die Anwesenheit von ein Restriktionsenzym geschnitten Website im Guide RNA, die sie versuchen, Einfügen in die U6 klonen Vektor sorgen. Dennoch kann dieses Protokoll für traditionelle Restriktionsenzym basierte Klonen auf Wunsch angepasst werden.
- Rekonstruieren Sie Führer RNS Oligos auf eine Endkonzentration von 100 μM Reinstwasser (RNase-frei, DNase). Es sollte mindestens 4 separate Anleitung RNS Oligos für jede Region von Interesse.
- Erstellen Sie für jede regulatorischen Region von Interesse einen Pool von allen Oligos entspricht der Region von Interesse. Verbinden Sie in einem Eppendorf-Rohr 5 μL jeder rekonstituierte Einzelführung RNA Oligo für jede Region. Mischen des Pools gut aufschütteln, dann entfernen Sie 1 μL und verdünnen dieser Aliquote 1: 200 in Reinstwasser.
Hinweis: Falls gewünscht, können diese Pools gezielt einzelne regulatorische Regionen weiter kombiniert werden um einen komplexen Pool für mehrere Regionen zu generieren. Bis zu 50 regulatorische Regionen können gleichzeitig in einem einzigen Pool (Abbildung 3) ausgerichtet sein. - Führen Sie eine kurze PCR mit der U6-Primern um Homologie Regionen Oligos vor der Gibson Montage anzubringen. Etwa 40 Basen werden jede Oligo, wodurch ein ~ 100 bp Produkt mit ausreichend Homologie zu den U6-Vektor an beiden Enden hinzugefügt werden.
- Für jede Anleitung RNA-Pool Einrichten einer 20 μL PCR mit einem High Fidelity Polymerase-master-Mix und die folgenden Komponenten: 1 μl verdünnter Oligo-Pool vom Schritt 3.2, 1 μL der U6 forward Primer (10 μM), 1 μL der U6 reverse Primer (10 μM) , und Wasser bis zu 20 μL.
- Brüten in einem Thermocycler mit den folgenden Bedingungen: 98 ° C für 30 s, 10 Zyklen (98 ° C für 10 s, 55 ° C für 30 s, 72 ° C für 2 min), 72 ° C für 5 min und Halt bei 4 ° C.
- Laufen Sie 5 μL der Reaktion auf eine 1-2 % Agarosegel mit niedrigem Molekulargewicht Leiter. Das Endprodukt sollte ~ 100 Basenpaaren (Abbildung 2).
- Bereinigen Sie die Erweiterung Reaktion mit einem spaltenbasierten DNA Reinigung Kit und eluieren in 20 μL der Elution Puffer im Kit enthalten.
Hinweis: Da das Produkt kurz ist, vermeiden Sie die Verwendung Wulst-basierte Bereinigungen, die kleine Fragmente von weniger als 100 ausschließen sollen bp. - Die gereinigte DNA mit einem Fluorometer oder Spektralphotometer zu quantifizieren (erwartete Ertrag liegt bei 10-20 ng/μl). Guide RNA-Einsätze können bei-20 ° C gelagert werden oder sofort in Gibson-Baugruppe mit einem linearisierten U6 Vektor eingesetzt werden.
- Um den Empfänger U6 klonen Vektor für Gibson Versammlung vorzubereiten, richten Sie ein Restriktionsenzym verdauen. Wenn viele Gibson Montage Reaktionen durchgeführt werden, richten Sie mehrere Übersichten um ausreichende Ausbeute an geschnittenen Vektor sicherzustellen.
- Verwenden Sie 20 Einheiten des AflII Enzyms und 1 µg Plasmid in 20 μL Reaktion mit dem entsprechenden Restriktionsenzym Puffer. Inkubation bei 37 ° C für 1-2 h.
- Bereinigen der Digest mit Perlen oder einem spaltenbasierten Kit für DNA-Reinigung und eluieren in 20 μL der Elution Puffer. Quantifizieren Sie die gereinigte DNA mit einem Fluorometer oder Spektralphotometer. Proben können bei-20 ° C für den späteren Gebrauch eingefroren werden.
- Führen Sie Gibson Montage auf dem vorbereiteten Vektor und einfügen.
- Richten Sie Gibson Montage Reaktionen auf Eis. Einsatz 50 ng des Vektors und 7 ng des Einsatzes in einer 20 μL Reaktion. Verdünnen Sie die Einsätze 01:10 in Reinstwasser, pipettieren zu erleichtern. Richten Sie einen Vektor nur Gibson Montage-Reaktion, mit 50 ng des Vektors und die Einlage durch Wasser ersetzen.
- Inkubieren Sie die Gibson Montage Reaktionen für 15 min bei 50 ° C, gefolgt von einen Halt bei 4 ° c
- Übertragen der zusammengesetzten Produkte zu Eis. Verdünnen Sie die zusammengesetzten Produkte 1:4 in Reinstwasser auf Eis. Z. B. Fügen Sie 5 μL der Gibson Montage Produkt 15 μl von Reinstwasser hinzu.
- Die verdünnte Gibson Montage Produkte zu verwandeln.
- Hohe Effizienz kompetente Zellen auf Eis Auftauen und 25 μl-Aliquots für jede Transformation zu machen. Auf Wunsch ein komplexen Pool für mehrere Standorte ist tauen Sie genügend Zellen in verschiedene Leitungen, mehrere unabhängige Transformationen des gleichen Komplexes gRNA Pools durchzuführen auf.
- Fügen Sie für jedes verdünnten Produkt 1 μl dieser Verdünnung eine 1,7 mL Eppendorf-Röhrchen mit 25 μl kompetente Zellen hinzu. Mischen Sie, indem Sie kurz das Rohr streichen. Inkubieren Sie die Rohre für 30 min auf Eis.
- Hitze Schock die Zellen für 30 s bei 42 ° C, dann sofort um zu Eis für 2 min übertragen.
- 300 μl SOC Media (2 % Tryptone, 0,5 % Hefeextrakt, 10 mM NaCl, 2,5 mM KCl, 10 mM MgCl2, 10 mM MgSO4und 20 mM Glukose) und lassen Sie die Zellen für 1 h bei 37 ° C mit schütteln (300 u/min) zu erholen. Während dieser Zeit warme Agarplatten mit Ampicillin/Carbenicillin auf 37 ° C im Brutschrank. Verwenden Sie eine Platte für jede Transformation.
- Platte 50 μL der Zellen und legen die Platten in einem Inkubator 37 ° C über Nacht. Legen Sie für die Ausrichtung auf einzelne Standorte-Pools direkt in 3-5 mL LB Brühe (Table of Materials) mit Ampicillin/Carbenicillin (1 mg/mL) und inkubieren Sie über Nacht mit schütteln bei 250 u/min bei 37 ° C für Minipreps.
- Ernten der Zellen und DNA zu isolieren.
- Verwenden Sie für große Führer RNA Bibliotheken auf mehrere Standorte Schabeisen Platte, um alle Kolonien von jeder einzelnen Platte in eine Maxiprep (150 mL Flüssigkultur) zu sammeln. Dies kann durch Gießen ~ 5 mL LB mit dem entsprechenden Antibiotikum in ein 50 mL Falcon-Röhrchen und kratzen die Kolonien in das Rohr erleichtert werden. Kratzen Sie für Bibliotheken für einzelne Websites die Platten in eine Miniprep (3-5 mL Flüssigkultur).
- Inkubieren Sie diese Kulturen mit dem entsprechenden Antibiotikum für 3-5 h bei 37 ° C mit schütteln bei 250 u/min.
- Durchführen Sie DNA-Extraktion mit einem Kit, die Ergebnisse in Endotoxin-freie Preps.
- DNA mit einem Fluorometer oder Spektralphotometer zu quantifizieren. Plasmide können in Transfektion sofort eingesetzt oder bei-20 ° C für eine spätere Verwendung gespeichert werden.
- Das Vorhandensein Guide RNA Sequenz innerhalb des Vektors U6 bestätigen für kleine Pools, die gezielt einzelne Websites verwenden Sanger Sequenzierung auf die vorbereiteten Miniprep mit der "U6_PCR_R" Primer in Tabelle 1aufgeführten. Durch Bündelung der Guide RNAs, die 19 Länge gRNA Zielsequenz führen zu gemischte Basen, aber U6 Förderer und Anleitung RNA Gerüst rund um diese Sequenz sollte intakt sein.
4. die Transfektion von Enhancer-i
Hinweis: Für die erfolgreiche Blockade einer Östrogen-Antwort mit Enhancer-i in Ishikawa Zellen, ist es notwendig, die Zellen von Östrogen zu entziehen, für 5-7 Tage vor Transfektion von Phenol rot warten kostenlose RPMI mit 10 % Kohle-FBS und 1 % abgestreift Penicillin/Streptomycin. Zellen sollten in diesem Medium während und nach der Transfektion kultiviert werden, wenn versuchen, eine Antwort von Östrogen blockieren. Wir empfehlen die Verwendung von Phenol rot frei Trypsin für die Passage von Zellen in komplette Phenol rot kostenlos RPMI.
- Am Vortag Transfection Platte Zellen (Wildtyp oder stabil mit dem Ausdruck SID4X-dCas9-KRABBE) in einer 24-Well-Platte bei 30-50 % Konfluenz (~ 60.000 Zellen pro Bohrloch für Ishikawa-Zellen). Platte genügend Zellen, so dass Transfektionen in zweifacher Ausfertigung durchgeführt werden können und Brunnen beinhalten um mit Kontrolle Guide RNAs transfiziert. Stellen Sie sicher, dass die Zellen gleichmäßig über die gut verteilt sind, durch sanft schütteln die Platte nach Zelle Beschichtung wie in Schritt 1.1.7.
Hinweis: Dieses Protokoll nimmt die Verwendung von Kationischen Liposomen-basiertes Transfektion Reagenzien. Electroporation bietet eine alternative Methode für Zelltypen, die sehr empfindlich auf diese Reagenzien. Transfektion Bedingungen müssen für die Zell-Linie von Interesse vor dem Versuch Enhancer-i Experimente optimiert werden. - Bereiten Sie am nächsten Tag Transfektionen gemäß Anweisungen des Transfection Reagens Wahl. Für Ishikawa Zellen, Verwendung 550 ng des gesamten Plasmid für jede Vertiefung einer 24-Well-Platte. Verdünnen Sie die Plasmide, eine Endkonzentration von 0,020 μg/μL in serumfreien Medien (1,1 µg DNA in 52 μL des Gesamtvolumens für transfecting 2 Brunnen). Verwenden Sie 3 μL Transfection Reagens für jeden 1 µg DNA, Wirbel und inkubieren Sie wie in Schritt 1.2 beschrieben. Jedes gut 25 μL der endgültige Mischung hinzufügen.
Hinweis: Ziel Kombinationen von Websites, verwenden Sie das gleiche Gewicht von Plasmid für jeden einzelnen Standort, und füllen Sie dann das übrig gebliebene Gewicht mit einer Kontrolle Plasmid (leere Guide RNA klonen Vektor oder Guide RNAs, die Ausrichtung einer Negativkontrolle Region wie IL1RN Promotor). Verwenden Sie für transiente Transfektionen ein Verhältnis von 3:2 Cas9 Fusion: Guide RNA Plasmid. Plasmide mit fluoreszierenden Reporter können hinzugefügt werden, um Transfektion Effizienz zu überwachen. - 36 h Post Transfektion, ändern die Medien mit Phenol rot kostenlos RPMI mit 10 % Kohle ausgezogen FBS und 1 % Penicillin/Streptomycin (für Ishikawa-Zellen) und liefern Puromycin (Endkonzentration: 1 μg/mL) und Neomycin (Endkonzentration: 300 ng/mL). Wenn Zellen empfindlich auf Transfection Reagens, die Medien zuvor geändert werden können, aber Antibiotika sollte frühestens 24 h Post Transfektion hinzugefügt werden.
Hinweis: Warten Sie mindestens 24 Stunden nach Zugabe von Antibiotika vor der Ernte der Zellen. Ausdruck Änderungen aufgrund von Enhancer-i können erkannt werden, da bereits im 48 h Transfektion buchen und bis zu 5 Tagen Transfektion. Wenn mit Ishikawa Zellen arbeiten, die von Östrogen beraubt wurden, führen Sie eine 8-h 10 nM 17β-Östradiol (E2) Induktion des Tages nach Behandlung mit Antibiotika und dann ernten Sie Zellen sofort.
5. Handy-Ernte und RNA-Extraktion
- Lyse-Puffer mit 1 % β-Mercaptoethanol (BME) vorzubereiten. Sicherstellen Sie, dass es genügend Lyse-BME-Mischung (300 μL für jedes gut geerntet).
- Aspirieren der Medien mit einer Vakuum-Sauger.
- Waschen Sie die Zellen einmal mit einem gleichen Volumen 1 X PBS (500 μL) und aufziehen, so viel PBS wie möglich zu entfernen.
- Geben Sie 300 μL Lyse-BME-Lösung in jedem mit einer Mehrkanal-Pipette. Pipette die Lyse Lösung oben und unten 8 - 10 Mal und Übertragung auf eine Deep-Well-Platte oder 1,7 mL Eppendorf tubes auf Eis. RNA sofort extrahiert werden kann, oder bei-80 ° C zur weiteren Verarbeitung Lysates eingefroren werden kann.
- Um RNA von Lysaten zu extrahieren, verwenden Sie eine kommerziell erhältliche Kit, die eine DNase-Behandlung beinhaltet. Eluieren in die kleinste empfohlene Volumen von Reinstwasser (RNase-frei, DNase) oder Elution buffer und RNA zu quantifizieren. Verwenden Sie für eine kleine Anzahl von Proben Fluorometer oder Spektralphotometer. Verwenden Sie für eine große Anzahl von Proben eine fluoreszierende Sonde, die RNA und Maßnahme auf eine Platte Leser erkennt. Proben können vor oder nach der Quantifizierung bei-80 ° C eingefroren werden.
(6) Quantifizierung von Veränderungen der Genexpression mit einstufigen qPCR und RNA-seq
- Erhalten Sie qPCR Primer für die Gene von Interesse und für mindestens ein Zimmermädchen-gen, das drückt sich in der Nähe der Ebene gezielt Gene und ändert sich nicht über experimentellen Bedingungen. Im Idealfall werden diese Primer eine Exon-Exon-Junction um Verstärkung der genomic DNA zu vermeiden erstrecken.
- Testen Sie diese Grundierungen auf RNA aus der Zell-Linie von Interesse gewonnen. Verwendung schmelzen Kurvenanalyse, die Produktion eines Produktes zu überprüfen. Wenn ein einzelnes Produkt nicht produziert wird, testen Sie zusätzliche Grundierung Paare.
- Für jede Enhancer-i und Kontrolle Guide RNA behandelt Probe ermitteln Sie wie viele Gene in dieser Probe untersucht werden müssen. Dieser Satz von Genen sollte Housekeeping-Gene, wie CTCF oder GAPDHenthalten.
- QPCR Reaktionen eingerichtet.
- Alle Proben, die gleiche Konzentration im Wasser zu verdünnen, dass 50 ng von Gesamt-RNS ist leicht pipettiert, und es gibt genug verdünnt RNA für jede Reaktion. Zum Beispiel zur ~16.6 ng/μl RNA zu verdünnen und 3 μl RNA in jeder Reaktion verwenden. Halten Sie RNA auf Eis beim Einrichten von master-Mixes.
- Bereiten Sie separate master Mischungen für jedes Gen gemessen werden mit handelsüblichen einstufige qPCR-Kits. Verwenden Sie eine 20 μl Reaktionsvolumen mit 1 μL jeder Grundierung (10 μM Stammlösung). Richten Sie diese Reaktionen auf Eis.
- Fügen Sie in eine Reaktion-Platte, die für die Thermocycler eignet, RNA-Proben, gefolgt von master-Mixes. Mit einer Platte Versiegelung versiegeln und vorsichtig mischen durch aufschütteln oder pipettieren. Die Platte kurz Zentrifugieren (140 X g für 60 s) um sicherzustellen, dass Flüssigkeit ist an der Unterseite der Brunnen.
- Die Platte in einem Thermocycler folgendermaßen inkubieren (oder als Kit weist): 48 ° C für 30 min, 95 ° C für 10 min, 40 Zyklen (95 ° C für 15 s, 60 ° C für 1 min).
- Erhalten Sie Ct-Werte für jedes Gen in jeder Probe gemessen. Verwenden Sie die vergleichende Ct-Methode, um Veränderungen in der Genexpression zu identifizieren.
- Subtrahieren Sie die Ct das Zimmermädchen-gen aus der Ct jedes Gen des Interesses für jede Probe, normalisierte Ct-Werte zu generieren.
- Für die behandelt Kontrollproben dauern Sie durchschnittlich normalisierte Ct-Werte für jedes Gen. Log Basis Maßstab 2 Falte Repression kann dann durch Subtraktion der normalisierten Enhancer-i behandelt Probe Ct für jedes Gen aus den gleichen Wert für die behandelt Kontrollprobe für das entsprechende gen berechnet werden.
- Um globale Änderungen in der Genexpression nach Enhancer-i Behandlung zu bestimmen, bereiten Sie die Proben für RNA-Sequenzierung mit einem kommerziell erhältlichen Kit kompatibel mit der Benutzer-Sequenzierung-Technologie vor. Verwendung ~ 500 ng RNA für Ausgangsmaterial und Bibliotheken für mindestens 2 biologische Wiederholungen vorbereiten.
7. Überprüfung der spezifischen genomischen Ausrichtung durch SID4X-dCas9-KRABBE mit ChIP-seq
Hinweis: Die SID4X-dCas9-KRAB Schmelzverfahren Protein enthält ein FLAG-Epitop-Tag und einem HA-Epitop-Tag, aber beste Ergebnisse für ChIP-Seq wurden mit Anti-FLAG Antikörper. Falls gewünscht, kann der Benutzer zusätzliche ChIP-Seq-Experimente durchführen, für Transkriptionsfaktoren, die potenziell betroffenen von Enhancer-i oder H3K27ac, eine Marke der Enhancer-Aktivität, die von Enhancer-i verringert wird. Jeder ChIP-Seq-Experiment erfordert jedoch 10 x 106 Zellen, also Plan entsprechend.
- Transfizieren Sie Zellen mit Enhancer-i Pools.
- Platte 10 x 106 Zellen in der Kulturschale 15 cm Gewebe. Jedes Gericht vertritt 1 ChIP-Seq-Experiment für 1 Faktor von Interesse.
- Transfizieren Sie am Folgetag die Zellen mit der Gesamt-DNA 20 μg pro Schale. Für Transfektionen in Zelllinien stabil SID4X-dCas9-KRABBE zum Ausdruck zu bringen sollte die DNA ein Plasmid-Pool des Guide RNAs, die Ausrichtung auf alle Seiten von Interesse sein, und optional ein Plasmid mit dem Ausdruck fluoreszierenden Proteins. Verwenden Sie für transiente Transfektionen ein Verhältnis von 3:2 dCas9 Fusion Protein: Guide RNA Pool. Führen Sie für ChIP-Seq anderer TFs oder Histon-Modifikationen mindestens eine zusätzliche Kontrolle Guide RNA Transfektion auf ein anderes Gericht.
- Behandeln Sie die Gerichte mit Puromycin (1 μg/mL) und Neomycin (300 ng/mL) in 24-48 h Post Transfektion. Warten Sie mindestens 24 Stunden vor der Ernte Chromatin.
- Chromatin von Gerichten zu ernten.
Hinweis: Studie über die Auswirkungen der Enhancer-i ER genomische Bindung in Ishikawa Zellen, führen Sie eine 1 h 10 nM E2 Behandlung auf Gerichte mit Kontrolle Guide RNAs und Enhancer-i vor Ernte transfiziert. Studie über die Auswirkungen von Enhancer-i auf H3K27ac, führen Sie eine 8 h 10 nM E2 Induktion auf Gerichte transfiziert mit Kontrolle Guide RNAs und Enhancer-i vor Ernte.- Wenden Sie 500 μL von 37 % Formaldehyd auf jedes Gericht (Endkonzentration von 1 %). Schwenken Sie die Platten kurz. Lassen Sie die Platten bei Raumtemperatur für 10 min sitzen.
- Fügen Sie 1 mL 2,5 M Glycin (Endkonzentration von 125 mM). Schwenken Sie die Platten kurz.
- Gießen Sie die Medien mit Formaldehyd und Glycin. Fügen Sie ein gleiches Volumen (~ 20 mL) kalt 1 X PBS.
- Gießen Sie die PBS. Aspirieren Sie mit Vakuum-Sauger, soviel PBS wie möglich zu entfernen. Legen Sie die Gerichte auf Eis.
- Fügen Sie 3-5 mL kalten 1 x PBS oder Farnham Lyse-Puffer (5mM Rohre pH 8.0, 85 mM KCl, 0,5 % NP-40) mit 1 x Protease-Hemmer (kurz vor dem Einsatz hinzugefügt) auf jede Platte. Kratzen Sie die Schüssel mit einem Teller Schaber und übertragen Sie die Lösung auf eine 15 mL konische Rohr auf Eis.
- Pellet-Chromatin durch Spinning Festlegung Rohre in einer Zentrifuge für 5 min bei 4 ° C bei 1000 X g. verwerfen des Überstands und die Pellets bei-80 ° C für eine spätere Verwendung zu speichern, oder fahren Sie mit dem ChIP-Seq-Protokoll der Wahl mit einem Anti-FLAG Antikörper oder Antikörper gezielt andere Transkriptionsfaktoren oder Histon-Modifikationen von Interesse (H3K27ac, H3K9me3).
Ergebnisse
Abbildung 1 zeigt eine schematische Darstellung des Workflows im Protokoll beschrieben. Um festzustellen, dass die Beiträge von ER-gebundenen Enhancer in der Nähe von Östrogen-regulierten Gens MMP17, hat 3 Bindungsstellen in der Nähe im Sinne von ChIP-Seq (Abbildung 2A) und RNAs leiten wurden für jede Region entwickelt. Design Guide RNAs, ein 600-900 bp Fenster rund um jede ER-Sequenz wurde Bindungsstelle von Interesse ausgewählt und ein Führer-RNA-Design-Programm. Resultierenden Guide Sequenzen RNA mit 0-2 vorausgesagt vom Zielwert, die Seiten des menschlichen Genoms mit BLAT ausgerichtet waren. Vier nicht-überlappende guide RNAs, die durch ChIP-Seq definierten Bereichs überspannt und DNaseI Überempfindlichkeit wurden ausgewählt, für die Ausrichtung (Abb. 2 b). Zusätzliche Sequenz (Tabelle 1) wurde hinzugefügt, um jedem Ende Erleichterung der nachgeschalteten Klonen und die daraus resultierenden bestellte waren 59 Nukleotid-Fragmente. Bei der Ankunft war Guide RNAs verdünnt und gebündelt durch die Website und eine kurze PCR wurden durchgeführt, um Homologie Regionen vor der Gibson-Montage hinzufügen. Abbildung 2 zeigt die erwarteten Guide RNA Produkt nach einer kurzen PCR unter Verwendung der "U6_internal" Zündkapseln (Tabelle 1), die an jedem Ende der 59 Länge 20 Basenpaaren Sequenz hinzufügen guide RNA Fragment, was in einer Länge von ~ 100-Sequenz. Anschluss an Gibson Vollversammlung diese Anleitung RNA-Pools verwandelten sich in Bakterien und Plasmid Minipreps waren am nächsten Tag vorbereitet. Abb. 2D zeigt Ergebnisse aus einem Enhancer Dissektion Experiment, wo mehrere Enhancer in der Nähe MMP17 allein ausgerichtet sind und in Kombination mit Enhancer-i. Seiten gezielt durch Enhancer-i sind mit einem schwarzen Sechseck gekennzeichnet. Guide RNA Plasmide Ausrichtung auf den angegebenen Seiten wurden in einer Östrogen-beraubt Ishikawa Zelllinie stabil mit dem Ausdruck SID4X-dCas9-KRAB transfiziert. Zwei Tage später änderte sich die Medien und Puromycin wurde hinzugefügt, um für transfizierte Zellen anzureichern. Am folgenden Tag wurden die Zellen geerntet, nach einem 8 h 10 nM Estradiol Behandlung. RNA wurde isoliert und eine einstufige qPCR erfolgte. In diesem Beispiel sind Seiten 1 und 2 erforderlich für eine vollständige Östrogene Antwort des MMP17, während Seite 3 nicht unter diesen Bedingungen (Abb. 2D, Fahrspuren Ii-iv) beiträgt. Wenn nur die Seiten 2 oder 3 aktiv sind (vi und Vii), die Östrogen-Reaktion ist ähnlich wie wenn keine Standorte sind aktiv (Viii), was darauf hindeutet, dass diese Seiten nicht selbständig beitragen. Seite 1 kann ein Ausdruck von selbst (V) beitragen, aber die größte Aktivität wird gesehen, wenn die Seiten 1 und 2 aktiv sind (iv).
10-Enhancer in der Nähe von 4 verschiedenen Genen gleichzeitig bearbeiten (Abbildung 3A), führen komplexe Pools von RNAs mit 42 Enhancer-Guides und 16 Veranstalter Guides generiert wurden. Anleitung RNS Oligos wurden vor der ersten Guide RNA Verlängerung PCR (Schritt 3.3) zusammengefasst und daraus resultierende PCR-Produkte wurden gereinigt und in Kombination mit der leeren Puromycin U6 klonen Vektor mit Gibson Assembly. Anschluss an die Vollversammlung Gibson wurden mehrere unabhängige Transformationen durchgeführt und vernickelt. Die Platten wurden in LB geschabt und erlaubt zu wachsen, für 2-4 h vor Maxiprep. Abbildung 3 b zeigt repräsentative Reduktionen in der Genexpression von qPCR wenn diese Anleitung RNA Pools waren in einer Östrogen-beraubt Ishikawa Zelllinie stabil mit dem Ausdruck SID4X-dCas9-KRAB transfiziert und behandelt wie beschrieben (Abb. 2D). Reduzierungen von Enhancer-i sind ähnlich denen, die durch die Ausrichtung des Projektträgers des vermeintlichen Zielgens. Abbildung 3 zeigt die Auswirkungen der Verdünnung des Guide RNAs auf Reduzierung der Östrogen-Antwort mit Enhancer-i. Eine 01:50, die Verdünnung eines Guide RNA-Pools, die Ausrichtung der Enhancer in der Nähe von G0S2 noch deutliche Reduzierung der Genexpression ergibt, was darauf hindeutet, dass Enhancer-i verwendet werden kann bis zu 50 Seiten auf einmal. Jedoch kann die Deaktivierung, verdünnt werden, darauf hinweist, dass Hunderte von Websites können nicht gleichzeitig ausgerichtet werden, es sei denn, empfindlichere Nachweisverfahren beschäftigt sind.

Abbildung 1: Protokoll für Multiplex-Enhancer Dissektion mit Enhancer-i. schematische Guide RNAs (rot und blau) sind mit e-knackig und ausgewählten Genom der UCSC Browser gestaltet. Vier Guide RNAs sind gewählt, die die Regionen von Interesse (Transkription Faktor Bindungsstellen definiert durch ChIP-Seq) umspannen. Guide RNA-Oligonukleotide, die von der Region von Interesse (rot und blau) gebündelt haben durchlaufen eine PCR um Homologie Regionen (Orange) vor der Gibson Montage und Transformation hinzuzufügen. Daraus resultierende Plasmid-Pools sind über Lipofektion in Zelllinien stabil mit dem Ausdruck SID4X-dCas9-KRABBE oder in Wildtyp Zellen in Verbindung mit SID4X-dCas9-KRAB Plasmid transfiziert. Guide RNA Plasmid Becken können individuell transfiziert werden, um eine Seite zu einem Zeitpunkt oder in Kombination mit mehreren Standorten gleichzeitig gezielt ansprechen. Transfizierte Zellen sind mit Antibiotika zu bereichern für Zellen mit Guide RNAs behandelt. ~ 72 h Post Transfektion sind die Zellen geerntet. Nukleinsäuren extrahiert werden können, für qPCR, RNA-Seq oder ChIP-Seq Klicken Sie bitte hier, um eine größere Version dieser Figur.
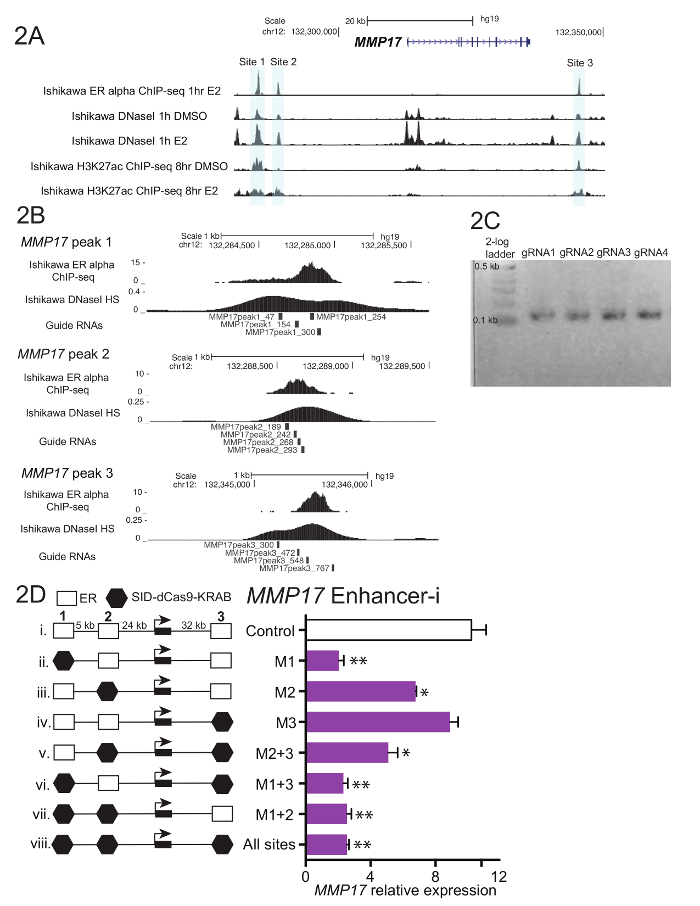
Abbildung 2. Führen RNA Design und Enhancer Dissektion für MMP17. (A) Genom Browser Screenshot von ER Alpha-gebundenen Enhancer (grau) in der Nähe von zielgerichtet sein MMP17. Diese Zahl wurde von Carleton, Et Al. modifiziert 18. (B) Guide RNA entwirft für die 3 Seiten18verbindlich. Die Bindungsstelle für ER definiert durch ChIP-Seq ist das Ziel und die 4 Guide RNAs Fliese in dieser Region. Die DNaseI Empfindlichkeit Signal, das die Bindungsstelle erstreckt, kann auch zur Zielsequenz für Guide RNA Design definieren. ChIP-Seq und DNaseI HS Daten stammen aus Ishikawa Zellen behandelt mit 10 nM Estradiol für 1 h (C) repräsentative Guide RNA Sequenzen, die montagebereit Gibson, eine kurze PCR Homologie Regionen hinzufügen unterzogen worden sind. (D) Relative Expression von MMP17 über qPCR nach Ausrichtung der einzelnen Regionen mit Enhancer-i und eine 8-h10 nM Estradiol Behandlung gemessen. Ausdruck ist relativ CTCF und Ausdruck Ebene der MMP17 in den Zellen nicht mit Estradiol behandelt. Kontrolle Guide RNAs der Veranstalter des Ziel IL1RN. Alle Fehlerbalken darzustellen SEM, doppelte Sternchen geben p < 0,01 und einzelne Sternchen geben p < 0,05 in ein gepaarter t-Test. Diese Zahl wurde von Carleton, geändert Et Al. 18. Bitte klicken Sie hier für eine größere Version dieser Figur.
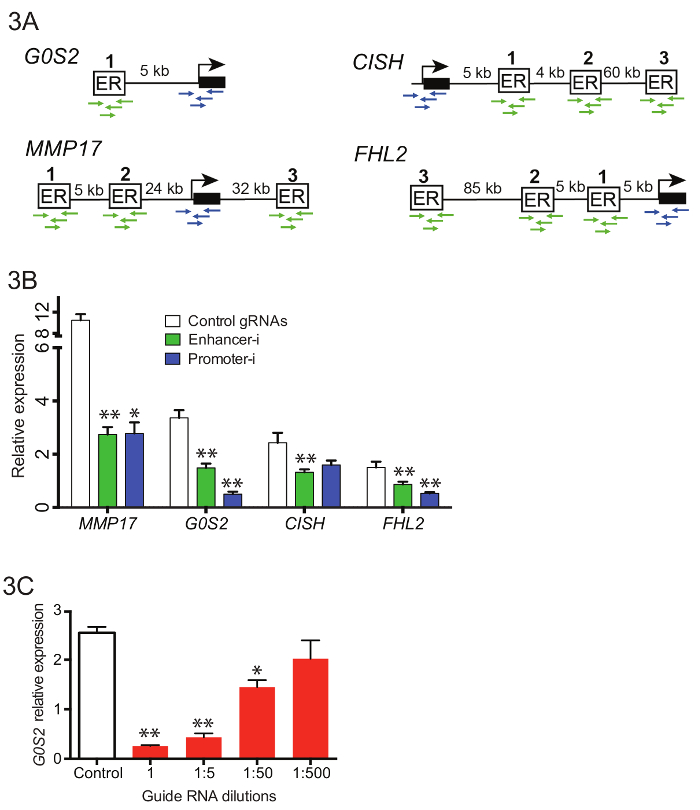
Abbildung 3. Ausrichtung auf mehrere Enhancer in der Nähe von verschiedenen Genen gleichzeitig mit gebündelt Enhancer-i. (A) schematische Darstellung der Bindungsstellen und Promotoren in gepoolten Enhancer-i ausgerichtet werden. (B) die Auswirkungen auf Ausdruck qPCR gemessen, nachdem E2 Behandlung auf Ishikawa Zellen mit Enhancer-i Plasmid Pool (grün), Promoter-i Plasmid Pool (blau) oder Kontrolle gRNAs (weiß)18transfiziert. Eine signifikante Reduktion bei aller Gene wird mit Enhancer-i beobachtet. Diese Zahl wurde modifiziert von Carleton, Et Al. 18. (C) die Effekte auf G0S2 Ausdruck Ebenen nach E2 Behandlung auf Ishikawa Zellen transfiziert mit unterschiedlichen Mengen an Guide RNAs gezielt G0S2. Eine signifikante Reduktion kann man schon mit kleinen Beträgen Guide RNA (01:50 Verdünnung), darauf hindeutet, dass bis zu 50 Seiten kann gleichzeitig ausgerichtet. Alle Fehlerbalken darzustellen SEM, doppelte Sternchen geben p < 0,01 und einzelne Sternchen geben p < 0,05 in ein gepaarter t-Test. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Name | Sequenz |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
Tabelle 1. Primer für RNA Verlängerung und Sequenzierung, qPCR und Erkennung des Fusionsproteins verwendet.
Diskussion
Dieses Protokoll beschreibt eine einfache und flexible Methode für sezieren Enhancer-Funktion bei der endogenen genomischen Lokus ohne physisch verändern die DNA-Sequenz. Zwar ähnlich im Konzept zuvor veröffentlichten CRISPR-Störungen-Protokolle mit dCas9-KRAB27, unterscheidet Enhancer-i sich diese Protokolle auf 3 Arten. Erstens nutzt Enhancer-i die SIN3A interagierende Domäne von MAD120 Enhancer Deaktivierung zu erreichen. Enhancer Deaktivierung kann gerettet werden, mit HDAC-Inhibitoren, was darauf hindeutet, dass der primäre Mechanismus der Deaktivierung HDAC angewiesen ist. Im Gegensatz zu CRISPR Störungen mit dCas9-KRABBE führen Enhancer-i nicht zur Ablagerung von H3K9me3. Dies dürfte die Enhancer-i stützt sich auf vorübergehende Einführung des Leitfadens, die RNAs, Transfektion mit den Zellen geerntet an 3 Tagen veröffentlichen. CRISPR Störungen wird eine Zunahme der H3K9me3 um 7 Tage Post Transduktion12beobachtet. Das Enhancer-i-Protokoll sieht schließlich eine Strategie, um mehrere Websites gleichzeitig Ziel und überwachen Sie die Effizienz des Zielens. Mosaik-Seq17dCas9-KRABBE wird verwendet, um mehrere Enhancer gleichzeitig, aber diese Technik stützt sich auf einzelne Zelle RNA Sequenzierung, Ausdruck Veränderungen zu erkennen und viele Gene (z. B. Östrogen-responsive Gene) unentdeckt aufgrund der geringen Empfindlichkeit der einzelligen RNA-FF Enhancer-i bietet eine zuverlässige Methode um Enhancer einzeln und in Kombination für jedes Gen zu untersuchen.
Der wichtigste Schritt der Enhancer-i ist Transfektion, die für die Zell-Linie von Interesse optimiert werden soll. Dieses Protokoll setzt auf Puromycin Behandlung für transfizierte Zellen zu bereichern, aber es ist möglich, dass Co transfecting Guide RNAs mit einem fluoreszierenden Proteins und Sortierung für fluoreszierende Zellen mittels Durchflusszytometrie erweisen eine bessere Methode der Anreicherung für einige Zelle Typen. Es wird empfohlen, Überwachung der Ausdruck der Guide RNAs und SID4x-dCas9-KRABBE durch qPCR zu beheben und Transfektion zu bestätigen. Wenn Richtmengen RNA niedrig sind (Zyklus Schwelle > 30), Benutzer können auch erwägen alternative Anleitung RNA Produktionsstrategien wie in Vitro Transkription28. Es ist auch möglich, dass trotz der hohen gRNA, Guide RNA Ausrichtung des SID4x-dCas9-KRABBE-Proteins ist ineffizient, können notwendig sein, in welchem Fall Auswahl verschiedener RNA-Sequenzen führen. Durch ChIP-Seq Fusionsprotein mit Chromatin von Enhancer-i behandelt Zellen ausführen, kann die Effizienz des Zielens überwacht werden. Wenn high-Signal des SID4x-dCas9-KRABBE in der Region von Interesse ist, und kein Ausdruck Änderungen in seiner vermeintlichen Zielgen werden erkannt, trägt die Region wahrscheinlich nicht zum Ausdruck, dass gen unter den untersuchten Bedingungen bei.
Eine mögliche Einschränkung der Enhancer-i ist, dass Ziel Effekte ansammeln können, wenn zu viele Seiten gleichzeitig gezielt eingesetzt werden. Nichtsdestotrotz haben CRISPR Störungen Strategien für Knockdown weniger Ziel-Effekte als RNAi29, besonders wenn eine polyklonale Zell-Linie mit dem Ausdruck dCas9-KRAB verwendet wird. Während wir off-Target genomische Bindung des SID4X-dCas9-KRABBE gesehen haben, wenn Sie 10 Seiten gleichzeitig auf, haben wir keine Veränderungen der Genexpression durch diese Bindung Ereignisse identifiziert. Da einige Enhancer mehrere Veranstalter bzw. andere Geschmacksverstärker kontaktieren können, ist es möglich, dass viele Gene Ausdruck bei der Ausrichtung der einzelnen Enhancer, ändern können, aber es unklar ist, ob diese Form der Genregulation üblich ist. Um zu bestätigen, dass der Ausdruck Veränderungen werden beobachtet durch Ausrichtung auf eine bestimmte Enhancer und nicht Ziel Effekte, können Benutzer Enhancer-i mit zwei unterschiedliche Sätze von nicht überlappenden Guide RNAs die gleiche Region durchführen. Darüber hinaus kann die genetische Löschung der Region mit Nuklease-kompetente Cas9 weiter seine Auswirkungen auf die Genexpression bestätigen.
Als Enhancer-i Funktionen durch Histon Deacetylation ist es möglich, dass seine Deaktivierung Fähigkeiten auf Geschmacksverstärker beschränken, die spürbare Histon Acetylierung verfügen. Es gibt eine Vielzahl von alternativen repressiven Fusionen, die möglicherweise effektiver bei Ausrichtung auf spezifische Enhancer. DNA-Methyltransferase Fusionen zu dCas9 können zur Genexpression bei distalen Enhancer30gezielt zu reduzieren, aber diese Unterdrückung ist oft nicht flüchtig. Eine andere repressive Fusion nutzt die Freund GATA1 (FOG1) Domäne, wodurch Histon H3 Lysin 27 Trimethylation und unterdrückt Genexpression auf Ebenen ähnlich wie dCas9-KRABBE in einer Vielzahl von Zelle Linien und Promotoren31. Interessanterweise reduziert hinzufügen mehr Kopien von FOG1 zu dCas9 das repressive Potential an Promotoren, was darauf hindeutet, dass eine einzelne Kopie der SID-Domäne möglicherweise mehr Enhancer Deaktivierung als die 4 Kopien derzeit in Enhancer-i verwendet. Es ist möglich, dass einige Loci von dual targeting durch unterschiedliche Kombinationen der oben genannten dCas9 Fusionen profitieren können. Zum Beispiel kann stabile langfristige Unterdrückung durch gleichzeitige Transduktion von dCas9-DNMT3a und dCas9-KRAB32erreicht werden. Die meisten dieser repressiven Fusionen haben nur auf einen einzigen Nenner zu einem Zeitpunkt gezielt, und es bleibt unklar, welche am effektivsten zu manipulieren gleichzeitig mehrere Enhancer ist.
Enhancer-i, während eine geeignete Methode für die Untersuchung von Kombinationen der Enhancer für eine Handvoll Genen, ist noch etwas im Durchsatz begrenzt, wenn der Benutzer wünscht, vermeintliche Enhancer für Hunderte von Genen zu studieren. Zukünftige Anwendungen dieser Technik werden Imaging-basierte Technologien um mehrere Gene in mehrere Proben gleichzeitig zu quantifizieren integrieren. Wichtiger ist, sind diese Technologien kompatibel mit direkte Nachweis der RNA-Moleküle aus lysate, wodurch die Notwendigkeit für zeitaufwändige RNA Isolierung. Diese Anpassungen werden das Verhör von größeren Mengen von Enhancer erleichtern.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von NIH/NHGRI R00 HG006922 NIH/NHGRI R01 HG008974, j.g. und Huntsman Cancer Institute unterstützt. J.B.C. wurde von NIH Trainingsprogramm in Genetik T32GM007464 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
Referenzen
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten