Method Article
CRISPR Guide RNA Klonen für Säugetier-Systeme
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier ist eine einfache, effiziente, und kosteneffektive Methode des Klonens SgRNA skizziert.
Zusammenfassung
Die skizzierte Protokoll beschreibt optimierte Methoden für die effiziente und kostengünstige Erzeugung von Cas9 verbundenen Guide RNAs. Zwei alternative Strategien für Guide RNA (gRNA) Klonen werden anhand ihrer Verwendung von Typ IIS Restriktionsenzym BsmBI in Kombination mit einer Reihe von kompatiblen Vektoren beschrieben. Außerhalb der Zugang zum Sanger-Sequenzierung Dienstleistungen um die erzeugten Vektoren zu überprüfen sind keine spezielle Ausrüstung oder Reagenzien abgesehen von diesen, die standard in modernen molekularbiologischen Labore sind erforderlich. Die beschriebenen Methode richtet sich vor allem für das Klonen einer einzigen gRNA oder einem gekoppelten gRNA exprimierenden Vektor zu einem Zeitpunkt. Dieses Verfahren ist nicht gut für die Erzeugung von Bibliotheken mit Tausenden von gRNAs skalierbar. Für diese Zwecke sind alternative Energiequellen Oligonukleotid-Synthese wie Oligo-Chip Synthese empfohlen. Schließlich, während dieses Protokoll konzentriert sich auf eine Reihe von Säugetieren Vektoren, die allgemeine Strategie ist aus Kunststoff und gilt für jeden Organismus, wenn der entsprechende gRNA Vektor zur Verfügung steht.
Einleitung
CRISPR-assoziierten Protein 9 (Cas9) ist eine RNA-geführte Endonuklease die gewünschte genomische Ziel wenn komplexiert mit einem entsprechend gestalteten kleinen Guide RNA (gRNA)1,2gerichtet ist. gRNAs umfassen eine 20-Nucleotid-Sequenz (Protospacer), die komplementär zu der genomischen Zielsequenz ist. Neben der genomischen Ziel ist Folge einer 3' Protospacer-assoziierten Motiv (PAM), die für Cas9 Bindung erforderlich ist. Im Falle von Streptococcus Pyogenes Cas9 (SpCas9), dies hat die Folge NGG. Bei der Bindung der DNA Ziel, spaltet Cas9 beide Stränge der DNA, so anregend Reparaturmechanismen, die ausgenutzt werden können, um den Ort von Interesse zu ändern. Die Aktivierung des Signalwegs fehleranfällige nicht-homologe Ende verbinden (NHEJ) kann die Ursache der Störung eines Ziel-Gens. Alternativ können Reparaturen über eine homologe Rekombination die gezielte Integration einer gewünschten DNA-Sequenz stimulieren, wenn eine Spender Vorlage6vorgesehen ist. Nuklease-Toten Cas9 (dCas9) Varianten kann auch generierte3 durch Mutationen die Rückstände innerhalb von Cas9, die für Endonucleolytic Tätigkeit unerlässlich sind. dCas9 bleibt zuständig für DNA-Bindung aber schneidet nicht gebundene Ziel. Durch die Verschmelzung verschiedener Effektor-Domains zu dCas9 und in der Nähe eines Gens transkriptionelle Startsite Regie, einsetzbar für selektive gen Induktion oder teilweise gen-silencing-4,5dCas9.
Wenn unglaublich leistungsfähig, erfordert die Fähigkeit, Cas9 zu verwenden, um gezielt eine genomische Sequenz von Interesse, dass ein Benutzer zunächst eine gRNA einzigartig für den Ziel-Websites generiert. Eine Vielzahl von Methoden für den Aufbau gRNA Expressionsvektoren zuvor beschrieben, viele unterschiedliche Effizienz und Kosten6,7,8. Stand-Alone-PCR Amplifikate kann als gRNAs für schnelle Screening von Oligonukleotid-Lieferung nach Transfektion innerhalb einiger Stunden verwendet werden; Dies erfordert jedoch Ultramer (mehr als 100 bp) Oligo-Synthese, die teure9ist. Es ist auch möglich, gBlocks zu kaufen, die kostengünstiger als Ultramers sind. Eine einfache, kostengünstige und hocheffiziente Methode zur gRNA Klonen wäre wie Cas9 integraler Bestandteil der modernen Molekularbiologie-Toolkit schnell geworden ist, ein Segen für das Feld. Die hier beschriebene Methode ist in unserer Gruppe und kooperierenden Labors seit mehreren Jahren, mehr als 2.000 einzigartige gRNAs4erzeugen beschäftigt.
Die beschriebenen Methode konzentriert sich auf Techniken zum Klonen von Lentivirale kompatibel Vektoren mit einem einzigen gRNA oder höchstens zwei gRNAs. Für die Generation der Plasmide, die mehr als zwei gRNAs enthalten oder eine Bibliothek von gRNAs Klonen werden alternative Ansätze9,10,11,12empfohlen.
Protokoll
Hinweis: Das folgende Protokoll wird beschrieben, wie gRNA Design mit online-Tools (Schritte 1.1-1.4), die gemeinsam alle Konstruktionsmethoden gRNA Plasmid detailliert in dieser Handschrift sind durchzuführen. Sobald die gewünschte gRNAs identifiziert werden, werden Schritte für die Bestellung notwendigen Oligonukleotide mit verschiedene Methoden für die Einführung der Oligonukleotide in Expressionsvektoren (z.B. pSB700) beschrieben. 2 Methoden zum Klonen von einzelnen Führer exprimierenden Vektoren basierend auf entweder Ligatur in einen vorverdauten Ausdruck Vektor (Schritte 2-7.3) oder Golden Gate Klonen (Schritte 8-8,4) werden vorgestellt. Weiter beschrieben, ist eine Strategie zur gekoppelten Reiseführer exprimierenden klonierungsvektoren mit Hilfe der Polymerase-Kettenreaktion (PCR) gefolgt von Golden Gate Klonen (Schritte 11-16). Schließlich werden auch gemeinsame Methoden für die Durchführung einer E. Coli chemische Umwandlung (Schritte 9-9.6) und ein Ausdruck Vektor Sequenz Validierung (Schritte 10 bis 10.2.3) beschrieben.
- GRNA Oligonukleotide mit online-Tools wie folgt zu entwerfen.
- Design gRNAs mit online-Tools wie der SgRNA Torschütze 2.0 Online-Tool (https://crispr.med.harvard.edu/sgRNAScorerV2/)13 oder Alternativen wie das breite SgRNA-Design-Tool (http://portals.broadinstitute.org/gpp/public/analysis-tools/ Sgrna-Design)14 und mehrere andere15.
- Verwenden Sie das Gen Name, Symbol oder rohe DNA-Zielsequenzen potenzielle gRNA Sequenzen generieren.
Hinweis: Eine Tabellenkalkulationsdatei (.csv/.xls) mit allen potenziellen gRNAs gegen das Gen oder die angegebene Reihenfolge wird erstellt. Die Qualität der potenziellen gRNAs wird als Ergebnis wenn das SgRNA Scorer 2.0 Online-Tool verwendet wird oder als %-Ziel Effizienz gemeldet werden, wenn die Breite SgRNA-Design-Tool verwendet wird. Beide Maßnahmen sind zur abgeleitet basierend auf der Ziel-schneiden-Aktivität. - Berücksichtigen Sie die gRNAs ranking der Off-Target-Aktivität in der gRNA Auswahl.
Hinweis: Optimale Führer sind diejenigen mit der größten Ziel-Effizienz und die wenigsten Ziel-Aktivität. Für die Aktivierung (CRISPRa) sollen gRNAs Ziel die Region von 1-200 bp stromaufwärts der transkriptionellen Start Seite (TSS)4,15. Für die Hemmung (CRISPRi), gRNAs sind entwickelt, um die Region von 50 Ziel bp stromaufwärts von der TSS bis 300 bp stromabwärts des TSS-5. Um ein Gen mit Cas9 schneiden zu deaktivieren, sind gRNAs entwickelt, um die ersten 10-50 % der kodierenden Sequenz stromabwärts der initiierenden Codon (als gRNAs targeting der 3'-Ende des Gens haben gezeigt, dass weniger wirksam)13Ziel. - Stellen Sie sicher, keine internen BsmBI Standorte liegen vor, wenn Oligonukleotide für die Golden Gate Klonen verwendet werden, sonst wird die geglühten Oligonucleotides verdaut13.
- Wählen Sie die genomische Zielsequenzen und entfernen Sie die 3' NGG PAM (wobei nur die Protospacer Sequenz) für die einzelnen gRNA Oligonukleotid Sequenz Änderung (z. B.Startvorgang: CGCGTGCTCTCCCTCATCCATGG).
Hinweis: Wenn Sie eine Tabelle verwenden, die folgende Formel wird entfernen 3' NGG: =LEFT(A2,LEN(A2)-3), wo befindet sich die Zelle A2 enthält die Zielsequenz und PAM.- CACCG an der 5'-Ende der Oligo anhängen.
Hinweis: Bei der Verbindung mit dem pSB700-Backbone, wird diese Sequenz 3' Ende der U6 Projektträger fahren die Transkription der gRNA umfassen. Die "CACC" Reihenfolge sorgt dafür, dass die Oligo mit den Überhängen des Vektors BsmBI verdaut pSB700 kompatibel ist. Das "G" ist eine Anforderung der Polymerase III Promotoren und sorgt für die effiziente Initiation der Transkription der gRNA. Verwenden Sie die folgende Formel in einer Tabelle zum Ausführen dieser Aufgabe: =CONCATENATE("CACCG",(B2)), wo B2 die Zelle ist mit dem Protospacer-Sequenz [z. B.Hinzufügen von 5' CACCG-Sequenz, die Protospacer: CACCGCGCGTGCTCTCCCTCATCCA (dies ist die endgültige vorwärts Oligo, die bestellt)]. - Eine umgekehrte Ergänzung (rc) der Protospacer Sequenz zu erstellen.
Hinweis: zum Beispiel ist die umgekehrte Ergänzung des oben genannten Protospacer TGGATGAGGGAGAGCACGCG. - 5'-Ende des Rc Protospacer AAAC hinzufügen. Fügen Sie eine zusätzliche C an der 3'-Ende des Rc Protospacer (reverse Oligo).
Hinweis: Die Sequenz "AAAC" sorgt dafür, dass die Oligo für die Klonierung in den Vektor BsmBI verdaut pSB700 kompatibel ist. Das zusätzliche "C" am 3'-Ende wird benötigt, um die Reihenfolge der initiierende "g" hinzugefügt, um die vorderen Oligo Tempern. Verwendung die folgende Formel in einer Tabelle, diese auszuführen Aufgabe =CONCATENATE("AAAC",(C2),"C"), wo C2 ist die Zelle mit der umgekehrte Ergänzung Sequenz [z.B.AAACTGGATGAGGGAGAGCACGCGC (Dies ist die letzte reverse Oligo, das ist bestellt)].
- CACCG an der 5'-Ende der Oligo anhängen.
- Bestellen Sie die Oligonucleotides, ohne zusätzliche Modifikationen auf die Oligonucleotides.
- Bestellen Sie die minimale Menge an Oligo erforderlich, da nur sehr geringe Mengen für das Klonen Reaktionen benötigt werden.
- Verdünnen Sie lyophilisierter Primer, eine Endkonzentration von 100 μM 1 x TE-Puffer (10 mM Tris-Cl, pH 7,5 mit 1 mM EDTA).
- Aliquoten 1:1 vorwärts und rückwärts Oligonukleotide (z.B. 10 μL jeder) in PCR-Streifen-Cap Röhrchen.
Hinweis: Dadurch wird das schnelle Klonen von vielen gRNAs zu einem Zeitpunkt mit einer Mehrkanal-Pipette.- Wirbel und Spin down die Oligo-Mischungen (100 X g für 15 s). Inkubieren Sie die Reaktion bei Raumtemperatur für 5 min vor dem Einrichten der Ligatur.
Hinweis: Es ist nicht notwendig zum Heizen und kühlen dann die Oligo-Mischungen zur Erleichterung ihrer glühen. Es ist erwähnenswert, dass 3-4 Paare von gRNA Oligonukleotide können gebündelt und in einer einzigen Reaktion geglüht (z.B.Mix 3 μL vorwärts und 3 μL der reverse Oligonukleotide zusammen von bis zu 4 gRNA Oligo Paaren, mit einem Gesamtvolumen von 24 μL). Das Volumen der Oligonukleotide verwendet für das Glühen kann an Bedürfnisse des Benutzers angepasst werden.
Hinweis: Schritte 6-7.3 beschreiben die Predigestion des pSB700. Eine alternative Methode finden Sie unter Schritt 8, das eine einstufige BsmBI Einschränkung Verbindung beschreibt.
- Wirbel und Spin down die Oligo-Mischungen (100 X g für 15 s). Inkubieren Sie die Reaktion bei Raumtemperatur für 5 min vor dem Einrichten der Ligatur.
- 1-5 μg des ausgewählten pSB700 Führer Expressionsvektors mit BsmBI zu verdauen (0,5 μl pro 1 μg) für 1 h bei 55 ° C15. Führen Sie die Verdauung des pSB700 gRNA Expressionsvektors in einem Gesamtvolumen von 40 μl (Tabelle 1).
- Führen Sie die Verdauung Produkte auf 1,5 % (wt/Vol) Niedrigschmelzende Agarose-Gel.
- Schneiden Sie die verdauten Vektor Rückgrat Band entspricht ein Fragment von ~ 9 kb groß und legen Sie die Gel-Scheibe in einem 1,5 mL Microcentrifuge Schlauch (Abbildung 1).
- Verwenden Sie eine kommerzielle Gel Reinigung Kit, um die DNA aus dem Agarosegel nach den Anweisungen des Herstellers zu extrahieren.
- Eluieren Sie die DNA in 10-15 μl TE-Puffer (10 mM Tris, pH 7,5, 10 mM Tris, pH 8.0) zu einer konzentrierten Eluat.
- Verbinden Sie die geglühten gRNA Oligonukleotide aus Schritt 4 in die BsmBI verdaut pSB700 Guide Expressionsvektor in einer 20 μL Reaktion (Tabelle 2) für das Klonen von gRNA Oligonukleotide in dem Expressionsvektor bereits verdaute pSB700.
- Zunächst fügen Sie Wasser, Puffer, und DNA, dann Wirbel die Mischung und fügen Enzym, Vortex 1 letztes Mal nach Zugabe des Enzyms und Spin-down die Lösung (100 X g für 15 s). Inkubieren Sie die Reaktionen bei Raumtemperatur über Nacht.
- Gehören Sie eine Negativkontrolle keine-Insert-Reaktion, die allein, ohne Beilage geglühten gRNA Oligo Vektor BsmBI verdaut hat. 1 μl Wasser kann anstelle der 1 μL gRNA Oligonukleotide für die keine-Insert-negativ-Kontrolle verwendet werden.
- Fahren Sie mit der Transformation (Schritt 9).
Hinweis: Schritt 8 ist eine alternative Methode zur Predigestion der pSB700 wie in den Schritten 6-7.3 beschrieben.
- Führen Sie gRNA Oligonukleotide (beschrieben in Schritten 1-5.1 ein) in der pSB700-Vektor durch den Einsatz von Golden Gate Klonen der einstufigen BsmBI Einschränkung Ligatur.
- Montieren Sie den master-Mix (1 X)-Wenn mehrere gRNAs geklont wird, sind bereiten den master-Mix ohne die Einlage Oligonucleotides hinzugefügt (Tabelle 3). Aliquoten Master in PCR-Röhrchen mischen und verwenden eine Mehrkanal-Pipette gRNA Oligonukleotide aus Schritt 4.2 hinzufügen. Enthalten Sie ein keine-Insert-Steuerelement mit 1 μl Wasser.
- Durch die Einbeziehung der BsmBI Enzym und T4 DNA-Ligase in die gleiche Reaktion, gleichzeitige Beschränkungsverdauung und Unterbindung erreicht (d.h., Golden Gate Klonen). Das folgende einfache Tor-Protokoll verwenden, um den Vektor zu verdauen und verbinden die Einsätze 1 Reaktion: halten Sie die Reaktion bei 37 ° C für 2 h bei 50 ° C für 10 min bei 65 ° C für 15 min bei 80 ° C für 15 min, und schließlich , halten Sie es bei 4 ° C.
- Beachten ist überprüfen Sie, ob die Oligonucleotides keine BsmBI Websites enthalten, bevor Sie diese Methode verwenden. Wenn die gRNAs, die geklont wird, sind BsmBI Restriktionsschnittstellen enthalten, werden sie auf die Zugabe von BsmBI verdaut. Dies wird verhindert, dass der Benutzer transformanten zu erhalten.
- Nach der ersten einfachen Tor Reaktion jede Reaktion eine zusätzliche 0,5 μL BsmBI Enzyms hinzu und legen Sie sie wieder bei 55 ° C für 1 h. Halten Sie nach der Verdauung die Reaktion bei 4 ° C oder frieren Sie ein, bis bereit zu verwandeln. Diese zweite Runde der Restriktionsenzym Inkubation entfernt jeden unverdaute oder religiert Wildtyp pSB700 Vektor Rückgrat aus dem Reaktionsgemisch, so dass nur Vektoren, die die Oligo-Einsätze wurden intakt bleiben und erbringt transformanten.
- Fahren Sie mit der Transformation (Schritt 9).
- Verwandeln Sie 0,5 μL des Reaktionsgemisches in 8 μl kompetente E. Coli [z. B.NEB-DH5a (1-3 x 109 KBE/µg pUC19 DNA) oder NEB stabile 1 - 3 x 109 KBE/µg pUC19 DNA). Für Lentivirale Plasmide werden NEB stabil Zellen konsequentere Plasmid Renditen bieten.
- E. Coli aus dem Speicher bei-80 ° C zu entfernen und für 5-10 min auf Eis Auftauen.
- 8 μl kompetente E. Coli 0,5 μL der Reaktion Mischung hinzu und halten Sie die Mischung für 30 min auf Eis.
- Hitze Schock die Mischung bei 42 ° C für 45 s.
- Ruhen sie sich für 2 min auf Eis.
- Die Kultur auf einen Dreh Shaker in 250 μl SOC-Medien für 1 h bei 37 ° C für NEB DH5a und bei 30 ° C für NEB Ställe zu erholen.
- Platte 80 μL der Kultur auf einer geeigneten Antibiotika-Resistenz Lysogeny Brühe (LB) Platte und inkubieren Sie es über Nacht bei 37 ° C für NEB DH5a und bei 30 ° C für NEB Ställe [z.B.pSB700 ist vergoldet auf LB mit Ampicillin (100 μg/mL) Platten] (Abbildung 3).
- Überprüfen Sie die Reihenfolge der gRNA expressionsplasmid: Überprüfen Sie die Reihenfolge der jede Kolonie von Sanger-Sequenzierung mit Hilfe der Primer 5'-GAGGGCCTATTTCCCATGATTCC-3 "welche Primzahlen an der U6 Projektträger stromaufwärts von der gRNA Oligo einfügen.
- Wählen Sie 2-3 Kolonien pro Reaktion für die Sequenzierung (Tabelle 4).
Hinweis: Mehrere Sequenzierung Anbieter nun das Angebot Dienstleistungen führen Sanger-Sequenzierung direkt von Bakterienkolonien, die reduziert Zeit und Kosten durch den Wegfall der Notwendigkeit, die Plasmiden vor der Sequenzierung zu reinigen. Alternativ kann eine Plasmid-Isolierung-Kit wieder der Plasmid-DNA aus den einzelnen bakterielle Klone verwendet werden. Gereinigte Plasmide können dann für die Sequenzierung eingereicht werden. Eine gepoolte Ligatur Reaktion erfolgt, verwenden Sie Tabelle 4 als Leitfaden für die Anzahl der Kolonien, die ausgewählt und zur Sequenzierung vorgelegt werden sollte. - Isolieren Sie nach der Bestätigung Sequenz gRNA Ausdruck Plasmide.
- Verwenden Sie eine sterilen PIPETTENSPITZE die gewünschte Kolonie in eine 5 mL LB-Medium mit 100 μg/mL Ampicillin für pSB700 Expressionsvektoren Kultur zu impfen.
- Wachsen Sie die Zellen in einem rotierenden Inkubator bei 100 u/min bei 37 ° C über Nacht (30 ° C Wenn NEB Ställe verwendet werden).
- Das Plasmid DNA aus den Kulturen mit einem Plasmid Prep Kit, nach der Hersteller-Protokolls zu isolieren.
Hinweis: Während der oben genannten Methoden Benutzern das Erstellen von einzelnen gRNA-haltigen Vektoren ermöglichen, dieses Protokoll erlaubt Benutzern, Vektoren zu erstellen, die 2 gRNAs, jeweils eigene RNA Polymerase-III-Promotor angetrieben enthalten.
- Wählen Sie 2-3 Kolonien pro Reaktion für die Sequenzierung (Tabelle 4).
- Für ein gekoppeltes gRNA Design die Output-Datei von oben (Schritte 1.1-1.4) beschriebenen SgRNA-Design-Tools und wählen Sie 2 Führungen von Interesse.
- Wählen Sie für eine Aktivierung oder Unterdrückung, Paare von Führern, die mindestens 30 nt auseinander.
- GRNAs zum Schneiden verwendet wählen Sie als Führer, die auf 2 verschiedenen Exons im Gen.
- Für die nachstehenden Anweisungen festlegen der ersten gRNA im Array gRNA-A und die zweite gRNA im Array gRNA-B, um eine gekoppelte gRNA Oligonukleotid Sequenz Modifikation zu erstellen.
Hinweis: Die Oligo zur Erstellung gRNA-a genannt fA und die umgekehrte Oligo verwendet, um einzuführen gRNA-B wird als rB bezeichnet werden. Die Klonmethode unter fügt automatisch eine initiierende G am 5'-Ende eines jeden der gRNAs.- Erstellen Sie wie folgt die entsprechende fA Oligo.
Hinweis: Die 20 Nukleotiden bestehend aus dem gRNA targeting-Sequenz für gRNA-A wird bilden die ersten fA-Sequenz auf der die folgenden Schritte (z. B.Protospacer - Aggggctacaccactcattg) aufbauen werden.- Fügen Sie TTTTCGTCTCTCACCG an der 5'-Ende des fA (z. B.der Sequenz TTTTCGTCTCTCACCGaggggctacaccactcattg).
- Fügen Sie GTTTTAGAGCTATGCTGAAAAGCA an die 3'-Ende des fA (z. B.der Sequenz TTTTCGTCTCTCACCGaggggctacaccactcattgGTTTTAGAGCTATGCTGAAAAGCA). Dies ist die endgültige Fassung des fA Oligo, die bestellt werden.
- Erstellen Sie die erforderlichen rB Oligo wie folgt.
Hinweis: Die 20 Nukleotiden bestehend aus gRNA targeting-Sequenz für gRNA-B werden die ersten rB-Sequenz darstellen, worauf die folgenden Schritte (z. B.der Start Sequenz Gtcccctccaccccacagtg) bauen werden.- Nehmen Sie die umgekehrte Ergänzung der rB (Revcom) = rB (z.B. Cactgtggggtggaggggac).
- Fügen Sie TTTTCGTCTCTAAAC zu den 5'-Ende (Revcom) rB (zB., TTTTCGTCTCTAAACcactgtggggtggaggggac).
- Anfügen der CGGTGACCCAGGCGGCGCACAAG an das 3'-Ende (Revcom) rB (z.B.TTTTCGTCTCTAAACcactgtggggtggaggggacCGGTGACCCAGGCGGCGCACAAG). Dies ist die Finale Version von rB-Oligo, die bestellt werden.
Hinweis: die folgenden Formeln können verwendet werden, mit gemeinsamen Tabellenkalkulations-Software automatisch die Oligo-Sequenzen bearbeiten: = CONCATENATE("TTTTCGTCTCTCACCG",(A2), "GTTTTAGAGCTATGCTGAAAAGCA"), wo A2 ist die Zelle mit der fA-Sequenz und = Concatenate("TTTTCGTCTCTAAAC",(B2),"CGGTGACCCAGGCGGCGCACAAG"), wo B2 ist die Zelle mit der rB-Sequenz (Revcom).
- Erstellen Sie wie folgt die entsprechende fA Oligo.
- Bestellen Sie die Oligonukleotide aus einer Synthese-Anbieter der Wahl. Es sind keine zusätzlichen Änderungen erforderlich.
- Verwenden Sie das vorbereitete Plasmid DNA um PCR mit fA und rB Primer durchzuführen. Dies wird sowohl die nach vorne legen und die umgekehrte gRNAs an das PCR-Fragment erzeugt aus diesem Plasmid und wird erlauben jedem von seinen eigenen Promotor ausgedrückt werden (U6 und 7SK Promotoren – verschiedene Promotoren werden in diesem Entwurf verwendet, um zu verhindern, dass virale Rekombination).
- Phusion GC spezielle PCR-Protokoll verwenden, um die benötigten Fragment erstellen: halten Sie die Mischung bei 98 ° C für 1 min (1 Zyklus), bei 98 ° C für 15 s bei 53 ° C für 15 s, bei 65 ° C für 2 min und bei 72 ° C für 4 min (30 Zyklen) , und halten Sie es dann bei 4 ° C (Tabelle 5).
- Führen das PCR-Produkt auf einem 1 % Agarosegel und überprüfen Sie, dass 1 Band gilt bei ~ 490 bp (Abbildung 2).
- Schneiden und diese PCR-Produkt mit einem Gel Extraction Kit Wahl zu extrahieren. Die DNA in 15 μl TE-Puffer (10 mM Tris, pH 7,5, 10 mM Tris, pH 8,0) eluieren oder destilliertes Wasser.
- Messen Sie die Konzentration von der extrahierten Produkt zusammen mit der pSB700-Vektor mit einem Spektralphotometer für die DNA Molarity Berechnung. Das PCR-Produkt und der Vektor zu einer Konzentration von 40 Femtomoles/μL zu normalisieren.
Hinweis: Mehrere Websites werden bei der Berechnung des Molarity einer bestimmten DNA-Lösung basierend auf der Länge der DNA und seine Konzentration in Lösung (z.B.Promega: http://www.promega.com/a/apps/biomath/index.html?calc=ugpmols). Der Rechner verwendet die Formel: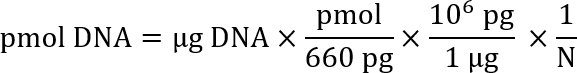
Hier ist N die Länge der Nukleotid-Sequenz.
- Richten Sie eine gleichzeitige Einschränkung Verdauung und Ligation Reaktion über das Protokoll beschriebenen Schritten 8-8,4, um PCR-Fragment in einen Vektor pSB700 zu klonen.
- Anstatt die Reaktion 1 μL der Primer-Lösung hinzufügen, fügen Sie 1 μl 40 Fmole/μl des PCR Produktes hinzu. Darüber hinaus verwenden Sie 1 μL des pSB700 Vektors in einer Konzentration von 40 Fmole/μL.
Hinweis: Gleiche molare Verhältnissen führen zu konsistenten Ergebnissen, obwohl sogar in Fällen, wo die genauen Verhältnisse nicht verwendet werden, Kolonien gewonnen werden können.
- Anstatt die Reaktion 1 μL der Primer-Lösung hinzufügen, fügen Sie 1 μl 40 Fmole/μl des PCR Produktes hinzu. Darüber hinaus verwenden Sie 1 μL des pSB700 Vektors in einer Konzentration von 40 Fmole/μL.
- Fahren Sie nach Abschluss des Golden Gate-Reaktion-Prozess (Schritte 8-8,4) mit der Transformation und Sequenz Überprüfung (Schritte 9 - 10.2.3).
Ergebnisse
Die in diesem Protokoll beschriebenen Methoden sind für die Erstellung von entweder einzeln oder paarweise gRNA Expressionsvektoren. Einzelne gRNA Vektoren Ausdrücken kann entweder vorverdauen Vektor Rückgrat (Abbildung 1) gefolgt von in einer Reihe von kurzen Oligonukleotide Ligation oder mit Golden Gate Klonen um gleichzeitig verdauen das Vektor-Rückgrat und verbinden es in erstellt werden ein Serie von kurzen Oligonukleotide in einer einzelnen Reaktion. Ebenfalls vorhanden ist ein Verfahren zur Herstellung von Vektoren, die 2 gRNAs enthalten jeweils eine eigene unabhängige Veranstalter durch Klonen einer benutzerdefinierten PCR-Fragment (Abbildung 2) angetrieben.
Eine erfolgreiche Klonen für einige der beschriebenen Protokolle führt das Aussehen deutlich mehr Kolonien für Transformationen mit den entsprechenden Einsatz DNA als auf die keine-Insert Steuerplatte (Abbildung 3).
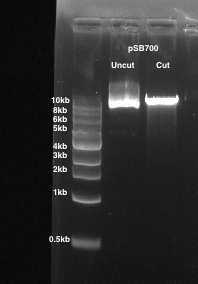
Abbildung 1: die Verdauung von einem pSB700 gRNA Expressionsvektor. 1 % Agarose-Gel wird verwendet. Bahn 1: ungeschnitten pSB700. Bahn 2: pSB700 mit BsmBI geschnitten.
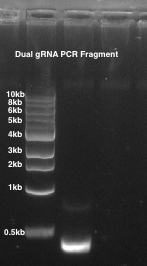
Abbildung 2: gRNA PCR gepaart. 1 % Agarose-Gel wird verwendet. Bahn 1: eine gekoppelte Reiseführer-PCR-Fragment ~ 490 bp.
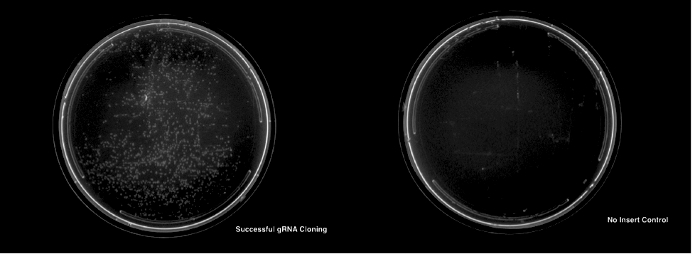
Abbildung 3: die erfolgreiche Klonen und Transformation von einem Plasmid gRNA. Die linke Tafel zeigt eine LB und Ampicillin-Platte mit erfolgreich Kolonien umgewandelt. Im Rechte Bereich zeigt eine keine-Insert Steuerungsplatine zeigt keine Kolonien. Bitte klicken Sie hier für eine größere Version dieser Figur.
| pSB700 Guide Expressionsvektor | 1-5 μg |
| NEB Puffer 3.1 | 4 ΜL |
| BsmBI | 1 ΜL |
| Destilliertes Wasser | bis zu 40 μL |
| Gesamtvolumen | 40 ΜL |
Tabelle 1: Die Beschränkungsverdauung eines pSB700 gRNA Ausdruck Vektors mit BsmBI.
| Geglühten gRNA Oligos (entweder einzelne oder gebündelte Reaktion) | 1 ΜL |
| BsmBI verdaut pSB700 Guide Expressionsvektor | 1 μl (~ 100-250 ng) |
| 10 x T4 DNA-Ligase Reaktion Puffer | 2 μL (1 x Endkonzentration) |
| T4-DNA-Ligase | 1 μl (120 Stück) |
| Destilliertes Wasser | 15 ΜL |
| Gesamtvolumen | 20 μl |
Tabelle 2: Eine Ligatur Reaktion gRNA Oligonukleotide in einen vorverdauten pSB700 gRNA Ausdruck Vektor.
| Reagenz | Volumen (1 X) |
| H2O | 6 ΜL |
| T4-Ligase | 0,5 ΜL |
| ATP | 1 ΜL |
| BsmBI | 0,5 ΜL |
| NEB Puffer 3.1 | 1 ΜL |
| Vektor (z.B. pSB700) (40 Fmol) | 1 μlL |
| Einfügen - vorwärts und rückwärts Oligos oder PCR-Fragment für dual Guide (40fmol) | 1 μl (0,5 μL nach vorne und 0,5 μl reverse) |
Tabelle 3: Eine einstufige BsmBI Einschränkung und gRNA Ligatur Reaktion mischen.
| 1 Anleitung | Sequenz 3 Kolonien |
| Für 2 guides | Sequenz 5-10 Kolonien |
| Für 3 guides | Sequenz 10-15 Kolonien |
| Für 4 guides | Folge 15-20 Kolonien |
Tabelle 4: Die empfohlene Anzahl der Kolonien zu Sequenz für gepoolte gRNA Klonen Reaktionen.
| PCR-Komponente | 50 µL Reaktion |
| 10 µM forward Primer | 2.5 ΜL |
| Rückwärts-Primer 10 µM | 2.5 ΜL |
| 5 X Phusion HF oder GC-Puffer | 10 ΜL |
| 10 mM dNTPs | 1 ΜL |
| Schablone DNA | 100 ng |
| Phusion-DNA-Polymerase | 0,5 ΜL |
| Nuklease-freies Wasser | Bis zu 50 µL |
Tabelle 5: Eine gekoppelte gRNA PCR Mischung.
Diskussion
An gRNA Ausdruck Vektor Bau wurden eine Reihe von Zeit- und kostensparendes Modifikationen vorgenommen. Ein Einzelschritt Einschränkung und Ligatur-Protokoll ist sowie eine Methode der gekoppelten gRNA Ausdruck Vektor Konstruktion skizziert. Der gekoppelten gRNA Ausdruck wird durch eine PCR-Amplifikation mit Oligonukleotide mit einer nach vorne gRNA Zielsequenz (n20) und die umgekehrte Ergänzung von einem anderen Ziel (n20) erreicht. Dies führt dann einen sekundären RNA Pol III-Promotor (7SK) sowie ein SgRNA Schweif in einzelne gRNA Ausdruck Plasmide konventionell zum Ausdruck zu bringen.
Eine sorgfältige Oligonukleotid-Design sorgt für eine erfolgreiche PCR für den gekoppelten gRNA Bau sowie eine klebrig-Ende-Verbindung. Nach dem einstufigen Einschränkung und Ligation Reaktion, die Zugabe von weiteren BsmBI wird sichergestellt, dass alle unveränderten Expressionsvektoren in der Reaktion geschnitten werden. Dies reduziert deutlich den Hintergrund auf die Transformation. NEB-Stable kompetente E. Coli mit einem Wachstum bei 30 ° C erhöht sich die Ausbeute an eine erfolgreiche Transformation.
Die Vorteile dieser Technik über gemeinsame Praktiken beinhalten eine Vereinfachung des Prozesses der Oligonukleotid glühen, eine Einzelschritt-Beschränkung des Expressionsvektors gRNA und die Unterbindung der Oligonukleotide und die Fähigkeit, herkömmliche nutzen einzelne gRNA Expressionsvektoren für den Ausdruck der gekoppelten Guides. Während das Protokoll hier konzentriert sich auf eine Reihe von Säugetieren Vektoren beschrieben, die allgemeine Strategie ist aus Kunststoff und gilt für jeden Organismus, vorausgesetzt die entsprechenden gRNA Vektor zur Verfügung steht. Alles in allem, das Protokoll beschrieben spart Zeit und Kosten.
Allerdings ist darauf hinzuweisen, dass die beschriebenen Verfahren für den Kauf von einzelnen Oligonukleotide nicht gut für die Erzeugung von Bibliotheken mit Tausenden von gRNAs skaliert. Für diese Zwecke sind alternative Energiequellen Oligonukleotid-Synthese wie Oligo Chip Synthese empfohlen.
Es ist vorteilhaft, eine keine-Insert-Steuerelement enthalten, beim Klonen gRNAs. Diese Reaktion kann parallel mit den Reaktionen von Interesse leicht eingerichtet werden und kann verwendet werden, um festzustellen, ob eine unvollständige Verdauung des Start Vektors zu den Kolonien beobachtet am Ende des Verfahrens beigetragen hat. Darüber hinaus, wenn eines der oben genannten Klonen Protokolle durch unvollständig verdaute Vektor Rückgrat oder schlechte Ligatur Effizienz ausgefallen ist, empfehlen wir, versuchen die Alternative Methode, die wir beschrieben haben Klonen.
Bei der Verwendung von Golden Gate Klonen, um gleichzeitig den Vektor zu verdauen und in die kleine Oligonucleotides innerhalb einer einzelnen Reaktion verbinden ist es wichtig zu überprüfen, haben die gRNA, die in den Vektor kloniert werden sind keine BsmBI Site verreisen, denn dies führt zu t He gRNA gekürzt und das Fehlen der Kolonien nach Umwandlung.
Die Strategie, die wir beschrieben haben Klonen gRNA ermöglicht eine schnelle und kostengünstige Generation von gRNAs bei ~ 10 US-Dollar pro Guide, wobei der Großteil der Kosten aus der Oligonukleotid-Synthese und die Sequenz-Überprüfung. Während die beschriebenen Methode Benutzern gRNAs für den Einsatz mit SpCas9 generieren soll, das Protokoll kann leicht für die Verwendung mit Cas9 Orthologe oder andere RNA-geführte jedoch wie Cpf1 oder C2C2, mit geringfügigen Änderungen an den Vektor-Backbone angepasst werden und die Überhang Oligonukleotid-Sequenzen16,17.
Das Protokoll beschriebenen bieten Sequenz verifiziert gRNA Ausdruck Plasmide in 3-d (beginnend mit entsprechend ausgelegten Oligonukleotide), die ist bedeutend schneller als bisherige Methoden. Dies beinhaltet die gRNA Gestaltung von 1 h (Stufen 1-2,3), die Verdünnung und Aliquot der Oligonukleotide von 10 min (Schritte 3-5.1), die Verdauung und Reinigung des pSB700 gRNA Expressionsvektors von 2 h (Schritte 6-6,4), das Klonen von gRNA Oligonukleotide in einem predi ter pSB700 gRNA Expressionsvektor über Nacht bei Raumtemperatur (Schritte 7-7,3), der einstufigen BsmBI Einschränkung Ligatur von 3 h und 40 min (Schritte 8-8,4), die Umwandlung von 16 h (Schritte 9-9,6), und die Validierung der Sequenz des Plasmids gRNA Ausdruck 24 h (mit der Dauer je nach der Sanger-Sequenzierung-Verfügbarkeit und die Geschwindigkeit nach der Einreichung der bakteriellen Kolonie; Schritt 10). Das gepaarte gRNA Design und die Änderung der Sequenz dauert 1 h (Schritte 11-13). Die gepaarten gRNA PCR dauert 3,5 h (Schritte 14-14,4). Das Klonen von PCR-Fragment in einen Vektor pSB700 dauert 3h 40 min (Schritt 15-16).
Die am wahrscheinlichsten Probleme, die mit diesem Protokoll konfrontiert werden können beziehen sich auf Ineffizienz in der Golden Gate Montage und Transformation. Sind ein keine-Insert-Steuerelement bei der Golden Gate-Montage, die Effizienz der Versammlung zu visualisieren. Während der Transformation wird die Positivkontrolle puc19 ein Umwandlung Leistungsfähigkeit Steuerelement bereitstellen. NEB-Stable E. Coli sollte für Lentivirale kompatibel Plasmide verwendet werden und erfordern häufig bis > 16 h bei 30 ° C zu sichtbare Kolonien zu führen.
Offenlegungen
George M. Church hat Gerechtigkeit in Editas und Caribou Biosciences, die darauf abzielen, CRISPR-Cas9 Genom Bearbeitung Technologie vermarkten (für eine vollständige Offenlegung-Liste, bitte siehe http://arep.med.harvard.edu/gmc/tech.html).
Danksagungen
Sathiji Nageshwaran stützt sich auf die Friedreich Ataxie Research Alliance (07340305 - 01) und National Ataxie Foundation Fellowships (7355538-01). Alejandro Chavez wurde von der National Cancer Institute Grant 5T32CA009216-34 und die Burroughs Wellcome Fonds Career Award für Mediziner finanziert. George M. Church wird unterstützt durch die US National Institute der Gesundheit (NIH) National Human Genome Research Institute Grant RM1 HG008525 und Wyss Institut für biologisch inspirierte Engineering. James J. Collins räumt Unterstützung der Defense Threat Reduction Agency Grant HDTRA1-14-1-0006 und Paul G. Allen Grenzen Group. Alejandro Chavez entwickelt die Klonmethode Dual-Guide. Sathiji Nageshwaran, Alejandro Chavez und Nan Cher Yeo schrieb das Manuskript mit Eingabe von allen Autoren.
Materialien
| Name | Company | Catalog Number | Comments |
| BsmBI | New England Biolabs | R0580L | |
| T4 DNA ligase | Enzymatic/New England Biolabs | L6030-LC-L/M0202S | |
| Buffer 3.1 | New England Biolabs | B7203S | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| QIAprep spin miniprep kit | Qiagen | 27104 | |
| Chemically competent E. coli | New England Biolabs | C3019l, C2987l, or C3040H | |
| Standard microcentrifuge tubes, 1.5 mL | Eppendorf | 0030 125.150 | |
| Axygen 8-Strip PCR tubes | Fischer Scientific | 14-222-250 | |
| Thermocycler with programmable temperature-stepping control | BioRad, | 1851148 | |
| UV spectrophotometer (NanoDrop 2000c) | Thermo Scientific | ||
| pSB700 plasmid | Addgene | #64046 | |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | c3040 | |
| Lysogeny broth (LB) | |||
| Ampicillin | |||
| TE buffer (1 mM EDTA, 10 mM Tris-Cl, pH 7.5) | |||
| pSN007 plasmid | Addgene | 102440 |
Referenzen
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America. 109 (39), E2579-E2586 (2012).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J., Charpentier, E. A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science. 337 (6096), 816-821 (2012).
- Shalem, O., Sanjana, N., Zhang, F. High-throughput functional genomics using CRISPR-Cas9. Nature Reviews Genetics. 16 (5), 299-311 (2015).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Gilbert, L., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Yang, L., Yang, J., Byrne, S., Pan, J., Church, G. CRISPR/Cas9-Directed Genome Editing of Cultured Cells. Current Protocols in Molecular Biology. , 31.1.1-31.1.17 (2014).
- Yang, L., Mali, P., Kim-Kiselak, C., Church, G. CRISPR-Cas-Mediated Targeted Genome Editing in Human Cells. Methods in Molecular Biology. 1114, 245-267 (2014).
- Vidigal, J., Ventura, A. Rapid and efficient one-step generation of paired gRNA CRISPR-Cas9 libraries. Nature Communications. 6, 8083 (2015).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nature Protocols. 12 (4), 828-863 (2017).
- Kabadi, A., Ousterout, D., Hilton, I., Gersbach, C. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Research. 42 (19), e147 (2014).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Scientific Reports. 4 (1), (2014).
- Chari, R., Yeo, N., Chavez, A., Church, G. sgRNA Scorer 2.0: A Species-Independent Model To Predict CRISPR/Cas9 Activity. ACS Synthetic Biology. 6 (5), 902-904 (2017).
- Doench, J., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2014).
- Engler, C., Marillonnet, S., Valla, S., Lale, R. Golden Gate Cloning. DNA Cloning and Assembly Methods. , 119-131 (2013).
- Zetsche, B., et al. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System. Cell. 163 (3), 759-771 (2015).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten