Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hochdurchsatz-Messung der Plasmamembran Wiederverschließen Effizienz in Säugerzellen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine Hochdurchsatz-Fluoreszenz basierende Assays, die die Plasmamembran Wiederverschließen Effizienz durch fluorometrisch und bildgebende Untersuchungen in lebenden Zellen misst. Dieser Test kann verwendet werden, für das screening von Drogen oder Zielgene, die Plasmamembran Wiederverschließen in Säugetierzellen zu regulieren.
Zusammenfassung
In ihrer physiologischen Umgebung sind Säugerzellen häufig mechanischen und biochemischen Belastungen ausgesetzt, die Plasmamembran Schäden zur Folge haben. In Reaktion auf diese Schäden verschließen komplexen molekularen Maschinen schnell der Plasmamembran für die Wiederherstellung der Barrierefunktion und Überleben der Zellen zu erhalten. Trotz 60 Jahre Forschung in diesem Bereich fehlen uns noch ein gründliches Verständnis der Zelle Wiederverschließen Maschinen. Mit dem Ziel der Identifizierung von Zellkomponenten haben dieser Kontrolle Plasmamembran Wiederverschließen oder Medikamenten, die Wiederverschließen verbessern können, wir einen Fluoreszenz-basierte Hochdurchsatz-Assay entwickelt, der der Plasmamembran Wiederverschließen Effizienz in Säugetierzellen misst kultiviert in Mikrotiterplatten. Als Modellsystem für die Plasmamembran Schaden, sind bakterielle Pore-forming Toxin Listeriolysin O (LLO), bildet große 30-50 nm Durchmesser proteinhaltige in Cholesterin-haltige Poren Zellen ausgesetzt Membranen. Die Verwendung eines Lesers temperaturgeführte Multimode Mikrotestplatte ermöglicht rasche und sensible Spectrofluorometric Messungen in Kombination mit Hellfeld und Fluoreszenz-Mikroskopie-Imaging lebender Zellen. Kinetische Analyse durch eine Membran impermeant Nukleinsäure-bindenden Fluorochrom emittiert Fluoreszenz-Intensität reflektiert das Ausmaß der Membran Verwundung und Wiederverschließen auf Bevölkerungsebene Zelle, so dass für die Berechnung der Zelle Wiederverschließen Effizienz . Fluoreszenz-Mikroskopie-Bildgebung ermöglicht die Aufzählung der Zellen, die konstitutiv ausdrücken eine fluoreszierende Chimäre aus der nuklearen Protein Histon 2 b, in jede Vertiefung der Mikrotestplatte, um mögliche Schwankungen in ihrer Anzahl zu berücksichtigen und können für eventuelle Identifizierung von unterschiedlichen Zellpopulationen. Dieser Hochdurchsatz-Assay ist ein leistungsfähiges Werkzeug erwartet, um unser Verständnis der Membran Reparaturmechanismen über Screening für Host Gene erweitern oder exogen hinzugefügt, dass Kontrolle Plasmamembran Wiederverschließen Verbindungen.
Einleitung
Säugerzellen unterliegen mechanischen, osmotische und biochemischer Stress, was zum Verlust der Integrität der Plasmamembran. Ohne schnelle und effiziente Wiederverschließen, würde geschädigte Zellen programmierten oder nekrotischen Tod schnell erliegen. Seit den 1960er Jahren haben Bemühungen um die Plasmamembran Wiederverschließen Prozess verstehen von den verheerenden Folgen, verbunden mit seiner Funktionsstörungen motiviert. In der Tat sind Krankheiten wie Muskeldystrophie Limb-Gürtel, Diabetes und Chediak-Higashi-Syndrom mit mangelhaft Plasmamembran Reparatur durch Mutationen im gen Kodierung Dysferlin, Produktion von advanced Glycation Endprodukte und defekte verbunden worden die lysosomalen Menschenhandel Regler CHS1, bzw.1,2,3,4,5,6. Allerdings ist bislang, unser Verständnis der Membran Wiederverschließen nach wie vor begrenzt7. Erste Studien haben gezeigt, dass die Membran Wiederverschließen durch den Zustrom von extrazellulären Ca2 + durch die beschädigte Plasmamembran8,9,10initiiert wird. Seitdem sind mehrere nicht gegenseitig Ca2 +-abhängige Mechanismen sind vorgeschlagen worden, um Zellen zu versiegeln. Die Patch-Hypothese schlägt vor, in der Nähe der Wunde, intrazelluläre Vesikeln miteinander und die beschädigten Plasmamembran verschmelzen zu handeln als ein Patch11,12,13,14. Ein zweites Modell schlägt vor, dass Kalzium-abhängige Exozytose Website Versionen von Lysosomen an der Wunde des lysosomalen Enzyms saure Sphingomyelinase, die Sphingomyelin Ceramid im äußeren Merkblatt der Plasmamembran umwandelt. Diese plötzliche Änderung in der Lipidzusammensetzung führt Ceramid-driven Endozytose von der beschädigten Region15,16,17. Schließlich geht der dritte vorgeschlagene Mechanismus eine Rolle für die Endosomal Sortierung komplexer erforderlich für den Transport (ESCRT) Förderung die Bildung von Vesikeln nach außen gerichtete, die Weg von der Plasmamembran18Knospe. Nur eine begrenzte Anzahl von Proteinen in diesen Modellen identifiziert wurde, und ihre Maschinen muss weiter aufgeklärt.
Hier beschreiben wir einen Hochdurchsatz-Assay, den Maßnahmen der Plasmamembran Wiederverschließen Effizienz bei anhaftenden Säugerzellen unterzogen um zu Schäden durch rekombinante Listeriolysin O (LLO)19vermittelt. LLO ist eine Pore-forming Toxin (PFT) abgesondert von der fakultativen intrazellulären Erreger Listeria Monocytogenes20,21,22 und gehört zu den MACPF/CDC (Membrane Angriff komplex, Perforin, und Cholesterin-abhängige Cytolysin)-Superfamilie. MACPF sind Säugetiere Pore-forming Proteine beteiligt, Immunabwehr, während CDCs bakterielle Toxine hauptsächlich sind produziert durch gram-positive Erreger, die Wirtszellen zur Förderung ihrer pathogenen Lebensstile23beschädigen. CDCs sind als wasserlösliche Monomere synthetisiert oder Dimere, die an Cholesterin binden in der Plasmamembran und oligomerize zu einem prepore Komplex von bis zu 50 Untereinheiten. Komplexe Prepore ordnet dann um β-Stränge über die Lipid-Bilayer bildet eine β-Fass-Pore, die 30-50 nm im Durchmesser24,25,26,27umfasst einzufügen. Diese großen Poren erlauben Fluten von Ionen und kleine Zellkomponenten innerhalb und außerhalb der Zelle; jedoch haben einige Studien vorgeschlagen, Poren kleiner Größen auch gebildete28,29,30 sind. Unter die CDCs zeigt LLO einzigartige Eigenschaften einschließlich irreversible pH-Wert und Temperatur-abhängige Aggregation, die förderlich für Hochdurchsatz-Analysen31,32ist. LLO kann Zellkulturmedium bei 4 ° c, eine Temperatur, die freizügig ihre Bindung an Zellen, aber nicht zur Bildung von komplexen Pore hinzugefügt werden. Einleitung der Porenbildung kann dann synchronisiert werden, durch Erhöhung der Temperatur um 37 ° c, so dass für die effiziente Verbreitung der Toxin-Moleküle in der Ebene der Membran zu Form Oligomere und zum Konformationsänderungen Umbau Pore Generation beteiligt. Daher wird nach dem Wechsel der Temperatur, der kinetische Zellschäden die Menge des Toxins an die Plasmamembran gebunden abhängen. Vor allem lösliche LLO (nicht an der Plasmamembran gebunden) rasch und irreversibel aggregiert, wenn die Temperatur 37 ˚C erreicht die verringert die Notwendigkeit ungebundenes Toxin Moleküle abzuwaschen und das Ausmaß der Membranschäden im Laufe der Zeit. Zu guter Letzt, weil LLO an Cholesterin bindet und Cholesterin-reiche Membranen Poren bildet, ist dieser Assay offen für eine Vielzahl von Säugerzellen. Es ist wichtig, im Auge zu behalten, die LLO betrifft Wirtszelle Signalisierung hauptsächlich über Porenbildung, mit wenigen Ausnahmen in die Pore-unabhängige Zelle Signalisierung kann auftreten,33,34,35,36 ,37,38,39. Es kann also nicht sein, dass LLO Signalisierung Aktivitäten beeinflussen den Prozess der Membran Reparatur ausgeschlossen.
Dieser Assay bewertet direkt das Ausmaß der Zelle Verwundung durch Messung der Einbau von einer Zelle impermeant Fluorochrom (z.B. Propidium Jodid), die passiv verletzte Zellen und wird stark fluoreszierende, sobald es mit Nukleinsäuren assoziiert . Daher kann die Fluorochrom in Zellkulturmedium während das Experiment, so dass Echtzeitanalysen der Zelle Verwundung gepflegt werden. Der Fluoreszenzintensität des Farbstoffs Nukleinsäure-Bindung erhöht die Konzentration des Toxins und für eine bestimmte Konzentration des Toxins wird im Laufe der Zeit erhöhen, bis alle Poren gebildet werden und Zellen sind vollständig repariert oder Sättigung erreicht ist. Der Zustrom von extrazellulären Ca2 + Poren der Membran ist eine conditio Sine Qua Non -Event für Wiederverschließen. Daher die resealing Effizienz kann indirekt nachgewiesen durch den Vergleich Zelle Verwundung in Kulturmedium mit Ca2 + (permissive Reparaturbedingung), Verwundung in einem Ca2 +-freie Mitte (Reparatur einschränkende Bedingung). Da der Fluoreszenzintensität der Nukleinsäure-Bindung Farbstoff direkt proportional zu der Zellkonzentration in jedes gut ist, ist es wichtig, Samenzellen bei der gleichen Konzentration in alle Wells. Es ist auch wichtig, Zellen in jedem gut vor und nach der Test um sicherzustellen, dass die Zelle Ablösung tritt nicht auf, als schwimmende, aggregierte Zellen Fluoreszenz Lesungen verdecken können die Interpretation der Daten erschweren kann, aufzählen. Um Zellen aufzuzählen, wurden Zellen mit dem Ausdruck ihrer nuklearen lokalisiert Histon 2 b-GLP (H2B-GFP) in diesem Test verwendet. Temperaturgeführte, Multimode, kombinieren Mikrotestplatte Leser schnelle Hochdurchsatz-Messungen (mit einem 96 oder 384-Well-Plattenformat) Fluoreszenz-Intensitäten mit Mikroskopie-Bildgebung von lebenden Zellen bei 37 ° C. Letzteres kann verwendet werden, zum Auflisten der Zellzahl und die eventuelle Bildung von unterschiedlichen Zellpopulationen zu beobachten.
Letztlich dieser Assay bietet dem Anwender die Möglichkeit, ihr Wissen über die Komplexität der Membran Reparaturmechanismen durch Screening für Host-Moleküle zu erweitern oder exogen zusätzliche Verbindungen, die Membran steuern können zu reparieren. Das folgende Protokoll beschreibt die experimentellen Schritte, um die resealing Effizienz der Zellen, LLO messen und bewerten die Auswirkungen einer bestimmten Droge oder zellulären Behandlung auf Wiederverschließen Effizienz.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Vorbereitung
- Zelle-Beschichtung
Hinweis: Menschlichen zervikale Epithelzellen, HeLa und HeLa auszudrücken Histon 2 b-GLP (H2B-GFP), wurden in diesem Protokoll verwendet, aber dieser Assay an anderen Säugerzellen19angepasst werden kann.- Nehmen Sie adhärente Zellen aus einem 75 cm2 Zelle Kultur Kolben durch Waschen der Zellen mit 2 mL Trypsin-EDTA 0,25 ab %. Ersetzen Sie die gebrauchte Trypsin mit 2 mL frische Trypsin-EDTA 0,25 %.
- Inkubieren Sie die Zellen bei 37 ° c für 5 min, bis die Zellen haben abgerundet und losgelöst von der Küvette.
- Aufschwemmen der Zellen in 8 mL Wachstumsmedium (DMEM mit 10 % Hitze-inaktivierten fötalen Rinderserum, 100 U/mL Penicillin und 100 µg/mL Streptomycin).
- Bestimmen Sie die Zellkonzentration mit einem Hemocytometer und 10 µL Zellsuspension.
- Die Zellen im Wachstumsmedium zu einer Konzentration von 2,5 x 105 Zellen/mL zu verdünnen.
- Gießen Sie die Zellsuspension in einer sterilen Pipette Schüssel und mischen Sie die Suspension mit einer serologischen 10 mL-Pipette.
- Mit einem 12-Mehrkanal-Mikropipette und 200 µL Spitzen, HeLa-Zellen (2,5 x 104 Zellen/100 µL/Well) in dreifacher Ausfertigung (oder Vierfachbestimmungen) in eine 96-Well flachen, klaren, schwarzen Polystyrol Gewebekultur behandelt Bodenplatte verteilen.
Hinweis: Eine Beschichtung-Anordnung wird als ein Beispiel in Abbildung 1dargestellt. - Kultur der Zellen für 24 h in einem befeuchteten Zelle Kultur Inkubator bei 37 ° c und 5 % CO2.
- Stammlösung Vorbereitung
- Bereiten Sie 1 L einer 10 X Aktie des Puffers M (zum Vorbereiten von M1 und M2) durch Zugabe von 95 g Hanks ausgewogen Salzlösung, 0,476 g MgCl2 (5 mM), und 23,83 g HEPES (100 mM) auf 900 mL Wasser. Passen Sie den pH auf 7,4 und erhöhen Sie die Lautstärke auf 1 L. Filter zu sterilisieren.
- Bereiten Sie 50 mL einer 50 x (1,25 M) Lager von Glukose durch Zugabe von 11,26 g D-(+)-Glucose auf insgesamt 50 mL Wasser. Filter zu sterilisieren die Lösung.
- Bereiten Sie 50 mL einer 100 x (120 mM) bestand von Kalzium durch Zugabe von 0,666 g CaCl2 auf insgesamt 50 mL Wasser vor. Filter zu sterilisieren die Lösung.
- Bereiten Sie 50 mL einer 10 x (50 mM) bestand von Ethylenglykol-bis(2-aminoethylether)-N, N, N', N', Tetraacetic Säure (EGTA) durch Zugabe von 0,951 g EGTA, 40 mL Wasser. Erhöhen Sie den pH-Wert auf 8 mit NaOH der EGTA aufzulösen, dann erhöhen Sie die Lautstärke auf 50 mL. Filter zu sterilisieren die Lösung.
- Vorbereitung einer einzelnen 96-Well-Platte, 50 mL Medium 1 (M1, enthält Ca2 +), 50 mL Medium 2 (M2, Ca2 +-frei), und 15 mL Medium 2 mit EGTA, entsprechend ergänzt:
- Fügen Sie für M1 5 mL 10 x 1 mL 50 X Glukose Puffer M und 0,5 mL 100 X CaCl2zu 43,5 mL Wasser hinzu.
- Fügen Sie für M2 5 mL 10 x Puffer M und 1 mL 50 X Glukose 44 mL Wasser hinzu.
- Für M2/EGTA, fügen Sie 1,5 mL 10 x Puffer M und 1,5 mL 10 X EGTA auf 12 mL Wasser.
Hinweis: Alle Lösungen mit Propidium Jodid (PI) sollten bereit sein, direkt vor dem Hinzufügen zu den Zellen.
- Platte Leser/Imaging Cytometer Einstellungen
Hinweis: Benutzen Sie einen Multi-Mode-Platte-Reader mit zwei Erkennungseinheiten ausgestattet: eine Spectrofluorometer und eine bildgebende Cytometer. Begrenzen Sie die Fluoreszenz-Exposition zur Vermeidung von Immunofluoreszenz die Fluorophore.- Vorwärmen des Platte Lesers auf 37 ° C vor der Durchführung des Tests.
- Richten Sie die Parameter für die kinetische Assay entsprechend im Modus " Einstellungen ":
- Wählen Sie Monochromator, FL (Fluoreszenz)und kinetische für die optische Konfiguration aus, lesen Sie Modi und lesen Sie Typ, bzw. zu.
- Wählen Sie unter Wellenlänge Einstellungeneine 9 und 15 nm Anregung und Emission Bandpass, beziehungsweise. Für Assays setzen mit Propidium Jodid (PI), Sie die Anregung und Emission Wellenlängen bis 535 und 617 nm.
- Wählen Sie unter Plattenart 96 Wells für das Plattenformat und eine voreingestellte Platte Konfiguration entspricht eine schwarze Wand klar Bodenplatte.
- Markieren Sie unter Bereich lesendie Brunnen, die während der kinetischen analysiert werden.
- Unter PMT und Optikdie Blitze pro gelesene auf 6 voreingestellt und aktivieren Sie das Kontrollkästchen Lesen von unten.
- Unter Timing 00:30:00 Uhr im Feld Laufen Gesamtzeit für einen 30 min. kinetischer Assay einfügen und 00:05:00 Uhr für das Intervalleinfügen.
Hinweis: Für jedes Mal zu zeigen und eine Wellenlänge, die Lesung eine volle 96-Well-Platte ist 30 s. - Überprüfen Sie die angegebenen Einstellungen in den Einstellungen auf der rechten Seite und wählen Sie "OK". Führen Sie Presse Lesen , die kinetischen einzuleiten.
- Richten Sie die bildgebenden Parameter entsprechend im Modus "Einstellungen":
- Wählen Sie Minimax, Imagingund Endpunkt für die optische Konfiguration aus, lesen Sie Modi und lesen Sie Typ, bzw. zu.
- Wellenlängen, wählen Sie unter Licht übertragen, und eine oder beide der Fluoreszenz Kontrollkästchen der entsprechenden Anregung und Emission Wellenlängen von 456/541 nm (GFP) und 625/713 nm (PI).
- Verwenden Sie die gleichen Optionen für das Plattenart und Lesen Sie , wie in den Schritten 1.3.2.3. und 1.3.2.4 definiert.
- Wählen Sie unter Gut Bereich Einstellungdie Anzahl der Standorte in einen Brunnen, abgebildet werden.
Hinweis: 12 Standorte entsprechen zu einem Full-Well-Bild. - Wählen Sie unter den Erwerb Bildeinstellungendie Belichtungszeiten für Durchlicht, 541 (GFP) und 713 (PI). Für GLP Bild, das ganze gut mit einer Belichtungszeit von 20 ms/Bild. Für übertragen Licht (TL) und PI Fluoreszenz, ein einzelnes Bild von der Mitte von jedem Bohrloch mit Belichtungszeiten von 8 bis 20 ms, bzw. zu erwerben.
- Überprüfen Sie die angegebenen Einstellungen in den Einstellungen -Informationen auf der rechten Seite und wählen Sie "OK". Die Erfassungszeit für imaging-die gesamte Oberfläche von jedem Bohrloch (12 Bilder/Na) einer 96-Well-Platte und für eine Wellenlänge ist ~ 15 min. Presse Lesen , Bildgebung zu initiieren.
Hinweis: Die Erfassungszeit von einem einzigen Bild/Brunnen einer 96-Well-Platte erfordert ~2.5 min/Platte für eine Wellenlänge. Die oben beschriebenen Parameter entsprechen den spezifischen Ausrüstung in unserem Labor. Spectrofluorometric Messungen: eine Xenon Blitzlampe 1,0 nm Schrittweite Erregung Wellenlängen (250-850 nm) mit einer verstellbaren 9 oder 15 nm Bandpass anzeigen, melden Sie sich ein Photomultiplier-Röhren-Detektor mit einer > 6 Dynamikbereich und eine verstellbare 15 oder 25 nm Emission Bandpass. Imaging Cytometer: eine Beleuchtung Lichtquelle in der Lage, des weißen Lichts, 460 nm und 625 nm Erregung Wellenlängen mit einem 20 nm Bandpass, Emission Filter zentriert auf 541 nm (108 nm Bandpass) und 713 nm (123 nm Bandpass), bzw., und eine 4 X Objektiv gekoppelt an ein 1,25 12-Bit Charge - Coupled Device Megapixelkamera.
(2) assay
Hinweis: Zum Zeitpunkt des Tests müssen Zellen Zusammenfluss von 70-90 % sein. Während der Wäsche Schritte sollte das Medium entnommen und an der Seitenwand des Brunnens (nicht direkt über die Zellen) angewendet. Behalten Sie die Temperatur der LLO auf < 4 ° c seine Aggregation bis Schritt 3.1.5 zu verhindern.
- Bereiten Sie einen Bestand von 30 µM PI in M1 und ein Bestand von 30 µM PI in M2 bei 37 ° c vorgewärmt.
- Vorsichtig Waschen der Zellen in Platte 1 mit einer 12-Mehrkanal-Mikropipette und 200 µL Tipps, wie folgt:
- Entfernen Sie für Reparatur-freizügigen Bedingungen die Wachstumsmedium und waschen, die die Zellen zweimal mit 200 µL/Well M1 bei 37 ° c vorgewärmt. Ersetzen Sie das Medium mit 100 µL/Well des warmen M1 mit 30 µM PI.
- Für reparieren Sie restriktive Bedingungen zu, entfernen Sie das Wachstumsmedium und waschen Sie die Zellen einmal mit 200 µL/Well warm M2 mit 5 mM EGTA, Ca2 +, gefolgt von einem waschen mit 200 µL/Well M2 Chelatkomplex. 30 µM PI-haltigem Medium mit 100 µL/Well warm M2 zu ersetzen.
- Nachdem das Wachstumsmedium gewaschen und mit Propidium Jodid-haltigem Medium ersetzt wurde, direkt zu 2.1.3 Schritt zu bewegen.
- Bild-Platte 1 unter Durchlicht, GLP und PI als detaillierte unter 1.3.3 (Pre-kinetische). Dieser Schritt dauert 15-20 min.
- Bereiten Sie den Zeitraum 15 min im Schritt 2.1.3 Platte 2 mit einem 12-Mehrkanal-Mikropipette und 200 µL Tipps wie folgt:
- Legen Sie eine 96-Well Rundboden Polypropylen Mikrotestplatte auf Eis. Konfigurieren Sie die Platte mit ein experimentelles Design entsprechende Platte 1 (Abbildung 1).
- Für Reparatur-freizügigen Bedingungen hinzufügen 100 µL/Well des eiskalten M1 mit 60 µM PI, gefolgt durch die Zugabe von 100 µL/Well des eiskalten M1, 4 x LLO oder nicht für das Steuerelement enthält.
- Für Reparatur-restriktiven Bedingungen hinzufügen 100 µL/Well des eiskalten M2 mit 60 µM PI, gefolgt durch die Zugabe von 100 µL/Well des eiskalten M2, 4 x LLO oder nicht für das Steuerelement enthält.
- Platzieren Sie nach der imaging-Platte 1 (Schritt 2.1.3), sofort es auf Eis, mit Alu-Folie, um die Platte vor dem direkten Kontakt mit Eis zu trennen. Lassen Sie Platte 1 bis 5 min abkühlen.
- Mit einem 12-Mehrkanal-Mikropipette und 200 µL Spitzen, 100 µL aus jedem Brunnen in Platte 2 (Schritt 2.1.4) in die entsprechenden Vertiefungen in die Platte 1 übertragen. Um das Toxin in den Medien der Platte 1 richtig zu verteilen, setzen Sie die Tipps unten der Meniskus und werfen Sie sanft die Lautstärke ohne Luftblasen.
Hinweis: Nicht rauf und runter, pipette, wie dies die Zellen versehentlich lösen kann. - Lassen Sie die Platte für eine zusätzliche 1 min um das Toxin an Zellen zu binden und Sofortüberweisung Platte 1 Platte Leser zum kinetischen Test mit dem Spectrofluorometer-Modus (Schritt 1.3.2) zu ermöglichen.
- Am Ende der kinetischen Test sofort Bild Platte 1 (Post-kinetische) mit Schritt 1.3.3.
3. Analyse: Cell Enumeration
- Bestimmen Sie die Zellzahl auf die nukleare Fluoreszenz unter Verwendung der Mikrotestplatte Zelle Enumeration Software basiert.
- Innerhalb der Einstellungen, wählen Sie Re-Analyseund unter der Kategorie Abschnitt innerhalb der Analyse Bildeinstellungen Diskrete Objekt-Analyse mit 541 als die Wellenlänge für die Suche nach Objekten.
- Innerhalb der Objekte finden-Option, mit der Suche nach Methode, Zeichnen Sie auf Bilder wählen Sie Kerne unter der Registerkarte "Einstellungen", und klicken Sie auf anwenden.
- Drücken Sie "OK" und Lesen Sie die Zelle zählen Algorithmus zu initiieren.
- Alternativ, wenn kein solches Tool verfügbar ist, verwenden Sie eine Bildanalyse-Software wie ImageJ Zellen auflisten.
- Öffnen Sie in ImageJ die Bilddatei als Stapel.
- Konvertieren des Stapels auf 8-Bit-Graustufen-Bilder durch Klick auf Bild in der Menüleiste, bewege den Mauszeiger über Typ, und wählen Sie 8-Bit.
- Subtrahieren Sie den Hintergrund: Klick Bild in der Menüleiste, zeigen Sie auf anpassen, und wählen Sie Helligkeit/Kontrast. Stellen Sie den Mindestwert um die Hintergrundgeräusche zu entfernen, und wählen übernehmen.
- Schwelle, binäre Abbilder zu erstellen: Klicken Sie Bild in der Menüleiste, zeigen Sie auf anpassen, und wählen Sie Schwelle. Wählen Sie dunklen Hintergrund aus, stellen Sie die minimale und maximale Grenzwerte, und klicken Sie auf anwenden.
- Bei Überschneidungen der Kerne, kann eine Wasserscheide Tool Segment Kerne verwendet werden. Klicken Sie auf Prozess im Menü, bewege den Mauszeiger über binäre und wählen Sie Wasserscheide.
Hinweis: Dies wird automatisch verbundenen Kernen trennen. - Analysieren Sie die maskierten Bilder nach benutzerdefinierten Kriterien (Größe und Zirkularität) die Identifizierung der Kerne zu verfeinern und Zellenrückstand ausschließen.
- Klicken Sie auf analysieren , in das Menü und dann analysieren Partikel. Legen Sie die gewünschte Größe (Pixel ^ 2) und Zirkularität (ein Wert von 1 ist ein perfekter Kreis) reicht, die ausreichen, um die einzelnen Zellen/Kerne enthalten.
- Wählen Sie im Feld Dropdown-Liste anzeigen die Option(en) gewünscht, überprüfen Sie zusammenfassenzu, und klicken Sie auf "OK" , um Zellen zu erhalten.
4. Analyse: Kinetische Kurven
- Übertragen Sie die kinetische Daten von der Platte-Reader-Software zum einen analytischen Daten-software
- Für jede Versuchsbedingung durchschnittlich die Fluoreszenz-Intensitäten der Wiederholungen bei jeder Timepoint, zusammen mit den entsprechenden Standardabweichung und Standardfehler des Mittelwerts für jede experimentelle Bedingung.
- Für jede Versuchsbedingung verfolgen die entsprechenden kinetische Kurve: PI Intensität (y-Achse) im Vergleich zur Zeit (x-Achse).
- Um die resealing Effizienz einer bestimmten Behandlung Bedingung zu berechnen, berechnen Sie die Fläche unter der Kurve (AUC) von der + LLO in M1 (AUC(M1)) und + LLO in M2 (AUC(M2)). Verwenden Sie den Ansatz zur Bewertung der Effizienz (E) der Wiederverschließen unten vorgeschlagen:
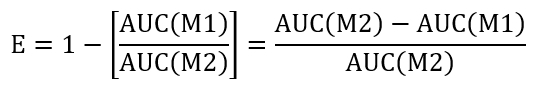
- Einen Abgleich zwischen Kontroll- und Behandlung durch die Bestimmung der Wirkungsgrad (REff) unten angegeben:
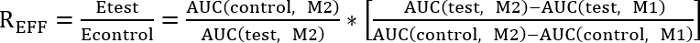
REFF = 1, Testbehandlung hat keine Auswirkung auf Reparatur
REFF < 1, Testbehandlung hemmt Reparatur
REFF > 1, Testbehandlung verbessert die Reparatur - Berechnen Sie die Fläche unter der Kurve unter Verwendung der folgenden Gleichung:
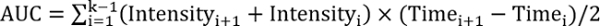 , wobei k ist die Gesamtzahl der Follow-ups.
, wobei k ist die Gesamtzahl der Follow-ups.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Zelle zählen Genauigkeit: HeLa-Zellen dienen häufig als eine Modellreihe Säugetier-Zelle Membran Reparaturmechanismen zu erkunden. Membran-Reparatur auf der Zellebene Bevölkerung zu beurteilen, ist es wichtig, Platte Zellen in der gleichen Konzentration in alle Wells für richtige Dateninterpretation. Es ist auch wichtig, die zum Zeitpunkt des Tests zu überprüfen, ob Zellzahlen über Brunnen gleichwertig sind. HeLa-Zellen, die konstitutiv Histon 2 b mit GFP (H2B-GFP) verschmolzen au...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Dieser Test misst die Effizienz der Membran Wiederverschließen auf Bevölkerungsebene Zelle mit hoher Durchsatzleistung. Es kann verwendet werden zum Bildschirm für zellulären Komponenten oder Droge-Bibliotheken, die Membran Reparatur beeinträchtigen könnten. Der beschriebenen Test verwendet eine 96-Well-Plattenformat, aber es kann auf 384-Well-Platten für höheren Durchsatz angepasst werden. Ein Vorteil dieser Assay ist seine Fähigkeit zur Fluoreszenz-Messungen von anhaftenden lebenden Zellen in Echtzeit ohne üb...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir bestätigen Dr. Jesse Kwiek (The Ohio State University) für freundlicherweise ermöglicht es uns, seine Multi-Mode-Erkennung-Plattform für einige Vorversuchen zu nutzen. Forschung berichtet in diesem Artikel wurde durch das National Institute of Allergy and Infectious Diseases von den National Institutes of Health unter Prämiennummer RO1AI107250, Stephanie Seveau unterstützt. Der Inhalt ist ausschließlich in der Verantwortung der Autoren und nicht unbedingt die offizielle Meinung der National Institutes of Health.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| SpectraMax i3x Multi-Mode Microplate Reader | Molecular Devices | i3x | |
| MiniMax 300 Imaging cytometer | Molecular Devices | 5024062 | |
| TO-PRO-3 | ThermoFisher Scientific | T3605 | |
| Propidium Iodide | ThermoFisher Scientific | P3566 | |
| HeLa | ATCC | CCL2 | |
| HeLa H2B-GFP | Millipore | SCC117 | |
| Trypsin-EDTA 0.25% | ThermoFisher Scientific | 25200056 | |
| 96-well Corning flat bottom black polystyrene tissue culture treated plate | Corning | 3603 | |
| Hanks' balanced Salts | Sigma-Aldrich | H4891 | |
| EGTA | ISC BioExpress | 0732-100G | |
| HEPES | Fisher Scientific | BP310-500 | |
| D-(+)-Glucose, HybriMax | Sigma-Aldrich | G5146-1KG |
Referenzen
- Demonbreun, A. R., McNally, E. M. Plasma Membrane Repair in Health and Disease. Current Topics in Membranes. 77, 67-96 (2016).
- Howard, A. C., McNeil, A. K., McNeil, P. L. Promotion of plasma membrane repair by vitamin E. Nature Communications. 2, 597(2011).
- Howard, A. C., et al. A novel cellular defect in diabetes: membrane repair failure. Diabetes. 60 (11), 3034-3043 (2011).
- Lozano, M. L., et al. Towards the targeted management of Chediak-Higashi syndrome. Orphanet Journal of Rare Diseases. 9, 132(2014).
- Vainzof, M., et al. Dysferlin protein analysis in limb-girdle muscular dystrophies. Journal of Molecular Neuroscience. 17 (1), 71-80 (2001).
- Huynh, C., et al. Defective lysosomal exocytosis and plasma membrane repair in Chediak-Higashi/beige cells. Proceeding of the National Academy of Sciences of the United States of America. 101 (48), 16795-16800 (2004).
- Cooper, S. T., McNeil, P. L. Membrane Repair: Mechanisms and Pathophysiology. Physiological Reviews. 95 (4), 1205-1240 (2015).
- Steinhardt, R. A., Bi, G., Alderton, J. M. J. M. Cell membrane resealing by a vesicular mechanism similar to neurotransmitter release. Science. 263 (5145), 390-393 (1994).
- De Mello, W. C. Membrane sealing in frog skeletal-muscle fibers. Proceedings of the National Academy of Sciences of the United States of America. 70 (4), 982-984 (1973).
- Fishman, H. M., Tewari, K. P., Stein, P. G. Injury-induced vesiculation and membrane redistribution in squid giant axon. Biochimica et Biophysica Acta. 1023 (3), 421-435 (1990).
- Davenport, N. R., Bement, W. M. Cell repair: Revisiting the patch hypothesis. Communicative & Integrative Biology. 9 (6), 1253643(2016).
- McNeil, P. L., et al. Patching plasma membrane disruptions with cytoplasmic membrane. Journal of Cell Science. 113 (11), 1891-1902 (2000).
- Terasaki, M., Miyake, K., McNeil, P. L. Large plasma membrane disruptions are rapidly resealed by Ca2+-dependent vesicle-vesicle fusion events. Journal of Cell Biology. 139 (1), 63-74 (1997).
- Bi, G. Q., Alderton, J. M., Steinhardt, R. A. Calcium-regulated exocytosis is required for cell membrane resealing. Journal of Cell Biology. 131 (6 Pt. 2), 1747-1758 (1995).
- Tam, C., et al. Exocytosis of acid sphingomyelinase by wounded cells promotes endocytosis and plasma membrane repair. Journal of Cell Biology. 189 (6), 1027-1038 (2010).
- Rodriguez, A., et al. Lysosomes behave as Ca2+-regulated exocytic vesicles in fibroblasts and epithelial cells. Journal of Cell Biology. 137 (1), 93-104 (1997).
- Reddy, A., Caler, E. V., Andrews, N. W. Plasma membrane repair is mediated by Ca(2+)-regulated exocytosis of lysosomes. Cell. 106 (2), 157-169 (2001).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Pathak-Sharma, S., et al. High-Throughput Microplate-Based Assay to Monitor Plasma Membrane Wounding and Repair. Frontiers in Cellular and Infection Microbiology. 7, 305(2017).
- Hamon, M. A., et al. Listeriolysin O: the Swiss army knife of Listeria. Trends in Microbiology. 20 (8), 360-368 (2012).
- Seveau, S. Multifaceted activity of listeriolysin O, the cholesterol-dependent cytolysin of Listeria monocytogenes. Subcellular Biochemistry. 80, 161-195 (2014).
- Osborne, S. E., Brumell, J. H. Listeriolysin O: from bazooka to Swiss army knife. Philosophical Transactions of the Royal Society of London B: Biological Sciences. 372 (1726), (2017).
- Lukoyanova, N., Hoogenboom, B. W., Saibil, H. R. The membrane attack complex, perforin and cholesterol-dependent cytolysin superfamily of pore-forming proteins. Journal of Cell Science. 129 (11), 2125-2133 (2016).
- Tweten, R. K. Cholesterol-dependent cytolysins, a family of versatile pore-forming toxins. Infection and Immunity. 73 (10), 6199-6209 (2005).
- Koster, S., et al. Crystal structure of listeriolysin O reveals molecular details of oligomerization and pore formation. Nature Communications. 5, 3690(2014).
- Duncan, J. L., Schlegel, R. Effect of streptolysin O on erythrocyte membranes, liposomes, and lipid dispersions. A protein-cholesterol interaction. Journal of Cell Biology. 67 (1), 160-174 (1975).
- Morgan, P. J., et al. Subunit organisation and symmetry of pore-forming, oligomeric pneumolysin. FEBS Letters. 371 (1), 77-80 (1995).
- Leung, C., et al. Stepwise visualization of membrane pore formation by suilysin, a bacterial cholesterol-dependent cytolysin. eLife. 3, (2014).
- Marchioretto, M., et al. What planar lipid membranes tell us about the pore-forming activity of cholesterol-dependent cytolysins. Biophysical Chemistry. 182, 64-70 (2013).
- Palmer, M., et al. Assembly mechanism of the oligomeric streptolysin O pore: the early membrane lesion is lined by a free edge of the lipid membrane and is extended gradually during oligomerization. European Molecular Biology Organization Journal. 17 (6), 1598-1605 (1998).
- Bavdek, A., et al. pH dependence of listeriolysin O aggregation and pore-forming ability. Federation of European Biochemical Society Journal. 279 (1), 126-141 (2012).
- Schuerch, D. W., Wilson-Kubalek, E. M., Tweten, R. K. Molecular basis of listeriolysin O pH dependence. Proceeding of the National Academy of Sciences of the United States of America. 102 (35), 12537-12542 (2005).
- Cassidy, S. K., O'Riordan, M. X. More than a pore: the cellular response to cholesterol-dependent cytolysins. Toxins (Basel). 5 (4), 618-636 (2013).
- Lam, J., et al. Host cell perforation by listeriolysin O (LLO) activates a Ca(2+)-dependent cPKC/Rac1/Arp2/3 signaling pathway that promotes L. monocytogenes internalization independently of membrane resealing. Molecular Biology of the Cell. , (2017).
- Gekara, N. O., Weiss, S. Lipid rafts clustering and signalling by listeriolysin O. Biochemical Society Transactions. 32 (Pt 5), 712-714 (2004).
- Magassa, N., Chandrasekaran, S., Caparon, M. G. Streptococcus pyogenes cytolysin-mediated translocation does not require pore formation by streptolysin O. European Molecular Biology Organization Reports. 11 (5), 400-405 (2010).
- Baba, H., et al. Induction of gamma interferon and nitric oxide by truncated pneumolysin that lacks pore-forming activity. Infection and Immunity. 70 (1), 107-113 (2002).
- Carrero, J. A., Vivanco-Cid, H., Unanue, E. R. Listeriolysin o is strongly immunogenic independently of its cytotoxic activity. Public Library of Science One. 7 (3), e32310(2012).
- Coconnier, M. H., et al. Listeriolysin O-induced stimulation of mucin exocytosis in polarized intestinal mucin-secreting cells: evidence for toxin recognition of membrane-associated lipids and subsequent toxin internalization through caveolae. Cell Microbiology. 2 (6), 487-504 (2000).
- Suzuki, T., et al. DNA staining for fluorescence and laser confocal microscopy. Journal of Histochemistry and Cytochemistry. 45 (1), 49-53 (1997).
- Bink, K., et al. TO-PRO-3 is an optimal fluorescent dye for nuclear counterstaining in dual-colour FISH on paraffin sections. Histochemistry and Cell Biology. 115 (4), 293-299 (2001).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nature Methods. 6 (8), 569-575 (2009).
- Zhang, X. D. A pair of new statistical parameters for quality control in RNA interference high-throughput screening assays. Genomics. 89 (4), 552-561 (2007).
- Zhang, X. D. A new method with flexible and balanced control of false negatives and false positives for hit selection in RNA interference high-throughput screening assays. Journal of Biomolecular Screening. 12 (5), 645-655 (2007).
- Idone, V., et al. Repair of injured plasma membrane by rapid Ca2+-dependent endocytosis. Journal of Cell Biology. 180 (5), 905-914 (2008).
- Davenport, N. R., et al. Membrane dynamics during cellular wound repair. Molecular Biology of the Cell. 27 (14), 2272-2285 (2016).
- Defour, A., Sreetama, S. C., Jaiswal, J. K. Imaging cell membrane injury and subcellular processes involved in repair. Journal of Visualized Experiments. (85), (2014).
- Lee, J. J. A., et al. Cell Membrane Repair Assay Using a Two-photon Laser Microscope. Journal of Visualized Experiments. (131), (2018).
- Weisleder, N., et al. Visualization of MG53-mediated cell membrane repair using in vivo and in vitro systems. Journal of Visualized Experiments. (52), (2011).
- Corrotte, M., et al. Toxin pores endocytosed during plasma membrane repair traffic into the lumen of MVBs for degradation. Traffic. 13 (3), 483-494 (2012).
- Kuismanen, E., Saraste, J. Low temperature-induced transport blocks as tools to manipulate membrane traffic. Methods in Cell Biology. 32, 257-274 (1989).
- Togo, T., et al. The mechanism of facilitated cell membrane resealing. Journal of Cell Science. 112, 719-731 (1999).
- Johnson, S. A., et al. Temperature-dependent phase behavior and protein partitioning in giant plasma membrane vesicles. Biochimica et Biophysica Acta. 1798 (7), 1427-1435 (2010).
- Lam, J. G. T., et al. Host cell perforation by listeriolysin O (LLO) activates a Ca(2+)-dependent cPKC/Rac1/Arp2/3 signaling pathway that promotes Listeria monocytogenes internalization independently of membrane resealing. Molecular Biology of the Cell. 29 (3), 270-284 (2018).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten