Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Design und Synthese eines rekonfigurierbaren DNA-Akkordeon-Racks
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir beschreiben das ausführliche Protokoll für Konstruktion, Simulation, nass-Laborversuche und Analyse für eine rekonfigurierbare DNA Akkordeon Rack 6 mal 6 Maschen.
Zusammenfassung
DNA-Nanostruktur-basierte mechanische Systeme oder DNA Nanomaschinen, die komplexe nanoskaligen Bewegung in 2D und 3D in den Nanometer Ångström Auflösung zu produzieren, zeigen großes Potenzial in verschiedenen Bereichen der Nanotechnologie wie die molekulare Reaktoren, Drug-Delivery, und Nanoplasmonic Systeme. Rekonfigurierbare DNA Akkordeon Rack, die gemeinsam eine 2D oder 3D nanoskaligen Netzwerk Elemente, in mehreren Stufen in Reaktion auf die DNA-Eingaben ändern können, wird beschrieben. Die Plattform hat Potenzial, die Anzahl der Elemente zu erhöhen, die DNA Nanomaschinen maßstabsgetreu ein Netzwerk mit mehreren Stufen der Neukonfiguration von ein paar Elemente kontrollieren kann.
In diesem Protokoll beschreiben wir den gesamten experimentellen Prozess der rekonfigurierbare DNA-Akkordeon-Rack 6 mal 6 Maschen. Das Protokoll enthält ein Berechnungsvorgang Regel und Simulation der Strukturen und eine nass-Laborversuch für Synthese und Neukonfiguration. Darüber hinaus ist die Analyse der Struktur mit TEM (Transmissions-Elektronenmikroskopie) und FRET (Fluoreszenz Resonanz Energietransfer) im Protokoll enthalten. Die neuartige Konstruktion und Simulation Methoden fallen in diesem Protokoll unterstützen Forscher, die DNA-Akkordeon-Rack für weitere Anwendungen zu verwenden.
Einleitung
Mechanische Systeme auf Basis von DNA Nanostrukturen oder DNA Nanomaschinen1,2,3,4,5 sind einzigartig, weil sie komplexe nanoskaligen Bewegung in 2D und 3D in den Nanometer zu produzieren Ångström Auflösung, nach verschiedenen biomolekularen Reize2,3,6. Durch Anbringen von funktionalen Materialien auf diese Strukturen und ihre Positionen zu kontrollieren, können diese Strukturen in verschiedenen Bereichen angewendet werden. Zum Beispiel wurden DNA-Nanomaschinen für eine molekulare Reaktor7, Drug-Delivery-8und Nanoplasmonic Systeme9,10vorgeschlagen.
Zuvor führten wir die rekonfigurierbare DNA-Akkordeon-Rack, die eine 2D oder 3D nanoskaligen Netzwerk Elemente11 (Abbildung 1A) manipulieren kann. Im Gegensatz zu anderen DNA-Nanomaschinen, die nur wenige Elemente steuern, kann die Plattform gemeinsam periodisch angeordneten 2D oder 3D Elemente in verschiedenen Stadien zu manipulieren. Wir erwarten, dass eine programmierbare chemische und biologische Reaktion-Netzwerk oder einem molekularen Computersystem aus unserem System gebaut werden kann durch die Erhöhung der Anzahl der steuerbaren Elemente. Das DNA-Akkordeon-Rack ist eine Struktur, in der das Netzwerk mehrere DNA-Balken mit Gelenken bestehend aus einzelsträngiger DNA (Abbildung 1 b) verbunden ist. Das Akkordeon Rack durch die DNA-Balken erzeugt wird durch die DNS-Sperren neu konfiguriert die hybridisieren auf den klebrigen Teil der Strahlen und ändern Sie den Winkel zwischen den Balken entsprechend der Länge der Brücke Teil der Schleusen (gesperrten Zustand). Darüber hinaus zeigt mehrstufigen Neukonfiguration durch das Hinzufügen von neuen Schleusen nach Bildung des Freistaates durch DNA-Sperren durch Brückenkopf-basierte Strang Verdrängung12,13abnehmen.
In diesem Protokoll beschreiben wir den Gesamtprozess für Design und die Synthese der rekonfigurierbare DNA-Akkordeon-Rack. Das Protokoll umfasst Konstruktion, Simulation, nass-Laborversuche und Analyse für die Synthese von DNA-Akkordeon Rack 6 mal 6 Maschen und eine Neukonfiguration von diesen. Die Struktur des Protokolls ist das Basismodell der bisherigen Forschung11 und 65 nm von 65 nm Größe, bestehend aus 14 Strahlen. In Bezug auf die Konstruktion und Simulation unterscheidet sich der konstruktive Aufbau des Akkordeon Racks von konventionellen DNA Origami14,15 (d. h. dicht gepackt). Daher wurden die Gestaltungsregel und molekulare Simulation von traditionellen Methoden modifiziert. Um zu demonstrieren, zeigen wir die Designtechnik mit der modifizierten Ansatz von CaDNAno14 und die Simulation des Akkordeon Racks mit zusätzliche Skripte OxDNA16,17 . Schließlich sind beide Protokolle der TEM und Bund für die Analyse der konfigurierten Akkordeon Rack Strukturen beschrieben.
Protokoll
1. Aufbau der 6 von 6 DNA-Akkordeon-Rack mit CaDNAno14
- Herunterladen und installieren CaDNAno 2.0 Software14 um ein DNA-Akkordeon-Rack zu entwerfen (CaDNAno 2.5 ist auch verfügbar auf https://github.com/cadnano/cadnano2.5). Öffnen Sie CaDNAno14 , und klicken Sie auf das Quadrat-Werkzeug fügen Sie ein neues Bauteil mit einem quadratischen Gitter.
- Nummer jeder Strahl des Racks, Akkordeon und zeichnen Sie auf der linken Gitter des CaDNAno14 (Abbildung 2).
- Klicken Sie auf das Bleistiftwerkzeug und ziehen Sie jeden Strahl auf die richtige Bearbeitungsfeld auf der CaDNAno14. Pause strahlt jede 32 bp, die für Fugen zwischen benachbarten Balken ist. Legen Sie Grundnahrungsmittel Frequenzweichen in der gleichen Position wie die Gelenke. Verwenden Sie das Tool einfügen und das Buntstift-Werkzeug in CaDNAno14 zusätzliche einsträngige Frequenzweichen haben Gelenke lassen.
- Klicken Sie auf das Bleistift-Werkzeug und verbinden Sie die Gelenke zu. Jeder Strahl hat sieben Gelenke.
- Gerüst Frequenzweichen um die einzelne Schleife die Gerüste zusammenführen, mithilfe der zuvor gemeldete Gerüst routing Algorithmus11zu generieren. Lassen Sie sich nicht die minimale bindende Domäne zwischen Gerüst und Grundnahrungsmittel Stränge werden weniger als 8 bp (Abbildung 3).
- Platzieren Sie die Gerüste, die nicht verwendet werden in der Baugruppe an den Scheitelpunkten auf gegenüberliegenden Seiten des Akkordeon Racks, gelegen, wie in Abbildung 3dargestellt.
- Klicken Sie auf das Werkzeug zu brechen. Brechen die Stränge wo Grundnahrungsmittel Stränge kreisförmige oder länger als 60 sind bp.
- Die DNA-Stränge Sperre zu entwerfen.
- Klicken Sie auf das Werkzeug zu brechen. Pause 8 bp ein Grundnahrungsmittel DNA-Region ein klebrigem Teil zu löschen 8 bp ein Grundnahrungsmittel DNA-Region. In der 6 x 6-Akkordeon-Rack gibt es 18 klebrigen Teile (Abbildung 1).
- Platzieren Sie Sequenzen, die umgekehrte sind an beiden Enden der Sperre Stränge komplementär zu den klebrigen Teile zu und verbinden sie durch eine Überbrückung Region, bestehend aus Poly T Stränge der gewünschten Länge (Abbildung 1 b).
- Für die Neukonfiguration hinzufügen 8 bp Brückenkopf Sequenzen am Ende des DNA sperrt für Strang Verschiebung. Die Brückenkopf-Sequenz verwendet, ist in Tabelle 2.
- Bereiten Sie Poly A Stränge, die komplementär zu der Überbrückung Region rückgängig zu machen sind.
- Design-Stränge, die komplementär zu der DNA-Schlösser für die Neukonfiguration Experiment rückgängig zu machen sind.
- Klicken Sie auf die Sequenz-Werkzeug und klicken Sie auf Gerüst DNA. Wählen Sie das Gerüst als standard M13mp18. Klicken Sie auf das Export-Tool und speichern Sie die Sequenz im Csv-Format (Tabelle 1).
(2) die Struktur mit dem OxDNA zu simulieren.
- Downloaden Sie und installieren Sie der OxDNA16,17. Die neuesten Quellcode ist verfügbar auf https://sourceforge.net/projects/oxdna/files/.
- Stellen Sie Start Konfigurations-Dateien aus der CaDNAno-14 -Datei mit Python-Skript "cadnano_interface.py", die in der OxDNA16,-17 -Paket bereitgestellt wird. Die Nutzung ist wie folgt: 'Python cadnano_interface.py cadnano_file.json sq'. Jetzt werden die Topologie und Konfigurationsdatei generiert.
Hinweis: Die topologiedatei enthält wie viele Stränge und Nukleotide sind in der Struktur und Informationen über Rückgrat-Backbone Verbindungen zwischen Nukleotiden. Die Konfigurationsdatei enthält allgemeine Informationen wie Zeitschritt, Energie und Boxgröße. Orientierungsinformationen wie Position Vektor, Rückgrat-Base Vektor, Normalenvektor, Velocity und Winkelgeschwindigkeit der Nukleotide ist auch enthalten (Abbildung 4). - Ändern Sie die Informationen in der Topologie und Konfiguration-Datei vom CaDNAno14 zu machen, die echte strukturelle Informationen des Akkordeon Racks zu reflektieren. Alle Balken sind parallel angeordnet, wenn die Topologie und Konfiguration-Dateien von CaDNAno14 visualisiert werden. Allerdings ist das Akkordeon Rack eine Gitterstruktur, so dass der Abstand zwischen verklebten Nukleotide sind weit für Simulation (Abbildung 5).
- Drehen Sie und verschieben Sie jeder Strahl auf die gewünschte Gitterstruktur. Die neun Spalten auf der linken Seite in der Konfigurationsdatei sind der Position Vektor, Rückgrat-Base Vektor und Normalenvektor (Abbildung 4). Um einen Lichtstrahl zu drehen, drehen Sie alle Stellung, Rückgrat-Base und normalen Vektoren mit rotatorischen Transformation. Dann bewegen Sie einen Strahl durch Veränderung des Position Vektors um es zu finden, wie in Abbildung 5dargestellt.
- Entspannen Sie die Struktur mit dem Skript in das OxDNA-Paket zur Verfügung gestellt (siehe Beispiel in $oxDNA/Beispiele/RELAX_INITIAL_CONFIGURATION für weitere Informationen).
- Molekulardynamik-Simulation für 10 Millionen Schritte mithilfe der entspannten Konfigurationsdatei ausgeführt. Die Nutzung ist wie folgt: '. / OxDNA < Eingabe >' speichern Daten alle 5000 oder 10000 Schritte.
- Visualisierung
Hinweis: Die Strukturen wurden mit Cogli visualisiert.- Downloaden Sie und installieren Sie die neueste Version von Cogli (https://sourceforge.net/projects/cogli1/).
- Führen Sie die Cogli mit der Topologie und Konfiguration Dateien aus der OxDNA-Simulation. Die Nutzung ist wie folgt: '. / cogli1 -t < topologiedatei >< Konfigurations-Datei > ".
- Verstecken Sie die Box mit der Taste b.
(3) Synthese der Struktur
Hinweis: Die Synthese-Methode wird von der vorherigen Protokoll15,18angepasst.
- Erwerben Sie die gestalteten DNA-Klammern bei einem Oligonukleotid-Anbieter.
- Passen Sie die Konzentration von diesen DNA-Klammern zu 100 μM mit Nuklease-freies Wasser.
- Bündeln Sie jeder DNA-Strang, die eine "free State" Struktur in eine Röhre bildet und passen Sie die Konzentration auf 2 μM für jeden Strang.
- Pool DNS Sperre Stränge durch Länge und Anzahl der Sperre Orte in Röhren und passen Sie die Konzentration auf 2 μM für jeden Strang. 18, 9 und 4 Sperre Websites dienen. Fügen Sie Poly-A-Stränge, die zu überbrückenden Region bei der gleichen Konzentration sind.
- Pool-Stränge, die komplementär zu der DNA umzukehren sind Sperren Stränge durch Länge in Röhren und passen Sie die Konzentration auf 2 μM für jeden Strang.
- Bereiten Sie MgCl2 Lösung von 300 nM durch Mischen von 70 μL der Nuklease-freies Wasser und 30 μl 1 μM MgCl2 Lösung. Bereiten Sie eine 5 x Tris-EDTA-Lösung durch Mischen von 95 μL der Nuklease-freies Wasser und 5 μL der 100 X-Tris-EDTA-Lösung.
- Fügen Sie 2 μL Heftklammer DNA, 1,1 μl MgCl2 Lösung, 2 μl Tris-EDTA-Lösung, 7,6 μl der Nuklease-freies Wasser und 7,3 μL Gerüst DNA von denen die Konzentration beträgt 110 nM 20 μL des gemischten Materials zu machen. Setzen Sie die Endkonzentration des Gerüstes DNA auf 40 nM, Heften DNA zu 200 nM, MgCl2 bis 16 mM und Tris-EDTA-Lösung 0,5 X.
- Erhitzen Sie schnell gemischte Vorratslösung in einem Thermocycler bis 80 ° C und kühlen bis 60 ° C mit einer Rate von 4 min pro ° C und kühlen von 60 ° C bis 4 ° C mit einer Rate von 40 min pro ° C.
4. Reinigung der Struktur
Hinweis: Die Proben aller Strukturen wurden vor der Analyse gereinigt. In diesem Abschnitt beschreiben wir das Protokoll der PEG Reinigung, vom früheren Literatur19angepasst ist. Die Probe kann auch durch Gelelektrophorese gereinigt werden, wie im vorherigen Literatur15,18beschrieben.
- 5 M NaCl und 100 X Tris-EDTA vorzubereiten.
- Bereiten Sie Niederschlag-Puffer durch das Mischen von 150 μL der PEG 8000, 500 μL 100 x Tris-EDTA und 101 μL 5 M NaCl und 249 μL der Nuklease-freies Wasser.
- Bereiten Sie Ziel-Puffer durch das Mischen von 5,5 μl 300 nM MgCl2 Lösung von Abschnitt 3.3, 10 μl 5 X Tris-EDTA-Lösung von Abschnitt 3.3 und 84,5 μL der Nuklease-freies Wasser.
- Mix 20 μL der synthetisierten Struktur aus Abschnitt 3 und 20 μL des Niederschlag-Puffer von Abschnitt 4.2. Dann drehen Sie den gemischten bestand bei 16000 X g bei 4 ° c Entfernen des Überstands und Pellet in den Zielpuffer aus Abschnitt 4.3 auflösen.
(5) Neukonfiguration des Akkordeon Racks von einem "freien Staat" zu "Gesperrten Zustand"
- Die Struktur ohne DNA-Schlösser für die Konfiguration-Experiment zu synthetisieren.
- Bereiten Sie DNS-Sperre Stränge aus Abschnitt 3.
- Fügen Sie 2 μL der DNA-Stränge der Sperre der gewünschten Länge in 20 μL der synthetisierten Struktur. Die DNA-Sperre Stränge Konzentration ist fünfmal höher als die Struktur.
- Inkubation der Probe für 0, 10, 25, 50 oder 100 Minuten zu sehen, wie schnell Neukonfiguration auftritt.
- Für die Inkubation von 100 Minuten 30 Minuten Probe bei 50 ° C inkubieren und bis zu 25 ° C mit einer Rate von 0,33 ° C/min langsam abkühlen.
- Für die 50-minütige Inkubation Inkubation der Probe bei 50 ° C für 15 Minuten und bis zu 25 ° C mit einer Rate von 0,66 ° C/min langsam abkühlen.
- Für die 25 minütige Inkubation 7,5 Minuten Probe bei 50 ° C inkubieren und bis zu 25 ° C mit einer Rate von 1,32 ° C/min langsam abkühlen.
- Für die 10 Minuten Inkubation 3 Minuten Probe bei 50 ° C inkubieren und bis zu 25 ° C mit einer Rate von 3,3 ° C/min langsam abkühlen.
- Für die 0-minütige Inkubation speichern Sie Muster 4 ° C rechts nachdem Schleusen DNA-Stränge hinzugefügt werden.
- Direkt nach dem anbringen Schritt rasch Abkühlen der Probe auf 4 ° C zu verhindern, dass unerwünschte Denaturierung.
6. Umgestaltung des Akkordeon Racks aus "Gesperrten Zustand" zu einem "freien Staat"
- Die Struktur mit DNA-Schlössern der gewünschten Länge für die Konfiguration-Experiment zu synthetisieren.
- Umgekehrte komplementäre Stränge aus Abschnitt 3 vorbereiten.
- Fügen Sie 2 μL der Stränge, die komplementär zu der Sperre Stränge der gewünschten Länge in 20 μL der synthetisierten Struktur rückgängig zu machen sind. Die DNA-Sperre Stränge Konzentration ist fünfmal höher als die Struktur.
- Inkubation der Probe für 0, 12, 60, 120, 240 Minuten zu sehen, wie schnell Neukonfiguration auftritt.
- Für die 12, 60, 120, 240 Minuten Inkubation, rasch die Probe auf 40 ° C erhitzen und langsam abkühlen auf 20 ° C für die Zeit, die für jede. Direkt nach dem Trennen Schritt rasch Abkühlen der Probe auf 4 ° C zu verhindern, dass unerwünschte Denaturierung.
- Für die 0-minütige Inkubation speichern Sie Muster 4 ° C rechts nach rückwärts komplementäre Stränge hinzugefügt werden.
(7) TEM Imaging
Hinweis: TEM-imaging-Protokoll wurde von früheren Literatur18,20angepasst.
- Bereiten Sie 1,25 M NaOH-Lösung durch Mischen von 87,5 μL der Nuklease-freies Wasser und 12,5 μl 10 M NaOH Lösung vor.
- 50 μL der 2 %-Uranyl-Formiat-Lösung 1 μl 1,25 M NaOH-Lösung hinzufügen.
- Vortex: die Lösung für 3 Minuten und Zentrifuge bei max. Drehzahl für 3 Minuten. Kaution 3 μL der gereinigten Probe in der Glut entladen TEM Startaufstellung für 3 Minuten und schnell auswaschen mit Filterpapier.
- 7 μL bereit Uranyl Formiat Lösung für 30 Sekunden zu hinterlegen und schnell auswaschen mit Filterpapier.
- Messen Sie die Länge und den Winkel der Akkordeon-Struktur durch TEM abgebildet.
(8) Bund-Analyse
- Verwenden Sie Atto 550 und Atto 647N Farbstoff, wofür die Förster-Entfernung 6,5 beträgt nm. Ersetzen Sie Grundnahrungsmittel 58 und Grundnahrungsmittel 117 in Tabelle 1 mit Gewebekulturen gekennzeichneten Adern. Dann synthetisieren Sie die Struktur mit Gewebekulturen gekennzeichneten Adern durch die in Abschnitt 3 beschriebenen Methode.
- Die Konzentration der gereinigten Probe zu messen.
- Normalisieren Sie die Probe 10 nM und Last 50 μL, 384 Mikroplatten gut.
- Begeistern Sie die Probe mit Donor und Akzeptor Farbstoffe bei 550 nm und messen die Fluoreszenz-Spektrum von 570 nm bis 800 nm mit einem Fluorometer.
- Die Fluoreszenz-Spektrum der Spender nur Probe auf die gleiche Weise zu messen.
- Begeistern die Farbstoffe der Probe bei 650 nm und die Fluoreszenz-Spektrum und Messen von 670 nm bis 800 nm. Dies ist zur Messung der Konzentration der Akzeptor.
- Erhalten Sie die Standardabweichungen durch Wiederholung des gleichen Experiments mit drei Proben, die synthetisiert werden und separat gereinigt.
- Berechnung der FRET-Effizienz mit dem Verhältnis eine Methode wie beschrieben durch die Gleichung unten21.
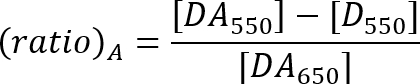
DA550: Akzeptor-Peak-Fluoreszenz-Intensität der Probe mit Donor und Akzeptor bei 550 nm Anregung.
D-550: Fluoreszenz-Intensität an den Akzeptor Emissionsbereich der Spender nur Probe bei 550 nm Anregung.
DA650: Akzeptor-Peak-Fluoreszenz-Intensität der Probe mit Donor und Akzeptor bei 650 nm Anregung.
Ergebnisse
Gestalteten 6 mal 6 DNA Akkordeon Rack wird von den OxDNA16,17 simuliert und die Ergebnisse sind in Abbildung 6dargestellt. Aus dem Simulationsergebnis bestätigte sich, dass die beabsichtigte Struktur ohne Verzerrung der Struktur gebildet wird.
Die TEM-Bilder in Abbildung 7 sind Bilder der konfigurierten Strukt...
Diskussion
Dieses Protokoll stellt den gesamten Prozess aus Konstruktion, Simulation, Synthese und Analyse von der 2D DNA Akkordeon Grundregal. Das geänderte Design und Simulation Regeln sind beschrieben worden weil standard DNA Origami Design Rule anders, dass die DNA-Akkordeon-Rack zusätzliche Nukleotide an die Frequenzweichen für Flexibilität14,15hat. Aus diesem Grund erwarten wir, dass das Protokoll verschiedene Forschungen mit DNA-Akkordeon Regale beschleunigen kan...
Offenlegungen
Die Autoren haben nichts preisgeben
Danksagungen
Diese Forschung wurde teilweise durch das Global Research Center Entwicklungsprogramm durch die National Research Foundation von Korea(NRF) gefördert durch das Ministerium für Wissenschaft und IKT (MSIT) (2015K1A4A3047345) und Nano· unterstützt. Material Technology Development Program durch die National Research Foundation von Korea (NRF) gefördert durch das Ministerium für Wissenschaft und IKT (MSIT) (2012M3A7A9671610). Das Institute of Engineering Research an der Seoul National University zur Verfügung gestellt Forschungseinrichtungen für diese Arbeit. Autoren erkennen Dankbarkeit gegenüber Tae-Young Yoon (Biological Sciences, Seoul National University) bezüglich der Fluoreszenz-Spektroskopie für die Bund-Analyse.
Materialien
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Referenzen
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten