Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Automatisierte bildbasierte Quantifizierung von neutrophilen extrazellulären Fallen mit NETQUANT
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Erzeugung von neutrophilen extrazellulären Fallen (NETs) und zum Betrieb von NETQUANT vor, einer vollautomatischen Softwareoption zur Quantifizierung von NETs in Immunfluoreszenzbildern.
Zusammenfassung
Neutrophile extrazelluläre Fallen (NETs) sind webartige antimikrobielle Strukturen, die aus DNA und Granulat abgeleiteten antimikrobiellen Proteinen bestehen. Immunfluoreszenzmikroskopie und bildbasierte Quantifizierungsmethoden bleiben wichtige Werkzeuge zur Quantifizierung der neutrophilen extrazellulären Trapbildung. Es gibt jedoch wesentliche Einschränkungen für die immunfluoreszenzbasierten Methoden, die derzeit für die Quantifizierung von NTs verfügbar sind. Manuelle Methoden der bildbasierten NET-Quantifizierung sind oft subjektiv, fehleranfällig und für Benutzer, insbesondere unerfahrene Benutzer, mühsam. Auch die derzeit verfügbaren Softwareoptionen zur Quantifizierung sind entweder halbautomatisch oder erfordern eine Schulung vor dem Betrieb. Hier zeigen wir die Implementierung einer automatisierten Immunfluoreszenz-basierten Bildquantifizierungsmethode zur Bewertung der NET-Bildung namens NETQUANT. Die Software ist einfach zu bedienen und verfügt über eine benutzerfreundliche grafische Benutzeroberfläche (GUI). Er berücksichtigt biologisch relevante Parameter wie eine Erhöhung der Oberfläche und das DNA:NET-Markerproteinverhältnis und die kerntechnische Verformung, um die NETZbildung zu definieren. Darüber hinaus ist dieses Tool als frei verfügbare App aufgebaut und ermöglicht eine Quantifizierung und Analyse der Einzelzellenauflösung.
Einleitung
Neutrophile sind entscheidende Vermittler von angeborenen Wirtsabwehrreaktionen gegen eine Vielzahl von mikrobiellen Erregern1. Sie führen ihre antimikrobiellen Funktionen aus, indem sie ihr Granulat freisetzen, das eine breite Palette von antimikrobiellen Proteinen2enthält, reaktive Sauerstoffspezies (ROS) und Hypochlorit1produziert und durch Phagozytose3. Darüber hinaus Brinkmann et al. 4 beschrieb neutrophile extrazelluläre Fallen (NETs) als einen neuartigen Mechanismus, durch den Neutrophile eindringende Krankheitserreger fangen und beseitigen. Seit ihrer Entdeckung vor etwas mehr als einem Jahrzehnt4,NETs wurden in einer Vielzahl von infektiösen5,6 und nicht-infektiösen7 Morbiditäten verwickelt. DIE NET-Bildung ist ein aktiver Prozess und führt zur Extrusion von Chromatin-DNA, die mit granulabgeleiteten antimikrobiellen Proteinen8beschichtet ist. Einige der wichtigsten Veränderungen in der zellulären und nuklearen Morphologie im Zusammenhang mit der NET-Bildung sind der Verlust der nuklearen Morphologie, Chromatin-Dekondensation, Mobilisierung von Granulatproteinen vom Zytoplasma zum Zellkern und eine Erhöhung des kern- und zellulären Durchmessers8,9.
Die webartigen NETs, die als diffuse Strukturen erscheinen können, die etwas größer als die Zelle sind, oder als Strukturen, die um ein Vielfaches größer sind als ein einzelnes Neutrophiler, werden als Indikatoren für NETosis5,10betrachtet. Mittels Fluoreszenzmikroskopie können NETs durch Diesprossen mit einer fluoreszierenden Sonde wie 4',6-Diamidino-2-Phenylindole (DAPI) und durch Immunfluoreszenzfärbung gegen NET-gebundene Proteine wie neutrophile Elastase nachgewiesen werden. Die Quantifizierung überlappender Gebeikbereiche für DNA und NET-gebundene Proteine bestimmt die Gesamtfläche unter NETs in einem Bild11.
Für die Fluoreszenzbild-basierte Quantifizierung von NETs11,12stehen eine Reihe von Bildanalyseoptionen zur Verfügung. Diese Softwareoptionen stellen jedoch Einschränkungen dar, da sie nicht benutzerfreundlich und/oder vollautomatisiert sind. In diesem Artikel zeigen wir den Betrieb von NETQUANT13, einer frei verfügbaren App, die unvoreingenommene vollautomatische Immunfluoreszenzmikroskopie bildbasierte NET-Quantifizierung durchführen kann. Die App verfügt über eine benutzerfreundliche grafische Oberfläche (GUI) und kann eine Einzelzellenanalyse durchführen. Die Software quantifiziert NETosis in einem Bild, indem sie die morphologischen Veränderungen im Bereich des DNA-NET-gebundenen Markers, der Chromatindekondensation im Zusammenhang mit der Verformung des Kerns und der Erhöhung des DNA:NET-gebundenen Proteinverhältnisses erkennt. Zusammengenommen ermöglichen die kriterien für mehrere NET-Definitionen eine strenge NET-Quantifizierung über mehrere Datensätze hinweg in unvoreingenommener Weise.
Protokoll
Die Ethikkommission der Universität Lund genehmigte die Entnahme von Venenblut von gesunden Freiwilligen gemäß der Erklärung von Helsinki (2013/728). Alle Freiwilligen erteilten ihre schriftliche Einwilligung in Kenntnis der Sachlage.
1. Isolierung von peripheren Blutneutrophilen mit Dichte-Gradientzentrifugation
- Sammeln Sie menschliches venöses Blut in Schläuchen, die Heparin enthalten, und lassen Sie die Röhren Raumtemperatur erreichen.
Hinweis: Mindestens 16 ml Blut von einem gesunden Spender sind erforderlich, um ein ausreichend großes Zellpellet zu ergeben. - Mischen Sie das Blut mit einem Volumen von 2% Dextran in Saline (0,9% NaCl) und lassen Sie Sediment bei Raumtemperatur für 30 min in einem sterilen 50 ml konischen Zentrifugenrohr sedimentieren.
- Den Überstand in ein steriles 50 ml kegelförmiges Zentrifugenrohr und zentrifugieren bei 200 x g für 10 min bei 4 °C aspirieren.
- Ab diesem Schritt weiter isoliert bei 4 °C oder auf Eis.
- Das Pellet in 5 ml eiskalter Kochchenschicht auf5 ml Leukozytenisolationsgradienten (9,1% Natriumdiatrizoat mit 5,7% Dextran, w/v) in einem sterilen 15 ml konischen Zentrifugenrohr wieder aufhängen.
- Zentrifuge für 30 min bei 400 x g bei 4 °C.
- Aspirieren Sie den Überstand und entsorgen Sie ihn.
- Lyse rote Blutkörperchen durch Wiederauflegen des Pellets in 3 ml eiskaltem Wasser für 30 s. Sofort 1 ml von 3,6% NaCl hinzufügen und dann mit 10 ml eiskalte Salzwäsche füllen.
- Zentrifugieren Sie die Zellsuspension für 10 min bei 350 x g.
- Entfernen Sie den Überstand, sammeln Sie das Zellpellet und setzen Sie es in 1 ml Saline wieder auf. Zur Beurteilung der Zellzahl und Lebensfähigkeit mit Trypanblau in einer Bürker-Kammer 10 l in einem Mikrozentrifugenrohr beiseite legen.
- Fügen Sie 10 l Zellsuspension zu 90 l von 0,4% Trypan-Blau-Lösung hinzu. Nehmen Sie 10 l der Zellsuspension in einer Bürker-Kammer. Zählen Sie die Zellen in den 4 Quadraten, die durch 3 Linien an jeder Ecke der Kammer gebunden sind. Zellen, die dunkelblau erscheinen, um Farbstoff aufzunehmen, sind nicht lebensfähig und schließen sie von der Gesamtzellenzahl aus.
- Geben Sie die Zellenzahl als Zellen/ml aus, wie in der folgenden Gleichung definiert.
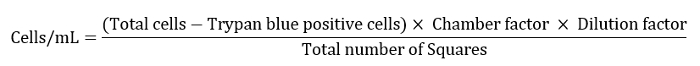
Hinweis: Hier betrug der Kammerfaktor 10.000, der Verdünnungsfaktor 10 und die Gesamtzahl der Quadrate 4. - Die verbleibende Zellsuspension für einen letzten Waschschritt auf 10 ml verdünnen.
- Zentrifuge für 5 min bei 200 x g.
- Setzen Sie die Neutrophilen in RPMI-1640 mit 2 mg/ml wärmeinaktiviertem humanem Serumalbumin (HSA) in einer Konzentration von 5 x 105 Zellen/ml aus.
2. Herstellung von Coverslips und Stimulation von Neutrophilen
- Legen Sie einen Deckelschlupf (10 mm, #1) in jede Bohrung einer 12-Well-Platte und beschichten Sie den Deckelschlupf, indem Sie 200 l 0,01 % Poly-L-Lysin-Lösung hinzufügen und ihn über Nacht bei 37 °C belassen.
- Die Deckellipsen mit 300 l Phosphatpuffer-Saline (PBS) einmal waschen und trocknen lassen.
- Fügen Sie 400 l von 5 x 105 Neutrophilen/ml zu jedem Brunnen hinzu und inkubieren Sie bei Raumtemperatur für 15 min.
- Bewegen Sie die Platte mit Neutrophilen in einen Inkubator bei 37 °C mit 5%CO2 für 15 min.
- Entfernen Sie den Überstand. Fügen Sie den Kontrollen 400 L vorgewärmtes RPMI-1640 Medium mit 2 mg/ml HSA hinzu. Fügen Sie 300 l vorgewärmten RPMI mit 20 nM Phorbol 12-Myristate 13-Acetat (PMA) zur Stimulation hinzu.
- Stimulieren Sie Neutrophile für 150 min bei 37 °C mit 5%CO2.
3. Visualisierung von NETs
- Entfernen Sie den Überstand und waschen Sie die Proben 2x mit 200 l PBS.
- Befestigen Sie Proben, indem Sie 200 l 4% Paraformaldehyd (PFA) in PBS für 20 min bei 37 °C hinzufügen.
Hinweis: PFA ist giftig und muss mit Vorsicht behandelt werden. - Waschen Sie Proben 3x mit 200 l PBS.
- Permeabilisieren Sie die Proben, indem Sie 50 l von 0,5% Triton X-100 für 30 s hinzufügen.
- Waschen Sie die Proben 3x mit 200 l PBS.
- Blockieren Sie die Proben mit 5% Ziegenserum in PBS für 1 h bei 37 °C.
- Fügen Sie 300 l primärkaninchen antihumane neutrophile Elastase in einer blockierenden Lösung bei einer Verdünnung von 1:500 für 90 min bei 37 °C hinzu.
- Waschen Sie die Proben 3x mit 300 l PBS.
- Fügen Sie 300 l sekundären Ziegen-Anti-Kaninchen-Fluoreszenz-Antikörper bei einer Verdünnung von 1:1000 für 90 min bei 37 °C hinzu.
- Waschen Sie die Abdeckungen 3x mit 300 l PBS.
- Entfernen Sie die Abdeckungen aus den Brunnen und montieren Sie den Deckelmitrutsch mit einem 10-L-Montagemedium, das DAPI enthält. Über Nacht bei Raumtemperatur im Dunkeln lagern, um die Proben zu trocknen.
Hinweis: Die Färbung von DNA mit DAPI ist sicherlich ein kritischer Schritt in der Methode. Die Benutzer können auch Fehler beheben, indem sie exogene DAPI-Lösung in einem endgültigen Konzentrationsbereich von 0,1,20120,5 g/ml für 2-u20123 min, gefolgt von 3 Waschschritten mit 300 l PBS. - Erfassen Sie Bilder mit einem Weitfeldfluoreszenzmikroskop mit einem 20-Fach-Objektiv.
4. Analyse und Quantifizierung von NETs mit NETQUANT
Hinweis: NETQUANT kann heruntergeladen werden, indem Sie auf die Installationsdatei im Zenodo Github Archiv oder auf der Nordenfelt Lab Website (https://nordlab.med.lu.se/?page_id=34) klicken.
- Importieren von Datensätzen zur Analyse, Benennung von Kanälen und Konvertierung von Bildern
- Öffnen Sie die Registerkarte Setup in NETQUANT.
- Wählen Sie den Quellordner für die Analyse aus, indem Sie im Quellmenü auf die Option Pfad abrufen klicken und den Ordner mit den zu analysierenden Bildsequenzen auswählen.
- Klicken Sie im Zielmenü auf die Option Pfad abrufen und wählen Sie den Ordner zum Speichern der Daten nach der Bildanalyse aus.
- Benennen Sie die Kanäle so, dass "DNA-Kanal" mit der DNA-Färbung(z.B.DNA oder DAPI) korrespondiert und "NET-Kanal" die NET-gebundene Proteinfärbung(z.B.NET, Neutrophilelastase) in den Bildern darstellt. Für das reibungslose Funktionieren der Software benennen Sie den Ordner, der die Systemabbilddateien enthält, als "Steuerung".
Hinweis: Der NET-Kanal bezieht sich nur auf die NET-gebundene Granulatprotein-Markerfärbung. - Führen Sie die Bildmetadaten in die Software ein, indem Sie im Untermenü Bildinformationen auf die Schaltfläche Bildinformationen laden klicken.
- Wählen Sie die richtige Kanalreihenfolge aus, die in den Bildern im Untermenü Kanalreihenfolge enthalten ist. Diese Option wurde als Ausfallschutz option, um versehentliche Inkongruenzen zu verhindern.
- Erfassen Sie primäre Bildeigenschaften aus den Rohdaten, und konvertieren Sie die Bilder, indem Sie auf die Schaltfläche Daten vorbereitenklicken . Die konvertierten Bilder werden im Untermenü "Beispieltyp" angezeigt. Klicken Sie auf das Menü Beispieltyp, um alle für die Analyse erworbenen Datensätze anzuzeigen und auszuwählen.
- Wählen Sie ein Bild aus dem Untermenü "Beispieltyp" aus, und klicken Sie auf die Schaltfläche Bilddaten anzeigen, um die in den DNA- bzw. NET-Kanal aufgeteilten Bilder anzuzeigen.
- Segmentierung von Zellen im DNA-Kanal und im NET-Kanal
- Wählen Sie die Segmentierungsmethode aus, indem Sie auf das Untermenü Methode sowohl im DNA-Kanal als auch im NET-Kanal klicken.
Hinweis: Die Standardmethode der Segmentierung ist auf adaptiv festgelegt und ist die empfohlene Einstellung. Weitere Optionen sind ebenfalls verfügbar, darunter global, edge und Chan-Vese. Eine Wasserscheide Option ist auch enthalten, um zu helfen, zwischen eng platzierten Zellen oder NETs zu unterscheiden. - Geben Sie die Registerkarte Segmentierung ein, um Steuerzellen zuerst in beiden Kanälen zu segmentieren, indem Sie auf die Option Segmentsteuerungsmuster klicken.
- Wählen Sie PMA aus dem Untermenü des Beispieltyps aus und klicken Sie auf die Option Batch (empfohlen), um mit der Segmentierung aller im Datensatz enthaltenen Bilder zu beginnen. Wählen Sie die Bilder im Beispieltypmenü aus, und klicken Sie auf die Schaltfläche Bilddaten anzeigen, um die nach der Segmentierung erzeugten binären Bildmasken (DNA-Maske und NET-Maske) zu visualisieren und zu validieren.
- Wählen Sie die Segmentierungsmethode aus, indem Sie auf das Untermenü Methode sowohl im DNA-Kanal als auch im NET-Kanal klicken.
- Einzelzellige Analyse identifizierbarer Eigenschaften
- Geben Sie die Registerkarte Analyse ein, und analysieren Sie die Steuerelementbeispiele, indem Sie auf die Schaltfläche Schwellenwert bestimmen klicken.
- Ändern Sie den Beispieltyp in PMA, und klicken Sie auf die Schaltfläche Zelleneigenschaften abrufen, um die Analyse der stimulierten Proben abzuschließen.
- Wählen Sie ein Bild aus dem Untermenü "Beispieltyp" aus, und klicken Sie auf die Schaltfläche Bilddaten anzeigen, um die Überlagerung und die Anzahl der Zellen und NET-bildenden Zellen im Bild anzuzeigen.
- Vergleich der Zelleigenschaften zur Identifizierung netbildender Zellen
- Wählen Sie das Beispiel aus dem Untermenü des Beispieltyps aus, und klicken Sie auf die Schaltfläche NETs analysieren, um die Analyse abzuschließen. Einzelne Bilder können aus dem Untermenü des Beispieltyps für die Analyse ausgewählt werden oder der gesamte Bildstapel kann durch Auswahl der Batch-Option (empfohlen) analysiert werden.
- Passen Sie die NET-Kriterien manuell an, um optimale Ergebnisse für eine bestimmte Stichprobe zu erzielen. Vergleichen Sie identifizierte NETs mit den Originalbildern, um die Qualität der Identifizierung zu bewerten.
Hinweis: Die NET-Kriterien können dann für alle Bilder im Datensatz verwendet werden. Alle Änderungen an den NET-Kriterien werden gleichzeitig auf alle Steuerelementstichproben angewendet. Dadurch wird die Möglichkeit möglicher Unterschiede begrenzt, die durch eine Überanpassung der NET-Parameter entstehen können. Die Einstellungen in den NET-Kriterien können entsprechend den Anforderungen des Benutzers angepasst werden. Die Beziehung zwischen falschen Ermittlungsraten und NETQUANT wurde zuvoruntersucht 13. Typische Bereiche für flächenerhöhen sind 2-u20124, Zirkularität auf 0,7-u20120,9 und das DNA/NET-Verhältnis 0,6–2,0. - Überprüfen Sie die Datenzusammenfassung im Untermenü "Zellendaten", in dem die Anzahl der Bilder, die Zellenanzahl pro Bild und der Prozentsatz der NETs pro Bild angezeigt werden.
Hinweis: Der Gesamtprozentsatz der NETs im gesamten Dataset wird durch das "NET-Gauge" angezeigt. Die Gesamtbildanzahl, die Zellanzahl, der Prozentsatz der NETs in der Stichprobe (NETs%) und NETs% in der Kontrollstichprobe werden in der Übersichtsstatistiktabelle unter dem NET-Spur angezeigt. Wir empfehlen, die Kontrolldaten zusammen mit den Daten aus stimulierten Proben zu melden.
- Ergebnis-Ausgaben
- Geben Sie die Registerkarte Ausgabe ein, um Ergebnisausgaben auszuwählen und anzuzeigen.
- Erkunden und vergleichen Sie die verschiedenen Datenausgänge, die aus der Analyse von Steuerung und PMA generiert wurden, indem Sie die Form der Ausgabe auswählen und auf die Schaltfläche Ausgabeergebnisse klicken.
Hinweis: Alle nach der Analyse generierten Daten für Steuerelemente und Stimulationen werden im Analyseordner gespeichert, wie im Zieluntermenü ausgewählt. Die Daten werden entweder im Format .csv oder .pdf gespeichert. - Starten Sie die Methodendatei, um die Für die Analyse verwendete Version der Software und NET-Kriterien zu erhalten (die für Veröffentlichungszwecke in den Abschnitt "Methoden" aufgenommen werden sollen).
- Klicken Sie auf die Tabelle Ergebnisse, um die einzelnen Datenpunkte in einer bestimmten Stichprobe zu visualisieren.
- Visualisieren Sie die NET-Flächenverteilung und das DNA:NET-Verhältnis in den Stichproben. Die rote Linie gibt den Schwellenwert in den Diagrammen an.
- Bestimmen Sie den NET-Bereich im Vergleich zur Form der DNA, indem Sie auf die Bivariate-Verteilungsdatei klicken.
- Vorherige Analyse und Batch alle Schritte laden
- Laden Sie zuvor erfolgreiche Analyseeinstellungen mit der Schaltfläche Vorherige Analyse laden in NETQUANT.
- Verwenden Sie die Im Setup-Menü enthaltene Schaltfläche "Alle Schritte" von Batch, um die Schritte 5-u201212 (Abbildung 1, Abbildung 2, Abbildung 3, Abbildung 4, Abbildung 5) direkt auszuführen, um die endgültige Ausgabe zu erhalten.
Ergebnisse
5 x 105 Neutrophile/ml wurden auf Decklippen gesät, die in einer 12-Well-Platte platziert und entweder mit 20 nM PMA stimuliert oder 150 min unstimuliert blieben. Die Proben wurden dann mit Primärkaninchen anti-human etrophil Elastase Antikörper, sekundäre Ziege Anti-Kaninchen Fluorophor konjugierte Antikörper und DAPI - ein fluoreszierend gekennzeichneter Farbstoff, der DNA färbt gefärbt (Siehe Tabelle der Materialien für Details). Mindestens 5 Bilder ...
Diskussion
NET-Bildung ist eine relativ neue Ergänzung zu den verschiedenen neutrophilen Armamentarium4 und es gab eine spürbare Zunahme des Interesses, die Implikation von NETs in einer breiten Palette von Forschungsbereichen5,7,14,15zu studieren. Die Erfassung von Bildern mittels Immunfluoreszenzmikroskopie und anschließender bildbasierter Quantifizierung ist eine weit verbrei...
Offenlegungen
TM und PN haben ein Patent angemeldet, das sich auf die in NETQUANT verwendeten Algorithmen bezieht.
Danksagungen
Finanziert wurde die Arbeit von der Crafoord Foundation (TM und PN), dem Schwedischen Regierungsforschungsstipendium (PN, TM), dem Schwedischen Forschungsrat (PN) und der Groschinsky Foundation (TM, PN).
Materialien
| Name | Company | Catalog Number | Comments |
| BD Vacutainer Heparinised plastic tubes | BD Biosciences | 367885 | |
| Lymphoprep | Axis-Shield | 114547 | |
| RPMI-1640 with L-Glutamine | Gibco | 11835-030 | |
| 50mL conical flasks | Sarstedt | 62.547.004 | |
| 15mL conical flasks | Sarstedt | 62.554.002 | |
| 12-well Tissue culture plates | Falcon | 10626491 | |
| #1 Coverslips 10mm | Menzel Glaser | CS10100 | |
| Glass slides | Menzel Glaser | 631-0098 | |
| Primary anti-human elastase | DAKO | DAKO rabbit 1373, contract immunization | |
| Secondary fluorophore conjugated goat anti-rabbit | Life technologies | A-11072, A-11070 | |
| PROLONG-Gold Antifade reagent with DAPI | Life technologies | P36930 | Mounting medium |
| Goat serum | Sigma-Aldrich | G9023 | |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma-Aldrich | 79346 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Nikon Ti-E Epifluorescence microscope | Nikon | ||
| CCD camera | Andor Zyla | ||
| Plan Apochromat 20x, 40x objectives | Nikon | ||
| Windows 10 | Microsoft | Operating system | |
| macOS Sierra 10.12 | Apple | Operating system | |
| MATLAB | Mathworks |
Referenzen
- Nauseef, W. M., Borregaard, N. Neutrophils at work. Nature Immunology. 15 (7), 602-611 (2014).
- Borregaard, N., Cowland, J. B. Granules of the human neutrophilic polymorphonuclear leukocyte. Blood. 89 (10), 3503-3521 (1997).
- Nordenfelt, P., Tapper, H. Phagosome dynamics during phagocytosis by neutrophils. Journal of Leukocyte Biology. 90 (2), 271-284 (2011).
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Sorensen, O. E., Borregaard, N. Neutrophil extracellular traps - the dark side of neutrophils. Journal of Clinical Investigation. 126 (5), 1612-1620 (2016).
- Yipp, B. G., Kubes, P. NETosis: how vital is it?. Blood. 122 (16), 2784-2794 (2013).
- Jorch, S. K., Kubes, P. An emerging role for neutrophil extracellular traps in noninfectious disease. Nature Medicine. 23 (3), 279-287 (2017).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. J Cell Biol. 176 (2), 231-241 (2007).
- Mohanty, T., et al. A novel mechanism for NETosis provides antimicrobial defense at the oral mucosa. Blood. 126 (18), 2128-2137 (2015).
- Hakkim, A., et al. Activation of the Raf-MEK-ERK pathway is required for neutrophil extracellular trap formation. Nature Chemical Biology. 7 (2), 75-77 (2011).
- Brinkmann, V., Goosmann, C., Kuhn, L. I., Zychlinsky, A. Automatic quantification of in vitro NET formation. Frontiers in Immunology. 3, 413 (2012).
- Coelho, L. P., et al. Automatic determination of NET (neutrophil extracellular traps) coverage in fluorescent microscopy images. Bioinformatics. 31 (14), 2364-2370 (2015).
- Mohanty, T., Sorensen, O. E., Nordenfelt, P. NETQUANT: Automated Quantification of Neutrophil Extracellular Traps. Frontiers in Immunology. 8, 1999 (2017).
- Kaplan, M. J., Radic, M. Neutrophil extracellular traps: double-edged swords of innate immunity. Journal of Immunology. 189 (6), 2689-2695 (2012).
- Cedervall, J., Olsson, A. K. Immunity Gone Astray - NETs in Cancer. Trends in Cancer. 2 (11), 633-634 (2016).
- Ginley, B. G., et al. Computational detection and quantification of human and mouse neutrophil extracellular traps in flow cytometry and confocal microscopy. Scientific Reports. 7 (1), 17755 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten