Method Article
Ein dreidimensionales thymisches Kultursystem zur Generierung von murine induzierten pluripotenten Stammzell-abgeleiteten Tumor-Antigen-spezifischen thymischen Auswanderern
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt eine neuartige Methode zur Erzeugung von Tumorantigen-spezifischen induzierten pluripotenten Stammzellen-abgeleiteten thymischen Auswanderern (iTE) durch ein dreidimensionales (3D) thymische Kultursystem. iTE sind eine homogene Teilmenge von T-Zellen, die eng mit naiven T-Zellen verwandt sind und die Fähigkeit zur Proliferation, Gedächtnisbildung und Tumorunterdrückung haben.
Zusammenfassung
Die Vererbung von vorneu angeordneten T-Zellrezeptoren (TCRs) und ihre epigenetische Verjüngung machen induzierte pluripotente Stammzellen (iPSC)-abgeleitete T-Zellen zu einer vielversprechenden Quelle für die Adoptiv-T-Zelltherapie (ACT). Klassische In-vitro-Methoden zur Herstellung regenerierter T-Zellen aus iPSC führen jedoch zu entweder angeborenen oder endlos differenzierten T-Zellen, die phänotypisch und funktionell von naiven T-Zellen unterschieden werden. Kürzlich wurde ein neuartiges dreidimensionales (3D) thymmisches Kultursystem entwickelt, um eine homogene Teilmenge von CD8- + antigenspezifischen T-Zellen mit einem naiven T-zellähnlichen funktionellen Phänotyp zu erzeugen, einschließlich der Fähigkeit zur Proliferation, Gedächtnisbildung und Tumorunterdrückung in vivo. Dieses Protokoll vermeidet abnorme Entwicklungsschicksale, wodurch die Erzeugung klinisch relevanter iPSC-abgeleiteter T-Zellen ermöglicht wird, die als iPSC-abgeleitete thymische Auswanderer (iTE) bezeichnet werden, und gleichzeitig ein wirksames Werkzeug zur Aufklärung der nachfolgenden Funktionen bietet, die für T-Zellreifung nach thymischer Selektion.
Einleitung
Die Adoptiv-T-Zelltherapie (ACT) kann eine wirksame Behandlung für einige Patienten mit fortgeschrittenem Krebs sein. Leider erleben viele Patienten keine Tumorregression, und übertragene Zellen bleiben nach der Infusion nicht bestehen. Dies kann auf die Qualität der infundierten T-Zellen zurückzuführen sein. Ein ACT-Mausmodell zeigte, dass im Vergleich zu naiven oder weniger differenzierten zentralen Gedächtnis-T-Zellen endlos differenzierte Effektorzellen aufgrund schlechter In-vivo-Persistenz1weniger potent sind, eine Beobachtung, die auch durch klinische Daten unterstützt wird2, 3.
Um die Wirksamkeit der aktuellen ACT zu verbessern, wurden T-Zell-abgeleitete induzierte pluripotente Stammzellen (T-iPSC) ausgiebig untersucht4,5. Wenn T-Zellen in T-iPSC umprogrammiert und in T-Zellen neu differenziert werden, wird die neu angeordnete Konfiguration von TCR-Genen von T-iPSC und anschließend den neu differenzierten T-Zellen vererbt. Daher ermöglicht die Fähigkeit von T-iPSC, eine unbegrenzte In-vitro-Erweiterung zu durchlaufen, die effiziente Reproduktion von unreifen T-Zellen, die die Neoantigen-spezifischen T-Zellrezeptoren (TCR) tragen, wenn solche Zellen aus Tumorantigen-spezifischen T-Zellen entwickelt werden6 ,7. Die genaue Methode zur Differenzierung von T-iPSC in reife T-Zellen, die die Produktion von krebsantigenspezifischen T-Zellen mit einem weniger differenzierten Phänotyp und einer besseren Antitumor-Potenz ermöglichen würde, muss jedoch noch erläutert werden.
Die T-iPSC-Differenzierung unter Verwendung der Kokultur von OP9-Murin-Stromalzellen, die den menschlichen Kerbligand DLL1 überexzättigen, ist eine etablierte Methode zur Herstellung von T-Zellen in vitro6,7. Bei Mäusen und Menschen kann dieses Kokultursystem iPSC konsequent unterscheiden und so Entwicklungsereignisse vom Blastozystenstadium bis zur unreifen T-Zelllinie6,7rekapitulieren. Trotz dieser biotechnologischen Fortschritte ist die physiologische Differenzierung nach der CD4+CD8+ Double Positive (DP) Phase immer noch schwer zu erreichen. Einer der Gründe ist, dass in vivo CD4+CD8- und CD4-CD8+ einzelne positive (SP) T-Zellen im Thymus erzeugt werden, einem Organ, das für die Reifung und Auswahl von T-Zellen verantwortlich ist, die eine fremde Antigenspezifität haben, nicht auto-reactivity8. Diese selektiven Prozesse werden als positive bzw. negative Selektion definiert. Die meisten molekularen Mechanismen, die für die Reifung von T-Zellen im Thymus erforderlich sind, sind jedoch noch nicht vollständig verstanden, was es schwierig macht, diesen Prozess in vitro zu rekonstruieren. Um diese physiologische Hürde zu überwinden, haben mehrere Gruppen den TCR-Komplex mit Anti-CD3-Antikörpern oder agonistischen Peptiden stimuliert. Diese In-vitro-Techniken erzeugen Zellprodukte, die wichtige T-Zellmarker wie CD3, CD8, TCR und CD62L exprimieren und dennoch die Tumorantigen-Spezifität beibehalten. Leider stellen T-Zellen, die durch diese extrathymischen Methoden erzeugt werden, eine breite heterogene Population von Zellen dar, die durch eine unvollständige positive Selektion, angeborene Merkmale, TCR-unspezifische Tötung, Unfähigkeit zur Gedächtnisbildung und nicht-persistente Anti-Tumor-Effekte in vivo8,9,10,11. Diese Anomalien haben Bedenken geweckt, dass solche Zellen eine Vielzahl von Nebenwirkungen auslösen könnten, einschließlich Lymphom und sowohl Haut- und Knochenanomalien, wenn sie für therapeutische Anwendungen verwendet werden12,13,14 .
Um die physiologischen Signale nachzubilden, die in aktuellen In-vitro-Differenzierungssystemen fehlten, wurden tumorantigenspezifisches T-iPSC mit einem geernteten Thymus unterschieden. Das klassische fetale Thymus-Organkultursystem (FTOC), das entwickelt wurde, um die intrathymische Entwicklung von T-Zellen zu untersuchen, wurde durch die Verwendung eines 3D-Kultursystems verbessert, das erfolgreich T-Zellen produzierte, die die thymische Ausbildung abschlossen. Diese postthymischen T-Zellen, die als iPSC-abgeleitete thymische Auswanderer (iTE) bezeichnet wurden, wiesen naive Eigenschaftenauf 15. iTE zeigte Proliferation, Gedächtnisbildung und angemessene Anti-Tumor-Effekte in einem Mausmodell gegen etablierte B16-Melanom-Tumoren. Dieser Artikel beschreibt ausführlich das Protokoll dieses neuartigen FTOC-Systems mit einem 3D-Kultursystem (Abbildung 1).
Protokoll
Alle Tierversuche wurden von den Institutional Animal Care and Use Committees des National Cancer Institute (NCI) genehmigt und in Übereinstimmung mit den NIH-Richtlinien durchgeführt.
1. Vorbereitung von OP9/DLL1-Zellen für die Co-Kultur mit iPSC
- Kultur OP9/DLL1-Zellen in OP9-Medien (-Mindestmittel medium ['-MEM]+ 20% nicht-hitzeaktiviertes fetales Rinderserum [FBS] + 1x Penicillin-Streptomycin + Ascorbinsäure [50 ng/mL] und Monothioglycerol [100 nM]) bei 37 °C. Wenn OP9/DLL1-Zellen 80–95% Koninfluenza erreichen, einmal mit 1x Magnesium, Calcium und phenolrot freiphosphatgepufferter Saline waschen (nachfolgend PBS genannt).
- 4 ml 0,05% Trypsin hinzufügen und 5 min bei 37 °C brüten. Fügen Sie dann 4 ml OP9-Medien hinzu, indem Sie die Zellschicht durch Pipettieren trennen, um eine einzelne Zellsuspension zu bilden.
- Übertragen Sie die Zellsuspension durch ein 100 m Zellsieb in ein 50 ml konisches Rohr. Zentrifuge bei 300 x g für 5 min bei 4 °C, den Überstand ansaugen und in 12 ml OP9-Medien wieder aussetzen.
- Platte 2 ml OP9/DLL1 Zellsuspension auf eine neue 10 cm Zellkultur Petrischale und fügen Sie zusätzliche 8 ml OP9-Medien hinzu. Wiederholen Sie den Durchgang alle 2–3 Tage.
HINWEIS: Die Qualität der FBS- und Kulturbedingungen ist entscheidend, um die Erweiterung von OP9/DLL1-Zellen aufrechtzuerhalten, ohne ihre Fähigkeit zur Unterstützung der iPSC-Differenzierung zu verlieren. Daher wird empfohlen, die Menge von FBS und Durchgang konsequent bei 80% Koninfluenza vorzubewerten, um Zelldifferenzierung und Seneszenz zu verhindern. Es ist auch wichtig, genügend gefrorene Bestände an OP9/DLL1-Zellen zu machen und alle 4–6 Wochen einen neuen Bestand aufzutauen.
2. In Vitro Differenzierung von iPSC in unreife T-Zellen
-
Beginnen Sie am Tag 0 mit der iPSC-Kokultur auf OP9/DLL1-Konfluent-Gerichten.
- Ernte iPSC als Einzelzellsuspension durch Trypsinisierung (5 min in 0,05% Trypsin bei 37 °C), die Zellen sammeln und bei 300 x g bei 300 x g für 5 min bei 4 °C zentrifugieren.
- Aspirieren Sie den Überstand und setzen Sie Zellen bei 1,0 x 105 iPSC pro 10 ml OP9-Medien aus. Platte 1,0 x 105 iPSC auf eine konfluente OP9/DLL1 10 cm Schale.
HINWEIS: OP9/DLL1 10 cm Geschirr werden zur iPSC-Differenzierung verwendet, wenn sie 90–100% Konfluenz erreichen. Unterschiede in der Konfluenz können die Effizienz der iPSC-Differenzierung beeinflussen.
- An Tag 3, aspirieren alte Medien und ersetzen sie durch 10 ml frische OP9-Medien.
-
Am 6. Tag, Durchgangszellen.
- Waschen Sie jede 10 cm konfluente OP9 Schale mit 10 ml PBS. Fügen Sie 3 ml 0,05% Trypsin pro Gericht hinzu und brüten für 3–5 min bei Raumtemperatur (RT).
- Fügen Sie 4 ml OP9-Medien hinzu und sammeln Sie Zellen durch sanftepipettierende. Übergeben Sie Zellen durch ein 100-m-Zellsieb und eine Zentrifuge bei 300 x g für 5 min bei 4 °C. Entsorgen Sie Überstand.
- Resuspend Zellen in 10 ml Differenzierungsmedien (OP9-Medien mit 5 ng/ml-Maus Flt3 Ligand [FLT3L] und 5 ng/ml-Maus IL-7) und Plattenzellaufhängung auf einer neuen 10 cm OP9/DLL1-Konfluentschale.
- An Tag 9, aspirieren alte Medien und ersetzen mit 10 ml frische Differenzierung Medien.
-
Am 11. Tag, an dem Kardiomyozyten in iPSC-Kolonien beobachtet werden, lösen Sie nicht haftende Zellen mechanisch durch Pipettieren und filtern Sie durch ein 100 m großes Zellsieb. Drehen Bei 300 x g für 5 min bei 4 °C.
- Aspirieren Sie den Überstand und resuspendieren Sie in 24 ml Differenzierungsmedien. Platte iPSC in eine konfluente OP9/DLL1 6-Well-Platte (4 ml/well).
-
Sammeln Sie am 15. Tag alle nicht haftenden Zellen und filtern Sie sie durch ein 40-m-Zellsieb.
- Drehen Bei 300 x g für 5 min bei 4 °C.
- Fahren Sie alle 3–4 Tage mit der Passierung nicht anhaftender Zellen fort, indem Sie Schritt 2.5.1 wiederholen.
3. 3D-Thymische Orgelkultur zur Generierung von iTE
- Ernte Maus fetale thymische Lappen und Einsatz von endogenen Lymphozyten durch Deoxyguanosin (dGUO) Behandlung wie zuvor beschrieben16.
- Nehmen Sie am 7. Tag der dGUO-Behandlung vier neue 10 cm-Gerichte und füllen Sie jeweils 20 ml komplette Medien (Rosswell Park Memorial Institute Media 1640 [RPMI 1640] + 10% FBS + 1x L-Alanyl-L-Glutamin + 1x Natriumpyruvat + 1x minimales essentielles Medium mit nicht-essentiellen Aminosäuren (MEM-NEAA) + 1x Penicillin-Streptomycin + [1:1000] 2-Mercapto Ethanol).
- Alle Nitrozellulosemembranen mit Thymianlappen in eine 10 cm große Schale geben. Lösen Sie die einzelnen Lappen mit Zangen von der Membran, so dass sie in Medien getaucht werden können. Entsorgen Sie die Membranen. 1 h bei RT inkubieren.
- Die Thymianlappen in eine neue 10 cm Schale mit komplettem Medium geben und 1 H bei RT inbrünstigen. Wiederholen Sie diesen Schritt noch 2 Mal.
- Mit Zangen, fixieren Sie die thymischen Lappen auf die Schale (eine nach der anderen), und mit der anderen Hand machen Sie einen 100-200 m tiefen Schnitt in der Mitte und verlängern den halben Durchmesser des Lappens, um T-Zell-Vorläufer Migration in den Lappen zu erleichtern.
- Übertragen Sie die thymischen Lappen auf eine neue 10 cm Schale, gefüllt mit kompletten Differenzierungsmedien (komplette Medien + 5 ng/mL Maus IL-7 + 5 ng/mL Maus FLT3L + 5 ng/mL SCF).
- Optional, wenn 3D-Kulturplatten mit unteren und oberen Ebenen Gitter verwenden, füllen Sie beide Gitter mit sterilen PBS, um die Verdunstung und Trocknung der hängenden Tropfen zu verhindern.
- Übertragen Sie 30 l vollständiger Medien, die einen dGuo-behandelten Thymsamen aus Schritt 3,6 enthalten, in jeden Brunnen der 3D-Kulturplatte.
- Sammeln Sie nicht-haftende T-Linienzellen (iPSC-abgeleitete unreife T-Zellen) aus der OP9/DLL1-Kokultur (Tage 16-21) (Schritt 2.6.2) und suspendieren Sie bei 2–5 x 103 T-Linienzellen pro 20 L-Medium.
- Fügen Sie jedem Thymianlappen in der 3D-Kulturplatte 20 L T-Linienzellsuspension hinzu. Über Nacht bei 37 °C mit 5% CO2 inkubieren.
- Stellen Sie die P200-Pipetten auf 30 L und aspirieren Sie das Medium nach dem Pipettieren mehrmals aus jedem Brunnen, um alle Zellen zu entfernen, die die Thymianlappen umgeben. Entsorgen Sie die Medien, und fügen Sie 30 L vollständige Medien hinzu. Wiederholen Sie diesen Vorgang 5–7 Mal, um alle extra unreifen T-Zellen zu entfernen, die nicht in die Lappen migrieren. Danach täglich 25–30 L Medien wechseln.
- Bestätigen Sie die Bildung eines Halos von iPSC-abgeleiteten thymischen Auswanderern (iTE) um die Lappen, beginnend am Tag 4 bis 5 durch Lichtmikroskopie.
- Sammeln Sie iTE täglich, indem Sie Medien ohne Lappenunterbrechung pfeifen. Wechseln Sie die Medien jeden Tag und fahren Sie mit der Sammlung bis zu ca. 12 Tagen fort.
- Geerntete iTE sind einsatzbereit für molekulare Analysen (Abbildung2, Abbildung 3, Abbildung 4und Abbildung 5) oder In-vivo-Transplantationsexperimente.
4. Herstellung von Antigen-Präsentationszellen (APC)
- Opfern Sie eine C57BL/6 Maus durch Zervixdislokation und legen Sie sie wie oben beschrieben auf eine Lab-Soaker-Matte.
- Entfernen Sie die Milz und legen Sie sie auf ein 100 m Zellsieb. Komprimieren Sie die Milz auf das Sieb mit einem 12 ml Spritzenkolben, um eine Einzelzellsuspension herzustellen.
- Übertragen Sie die Zellsuspension durch ein steriles 40-m-Zellsieb. Zentrifugieren Sie die Suspension bei 300 x g für 5 min bei 4 °C, um die Zellen zu pellet.
- Aspirieren Sie den Überstand und setzen Sie das Zellpellet in 2 ml Ammoniumchlorid-Kalium (ACK) Lysepuffer wieder aus, um rote Blutkörperchen (RBC) auszuschließen. 5 min bei RT inkubieren.
- Löschen Sie den ACK-Lysepuffer, indem Sie 10 ml PBS hinzufügen. Pellet die Zellen durch Zentrifugation bei 300 x g für 5 min bei 4 °C.
- Den Überstand ansaugen und das Zellpellet in 10 ml Komplettmedien wieder aufhängen und auf eine 10 cm sterile Petrischale übertragen.
- Bestrahlen Sie Splenozyten mit 3500 rad mit hilfe einer Bestrahlungsvorrichtung (Strahlung), um die Zellproliferation zu verhindern.
- Die bestrahlten Zellen sofort über Nacht in einen 37 °C-Inkubator und eine Kultur zurückbringen.
- Verwenden Sie bestrahlte Zellen als APC oder einfrieren Sie in Zellbanker.
5. Pulsierendes APC mit Antigen
- Zählen Sie live bestrahlte APC mit einem Neubauer Hämozytometer und Trypan blau Farbstoff. APC mit Peptiden (hgp100) oder Nukleoprotein 30 min bei 37 °C inkubieren.
- Waschen Sie APC zweimal mit 10 ml PBS, um zusätzliches Peptid zu entfernen.
- Zählen Sie iTE und mischen Sie mit APC in einem 1:1-Verhältnis in kompletten Medien mit 100 I.E. IL-2 und 5 ng/ml IL-7. Aliquot 100 l des Zellgemischs (Gesamtkonzentration: 1 x 106 Zellen/ml) in jeden Brunnen einer ultraniedrigen Befestigung U boden 96 Wellplatte und Kultur für 48 h bei 37 °C.
- Nach 48 h, übertragen Zellen auf eine neue Platte mit einer Mehrkanalpipette und Durchgang alle 2 bis 3 Tage danach.
-
Analysieren Sie am 3. Tag das Cytokin-Sekretionsprofil, indem Sie die Zellen mit intrazellulärem Antikörper beflecken und durch Durchflusszytometrie analysieren (Abbildung 3).
- Fügen Sie 0,67 L/ml Proteintransporthemmer (z. B. GolgiStop) hinzu und brüten Sie bei 37 °C für 6 h, um die intrazelluläre Akkumulation von Zytokinen zu verbessern. Mit 10 ml PBS waschen.
- Zellen in 3 ml kaltem (4 °C) PBS aussetzen und langsam 1 ml kalte 4%-Paraformaldehyd(PFA)-Lösung hinzufügen.
- Nach 10 min Zellen bei 300 x g für 5 min bei 4 °C abwerfen, Überstand entsorgen und mit 10 ml PBS waschen.
- Zellen in 1 ml PBS + 1% FBS + 0,1% nichtionisches Tensid wieder aufsetzen und 10–15 min in 4 °C platzieren.
- Antikörper hinzufügen, Proben vor Licht schützen und 30 min in 4 °C stellen.
- Zellen bei 300 x g für 5 min bei 4 °C abdrehen, Überstand entsorgen und mit 10 ml PBS waschen.
- Zellen bei 300 x g für 5 min bei 4 °C abdrehen und Zellen in 1 ml PBS wieder aufsetzen. Die Zellen können in einem Durchflusszytometer analysiert werden.
Ergebnisse
Co-kultivierte fetale Thymuse wurden geschnitten, um zu analysieren, ob iPSC-abgeleitete T-Linienzellen in die thymischen Lappen wandern können. Unsättliche Kontrolllappen hatten eine Gewebearchitektur, die sich durch ein astrozytartiges thymisches Epithelweb17aus endogenen CD3+ Zellen auszeichnete. Auf der anderen Seite wurden thymische Lappen, die mit iPSC-abgeleiteten unreifen T-Zellen gesät wurden, mit CD3+ mononukleären Zellen wieder aufgefüllt, was auf die Migration von iPSC-abgeleiteten unreifen T-Zellen in die Lappen hindeutet (Abbildung 2A).
T-Zellen, die in die thymische Mikroumgebung migrierten und reiften, wanderten anschließend als iTE aus. Um ihre phänotypische Charakterisierung zu testen, wurde eine zytometrische Durchflussanalyse von C57BL6-Thymoseten, Pmel iPSC-abgeleiteten unreifen T-Zellen (extrathymisch) und Zellen durchgeführt, die aus thymischen Lappen (iTE) austraten. Extrathymische T-Zellen auf OP9/DLL1 zeigten CD4+CD8+ (DP) T-Zellen und CD8-SP-T-Zellen ohne Expression des positiven Selektionsmarkers MHC-I, während iTE eine klare Population des CD8-SP-MHC-I+ T-Zellphänotyps hatte, was ihre erfolgreichen Durchgang durch positive Selektion vor dem Austreten aus den thymischen Lappen. iTE beständig ausdrücken MHC-I und CD62L, die Marker mit hoher proliferativer Kompetenz, Zytokinproduktion, peripherem Überleben und lymphoidem Homing18,19,20assoziiert sind. Dieser Phänotyp stimmt mit M2 SP-Thymozyten überein, die die ausgereifteste Population einzelner positiver T-Zellen im Thymus20sind, was darauf hindeutet, dass iTE durch ein normales thymisches Entwicklungsprogramm übergegangen ist (Abbildung 3). Um die Effizienz der iTE-Generierung zu überwachen, wurden Zellen isoliert, die aus einzelnen thymischen Lappen ausgestiegen waren. An Tag 7 erzeugten thymische Lappen durchschnittlich 1 x 103 Live-CD8SP CD45.1+ CD3+ iTE pro Tag (Abbildung 3B). Eine ähnliche Rate der iTE-Produktion wird vom 6. bis zum Tag 12 der 3D-Thymischen Co-Kultur beobachtet.
Antigenabhängige Aktivierung und Sekretion von Zytokinen wurden analysiert, um die funktionellen Eigenschaften von thymisch gebildeten iPSC-abgeleiteten unreifen T-Zellen zu beobachten. In Gegenwart eines irrelevanten Peptids (Nukleoprotein) gab Pmel-iTE keine signifikanten Mengen an TNF-, IL-2 oder IFN-A frei. Bei stimulierter Stimulation mit dem Cognate-Peptid für Pmel-T-Zellen (hgp100) gab Pmel-iTE robuste Mengen an TNF-A und IL-2 frei, während gleichzeitig geringe Mengen an IFN-A (Abbildung4)produziert wurden, was darauf hindeutet, dass thymisch gebildetes iTE ihr kogatiertes Peptid und Sezerwirkungseffekte mit einem Profil, das dem von natürlichen thymischen Auswanderern (RTE) ähnelt.
Um die Transkriptionsunterschiede zwischen iPSC-abgeleiteten T-Linienzellen zu untersuchen, die auf OP9/DLL1 mit oder ohne thymische Bildung differenziert wurden (d. h. iTE versus extrathymische T-Zellen), wurde eine RNA-Seq-Analyse an diesen beiden Populationen durchgeführt und ZU DP T-Abstammungszellen, die mit OP9/DLL1 (DP) und primären naiven CD8+ Pmel T-Zellen differenziert sind. Die Expression von 102 Genen, die eine entscheidende Rolle in T-Zell-Ontogenie, Thymose-Aktivierung und Gedächtnisbildung spielen, wurdenanalysiert 15,20,21,22. Eine Hauptkomponentenanalyse dieser vier untersuchten Populationen zeigte, dass extrathymisch erzeugte DP- und CD8SP-T-Zellen zusammengruppiert wurden, während iTE sich näher an naiven T-Zellen gruppierte (Abbildung 5). Zusammen zeigen diese Daten, dass iTE einen Phänotyp hat, der näher an naiven T-Zellen liegt als T-Linienzellen, die durch extrathymische Methoden erzeugt werden.
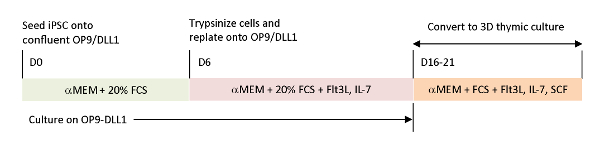
Abbildung 1 : Schematische Übersicht über die Differenzierung von iPSC zu iTE mit OP9/DLL1 und 3D-Thymkultur. Das Protokoll umfasst drei separate Differenzierungsschritte; (Links) von iPSC-Zellen zu hämatopoetischen Abstammungszellen auf OP9/DLL1 (Tag 0 bis 6), (Mitte) von hämatopoetischen Abstammungszellen bis zu unreifen T-Zellen auf OP9/DLL1 mit Zytokinen (Tag 6 bis 16–21) und (Rechts) von unreifen T-Zellen (Tag 16–21) bis iTE mit einem 3D-Thymkultursystem. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
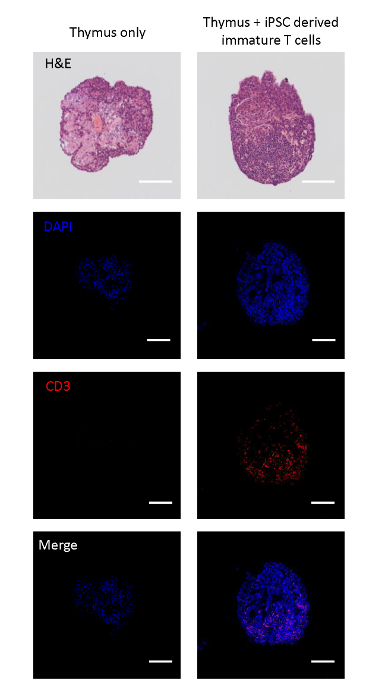
Abbildung 2 : Immunhistochemie von Thymianlappen, die mit iPSC-abgeleiteten unreifen T-Zellen gesät wurden. Oben: H&E-Färbung eines Thymianlappens mit und ohne Aussaat von iPSC-abgeleiteten unreifen T-Zellen. Von der zweiten Oben nach unten: konfokale Bilder der mit DAPI (Kern), CD3 (T-Zelle) befleckten Schnittlappen und Zusammenführen. Skalenbalken = 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
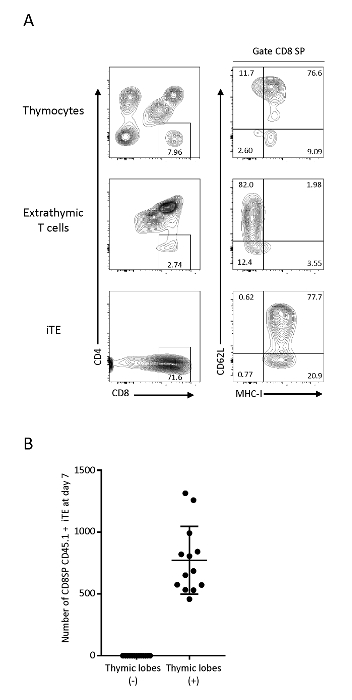
Abbildung 3 : iTE zeigen einen postthymischen T-Zell-Phänotyp. (A) FACS-Analysen von Thymosyten, extrathymischen T-Zellen (OP9/DLL1-Kokultursystem) und Pmel-iTE. Lebende Zellen wurden auf kongenen CD45+abgesperrt. CD8 SP-Populationen wurden weiter auf CD62L- und MHC-I-Expression analysiert. (B) Durchschnittliche Anzahl der CD8SP CD45.1 iTE, die über Nacht pro Lappen produziert wird, 7 Tage nach der Vorsaat. Die Daten wurden aus 12 unabhängigen Experimenten gesammelt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
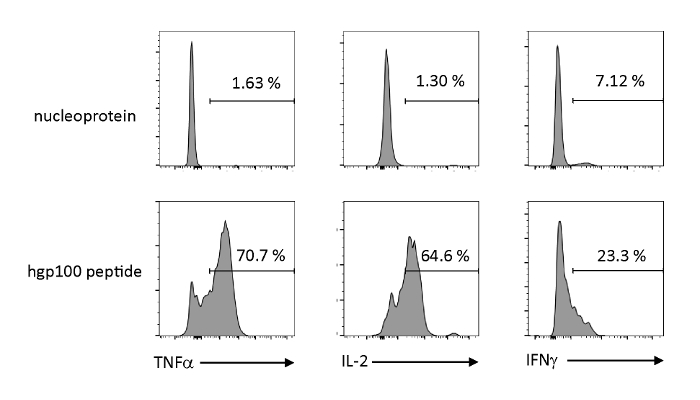
Abbildung 4 : iTE produzieren verschiedene Zytokine durch antigenspezifische Stimulation. FACS-Analysen der intrazellulären Produktion von Zytokinen durch iTE. iTE wurden mit APCs ko-kultiviert, die drei Tage lang mit irrelevantem (Nukleoprotein) oder Cognate (hgp100) Peptid vorinstalliert waren. Die zahlen in den oberen rechten Quadranten geben die Prozentsätze von iTE an, die Zytokin produzieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
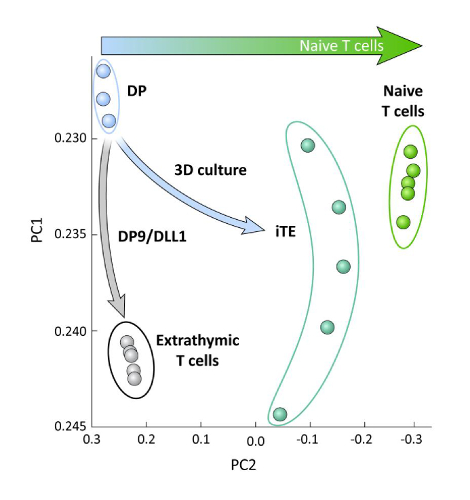
Abbildung 5 : Die Whole-Transkriptom-Analyse zeigt eine Verschiebung der iTE-Genexpression hin zu einer naiven CD8 + T-Zell-Programm. Prinzipiellkomponentenanalyse (PCA) von RNA-seq-Daten aus DP-, extrathymischen CD8-SP-, iTE- und naiven T-Zellen. (Analyse von 102 Genen im Zusammenhang mit thymischer Differenzierung unter Verwendung der öffentlichen Datenbank GSE105110) 15. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Verwendung von T-iPSC zur Regeneration von Tumorantigen-spezifischen T-Zellen kann viele der aktuellen Hindernisse von ACT überwinden, indem junge Zellen mit verbesserter Persistenz erzeugt werden. Obwohl mehrere Methoden mit dem OP9/DLL1-Kokultursystem berichtet wurden, um CD8 SP-Zellen6,7,10,13 zu erzeugen, die CD8-Moleküle und Tumor-Antigen-spezifische TCRs, globale Gen Expressionsmuster und funktionelle Analysen zeigen, dass sich diese extrathymisch regenerierten CD8-SP-Zellen von naiven T-Zellen unterscheiden (Abbildung 4). Hier beschreiben wir ein 3D-Thymkultursystem, das iPSC-abgeleitete thymische Auswanderer (iTE) mit hoher Genauigkeit und Homogenität aus murinem T-iPSC erzeugen kann. iTE ähneln naiven T-Zellen im globalen Genexpressionsmuster und in der Funktionalität, wie Gedächtnisbildung und in vivo Anti-Tumor-Effekt gegen etablierten Tumor15.
Das klassische FTOC-System ist eine Möglichkeit, die thymische Selektion in vitrozu rekapitulieren. Es wurde für die Untersuchung der intrathymischen Entwicklung von Thymosyten23verwendet, und es gibt ein paar Berichte über FTOC verwendet, um RTE24zu generieren. Das FTOC-System weist jedoch mehrere Einschränkungen auf. Um mit dem Sauerstoffmangel in einer künstlichen Organkultur fertig zu werden, haben mehrere Gruppen entweder eine halbtrockene Membrankultur23oder Hochsauerstoff-Untergetauchtkultursysteme25verwendet. Allerdings können keine aktuellen Methoden ständig eine homogene Population von postthymischen T-Zellen erzeugen. Um die Grenzen des klassischen FTOC-Systems zu überwinden, haben wir ein 3D-Thymkultursystem entwickelt, das technische Verbesserungen gegenüber herkömmlichen Methoden bietet15. Zum Beispiel, mit unserer 3D-Thymkultur-Methode, maximaler Sauerstoffaustausch und das Fehlen von Oberflächen-Lappen mechanische Beanspruchung halten die Thymianlappen in einer physiologischeren Umgebung. Darüber hinaus ermöglicht die Langzeitkultur, dass reife T-Zellen auf natürliche Weise aus den thymischen Lappen austreten. Schließlich ermöglichen Echtzeitbeobachtung und Mikromanipulation den Medienaustausch und eine konstante Sammlung von iTE, ohne die Thymianlappen physisch zu stören. So bietet die 3D-Thymkultur-Methode signifikante technische Verbesserungen sowie einen Weg, thymisch ausgewählte naive T-Zellen zu studieren, die zuvor nicht verfügbar waren.
Es gibt mehrere Wichtige für die erfolgreiche ITE-Generierung mit diesem 3D-Thymkultursystem. Die Qualität der FBS- und Kulturbedingungen ist entscheidend, um die Erweiterung von OP9/DLL1-Zellen aufrechtzuerhalten, ohne ihre Fähigkeit zur Unterstützung der iPSC-Differenzierung zu verlieren. Daher empfehlen wir eine Vorbewertung des FBS-Los sowie eine konsequente Passierung bei 80% Koninfluenza, um Zelldifferenzierung und Seneszenz zu verhindern. Zusätzlich ist eine konfluente OP9/DLL1-Kultur für die In-vitro-Differenzierung von iPSC in unreife T-Zellen erforderlich, da Unterschiede in der Konfluenz ihre Effizienz beeinflussen können. Schließlich ist das embryonale Alter der Thymschenlappen entscheidend für die Erzeugung von iTE. Wir empfehlen die Verwendung von E14.5 - 15.5 Thymischen Lappen.
Wie bei jedem neuen Protokoll hat diese Methode Einschränkungen und kann verbessert werden. Die hier vorgestellte Kulturtechnik erzeugt ca. 1000 iTE pro Thymianlappen pro Tag für einen Zeitraum von zwei Wochen. Eine erhöhte iTE-Generierung kann mit weiteren Modifikationen möglich sein, einschließlich der Optimierung der Sauerstoffkonzentration, des Medienvolumens und der Art der 3D-Kulturplatte. Die Addition oder Entfernung von Zytokinen sowie Veränderungen der Zytokinkonzentration können ebenfalls zu einer verbesserten iTE-Ausbeute beitragen.
Da das hier vorgestellte 3D-Thymkultursystem thymische Auswanderer in einem vollständig ex vivo-System erzeugen kann, kann diese Technik auf eine Vielzahl von immunologischen und Adoptivzelltransfer-Forschungsprojekten angewendet werden, einschließlich, aber nicht beschränkt auf T Zelldifferenzierung, postthymische T-Zellreifung und Generierung antigenspezifischer T-Zellen aus hämatopoetischem Vorläufer oder Stammzellen. Obwohl diese Methode nicht direkt auf menschliche Proben anwendbar ist, haben iTE und das 3D-Thymkultursystem ein großes Potenzial zur Aufklärung der molekularen Mechanismen positiver und negativer Selektion und können die Schaffung eines Kultursystems erleichtern, das es ermöglicht, die Erzeugung klinisch relevanter Tumorantigen-spezifische naive T-Zellen für ACT.
Offenlegungen
Die Autoren Raul Vizcardo, Nicholas D. Klemen und Nicholas P. Restifo sind Erfinder der anhängigen internationalen Patentanmeldung PCT/US2017/65986, eingereicht am 13. Dezember 2017 mit dem Titel "Methods of Preparing an Isolated or Purified Population of Thymic Emigrant Cells und Behandlungsmethoden mit dem gleichen."
Danksagungen
Wir danken Hiroshi Kawamoto und Kyoko Masuda für die bereitstellung der OP9/DLL1-Zelllinie. Wir danken Alan B. Hoofring und Erina Z. Er für grafische Unterstützung. Diese Forschung wurde durch das Intramural Research Program des US National Cancer Institute (ZIA BC010763) und das Cancer Moonshot Programm für das Center for Cell-based Therapy am NCI, NIH, unterstützt. Die Arbeit wurde auch von der Milstein Family Foundation unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals, Peptides and Recombinant Proteins | |||
| 2-deoxyguanosine | Sigma-Aldrich | 312693-72-4 | |
| 2-Mercaptoethanol (1,000x) | Thermo Fisher Scientific | 21985-023 | |
| ACK Lysing Buffer | Gibco | A1049201 | |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| Blasticidin | Thermo Fisher Scientific | R21001 | |
| FBS | Gemini | 100-500 | |
| Flt-3 ligand | R&D Systems | 427-FL | |
| GlutaMAX (100x) | Thermo Fisher Scientific | 35050-061 | |
| hgp100 | Genscript | 282077-1, KVPRNQDWL | |
| Interleukin-2 | R&D Systems | 402-ML | |
| Interleukin-7 | R&D Systems | 407-ML | |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| MEM powder | Gibco | 61100061 | |
| Monothioglycerol | Sigma-Aldrich | M-6145 | |
| Nucleoprotein | Global Peptides | ASNENMETM | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140-122 | |
| Phosphate buffered saline pH 7.4 (1x) | Thermo Fisher Scientific | 10010-023 | |
| Puromycin | Thermo Fisher Scientific | A1113803 | |
| RPMI 1640 | Gibco | 11875093 | |
| Sodium Pyruvate | Thermo Fisher Scientific | 11360-070 | |
| Stem Cell Factor (SCF) | R&D Systems | 455-MC | |
| Stemfactor LIF, Mouse Recombinant | STEMGENT | 03-0011-100 | |
| Trypsin-EDTA (0.05%), phenol red | Thermo Fisher Scientific | 25300-062 | |
| Cell Culture Vessels and others | |||
| 10 cm dish | Corning, Inc. | 353003 | |
| 12 mL Syringe | Covidien Monoject | 22-652-090 | |
| 6 well plate | Corning/Coster | 3516 | |
| Cell strainer 100 μm | Fisher Scientific | 22-363-549 | |
| Cell strainer 40 μm | Fisher Scientific | 22-363-547 | |
| Forceps | DUMONT | 0108-5PO | |
| Lab soaker mat | Versi-Dry | Cat. EF2175CX 74018-00 | |
| Membrane filters ( 0.8 μm, 47diam) | Whatman | WHA7408004 ALDRICH | |
| Perfecta3D Hanging Drop Plate | Sigma-Aldrich | HDP1096 | |
| U Bottom 96 well plate | Corning/Coster | 3799 | |
| Experimental Cell lines | |||
| CD3-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| MEF-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Mouse Embryonic Fibroblasts (MEF) | ATCC | SCRC-1040; RRID:MGI:5007926 | |
| OP9/N-DLL1 | Riken Bioresource center | Cat# RCB2927; RRID:CVCL_B220 | |
| Pmel-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Experimental mouse models | |||
| B6.SJL-PtprcaPepcb/BoyCrCrl | Charles River | Strain Code 564; RRID:IMSR_CRL:564 | |
| C57BL/6N | NCI/Charles River | N/A | |
| Pmel-1 mice | Overwijk et al. | J Exp Med 198(4):569-80 | |
| Antibodies | |||
| Anti-aTCR | Biolegend | 109202; RRID:AB_313425 | |
| Anti-CD3 | abcam | ab11089; RRID:AB_369097 | |
| Anti-CD4 | BD Biosciences | 553730; RRID:AB_395014 | |
| Anti-CD44 | BD Biosciences | 559250; RRID:AB_398661 | |
| Anti-CD45.1 | BD Biosciences | 553775; RRID:AB_395043 | |
| Anti-CD45.2 | BD Biosciences | 553772; RRID:AB_395041 | |
| Anti-CD62L | BD Biosciences | 560516; RRID:AB_1645257 | |
| Anti-CD69 | BD Biosciences | 552879; RRID:AB_394508 | |
| Anti-CD8a | BD Biosciences | 557959; RRID:AB_396959 | |
| Anti-CD8b | BD Biosciences | 550798; RRID:AB_393887 | |
| Anti-H-2Kb | BD Biosciences | 553570; RRID:AB_394928 | |
| Anti-IFN-g | BD Biosciences | 557998; RRID:AB_396979 | |
| Anti-IL-2 | BD Biosciences | 554428; RRID:AB_395386 | |
| Anti-TCRb | Thermo Fisher Scientific | 35-5961-81; RRID:AB_469741 | |
| Anti-TCRVb13 | BD Biosciences | 553204; RRID:AB_394706 | |
| Anti-TNFa | BD Biosciences | 557644; RRID:AB_396761 |
Referenzen
- Gattinoni, L., et al. Wnt signaling arrests effector T cell differentiation and generates CD8+ memory stem cells. Nature Medicine. 15 (7), 808-813 (2009).
- Rosenberg, S. A., et al. Durable complete responses in heavily pretreated patients with metastatic melanoma using T-cell transfer immunotherapy. Clinical Cancer Research. 17 (13), 4550-4557 (2011).
- Singh, N., Perazzelli, J., Grupp, S. A., Barrett, D. M. Early memory phenotypes drive T cell proliferation in patients with pediatric malignancies. Science Translational Medicine. 8 (320), 320-323 (2016).
- Crompton, J. G., Clever, D., Vizcardo, R., Rao, M., Restifo, N. P. Reprogramming antitumor immunity. Trends of Immunology. 35 (4), 178-185 (2014).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Vizcardo, R., et al. Regeneration of human tumor antigen-specific T cells from iPSCs derived from mature CD8(+) T cells. Cell Stem Cell. 12 (1), 31-36 (2013).
- Nishimura, T., et al. Generation of rejuvenated antigen-specific T cells by reprogramming to pluripotency and redifferentiation. Cell Stem Cell. 12 (1), 114-126 (2013).
- Takada, K., Kondo, K., Takahama, Y. Generation of Peptides That Promote Positive Selection in the Thymus. Journal of Immunology. 198 (6), 2215-2222 (2017).
- Yamagata, T., Mathis, D., Benoist, C. Self-reactivity in thymic double-positive cells commits cells to a CD8 alpha alpha lineage with characteristics of innate immune cells. Nature Immunology. 5 (6), 597-605 (2004).
- Themeli, M., et al. Generation of tumor-targeted human T lymphocytes from induced pluripotent stem cells for cancer therapy. Nature Biotechnology. 31 (10), 928-933 (2013).
- Serwold, T., Hochedlinger, K., Inlay, M. A., Jaenisch, R., Weissman, I. L. Early TCR expression and aberrant T cell development in mice with endogenous prerearranged T cell receptor genes. Journal of Immunology. 179 (2), 928-938 (2007).
- Lei, F., et al. In vivo programming of tumor antigen-specific T lymphocytes from pluripotent stem cells to promote cancer immunosurveillance. Cancer Research. 71 (14), 4742-4747 (2011).
- Maeda, T., et al. Regeneration of CD8alphabeta T Cells from T-cell-Derived iPSC Imparts Potent Tumor Antigen-Specific Cytotoxicity. Cancer Research. 76 (23), 6839-6850 (2016).
- Serwold, T., et al. T-cell receptor-driven lymphomagenesis in mice derived from a reprogrammed T cell. Proceedings of the National Academy of Sciences of the United States of America. 107 (44), 18939-18943 (2010).
- Vizcardo, R., et al. Generation of Tumor Antigen-Specific iPSC-Derived Thymic Emigrants Using a 3D Thymic Culture System. Cell Reports. 22 (12), 3175-3190 (2018).
- Jenkinson, W., Jenkinson, E., Anderson, G. Preparation of 2-dGuo-treated thymus organ cultures. Journal of Visualized Experiments. (18), (2008).
- Hamazaki, Y., Sekai, M., Minato, N. Medullary thymic epithelial stem cells: role in thymic epithelial cell maintenance and thymic involution. Immunological Reviews. 271 (1), 38-55 (2016).
- Vivier, E., et al. Innate or adaptive immunity? The example of natural killer cells. Science. 331 (6013), 44-49 (2011).
- Rosen, S. D. Ligands for L-selectin: homing, inflammation, and beyond. Annual Review of Immunology. 22, 129-156 (2004).
- Hogquist, K. A., Xing, Y., Hsu, F. C., Shapiro, V. S. T Cell Adolescence: Maturation Events Beyond Positive Selection. Journal of Immunology. 195 (4), 1351-1357 (2015).
- Best, J. A., et al. Transcriptional insights into the CD8(+) T cell response to infection and memory T cell formation. Nature Immunology. 14 (4), 404-412 (2013).
- Schmitz, I., Clayton, L. K., Reinherz, E. L. Gene expression analysis of thymocyte selection in vivo. International Immunology. 15 (10), 1237-1248 (2003).
- Nitta, T., Ohigashi, I., Takahama, Y. The development of T lymphocytes in fetal thymus organ culture. Methods in Molecular Biology. 946, 85-102 (2013).
- Ueno, T., et al. Role for CCR7 ligands in the emigration of newly generated T lymphocytes from the neonatal thymus. Immunity. 16 (2), 205-218 (2002).
- Watanabe, Y., Katsura, Y. Development of T cell receptor alpha beta-bearing T cells in the submersion organ culture of murine fetal thymus at high oxygen concentration. European Journal of Immunology. 23 (1), 200-205 (1993).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten