Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Genetische Kartierung von Thermotoleranzunterschieden zwischen Spezies von Saccharomyces Hefe über Genom-Wide Reciprocal Hemizygosity Analysis
In diesem Artikel
Zusammenfassung
Die reziproke Hemizygosität durch Sequenzierung (RH-seq) ist eine leistungsstarke neue Methode, um die genetische Grundlage eines Merkmalsunterschieds zwischen den Arten abzubilden. Pools von Hemizygoten werden durch Transposon-Mutagenese erzeugt und ihre Fitness wird durch Wettbewerbswachstum mit einer durchgehenden Sequenzierung verfolgt. Die Analyse der resultierenden Daten zeigt Gene, die dem Merkmal zugrunde liegen.
Zusammenfassung
Ein zentrales Ziel der modernen Genetik ist es zu verstehen, wie und warum sich Organismen in freier Wildbahn im Phänotyp unterscheiden. Bis heute hat sich das Feld weitgehend auf die Stärke von Verknüpfungs- und Assoziationsmapping-Methoden entwickelt, die die Beziehung zwischen DNA-Sequenzvarianten und Phänotyp über rekombinante Nachkommen hinweg aus Paarungen zwischen Individuen einer Art verfolgen. Diese Ansätze sind zwar mächtig, aber nicht gut geeignet, um Unterschiede zwischen reproduktiv isolierten Arten zu charakterisieren. Hier beschreiben wir eine neue Methode zur genomweiten Zerlegung natürlicher Merkmalsvariationen, die leicht auf inkompatible Arten angewendet werden kann. Unsere Strategie, RH-seq, ist eine genomweite Umsetzung des gegenseitigen Hemizygote-Tests. Wir nutzten es, um die Gene zu identifizieren, die für das auffällige Hochtemperaturwachstum der Hefe Saccharomyces cerevisiae im Vergleich zu ihrer Schwesterart S. paradoxusverantwortlich sind. RH-seq nutzt Transposon-Mutagenese, um einen Pool von reziproken Hemizygoten zu schaffen, die dann durch einen Hochtemperaturwettbewerb mittels Hochdurchsatzsequenzierung verfolgt werden. Unser RH-Seq-Workflow, wie er hier dargelegt ist, bietet eine rigorose, unvoreingenommene Möglichkeit, alte, komplexe Merkmale in der angehenden Hefeklade zu sezieren, mit dem Vorbehalt, dass eine ressourcenintensive tiefen Sequenzierung erforderlich ist, um eine genomische Abdeckung für die genetische Kartierung zu gewährleisten. Da die Sequenzierungskosten sinken, ist dieser Ansatz für die zukünftige Verwendung in eukaryotes vielversprechend.
Einleitung
Seit beginne des Feldes, ist es ein hauptziel in der Genetik, die mechanistische Grundlage der Variation zwischen wilden Individuen zu verstehen. Wenn wir Loci, die einem Merkmal von Interesse zugrunde liegen, kartieren, können die entstehenden Gene als Ziele für Diagnostika und Medikamente sofort von Nutzen sein und Licht auf die Prinzipien der Evolution werfen. Der Industriestandard zu diesem Zweck besteht darin, eine Beziehung zwischen Genotyp und Phänotyp in einer Population über Verbindung oder Assoziation zu testen1. So mächtig diese Ansätze auch sind, sie haben eine wichtige Einschränkung – sie verlassen sich auf große Platten rekombinanter Nachkommen aus Kreuzungen zwischen interfertilen Individuen. Sie nützen nichts bei der Untersuchung von Arten, die sich überhaupt nicht zur Nachkommenschaft paaren können. Als solches hatte das Feld wenig Kapazität für unvoreingenommene Zerlegung von Merkmalsunterschieden zwischen reproduktiv isolierten Arten2.
In dieser Arbeit berichten wir über die technischen Grundlagen einer neuen Methode, RH-seq3, für genombasierte Erhebungen der genetischen Grundlagen der Merkmalsvariation zwischen den Arten. Dieser Ansatz ist eine massiv parallele Version des gegenseitigen Hemizygotentests4,5, der zuerst als eine Möglichkeit konzipiert wurde, die phänotypischen Auswirkungen von allelischen Unterschieden zwischen zwei genetisch unterschiedlichen Hintergründen an einem besonderer Ort (Abbildung 1A). In diesem Schema werden die beiden divergierenden Individuen zunächst zu einem Hybriden verpaart, dessen Genom zur Hälfte von jedem der jeweiligen Elternteile stammt. In diesem Hintergrund werden mehrere Stämme generiert, die jeweils eine unterbrochene oder gelöschte Kopie des Alleels des Ortes enthalten. Diese Stämme sind hemizygot, da sie überall im Genom diploid bleiben, außer am Ort des Interesses, wo sie als haploid gelten, und werden als reziprok bezeichnet, da jeder nur ein Elternallel fehlt, wobei sein verbleibendes Allel von der anderen Elternteilen. Durch den Vergleich der Phänotypen dieser reziprok-hemizygoten Stämme kann man feststellen, ob DNA-Sequenzvarianten am manipulierten Ort zum Merkmal des Interesses beitragen, da Varianten am Ort der Klagegang der einzige genetische Unterschied zwischen dem hemizygote Stämme. Auf diese Weise ist es möglich, genetische Unterschiede zwischen den Arten mit einem phänotypischen Unterschied zwischen ihnen in einem gut kontrollierten Versuchsaufbau zu verknüpfen. Bis heute wurden die Anwendungen dieses Tests in einem Kandidaten-Gen-Framework eingereicht, d. h. in Fällen, in denen die Hypothese bereits in der Hand ist, dass natürliche Variationen an einem Kandidatenort eine Eigenschaft beeinflussen könnten.
Im Folgenden legen wir das Protokoll für einen genomgroßen gegenseitigen Hemizygositätsbildschirm fest, bei dem Hefe als Modellsystem verwendet wird. Unsere Methode schafft eine genomische Ergänzung von Hemizygotenmutanten, indem sie lebensfähige, sterile F1-Hybriden zwischen Arten erzeugt und sie der Transposon-Mutagenese aussetzt. Wir bündeln die Hemizygoten, messen ihre Phänotypen in Sequenzierungs-basierten Assays und testen auf Frequenzunterschiede zwischen Klonen des Pools, die die Allele der beiden Eltern eines bestimmten Gens tragen. Das Ergebnis ist ein Katalog von Loci, bei dem Varianten zwischen Denspezies das Merkmal des Interesses beeinflussen. Wir implementieren den RH-seq-Workflow, um die genetische neratorischen Grundlage für die Thermoverträglichkeitsunterschiede zwischen zwei angehenden Hefearten, Saccharomyces cerevisiae und S. paradoxus, aufzuklären, die vor 5 Millionen Jahren auseinandergingen.
Protokoll
1. Vorbereitung des piggyBac-haltigen Plasmids zur Transformation
- Streifen Sie zu einzelnen Kolonien den E. coli-Stamm, der Plasmid pJR487 auf einer LB + Carbenicillin Agarplatte beherbergt. 1 Nacht bei 37 °C oder bis einzelne Kolonien auftauchen.
HINWEIS: Eine Beschreibung, wie Plasmid pJR487 geklont wurde, finden Sie in unserer vorherigen Arbeit3. - Impfung 1 L LB + Carbenicillin bei 100 g/ml mit einer einzigen Kolonie von E. coli, die pJR487 in einem 2-L-Glaskolben enthält. Über Nacht bei 37 °C mit Schütteln bei 200 Umdrehungen von Euro bis gesättigt (OD600 bei 1,0) wachsen.
- Reinigen Sie Plasmid-DNA aus der Kultur mit einem groß angelegten Plasmid-Vorbereitungskit, wie im veröffentlichten Protokoll des Herstellers angegeben (Details siehe Tabelle der Materialien). Elute die DNA nach einer 10-minütigen Inkubation mit 5 ml Elutionspuffer erwärmt auf 37 °C.
- Messen Sie die Menge und Qualität der Plasmid-DNA mit einem Spektralphotometer (Details siehe Materialtabelle).
- Wiederholen Sie die Schritte 1,2 – 1,4, bis insgesamt mindestens 11 mg Plasmid-DNA bei einem A260:A280-Verhältnis von mindestens 1,8 isoliert sind. Dies kann ein paar Vorbereitungen dauern, je nach Effizienz.
- Mischen Sie alle Plasmidpräparationen in einem einzigen Rohr und bringen Sie das Gesamtvolumen bis zu 20 ml mit Elutionspuffer oder Wasser. Messen Sie die endgültige Menge und Qualität erneut mit einem Spektralphotometer. Die Konzentration des Plasmids sollte in diesem endgültigen Volumen von 20 ml mindestens 538 ng/l betragen. Wenn die Konzentration höher als 538 ng/l ist, verdünnen Sie das Plasmid mit Elutionspuffer oder Wasser auf 538 ng/L. Plasmid kann bis zur Anwendung bei 4 °C bis zu einigen Wochen gelagert werden.
2. Schaffung eines Pools von ungezielten genomweiten reziproziproken Hemizygoten
- Vorbereitung von Hybridhefezellen zur Transformation
- Jr507 von einem -80 °C Gefrierstockstamm auf einzelne Kolonien auf einer YPD-Agarplatte herausstreichen. 2 Tage lang bei 26 °C bebrüten oder bis Kolonien auftreten.
HINWEIS: JR507 ist ein Hybridstamm, der durch einzellige Paarung von haploiden Sporen von S. cerevisiae DBVPG1373 und S. paradoxus Z1 (mit einem Tetrad-Dissektionsmikroskop)3hergestellt wird. - 100 ml flüssiges YPD in einem 250 ml Glaskolben mit einer einzigen Kolonie von JR507 impfen und bei 28 °C, 200 U/min für 24 Stunden oder bis zur stationären Phase schütteln.
- Messen Sie am nächsten Tag die optische Dichte bei 600 nm (OD600) der Übernachtungskultur. Schaffen Sie eine neue Kultur, indem Sie einen Teil der Nachtkultur mit frischer Flüssigkeit YPD in einen neuen 1 L Glaskolben zu einem OD600 von 0,2 und einem Volumen von 500 ml verdünnen.
ANMERKUNG: Beispielberechnung einer Rückverdünnung, wenn die Nachtkultur eine OD600 von 5,0 aufweist, wobei C optische Dichte und V Volumen ist: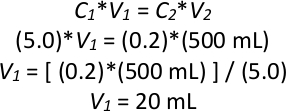
So würden 20 ml gesättigte Nachtkultur zu 480 ml flüssiger YPD hinzugefügt, um insgesamt 500 ml Kultur bei einem OD600 von 0,2 zu machen. - Wiederholen Sie Schritt 2.1.3 noch dreimal, um insgesamt vier 500 ml Kulturen bei einem OD600 von 0,2 in vier 1-L-Glaskolben zu machen, die die gleiche Nachtkultur für alle vier neuen Kulturen verwenden. Inkubieren Sie sie alle bei 28 °C für 6 Stunden (2-3 Generationen) Schütteln bei 200 Rpm.
- Kombinieren Sie zwei der 500 ml Kulturen, um eine 1 L-Kultur zu schaffen. Kombinieren Sie die verbleibenden zwei 500 ml-Kulturen, um eine weitere 1 L-Kultur zu erstellen. An dieser Stelle gibt es zwei 1 L Kulturen. Jede dieser 1 L-Kulturen wird in den folgenden Schritten mit pJR487 transformiert.
- Jr507 von einem -80 °C Gefrierstockstamm auf einzelne Kolonien auf einer YPD-Agarplatte herausstreichen. 2 Tage lang bei 26 °C bebrüten oder bis Kolonien auftreten.
- Umwandlung von pJR487 in hybride Hefezellen
- Teilen Sie jede der 1 L-Kulturen in zwanzig 50 ml-Aliquots in 20 Kunststoffkonischen Rohren für insgesamt 40 Rohre auf. Stellen Sie 20 Rohre beiseite und führen Sie die folgenden Schritte auf 20 Röhren gleichzeitig aus.
- Zentrifugieren Sie jede der zwanzig Röhren für 3 min bei 1.000 x g, um die Hefezellen zu pellet. Entsorgen Sie den Überstand.
- Jedes Pellet mit 25 ml sterilemH2 O durch Wirbeln aufheben. Zentrifuge für 3 min bei 1.000 x g. Entsorgen Sie den Überstand.
- Setzen Sie jedes Pellet mit 5 ml 1x TE, 0,1 M LiOAc Puffer durch Wirbel auf. Zentrifuge für 3 min bei 1.000 x g. Entsorgen Sie den Überstand.
- Wiederholen Sie Schritt 2.2.4. Während die Zellen zentrifugieren, bereiten Sie mindestens 120 ml einer Lösung von 39,52% Polyethylenglykol, 0,12 M LiOAc und 1,2x Tris-EDTA Puffer (12 mM Tris-HCl und 1,2 mM EDTA) vor. Auf Eis aufbewahren.
- Um die Plasmid-DNA für die Transformation vorzubereiten, kochen Sie zunächst 4 ml Lachssperma-DNA bei 100 °C für 5 min und kühlen Sie sie sofort auf Eis für 5 min ab. Mischen Sie dann 20 ml pJR487 (in Abschnitt 1) in einer Konzentration von 538 ng/l mit der 4 ml gekühlten Lachssperma-DNA für ein Gesamtvolumen von 24 ml. Auf Eis halten, bis es verwendet wird.
- Fügen Sie 600 L Plasmid-DNA mit Lachssperma-DNA auf jedem Zellpellet gemischt. Lassen Sie sich noch nicht aussetzen.
- Fügen Sie 3 ml PEG-LiOAc-TE-Lösung in Schritt 2.2.5 zu jedem Pellet hinzu. Setzen Sie das Pellet durch Pipettieren nach oben und unten und Wirbeln.
- Inkubieren Sie jedes Rohr für 10 min bei Raumtemperatur.
- Hitzeschock jedes Rohr für 26 min in einem Wasserbad auf 39 °C eingestellt.
HINWEIS: Alle paar Minuten jede Röhre umkehren, um zu verhindern, dass sich die Zellen auf der Unterseite des Rohres absetzen. - Zentrifugieren Jedes Rohr für 3 min bei 1.000 x g. Entsorgen Sie den Überstand und setzen Sie jedes Pellet in 10 ml YPD durch Wirbel wieder aus. Kombinieren Sie alle zwanzig Rohre zu einem neuen Glaskolben. Das Gesamtvolumen der Zellen sollte 200 ml betragen.
- 66,6 ml Zellen in einen neuen 1 L Glaskolben übertragen und mit flüssigem YPD auf ein Volumen von 500 ml bringen. Wiederholen Sie zwei weitere Wiederholungen, um die gesamten 200 ml transformierter Zellen zu verwenden. Messen Sie die OD600 jeder neuen 500-ml-Kultur (erwarten Sie eine OD600 von 0,35-4).
- Schütteln Sie alle drei Kolben bei 28 °C für 2 Stunden, um sich (<1 Generation) bei 200 U/min zu erholen.
- Fügen Sie 0,5 ml 300 mg/ml G418 zu jedem der drei Kolben hinzu, zu einer Endkonzentration von 300 g/ml G418 und bei 28 °C, 200 U/min wieder zum Schütteln bringen.
HINWEIS: Vor diesem Schritt haben sich die transformierten Hybridzellen von der Transformation erholt. Nach Zugabe von G418 wird das Vorhandensein des Plasmids pJR487 ausgewählt. Alle Zellen, die das Plasmid während der Transformation nicht aufgenommen haben, beginnen zu sterben. - Wiederholen Sie die Schritte 2.2.2 – 2.2.14 mit den restlichen 20 konischen Zellröhren. An dieser Stelle sollten sechs 1 L Glaskolben mit jeweils 500 ml Zellen mit G418 hinzugefügt werden.
- Inkubieren Sie alle sechs Zellkolben bei 28 °C, schütteln bei 200 U/min, für ca. 2 Tage oder bis in jedem Kolben ein OD600 von 2,3 USD erreicht ist. Kombinieren Sie alle sechs Kolben zu einer einzigen Kultur.
HINWEIS: Obwohl nicht alle Zellen in dieser Kultur in nachgelagerten Schritten verwendet werden, besteht das Ziel der Verwendung solch großer Volumes darin, so viele eindeutige Transformationsereignisse wie möglich zu erstellen und alle Verzerrungen in einer einzelnen Transformation zu normalisieren, indem sie alle zusammen. - Verwenden Sie die in 2.2.16 erstellte Kultur, um zwei neue 1 L-Kolben mit 500 ml YPD + G418 (300 g/ml) auf eine OD600 von 0,2 zu impfen. Es wird übrig gebliebene Kultur geben, die verworfen werden kann.
- Inkubieren Sie beide 1-L-Kolben bei 28 °C über Nacht, mit Schütteln bei 200 U/min, bis jeder eine OD600 von 2,2 USD (3,5 Generationen) erreicht. Kombinieren Sie beide Kulturen zu einer einzigen Kultur und messen Sie die OD600 der kombinierten Kultur wieder.
HINWEIS: An dieser Stelle sollte die Kultur fast vollständig aus Zellen bestehen, die Plasmid pJR487 beherbergen. In einem Teil der Zellpopulation wird das PiggyBac-Transposon durch die aus dem Plasmid exprimierte Transposase aus dem Plasmid aus dem Plasmid in das Genom transponiert worden sein. Eine fortgesetzte Expression der Transposase kann jedoch im Laufe einer Selektion zur Transposition führen, was die Beziehung zwischen Genotyp und Phänotyp verschleiern würde. Das Ziel der nächsten Schritte ist es, eine Gegenauswahl gegen das Vorhandensein des Plasmids durchzuführen, um sicherzustellen, dass es keine expressionionium mehr der Transposase gibt. Der resultierende Pool ist eine Mischung von Zellen mit oder ohne das in das Genom integrierte Transposon, aber nur Zellen, die das Transposon enthalten, werden während der nachfolgenden Mapping-Schritte detektiert. Die Zeit in der Transformation, in der Transposase exprimiert wird, bevor die Plasmid-Codierung verloren geht, kann die Wahrscheinlichkeit bestimmen, dass ein gegebener Klon nach Mutagenese mehr als eine Transposon-Einfügung beherbergt. Die Häufigkeit dieser, die sich bei der Analyse eines Gens zu einem Zeitpunkt als "sekundäre" Mutationen manifestieren, kann geschätzt werden, indem eine definierte Anzahl von Kolonien nach der Mutagenese angeordnet wird, dann ihre DNA kombiniert und die Anzahl der unabhängigen Einfügungen sequenziert wird. Positionen im Pool. - Zentrifuge 25 ml dieser Kultur für 3 min bei 1.000 x g. Berechnen Sie die Anzahl der gesamten OD600-Einheiten von Zellen, die sich in der 25-km-Tabelle befinden (siehe Beispielberechnung unten). Entsorgen Sie den Überstand und setzen Sie sich in genügend H2O wieder aus, um eine Zellsuspension von 1,85 OD600/ml durch Wirbeln zu erzeugen.
ANMERKUNG: Beispielberechnung für die Resuspension von Zellen in Wasser, wenn OD600 der kombinierten Kultur 2.2 war: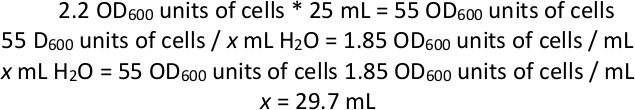
Fügen Sie also nach dem Spinnen von 25 ml Zellkultur unddem Wegwerfen des Überstandes genügend H2 O zu den Zellen hinzu, um das Gesamtvolumen von Zellen und Wasser auf 29,7 ml zu erhöhen (da das Zellpellet auch ein Volumen haben wird, fügen Sie weniger als 29,7 ml H2O hinzu). - Mit Glasperlen, Platte 1 ml resuspendierte Zellen in Wasser auf jede der 12 großen quadratischen kompletten synthetischen Agarplatten mit 5-FOA. Jede Platte 1-2 Tage lang bei 28 °C bebrüten oder bis sich ein Rasen auf dem Teller bildet.
- Mit kleinen sterilen Rakel, kratzen Sie die Zellen von jeder der 6 Platten in ein Rohr mit 35 ml sterilem Wasser. Wiederholen Sie dies mit den anderen 6 Platten für insgesamt zwei Zellröhren und Wasser. Kombinieren Sie alle Zellsuspensionen in einem einzigen Rohr. Messen Sie die OD600 dieser Suspension, indem Sie Wasser als Rohling verwenden. Bringen Sie die OD600/ml-Konzentration der Zellen auf 44,4 OD600 Einheiten/ml mit Wasser. Nach unserer Erfahrung beträgt die Umsetzungseffizienz (der Anteil der KAN+-Zellen, die URA-) im Durchschnitt 50%.
- Bestimmen Sie die Anzahl der -80 °C Gefriervorräte von Zellen zu speichern. Jedes Aliquot kann in Zukunft für ein einzelnes Experiment verwendet werden.
HINWEIS: Da die Generierung des Pools zeitaufwändig ist, sollten Sie mehrere Durchstechflaschen bei versehentlichem Missbrauch oder für DieReplikationsexperimente speichern. 20-30 Bestände sind eine vernünftige Zahl. - Jeder Gefrierbestand wird 40 OD600 Einheiten von Zellen in 1 ml von 10% DMSO enthalten. Fügen Sie 900 L Zellen zu 100 L DMSO hinzu. Wiederholen Sie diesen Vorgang für die Gesamtzahl der erstellten Gefriervorräte. Lagern Sie jeweils bei -80 °C für die zukünftige Verwendung.
3. Auswahl der wechselseitigen Hemizygoten in einem gepoolten Format
- Vom -80 °C Gefrierschrank ein einzelnes Aliquot aus gepoolten wechselseitigen Hemizygoten aus Abschnitt 2 bei Raumtemperatur auftauen.
HINWEIS: Lassen Sie den Aliquot nicht lange bei Raumtemperatur sitzen, sobald es auftaut, verwenden Sie es sofort. - Verwenden Sie die gesamte 1 ml aliquot, um 150 ml flüssige YPD in einem 250 ml Glaskolben zu impfen. Messen Sie die OD600 dieser Kultur, und brüten Sie dann bei 28 °C, schütteln bei 200 Umdrehungen pro Minute, für 7 Stunden, oder bis die Kultur durch 2-3 Bevölkerungsverdoppelungen gegangen ist. An diesem Punkt ist die Kultur bereit, verwendet zu werden, um Kulturen zu impfen, die sich einer Auswahl unterziehen.
HINWEIS: Beispielberechnung: Wenn der OD600 des ursprünglichen Kolbens 0,25 misst, inkubieren Sie die Kultur, bis sie eine OD600 von mindestens 1,0 erreicht. Wenn bei "Time-Zero" (T-0) Probenpunkte gewünscht werden, um die Hemizygotenpopulation vor der Selektion zu untersuchen, können Zellpellets jetzt durch Zentrifugieren von 5-10 ml Kultur pro Pellet bei 1.000 x g für 3 min eingenommen werden, indem der Überstand verworfen und Gefrieren bei -80 °C. - Verwenden Sie den angebauten Hemizygote-Pool, um Kulturen zur Auswahl in einem geeigneten Replikationsschema zu impfen, sowohl bei hoher Temperatur (39 °C) als auch bei freizügiger Temperatur (28 °C). Richten Sie mindestens drei biologische Replizierenselektionskulturen bei jeder Temperatur für insgesamt sechs Selektionskulturen ein.
- Erstellen Sie jede Selektionskultur mit 500 ml insgesamt in einem 2 L Glaskolben mit flüssigem YPD und impfen Sie zu einem OD600 von 0,02. Schütteln Sie jede Selektionskultur bei 100 Umdrehungen pro Minute bei 28 °C oder 39 °C, bis sich 6-7 Populationsverdoppelungen verdoppelt haben (entsprechend einem OD600 von 1,28-2,56). Versuchen Sie, so nah wie möglich die endgültige OD600 aller Auswahlkulturen zu entsprechen.
HINWEIS: Selektionskulturen bei 28 °C wachsen bei 39 °C schneller als Selektionskulturen. Folglich werden Selektionskulturen bei 39 °C eine längere Zeit im Inkubator verbringen. Fahren Sie mit den folgenden Schritten mit jedem Kolben fort, sobald er bereit ist, unabhängig von der Gesamtanzahl der Stunden, die im Inkubator verbracht werden. Nach unserer Erfahrung brauchten Kulturen bei 28 °C bzw. 39 °C 12 bzw. 18 Stunden, um eine OD von 2,0 zu erreichen. Lange Auswahlen könnten den Vorteil haben, kleine Fitnesseffekte zu verstärken, aber auch de novo Hintergrundmutationen entstehen lassen, die Lärm in die endgültige Verteilung von Fitnessen über Transposonmutanten in einem Gen/Allel führen würden. Daher ist es wichtig, die Auswahlzeit in einem RH-Seq-Experiment zu begrenzen.
- Erstellen Sie jede Selektionskultur mit 500 ml insgesamt in einem 2 L Glaskolben mit flüssigem YPD und impfen Sie zu einem OD600 von 0,02. Schütteln Sie jede Selektionskultur bei 100 Umdrehungen pro Minute bei 28 °C oder 39 °C, bis sich 6-7 Populationsverdoppelungen verdoppelt haben (entsprechend einem OD600 von 1,28-2,56). Versuchen Sie, so nah wie möglich die endgültige OD600 aller Auswahlkulturen zu entsprechen.
- Erntezellpellets aus jeder Selektionskultur. Berechnen Sie das Volumen, das erforderlich ist, um 7 OD600 Einheiten von Zellen und Zentrifugen bei 1.000 x g für 3 min mindestens vier Pellets dieses Bandes aus jeder Selektionskultur als technische Replikationen für die Bibliotheksvorbereitung und -sequenzierung zu erhalten (siehe Abschnitte 4 und 5 , unten). Den Überstand entsorgen und bei -80 °C lagern.
HINWEIS: Beispiel, wenn ein Auswahlkolben eine endgültige OD600 von 2.0 hat: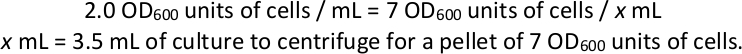
4. Tn-seq Bibliotheksbau und Illumina-Sequenzierung zur Bestimmung der Fülle von Transposon-Mutantenhemizygoten
- Tauen Sie auf Eis jedes Zellpellet aus Abschnitt 3, die sequenziert werden soll.
- Isolieren Sie die gesamte genomische DNA (gDNA) aus jedem Zellpellet mit einem Hefe-gDNA-Reinigungskit nach den Anweisungen des Herstellers. Resuspend ieren die DNA in 50 l Elutionspuffer erwärmt auf 65 °C.
- Quantifizieren Sie die Menge an gDNA aus jedem Pellet mit einem Fluorimeter. Die minimale Gesamtmenge an gDNA, die für jedes Zellpellet erforderlich ist, um eine NGS-Bibliothek der nächsten Generation für Tn-seq zu erstellen, beträgt das folgende Verfahren 1 g.
ANMERKUNG: Es können weniger als 1 gDNA verwendet werden, um eine Bibliothek zu erstellen, aber die endgültige Quantität und Qualität der Bibliothek wird leiden. - Befolgen Sie ein etabliertes Protokoll zum Erstellen von Tn-seq-Bibliotheken7. Beachten Sie die folgenden relevanten Informationen, die für dieses Protokoll eindeutig sind:
- Nach gDNA-Scheren, Endreparatur und Adapterligation die gDNA, die das Transposon enthält, über PCR verstärken. Verwenden Sie für diese PCR die folgende Vorwärts- und Rückwärtsgrundierung, die für die PiggyBac-Transposon- bzw. NGS-Adapter spezifisch sind:
Vorwärts (N – zufälliges Nukleotid)
5' ATGATACGGCGACCACCGAGATCTACCTCTCCTACACGACG
CTCTTCCGATCTNNNNNNAGCAATATTTCAAGAATGCATGCGTCAAT 3'
Umgekehrt (die Strecke von Ns stellt einen eindeutigen 6-Bp-Index dar, der für Multiplexing verwendet wird. Weitere Informationen zu Indizes finden Sie weiter unten:
5' CAAGCAGAAGACGGCATACGAGATNNNNNNNGTGACTGGAGTTCAG
ACGTGTGCTCTTCCGATCT 3' - Verwenden Sie die mitgelieferten Bereinigungsschritte mit größenselektiven Perlen, um den Anteil der geklonten Fragmente in der endgültigen Bibliothek zu minimieren, die zu kurz wären, um eine zu beschwichtigende genomische Sequenz einzuschließen.
HINWEIS: Nachdem die minimalen Replikationsanforderungen für Selektionskulturen bisher erfüllt wurden, werden 24 einzelne gDNA-Proben für die Sequenzierung vorhanden sein. Angesichts der aktuellen Kosten für die Sequenzierung ist es unwahrscheinlich, dass jede Probe eigennfürschweise ausgeführt wird. Um Samples auf derselben Spur zu kombinieren, erstellen Sie mehrere Reverse Primer mit jeweils einem eindeutigen 6-Basis-Paarindex. Samples mit unterschiedlichen Indizes können in dieselbe Sequenzierungsspur kombiniert und anschließend rechnerisch getrennt werden.
- Nach gDNA-Scheren, Endreparatur und Adapterligation die gDNA, die das Transposon enthält, über PCR verstärken. Verwenden Sie für diese PCR die folgende Vorwärts- und Rückwärtsgrundierung, die für die PiggyBac-Transposon- bzw. NGS-Adapter spezifisch sind:
- Sequenz-Single-End-150-Bp-Lesevorgänge aus jeder Bibliothek mit NGS-Technologien über acht Spuren hinweg.
HINWEIS: Die Menge der erforderlichen Sequenzierungs-Lesevorgänge hängt stark von der Qualität der bibliotheken ab, die im vorherigen Schritt erstellt wurden (d. h. der Anteil der DNA in der Bibliothek, die tatsächlich Transposon-DNA enthält und DNA darstellt, die aus reziproken Hemizygoten stammt). Dazu tragen zwei Hauptfaktoren bei. Erstens, da Zellen ohne integriertes Transposon während der Poolerstellung nicht gegengewählt werden, wird jede Kultur eine Mischung von Zellen mit und ohne Transposon sein. Zweitens ist selbst innerhalb der Genome von transposonhaltigen reziprokhaltigen reziproken hemizygoten, der größte Teil des Genoms ist nicht transposon-enthaltende Sequenz, und diese gDNA wird unweigerlich Teil der Bibliotheksvorbereitung sein. Das Ziel der endgültigen PCR-Verstärkung transposonhaltiger DNA ist es, das Verhältnis der transposonhaltigen DNA zu diesen beiden Quellen von Hintergrund-gDNA zu erhöhen. Je effizienter diese Verstärkung ist, desto höher wird der Anteil der Lesevorgänge in der nachgelagerten Analyse verwendet werden können. Je niedriger die Qualität der Bibliotheken ist, desto mehr Sequenzierung muss durchgeführt werden, da ein zunehmender Anteil der Lesevorgänge keine Transposon-DNA enthält und nicht nützlich ist. Angesichts der oben genannten Einschränkungen waren acht Bahnen der Sequenzierung in der Lage, die gegenseitigen Hemizygote-Übermengen in einem angemessenen Maße zu verfolgen. Eine genauere Sequenzierung würde eine tiefere Analyse ermöglichen.
5. Abbildung der Positionen von Transposon-Einfügungen und RH-Seq-Analysen
HINWEIS: Die folgende Datenanalyse wurde mit benutzerdefinierten Python-Skripten durchgeführt (online unter https://github.com/weiss19/rh-seq gefunden), konnte aber mit anderen Skriptsprachen neu erstellt werden. Im Folgenden werden die wichtigsten Schritte im Prozess beschrieben. Führen Sie die folgenden Schritte für jede einzelne Replizionlesedatei aus, es sei denn, es wird angegeben, sie zu kombinieren.
- Trimmen Sie Adaptersequenzen von Lesevorgängen ab und trennen Sie die Lesevorgänge jeder Replikation nach Index.
- Suchen Sie Lesevorgänge, die Transposon-Genom-Kreuzungen enthalten. Um dies zu erreichen, suchen Sie in jedem Gelesennach nach den letzten 20 Basispaaren des Transposons, CAGACTATCTCTTAGGGTTAA. Verwerfen Sie alle Lesevorgänge, die diese Sequenz nicht enthalten.
HINWEIS: Unserer Erfahrung nach beträgt der Anteil der Lesezuordnungen bis zum Ende des Transposons 83-95%. - Schneiden Sie die verbleibenden, transposonhaltigen Lesevorgänge so ab, dass sie nur die Sequenz nach dem 3' Ende des Transposons enthalten. Durch Mapping dieser Sequenz zum Hefegenom, bestimmen Sie den genomischen Kontext der Transposon-Insertion für jeden Gelesenen (Schritt 5.4 unten).
- Verwenden Sie BLAT oder ein gleichwertiges Mapping-Tool, um die Sequenz nach dem Transposon dem S. cerevisiae DBVPG1373 x S. paradoxus Z1 Hybridgenom zuzuordnen (Skriptname: map_and_pool_BLAT.py).
- Verwerfen Sie alle Lesevorgänge, für die weniger als 50 Basispaare verwendbarer Sequenz nach dem 3'-Ende des Transposons vorhanden sind. Kurze Sequenzen sind schwer auf einzigartige Weise abzubilden.
- Wenn Sie BLAT verwenden, verwenden Sie die folgenden Parameter: identity = 95, tile size = 12.
- Erstellen Sie ein grundlegendes Hybridgenom, das für die Kartierung verwendet werden kann, indem Sie die neuesten Versionen der Referenzgenome von S. cerevisiae S288c und S. paradoxus CBS432 verketten.
HINWEIS: Eine grundlegende Anmerkungsdatei, die die genomischen Grenzen einzelner Gene im Hybridgenom beschreibt, befindet sich im oben aufgeführten Github-Repository (Dateiname: YS2+CBS432+plasmid_clean). Verwenden Sie nur Lesevorgänge, welche Karte zu einem einzigen Ort im Hybridgenom (d.h. sind einzigartig für S. cerevisiae oder S. paradoxus). Es wird eine gleichmäßige Häufigkeit von Insertionsereignissen im genomischen Bereich erwartet; die Verteilung der Einfügepositionen auf die Genome wird an anderer Stelle berichtet3.
- Tally die Gesamtzahl der Lesevorgänge Zuordnung zu jeder eindeutigen Transposon-Einfügeposition, die wir alle aus Zellen eines einzelnen Transposon-Einfügungmutanten-Klon sstammen. Die Summe aller dieser Werte aus einer einzelnen Bibliothek wird als Gesamtanzahl der zugeordneten Lesevorgänge für diese Bibliothek bezeichnet.
- In Fällen, in denen mehrere Einfügungen innerhalb von 3 Basispaaren voneinander zugeordnet sind, kombinieren Sie sie alle zu einer einzigen Einfügemarke, indem Sie alle Lesevorgänge der einzelnen Position mit der höchsten Leseanzahl zuweisen. Dieser Wert, ninsert, stellt die Häufigkeit des Insertionklons im Zellpellet dar, aus dem gDNA sequenziert wurde. An diesem Punkt wird es Listen von nInsertgeben, jeder die Fülle einer eindeutigen zugeordneten Transposon-Einfügung, eine Liste für jedes Zellpellet sequenziert.
HINWEIS: Das PiggyBac-Transposon fügt in TTAA-Sequenzen im Genom, einer 4-Basis-Paar-Sequenz, ein. Daraus wird daraus geschlossen, dass Einfügungen, die innerhalb von 3 Basispaaren voneinander zugeordnet sind, von derselben TTAA-Site stammen müssen. - Da es eine etwas andere Anzahl von Gesamtlesevorgängen aus jeder sequenzierten Bibliothek gibt, normalisieren Sie dien-Einfügewerte für alle Dateien, wenn sie verglichen werden sollen. Führen Sie dies aus, indem Sie die Gesamtzahl der zugeordneten Lesevorgänge aus jeder einzelnen Bibliothek, nPellet,aufführen, und nehmen Sie den Durchschnitt aller nPellets über alle Bibliotheken hinweg, Pellet>. Multiplizieren Sie jedenn-Einsatz in den Daten einer einzelnen Bibliothek mit dem Verhältnis von Pellet> / nPellet, um einenInsertzu berechnen, die normalisierte Häufigkeit eines gegebenen Transposon-Insertionklons.
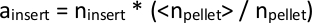
Alternativ kann die Bibliotheksgröße mit verfügbaren Tools wie DESeq28 (Skriptname: total_reads_and_normalize.py) geschätzt werden. - Tabuulieren Sie den Satz aller Einfügungen, die über alle Bibliotheken zugeordnet sind. Legen Sie für Einfügungen, die in einigen Bibliotheken gefunden wurden, aber nicht in anderen, eineEinfügung = 1 für nachgelagerte Berechnungen.
- Filtern Sie die Lesevorgänge, um die Einfügungen zu finden, die gemäß der Anmerkungsdatei innerhalb von Genen liegen (Skriptname: remove_NC_and_plasmid_inserts.py).
- Berechnen Sie für jede einzelne Einfügung die durchschnittliche Häufigkeit über technische Replikationen jeder Auswahl (jede Kultur bei 28 °C oder 39 °C), Einfügen>technical (Skriptname: combine_tech_reps_V2.py).
- Berechnen Sie für jede einzelne Einfügung die durchschnittliche Häufigkeit über biologische Replikationen jeder Temperatur, Einfügen>Gesamt, indem Sie den Mittelwert aller Insert>technical bei jeder Temperatur. Berechnen Sie gleichzeitig den Variationskoeffizienten für jede Einfügung, CV-Einfügung,Summe über die technisch (Skriptname: combine_bio_reps.py).
ANMERKUNG: An dieser Stelle gibt es für jede Temperatur, 28 °C und 39 °C, eine Liste einzigartiger Transposoneinfügungen, ihre durchschnittliche Häufigkeit und den Variationskoeffizienten zwischen biologischen Replikationen für jede. Diese Daten für unser Experiment werden an anderer Stelle gemeldet3. - Filtern Sie die Liste aller Einfügungen für ein einzufügen, das entweder bei 28 °C oder 39 °C, Insert>total > 1.1 und CVinsert,insgesamt 1,5 (Skriptname: filter_inserts.py) enthalten ist.
- Berechnen Sie für jede eindeutige Einfügung das Protokoll2(Insert>total,28 °C / Insert>total,39 °C). Dieser Wert stellt die "Thermotoleranz" eines gegebenen Transposon-Einfügemutantenklons dar (Skriptname: fitness_ratios.py).
- Sortieren Sie alle einzigartigen Einfügungen nach Gen und Allel (S. cerevisiae oder S. paradoxus), und tabellarisch die Anzahl der Einfügungen in jedem Allel. Filtern Sie Gene so, dass nur Gene analysiert werden, die mindestens 5 Einfügungen in jedes Allel haben (Skriptname: organize_and_filter_genes.py).
HINWEIS: Mehrere eindeutige Einfügungen über jedes Allel ermöglichen ein genaueres Maß für die Thermotoleranz dieser wechselseitigen Hemizygote. Die Verringerung der Anzahl der pro Allel erforderlichen Einfügungen ist möglich, beeinträchtigt jedoch die Genauigkeit dieser Maßnahme und erhöht die Mehrfachtestlast, indem mehr Gene getestet werden können. Darüber hinaus wird das Herausfiltern von Genen mit zu wenigen Einfügungen pro Allel dazu beitragen, die Auswirkungen auf die Testergebnisse eines einzelnen Hämizygoten-Klons zu reduzieren, der eine sekundäre Standortmutation enthält, die einen sehr unterschiedlichen Phänotyp verleiht. - Vergleichen Sie für jedes Gen, das nach der obigen Filterung im Datensatz verbleibt, die Thermotoleranzen (Log 2-Verhältnisse) aller Einfügungen im S. cerevisiae-Allel mit denen im S.-Paradoxus-Allel mit einem Mann-Whitney U-Test. Alternativ könnte ein Regressionsmodell implementiert werden, das von DESeq28 (Scriptname: mann_whitney_u.py) angepasst wurde.
- Korrekte p-Werte für mehrfache Tests mit der Benjamini-Hochberg-Methode.
- Gene mit signifikanten p-Werten (z. B. 0,01) sind Kandidaten für Gene, die für Unterschiede in der Thermoverträglichkeit zwischen den beiden Arten wichtig sind.
Ergebnisse
Wir verpaarten S. cerevisiae und S. paradoxus zu einer sterilen Hybride, die wir der Transposon-Mutagenese unterzogen haben. Jeder mutagenisierte Klon war ein Hemizygote, ein diploider Hybrid, bei dem ein Allel eines Gens gestört ist (Abbildung 1A, Abbildung 2). Wir konkurrierten die Hemizygoten gegeneinander durch Wachstum bei 39 °C und, in einem separaten Experiment als Kontrolle, bei 28 °C (Abbildung1...
Diskussion
Die Vorteile von RH-seq gegenüber früheren statistisch-genetischen Methoden sind vielfältig. Im Gegensatz zur Verknüpfungs- und Assoziationsanalyse bietet RH-seq eine Single-Gen-Mapping-Auflösung; als solche wird es wahrscheinlich von erheblichem Nutzen sein, auch in Studien über Merkmalsvariationen zwischen Individuen einer bestimmten Art, sowie interspezifische Unterschiede. Auch frühere Versuche der genomweiten reziproklichen Hemizygositätsanalyse verwendeten Sammlungen von Gen-Deletion-Mutanten, von denen ein...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken J. Roop, R. Hackley, I. Grigoriev, A. Arkin und J. Skerker für ihre Beiträge zur Originalstudie, F. AlZaben, A. Flury, G. Geiselman, J. Hong, J. Kim, M. Maurer und L. Oltrogge für technische Hilfe, D. Savage für seine Großzügigkeit mit Mikroskopie Und B. Blackman, S. Coradetti, A. Flamholz, V. Guacci, D. Koshland, C. Nelson und A. Sasikumar für Gespräche; Wir danken auch J. Dueber (Department of Bioengineering, UC Berkeley) für das PiggyBac Plasmid. Diese Arbeit wurde von R01 GM120430-A1 und vom Community Sequencing Project 1460 an das U.S. Department of Energy Joint Genome Institute, einer DOE Office of Science User Facility, unterstützt. Die von letzterem durchgeführte Arbeit wurde vom Office of Science des US-Energieministeriums unter Vertrag Nr. DE-AC02-05CH11231.
Materialien
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
Referenzen
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten