Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine semi-High-Throughput-Anpassung des NADH-gekoppelten ATPase-Assays zum Screening kleiner Molekülinhibitoren
In diesem Artikel
Zusammenfassung
Ein Nicotinamid-Adenin-Dinukleotid (NADH)-gekoppelter ATPase-Assay wurde an das semihohe Durchsatzscreening von kleinen Molekül-Myosininhibitoren angepasst. Dieser kinetische Assay wird in einem 384-Well-Mikroplattenformat mit einem Gesamtreaktionsvolumen von nur 20 l pro Brunnen ausgeführt. Die Plattform sollte für praktisch jedes ADP-produzierende Enzym gelten.
Zusammenfassung
ATPase-Enzyme nutzen die in Adenosintriphosphat gespeicherte freie Energie, um eine Vielzahl von endergonischen biochemischen Prozessen in vivo zu katalysieren, die nicht spontan auftreten würden. Diese Proteine sind entscheidend für alle Aspekte des Zelllebens, einschließlich Stoffwechsel, Zellteilung, Reaktionen auf Umweltveränderungen und Bewegung. Das hier vorgestellte Protokoll beschreibt einen nicotinamidadenin-Dinukleotid (NADH)-gekoppelten ATPase-Assay, der an das semi-hohe Durchsatzscreening kleiner Molekül-ATPase-Inhibitoren angepasst wurde. Der Assay wurde als Beweis für das Prinzip auf Herz- und Skelettmuskel Myosin II, zwei aktinbasierte molekularmotorische ATPases, angewendet. Die Hydrolyse von ATP wird mit der Oxidation von NADH durch enzymatische Reaktionen im Assay gekoppelt. Zunächst wird die von der ATPase erzeugte ADP durch Pyruvatkinase (PK) zu ATP regeneriert. PK katalysiert den Übergang von Phosphoenolpyruvat (PEP) zu Pyruvat parallel. Anschließend wird Pyruvat durch Laktatdehydrogenase (LDH) zu Laktat reduziert, was die Oxidation von NADH parallel katalysiert. Somit steht die Abnahme der ATP-Konzentration in direktem Zusammenhang mit der Abnahme der NADH-Konzentration, gefolgt von einer Veränderung der intrinsischen Fluoreszenz von NADH. Solange PEP im Reaktionssystem verfügbar ist, bleibt die ADP-Konzentration sehr gering, wodurch eine Hemmung des ATPase-Enzyms durch sein eigenes Produkt vermieden wird. Darüber hinaus bleibt die ATP-Konzentration nahezu konstant, was zu linearen Zeitkursen führt. Die Fluoreszenz wird kontinuierlich überwacht, was eine einfache Abschätzung der Datenqualität ermöglicht und dabei hilft, potenzielle Artefakte herauszufiltern (z. B. durch zusammengesetzte Fällungen oder thermische Veränderungen).
Einleitung
Myosine sind mechanochemische Energiewandler, die Adenosintriphosphat (ATP) hydrolysieren, um richtungsweisende Bewegungen entlang der Filamente des Aktin-Zytoskeletts in Eukaryoten1,2zu erzeugen. Sie haben sich sowohl strukturell als auch kinetisch an ihre verschiedenen intrazellulären Funktionen angepasst, wie z.B. den Transport von Organellen, Muskelkontraktion oder die Erzeugung von Zytoskelettspannung1,2. Die Myosin-Überfamilie wird durch 40 Myosin-Gene repräsentiert, die zu 12 verschiedenen Myosin-Klassen im menschlichen Genom3,4gehören. Mitglieder der Myosin-Klassen spielen verschiedene Rollen in einer sehr unterschiedlichen Reihe von Erkrankungen, wie mehrere Krebsarten, neurologische Störungen, Skelettmyopathien, und hypertrophe Kardiomyopathie5,6. Angesichts der großen Anzahl von physiologischen und pathologischen Funktionen dieser molekularen Motoren, ist es nicht verwunderlich, dass sie zunehmend als Wirkstoffziele für eine Vielzahl von Bedingungen erkannt werden7. Erhebliche Fortschritte wurden in letzter Zeit bei der Entdeckung neuer Myosininhibitoren8,9,10 und Aktivatoren11, und zur Verbesserung der Eigenschaften der bestehenden12 , 13 , 14 , 15.
Der nicotinamidadenin-Dinukleotid (NADH)-gekoppelte ATPase-Assay wird seit langem verwendet, um die ATPase-Aktivität verschiedener Enzyme zu messen, wie z.B. die sarkoplasmische Retikulum-Ca 2+-Pumpe ATPase16, die DNA-Reparatur ATPase Rad5417, die AAA+ ATPase p9718 oder der Mikrotubuli-Motor Kinesin19. Der Test verwendet einen ATP-Regenerationszyklus. Das von der ATPase erzeugte Adenosindiphosphat (ADP) wird durch Pyruvatkinase (PK) zu ATP regeneriert, wodurch ein Molekül Phosphoenolpyruvat (PEP) parallel in Pyruvat umwandelt. Anschließend wird Pyruvat durch Laktatdehydrogenase (LDH) zu Laktat reduziert. Das wiederum oxidiert ein Molekül von NADH zu NAD. Daher entspricht die Abnahme der NADH-Konzentration in Abhängigkeit von der Zeit der ATP-Hydrolyserate. Der ATP-Regenerationszyklus hält die ATP-Konzentration nahezu konstant und die ADP-Konzentration niedrig, solange PEP verfügbar ist. Dies führt zu linearen Zeitkursen, was es einfach macht, die anfänglichen Reaktionsraten zu bestimmen und hilft, Produkthemmung durch ADP19zu vermeiden. Obwohl der NADH-gekoppelte ATPase-Assay bereits an ein 96-Well-Format20angepasst wurde, machen die hohen Reaktionsvolumina (ca. 150 l) es aufgrund der hohen Nachfrage nach Reagenzien relativ teuer, so dass er weniger anfällig für eine schnelle Abschirmung großer Verbindungen. Alternative Methoden wie der Malachit-Grüntest19,21, der auf dem Nachweis des vom ATPase-Enzym produzierten Phosphats beruht, erwiesen sich als besser geeignet für die Miniaturisierung und das Hochdurchsatzscreening22 , 23 , 24. Ein Endpunkttest wird jedoch eher von mehreren Artefakten beeinflusst (siehe unten), die ohne Vollzeitkurse unentdeckt bleiben können.
Hier wurde der NADH-gekoppelte ATPase-Assay für das semi-hohe Durchsatzscreening kleiner Molekülinhibitoren optimiert. Skelett- und Herzmuskelmyosin II und die Myosininhibitoren Blebbistatin8, Para-Aminoblebbistatin13 und Para-Nitroblebbistatin12 werden verwendet, um die Kraft des Assays zu demonstrieren, der auf NADH beruht Fluoreszenz als Auslesung. Dieses Protokoll ist für Screening-Projekte zugänglich, die sich auf alle ADP-produzierenden Enzyme konzentrieren.
Protokoll
1. Vorbereitung von Lagerlösungen und Reagenzien
- Bereiten Sie die Lagerlösung Dithiothreitol (DTT) vor, indem Sie kristallines DTT in destilliertem Wasser auf eine Endkonzentration von 1000 mM auflösen. Stellen Sie den pH-Wert auf 7,0 mit 1 M NaOH-Lösung ein. Aliquot und lagern bei -20 °C.
- Bereiten Sie die ATP-Lagerlösung vor, indem Sie kristallines ATP in destilliertem Wasser auf eine Endkonzentration von 100 mM auflösen. Stellen Sie den pH-Wert auf 7,0 mit 1 M NaOH-Lösung ein. Aliquot und lagern bei -20 °C.
- 10x NADH-Puffer mit 70 mM 3-(N-Morpholino)propanesulfonsäure (MOPS), 10 mM MgCl2, 0,9 mM Ethylenglykol-Bis(-Aminoethylether)-N,N,N,N-Tetraessigsäure (EGTA) und 3 mM NaN3vorbereiten. Stellen Sie den pH-Wert auf 7,0 mit 1 M NaOH-Lösung ein. Bei 4 °C lagern.
- Bereiten Sie 1x Myosinpuffer mit 10 mM MOPS und 0,1 mMEGTA vor. Stellen Sie den pH-Wert auf 7,0 mit 1 M NaOH-Lösung ein. Bei 4 °C lagern. Rinderserumalbumin (BSA) und DTT zu einer Endkonzentration von 0,1% (w/v%) hinzufügen bzw. 1 mM vor Der Anwendung.
- Bereiten Sie 1x Aktinpuffer mit 4 mM MOPS, 0,1 mM EGTA, 2 mM MgCl2und 3 mM NaN3vor. Stellen Sie den pH-Wert auf 7,0 mit 1 M NaOH-Lösung ein. Bei 4 °C lagern. BSA und DTT zu einer Endkonzentration von 0,1% hinzufügen (w/v%) bzw. 1 mM vor Der Anwendung.
- Bereiten Sie die NADH-Lagerlösung vor, indem Sie kristalline NADH in 10x NADH-Puffer auf eine Endkonzentration von 5,5 mM auflösen. Aliquot und lagern bei -20 °C.
- Bereiten Sie die PEP-Lagerlösung vor, indem Sie kristallines PEP in 10x NADH-Puffer auf eine Endkonzentration von 50 mM auflösen. Aliquot und lagern bei -20 °C.
- Herstellung der LDH-Stammlösung durch Auflösen von lyophilisiertem LDH-Pulver in einer Mischung aus Glycerin und 10x NADH-Puffer (50%:50%) einer Endkonzentration von 2000 U/ml. Zentrifugieren Sie die Lösung, um das vorhandene ungelöste Protein zu entfernen (7.197 x g, 20 °C, 10 min). Übertragen Sie den Überstand vorsichtig in ein sauberes Zentrifugenrohr. Aliquot und lagern bei -20 °C.
- Bereiten Sie PK-Stammlösung durch Auflösen von lyophilisiertem PK-Pulver in einer Mischung aus Glycerin und 10x NADH-Puffer (50%:50%) einer Endkonzentration von 10000 U/ml. Zentrifugieren Sie die Lösung, um das vorhandene ungelöste Protein zu entfernen (7.197 x g, 20 °C, 10 min). Übertragen Sie den Überstand vorsichtig in ein sauberes Zentrifugenrohr. Aliquot und lagern bei -20 °C.
- Rekonstituieren Sie die lyophilisierten Herz- und Skelettmuskelmyosin-II-Proben, indem Sie 100 l destilliertes Wasser hinzufügen, um 10 mg/ml-Stammlösungen zu erhalten, die 37,9 M bzw. 40,8 M Myosinkonzentrationen (monomer) entsprechen. Weitere Informationen finden Sie in den Anweisungen des Herstellers.
- Bereiten Sie F-Actin aus Kaninchenmuskel AcetonPulver, wie von Pardee und Spudich25beschrieben.
2. Messung von ATPase-Aktivitäten und hemmenden Wirkungen von kleinen Molekülinhibitoren
- Bereiten Sie zusammengesetzte Platte.
- Lösen Sie Verbindungen von Interesse in hochwertigem Dimethylsulfoxid (DMSO).
- Erstellen Sie fünfzehnstufige serielle 1:2 Verdünnungen ab 10 mM Compoundkonzentration in DMSO.
- Übertragen Sie die Proben mit einer Mehrkanalpipette auf eine 384-Well-Polypropylenplatte in Dreisprachigen (je 12,5 l). Verwenden Sie zwei Zeilen auf der Verbundplatte für eine Verbindung (anstelle von drei Spalten), um die Anzahl der Brunnen zu minimieren, die potenziell von Kanteneffekten betroffen sind. Verwenden Sie die letzten drei Bohrungen in der zweiten Reihe für jede Verbindung als Negativkontrolle (nur DMSO). Verwenden Sie nicht die erste und die letzte Reihe auf der Platte für zusammengesetzte Verdünnungen.
- Übertragen Sie reines DMSO in die Brunnen der ersten Reihe (reserviert für die NADH-Kalibrierung).
- Verwenden Sie die letzte Zeile für die positivsteuernde Steuerung.
HINWEIS: Para-Aminoblebbistatin bei 4 mM Konzentration in DMSO wurde hier verwendet.
- Bereiten Sie für jede Assayplatte (384-well Black-Wall-Polystyrol-Mikroplatte) 4500 l von 20 M verdünnte Aktinlösung vor, indem Sie die Actin-Lagerlösung im Aktinpuffer verdünnen. Mischen Sie die Lösung gründlich, indem Sie 30x mit einer 5 ml Pipette nach oben und unten pipetieren, um Viskosität und Heterogenität durch Brechen von Aktinfilamenten zu reduzieren. Zentrifugieren Sie die Lösung, um vorhandene gefällte Proteine zu entfernen (7.197 x g, 20 °C, 10 min). Den Überstand vorsichtig in ein sauberes Zentrifugenrohr geben.
- Bereiten Sie Master-Mix mit LDH- und PK-Enzymen ("Enzymmix") vor. Kombinieren Sie für jede Testplatte 171,4 l LDH-Lösung, 171,4 l PK-Lösung und 3189,3 l oder 3252,9 l Myosinpuffer für Assays mit Herz- oder Skelettmuskel-Myosin-II-Lösung in einem 15 ml konischen Zentrifugenrohr. Fügen Sie an dieser Stelle kein Myosin hinzu, um Aggregation und Niederschlag zu vermeiden.
- Bereiten Sie master mix mit allen Substraten ("Substratmischung"). Kombinieren Sie für jede Platte 162,1 l ATP, 162,1 l PEP und 324,1 l NADH-Lösung in einem 15 ml konischen Zentrifugenrohr. Fügen Sie an dieser Stelle kein Actin hinzu, um Aggregation und Niederschlag zu vermeiden.
- Erstellen Sie siebenstufige serielle 1:2 Verdünnungen von NADH für die Kalibrierung ab 250 M.
- Mischen Sie 12,3 l NADH-Stammlösung mit 257,7 l Myosinpuffer in einem 1,5 ml Mikrozentrifugenrohr.
- Aliquot 135 l Myosinpuffer in sieben 1,5 ml Mikrozentrifugenröhren.
- 135 L Lösung aus dem ersten Rohr in das zweite geben und durch Pipettieren mischen. Wiederholen Sie dies, bis sie das7. Rohr erreicht.
- Verwenden Sie die letzte Röhre als No-NADH-Steuerelement (nur Puffer).
- Übertragen Sie mit einer 8-Kanal-Pipette 20 l der NADH-Kalibrierlösungen in die erste Reihe der Assayplatte in Triplicaten.
- Fügen Sie dem Enzymmix 68 l Kardial oder 4,2 l SkelettmuskelMyosin II hinzu. Wirbel kurz.
- Mit Ausnahme der ersten Reihe 8,4 l des vorbereiteten Myosin-Enzym-Mixes in jeden Brunnen der Assayplatte mit einem automatisierten Spender.
- Übertragen Sie 100 nL Lösungen von der Verbundplatte auf die Assayplatte, die Enzymmischung enthält, mit einem automatisierten Flüssigkeitshandhabungssystem, das mit einem 100 nL-Stiftwerkzeugkopf ausgestattet ist.
- Schütteln Sie die Assayplatte 1 min bei Raumtemperatur bei 1200 Umdrehungen pro Minute mit einem Mikroplatten-Shaker.
- Fügen Sie 4.052 l der zentrifugierten Aktinlösung in die Substratmischung ein. Wirbel kurz.
- Geben Sie 11,6 l Actin-Substrat-Mix in jede Bohrung der Assayplatte (außer der ersten Reihe) ein, um die enzymatische Reaktion mit einem automatisierten Spender zu starten.
- Schütteln Sie die Assayplatte 1 min bei Raumtemperatur bei 1200 Umdrehungen pro Minute mit einem Mikroplatten-Shaker.
- Zentrifugieren Sie die Assayplatte bei 101 x g für 30 s.
- Stellen Sie sicher, dass die Innentemperatur des Plattenlesers bei 25 °C stabilisiert wurde. Laden Sie die Platte und schütteln Sie für weitere 30 s. Dieser Schüttelschritt ist notwendig, um die Form der flüssigen Oberfläche in jedem Brunnen ähnlich zu machen und lässt der Platte Zeit, um die Messtemperatur zu erreichen.
- Nehmen Sie die NADH-Fluoreszenz für 30 min auf, die Platte in 45 s Intervallen zu scannen. Verwenden Sie einen 380 nm, 10 nm Bandbreiten-Erregungsfilter und einen 470 nm, 24 nm Bandbreiten-Emissionsfilter in Verbindung mit einem 425 nm abgesenkten dichroitischen Spiegel. Führen Sie die Messung im Hochkonzentrationsmodus aus. Optimieren Sie die Anzahl der Blitze, die Detektorverstärkung, die Plattenabmessungen und die Messhöhe, bevor Sie die Tests ausführen.
HINWEIS: Die Endaufnahmebedingungen sind 300 nM Herz/20 nM Skelettmuskel Myosin II, 10 M Actin, 40 U/mL LDH, 200 U/mL PK, 220 M NADH, 1 mM PEP, 1 mM ATP in einem Puffer mit 10 mM MOPS (pH = 7,0), 2 mM MgCl2 0,15 mM EGTA, 0,1 mg/ml BSA, 0,5% (v/v) DMSO und 1 mM DTT. Das Gesamtvolumen beträgt 20 L/Well. Die höchste Endkonzentrationskonzentration liegt bei 50 M. 20 M Para-Aminoblebbistatin in 0,5% DMSO dient als Positivkontrolle und 0,5% DMSO allein ist die negative Kontrolle. Alle Messungen werden in Dreiern durchgeführt.
3. Analysieren von Daten
- Plotten Sie die beobachtete Fluoreszenzintensität gegen die Zeit für jeden Brunnen.
- Führen Sie eine einfache lineare Regression durch, um die Neigung und das Abfangen der Fluoreszenzantworten für jeden Brunnen zu bestimmen. Die Steigung ist proportional zur ATP-Verbrauchsrate (NADH), während der Abfangproportional zur NADH-Konzentration zu Beginn der Messung ist (t = 0 s).
- Konstruieren Sie eine Kalibrierkurve für NADH, indem Sie die für die erste Reihe der Platte erhaltenen Abschnitte gegen die Konzentration von NADH zeichnen. Stellen Sie sicher, dass die Abfangabschnitte linear von der NADH-Konzentration abhängen.
ANMERKUNG: Die Intercepts schätzen die realen Fluoreszenzintensitäten auf t = 0 s mit viel mehr Vertrauen als der Durchschnitt der Rohfluoreszenzintensität liest bei t 0 s. - Führen Sie eine einfache lineare Regression durch, um die Steigung und den Abfang der NADH-Kalibrierungslinie zu erhalten.
ANMERKUNG: Der Intercept beschreibt das Fluoreszenz-Hintergrundsignal (kein NADH vorhanden), während die Steigung der extrapolierten/theoretischen Fluoreszenzintensität einer 1 M NADH-Lösung in diesem speziellen Experiment entspricht. - Teilen Sie die Steigung der Fluoreszenzantwort, die für den Rest der Brunnen erhalten wird, durch die Steigung der NADH-Kalibrierlinie, um Fluoreszenzänderungen in ATP-Verbrauchsraten umzuwandeln.
- Zeichnen Sie die ATP-Verbrauchsraten gegen die Konzentration des Inhibitors.
- Um hemmende Konstanten zu bestimmen, verwenden Sie geeignete statistische Software, um die Dosis-Wirkungs-Daten an die folgende quadratische Gleichung anzupassen, die einem einfachen Eins-zu-Eins-Bindungsgleichgewichtsmodell entspricht:
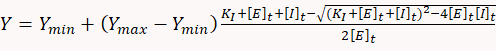
wobei Y die ATP-Verbrauchsrate ist, Ymin die ATP-Verbrauchsrate int das Fehlen eines Inhibitors ist, Ymax ist die theoretische ATP-Verbrauchsrate bei 100% Hemmung, KI ist die hemmende Konstante , [E]t und [I]t sind die Gesamtkonzentration des Enzyms (Myosin) bzw. des Inhibitors.
Ergebnisse
Die typische Plattenlayoutkarte, die für Screening-Experimente verwendet wird, ist in Abbildung 1dargestellt. Die erste und letzte Zeile sind für die NADH-Kalibrierung und Positivkontrolle reserviert (20 M Para-Aminoblebbistatin, 0,5% DMSO). Die übrigen Reihen (B bis O) werden verwendet, um die hemmende Aktivität von Verbindungen zu testen. Hierbei werden fünfzehnstufige serielle 1:2 Verdünnungen ab 10 mM Compoundkonzentration in DMSO vorberei...
Diskussion
Kritische Schritte im Protokoll
Optimieren Sie das Plattenlayout, indem Sie mehrere Platten nur mit Negativkontrolle betreiben (ATPase-Reaktion ohne Inhibitor). Prüfen Sie die Ergebnisse sorgfältig auf Muster in Reaktionsgeschwindigkeiten. Diese können z. B. durch Kanteneffekte und/oder Unvollkommenheiten in der hydrophilen Oberflächenbeschichtung von "unverbindlichen" Platten entstehen. Wenn ein Muster beobachtet wird, ändern Sie den Plattentyp und/oder das Plattenlayout...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch ein Stipendium des National Institute of Neurological Disorders and Stroke und des National Institute on Drug Abuse NS096833 (CAM) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 384-well Low Flange Black Flat Bottom Polystyrene NBS Microplate | Corning | 3575 | |
| ATP (Adenosine 5′-triphosphate disodium salt hydrate) | Sigma | A7699 | |
| Aurora FRD-IB Dispenser | Aurora Discovery, Inc. | 00017425 | |
| Biomek NXP Multichannel Laboratory Automation Workstation | Beckman Coulter | A31841 | |
| Blebbistatin | AMRI | N/A | Custom synthesis |
| BSA (Bovine Serum Albumin, Protease-Free) | Akron Biotech | AK1391 | |
| Centrifuge 5430 R, refrigerated, with Rotor FA-35-6-30 | Eppendorf | 022620663 | |
| Centrifuge 5430, non-refrigerated, with Rotor A-2-MTP | Eppendorf | 022620568 | |
| DMSO (Dimethyl sulfoxide) | Sigma | D2650 | |
| DTT (DL-Dithiothreitol) | Sigma | D5545 | |
| E1 ClipTip Multichannel Pipette; 384-format; 8-channel | Thermo Scientific | 4672010 | |
| E1 ClipTip Multichannel Pipette; 96-format; 8-channel | Thermo Scientific | 4672080 | |
| EGTA (Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid) | Sigma | E3889 | |
| EnVision 2104 Multilabel Plate Reader | PerkinElmer | 2104-0010 | |
| Glycerol | Sigma | G2025 | |
| LDH (L-Lactic Dehydrogenase from rabbit muscle) | Sigma | L1254 | |
| MgCl2.6H2O (Magnesium chloride hexahydrate) | Sigma | M2670 | |
| Microplate Shaker | VWR | 12620-926 | |
| Microplate, 384 well, PP, Small Volume, Deep Well, Natural | Greiner Bio-One | 784201 | |
| MOPS (3-(N-Morpholino)propanesulfonic acid) | Sigma | M1254 | |
| Myosin Motor Protein (full length) (Bovine cardiac muscle) | Cytoskeleton | MY03 | |
| Myosin Motor Protein (full length) (Rabbit skeletal muscle) | Cytoskeleton | MY02 | |
| NADH (β-Nicotinamide adenine dinucleotide, reduced disodium salt hydrate) | Sigma | N8129 | |
| NaN3 (Sodium azide) | Sigma | 71289 | |
| NaOH (Sodium hydroxide) | Sigma | S8045 | |
| Optical Filter CFP 470/24nm (Emission) | PerkinElmer | 2100-5850 | Barcode 240 |
| Optical Filter Fura2 380/10nm (Excitation) | PerkinElmer | 2100-5390 | Barcode 112 |
| Optical Module: Beta Lactamase | PerkinElmer | 2100-4270 | Barcode 418 |
| OriginPro 2017 software | OriginLab | N/A | |
| para-Aminoblebbistatin | AMRI | N/A | Custom synthesis |
| para-Nitroblebbistatin | AMRI | N/A | Custom synthesis |
| PEP (Phospho(enol)pyruvic acid monopotassium salt) | Sigma | P7127 | |
| PK (Pyruvate Kinase from rabbit muscle) | Sigma | P9136 | |
| Rabbit Muscle Acetone Powder | Pel Freez Biologicals | 41995-2 |
Referenzen
- Heissler, S. M., Sellers, J. R. Kinetic Adaptations of Myosins for Their Diverse Cellular Functions. Traffic. 17 (8), 839-859 (2016).
- Hartman, M. A., Spudich, J. A. The myosin superfamily at a glance. Journal of Cell Science. 125 (Pt 7), 1627-1632 (2012).
- Berg, J. S., Powell, B. C., Cheney, R. E. A millennial myosin census. Molecular Biology of the Cell. 12 (4), 780-794 (2001).
- Sebe-Pedros, A., Grau-Bove, X., Richards, T. A., Ruiz-Trillo, I. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution. 6 (2), 290-305 (2014).
- Newell-Litwa, K. A., Horwitz, R., Lamers, M. L. Non-muscle myosin II in disease: mechanisms and therapeutic opportunities. Disease Models & Mechanisms. 8 (12), 1495-1515 (2015).
- He, Y. M., Gu, M. M. Research progress of myosin heavy chain genes in human genetic diseases. Yi Chuan. 39 (10), 877-887 (2017).
- Rauscher, A. A., Gyimesi, M., Kovacs, M., Malnasi-Csizmadia, A. Targeting Myosin by Blebbistatin Derivatives: Optimization and Pharmacological Potential. Trends in Biochemical Sciences. 43 (9), 700-713 (2018).
- Straight, A. F., et al. Dissecting temporal and spatial control of cytokinesis with a myosin II Inhibitor. Science. 299 (5613), 1743-1747 (2003).
- Sirigu, S., et al. Highly selective inhibition of myosin motors provides the basis of potential therapeutic application. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), E7448-E7455 (2016).
- Green, E. M., et al. A small-molecule inhibitor of sarcomere contractility suppresses hypertrophic cardiomyopathy in mice. Science. 351 (6273), 617-621 (2016).
- Morgan, B. P., et al. Discovery of omecamtiv mecarbil the first, selective, small molecule activator of cardiac Myosin. ACS Medicinal Chemistry Letters. 1 (9), 472-477 (2010).
- Kepiro, M., et al. para-Nitroblebbistatin, the non-cytotoxic and photostable myosin II inhibitor. Angewandte Chemie International Edition. 53 (31), 8211-8215 (2014).
- Varkuti, B. H., et al. A highly soluble, non-phototoxic, non-fluorescent blebbistatin derivative. Scientific Reports. 6, 26141 (2016).
- Verhasselt, S., et al. Discovery of (S)-3'-hydroxyblebbistatin and (S)-3'-aminoblebbistatin: polar myosin II inhibitors with superior research tool properties. Organic and Biomolecular Chemistry. 15 (9), 2104-2118 (2017).
- Verhasselt, S., Roman, B. I., Bracke, M. E., Stevens, C. V. Improved synthesis and comparative analysis of the tool properties of new and existing D-ring modified (S)-blebbistatin analogs. European Journal of Medicinal Chemistry. 136, 85-103 (2017).
- Warren, G. B., Toon, P. A., Birdsall, N. J., Lee, A. G., Metcalfe, J. C. Reconstitution of a calcium pump using defined membrane components. Proceedings of the National Academy of Sciences of the United States of America. 71 (3), 622-626 (1974).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. Rad54 protein exerts diverse modes of ATPase activity on duplex DNA partially and fully covered with Rad51 protein. Journal of Biological Chemistry. 277 (48), 46205-46215 (2002).
- Hanzelmann, P., Schindelin, H. Structural Basis of ATP Hydrolysis and Intersubunit Signaling in the AAA+ ATPase p97. Structure. 24 (1), 127-139 (2016).
- Hackney, D. D., Jiang, W. Assays for kinesin microtubule-stimulated ATPase activity. Methods in Molecular Biology. 164, 65-71 (2001).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. NADH-coupled microplate photometric assay for kinetic studies of ATP-hydrolyzing enzymes with low and high specific activities. Analytical Biochemistry. 321 (2), 266-271 (2003).
- Carter, S. G., Karl, D. W. Inorganic phosphate assay with malachite green: an improvement and evaluation. Journal of Biochemical and Biophysical Methods. 7 (1), 7-13 (1982).
- Henkel, R. D., VandeBerg, J. L., Walsh, R. A. A microassay for ATPase. Analytical Biochemistry. 169 (2), 312-318 (1988).
- Rowlands, M. G., et al. High-throughput screening assay for inhibitors of heat-shock protein 90 ATPase activity. Analytical Biochemistry. 327 (2), 176-183 (2004).
- Rule, C. S., Patrick, M., Sandkvist, M. Measuring In Vitro ATPase Activity for Enzymatic Characterization. Journal of Visualized Experiments. (114), 54305 (2016).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Cell Biology. 24, 271-289 (1982).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Kovacs, M., Toth, J., Hetenyi, C., Malnasi-Csizmadia, A., Sellers, J. R. Mechanism of blebbistatin inhibition of myosin II. Chem Journal of Biological Chemistry. 279 (34), 35557-35563 (2004).
- Allingham, J. S., Smith, R., Rayment, I. The structural basis of blebbistatin inhibition and specificity for myosin II. Nature Structural & Molecular Biology. 12 (4), 378-379 (2005).
- Kettlun, A. M., et al. Purification and Characterization of 2 Isoapyrases from Solanum-Tuberosum Var Ultimus. Phytochemistry. 31 (11), 3691-3696 (1992).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. British Journal of Pharmacology. 161 (6), 1219-1237 (2010).
- Motulsky, H. J., Neubig, R. R. Analyzing binding data. Current Protocols in Neuroscience. 52 (1), 7.5.1-7.5.65 (2010).
- Sehgal, P., Olesen, C., Moller, J. V. ATPase Activity Measurements by an Enzyme-Coupled Spectrophotometric Assay. Methods in Molecular Biology. 1377, 105-109 (2016).
- Solinger, J. A., Lutz, G., Sugiyama, T., Kowalczykowski, S. C., Heyer, W. D. Rad54 protein stimulates heteroduplex DNA formation in the synaptic phase of DNA strand exchange via specific interactions with the presynaptic Rad51 nucleoprotein filament. Journal of Molecular Biology. 307 (5), 1207-1221 (2001).
- Banik, U., Roy, S. A continuous fluorimetric assay for ATPase activity. Biochemistry Journal. 266 (2), 611-614 (1990).
- Xiao, Y. X., Yang, W. X. KIFC1: a promising chemotherapy target for cancer treatment?. Oncotarget. 7 (30), 48656-48670 (2016).
- See, S. K., et al. Cytoplasmic Dynein Antagonists with Improved Potency and Isoform Selectivity. ACS Chemical Biology. 11 (1), 53-60 (2016).
- Datta, A., Brosh, R. M. New Insights Into DNA Helicases as Druggable Targets for Cancer Therapy. Frontiers in Molecular Biosciences. 5, 59 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten