Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
A High-Throughput In Situ Method for Estimation of Hepatocyte Nuclear Ploidy in Mice
In diesem Artikel
Zusammenfassung
Wir präsentieren eine robuste, kostengünstige und flexible Methode zur Messung von Veränderungen der Hepatozytenzahl und der Kernploidie in festen/kryokonservierten Gewebeproben, die keine Durchflusszytometrie erfordern. Unser Ansatz bietet eine leistungsstarke probenweite Signatur der Leberzytologie, ideal für die Verfolgung des Fortschreitens von Leberverletzungen und Krankheiten.
Zusammenfassung
Wenn die Leber verletzt ist, hepatozyten Zahlen abnehmen, während Zellgröße, Kerngröße und Ploidy zu erhöhen. Die Ausdehnung von nicht-parenchymalen Zellen wie Cholangiozyten, Myofibroblasten, Vorläufern und Entzündungszellen weist auch auf chronische Leberschäden, Gewebeumbau und Krankheitsprogression hin. In diesem Protokoll beschreiben wir einen einfachen Ansatz mit hohem Durchsatz zur Berechnung von Veränderungen in der zellulären Zusammensetzung der Leber, die mit Verletzungen, chronischen Krankheiten und Krebs verbunden sind. Wir zeigen, wie Informationen aus zweidimensionalen (2D) Gewebeabschnitten verwendet werden können, um hepatozyte Kernploidie innerhalb einer Probe zu quantifizieren und zu kalibrieren und es dem Benutzer zu ermöglichen, bestimmte Ploidie-Teilmengen innerhalb der Leber in situ zu lokalisieren. Unsere Methode erfordert Zugang zu festem/gefrorenem Lebermaterial, grundlegenden immunzytochemischen Reagenzien und jeder Standardplattform für bildende Bildgebungsmittel mit hohem Inhalt. Es dient als leistungsstarke Alternative zu Standard-Flow-Zytometrie-Techniken, die Eine Unterbrechung des frisch gesammelten Gewebes, Verlust räumlicher Informationen und potenzielle Disaggregationsverzerrung erfordern.
Einleitung
Hepatozyten in der Säugetierleber können einer stockenden Zytokinese unterzogen werden, um binukleare Zellen zu produzieren, und DNA-Endoreplikation, um polyploide Kerne zu produzieren, die bis zu 16N DNA-Gehalt enthalten. Insgesamt zelluläre und nukleare Ploidie erhöhen während der postnatalen Entwicklung, Alterung und als Reaktion auf verschiedene zelluläre Belastungen1. Der Prozess der Polyploidisierung ist dynamisch und reversibel2, obwohl seine genaue biologische Funktion unklar bleibt3. Erhöhte Ploidie ist verbunden mit reduzierter Proliferativfähigkeit4, genetische Vielfalt2, Anpassung an chronische Verletzungen5 und Krebsschutz6. Hepatozytenploidie Veränderungen treten als Folge von veränderten zirkadianen Rhythmus7, und Entwöhnung8. Vor allem wird das Ploidy-Profil der Leber durch Verletzung und Krankheitverändert 9, und überzeugende Beweise deuten darauf hin, dass spezifische Ploidy-Änderungen, wie erhöhte 8N-Kerne oder Verlust von 2N Hepatozyten, nützliche Signaturen für die Verfolgung der nicht-alkoholischen Fettleber -Progression (NAFLD) Progression3,10oder die differentialen Auswirkungen von Virusinfektionen11.
Im Allgemeinen sind Leberverletzung und Regeneration mit erhöhter Hepatozytenzellgröße und Kernfläche12verbunden, zusammen mit einer reduzierten Gesamtzahl von Hepatozyten, insbesondere solchen mit 2N-DNA-Gehalt10,11. Parenchymale Verletzungen in der Leber gehen häufig auch mit einer Ausdehnung nicht-parenchymaler Zellen (NPCs) einher, einschließlich stromaler Myofibroblasten, Entzünderzellen und bipotenten Lebervorläuferzellen. Hochdurchsatzmethoden, die ein quantitatives zytologisches Profil der parenchymalen Zellzahl und der Kernploidie liefern und gleichzeitig Veränderungen der NPCs berücksichtigen, haben daher ein beträchtliches Potenzial als Forschungs- und klinische Instrumente, um das Reaktionsverhalten der Leber während von Verletzungen und Krankheiten zu verfolgen. Überzeugende jüngste In-situ-Analysen von Ploidy-Spektren in menschlichen Proben des hepatozellulären Karzinoms zeigen auch, dass die Kernploidie innerhalb von Tumoren dramatisch erhöht ist und speziell in aggressiveren Tumorsubtypen mit reduzierter Differenzierung und Verlust von TP5313verstärkt wird. Daher besteht die große Möglichkeit, dass methodische Fortschritte bei der quantitativen Bewertung der Kernploidie bei der zukünftigen prognostischen Profilierung von Leberkrebs helfen werden.
In diesem Protokoll wird eine flexible Hochdurchsatzmethodik für die vergleichende Analyse von Lebergewebeabschnitten von Mäusen beschrieben, die eine detaillierte zytometrische Profilierung von Hepatozytenzahlen, die NPC-Antwort und eine intern kalibrierte Methode zur Schätzung der Kernploidie bietet (Abbildung 1). Hepatozyten unterscheiden sich von NPCs durch Hepatozyten-Atomfaktor 4 alpha (HNF4) Immunlabelling, vor der Charakterisierung der nuklearen Größe und der nuklearen Morphometrie. Der "Minimale DNA-Gehalt" wird für alle kreisförmigen Kernmasken geschätzt, indem die mittlere Hoechst 33342-Intensität (ein Proxy für DNA-Dichte) mit interpoliertem dreidimensionalem (3D) Kernvolumen integriert wird. Hepatozyten minimaler DNA-Gehalt wird dann mit NPCs kalibriert, um ein nukleares Ploidy-Profil zu erzeugen.
Die Bildaufnahme, die nukleare Segmentierung und die Bildanalyse werden mittels hochauflösender Bildgebung durchgeführt, sodass große Bereiche zweidimensionaler (2D) Leberabschnitte mit Zehntausenden von Zellen gescreent werden können. Für die automatisierte Nachbearbeitung von Bildanalysedaten mit hohem Inhalt wird ein kundenspezifisches Programm bereitgestellt, um ein stichprobenweites Ploidy-Profil für alle kreisförmigen Hepatozytenkerne zu erstellen. Dies wird mit kostenlosen Software zum Herunterladen von Software durchgeführt, um kerntechnische Ploidie basierend auf stereologischer Bildanalyse (SIA)10,11,14,15zu berechnen. Die SIA-Methodik wurde zuvor durch Diedurchflusszytometrie als eine genaue, wenn auch mühsame Methode zur Schätzung der Hepatozyten-Kernploidie in der Leber14validiert, wobei die zirkuläre Kernmorphologie und eine monotone Beziehung zwischen nuklearer Größe und DNA-Gehalt vorausgesetzt werden. In diesem Protokoll werden beide nuklearen Parameter durch Diebewertung der nuklearen Morphometrie und der Hoechst 33342-Kennzeichnung gemessen. Auf die Berechnung des "minimalen DNA-Gehalts" für jede Kernmaske folgt die Kalibrierung der Hepatozyten-Kernploidie mit NPCs, die einen bekannten 2-4N-DNA-Gehalt aufweisen und daher als nützliche interne Kontrolle dienen.
Im Vergleich zu herkömmlichen Durchflusszytometriemethoden16 ermöglicht der beschriebene Ansatz die Bewertung der Hepatozyten-Kernploidie vor Ort und erfordert keinen Zugang zu frischem Gewebe oder Disaggregationsmethoden, die die Ergebnisse verzerrt und schwer zu standardisieren sind. Wie bei allen SIA-basierten Ansätzen sind die Kernploidie-Unterklassen >2N durch 2D-Probenahme aufgrund der Schnittung größerer Kerne außerhalb der äquatorialen Ebene unterrepräsentiert. Das gewebeweite Ploidy-Profil beschreibt auch den minimalen DNA-Gehalt für alle kreisförmigen Hepatozyten-Kernmasken und unterscheidet nicht direkt zwischen mononukleären Hepatozyten und binukleären Zellen, die zwei diskrete ("nicht berührende") Kerne derselben Ploidie haben. Die Einfachheit dieses Protokolls lässt jedoch einen beträchtlichen Spielraum für eine Anpassung an zusätzliche Parameter wie den internuklearen Abstand oder die Zellperimeteranalyse, die die Identifizierung binuklearer Zellen erleichtern würde, was eine detailliertere Bewertung der zellulären Ploidie ermöglichen würde.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Tierversuche wurden zuvor von der CIPF-Ethikkommission genehmigt. Mäuse wurden in einer pathogenfreien Anlage im Centro de Investigacion Principe Felipe (Valencia, Spanien) untergebracht, registriert als Versuchstierzüchter, Anwender und Versorgungszentrum (reg. ES 46 250 0001 002) nach den geltenden europäischen und spanischen Tierschutzvorschriften (FU 53/2013).
1. Gewebeernte und Probenvorbereitung
HINWEIS: Dieses Protokoll beschreibt, wie Gewebe ohne vorherige Fixierung oder Kryokonservierung eingefroren wird. Für zuvor fixierte/kryokonservierte Proben fahren Sie mit Abschnitt 2 fort und lassen Sie Schritt 3.1 weg. Alle Analysen wurden mit erwachsenen weiblichen C57BL/6-Mäusen im Alter von 12 bis 16 Wochen durchgeführt.
- Opfern Sie Tiere durch Fentanyl/Pentobarbital intraperitoneale Injektion gefolgt von zervikaler Dislokation. Mit der Maus nach oben gerichtet, öffnen Sie die Bauchhöhle und belichten die Leber, indem Sie die Haut mit einer Pinzette greifen und einen vertikalen Schnitt von der Basis des Unterbauchs bis zur Basis des Brustbeins mit einer chirurgischen Schere durchführen.
- Entfernen Sie vorsichtig die Gallenblase mit einer feinen Pinzette, sezieren Sie die Leber aus und spülen Sie die ausgewählte Leberlobule in einem 10 cm Petriteller, gefüllt mit Phosphat-gepufferter Kochsaline (PBS).
HINWEIS: Es wird empfohlen, den gleichen Leberlappen für jedes Tier zu vergleichen, in diesem Fall wurde der Medianlappen verwendet. - Füllen Sie ein beschriftetes Kryomold mit optimaler Schnitttemperatur (OCT) Medium bei Raumtemperatur (RT). Vermeiden Sie OCT-Blasen. Wenn sie erscheinen, schieben Sie sie mit einer Nadel oder Pipettespitze an den Rand der Form.
- Leberlobule in einen gefüllten OCT-Kryomold einbetten und sofort auf Trockeneis legen, um ein schnelles Einfrieren zu gewährleisten. Kryomolden bei -80 °C bis zum Kryosektionen lagern.
2. Kryosektion
- Transportieren Sie Kryomolde auf Trockeneis, um Gewebeabbau zu vermeiden. Vor dem Kryosektionsgleichgewicht im Cryostat auf -20 °C für 20 min eingestellt.
- Auswerfen der Probe durch Druck auf die Basis des Kunststoff-Kryomolds. Verwenden Sie flüssiges OCT auf warme Probenscheibe bei RT, Position in Kryostat und befestigen Sie eine OCT eingebettete Leberprobe. Tragen Sie sanften Druck und warten Sie 3 min auf OCT, um einzufrieren, um sicherzustellen, dass die Probe auf der Scheibe klebt.
HINWEIS: Vermeiden Sie es, die Probe so weit wie möglich mit den Fingern zu behandeln, um dem Gewebeabbau zu entgehen. - Verriegeln Sie die Probe in den Arm des Kryostats und passen Sie die Ausrichtung so an, dass der Rand der Probe parallel zur Kryostatklinge verläuft. In die Probe schneiden, bis das Gewebe erreicht ist.
- Schneiden Sie die Probe mit einer Dicke von 6 m ab. Legen Sie einen beschrifteten polyamidbeschichteten Schlitten für 5 s über die Probe, damit die Probe auf den Schlitten kleben bleibt. Platzieren Sie die Folie bei RT für 3 x 5 min, und fahren Sie dann für beste Ergebnisse direkt zu Abschnitt 3.
HINWEIS: Für die Verarbeitung mehrerer frisch gefrorener Proben wurden reproduzierbare Ergebnisse erzielt, indem Dias vorübergehend in einer Diabox auf Trockeneis gelagert wurden, bis alle Proben verarbeitet wurden. Bei Verwendung dieses Ansatzes lassen Sie alle Folien mit RT ausdemalibrisieren, bevor Sie mit Abschnitt 3 fortfahren. Formalin fixed paraffin embedded (FFPE) Proben können verwendet werden, obwohl die Hintergrundautofluoreszenz durch diese Methode erhöht wird. Um von FFPE-Proben zu gehen, Abschnitt bei 4 m. Montage durch Fangen von Abschnitten aus 40 °C Wasserbad auf Polyamid-behandelten Dias. Wärmegleitet für 1 h bei 60 °C, dann durch serielle RT-Waschungen (5 min) in Coplin-Gläsern, die Xylol (x2), Ethanol 100% (x2), 96% (x2), 70% (x1) und dH2 O(x1) enthalten. Um Antigene freizulegen, platzieren Sie Dias 20 min bei 90 °C in citratpuffer, bevor Sie Dias in PBS bei RT temperieren. Fahren Sie mit Schritt 3.2 fort.
3. Fluoreszenz-Immunlabelling
- Befestigen Sie Gewebeabschnitte in einer Rauchhaube, indem Sie 1 ml 4% Paraformaldehyd (PFA) in PBS für 10 min bei RT auftragen. Dias in ein PBS gefülltes Coplin-Glas übertragen und 3 min mit sanfter Rührung waschen (wiederholen Sie 3x).
HINWEIS: Vermeiden Sie von jetzt an bis zum Ende des Immunstaining-Prozesses das Trocknen der Probe. - Trocknen Sie den Bereich um jeden Gewebeabschnitt und umgeben Sie ihn mit einem hydrophoben Pen. Permeabilisieren Sie mit 0,5% nichtionischem Tensid (d.h. Triton X-100) in PBS für 15 min bei RT. Dann waschen Sie in PBS gefüllt Ecoplin Glas für 3 min mit sanfter Rührung (wiederholen 2x).
- Block mit einer gefilterten Lösung von 1% Rinderserumalbumin (BSA), 5% Pferdeserum, 0,2% nichtionischem Tensid in PBS (für mindestens 1 h bei RT).
- Inkubieren Sie mit dem primären HNF4-Antikörper, der über Nacht bei 4 °C in einer dunklen feuchten Färbekammer im Sperrpuffer verdünnt wird (siehe Tabelle der Materialien für Antikörper und spezifische Verdünnungen).
- Dias in ein PBS gefülltes Coplin-Glas geben und 3 min mit sanfter Rührung waschen (4x wiederholen).
- Inkubieren Sie mit Alexa-488 konjugierten Sekundärantikörpern und Hoechst verdünnt in gefilterten 1% BSA und 0,2% nichtionischem Tensid in PBS für 2 h bei RT in einer dunklen feuchten Färbekammer (siehe Tabelle der Materialien für Antikörper und spezifische Verdünnungen).
- Dias in ein PBS gefülltes Coplin-Glas geben und 3 min mit sanfter Rührung waschen (4x wiederholen). Waschen Sie in ddH2O für 3 min mit sanfter Rührung (wiederholen 2x).
- Montieren Sie Dias, indem Sie zwei Tropfen fluoreszierender Montagemedien auf einen Deckel (24 x 60 mm) legen und Rutschen darüber legen, wodurch Blasen durch sanften Druck vermieden werden. Zur Langzeitlagerung an Kanten mit klarem Nagellack abdichten und bei 4 °C im Dunkeln lagern.
- Bevor Sie fortfahren, überprüfen Sie Dias mit einem herkömmlichen Fluoreszenzmikroskop, um eine gute Fixierung und Immunkennzeichnung zu gewährleisten.
HINWEIS: Siehe Abbildung 2A,B für erwartete Ergebnisse.
4. Fluoreszenz-Bildaufnahme
HINWEIS: Für diesen Schritt ist eine bildende Plattform mit hohem Inhalt (Materialtabelle) erforderlich, die die automatische Fluoreszenzbildaufnahme unterstützt.
- Schalten Sie das Bildgebungssystem ein, und öffnen Sie ein neues Erfassungsprotokoll.
- Wählen Sie das 10x Objektiv, beachten Sie den Bereich des Sichtfeldes (in diesem Fall 0,6 mm2).
- Legen Sie Parameter fest, um Fluoreszenzbilder mit den entsprechenden Anregungs- und Emissionsfiltern (gemäß Schritt 3.6) zu erfassen. Wählen Sie für Hoechst und Alexa-488 die Kanäle "DAPI" und "GFP" mit 390/18 und 438/24 nm Anregung und 432,5/48 bzw. 475/24 nm Emission aus.
- Konzentrieren Sie die Probe und stellen Sie sicher, dass die Signalintensität nicht sättigend ist. Stellen Sie sicher, dass die Bildaufnahme mit der gleichen Belichtungszeit für alle Bilder erfolgt, oder verwenden Sie ein System, bei dem die Fluoreszenzintensität für die Belichtungszeit korrigiert wird.
- Scannen Sie die Probe und erfassen Sie genügend Bilder, um eine vollständige Abdeckung des Gewebeabschnitts zu erhalten (je nach Stichprobengröße ca. 20 bis 50 Sichtfelder).
- Überprüfen Sie die Bilddatenbank, indem Sie (i) schlecht fokussierte Felder, (ii) die Felder an den Rändern jedes Gewebeabschnitts (um verzerrungsorientierte Zelldichteberechnungen zu vermeiden) und (iii) diejenigen, die gefaltete/physisch beschädigte Bereiche des Gewebeabschnitts enthalten, wenn vorhanden, manuell eliminieren.
5. Automatisierte Fluoreszenzbildanalyse
ANMERKUNG: Dieser Schritt erfordert eine geeignete Bildanalysesoftware (Materialtabelle), die in der Lage ist: (1) hoechst-markierte Kerne in Bildern mit 405 nm (nukleare Segmentierung), (2) die Bewertung der mittleren Hoechst-Kernintensität und -morphometrie und (3) die Schwellenanalyse zur Bestimmung des +/- Status der Kernfluoreszenz bei 488 nm (HNF4) zu ermitteln. Zur visuellen Bewertung und Anpassung der Segmentierungs- und Schwellenwertparameter innerhalb des Programms ist eine grundlegende Bedienerschulung/-expertise erforderlich, um sicherzustellen, dass die Kerne und der HNF4-Status optimal abgesperrt sind (Abbildung 2).
- Öffnen Sie in der Bildanalyse-Software die Erfassungsdatei mit Hochst-Bildern (405 nm) und HNF4 (488 nm) aus Schritt 4.5, und erstellen Sie ein neues Analyseprotokoll.
- Definieren Sie Wellenlängen, die für die kernnukleare Segmentierung (Hoechst, 405 nm) und für die Hepatozyten-/NPC-Schwellenanalyse (HNF4, 488 nm) verwendet werden sollen.
- Passen Sie die nuklearen Segmentierungsparameter der Software (z. B. "Mindestkernfläche" und Kernerkennung "Empfindlichkeit") an, um sicherzustellen, dass die Kerne optimal getrennt sind.
HINWEIS: Eine gute Segmentierung von Hepatozyten sollte Vorrang vor der von NPCs haben. Hepatozytenkerne sind charakteristisch abgerundet (interquartiler Größenbereich: 40 x 64 m2). NPC-Kerne, wie die der sinusförmigen Endothelie, sind abgeflacht/elliptisch oder unregelmäßig in der Form und im Allgemeinen kleiner und dichter verpackt als die von Hepatozyten (interquartiler Größenbereich: 30 x 43 m2). Für die Mausleber wurden eine minimale Kernfläche von 23 m2 und eine "Empfindlichkeit" von 65 % verwendet (siehe Abbildung 2C,D für erwartete Ergebnisse). Die Sensitivität bestimmt, wie Pixelcluster basierend auf ihrer Intensität als einzelne Kerne erkannt werden und sollte empirisch für jede vom Benutzer festgelegte Probe getestet werden, bevor die automatisierte Bildanalyse fortgesetzt wird. - Ändern Sie die Schwellenintensität bei 488 nm, um eine optimale Gatingung von Hepatozyten (HNF4+) und nicht parenchymalen Zellen (HNF4-) zu gewährleisten.
HINWEIS: Siehe Abbildung 2C,D für erwartete Ergebnisse. Der Wert der Schwellenintensität ist relativ und hängt von der Färbeeffizienz und Erfassungseinstellungen wie Laserintensität ab. Es sollte daher vom Benutzer standardisiert werden. Verwenden Sie bekannte HNF4-Zellen wie Endothelzellen und Periportal-NPCs als interne Negativkontrolle und binukleare Hepatozytenkerne als positive Referenz für die Färbung. Testen Sie die Analyseparameter mit einer kleinen Anzahl von Bildern, um eine gute kernnukleare Segmentierung und DieIntensitätsschwellentrennung sicherzustellen, bevor Sie Analyseparameter auf den gesamten Datensatz anwenden. - Wählen Sie die folgenden zu quantifizierenden Kernparameter aus: (1) Kernfläche auf der Grundlage der Hoechst-Färbung (m2), (2) mittlerekerne Hoechst-Intensität (RU), (3) kernischer Dehnungsfaktor (mittleres Verhältnis der kurzen Achse des Kerns zur langen Achse des Kerns; wenn ein zentrumssymmetrisches [nicht-lästiges] Objekt den Wert 1, (4) Nuc 1/(Formfaktor) hat, bedeutet der kerntechnische "Rundungsindex", der nach Umkreis 2/(4x x Fläche) berechnet wird. Die Werte reichen von 1 bis unendlich, wobei 1 ein perfekter Kreis ist, (5) HNF4-Status (positiv-1 oder negativ-0) und (6) nukleare x/y-Koordinaten basierend auf "Schwerpunkt" (cg), einer Methode zum Einstellen des Objektmittelpunkts aus Graustufenbildern mit Subpixelgenauigkeit.
- Führen Sie die Analyse für alle Beispiel-Datasets aus, und exportieren Sie numerische Daten aus Schritt 5.5 in Tabellenkalkulationssoftware.
6. Datenanalyse
HINWEIS: Der Datenanalyseschritt kann mit jeder Standard-Tabellenkalkulationssoftware durchgeführt werden.
- Berechnen Sie Hepatozyten- und Nicht-Hepatozyten-Zellzahlen.
- Berechnen Sie die Gesamtfläche des für jede Stichprobe analysierten Leberabschnitts, indem Sie die Anzahl der Ansichtsfelder mit dem Bereich des Ansichtsfeldes multiplizieren (Schritt 4.2).

- Wenn Sie mit Tabellenkalkulationsdateien arbeiten, die für jeden Leberabschnitt generiert werden, filtern Sie die Daten, indem Sie nur HNF4+-Kerne auswählen. Berechnen Sie die Gesamtzahl der analysierten HNF4+-Kerne und dividieren Sie diese durch die Gesamtfläche, die analysiert wurde, um eine mittlere Hepatozytendichte für jede Probe zu erhalten (Abbildung 2F).

- Führen Sie die gleiche Berechnung für nicht-parenchymale Zellen durch, indem Sie die Kalkulationstabelle nach HNF4-Zellen filtern (Abbildung 2E).
- Berechnen Sie die Gesamtfläche des für jede Stichprobe analysierten Leberabschnitts, indem Sie die Anzahl der Ansichtsfelder mit dem Bereich des Ansichtsfeldes multiplizieren (Schritt 4.2).
- Berechnen Sie die Verteilung der nuklearen Größe von Hepatozyten.
- Filtern Sie daten mit Tabellenkalkulationssoftware, um nur HNF4+-Kerne auszuwählen.
- Plotwerte des Kernbereichs in einem Histogramm (Abbildung 2G). Stellen Sie die Behälterbreite auf 5 m2ein.
HINWEIS: Frequenzwerte können für Fläche (Kerne/mm2) gemäß Schritt 6.1.1 korrigiert werden.
- Führen Sie hepatozyte Kernploidie-Analyse.
HINWEIS: Die Tabellendaten aus Schritt 5.6 werden verwendet, um für jede Probe ein kerntechnisches Ploidy-Profil zu generieren. Dieser Prozess wurde automatisiert und kann mit einer benutzerdefinierten geschriebenen Software durchgeführt werden, die frei zum Download mit unterstützenden Informationen und Demonstrations-Datasets unterhttps://github.com/lukeynoon(sieheErgänzende Dateien). Der Quellcode wird für Benutzer bereitgestellt, die die Methodik anpassen möchten. Eine Beschreibung des Algorithmus sowie Installations- und Gebrauchsanweisungen sind unten aufgeführt. Das Programm verwendet Tabellenkalkulationsdaten, um Hepatozytenkerne automatisch in zwei Gruppen zu trennen; (1) solche mit "einfachen" kreisförmigen Kernen und (2) "komplexen" nichtkreisförmigen Kernen, die für binukleare Zellen mit >2c Ploidie repräsentativ sind. Der minimale Kern-DNA-Gehalt (eine Funktion des Kernbereichs und der DNA-Dichte) wird als nächstes für alle "einfachen" Kerne berechnet. Ein nachfolgender Schritt kalibriert dann automatisch die KERNploidie HNF4+ Hepatozyten mit HNF4-Kernen als bekannte 2-4N-interne Kontrolle.- Herunterladen und Installieren von Software.
- Laden Sie die verpackte Anwendung herunter von: https://github.com/lukeynoon
- Starten Sie MATLAB. Navigieren Sie zur REGISTERKARTE APP des Toolstrips, klicken Sie auf App installieren und öffnen Sie die heruntergeladene Anwendung mit dem Begriff"Ploidy_Application.mlappinstall". Es wird eine Meldung angezeigt, um die erfolgreiche Installation zu bestätigen.
HINWEIS: Die Anwendung ist jetzt einsatzbereit und verbleibt in der APP-Registerkarte des Toolstrips.
- Formatieren Sie Eingabedaten.
HINWEIS: Vor der automatisierten Kernploidie-Analyse sollten alle Tabellenkalkulationsdateien mit bildhafter Daten mit hohem Inhalt (Schritt 5.6) gemäß den folgenden Anweisungen gespeichert und formatiert werden.- In jeder exportierten Datendatei (. XLS 97-2004 Arbeitsmappe) aus Schritt 5.6, enthalten ein Blatt mit dem Begriff "Zellmaße" mit allen Daten, die für die ploidy-Analyse erforderlich sind, die in Spalten aufgeführt sind (Abbildung 3A). Stellen Sie sicher, dass das Tabellenkalkulationslayout einschließlich der Spaltenkopfnamen gegenüber dem von Abbildung 3Aunverändert bleibt, da die Analysemethode die richtigen Spaltendaten findet, indem sie nach diesen Namen sucht (siehe Demo-Datasets in Ergänzenden Dateien als Referenz). Wenn z. B. die Bildanalysesoftware mit hohem Inhalt keine Spalte "Lichtfluss" erzeugt (Abbildung 3A), fügen Sie manuell eine Spalte "Lichtfluss" an derselben Stelle ein, d. h. Spalte K, und füllen Sie sie mit Nullen.
- Für jede Versuchsbedingung (z. B. "Verletzte-d14") stellen Sie einen Kontrolldatensatz bereit, der zur Berechnung der internen Kontrolle für die Kernploidienkalibrierung verwendet wird (Schritt 6.3.4.3). Wählen Sie hier Leberproben von unbehandelten erwachsenen Littermaten ("Control-d0"; Abbildung 3B-D).
- Speichern Sie für biologische Replikationen (pro Bedingung) jede Kalkulationstabelle in einem eigenen Ordner (wie in Abbildung 3B). Benennen Sie die Ordnerpräfixe inkrementell, z. B. "Sample1, Sample2, Sample3... SampleN", wie es die darin enthaltenen Dateinamen beziffern. Daher sollte jeder Datensatzordner (z. B. "Control-d0") eine Reihe von Unterordnern ("Sample1", "Sample2", etc.) enthalten, die jeweils eine Tabellenkalkulationsdatei mit demselben entsprechenden Namen enthalten.
- Führen Sie die Anwendung aus.
- Starten Sie in MATLAB die "Ploidy_Application", indem Sie auf das Symbol in der Registerkarte MY APPS des Toolstrips klicken (Abbildung 3C). Die Ploidy_Application grafische Benutzeroberfläche (GUI) wird angezeigt (Abbildung 3C).
- Klicken Sie auf die Schaltfläche Pfad, um Daten zu steuern, um zu dem Ordner zu navigieren, in dem sich die Steuerelementdaten replizieren (z. B. "Control-d0"). Dieser Datenpfad wird dann in der Schnittstelle angezeigt (z. B. /Users/Desktop/Control-d0).
- Geben Sie im "Ordnerpräfix" den Namen ein, der den Ausgabedateien gegeben werden soll (z. B. "Beispiel").
HINWEIS: Dieses Präfix kann in einen beliebigen Text geändert werden, vorausgesetzt, dass die Ordner und Dateinamen inkrementell benannt bleiben. - Klicken Sie auf die Schaltfläche Pfad zu anderen Daten, und navigieren Sie zu dem Ordner, in dem sich die Vergleichsdaten replizieren (z. B. "Verletzte-d14"). Dieser Datenpfad wird dann in der Schnittstelle angezeigt (z. B. /Users/Desktop/Injured-d14).
- Klicken Sie auf Ausführen!. Wenn die Analyse abgeschlossen ist, lautet die Statusleiste "Analysis Complete!..".
ANMERKUNG: Der Antrag meldet für jede Probe die Schichtung der "einfachen" Kerne in 2n, 2n, 4n, 4n 8n und 8n+ in Absoluten Zahlen und als Prozentsatz der Gesamtanzahl(Abbildung 3D). Diese Dateien werden automatisch in jedem Beispielordner gespeichert als: "Count_2n.txt", "Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". Die Ploidy_Application speichert automatisch eine Liste für jede Probe, aller individuellen Ploidy-Schätzungen für "einfache" Hepatozyten- und Nicht-Hepatozytenkerne in "Ploidy_All_Hepatocytes.txt" und "Ploidy_NonHepatocytes.txt". Für das Kontroll-Dataset speichert die Methode auch die minimalen DNA-Gehalt-Schwellenwerte, die für die Schichtung der Ploidie berechnet wurden (siehe Schritt 6.3.4.3.7) in einer Datei mit dem Namen "Normalised_Thresholds_Control". Schließlich erstellt die Anwendung einen Ordner für das Steuerelement und die ausgewählten vergleichenbedingungenDaten mit dem Namen "Zusammenfassung". Dieser Ordner enthält zwei Unterordner, "Ploidy" und "Stratification", die die Durchschnittswerte aller bereitgestellten Stichproben enthalten (Abbildung 3D).
- Beschreibung der Methodik.
HINWEIS: Im folgenden Abschnitt wird die von der Software "Nukleare Ploidie-Analyse" verwendete Methodik ausführlich beschrieben. Wenn der Benutzer sich entscheidet, die Anwendung nicht zu verwenden, können diese Schritte mit Tabellenkalkulationssoftware befolgt werden, um das Kernploidy-Profil manuell zu berechnen.- Trennen Sie Kerne in "einfach" oder "komplex" nach nuklearer Morphometrie.
- Berechnen Sie einen "Zirkularitätsindex" für alle Kerne, definiert als der nukleare "Dehnungsfaktor" dividiert durch den "Nuc 1/(Formfaktor)", wobei ein Wert von 1,0 einen perfekten Kreis angibt.
ANMERKUNG: "Nukleare Dehnung" und "Nuc 1/(Formfaktor)" sind zwei diskrete Kennzahlen der "Zirkularität" eines Objekts, die ergänzende, nicht überlappende morphometrische Kriterien bewerten. Erstere misst die Lang- und Kurzachsen eines Objekts, während letztere die Länge des Umfangs eines Objekts mit der seines Bereichs vergleicht. Um die in diesem Protokoll verwendete Definition der nuklearen Zirkularität zu stärken, wurden diese beiden Messungen zu einem einzigen "Zirkularitätsindex" zusammengefasst. Ein früherer Ansatz zur Schätzung der Kernkraftploidie unter Verwendung der beschriebenen Methode, die nur die nukleare Dehnung verwendethat 17. Während mit diesem Ansatz akzeptable Ergebnisse erzielt wurden, haben die Autoren beobachtet, dass ein zusammengesetzter "Zirkularitätsindex" die Diskriminierung manuell ausgewählter Kerne von mononuklearen und binukleären Hepatozyten verbessert (Daten werden nicht angezeigt). - Klassifizieren Sie Kerne mit einem Zirkularitätsindex von 0,8 als "komplex" und die von > 0,8 als "einfach".
- Berechnen Sie einen "Zirkularitätsindex" für alle Kerne, definiert als der nukleare "Dehnungsfaktor" dividiert durch den "Nuc 1/(Formfaktor)", wobei ein Wert von 1,0 einen perfekten Kreis angibt.
- Schätzen Sie den "minimalen" DNA-Gehalt (m) für alle "einfachen" Kerne.
- Berechnen Sie den Kernradius (r) mit der Formel:

- Berechnen Sie das Kernvolumen (v) mit dem Volumen einer Kugelformel:
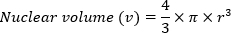
- Generieren Sie einen relativen Wert für minimalen DNA-Gehalt (m) mit der Formel:

- Berechnen Sie den Kernradius (r) mit der Formel:
- Kalibrieren Sie den Datensatz mit den NPC-Kernen (HNF4-) als interne 2-4N-Steuerung.
HINWEIS: NPCs haben je nach Zellzyklusstatus einen DNA-Gehalt von 2 bis 4 N. Daher steigt der Mittelwert des "minimalen" DNA-Gehalts von NPC (NPCm) mit der Verletzung (Abbildung 4A). Der Kalibrierungsfehler wird minimiert, indem eine Obergrenze von NPCm festgelegt wird, die einen Schwellenwert von 4c darstellt (Abbildung 4B).- Wählen Sie in der Kalkulationstabelle nur NPC-Kerne mit Werten für "m" aus, die innerhalb von 1 Standardabweichung (SD) des Modus liegen (dies filtert Rauschen aus einem möglichen Segmentierungsfehler heraus).
- Untersuchen Sie innerhalb dieses gefilterten Bereichs Kerngebiete und deren entsprechende mittlere Hoechst-Intensitäten (Abbildung 4C).
- Schätzen Sie den kleinsten Kernbereich innerhalb dieses gefilterten Bereichs mit maximaler kernkrafter Hoechst-Intensität (d. h. dem Punkt, an dem die Linie der Kurve die Richtung im gefilterten Datensatz ändert, wie durch den roten Kreis in Abbildung 4Cdargestellt). Dieser Wert stellt einen 2N-4N-Übergangszustand (t) dar, über dem die Probenahme von 4c-Kernen über 2c-Kernen überwiegt, was zu einer Maxima der mittleren Hoechst-Intensität führt.
HINWEIS: Dieser Wert wird automatisch von der Software bestimmt; Tabellenkalkulationsbenutzer können diesen Punkt jedoch manuell als Übergangsgröße auswählen. - Berechnen Sie den minimalen DNA-Gehalt, der durch diese Übergangsgröße (tm) dargestellt wird, indem Sie Schritt 6.3.4.2 folgen.
- Um die 4N-Schulter des NPCm-Datasets zu schätzen, fügen Sie dem Wert von tm1 SD hinzu. Die resultierende Zahl (Abbildung 4B) beschreibt die obere Grenze des minimalen DNA-Gehalts von NPC, die für die Kernploidienschichtung verwendet werden soll (S4c).
- Wiederholen Sie die Schritte 6.3.4.3.1 bis 6.3.4.3.5 für alle "Kontroll"-Proben.
HINWEIS: In Abbildung 3werden beispielsweise unverletzte Kontrolllebern ("Control-d0") als Kontrollbedingung verwendet. - Berechnen Sie einen durchschnittlichen 4c-Schichtungsschwellenwert (S4c) für "Kontroll"-Proben und verwenden Sie diese, um die Grenzen 2c (S2c) und 8c (S8c) für minimalen DNA-Gehalt (m) zu extrapolieren. Schichtungsschwellen werden automatisch von der Software generiert und gespeichert (Schritt 6.3.3.3).
HINWEIS: Je nach Studienentwurf können die durchschnittlichen Schichtungsschwellenwerte für jede Bedingung oder für bestimmte Bedingungen (z. B. gesunde Kontrollleber) berechnet werden. Die Software Zur Analyse der Nuklearen Ploidie erfordert jedoch, dass eine von 2 Dateien als "Kontrolle" für die Berechnung relativer Ploidy-Werte bezeichnet wird. - Berechnen Sie einen Ploidy-Wert für alle Kerne unter Verwendung des in Schritt 6.3.4.3.7 erzeugten S2c-Werts gemäß:

- "einfache" Hepatozyten (HNF4+) Kerne nach folgenden Kriterien in 2c/4c/8c/>8c-Klammern stifizieren: "2c" HNF4+ = p 2; "4c" HNF4-+ = 2 < p 4; "8c" HNF4-+ = 4 < p - 8; ">8c" HNF4- + = 8 < p.
- Um die räumliche Strukturierung von Ploidy-Untergruppen zu rekonstruieren, trennen Sie die Kerndaten in jeder Stichprobentabelle nach den entsprechenden Feldern, in denen sie erworben wurden. Verwenden Sie dann die zugehörigen kernnuklearen x/y-Koordinaten (ab Schritt 5.5), um Ploidy-Untergruppen in 2D zu zeichnen (Abbildung 5C).
- Trennen Sie Kerne in "einfach" oder "komplex" nach nuklearer Morphometrie.
- Herunterladen und Installieren von Software.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Diese Methode wurde verwendet, um die Auswirkungen von cholestatischen Verletzungen auf die erwachsene Mausleber zu messen, indem Tiere für 0-21 Tage mit einer hepatotoxischen Diät gefüttert wurden, die 0,1% 3,5-Diethoxycarbonyl-1,4-Dihydrocollidin (DDC)17enthält. Chronische DDC-Fütterung führt zu hepatozellulären Verletzungen erhöhte Ploidie und Periportal Expansion von NPCs. Der Benutzer sollte sich bewusst sein, dass Mausstamm und altersabhängige Unters...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Ein hochgehaltiger, hochdurchsatzhoher Ansatz für die Analyse der Gewebeumgestaltung und Schätzung der Hepatozyten-Atomploidie in der murinen Leber wird beschrieben. Sobald ein Benutzer mit dem Verfahren vertraut ist, kann er mehrere Proben in einem Zeitraum von 3 bis 5 Tagen verarbeiten, abbilden und analysieren, wodurch große testbare Datasets generiert werden, die eine detaillierte Signatur des Leberzustands bereitstellen. Angesichts der Einfachheit der Probenvorbereitungsmethode, zusammen mit der großen Anzahl de...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von der spanischen MINECO Regierung Zuschüsse BFU2014-58686-P (LAN) und SAF-2017-84708-R (DJB) finanziert. LAN wurde unterstützt durch ein nationales MINECO Ramon y Cajal Fellowship RYC-2012-11700 und Plan GenT Award (Comunitat Valenciana, CDEI-05/20-C) und FMN von einem regionalen ValI+D Studentenschaft der Valencianischen Generalitat ACIF/2016/020. Das RP würdigt Prof. Ewa K. Paluch für die Finanzierung. Wir danken Dr. Alicia Martinez-Romero (CIPF Cytometry Service) für die Hilfe bei der IN Cell Analyzer Plattform.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Referenzen
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten